JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
오픈 소스 디지털 이미지 상관관계를 통한 힘줄의 국소 조직 변형 측정
요약
이 논문은 힘줄 외식편 내에서 국소 2D 조직 변형을 측정하기 위한 오픈 소스 디지털 이미지 상관 알고리즘에 대해 설명합니다. 이 기술의 정확성은 여러 기술을 사용하여 검증되었으며 공개적으로 사용할 수 있습니다.
초록
힘줄 세포가 현장에서 경험하는 균주와 이러한 균주가 조직 리모델링에 미치는 영향을 이해하는 데 상당한 과학적 관심이 있습니다. 이러한 관심을 바탕으로, 로딩 동안 힘줄 외식편 내의 국소 조직 변형을 측정하기 위한 몇 가지 분석 기술이 개발되었습니다. 그러나, 몇몇 경우에, 이들 기술들의 정확성 및 민감도는 보고되지 않았으며, 알고리즘들 중 어느 것도 공개적으로 이용가능하지 않다. 이로 인해 힘줄 외식편에서 국소 조직 변형을 보다 광범위하게 측정하는 것이 어려워졌습니다. 따라서 이 논문의 목적은 쉽게 사용할 수 있고 사용하기 쉬운 힘줄 외식편에서 국소 조직 변형을 측정하기 위한 검증된 분석 도구를 만드는 것이었습니다. 특히, 공개적으로 사용 가능한 ALDIC(Augmented-Lagrangian Digital Image Correlation) 알고리즘은 단축 장력 하에서 마우스 아킬레스건 내 세포핵의 변위를 추적하여 2D 변형을 측정하는 데 적용되었습니다. 또한, 계산된 균주의 정확도는 디지털로 변환된 이미지를 분석하고 변형을 독립적인 기술(즉, 광표백 선)에서 결정된 값과 비교하여 검증되었습니다. 마지막으로, 계산된 변위 필드를 사용하여 참조 이미지를 재구성하는 기술이 알고리즘에 통합되었으며, 이는 알려진 변형률 값 또는 2차 측정 기술이 없는 경우 알고리즘의 정확도를 평가하는 데 사용할 수 있습니다. 이 알고리즘은 0.00015의 정확도로 최대 0.1의 변형률을 측정할 수 있습니다. 재구성된 참조 이미지를 실제 참조 이미지와 비교하는 기술은 잘못된 데이터가 있는 샘플을 성공적으로 식별했으며 데이터가 좋은 샘플에서 변위 필드의 약 85%가 정확함을 나타냅니다. 마지막으로, 마우스 아킬레스건에서 측정된 균주는 이전 문헌과 일치했습니다. 따라서 이 알고리즘은 힘줄의 국소 조직 변형을 정확하게 측정하는 데 매우 유용하고 적응력이 뛰어난 도구입니다.
서문
힘줄은 기계적 하중 1,2,3,4에 반응하여 적응하고 퇴화하는 기계에 민감한 조직입니다. 힘줄 세포 생물학에서 기계적 자극이 하는 역할로 인해 로딩 중에 힘줄 세포가 천연 조직 환경에서 경험하는 균주를 이해하는 데 큰 관심이 있습니다. 힘줄의 국소 조직 변형을 측정하기 위해 몇 가지 실험 및 분석 기술이 개발되었습니다. 여기에는 스페클 패턴 또는 광표백 선(PBL)을 사용한 표면 변형의 2D/3D 디지털 이미지 상관관계(DIC) 분석5,6,7,8, 조직 내 개별 핵의 중심 대 중심 거리 변화 측정(9,10), 면외 운동 및 3D 변형을 고려하는 최근의 전체 필드 3D DIC 방법이 포함됩니다 11 . 그러나 이러한 기술의 정확성과 민감도는 소수의 경우에만 보고되었으며 이러한 기술 중 어느 것도 공개적으로 사용할 수 없었기 때문에 이러한 기술의 광범위한 채택 및 활용이 어렵습니다.
이 작업의 목적은 쉽게 사용할 수 있고 사용하기 쉬운 힘줄 외식편에서 국소 조직 변형을 측정하기 위한 검증된 분석 도구를 만드는 것이었습니다. 선택된 방법은 Yang과 Bhattacharya12가 개발한 MATLAB으로 작성된 공개적으로 사용 가능한 ALDIC(Augmented-Lagrangian Digital Image Correlation) 알고리즘을 기반으로 합니다. 이 알고리즘은 힘줄 샘플 분석에 적용되었으며 디지털 변환된 이미지에 적용하고 실제 힘줄 샘플에서 측정된 변형을 광표백 라인에서 얻은 결과와 비교하여 검증했습니다. 또한, 알려진 변형률 값이나 2차 측정 기술이 없는 경우에도 계산된 변위 필드의 정확도를 확인하기 위해 알고리즘에 추가 기능이 구현되었습니다. 따라서 이 알고리즘은 힘줄의 국소 2D 조직 변형을 정확하게 측정하기 위한 매우 유용하고 적응 가능한 도구입니다.
프로토콜
이 연구는 펜실베니아 주립 대학 기관 동물 관리 및 사용 위원회의 승인을 받았습니다.
1. 조직 준비
- 이 프로토콜을 위해 2-4개월 된 수컷 C57BL/6 마우스에서 아킬레스건을 채취합니다.
참고: 생쥐나 다른 작은 동물의 다른 힘줄이나 인대도 사용할 수 있습니다.- 아킬레스건 표면의 피부를 절개하여 족저힘줄과 주변 결합조직을 노출시킵니다. 그런 다음 수술용 칼날을 사용하여 제거합니다.
- 노출된 가자미근과 비복근을 뒷다리에서 분리하고 수술용 칼날로 아킬레스건을 조심스럽게 긁어냅니다
- 회전 도구에 절단 휠 부착물을 사용하여 발의 나머지 부분에서 종골을 분리합니다.
- 실온에서 회전 믹서에서 5-(4,6-디클로로트리아지닐) 아미노플루오레세인(DTAF) 및 0.1M 중탄산나트륨 완충액의 5μg/mL 용액 1.5mL에 조직을 20분 동안 염색합니다. 이 용액은 조직에서 단백질(예: 세포외 기질)을 염색합니다.
참고: 이 20분 동안 1.3단계를 완료해야 합니다. - 인산염 완충 식염수(PBS)에 DRAQ5의 1:1,000 용액을 준비하여 핵을 염색합니다. 와류 믹서를 사용하여 용액을 균질화하십시오.
- 단계 1.2에서 20분의 배양 기간 후, DTAF 용액으로부터 DRAQ5 용액으로 조직을 옮기고, 실온에서 10분 동안 암실에서 배양한다.
2. 힘줄 로딩 및 이미지 획득
참고: 이 프로토콜에는 컨포칼 현미경 위에 장착할 수 있는 인장 장치가 필요합니다. 이 연구를 위해, Peterson과 Szczesny13 에 의해 기술된 미세 인장 장치가 사용되었다.
- 힘줄을 인장 하중 장치의 그립에 놓습니다. 로딩 장치에 그립을 장착하기 전에 디지털 캘리퍼스를 사용하여 종골 부착물과 반대쪽 그립 사이의 거리를 측정하십시오. 이 거리는 힘줄 표점 거리입니다.
- 또는 힘줄을 삽입하기 전에 그립을 로딩 장치에 장착하고 접촉하여 무변위 모터 위치를 정의합니다. 힘줄을 삽입한 후 모터의 변위는 잠재적으로 더 정확한 그립 투 그립 게이지 거리를 제공할 수 있습니다.
- 조직 수화를 유지하기 위해 PBS가 포함된 로딩 장치에 그립을 장착합니다. 알고리즘의 x-스트레인 및 y-스트레인 출력이 힘줄 축과 일치하도록 현미경 이미지의 x축 또는 y축에 힘줄을 최대한 정렬합니다.
참고: 이 연구에서 힘줄은 x축과 정렬되었습니다. 힘줄을 이미지 축과 완벽하게 정렬할 수 없는 경우 표준 변형 변환 방정식14를 사용하여 알고리즘의 x-스트레인 및 y-스트레인 출력을 힘줄의 세로/수직 축과 정렬하도록 변환할 수 있습니다. - 힘줄에 1g의 장력을 가하고, 원하는 경우 순환 하중을 가하여 시료를 사전 컨디셔닝합니다. 이 프로토콜에서는 연구 목적이 조직 재료 특성을 측정하는 것이 아니라 측정된 국소 조직 변형을 검증하는 것이었기 때문에 사전 조건을 사용하지 않았습니다. 로딩 이력에 따라 달라지는 거시적 재료 특성을 측정하는 데 관심이 있는 경우 사전 조정을 권장합니다. 프리컨디셔닝 및 복구 후 1g 프리로드를 다시 적용합니다.
- 원하는 경우 조직의 중앙 영역에서 80μm 간격으로 4개의 선 세트를 광표백합니다(자세한 내용은 Peterson 및 Szczesny13 참조).
알림: 광표백된 선은 ALDIC 알고리즘의 측정을 검증하는 데 사용되었으며 ALDIC 자체를 수행하는 데 필요하지 않습니다. 선의 수와 간격을 조정할 수 있으며 선의 선명도를 감소시키는 샘플의 아티팩트를 방지하기 위해 선의 위치를 선택해야 합니다. - 그립 근처 조직의 왼쪽과 오른쪽 끝에서 광표백 절차를 반복합니다.
- 컨포칼 현미경을 사용하여 1g의 예압에서 DTAF 및 DRAQ5 형광의 체적 이미지(x,y: 1.25μm/픽셀, z: 2.5μm/픽셀)를 획득합니다.
- 0.5%/s에서 2% 변형률로 스트레인 램프를 수행합니다. 변형률 속도와 증분 변형률 크기를 조정할 수 있습니다.
- 조직이 스트레스를 받도록 10분 동안 긴장을 푸십시오.
참고: 응력 완화 기간은 s가 amp이미지 획득 중에 대략 준정적 하중을 받습니다. 응력 완화 지속 시간이 허용 가능한지 확인하려면 응력 완화의 마지막 순간 동안 힘-시간 곡선의 기울기를 결정하고(보충 그림 1) 이 기울기에 총 이미징 지속 시간을 곱합니다. 이 연구에서 가장 큰 변형률 증가에 가해지는 힘은 5% 이상 변하지 않았습니다. - 변형 후 조직의 또 다른 체적 이미지를 찍습니다.
- 원하는 최종 변형률에 도달할 때까지 2.7-2.9단계를 반복합니다. 본 논문에서는 최종 변형률 값 12%를 선택하였다.
3. 이미지 처리
- ImageJ 또는 Fiji를 사용하여 DRAQ5(핵) 채널의 각 체적 이미지에 대한 최대 z-프로젝션을 만듭니다. 이것은 ALDIC의 2D 얼룩덜룩 한 이미지로 사용됩니다.
- max-intensity z-투영을 .tiff 파일로 저장하고 다음 명명 규칙에 따라 이름을 지정합니다.
- 이미지 이름의 첫 글자로 숫자를 사용합니다.
- 숫자가 변형률 분석 중에 이미지가 고려되는 순서와 일치하도록 합니다. 예를 들어 첫 번째 이미지는 1로 시작하고 두 번째 이미지는 2로 시작해야 합니다. 다른 숫자를 선택할 수 있지만 순차적으로 증가해야 합니다. 명명 규칙의 예는 "0_Experiment1_MaxZProjection"입니다.
- 이름이 바뀐 모든 max-intensity z-투영을 폴더에 저장합니다.
4. Photo표백 라인 분석 코드 설치 및 적용
알림: 이 단계는 광표백선을 사용하여 ALDIC 알고리즘의 정확성을 확인하려는 경우에만 필요합니다. 이 코드는 광표백 라인 세트 내에서 각 광표백 라인 사이의 평균 정규화된 거리 변화로 국소 조직 변형을 계산합니다. 이 연구에서, 평균 국소 값은 모든 광표백 라인 세트 (즉, 중앙 및 왼쪽 / 오른쪽 끝)에 걸쳐 평균을 구하여 각 샘플에 대한 단일 평균 국소 조직 변형 값을 결정했습니다. 그런 다음 이 값을 사용하여 ALDIC 알고리즘의 정확도를 추정했습니다.
- GitHub(https://github.com/Szczesnytendon/TendonStrainCalc)에서 "PBL Code" 폴더를 다운로드하고 모든 내용을 MATLAB의 작업 디렉터리로 이동합니다.
- "Micro_Mech_Template.m" MATLAB 스크립트를 엽니다.
- Run(실행)을 누르고 볼륨 이미지가 포함된 이미지 파일 중 하나를 선택합니다. 체적 이미지는 .lsm, .tiff, .nd2 파일 형식 중 하나일 수 있습니다.
- 소프트웨어는 폴더의 모든 이미지를 자동으로 로드하고 참조 체적 이미지의 투영된 이미지를 표시합니다. 메시지가 표시되면 왼쪽 클릭하여 샘플의 왼쪽 끝과 오른쪽 끝을 추적하는 다점 선을 만듭니다. 마우스 오른쪽 버튼을 클릭하여 줄을 종료합니다. 입력이 처리된 후 가장자리가 올바르면 확인을 눌러 결과를 수락합니다.
- 메시지가 표시되면 샘플을 가로질러 임의의 대각선을 기준선으로 그립니다.
- 생성된 광표백 선의 수를 입력하고 다점 선으로 광표백 선을 추적합니다.
- 결과가 수용 가능하면 수락하십시오. 결과가 잘못된 경우 조정하고 다시 처리하십시오.
- 모든 이미지에 대해 4.2단계를 반복하고 추적된 선의 모든 이미지를 단일 폴더로 이동합니다.
- 스크립트 "Micro_Mech_Strain.m"을 엽니다.
- Run을 눌러 코드를 실행하고 포토 표백 된 선이 추적되는 저장된 이미지 중 하나를 선택합니다.
- Ok(확인)를 눌러 이미지를 선택한 후 선택한 이미지가 올바른지 확인합니다.
5. 디지털로 변환된 이미지 만들기
참고: 이 단계는 디지털 변환된 이미지를 사용하여 ALDIC 알고리즘의 정확성을 확인하려는 경우에만 필요합니다. 이러한 이미지는 참조 이미지를 인위적으로 변환하여 알려진 크기의 균질한 2D 변형장을 시뮬레이션합니다.
- GitHub(https://github.com/Szczesnytendon/TendonStrainCalc)에서 코드 "Digital_strain.m"을 다운로드합니다.
- 코드를 열고 실행합니다.
- 프롬프트가 표시되면 최대 적용 변형률, 적용된 변형률 증가 및 푸아송 비율에 대해 원하는 값을 삽입합니다. 확인을 누릅니다.
참고: 이 실험의 경우 최대 적용 변형률은 0.1(10%), 적용된 변형률 증가는 0.02(2%), 푸아송 비율 1이 사용되었으며 이는 힘줄 인장 시험15,16의 실험 데이터와 일치합니다. 이 코드는 내장된 MATLAB 함수 imwarp와 입력값(예: 변형률 증분, 푸아송 비율)을 사용하여 디지털로 변환된 이미지를 만듭니다. - 프롬프트가 표시되면 변형되지 않은 참조 이미지를 선택합니다.
- 각 변형률 증가에 대해 참조 이미지와 변환된 이미지의 오버레이가 표시됩니다. 변환된 이미지는 "DigitallyTransformedX%Strain"이라는 제목으로 디렉토리에 저장되며, 여기서 X는 변형률 증분입니다.
6. 변형률 계산 및 검증 코드 설치 및 적용
- GitHub(https://github.com/Szczesnytendon/TendonStrainCalc)에서 "Strain Calculation and Validation Code" 폴더를 다운로드하고 모든 내용을 MATLAB 작업 디렉터리로 옮깁니다
- Yang 및 Bhattacharya12에 따라 mex C/C++ 컴파일러를 설치합니다. 단계는 아래에 요약되어 있습니다.
- MATLAB 명령 창에 "mex -setup"을 입력하고 Enter 키를 눌러 MATLAB에서 mex C/C++ 컴파일러가 설치되었는지 확인합니다.
- 컴파일러가 지원되지 않거나 존재하지 않음을 나타내는 오류가 나타나면 6.3단계와 6.4단계로 진행합니다.
- 오류가 없으면 6.5단계로 진행합니다
- mex C/C++ 컴파일러를 다운로드하려면 "https:/tdm-gcc.tdragon.net/"로 이동하여 TDM-gcc 컴파일러를 선택하십시오.
- 다운로드한 컴파일러를 알려진 위치에 설치합니다.
- MATLAB 명령 창으로 돌아가서 " setenv("MW_MINGW64_LOC","[여기에 설치 경로 입력]")"을 입력합니다. 예를 들어 "setenv("MW_MINGW64_LOC","C:\TDM-GCC-64")"일 수 있습니다. 이 명령이 성공적으로 실행되면 mex 컴파일러가 제대로 설치된 것입니다.
- "main_aldic.m" 함수 스크립트를 입력하고 6.5단계에서 실행된 명령과 일치하도록 22행을 변경합니다.
- 스크립트 "Strain_calc_and_validate.m"을 엽니다.
- Run(실행)을 눌러 이미지 분석을 시작합니다.
- 메시지가 표시되면 ALDIC 매개변수의 값을 원하는 대로 변경합니다.
참고: 창 크기는 0.25-1 곱하기 하위 집합 크기여야 합니다. 매개변수 선택에 대한 자세한 내용은 온라인 사용 설명서를 참조하십시오(https://www.researchgate.net/publication/344796296_Augmented_Lagrangian_Digital
_Image_Correlation_AL-DIC_Code_Manual).- 이 연구에서는 다음 값이 사용되었습니다.
하위 집합 크기(픽셀): 20
창 크기(픽셀): 10
ALDIC를 푸는 방법 : 유한 차이 (1)
병렬 컴퓨팅이 사용되지 않음 (1)
초기 추측을 계산하는 방법: 이미지 피라미드를 기반으로 하는 멀티그리드 검색 (0)
- 이 연구에서는 다음 값이 사용되었습니다.
- 프롬프트가 표시되면 "예" 확인란을 선택하여 알고리즘이 원하는 변수 모음(예: x-변형률, y-변형률, 전단 변형률, 불량 영역 등)에 대한 평균값, 표준 편차 및 2D 맵을 자동으로 저장하도록 합니다. 저장할 변수를 선택하고 확인을 누릅니다.
- 메시지가 표시되면 원하는 대로 매개 변수를 변경합니다.
- 이 실험에는 다음 값이 사용되었습니다.
변형률 계산을 위한 주변점(numP): 12
불량 영역 식별에 대한 상관 계수(corr_threshold): 0.5
불량 영역 분석을 위한 하위 영역 크기(픽셀)(Subsize): 32
- 이 실험에는 다음 값이 사용되었습니다.
- 프롬프트가 표시되면 이름이 바뀐 max-intensity z-프로젝션이 포함된 폴더를 선택합니다. 소프트웨어는 변형된 이미지의 변형률 필드를 결정하기 위해 증분 ALDIC를 자동으로 수행합니다. 즉, 각각의 변형된 이미지는 다음 변형된 이미지에 대한 새로운 "참조" 이미지로서 기능한다. 이는 누적 ALDIC를 수행하는 것과 비교하여 결과의 정확도를 향상시키며(보충 그림 2), 여기서 각 변형된 이미지는 원본(0% 변형률) 참조 이미지와 다시 비교됩니다. 누적 분석을 수행하려면 이미지를 로드하되 원본 참조 이미지와 관심 있는 변형된 이미지만 선택합니다.
참고: 정상 변형률은 λ - 1로 계산되며, 여기서 λ는 조직 스트레치입니다. 조직 스트레치는 에 따라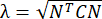 계산되며, 여기서 n = [1 0]T 또는 [0 1]T는 각각 x 방향 및 y 방향에 대해 C = F T F이며, 여기서 F는 ALDIC 알고리즘에 의해 출력된 각 데이터 포인트를 둘러싸는 "numP" 포인트를 사용하여 계산된 변형 기울기입니다. 전단 변형률은 , 여기서
계산되며, 여기서 n = [1 0]T 또는 [0 1]T는 각각 x 방향 및 y 방향에 대해 C = F T F이며, 여기서 F는 ALDIC 알고리즘에 의해 출력된 각 데이터 포인트를 둘러싸는 "numP" 포인트를 사용하여 계산된 변형 기울기입니다. 전단 변형률은 , 여기서 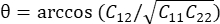 로 계산됩니다
로 계산됩니다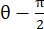 .
. - 메시지가 표시되면 왼쪽 클릭하여 변형을 측정하기 위한 관심 영역을 정의하는 4점 다각형을 만듭니다. 왼쪽 위 모서리에 있는 점부터 시작하여 시계 방향으로 후속 점을 할당합니다.
참고: MATLAB 작업 공간에 저장된 변수 "Storage"에는 평균 x-변형률, x-변형률 표준 편차, 평균 y-변형률, y-변형률 표준 편차, 평균 전단 변형률, 전단 변형률 표준 편차 및 불량 영역의 백분율에 대한 모든 값이 포함되어 있습니다. 불량 영역은 6.13단계에서 선택된 관심 영역 내의 상관 계수 분석에 따라 정의됩니다. "NuclearTrackingResults" 폴더(555 및 556 행을 조정하여 이름을 바꿀 수 있음)는 6.10단계에서 지정한 모든 플롯을 저장합니다. 이 폴더에는 6.10단계에서 지정한 모든 평균과 표준 편차를 저장하는 "Results"라는 스프레드시트 파일도 포함되어 있습니다.
결과
실제 조직 샘플의 변형 필드를 분석하기 전에 ALDIC 프로토콜은 먼저 마우스 아킬레스건 내 핵의 디지털 변형/변환 이미지를 사용하여 검증되었습니다. 구체적으로, 이미지는 시뮬레이션된 푸아송 비율 1 15,16으로 2%, 4%, 6%, 8% 및10% 변형률의 x 방향으로 균일한 변형을 디지털 방식으로 생성하도록 변환되었습니다. 그런 다음 ALDIC 알고리즘의 정확도는 평균...
토론
이 논문의 목적은 인장 하중 하에서 힘줄의 2D 변형률을 측정하기 위한 오픈 소스의 검증된 방법을 제공하는 것이었습니다. 소프트웨어의 기초는 공개적으로 이용 가능한 ALDIC 알고리즘12을 기반으로합니다. 이 알고리즘은 증분(누적) 변형률 분석의 기능이 추가된 더 큰 MATLAB 코드에 포함되었습니다. 이 적응 된 알고리즘은 힘줄의 인장 시험에 적용되었으며, 그 정확도는 두 가지 ...
공개
모든 저자는 공개할 이해 상충이 없습니다.
감사의 말
이 연구는 국립 보건원 (R21 AR079095)과 국립 과학 재단 (2142627)의 지원을 받았습니다.
자료
| Name | Company | Catalog Number | Comments |
| 5-DTAF (5-(4,6-Dichlorotriazinyl) Aminofluorescein), single isomer | ThermoFisher | D16 | |
| Calipers | Mitutoyo | 500-196-30 | |
| Confocal Microscope | Nikon | A1R HD | |
| Corning LSE Vortex Mixer | Coning | 6775 | |
| DRAQ5 Fluorescent Probe Solution (5 mM) | ThermoFisher | 62554 | |
| MATLAB | MathWorks | R2022b | |
| Tensile Loading Device | N/A | N/A | Tensile loading device described in Peterson et al, 2020. (ref 13) |
| Tube Revolver Rotator | ThermoFisher | 88881001 |
참고문헌
- Devkota, A. C. Distributing a fixed amount of cyclic loading to tendon explants over longer periods induces greater cellular and mechanical responses. Journal of Orthopaedic Research. 11 (4), 1609-1612 (2007).
- Sun, H. B., et al. Cycle-dependent matrix remodeling gene expression response in fatigue-loaded rat patellar tendons. Journal of Orthopaedic Research. 28 (10), 1380-1386 (2010).
- Shepherd, J. H., Screen, H. R. C. Fatigue loading of tendon. International Journal of Experimental Pathology. 94 (4), 260-270 (2013).
- Paschall, L., Pedaprolu, K., Carrozzi, S., Dhawan, A., Szczesny, S. Mechanical stimulation as both the cause and the cure of tendon and ligament injuries. Regenerative Rehabilitation: From Basic Science to the Clinic. , 359-386 (2022).
- Andarawis-Puri, N., Ricchetti, E. T., Soslowsky, L. J. Rotator cuff tendon strain correlates with tear propagation. Journal of Biomechanics. 42 (2), 158-163 (2009).
- Cheng, V. W. T., Screen, H. R. C. The micro-structural strain response of tendon. Journal of Materials Science. 42 (21), 8957-8965 (2007).
- Luyckx, T., et al. Digital image correlation as a tool for three-dimensional strain analysis in human tendon tissue. Journal of Experimental Orthopaedics. 1 (1), 7 (2014).
- Duncan, N. A., Bruehlmann, S. B., Hunter, C. J., Shao, X., Kelly, E. J. In situ cell-matrix mechanics in tendon fascicles and seeded collagen gels: Implications for the multiscale design of biomaterials. Computer Methods in Biomechanics and Biomedical Engineering. 17 (1), 39-47 (2014).
- Arnoczky, S. P., Lavagnino, M., Whallon, J. H., Hoonjan, A. In situ cell nucleus deformation in tendons under tensile load; A morphological analysis using confocal laser microscopy. Journal of Orthopaedic Research. 20 (1), 29-35 (2002).
- Screen, H. R. C., Bader, D. L., Lee, D. A., Shelton, J. C. Local strain measurement within tendon. Strain. 40 (4), 157-163 (2004).
- Fung, A. K., Paredes, J. J., Andarawis-Puri, N. Novel image analysis methods for quantification of in situ 3-D tendon cell and matrix strain. Journal of Biomechanics. 67, 184-189 (2018).
- Yang, J., Bhattacharya, K. Augmented Lagrangian digital image correlation. Experimental Mechanics. 59 (2), 187-205 (2019).
- Peterson, B. E., Szczesny, S. E. Dependence of tendon multiscale mechanics on sample gauge length is consistent with discontinuous collagen fibrils. Acta Biomaterialia. 117, 302-309 (2020).
- Humphrey, J. D., O'Rourke, S. L. . An Introduction to Biomechanics. , (2015).
- Reese, S. P., Weiss, J. A. Tendon fascicles exhibit a linear correlation between Poisson's ratio and force during uniaxial stress relaxation. Journal of Biomechanical Engineering. 135 (3), 34501 (2013).
- Ahmadzadeh, H., Freedman, B. R., Connizzo, B. K., Soslowsky, L. J., Shenoy, V. B. Micromechanical poroelastic finite element and shear-lag models of tendon predict large strain dependent Poisson's ratios and fluid expulsion under tensile loading. Acta Biomaterialia. 22, 83-91 (2015).
- Szczesny, S. E., Elliott, D. M. Interfibrillar shear stress is the loading mechanism of collagen fibrils in tendon. Acta Biomaterialia. 10 (6), 2582-2590 (2014).
- Han, W. M., et al. Macro- to microscale strain transfer in fibrous tissues is heterogeneous and tissue-specific. Biophysical Journal. 105 (3), 807-817 (2013).
- Pedaprolu, K., Szczesny, S. E. A novel, open-source, low-cost bioreactor for load-controlled cyclic loading of tendon explants. Journal of Biomechanical Engineering. 144 (8), 084505 (2022).
- Gatt, R., et al. Negative Poisson's ratios in tendons: An unexpected mechanical response. Acta Biomaterialia. 24, 201-208 (2015).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유