Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Ultrastrukturelle Expansionsmikroskopie in drei in vitro Lebenszyklusstadien von Trypanosoma cruzi
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Diese Studie zeigt ein detailliertes Protokoll zur Durchführung der Ultrastrukturexpansionsmikroskopie in drei in vitro Lebenszyklusstadien von Trypanosoma cruzi, dem Erreger, der für die Chagas-Krankheit verantwortlich ist. Wir schließen die optimierte Technik für Zytoskelettproteine und die Pan-Proteom-Markierung ein.
Zusammenfassung
Wir beschreiben hier die Anwendung der Ultrastrukturexpansionsmikroskopie (U-ExM) bei Trypanosoma cruzi, einer Technik, die es ermöglicht, die räumliche Auflösung einer Zelle oder eines Gewebes für die mikroskopische Bildgebung zu erhöhen. Dies geschieht durch die physikalische Erweiterung einer Probe mit handelsüblichen Chemikalien und gängigen Laborgeräten.
Die Chagas-Krankheit ist ein weit verbreitetes und drängendes Problem der öffentlichen Gesundheit, das durch T. cruzi verursacht wird. Die Krankheit ist in Lateinamerika weit verbreitet und hat sich in nicht-endemischen Regionen aufgrund der zunehmenden Migration zu einem erheblichen Problem entwickelt. Die Übertragung von T. cruzi erfolgt durch hämatophage Insektenvektoren, die zu den Familien Reduviidae und Hemiptera gehören. Nach der Infektion vermehren sich T. cruzi amastigoten im Säugetierwirt und differenzieren sich zu Trypomastigoten, der nicht-replikativen Blutbahnform. Im Insektenvektor verwandeln sich Trypomastigoten in Epimastigoten und vermehren sich durch binäre Spaltung. Die Unterscheidung zwischen den Lebenszyklusstadien erfordert eine umfangreiche Neuanordnung des Zytoskeletts und kann im Labor mit verschiedenen Zellkulturtechniken vollständig nachgebildet werden.
Wir beschreiben hier ein detailliertes Protokoll für die Anwendung von U-ExM in drei in vitro Lebenszyklusstadien von Trypanosoma cruzi, wobei der Schwerpunkt auf der Optimierung der Immunlokalisation von Zytoskelettproteinen liegt. Wir haben auch die Verwendung von N-Hydroxysuccinimidester (NHS) optimiert, einer Pan-Proteom-Markierung, die es uns ermöglicht hat, verschiedene Parasitenstrukturen zu markieren.
Einleitung
Die Expansionsmikroskopie (ExM) wurde 2015 von Boyden et al.1 erstmals beschrieben. Es handelt sich um ein bildgebendes Protokoll, mit dem ein herkömmliches Mikroskop eine räumliche Auflösung unterhalb der Beugungsgrenze erreichen kann. Diese höhere Auflösung wird durch eine physikalische Vergrößerung der Probe erreicht. Dazu werden fluoreszenzmarkierte Moleküle mit einem Hydrogel vernetzt, das anschließend isotrop mit Wasser expandiert wird. Durch diese Expansion werden die Signale in allen drei Dimensionen nahezu isotrop getrennt. Diese Methode verwendet kostengünstige Chemikalien und ermöglicht eine räumliche Auflösung von ca. 65 nm mit herkömmlichen (konfokalen) Mikroskopen, was etwa viermal besser ist als die Standardauflösung eines konfokalen Mikroskops (ca. 250 nm)1.
Der nächste Meilenstein, der den Einsatz der Expansionsmikroskopie in vielen biologischen Bereichen ermöglicht hat, war die Adaption der Immunfluoreszenzmarkierung mit herkömmlichen Antikörpern2. Eine weitere Adaption des ursprünglich veröffentlichten ExM-Protokolls ist die vergrößerte Analyse des Proteoms (MAP)3. Bei dieser Methode wurden hohe Konzentrationen von Acrylamid und Paraformaldehyd vor dem Eintauchen in das Hydrogel der Probe verwendet, um eine Intra- und Interproteinvernetzung zu verhindern, was zu einer besseren Erhaltung des Proteingehalts und der subzellulären Architektur der Proben führte. Dieses alternative Protokoll wurde optimiert, um eine verbesserte Konservierung der Gesamtultrastruktur isolierter Organellen durch die Verwendung niedrigerer Konzentrationen der Fixiermittel (Formaldehyd/Paraformaldehyd und Acrylamid) zu erreichen; Dieser Ansatz wurde als Ultrastrukturexpansionsmikroskopie (U-ExM)4 bezeichnet.
Um noch mehr Auflösung zu erreichen, wurde auch über die Kombination von ExM mit superauflösenden Mikroskopietechniken, einschließlich der stimulierten Emissionsverarmungsmikroskopie oder der Einzelmolekül-Lokalisationsmikroskopie, berichtet, um Auflösungen unter 20 nmzu erreichen 5.
Die Verwendung von ExM wurde in den Bereichen der Neurowissenschaften und der Zytoskelettforschungausführlich berichtet 6, aber es wurden nur wenige Studien an parasitären Protisten durchgeführt. Unser Labor war das erste, das über die Anwendung von U-ExM bei T. cruzi7 berichtete. Das Grundlagenprotokoll basiert hauptsächlich auf den vorangegangenen U-ExM-Berichten zu Toxoplasma gondii, Plasmodium ssp. und Trypanosoma brucei 8,9,10,11.
Einer der größten Vorteile von ExM ist sein modularer Charakter, der eine große Flexibilität bei der Anpassung an verschiedene biologische Proben ermöglicht. Das Protokoll kann in Schritte unterteilt werden (z. B. Fixierung, Vernetzungsverhinderung oder Gelierung), die vom Benutzer leicht an seine experimentellen Anforderungen angepasst werden können. Darüber hinaus kann diese Pipeline modifiziert werden, um die Kompatibilität mit dem Modellorganismus zu verbessern oder eine bestimmte Auflösung zu erreichen. Infolgedessen bietet ExM ein enormes Potenzial sowohl für fortschrittliche als auch für nicht-fortschrittliche optische Systeme und gewährleistet so breitere Anwendungen in der Zukunft.
Die Chagas-Krankheit, auch Amerikanische Trypanosomiasis genannt, ist eine endemische Krankheit in Lateinamerika, die durch Trypanosoma cruzi, einen Protozoen-Parasiten, verursacht wird. Der Lebenszyklus des Parasiten ist komplex und umfasst zwei Entwicklungsstadien bei Säugetieren und zwei beim Insektenwirt (Mitglieder der Familie der Triatominidae), dem biologischen Überträger dieser Krankheit. Die Chagas-Krankheit gehört zu der Gruppe der vernachlässigten Tropenkrankheiten, die von der Weltgesundheitsorganisation gelistet werden, und stellt ein bedeutendes wirtschaftliches und soziales Problem in Lateinamerika dar. Epidemiologische Studien schätzen, dass weltweit 8 Millionen Menschen mit der Chagas-Krankheit leben und über 10.000 Menschen pro Jahr sterben. Diese Zahlen verdeutlichen die Bedeutung der Chagas-Krankheit als Problem der öffentlichen Gesundheit weltweit. Die geografische Verteilung der Chagas-Krankheit hat sich in den letzten Jahrzehnten verändert, wobei viele infizierte Personen aufgrund der zunehmenden Migration heute weltweit in großen städtischen Gebieten leben, im Gegensatz zu den hauptsächlich ländlichen Gebieten Lateinamerikas, wo sie ursprünglich gefunden wurde12.
Die Entwicklungsstadien von T. cruzi unterscheiden sich im Laufe seines Lebenszyklus, was in vitro vollständig repliziert werden kann. Epimastigoten sind replikative Formen im Insektenvektor und haben einen kugelförmigen Kern in der zentralen Region des Zellkörpers und einen balkenförmigen Kinetoplast (eine mitochondriale DNA-enthaltende Struktur, die nur bei Kinetoplastiden vorkommt) in der vorderen Region relativ zum Zellkern mit einem freien Flagellum. Trypomastigoten sind die infektiöse, nicht-replikative Form und haben einen länglichen Kern, einen abgerundeten hinteren Kinetoplasten und ein Flagellum, das über die gesamte Länge des Parasiten an der Plasmamembran befestigt ist. Amastigoten sind die intrazelluläre replikative Form; Sie haben einen Zellkern in der zentralen Region, einen stäbchenförmigen Kinetoplasten im vorderen Teil des Zellkörpers und ein reduziertes Flagellum. Die Anpassungsfähigkeit des Parasiten an unterschiedliche Umgebungen ist ein Spiegelbild dieser morphologischen Variationen. Erwähnenswert ist auch, dass dieser Lebenszyklus eine symmetrische Teilung und unterschiedliche Übergangsentwicklungsstadien beinhaltet13. Bei der Differenzierung spielt das Zytoskelett der Trypanosomatiden eine entscheidende Rolle. Diese Struktur besteht aus einem Korsett aus subpellikulären Mikrotubuli, die in einer geordneten Anordnung stabiler Mikrotubuli unterhalb der Plasmamembran angeordnet sind. Außerdem ist bei diesen Organismen ein paraflagellärer Stab vorhanden, bei dem es sich um eine gitterartige Struktur handelt, die parallel verläuft und an das flagelläre Axonem14 gebunden ist. Die präzise Organisation des Zytoskeletts und die strukturellen Veränderungen des Kerns entlang der Zellzyklusstadien beinhalten einzigartige Genregulationsmechanismen, die für Trypanosomatiden spezifisch sind, was sie zu interessanten Modellen für zellbiologische Studien macht.
Angesichts der geringen Größe von T. cruzi und anderen protozoischen Parasiten stellt U-ExM ein hervorragendes Werkzeug dar, um die strukturellen Merkmale dieser wichtigen Krankheitserreger zu analysieren. Wie bereits erwähnt, wurde die Anwendbarkeit dieser Technik auf T. cruzi zum ersten Mal von Dr. Alonsovalidiert 7. Dieser Bericht beschreibt ein vollständiges U-ExM-Protokoll, wobei der Schwerpunkt auf der Immunlokalisierung von Zytoskelettproteinen während der verschiedenen Lebenszyklusstadien von T. cruzi liegt. Außerdem haben wir die Verwendung von N-Hydroxysuccinimidester (NHS) optimiert, einer Pan-Proteom-Markierung, die es uns ermöglicht, verschiedene Parasitenstrukturen zu markieren. Darüber hinaus wird eine in vitro Methodik beschrieben, um die drei Stadien des Parasiten zu erhalten.
Protokoll
HINWEIS: Abbildung 1 veranschaulicht das vollständige Versuchsdesign.
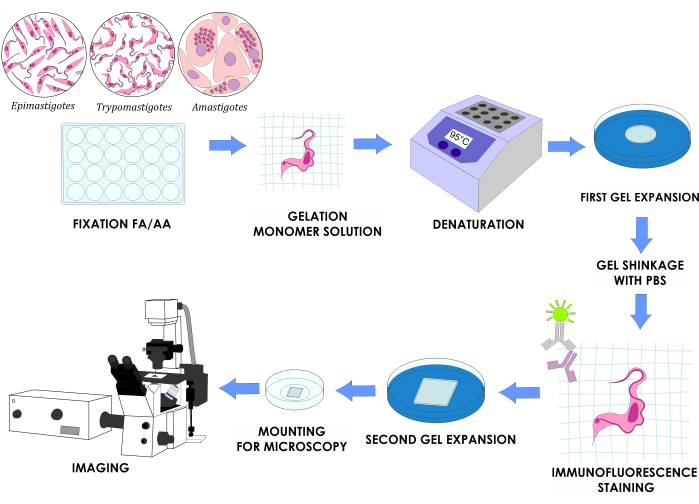
Abbildung 1: U-ExM-Workflow für drei in vitro Lebenszyklusstadien von T. cruzi. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
1. Vorbereitung der Poly-D-Lysin-beschichteten Deckgläser
- Legen Sie ein 10 cm x 10 cm großes Quadrat Siegelfolie in eine Petrischale. Waschen Sie die Deckgläser, indem Sie sie in einer 35 mm Glas-Petrischale in absolutem Ethanol baden.
- Entfernen Sie die Deckgläser mit einer Pinzette aus dem Ethanolbad und lassen Sie die überschüssige Flüssigkeit mit Seidenpapier abtropfen. Legen Sie die Deckgläser über die Siegelfolie.
- Absorbieren Sie den Rest des Ethanols mit mikroskopischem Papier. Geben Sie eine 0,1%ige v/v-Lösung von Poly-D-Lysin auf die Mitte des Deckglases und verteilen Sie es mit der Spitze, so dass es etwa 80% seiner Oberfläche bedeckt. Die Petrischale verschließen und 1 h bei 37 °C inkubieren.
HINWEIS: Verwenden Sie für 22 mm2 Deckgläser 200 μl Poly-D-Lysin-Lösung; Für 12 mm runde Deckgläser 100 μl Poly-D-Lysin-Lösung verwenden. - Waschen Sie die Deckgläser dreimal mit Reinstwasser. Verwenden Sie eine Absaugung mit einem Vakuum, um das Wasser zwischen jedem Waschen zu entfernen. Bis zu 1 Woche bei 4 °C aufbewahren.
2. Vorbereitung der Lösung
- Bereiten Sie Stammlösungen mit 38 % (w/w) Natriumacrylat (SA) vor.
- Geben Sie langsam 19 g SA unter Rühren in 31 ml nukleasefreies Wasser.
HINWEIS: Diese Lösung ist sehr viskos; Seien Sie vorsichtig beim Pipettieren. - Sobald sich das SA vollständig aufgelöst hat, lagern Sie es in einem sterilen Behälter. Halten Sie es bei 4 °C und wechseln Sie es alle 6 Monate.
HINWEIS: SA zeigt je nach Marke manchmal Anzeichen einer Kontamination mit Natriumpolyacrylat, was sich bei der Herstellung der Stammlösung bemerkbar macht, da sie gelblich und trüb wird. Verwenden Sie es vorsichtig, wenn dies der Fall ist.
- Geben Sie langsam 19 g SA unter Rühren in 31 ml nukleasefreies Wasser.
- Bereiten Sie eine Stammlösung aus 40 % (w/v) Acrylamid (AA) und eine Stammlösung aus 2 % (w/v) N,N'-Methylenbisacrylamid (BIS) vor. Jede Verbindung wird in Reinstwasser aufgelöst und mit einem 0,22 μm sterilen Spritzenvorsatzfilter filtriert. In einem sterilen Behälter bei 4 °C lagern.
ACHTUNG: AA und BIS sind hochgiftige Substanzen. Arbeiten Sie unter einem Abzug und verwenden Sie geeignete Schutzelemente (Handschuhe, Schutzkleidung, Maske und Schutzbrille). - Bereiten Sie die Lösung zur Verhinderung der Proteinvernetzung (CP) vor, indem Sie 38 μl einer 37%igen Formaldehyd (FA)-Lösung mit 50 μl der 40%igen Acrylamid-Stammlösung (Schritt 2.2) in 912 μl phosphatgepufferter Kochsalzlösung (PBS; Tabelle 1) um eine Endkonzentration von 1,4 % Formaldehyd und 2 % Acrylamid zu erhalten.
HINWEIS: Bereiten Sie die CP-Lösung immer frisch vor und seien Sie beim Pipettieren äußerst präzise. Verwenden Sie beispielsweise eine P1000-Pipette, um 900 μl PBS zu entnehmen, und eine P20-Pipette, um die restlichen 12 μl PBS zu entnehmen. - Bereiten Sie die monomere Lösung vor
HINWEIS: Verwenden Sie die Lösung nicht sofort nach der Zubereitung; Vor der Verwendung mindestens 24 Stunden bei -20 °C lagern.- Mischen Sie 500 μl der 38%igen SA-Lösung (Schritt 2.1), 250 μl der 40%igen AA-Lösung und 50 μl der BIS-Lösung (Schritt 2.2) sowie 100 μl 10x PBS (Tabelle 1).
- Aliquotieren Sie dieses endgültige Volumen von 900 μl in 10x 1,5 mL Mikrozentrifugenröhrchen mit je 90 μl monomerer Lösung. Bei -20 °C bis zu 2 Wochen lagern.
HINWEIS: Die SA-Lösung kann beim Pipettieren an der Spitze verbleiben, da sie sehr viskos ist. Seien Sie vorsichtig und verzichten Sie auf alles.
- Bereiten Sie die Denaturierungslösung vor
- Mischen Sie 114,3 ml einer 350 mM Natriumdodecylsulfat (SDS)-Lösung, die in Reinstwasser hergestellt wurde, mit 10 ml einer 4 M Natriumchloridlösung, die in Reinstwasser hergestellt wurde. 12 g Tris unter Rühren in einem 250-ml-Becherglas hinzufügen.
ACHTUNG: SDS ist hochgiftig; Verwenden Sie es unter einem Abzug und tragen Sie Handschuhe, Schutzkleidung, eine Maske und eine Schutzbrille. - Stellen Sie den pH-Wert mit einer konzentrierten Salzsäurelösung auf 9 ein. Mit Reinstwasser auf 200 ml auffüllen und in einem sterilen Kolben bei 4 °C lagern.
- Mischen Sie 114,3 ml einer 350 mM Natriumdodecylsulfat (SDS)-Lösung, die in Reinstwasser hergestellt wurde, mit 10 ml einer 4 M Natriumchloridlösung, die in Reinstwasser hergestellt wurde. 12 g Tris unter Rühren in einem 250-ml-Becherglas hinzufügen.
- Bereiten Sie eine 10%ige Ammoniumpersulfat (APS)-Lösung und eine 10%ige TEMED-Lösung vor.
- 0,1 g APS in 1 ml Reinstwasser auflösen.
- Bereiten Sie 1 ml einer 10%igen TEMED-Lösung in Reinstwasser vor.
- Bereiten Sie 100 μl Aliquots beider Lösungen in sterilen 1,5-ml-Mikrozentrifugenröhrchen vor und lagern Sie sie bis zu 1 Monat lang bei -20 °C.
- Bereiten Sie die Paraformaldehyd-Lösung vor.
- 2 g Paraformaldehyd in 40 ml PBS unter Rühren bei 60 °C lösen. Fügen Sie 1 M NaOH tropfenweise hinzu, bis die Lösung von weiß zu farblos wird.
ACHTUNG: Formaldehyd ist hochgiftig; Verwenden Sie es unter einem Abzug und tragen Sie Handschuhe, Schutzkleidung, eine Maske und eine Schutzbrille. - Kühlen Sie die Lösung bei Raumtemperatur ab und stellen Sie den pH-Wert mit NaOH auf 7,2 in einem Endvolumen von 50 ml ein. Mit einem 0,22 μm sterilen Spritzenvorsatzfilter filtrieren und in einem sterilen Behälter aufbewahren.
- Aliquots von 1 ml in sterilen 1,5-ml-Mikrozentrifugenröhrchen vorbereiten und bei -20 °C lagern.
- 2 g Paraformaldehyd in 40 ml PBS unter Rühren bei 60 °C lösen. Fügen Sie 1 M NaOH tropfenweise hinzu, bis die Lösung von weiß zu farblos wird.
- Bereiten Sie die Paraformaldehyd/Glutaraldehyd-Lösung vor.
- 0,2 g Paraformaldehyd in 3,5 ml Reinstwasser und 50 μl 16 M NaOH-Lösung geben. Erhitzen Sie die Lösung auf 60 °C, um das Paraformaldehyd aufzulösen.
- Abkühlen lassen und 300 μl 70% Glutaraldehyd hinzufügen. Erhöhen Sie das Volumen mit Reinstwasser auf 5 mL und schließlich mit PBS auf 10 mL.
ACHTUNG: Paraformaldehyd und Glutaraldehyd sind hochgiftig; Tragen Sie sie unter einem Abzug und tragen Sie Handschuhe, Schutzkleidung, eine Maske und eine Schutzbrille. - Aliquots von 1 ml in sterilen 1,5-ml-Mikrozentrifugenröhrchen vorbereiten und bei -20 °C lagern.
3. Vorbereitung der Parasitenkulturen
- Anbau von T. cruzi epimastigotes.
- Verwenden Sie eine axenische Kultur in einem T-25-Kolben (25 cm2 Wachstumsfläche) und halten Sie die Kulturen in der logarithmischen Phase, indem Sie alle 48-72 Stunden in Leberinfusions-Tryptose (LIT)-Medium mit 10 % fötalem Kälberserum (FCS; Tabelle 1).
- Stellen Sie sicher, dass der Deckel fest verschlossen ist, und halten Sie den Kulturkolben zur Inkubation senkrecht bei 28 °C. Überwachen Sie das Parasitenwachstum durch Zellzählung in einer Neubauer-Kammer während jeder Subkultur.
HINWEIS: Für den in dieser Studie verwendeten Dm28c-Stamm liegt die Konzentration von Epimastigoten in Log-Phasen-Kulturen zwischen 1-5 x 107 Parasiten/ml. - Bereiten Sie eine Suspension von 2 x 106 Epimastigoten/ml aus einer logarithmischen Phasenkultur in LIT-Medium vor, die mit 10 % FCS ergänzt wurde. Die Suspension wird bei 5.000 x g 10 min bei Raumtemperatur (RT) zentrifugiert. Ein- bis zweimal mit PBS waschen und in 200 μl PBS resuspendieren.
- Kleben Sie das runde 12 mm Deckglas auf, das zuvor mit Poly-D-Lysin beschichtet war (Abschnitt 1). Inkubieren Sie 15-20 Minuten bei RT. Fahren Sie mit dem Schritt zur Verhinderung von Vernetzungen fort (Abschnitt 4).
HINWEIS: Alternativ können die Epimastigoten 7 min lang mit kaltem Methanol oder 10 min mit Paraformaldehyd/Glutaraldehyd-Lösung (Schritt 2.8) bei RT fixiert werden, bevor die Parasiten am Deckglas haften. Es ist möglich, die fixierten Parasiten bis zu 1 Woche bei 4 °C zu lagern.
- Gewinnen Sie die Amastigoten aus infizierten Vero-Zellen.
- Legen Sie ein steriles 12 mm rundes Deckglas auf den Boden einer 24-Well-Gewebekulturplatte. Bereiten Sie eine Suspension von 2 x 105 Vero-Zellen/ml in Dulbeccos modifiziertem Eagle-Medium (DMEM) vor, ergänzt mit 2 % fötalem Kälberserum (FCS). 500 μl der Suspension pro Vertiefung aussäen.
HINWEIS: In dieser Studie wurde DMEM mit 2 % FCS verwendet, damit die Zellen langsamer wachsen können. - Über Nacht (ON; 12-16 h) bei 37 °C und 5 % CO2 inkubieren, um die Zelladhäsion sicherzustellen.
- Waschen Sie die Zellen nach der Inkubation zweimal mit 500 μl sterilem PBS. Geben Sie T. cruzi Trypomastigoten in den Zellen mit einer Infektionsmultiplizität (MOI) von 10 zu 100 μl DMEM mit 2 % FCS pro Vertiefung, was einer Million Trypomastigoten pro Vertiefung entspricht. Bei 37 °C und 5 % CO2 6 h inkubieren.
- Spülen Sie die Platten nach der Inkubation zweimal mit PBS ab. Fügen Sie 500 μl DMEM hinzu, ergänzt mit 2 % FCS. Fahren Sie an dieser Stelle mit dem Schritt zur Verhinderung von Vernetzungen fort (Abschnitt 4).
HINWEIS: Intrazytoplasmatische Amastigoten sind 2 Tage nach der Infektion durch ein inverses optisches Mikroskop sichtbar (Ergänzende Abbildung 1). Alternativ können die Trypomastigoten 7 min lang bei -20 °C mit kaltem Methanol oder 10 min bei RT mit Paraformaldehyd/Glutaraldehyd fixiert werden, bevor die Parasiten am Deckglas anhaften.
- Legen Sie ein steriles 12 mm rundes Deckglas auf den Boden einer 24-Well-Gewebekulturplatte. Bereiten Sie eine Suspension von 2 x 105 Vero-Zellen/ml in Dulbeccos modifiziertem Eagle-Medium (DMEM) vor, ergänzt mit 2 % fötalem Kälberserum (FCS). 500 μl der Suspension pro Vertiefung aussäen.
- Gewinnung von Trypomastigoten aus infizierten Vero-Zellen.
- Sammeln Sie den Überstand einer Vero-Zell-Monoschicht (30%-40% Konfluenz), die mit Trypomastigoten infiziert ist (MOI 1:10; IN-Inkubation) 4 Tage nach der Infektion.
HINWEIS: Für einen T-25-Kolben beträgt die Anfangskonzentration der verwendeten Vero-Zellen 800.000 Zellen und für einen T-75-Kolben zwei Millionen Zellen. - Bestimmen Sie die Trypomastigoten-Konzentration mit einer Neubauer-Kammer zur Zellzählung. Zentrifugieren Sie 4 x 106 Trypomastigoten à 7.000 x g bei RT für 10 min.
- Zweimal mit PBS spülen und in 200 μl PBS resuspendieren. Auf ein rundes, 12 mm Deckglas auftragen, das mit Poly-D-Lysin beschichtet ist (Abschnitt 1). Inkubieren Sie 10-15 Minuten bei RT. Fahren Sie mit dem Schritt zur Vernetzungsprävention fort (Abschnitt 4).
HINWEIS: Alternativ können die Trypomastigoten mit kaltem Methanol für 7 min bei -20 °C oder Paraformaldehyd/Glutaraldehyd für 10 min bei RT fixiert werden, bevor die Parasiten am Deckglas anhaften.
- Sammeln Sie den Überstand einer Vero-Zell-Monoschicht (30%-40% Konfluenz), die mit Trypomastigoten infiziert ist (MOI 1:10; IN-Inkubation) 4 Tage nach der Infektion.
4. Durchführung der Crosslinking-Prävention (TAG 1)
- Tauchen Sie das 12-mm-Deckglas mit den anhaftenden Parasiten oder infizierten Zellen (nach oben) in eine 24-Well-Platte mit 0,5 ml CP-Lösung (Schritt 2.3) in jeder Well.
- Füllen Sie die leeren Vertiefungen mit Wasser, um die Verdunstung zu reduzieren. Verschließen Sie die Platte mit einer Siegelfolie. 5 h bei 37 °C inkubieren. Dieser Schritt kann bis zu einer ON-Inkubation bei 4 °C verlängert werden.
HINWEIS: Tauchen Sie das Deckglas immer in die Lösung; Pipettieren Sie die Fixierlösung nicht über die Deckgläser.
5. Durchführung der Gelierung der Probe
- Bauen Sie eine feuchte Kammer in eine Petrischale mit einer Siegelfolie auf Seidenpapier ein (Abbildung 2A). Das Seidenpapier mit Wasser aufgießen und bei -20 °C 20 min abkühlen lassen. Tauen Sie ein TEMED und ein APS-Aliquot 20 min lang auf Eis auf (Schritt 2.6).
HINWEIS: Frieren und tauen Sie das APS nicht mehr als dreimal auf. - Legen Sie die kühle, feuchte Kammer auf Eis (Abbildung 2A). Nehmen Sie die in Abschnitt 4 vorbereitete 24-Well-Platte aus dem Inkubator. Aspirieren Sie die CP-Lösung mit einer 3-ml-Pasteur-Pipette und lassen Sie etwas Lösung übrig, da sonst die Deckgläser schwer zu entfernen sind.
- Entfernen Sie die 12 mm Deckgläser mit einer Pinzette von der Fixierlösung und legen Sie sie mit den Parasiten nach oben auf Seidenpapier.
HINWEIS: Es ist hilfreich, das Deckglas mit einer sterilen Nadel anzuheben und dann mit der Pinzette festzuhalten. - Zu einem Aliquot von 90 μl monomerer Lösung (Schritt 2.4) werden 5 μl TEMED und 5 μl APS hinzugefügt, die zuvor aufgetaut wurden. Mit einem Wirbelmischer nicht länger als 2-3 s mischen; Es ist nicht notwendig, die Tube mit dem Deckel zu verschließen.
HINWEIS: Fügen Sie TEMED immer zuerst und APS zuletzt hinzu. Die Zugabe von APS zuerst und TEMED zuletzt zur monomeren Lösung beschleunigt den Gelierungsprozess, so dass keine Zeit bleibt, ihn zu manipulieren. - Für jedes Deckglas schnell einen Tropfen von 35 μL über die Siegelfolie der Feuchtkammer geben. Nehmen Sie das Deckglas sofort mit einer Pinzette auf und legen Sie es mit den Parasiten nach unten über den Tropfen (Abbildung 2B).
HINWEIS: Erstellen Sie maximal zwei Deckgläser gleichzeitig; Es ist wichtig, diesen Schritt nicht zu verzögern, da die Lösung sehr schnell polymerisiert. - Die Feuchtkammer 5 Minuten auf Eis und dann 1 Stunde bei 37 °C inkubieren. Schalten Sie einen Heizblock bei 95 °C ein, um die richtige Temperatur für den nächsten Schritt zu gewährleisten.
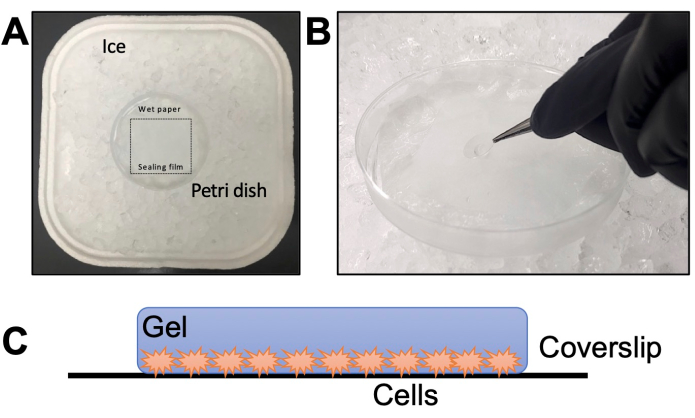
Abbildung 2: Details zum Gelierungsschritt. (A) Montage der Feuchtkammer. (B) Tropfen der Deckgläser auf die Monomerlösung mit TEMED und APS zur Gelierung. (C) Schematische Darstellung des für die Bildgebung zusammengesetzten Gels. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
6. Denaturierung der gelierten Proben und Durchführung der isotropen Expansion
- Die Denaturierungslösung (Schritt 2.5) aus 4 °C entfernen. Wenn es ausgefällt wird, legen Sie es in ein heißes Wasserbad, bis es sich vollständig aufgelöst hat.
- Geben Sie 2 ml der Denaturierungslösung in jede Vertiefung einer 6-Well-Platte. Übertragen Sie die Deckgläser aus Schritt 5.3 auf die Platte mit der Denaturierungslösung. Inkubieren Sie 15 Minuten lang bei RT unter leichtem Schütteln, damit sich das Gel vom Deckglas löst.
- Übertragen Sie das Gel vorsichtig mit einem Metallspatel (entfernen Sie es vom 12-mm-Deckglas) in ein steriles 1,5-ml-Mikrozentrifugenröhrchen mit 1 mL denaturierender Lösung. Verwenden Sie Kappenverschlüsse, um die Schläuche zu sichern. 1 h und 30 min bei 95 °C in einem Heizblock inkubieren.
HINWEIS: Die Gele beginnen sich während dieses Schritts auszudehnen; Seien Sie vorsichtig, wenn Sie das Gel in das 1,5-ml-Mikrozentrifugenröhrchen übertragen.
ACHTUNG: Nach der Inkubation hat das Röhrchen mit dem Gel eine Temperatur von 95 °C, was gefährlich sein kann. Tragen Sie Schutzhandschuhe und lassen Sie die Röhrchen abkühlen, bevor Sie sie anfassen, um Verbrennungen und Spritzer zu vermeiden. - Führen Sie die erste Erweiterungsrunde durch
- Aspirieren Sie die denaturierende Lösung aus dem 1,5-ml-Mikrozentrifugenröhrchen mit dem Gel mit einer P1000-Pipette. Übertragen Sie das Gel aus dem 1,5 mL Mikrozentrifugenröhrchen mit einem kleinen Spatel für 30 min in eine Petrischale mit 10 mL Reinstwasser.
- Wechseln Sie das Reinstwasser mit einer Einwegpipette von 3 mL Pasteur. Inkubieren Sie ON bei RT.
HINWEIS: Seien Sie vorsichtig mit den Gelen, da sie nach den ersten 30 Minuten der Inkubation brüchig wurden. - Wechseln Sie das Reinstwasser noch einmal mit einer 3-ml-Einwegpipette von Pasteur.
HINWEIS: Drei Wasserinkubationen von jeweils 30 Minuten sind ausreichend, aber aus praktischen Gründen ist es besser, die zweite Inkubation eingeschaltet zu lassen.
7. Durchführung der Fluoreszenzmarkierung der Zielproteine (TAG 2)
- Entfernen Sie das Wasser aus der Petrischale mit dem Gel mit einer Einweg-3-ml-Pasteurpipette. Messen Sie den Durchmesser des Gels mit einem Messschieber, um die Ausdehnung zu berechnen (zwischen vier und fünf Mal).
- Zweimal 15 Minuten lang mit 10 ml PBS waschen. Schneide das Gel mit einer Rasierklinge in der Mitte des kreisförmigen Gels in Quadrate von ca. 10 mm x 10 mm. Verwenden Sie ein Quadrat pro zu testender Bedingung.
HINWEIS: Das Gel schrumpft nach den PBS-Inkubationen. Gele können in PBS bei 4 °C bis zu 1 Woche gelagert werden. - Jedes Quadrat wird auf eine 12-Well-Platte umgefüllt und mit 500 μl Primärantikörper, verdünnt in 2 % PBS-Rinderserumalbumin (BSA), für 2 h und 30 min bei 37 °C unter Schütteln inkubiert.
HINWEIS: Die Inkubation alternativer Antikörper kann ON bei 4 °C durchgeführt werden. Das minimale Antikörpervolumen, das verwendet werden kann, beträgt 300 μl in einer 24-Well-Platte. In der Regel sollten Sie die doppelte Konzentration von Antikörpern verwenden, die für die herkömmliche Immunmarkierung verwendet werden. - Waschen Sie dreimal mit 2 mL PBS mit 0,1 % Polysorbat 20 für 10 Minuten unter Schütteln in einer 6-Well-Platte.
- Das Gel wird auf eine 12-Well-Platte umgefüllt und mit 500 μl Sekundärantikörper in PBS mit DAPI und 10 μg/mL NHS-Ester, konjugiert mit dem gewünschten Fluorophor für 2 h und 30 min bei 37 °C unter leichtem Schütteln inkubiert.
HINWEIS: Verwenden Sie in der Regel die doppelte Konzentration von Antikörpern, die für die herkömmliche Immunmarkierung verwendet werden. Alternativ kann diese Inkubation auch ON bei 4 °C durchgeführt werden. - Waschen Sie dreimal mit 2 mL PBS mit 0,1 % Polysorbat 20 für 10 Minuten unter Schütteln in einer 6-Well-Platte.
- Übertragen Sie das Gel in eine Petrischale mit Reinstwasser. 30 min inkubieren. Wechseln Sie das Wasser zweimal mit einer 3-ml-Einwegpipette von Pasteur, wie in Schritt 6.4 beschrieben.
8. Bildgebung und Bildverarbeitung (TAG 3)
- Entfernen Sie das Wasser aus den Petrischalen mit einer Einwegpipette von 3 mL Pasteur und messen Sie den Geldurchmesser mit einem Messschieber, um den Expansionsfaktor zu berechnen.
- Schneide mit einer Rasierklinge ein kleines Stück von ~10 mm x 10 mm ab und lege es auf eine 35 mm Glasbodenschale.
HINWEIS: Alternativ können Sie das Gel zwischen einen Objektträger und ein Deckglas (ohne Poly-D-Lysin) legen. Der Objektträger sollte zwei kleinere Objektträgerstücke haben, die mit einem Diamantmesser an den Seiten geschnitten werden, um eine Kammer von etwa der Dicke des Gels zu bilden. - Überprüfen Sie die Ausrichtung bei 10-facher oder 20-facher Vergrößerung. Um sich richtig zu fokussieren, stellen Sie sicher, dass die Parasiten dem Deckglas zugewandt sind. Andernfalls muss man das Gel umdrehen und erneut kontrollieren (Abbildung 2C).
- Sobald die richtige Ausrichtung gefunden ist, trocknen Sie das restliche Gel mit Mikroskopiepapier. Decken Sie das Gel mit einem Poly-D-Lysin-beschichteten Deckglas ab.
- Geben Sie einen kleinen Tropfen Reinstwasser in das Gel, um es mit einem Konfokalmikroskop zu visualisieren.
HINWEIS: Um eine korrekte Bildgebung zu gewährleisten, ist es wichtig, die Gelausrichtung während des gesamten Prozesses beizubehalten, so dass die Seite des Gels, das Zellen in der Nähe seiner Oberfläche enthält, am Ende der Glasbodenschale zugewandt ist. Bei der Verwendung von 35-mm-Schalen wird durch die Zugabe von Poly-D-Lysin zum Deckglas im Boden die Gelverschiebung während der Bildaufnahme reduziert. - Parameter für die Bilderfassung
- Nehmen Sie die Proben in einem konfokalen Mikroskop (Materialtabelle) mit einem 63-fachen Ölimmersionsobjektiv mit 1,4 numerischer Apertur (NA) auf.
- Erfassen Sie Z-Stacks, die Breite jedes Z-Schritts und eine Belichtungszeit pro Pixel, die empirisch in Abhängigkeit von der Abtastung, der Signalintensität und der Optimierung der Erfassungszeiten bestimmt wird. Verwenden Sie bei Bedarf den Scan-Zoom für eine effektive Vergrößerung.
- Öffnen Sie den Z-Stapel mit einer Bildverarbeitungssoftware (Materialtabelle). Gruppieren Sie für jeden Kanal die gestapelten Bilder mit der Option Z-Projekt gruppieren . Wählen Sie die maximale Intensitätsprojektion aus.
- Um Bilder zusammenzuführen, verwenden Sie das Werkzeug "Kanäle zusammenführen " und wählen Sie die Farbe der einzelnen Kanäle aus. Fügen Sie eine Maßstabsleiste mit dem Werkzeug Maßstabsleiste in der Verarbeitungssoftware hinzu.
HINWEIS: Alternativ kann man jeden Kanal nach Belieben einfärben (siehe Beispiele in den repräsentativen Ergebnissen).
Ergebnisse
Wenn das Protokoll ordnungsgemäß ausgeführt wurde (Abbildung 1), sind die Proben als planares und durchscheinendes Gel sichtbar, das in Wasser bis zu einem Faktor von 4-4,5x expandiert werden kann (Abbildung 3A). Diese Expansion ergab eine effektive Auflösung von etwa 70 nm, die je nach endgültigem Expansionsfaktor und verwendetem Bildgebungssystem variieren kann. Nach dem zweiten Expansionsprozess und der Bildaufnahme in e...
Diskussion
Die Ultrastrukturexpansionsmikroskopie ist eine Technik, die es ermöglicht, hochauflösende Bilder von biologischen Proben zu erhalten, indem sie physikalisch auf ein Vielfaches ihrer ursprünglichen Größe erweitert werden. Das U-ExM-Protokoll umfasst mehrere kritische Schritte, die sorgfältig ausgeführt werden müssen, um optimale Ergebnisse zu erzielen4. Zunächst muss die Probe mit einem CP-Mittel fixiert und in eine quellfähige Hydrogel-Matrix eingebette...
Offenlegungen
Die Autoren haben keine Interessenkonflikte offenzulegen.
Danksagungen
Wir danken Dolores Campos für die Unterstützung bei der Vero-Zellkultur und Romina Manarin für die Unterstützung bei der T. cruzi-Kultur . Diese Arbeit wurde unterstützt von der Agencia Nacional de Promoción Científica y Tecnológica, dem Ministerio de Ciencia e Innovación Productiva aus Argentinien (PICT2019-0526), dem Consejo Nacional de Investigaciones Científicas y Técnicas (PIBAA 1242) und dem Research Council United Kingdom [MR/P027989/1].
Materialien
| Name | Company | Catalog Number | Comments |
| 0.22 micrometers sterile syringe filters PES | Membrane solutions | SFPES030022S | |
| 1 L beaker | Schott Duran | 10005227 | |
| 1.5-mL SPINWIN Micro Centrifuge Tube | Tarson | T38-500010 | |
| 10 mL disposable sterile serynge | NP | 66-32 | |
| 10 mL serological pipette sterile | Jet Biofil | GSP211010 | |
| 12-mm coverslips | Merienfeld GmbH | 01 115 20 | Round coverslips |
| 12-well plates | Jet Biofil | TCP011012 | |
| 22-mm coverslips | Corning | 2845-22 | Square coverslips |
| 24-well plates | Jet Biofil | TCP-011-024 | |
| 250 mL beaker | Schott Duran | C108.1 | |
| 3 mL Pasteur pipette | Deltalab | 200037 | |
| 35-mm glass bottom dishes | Matsunami glass ind | D11130H | |
| 4′,6-Diamidine-2′-phenylindole dihydrochloride | Sigma Aldrich | D9542 | DAPI |
| 5 ml serological pipette sterile | Jet Biofil | GSP010005 | |
| 6-well plates | Sarstedt | 83.3920 | |
| Acrilamide | BioRad | 1610101 | |
| Ammonium persulfate | Sigma Aldrich | A3678-25G | APS |
| ATTO 647 NHS ester | BOC Sciences | F10-0107 | For pan-proteome labelling |
| Biosafty Cabinet | Telstar | Bio II A/P | |
| Bovine Sodium Albumine | Sigma Aldrich | A7906 | BSA |
| CO2 Incubator | Sanyo | MCO-15A | |
| Confocal Microscope | Zeiss | LSM 880 | |
| Disposable Petridish | Tarsons | 460095 | 90 mm diameter |
| DMEM, High Glucose | Thermo Fisher Cientific | 12100046 | Powder |
| Electronic digital caliper | Radar | RADAR-SLIDE-CALIPER | |
| Ethanol Absolute | Supelco | 1,00,98,31,000 | |
| Fetal Calf Serum | Internegocios SA | FCS FRA 500 | Sterile and heat-inactivated |
| Fiji image processing package | ImageJ | doi:10.1038/nmeth.2019 | |
| Formaldehyde 37% | Sigma Aldrich | F8775 | FA |
| Glass Petridish | Marienfeld Superior | PM-3400300 | 60 mm diameter |
| Glucosa D(+) | Cicarelli | 716214 | |
| Glutaraldehyde 70% | Sigma Aldrich | G7776 | |
| Goat anti-Mouse IgG Secondary Antibody Alexa Fluor 555 | Invitrogen | A-21422 | |
| Goat anti-Rabbit IgG Secondary Antibody FICT | Jackson Immunoresearch | 115-095-003 | |
| Graduated cylinder | Nalgene | 3663-1000 | |
| Graduated glass flask | Glassco | GL-274.202.01 | 100 mL |
| Heating Block | IBR | Made in house | |
| Hemin | Frontier Scientific | H651-9 | |
| Hydrochloric acid 36.8-38.0% | Ciccarelli | 918110 | |
| Ice bucket | Corning | 1167U68 | |
| Incubator | Tecno Dalvo | TOC130 | |
| Liver Infusion | Difco | 226920 | |
| Magnetic stirrer and heater | Lab companion | HP-3000 | |
| Metal spatula | SALTTECH | 200MM | |
| Metal tweezers | Marienfeld Superior | PM-6633002 | |
| Methanol absolut | Cicarelli | 897110 | |
| Microcentrifuge tube 1.5 mL | Tarson | 500010-N | |
| Microscopy grade paper KimWipes | Kimtech Science | B0013HT2QW | |
| Milli-Q water sistem | Merk Millipore | IQ-7003 | |
| mouse anti- alpha tubulin clone DM1A | Sigma Aldrich | T9026 | |
| mouse anti-PFR | Purified antibodies | Donated by Dr. Ariel Silber (USP) | |
| N,N´-methylenbisacrilamide | ICN | 193997 | BIS |
| Na2HPO4 | Cicarelli | 834214 | |
| Neubauer chamber | Boeco | BOE 01 | |
| p1000 pipette | Gilson | PIPETMAN P1000 | |
| p1000 pipette tips | Tarson | TAR-521020B | |
| p20 pipette | Gilson | PIPETMAN P20 | |
| p20 pipette tips | Tarson | TAR-527108 | |
| p200 pipette | Gilson | PIPETMAN P200 | |
| p200 pipette tips | Tarson | TAR-521010Y | |
| Paraformaldehyde | Sigma Aldrich | P6148 | PFA |
| pH / ORP / °C meter | HANNA Instruments | HI 2211 | |
| Poly-D-Lysine 0.1% | Sigma Aldrich | P8920 | |
| Potassium Chloride | Cicarelli | 867212 | KCl |
| Razor blade | Printex | BS 2982:1992 | |
| Sealing FIlm "Parafilm M" | Bemis | PM996 | |
| Sodium Acrilate | Sigma Aldrich | 408220-25G | SA |
| Sodium Bicarbonate | Cicarelli | 929211 | NaHCO3 |
| Sodium Chloride | Cicarelli | 750214 | NaCl |
| Sodium Dodecyl Sulfate | BioRad | 1610302 | SDS |
| Sodium Hidroxide | Merk | 1-06498 | NaOH |
| Sorvall ST 16 Centrifuge | Thermo Fisher Scientific | 75004380 | |
| T-25 flasks | Corning | 430639 | |
| TEMED | Invitrogen | 15524-010 | |
| Tissue paper | Elite | ||
| Triptose | Merck | 1106760500 | |
| Tris | BioRad | 1610719 | |
| Tween-20 | Biopack | 2003-07 | Polysorbate 20 |
| Vaccum pump | Silfab | N33-A | |
| Vero cells | ATCC | CRL-1587 | |
| Vortex MIxer | Dragon Lab | MX-S |
Referenzen
- Chen, F., Tillberg, P. W., Boyden, E. S. Expansion microscopy. Science. 347 (6221), 543-548 (2015).
- Chozinski, T. J., et al. Expansion microscopy with conventional antibodies and fluorescent proteins. Nature Methods. 13 (6), 485-488 (2016).
- Ku, T., et al. Multiplexed and scalable super-resolution imaging of three-dimensional protein localization in size-adjustable tissues. Nature Biotechnology. 34 (9), 973-981 (2016).
- Gambarotto, D., et al. Imaging cellular ultrastructures using expansion microscopy (U-ExM). Nature Methods. 16 (1), 71-74 (2018).
- Tillberg, P. W., Chen, F. Expansion microscopy: Scalable and convenient super-resolution microscopy. Annual Review of Cell and Developmental Biology. 35, 683-701 (2019).
- Jurriens, D., van Batenburg, V., Katrukha, E. A., Kapitein, L. C. Mapping the neuronal cytoskeleton using expansion microscopy. Methods in Cell Biology. 161, 105-124 (2021).
- Alonso, V. L. Ultrastructure expansion microscopy (U-ExM) in Trypanosoma cruzi: localization of tubulin isoforms and isotypes. Parasitology Research. 121 (10), 3019-3024 (2022).
- Kalichava, A., Ochsenreiter, T. Ultrastructure expansion microscopy in Trypanosoma brucei. Open Biology. 11 (10), 210132 (2021).
- Amodeo, S., et al. Characterization of the novel mitochondrial genome segregation factor TAP110 in Trypanosoma brucei. Journal of Cell Science. 134 (5), (2021).
- Liffner, B., Absalon, S. Expansion microscopy reveals plasmodium falciparum blood-stage parasites undergo anaphase with a chromatin bridge in the absence of mini-chromosome maintenance complex binding protein. Microorganisms. 9 (11), 2306 (2021).
- Dos Santos Pacheco, N., Soldati-Favre, D. Coupling auxin-inducible degron system with ultrastructure expansion microscopy to accelerate the discovery of gene function in Toxoplasma gondii. Methods in Molecular Biology. 2369, 121-137 (2021).
- Lidani, K. C. F., et al. Chagas disease: From discovery to a worldwide health problem. Frontiers in Public Health. 7, 166 (2019).
- Carrea, A., Diambra, L. Systems biology approach to model the life cycle of Trypanosoma cruzi. PloS One. 11 (1), e0146947 (2016).
- Vidal, J. C., De Souza, W. . Morphological and Functional Aspects of Cytoskeleton of Trypanosomatids. Cytoskeleton - Structure, Dynamics, Function and Disease. , (2017).
- Kunz, T. C., Götz, R., Gao, S., Sauer, M., Kozjak-Pavlovic, V. Using expansion microscopy to visualize and characterize the morphology of mitochondrial cristae. Frontiers in Cell and Developmental Biology. 8, 617 (2020).
- Laporte, M. H., Klena, N., Hamel, V., Guichard, P. Visualizing the native cellular organization by coupling cryofixation with expansion microscopy (Cryo-ExM). Nature Methods. 19 (2), 216-222 (2022).
- Sahabandu, N., et al. Expansion microscopy for the analysis of centrioles and cilia. Journal of Microscopy. 276 (3), 145-159 (2019).
- Wen, G., Leen, V., Rohand, T., Sauer, M., Hofkens, J. Current progress in expansion microscopy: chemical strategies and applications. Chemical Reviews. 123 (6), 3299-3323 (2023).
- Trinks, N., et al. Subdiffraction-resolution fluorescence imaging of immunological synapse formation between NK cells and A. fumigatus by expansion microscopy. Communications Biology. 4 (1), 1151 (2021).
- White, B. M., Kumar, P., Conwell, A. N., Wu, K., Baskin, J. M. Lipid expansion microscopy. Journal of the American Chemical Society. 144 (40), 18212-18217 (2022).
- Steib, E., et al. TissUExM enables quantitative ultrastructural analysis in whole vertebrate embryos by expansion microscopy. Cell Reports Methods. 2 (10), 100311 (2022).
- Shaib, A. H., et al. Visualizing proteins by expansion microscopy. bioRxiv. , (2023).
- M'Saad, O., et al. All-optical visualization of specific molecules in the ultrastructural context of brain tissue. bioRxiv. , (2022).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten