È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Microscopia ad espansione ultrastrutturale in tre fasi del ciclo di vita in vitro di Trypanosoma cruzi
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Questo studio mostra un protocollo dettagliato per eseguire la microscopia ad espansione ultrastrutturale in tre fasi del ciclo di vita in vitro di Trypanosoma cruzi, il patogeno responsabile della malattia di Chagas. Includiamo la tecnica ottimizzata per le proteine del citoscheletro e la marcatura del pan-proteoma.
Abstract
Descriviamo qui l'applicazione della microscopia ad espansione ultrastrutturale (U-ExM) in Trypanosoma cruzi, una tecnica che consente di aumentare la risoluzione spaziale di una cellula o di un tessuto per l'imaging microscopico. Ciò viene eseguito espandendo fisicamente un campione con sostanze chimiche standard e attrezzature di laboratorio comuni.
La malattia di Chagas è un problema di salute pubblica diffuso e pressante causato da T. cruzi. La malattia è prevalente in America Latina ed è diventata un problema significativo nelle regioni non endemiche a causa dell'aumento della migrazione. La trasmissione di T. cruzi avviene attraverso insetti vettori ematofagi appartenenti alle famiglie Reduviidae ed Hemiptera. Dopo l'infezione, gli amastigoti di T. cruzi si moltiplicano all'interno dell'ospite mammifero e si differenziano in tripomastigoti, la forma del flusso sanguigno non replicativa. Nell'insetto vettore, i tripomastigoti si trasformano in epimastigoti e proliferano attraverso la fissione binaria. La differenziazione tra le fasi del ciclo di vita richiede un ampio riarrangiamento del citoscheletro e può essere ricreata completamente in laboratorio utilizzando diverse tecniche di coltura cellulare.
Descriviamo qui un protocollo dettagliato per l'applicazione di U-ExM in tre fasi del ciclo di vita in vitro di Trypanosoma cruzi, concentrandosi sull'ottimizzazione dell'immunolocalizzazione delle proteine del citoscheletro. Abbiamo anche ottimizzato l'uso dell'estere N-idrossisuccinimide (NHS), un'etichetta pan-proteoma che ci ha permesso di marcare diverse strutture parassitarie.
Introduzione
La microscopia ad espansione (ExM) è stata descritta per la prima volta nel 2015 da Boyden et al.1. Si tratta di un protocollo di imaging con il quale un microscopio convenzionale può raggiungere una risoluzione spaziale inferiore al limite di diffrazione. Questa risoluzione più elevata è ottenuta a causa di un ingrandimento fisico del campione. Per raggiungere questo obiettivo, le molecole marcate in fluorescenza vengono reticolate in un idrogel, che viene successivamente espanso isotropicamente con acqua. Come risultato di questa espansione, i segnali vengono separati quasi isotropicamente in tutte e tre le dimensioni. Questo metodo impiega sostanze chimiche a basso costo e consente una risoluzione spaziale di circa 65 nm utilizzando microscopi convenzionali (confocali), che è circa quattro volte migliore della risoluzione standard di un microscopio confocale (circa 250 nm)1.
La pietra miliare successiva, che ha permesso l'uso della microscopia ad espansione in molti campi biologici, è stato l'adattamento della marcatura in immunofluorescenza con anticorpi convenzionali2. Un altro adattamento rispetto al protocollo ExM pubblicato inizialmente è l'analisi ingrandita del proteoma (MAP)3. Questo metodo ha introdotto l'uso di alte concentrazioni di acrilammide e paraformaldeide prima dell'immersione del campione nell'idrogel per prevenire la reticolazione intra e interproteica, il che ha portato a una migliore conservazione del contenuto proteico dei campioni e dell'architettura subcellulare. Questo protocollo alternativo è stato ottimizzato per ottenere una maggiore conservazione dell'ultrastruttura complessiva di organelli isolati utilizzando concentrazioni più basse di agenti fissativi (formaldeide/paraformaldeide e acrilammide); questo approccio è stato chiamato microscopia ad espansione ultrastrutturale (U-ExM)4.
Per ottenere una risoluzione ancora maggiore, è stata segnalata anche la combinazione di ExM con tecniche di microscopia a super-risoluzione, tra cui la microscopia a deplezione di emissione stimolata o la microscopia a localizzazione di una singola molecola, al fine di raggiungere risoluzioni inferiori a 20 nm5.
L'uso di ExM è stato ampiamente riportato nel campo delle neuroscienze e della ricerca sul citoscheletro6, ma solo pochi studi sono stati condotti su protisti parassiti. Il nostro laboratorio è stato il primo a segnalare l'applicazione di U-ExM in T. cruzi7. Il protocollo di base si basa principalmente sui precedenti rapporti U-ExM in Toxoplasma gondii, Plasmodium ssp. e Trypanosoma brucei 8,9,10,11.
Uno dei maggiori vantaggi di ExM è la sua natura modulare, che consente una grande flessibilità per adattarsi a diversi campioni biologici. Il protocollo può essere suddiviso in fasi (come la fissazione, la prevenzione della reticolazione o la gelificazione) che possono essere facilmente regolate dall'utente per soddisfare le proprie esigenze sperimentali. Inoltre, questa pipeline può essere modificata per migliorare la compatibilità con l'organismo modello o per ottenere una risoluzione specifica. Di conseguenza, ExM offre un enorme potenziale sia per i sistemi ottici avanzati che per quelli non avanzati, garantendo applicazioni più ampie in futuro.
La malattia di Chagas, chiamata anche tripanosomiasi americana, è una malattia endemica dell'America Latina causata dal Trypanosoma cruzi, un parassita protozoo. Il ciclo vitale del parassita è complesso e prevede due stadi di sviluppo nei mammiferi e due nell'insetto ospite (membri della famiglia dei Triatominidae), che è il vettore biologico di questa malattia. La malattia di Chagas appartiene al gruppo delle malattie tropicali neglette elencate dall'Organizzazione Mondiale della Sanità e rappresenta un problema economico e sociale significativo in America Latina. Studi epidemiologici stimano che 8 milioni di persone in tutto il mondo convivono con la malattia di Chagas e oltre 10.000 decessi all'anno. Questi numeri esemplificano l'importanza della malattia di Chagas come problema di salute pubblica in tutto il mondo. La distribuzione geografica della malattia di Chagas è cambiata negli ultimi decenni, con molti individui infetti che ora risiedono nelle grandi aree urbane a livello globale a causa dell'aumento delle migrazioni, al contrario delle aree principalmente rurali dell'America Latina dove è stata originariamente trovata12.
Le fasi di sviluppo di T. cruzi differiscono durante il suo ciclo di vita, che può essere replicato completamente in vitro. Gli epimastigoti sono forme replicative nell'insetto vettore e hanno un nucleo sferico nella regione centrale del corpo cellulare e un cinetoplasto a forma di barra (una struttura contenente DNA mitocondriale unica per i cinetoplastidi) nella regione anteriore rispetto al nucleo, con un flagello libero. I tripomastigoti sono la forma infettiva, non replicativa, e hanno un nucleo allungato, un cinetoplasto posteriore arrotondato e un flagello attaccato alla membrana plasmatica lungo l'intera lunghezza del parassita. Gli amastigoti sono la forma replicativa intracellulare; Hanno un nucleo nella regione centrale, un cinetoplasto a forma di bastoncino nella parte anteriore del corpo cellulare e un flagello ridotto. L'adattabilità del parassita a diversi ambienti è un riflesso di queste variazioni morfologiche. Vale anche la pena ricordare che questo ciclo di vita comporta una divisione simmetrica e diverse fasi di sviluppo transitorio13. Durante la differenziazione, il citoscheletro dei tripanosomatidi svolge un ruolo fondamentale. Questa struttura è formata da un corsetto di microtubuli subpellicolari disposti in una serie ordinata di microtubuli stabili sotto la membrana plasmatica. Inoltre, in questi organismi è presente un'asta paraflagellare, che è una struttura reticolare che corre parallela ed è attaccata all'assonema flagellare14. L'organizzazione precisa del citoscheletro e i cambiamenti strutturali nucleari lungo le fasi del ciclo cellulare coinvolgono meccanismi di regolazione genica unici specifici per i tripanosomatidi, rendendoli modelli interessanti per gli studi di biologia cellulare.
Date le piccole dimensioni di T. cruzi e di altri protozoi parassiti, U-ExM rappresenta un ottimo strumento per analizzare le caratteristiche strutturali di questi importanti patogeni. Come accennato in precedenza, l'applicabilità di questa tecnica su T. cruzi è stata convalidata per la prima volta dal Dr. Alonso7. Questo rapporto descrive in dettaglio un protocollo U-ExM completo, con particolare attenzione all'immunolocalizzazione delle proteine del citoscheletro durante le diverse fasi del ciclo di vita di T. cruzi. Inoltre, abbiamo ottimizzato l'uso dell'estere N-idrossisuccinimide (NHS), un'etichetta pan-proteoma che ci consente di marcare varie strutture parassitarie. Inoltre, viene descritta una metodologia in vitro per ottenere i tre stadi del parassita.
Protocollo
NOTA: La Figura 1 illustra l'intero disegno sperimentale.
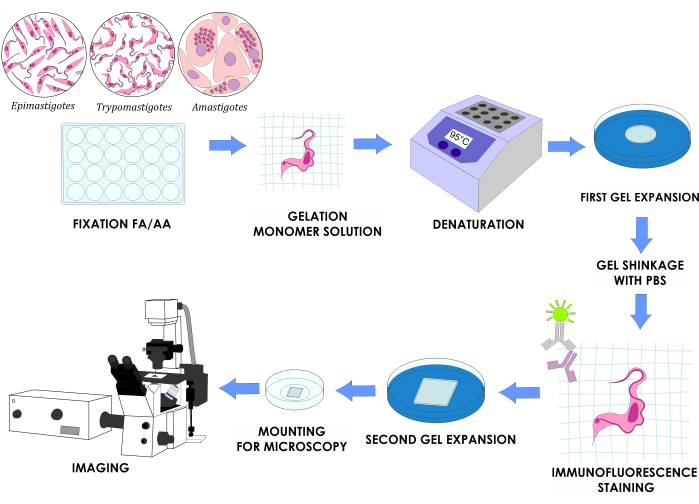
Figura 1: Flusso di lavoro U-ExM per tre fasi del ciclo di vita in vitro di T. cruzi. Fare clic qui per visualizzare una versione più grande di questa figura.
1. Preparazione dei vetrini coprioggetti rivestiti di poli-D-lisina
- Mettere un quadrato di 10 cm x 10 cm di pellicola sigillante in una capsula di Petri. Lavare i vetrini coprioggetti immergendoli in etanolo assoluto in una capsula Petri di vetro da 35 mm.
- Rimuovere i vetrini coprioggetti con una pinzetta dal bagno di etanolo e scolare il liquido in eccesso con carta velina. Posizionare i vetrini coprioggetti sulla pellicola sigillante.
- Assorbire il resto dell'etanolo con carta per microscopia. Aggiungere una soluzione allo 0,1% v/v di poli-D-lisina al centro del vetrino coprioggetti e stenderla con la punta fino a coprire circa l'80% della sua superficie. Chiudere la capsula di Petri e incubare per 1 ora a 37 °C.
NOTA: Per vetrini coprioggettida 22 mm 2, utilizzare 200 μL di soluzione di poli-D-lisina; per vetrini coprioggetti rotondi da 12 mm, utilizzare 100 μL di soluzione di poli-D-lisina. - Lavare i vetrini coprioggetti con acqua purissima tre volte. Utilizzare l'aspirazione con l'aspirapolvere per rimuovere l'acqua tra un lavaggio e l'altro. Conservare a 4 °C per un massimo di 1 settimana.
2. Preparazione della soluzione
- Preparare soluzioni madre di acrilato di sodio (SA) al 38% (p/p).
- Aggiungere lentamente 19 g di SA a 31 ml di acqua priva di nucleasi mescolando.
NOTA: Questa soluzione è molto viscosa; Prestare attenzione durante il pipettaggio. - Una volta che l'SA è completamente sciolto, conservarlo in un contenitore sterile. Conservarla a 4 °C e cambiarla ogni 6 mesi.
NOTA: SA a volte mostra segni di contaminazione con poliacrilato di sodio a seconda della marca, il che è evidente durante la preparazione della soluzione madre poiché diventa giallastra e torbida. Usalo con cautela se questo è il caso.
- Aggiungere lentamente 19 g di SA a 31 ml di acqua priva di nucleasi mescolando.
- Preparare una soluzione madre di acrilammide (AA) al 40% (p/v) e una soluzione madre di N'-metilene bisacrilammide (BIS) al 2% (p/v). Sciogliere ogni composto in acqua ultrapura e filtrare con un filtro per siringa sterile da 0,22 μm. Conservare in contenitore sterile a 4 °C.
ATTENZIONE: AA e BIS sono sostanze altamente tossiche. Lavorare sotto una cappa aspirante e utilizzare elementi di protezione adeguati (guanti, indumenti protettivi, maschera e occhiali di sicurezza). - Preparare la soluzione di prevenzione della reticolazione proteica (CP) mescolando 38 μL di una soluzione di formaldeide al 37% (FA) con 50 μL della soluzione madre di acrilammide al 40% (fase 2.2) in 912 μL di soluzione salina tamponata con fosfato (PBS; Tabella 1) per ottenere una concentrazione finale dell'1,4% di formaldeide e del 2% di acrilammide.
NOTA: Preparare sempre la soluzione CP fresca e prestare la massima precisione durante il pipettaggio. Ad esempio, utilizzare una pipetta P1000 per prelevare 900 μL di PBS e una pipetta P20 per prelevare i restanti 12 μL di PBS. - Preparare la soluzione monomerica
NOTA: Non utilizzare la soluzione subito dopo essere stata preparata; conservare a -20 °C almeno 24 ore prima dell'uso.- Miscelare 500 μl della soluzione di SA al 38% (fase 2.1), 250 μl della soluzione di AA al 40% e 50 μl della soluzione BIS (fase 2.2), nonché 100 μl di PBS 10x (Tabella 1).
- Aliquotare questo volume finale di 900 μl in 10 provette per microcentrifuga da 1,5 mL con 90 μl di soluzione monomerica ciascuna. Conservare a -20 °C per un massimo di 2 settimane.
NOTA: La soluzione SA può rimanere sul puntale durante il pipettaggio perché è molto viscosa; Fai attenzione e dispensa tutto.
- Preparare la soluzione denaturante
- Miscelare 114,3 mL di una soluzione di sodio dodecil solfato (SDS) da 350 mM preparata in acqua ultrapura con 10 mL di una soluzione di cloruro di sodio 4 M preparata in acqua ultrapura. Aggiungere 12 g di Tris mescolando in un becher da 250 ml.
ATTENZIONE: La SDS è altamente tossica; Usalo sotto una cappa aspirante indossando guanti, indumenti protettivi, maschera e occhiali di sicurezza. - Regolare il pH a 9 con una soluzione concentrata di acido cloridrico. Preparare fino a 200 ml con acqua ultrapura e conservare in un pallone sterile a 4 °C.
- Miscelare 114,3 mL di una soluzione di sodio dodecil solfato (SDS) da 350 mM preparata in acqua ultrapura con 10 mL di una soluzione di cloruro di sodio 4 M preparata in acqua ultrapura. Aggiungere 12 g di Tris mescolando in un becher da 250 ml.
- Preparare una soluzione di persolfato di ammonio (APS) al 10% e una soluzione TEMED al 10%.
- Sciogliere 0,1 g di APS in 1 mL di acqua ultrapura.
- Preparare 1 mL di una soluzione TEMED al 10% in acqua ultrapura.
- Preparare 100 μL di aliquote di entrambe le soluzioni in provette sterili per microcentrifuga da 1,5 mL e conservarle a -20 °C per un massimo di 1 mese.
- Preparare la soluzione di paraformaldeide.
- Sciogliere 2 g di paraformaldeide in 40 mL di PBS mescolando a 60 °C. Aggiungere 1 M di NaOH goccia a goccia fino a quando la soluzione diventa bianca o incolore.
ATTENZIONE: La formaldeide è altamente tossica; Usalo sotto una cappa aspirante indossando guanti, indumenti protettivi, maschera e occhiali di sicurezza. - Raffreddare la soluzione a temperatura ambiente e regolare il pH con NaOH a 7,2 in un volume finale di 50 mL. Filtrare con un filtro per siringa sterile da 0,22 μm e conservare in un contenitore sterile.
- Preparare aliquote da 1 mL in provette sterili da microcentrifuga da 1,5 mL e conservare a -20 °C.
- Sciogliere 2 g di paraformaldeide in 40 mL di PBS mescolando a 60 °C. Aggiungere 1 M di NaOH goccia a goccia fino a quando la soluzione diventa bianca o incolore.
- Preparare la soluzione di paraformaldeide/glutaraldeide.
- Aggiungere 0,2 g di paraformaldeide in 3,5 mL di acqua ultrapura e 50 μL di soluzione 16 M NaOH. Riscaldare la soluzione a 60 °C per sciogliere la paraformaldeide.
- Raffreddare e aggiungere 300 μl di glutaraldeide al 70%. Aumentare il volume a 5 ml con acqua ultrapura e infine a 10 ml con PBS.
ATTENZIONE: La paraformaldeide e la glutaraldeide sono altamente tossiche; Usali sotto una cappa aspirante indossando guanti, indumenti protettivi, maschera e occhiali di sicurezza. - Preparare aliquote da 1 mL in provette sterili da microcentrifuga da 1,5 mL e conservare a -20 °C.
3. Preparazione delle colture di parassiti
- Coltiva gli epimastigoti di T. cruzi .
- Utilizzare una coltura axenica in un pallone T-25 (area di crescita di 25cm2 ) e mantenere le colture nella fase logaritmica sub-colturando ogni 48-72 ore in terreno di infusione epatica triptosio (LIT) con il 10% di siero fetale di vitello (FCS; Tabella 1).
- Assicurarsi che il tappo sia ben chiuso e tenere il pallone di coltura in verticale a 28 °C per l'incubazione. Monitorare la crescita del parassita mediante conta cellulare in una camera di Neubauer durante ogni sottocoltura.
NOTA: Per il ceppo Dm28c utilizzato in questo studio, la concentrazione di epimastigoti nelle colture in fase logaritmica è compresa tra 1-5 x 107 parassiti/mL. - Preparare una sospensione di 2 x 106 epimastigoti/mL da una coltura in fase logaritmica in terreno LIT integrata con FCS al 10%. Centrifugare la sospensione a 5.000 x g per 10 minuti a temperatura ambiente (RT). Lavare con PBS una o due volte e risospendere in 200 μL di PBS.
- Aderire al vetrino coprioggetti rotondo da 12 mm precedentemente rivestito con poli-D-lisina (sezione 1). Incubare a RT per 15-20 min. Continuare con la fase di prevenzione della reticolazione (sezione 4).
NOTA: In alternativa, fissare gli epimastigoti con metanolo freddo per 7 minuti o con una soluzione di paraformaldeide/glutaraldeide (fase 2.8) per 10 minuti a RT prima dell'adesione dei parassiti al vetrino. È possibile conservare i parassiti fissi a 4 °C per un massimo di 1 settimana.
- Ottieni gli amastigoti dalle cellule Vero infette.
- Stendere un vetrino coprioggetto rotondo sterile da 12 mm sul fondo di una piastra di coltura tissutale a 24 pozzetti. Preparare una sospensione di 2 x 105 cellule Vero/mL nel terreno Eagle modificato (DMEM) di Dulbecco integrato con siero fetale di vitello (FCS) al 2%. Seminare 500 μl di sospensione per pozzetto.
NOTA: Questo studio utilizza DMEM con FCS al 2% per consentire alle cellule di crescere più lentamente. - Incubare per una notte (ON; 12-16 h) a 37 °C e 5% di CO2 per garantire l'adesione delle cellule.
- Dopo l'incubazione, lavare le cellule due volte utilizzando 500 μL di PBS sterile. Aggiungere T. cruzi tripomastigoti nelle cellule a una molteplicità di infezione (MOI) di 10 in 100 μL di DMEM con il 2% di FCS per pozzetto, corrispondente a un milione di tripomastigoti per pozzetto. Incubare a 37 °C e 5% CO2 per 6 ore.
- Dopo l'incubazione, sciacquare le piastre due volte con PBS. Aggiungere 500 μl di DMEM integrato con FCS al 2%. A questo punto, continuare con la fase di prevenzione della reticolazione (sezione 4).
NOTA: Gli amastigoti intracitoplasmatici saranno visibili attraverso un microscopio ottico invertito 2 giorni dopo l'infezione (Figura 1 supplementare). In alternativa, fissare i tripomastigoti con metanolo freddo per 7 minuti a -20 °C, o paraformaldeide/glutaraldeide per 10 minuti a RT, prima dell'adesione dei parassiti al vetrino.
- Stendere un vetrino coprioggetto rotondo sterile da 12 mm sul fondo di una piastra di coltura tissutale a 24 pozzetti. Preparare una sospensione di 2 x 105 cellule Vero/mL nel terreno Eagle modificato (DMEM) di Dulbecco integrato con siero fetale di vitello (FCS) al 2%. Seminare 500 μl di sospensione per pozzetto.
- Ottenere tripomastigoti dalle cellule Vero infette.
- Raccogliere il surnatante di un monostrato di cellule Vero (confluenza 30%-40%) infettato da tripomastigoti (MOI 1:10; ON incubazione) 4 giorni dopo l'infezione.
NOTA: Per un pallone T-25, la concentrazione iniziale di celle Vero utilizzate è di 800.000 cellule, mentre per un pallone T-75 è di due milioni di cellule. - Determinare la concentrazione di tripomastigote utilizzando una camera di Neubauer per il conteggio delle cellule. Centrifugare 4 x 106 tripomastigoti a 7.000 x g in RT per 10 min.
- Risciacquare due volte con PBS e risospendere in 200 μL di PBS. Applicare su un vetrino coprioggetti rotondo da 12 mm rivestito con poli-D-lisina (sezione 1). Incubare per 10-15 minuti a RT. Procedere con la fase di prevenzione della reticolazione (sezione 4).
NOTA: In alternativa, fissare i tripomastigoti con metanolo freddo per 7 minuti a -20 °C o paraformaldeide/glutaraldeide per 10 minuti a RT prima dell'adesione dei parassiti al vetrino.
- Raccogliere il surnatante di un monostrato di cellule Vero (confluenza 30%-40%) infettato da tripomastigoti (MOI 1:10; ON incubazione) 4 giorni dopo l'infezione.
4. Esecuzione della prevenzione della reticolazione (GIORNO 1)
- Immergere il vetrino coprioggetti da 12 mm con i parassiti aderenti o le cellule infette (rivolte verso l'alto) in una piastra a 24 pozzetti con 0,5 mL di soluzione CP (fase 2.3) in ciascun pozzetto.
- Riempire i pozzetti vuoti con acqua per ridurre l'evaporazione. Sigillare la piastra con una pellicola sigillante. Incubare per 5 ore a 37 °C. Questa fase può essere estesa fino a un'incubazione ON a 4 °C.
NOTA: Immergere sempre il vetrino coprioggetti nella soluzione; Non pipettare la soluzione fissativa sui vetrini.
5. Esecuzione della gelificazione del campione
- Assemblare una camera umida in una capsula di Petri con una pellicola sigillante sopra la carta velina (Figura 2A). Aggiungere acqua alla carta velina e incubare a -20 °C per 20 minuti per raffreddare. Scongelare un'aliquota TEMED e una APS sul ghiaccio per 20 minuti (passaggio 2.6).
NOTA: Non congelare-scongelare l'APS più di tre volte. - Stendere la camera fresca e umida sul ghiaccio (Figura 2A). Estrarre dall'incubatrice la piastra a 24 pozzetti preparata nella sezione 4. Aspirare la soluzione CP con una pipetta Pasteur da 3 mL, lasciando un po' di soluzione, altrimenti i vetrini coprioggetti saranno difficili da rimuovere.
- Rimuovere i vetrini coprioggetti da 12 mm dalla soluzione fissativa con una pinzetta e adagiarli su carta velina con i parassiti rivolti verso l'alto.
NOTA: È utile utilizzare un ago sterile per sollevare il vetrino coprioggetti e quindi tenerlo con le pinzette. - Ad un'aliquota di 90 μL di soluzione monomerica (fase 2.4), aggiungere 5 μL di TEMED e 5 μL di APS precedentemente scongelato. Miscelare con un miscelatore a vortice per non più di 2-3 s; Non è necessario chiudere il tubo con il coperchio.
NOTA: Aggiungi sempre TEMED per primo e APS per ultimo. L'aggiunta di APS prima e TEMED per ultimo alla soluzione monomerica rende il processo di gelificazione più veloce, senza dare il tempo di manipolarlo. - Versare rapidamente una goccia di 35 μl sulla pellicola sigillante della camera umida per ogni vetrino coprioggetti. Raccogliere immediatamente il vetrino coprioggetti con una pinzetta e stenderlo sopra la goccia (Figura 2B) con i parassiti rivolti verso il basso.
NOTA: Effettuare un massimo di due vetrini coprioggetti alla volta; È fondamentale non ritardare questo passaggio perché la soluzione polimerizza molto rapidamente. - Incubare la camera umida per 5 minuti con ghiaccio e poi per 1 ora a 37 °C. Accendere un blocco riscaldante a 95 °C per garantire la temperatura corretta per il passaggio successivo.
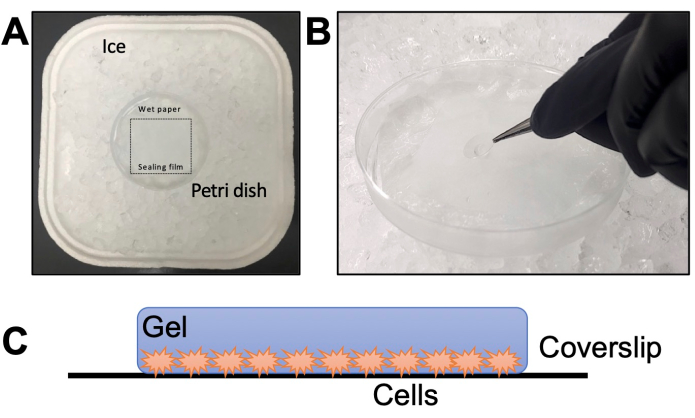
Figura 2: Dettagli della fase di gelificazione. (A) Assemblaggio della camera umida. (B) Far cadere i vetrini sulla soluzione monomerica con TEMED e APS per la gelificazione. (C) Rappresentazione schematica del gel assemblato per l'imaging. Clicca qui per visualizzare una versione più grande di questa figura.
6. Denaturazione dei campioni gelificati ed esecuzione dell'espansione isotropa
- Rimuovere la soluzione di denaturazione (passaggio 2.5) a 4 °C. Se è precipitato, metterlo a bagnomaria fino a quando non si scioglie completamente.
- Aggiungere 2 mL di soluzione denaturante a ciascun pozzetto di una piastra a 6 pozzetti. Trasferire i vetrini coprioggetti dal passaggio 5.3 alla piastra con la soluzione di denaturazione. Incubare per 15 minuti a RT agitando delicatamente in modo che il gel si stacchi dal vetrino.
- Trasferire con cautela il gel (rimuovendolo dal vetrino coprioggetti da 12 mm) con una spatola metallica in una provetta da microcentrifuga sterile da 1,5 mL con 1 mL di soluzione denaturante. Utilizzare i blocchi del tappo per fissare le provette. Incubare per 1 ora e 30 minuti a 95 °C in un blocco riscaldante.
NOTA: I gel iniziano ad espandersi durante questa fase; Prestare attenzione quando si trasferisce il gel nella provetta da microcentrifuga da 1,5 mL.
ATTENZIONE: Dopo l'incubazione, la provetta contenente il gel è a 95 °C, il che può essere pericoloso. Utilizzare guanti protettivi e lasciare raffreddare i tubi prima di maneggiarli per evitare ustioni e sporgenze. - Completa il primo round di espansione
- Aspirare la soluzione denaturante dalla provetta da microcentrifuga da 1,5 mL con il gel con una pipetta P1000. Trasferire il gel dalla provetta da microcentrifuga da 1,5 ml a una piastra di Petri con 10 ml di acqua ultrapura per 30 minuti utilizzando una piccola spatola.
- Sostituire l'acqua ultrapura utilizzando una pipetta Pasteur usa e getta da 3 ml. Incubare ON a RT.
NOTA: Essere delicati con i gel perché, dopo i primi 30 minuti di incubazione, sono diventati fragili. - Sostituire l'acqua ultrapura utilizzando ancora una volta una pipetta Pasteur usa e getta da 3 ml.
NOTA: Tre incubazioni in acqua di 30 minuti ciascuna sono sufficienti, ma per praticità è meglio lasciare accesa la seconda incubazione.
7. Esecuzione della marcatura a fluorescenza delle proteine bersaglio (GIORNO 2)
- Rimuovere l'acqua dalla capsula di Petri con il gel con una pipetta Pasteur usa e getta da 3 ml. Misurare il diametro del gel con un calibro per calcolare l'espansione (tra quattro e cinque volte).
- Lavare due volte con 10 ml di PBS per 15 minuti. Tagliare il gel con una lametta da barba in quadrati di circa 10 mm x 10 mm al centro del gel circolare. Utilizzare un quadrato per ogni condizione da testare.
NOTA: Il gel si restringe dopo l'incubazione del PBS. I gel possono essere conservati in PBS a 4 °C per un massimo di 1 settimana. - Trasferire ogni quadrato in una piastra a 12 pozzetti e incubare con 500 μl di anticorpo primario diluito in PBS-albumina sierica bovina (BSA) al 2% di PBS per 2 ore e 30 minuti a 37 °C con agitazione.
NOTA: L'incubazione alternativa degli anticorpi può essere eseguita ON a 4 °C. Il volume minimo di anticorpi che può essere utilizzato è di 300 μl in una piastra a 24 pozzetti. Come regola generale, utilizzare una concentrazione di anticorpi doppia rispetto a quella utilizzata per l'immunomarcatura convenzionale. - Lavare tre volte con 2 mL di PBS con polisorbato 20 allo 0,1% per 10 minuti agitando in una piastra a 6 pozzetti.
- Trasferire il gel in una piastra a 12 pozzetti e incubare con 500 μL di anticorpo secondario in PBS con DAPI e 10 μg/mL di estere NHS coniugato al fluoroforo desiderato per 2 ore e 30 minuti a 37 °C agitando delicatamente.
NOTA: Come regola generale, utilizzare una concentrazione di anticorpi doppia rispetto a quella utilizzata per l'immunomarcatura convenzionale. In alternativa, questa incubazione può essere eseguita ON a 4 °C. - Lavare tre volte con 2 mL di PBS con polisorbato 20 allo 0,1% per 10 minuti agitando in una piastra a 6 pozzetti.
- Trasferire il gel in una capsula di Petri con acqua ultrapura. Incubare per 30 min. Cambiare l'acqua due volte con una pipetta Pasteur usa e getta da 3 mL, come fatto al punto 6.4.
8. Imaging ed elaborazione delle immagini (GIORNO 3)
- Rimuovere l'acqua dalle piastre di Petri con una pipetta Pasteur usa e getta da 3 ml e misurare il diametro del gel con un calibro per calcolare il fattore di espansione.
- Taglia un pezzetto di ~10 mm x 10 mm con una lama di rasoio e posizionalo su un piatto con fondo di vetro da 35 mm.
NOTA: In alternativa, posizionare il gel tra un vetrino e un vetrino coprioggetti (senza poli-D-lisina). Il vetrino dovrebbe avere due pezzi di vetrino più piccoli tagliati con un coltello diamantato attaccato ai lati per formare una camera dello spessore del gel. - Controllare l'orientamento con un ingrandimento di 10x o 20x. Per mettere a fuoco correttamente, assicurarsi che i parassiti siano rivolti verso il vetrino; in caso contrario, è necessario girare il gel e ricontrollare (Figura 2C).
- Una volta trovato l'orientamento corretto, asciugare il gel rimanente con carta per microscopia. Coprire il gel con un vetrino coprioggetti rivestito di poli-D-lisina.
- Aggiungere una piccola goccia di acqua ultrapura al gel per visualizzarlo con un microscopio confocale.
NOTA: Per garantire un imaging corretto, è fondamentale mantenere l'orientamento del gel durante tutto il processo in modo che il lato del gel contenente le cellule vicino alla sua superficie sia rivolto verso il piatto con fondo di vetro all'estremità. Quando si utilizzano piastre da 35 mm, l'aggiunta di poli-D-lisina al vetrino coprioggetti posto nella parte inferiore riduce lo spostamento del gel durante l'acquisizione dell'immagine. - Parametri di acquisizione delle immagini
- Visualizzare i campioni in un microscopio confocale (Tabella dei materiali) utilizzando un obiettivo 63x ad immersione in olio con apertura numerica (NA) 1,4x.
- Acquisisci stack Z, la larghezza di ogni passo z e un tempo di esposizione per pixel, da determinare empiricamente in base al campione, all'intensità del segnale e all'ottimizzazione dei tempi di acquisizione. Se lo si desidera, utilizzare lo zoom di scansione per un ingrandimento efficace.
- Apri la pila Z utilizzando il software di elaborazione delle immagini (Tabella dei materiali). Per ogni canale, raggruppate le immagini impilate utilizzando l'opzione Raggruppa progetto Z . Selezionare la proiezione dell'intensità massima.
- Per unire le immagini, usa lo strumento Unisci canali e seleziona il colore di ciascun canale. Aggiungere una barra di scala utilizzando lo strumento Barra di scala nel software di elaborazione.
NOTA: In alternativa, è possibile colorare ogni canale come desiderato (vedere esempi nei risultati rappresentativi).
Risultati
Se il protocollo è stato eseguito correttamente (Figura 1), i campioni saranno visibili come un gel planare e traslucido che può essere espanso fino a un fattore di 4-4,5x in acqua (Figura 3A). Questa espansione ha fornito una risoluzione effettiva di circa 70 nm, che può variare a seconda del fattore di espansione finale e del sistema di imaging impiegato. Dopo il secondo processo di espansione e l'acquisizione dell'immagine...
Discussione
La microscopia ad espansione ultrastrutturale è una tecnica che consente di ottenere immagini ad alta risoluzione di campioni biologici espandendoli fisicamente fino a diverse volte la loro dimensione originale. Il protocollo U-ExM prevede diversi passaggi critici che devono essere eseguiti con attenzione per ottenere risultati ottimali4. In primo luogo, il campione deve essere fissato con un agente CP e incorporato in una matrice di idrogel rigonfiabile. La form...
Divulgazioni
Gli autori non hanno conflitti di interesse da rivelare.
Riconoscimenti
Ringraziamo Dolores Campos per l'assistenza alla coltura cellulare Vero e Romina Manarin per l'assistenza alla coltura di T. cruzi . Questo lavoro è stato sostenuto dall'Agencia Nacional de Promoción Científica y Tecnológica, dal Ministerio de Ciencia e Innovación Productiva dell'Argentina (PICT2019-0526), dal Consejo Nacional de Investigaciones Científicas y Técnicas (PIBAA 1242) e dal Research Council United Kingdom [MR/P027989/1].
Materiali
| Name | Company | Catalog Number | Comments |
| 0.22 micrometers sterile syringe filters PES | Membrane solutions | SFPES030022S | |
| 1 L beaker | Schott Duran | 10005227 | |
| 1.5-mL SPINWIN Micro Centrifuge Tube | Tarson | T38-500010 | |
| 10 mL disposable sterile serynge | NP | 66-32 | |
| 10 mL serological pipette sterile | Jet Biofil | GSP211010 | |
| 12-mm coverslips | Merienfeld GmbH | 01 115 20 | Round coverslips |
| 12-well plates | Jet Biofil | TCP011012 | |
| 22-mm coverslips | Corning | 2845-22 | Square coverslips |
| 24-well plates | Jet Biofil | TCP-011-024 | |
| 250 mL beaker | Schott Duran | C108.1 | |
| 3 mL Pasteur pipette | Deltalab | 200037 | |
| 35-mm glass bottom dishes | Matsunami glass ind | D11130H | |
| 4′,6-Diamidine-2′-phenylindole dihydrochloride | Sigma Aldrich | D9542 | DAPI |
| 5 ml serological pipette sterile | Jet Biofil | GSP010005 | |
| 6-well plates | Sarstedt | 83.3920 | |
| Acrilamide | BioRad | 1610101 | |
| Ammonium persulfate | Sigma Aldrich | A3678-25G | APS |
| ATTO 647 NHS ester | BOC Sciences | F10-0107 | For pan-proteome labelling |
| Biosafty Cabinet | Telstar | Bio II A/P | |
| Bovine Sodium Albumine | Sigma Aldrich | A7906 | BSA |
| CO2 Incubator | Sanyo | MCO-15A | |
| Confocal Microscope | Zeiss | LSM 880 | |
| Disposable Petridish | Tarsons | 460095 | 90 mm diameter |
| DMEM, High Glucose | Thermo Fisher Cientific | 12100046 | Powder |
| Electronic digital caliper | Radar | RADAR-SLIDE-CALIPER | |
| Ethanol Absolute | Supelco | 1,00,98,31,000 | |
| Fetal Calf Serum | Internegocios SA | FCS FRA 500 | Sterile and heat-inactivated |
| Fiji image processing package | ImageJ | doi:10.1038/nmeth.2019 | |
| Formaldehyde 37% | Sigma Aldrich | F8775 | FA |
| Glass Petridish | Marienfeld Superior | PM-3400300 | 60 mm diameter |
| Glucosa D(+) | Cicarelli | 716214 | |
| Glutaraldehyde 70% | Sigma Aldrich | G7776 | |
| Goat anti-Mouse IgG Secondary Antibody Alexa Fluor 555 | Invitrogen | A-21422 | |
| Goat anti-Rabbit IgG Secondary Antibody FICT | Jackson Immunoresearch | 115-095-003 | |
| Graduated cylinder | Nalgene | 3663-1000 | |
| Graduated glass flask | Glassco | GL-274.202.01 | 100 mL |
| Heating Block | IBR | Made in house | |
| Hemin | Frontier Scientific | H651-9 | |
| Hydrochloric acid 36.8-38.0% | Ciccarelli | 918110 | |
| Ice bucket | Corning | 1167U68 | |
| Incubator | Tecno Dalvo | TOC130 | |
| Liver Infusion | Difco | 226920 | |
| Magnetic stirrer and heater | Lab companion | HP-3000 | |
| Metal spatula | SALTTECH | 200MM | |
| Metal tweezers | Marienfeld Superior | PM-6633002 | |
| Methanol absolut | Cicarelli | 897110 | |
| Microcentrifuge tube 1.5 mL | Tarson | 500010-N | |
| Microscopy grade paper KimWipes | Kimtech Science | B0013HT2QW | |
| Milli-Q water sistem | Merk Millipore | IQ-7003 | |
| mouse anti- alpha tubulin clone DM1A | Sigma Aldrich | T9026 | |
| mouse anti-PFR | Purified antibodies | Donated by Dr. Ariel Silber (USP) | |
| N,N´-methylenbisacrilamide | ICN | 193997 | BIS |
| Na2HPO4 | Cicarelli | 834214 | |
| Neubauer chamber | Boeco | BOE 01 | |
| p1000 pipette | Gilson | PIPETMAN P1000 | |
| p1000 pipette tips | Tarson | TAR-521020B | |
| p20 pipette | Gilson | PIPETMAN P20 | |
| p20 pipette tips | Tarson | TAR-527108 | |
| p200 pipette | Gilson | PIPETMAN P200 | |
| p200 pipette tips | Tarson | TAR-521010Y | |
| Paraformaldehyde | Sigma Aldrich | P6148 | PFA |
| pH / ORP / °C meter | HANNA Instruments | HI 2211 | |
| Poly-D-Lysine 0.1% | Sigma Aldrich | P8920 | |
| Potassium Chloride | Cicarelli | 867212 | KCl |
| Razor blade | Printex | BS 2982:1992 | |
| Sealing FIlm "Parafilm M" | Bemis | PM996 | |
| Sodium Acrilate | Sigma Aldrich | 408220-25G | SA |
| Sodium Bicarbonate | Cicarelli | 929211 | NaHCO3 |
| Sodium Chloride | Cicarelli | 750214 | NaCl |
| Sodium Dodecyl Sulfate | BioRad | 1610302 | SDS |
| Sodium Hidroxide | Merk | 1-06498 | NaOH |
| Sorvall ST 16 Centrifuge | Thermo Fisher Scientific | 75004380 | |
| T-25 flasks | Corning | 430639 | |
| TEMED | Invitrogen | 15524-010 | |
| Tissue paper | Elite | ||
| Triptose | Merck | 1106760500 | |
| Tris | BioRad | 1610719 | |
| Tween-20 | Biopack | 2003-07 | Polysorbate 20 |
| Vaccum pump | Silfab | N33-A | |
| Vero cells | ATCC | CRL-1587 | |
| Vortex MIxer | Dragon Lab | MX-S |
Riferimenti
- Chen, F., Tillberg, P. W., Boyden, E. S. Expansion microscopy. Science. 347 (6221), 543-548 (2015).
- Chozinski, T. J., et al. Expansion microscopy with conventional antibodies and fluorescent proteins. Nature Methods. 13 (6), 485-488 (2016).
- Ku, T., et al. Multiplexed and scalable super-resolution imaging of three-dimensional protein localization in size-adjustable tissues. Nature Biotechnology. 34 (9), 973-981 (2016).
- Gambarotto, D., et al. Imaging cellular ultrastructures using expansion microscopy (U-ExM). Nature Methods. 16 (1), 71-74 (2018).
- Tillberg, P. W., Chen, F. Expansion microscopy: Scalable and convenient super-resolution microscopy. Annual Review of Cell and Developmental Biology. 35, 683-701 (2019).
- Jurriens, D., van Batenburg, V., Katrukha, E. A., Kapitein, L. C. Mapping the neuronal cytoskeleton using expansion microscopy. Methods in Cell Biology. 161, 105-124 (2021).
- Alonso, V. L. Ultrastructure expansion microscopy (U-ExM) in Trypanosoma cruzi: localization of tubulin isoforms and isotypes. Parasitology Research. 121 (10), 3019-3024 (2022).
- Kalichava, A., Ochsenreiter, T. Ultrastructure expansion microscopy in Trypanosoma brucei. Open Biology. 11 (10), 210132 (2021).
- Amodeo, S., et al. Characterization of the novel mitochondrial genome segregation factor TAP110 in Trypanosoma brucei. Journal of Cell Science. 134 (5), (2021).
- Liffner, B., Absalon, S. Expansion microscopy reveals plasmodium falciparum blood-stage parasites undergo anaphase with a chromatin bridge in the absence of mini-chromosome maintenance complex binding protein. Microorganisms. 9 (11), 2306 (2021).
- Dos Santos Pacheco, N., Soldati-Favre, D. Coupling auxin-inducible degron system with ultrastructure expansion microscopy to accelerate the discovery of gene function in Toxoplasma gondii. Methods in Molecular Biology. 2369, 121-137 (2021).
- Lidani, K. C. F., et al. Chagas disease: From discovery to a worldwide health problem. Frontiers in Public Health. 7, 166 (2019).
- Carrea, A., Diambra, L. Systems biology approach to model the life cycle of Trypanosoma cruzi. PloS One. 11 (1), e0146947 (2016).
- Vidal, J. C., De Souza, W. . Morphological and Functional Aspects of Cytoskeleton of Trypanosomatids. Cytoskeleton - Structure, Dynamics, Function and Disease. , (2017).
- Kunz, T. C., Götz, R., Gao, S., Sauer, M., Kozjak-Pavlovic, V. Using expansion microscopy to visualize and characterize the morphology of mitochondrial cristae. Frontiers in Cell and Developmental Biology. 8, 617 (2020).
- Laporte, M. H., Klena, N., Hamel, V., Guichard, P. Visualizing the native cellular organization by coupling cryofixation with expansion microscopy (Cryo-ExM). Nature Methods. 19 (2), 216-222 (2022).
- Sahabandu, N., et al. Expansion microscopy for the analysis of centrioles and cilia. Journal of Microscopy. 276 (3), 145-159 (2019).
- Wen, G., Leen, V., Rohand, T., Sauer, M., Hofkens, J. Current progress in expansion microscopy: chemical strategies and applications. Chemical Reviews. 123 (6), 3299-3323 (2023).
- Trinks, N., et al. Subdiffraction-resolution fluorescence imaging of immunological synapse formation between NK cells and A. fumigatus by expansion microscopy. Communications Biology. 4 (1), 1151 (2021).
- White, B. M., Kumar, P., Conwell, A. N., Wu, K., Baskin, J. M. Lipid expansion microscopy. Journal of the American Chemical Society. 144 (40), 18212-18217 (2022).
- Steib, E., et al. TissUExM enables quantitative ultrastructural analysis in whole vertebrate embryos by expansion microscopy. Cell Reports Methods. 2 (10), 100311 (2022).
- Shaib, A. H., et al. Visualizing proteins by expansion microscopy. bioRxiv. , (2023).
- M'Saad, O., et al. All-optical visualization of specific molecules in the ultrastructural context of brain tissue. bioRxiv. , (2022).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon