Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Microscopie à expansion ultrastructurale en trois étapes du cycle de vie in vitro de Trypanosoma cruzi
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Cette étude montre un protocole détaillé pour effectuer la microscopie à expansion d’ultrastructure dans trois stades du cycle de vie in vitro de Trypanosoma cruzi, l’agent pathogène responsable de la maladie de Chagas. Nous incluons la technique optimisée pour les protéines cytosquelettiques et le marquage pan-protéome.
Résumé
Nous décrivons ici l’application de la microscopie à expansion d’ultrastructure (U-ExM) chez Trypanosoma cruzi, une technique qui permet d’augmenter la résolution spatiale d’une cellule ou d’un tissu pour l’imagerie microscopique. Ceci est réalisé en élargissant physiquement un échantillon avec des produits chimiques prêts à l’emploi et des équipements de laboratoire courants.
La maladie de Chagas est un problème de santé publique répandu et pressant causé par T. cruzi. La maladie est répandue en Amérique latine et est devenue un problème important dans les régions non endémiques en raison de l’augmentation des migrations. La transmission de T. cruzi se fait par l’intermédiaire d’insectes vecteurs hématophages appartenant aux familles des Reduviidae et des Hémiptères. Après l’infection, les amastigotes de T. cruzi se multiplient au sein de l’hôte mammifère et se différencient en trypomastigotes, la forme non réplicative de la circulation sanguine. Chez l’insecte vecteur, les trypomastigotes se transforment en épimastigotes et prolifèrent par fission binaire. La différenciation entre les étapes du cycle de vie nécessite un réarrangement important du cytosquelette et peut être entièrement recréée en laboratoire à l’aide de différentes techniques de culture cellulaire.
Nous décrivons ici un protocole détaillé pour l’application d’U-ExM dans trois stades du cycle de vie in vitro de Trypanosoma cruzi, en nous concentrant sur l’optimisation de l’immunolocalisation des protéines du cytosquelette. Nous avons également optimisé l’utilisation de l’ester de N-hydroxysuccinimide (NHS), un marqueur pan-protéome qui nous a permis de marquer différentes structures parasitaires.
Introduction
La microscopie à expansion (ExM) a été décrite pour la première fois en 2015 par Boyden et al.1. Il s’agit d’un protocole d’imagerie avec lequel un microscope conventionnel peut atteindre une résolution spatiale inférieure à la limite de diffraction. Cette résolution plus élevée est obtenue en raison d’un agrandissement physique de l’échantillon. Pour ce faire, des molécules marquées par fluorescence sont réticulées à un hydrogel, qui est ensuite expansé isotrope avec de l’eau. À la suite de cette expansion, les signaux sont séparés presque isotropement dans les trois dimensions. Cette méthode utilise des produits chimiques peu coûteux et permet une résolution spatiale d’environ 65 nm à l’aide de microscopes conventionnels (confocaux), ce qui est environ quatre fois supérieur à la résolution standard d’un microscope confocal (environ 250 nm)1.
L’étape suivante, qui a permis l’utilisation de la microscopie à expansion dans de nombreux domaines biologiques, a été l’adaptation du marquage par immunofluorescence avec des anticorps conventionnels2. Une autre adaptation du protocole ExM initialement publié est l’analyse magnifiée du protéome (MAP)3. Cette méthode a introduit l’utilisation de fortes concentrations d’acrylamide et de paraformaldéhyde avant l’immersion de l’échantillon dans l’hydrogel pour éviter la réticulation intra et interprotéique, ce qui a conduit à une meilleure préservation de la teneur en protéines et de l’architecture subcellulaire des échantillons. Ce protocole alternatif a été optimisé pour obtenir une meilleure conservation de l’ultrastructure globale des organites isolés en utilisant des concentrations plus faibles d’agents fixateurs (formaldéhyde/paraformaldéhyde et acrylamide) ; cette approche a été appelée microscopie à expansion d’ultrastructure (U-ExM)4.
Pour obtenir encore plus de résolution, la combinaison d’ExM avec des techniques de microscopie à super-résolution, y compris la microscopie à déplétion par émission stimulée ou la microscopie de localisation à molécule unique, a également été rapportée afin d’atteindre des résolutions inférieures à 20 nm5.
L’utilisation d’ExM a été largement rapportée dans les domaines des neurosciences et de la recherche sur le cytosquelette6, mais seules quelques études ont été menées sur des protistes parasites. Notre laboratoire a été le premier à signaler l’application de U-ExM chez T. cruzi7. Le protocole de base est principalement basé sur les rapports précédents de l’U-ExM chez Toxoplasma gondii, Plasmodium ssp. et Trypanosoma brucei 8,9,10,11.
L’un des plus grands avantages d’ExM est sa nature modulaire, qui permet une grande flexibilité pour s’adapter à différents échantillons biologiques. Le protocole peut être divisé en étapes (telles que la fixation, la prévention de la réticulation ou la gélification) qui peuvent être facilement ajustées par l’utilisateur pour répondre à ses exigences expérimentales. De plus, ce pipeline peut être modifié pour améliorer la compatibilité avec l’organisme modèle ou pour obtenir une résolution spécifique. Par conséquent, ExM offre un énorme potentiel pour les systèmes optiques avancés et non avancés, garantissant ainsi des applications plus larges à l’avenir.
La maladie de Chagas, également appelée trypanosomiase américaine, est une maladie endémique d’Amérique latine causée par Trypanosoma cruzi, un parasite protozoaire. Le cycle de vie du parasite est complexe et comporte deux stades de développement chez les mammifères et deux chez l’insecte hôte (membres de la famille des Triatominidae), qui est le vecteur biologique de cette maladie. La maladie de Chagas appartient au groupe des maladies tropicales négligées répertoriées par l’Organisation mondiale de la santé et représente un problème économique et social important en Amérique latine. Des études épidémiologiques estiment que 8 millions de personnes dans le monde vivent avec la maladie de Chagas et plus de 10 000 décès par an. Ces chiffres illustrent l’importance de la maladie de Chagas en tant que problème de santé publique dans le monde entier. La répartition géographique de la maladie de Chagas a changé au cours des dernières décennies, de nombreuses personnes infectées résidant désormais dans les grandes zones urbaines du monde entier en raison de l’augmentation des migrations, par opposition aux zones principalement rurales d’Amérique latine où elle était initialement présente12.
Les stades de développement de T. cruzi diffèrent tout au long de son cycle de vie, qui peut être reproduit entièrement in vitro. Les épimastigotes sont des formes répliquatives chez l’insecte vecteur, et ils ont un noyau sphérique dans la région centrale du corps cellulaire et un kinétophlast en forme de barre (une structure contenant de l’ADN mitochondrial unique aux kinétoplastides) dans la région antérieure par rapport au noyau, avec un flagelle libre. Les trypomastigotes sont la forme infectieuse non réplicative et ont un noyau allongé, un kinétoplast postérieur arrondi et un flagelle attaché à la membrane plasmique sur toute la longueur du parasite. Les amastigotes sont la forme réplicative intracellulaire ; Ils ont un noyau dans la région centrale, un kinétoplaste en forme de bâtonnet dans la partie antérieure du corps cellulaire et un flagelle réduit. L’adaptabilité du parasite à différents environnements est le reflet de ces variations morphologiques. Il convient également de mentionner que ce cycle de vie implique une division symétrique et différents stades de développement transitoires13. Au cours de la différenciation, le cytosquelette des trypanosomatidés joue un rôle critique. Cette structure est formée par un corset de microtubules sous-pelliculaires disposés en un réseau ordonné de microtubules stables sous la membrane plasmique. De plus, un bâtonnet paraflagellaire est présent dans ces organismes, qui est une structure en forme de treillis qui est parallèle et attachée à l’axonème flagellaire14. L’organisation précise du cytosquelette et les changements structurels nucléaires tout au long des étapes du cycle cellulaire impliquent des mécanismes uniques de régulation génique spécifiques aux trypanosomatidés, ce qui en fait des modèles intéressants pour les études de biologie cellulaire.
Compte tenu de la petite taille de T. cruzi et d’autres parasites protozoaires, U-ExM présente un excellent outil pour analyser les caractéristiques structurelles de ces agents pathogènes importants. Comme mentionné précédemment, l’applicabilité de cette technique sur T. cruzi a été validée pour la première fois par le Dr Alonso7. Ce rapport détaille un protocole U-ExM complet, en mettant l’accent sur l’immunolocalisation des protéines du cytosquelette au cours des différentes étapes du cycle de vie de T. cruzi. De plus, nous avons optimisé l’utilisation de l’ester de N-hydroxysuccinimide (NHS), un marqueur pan-protéome qui nous permet de marquer diverses structures parasitaires. De plus, une méthodologie in vitro pour obtenir les trois stades du parasite est décrite.
Protocole
REMARQUE : La figure 1 illustre le plan expérimental complet.
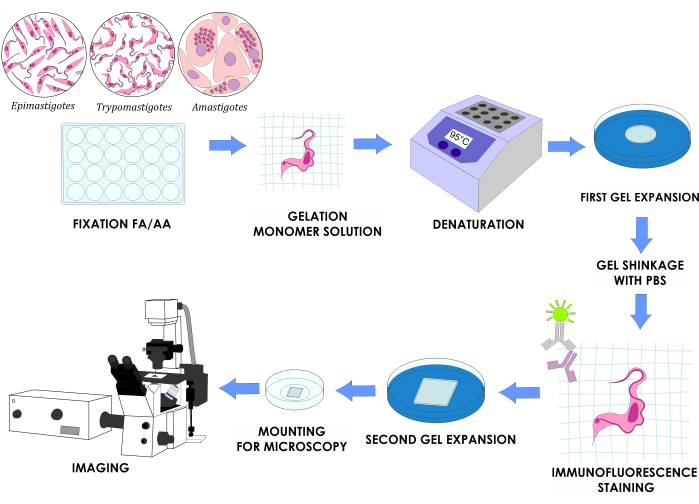
Figure 1 : Flux de travail U-ExM pour trois étapes du cycle de vie in vitro de T. cruzi. Veuillez cliquer ici pour voir une version agrandie de cette figure.
1. Préparation des lamelles enrobées de poly-D-lysine
- Placez un carré de film d’étanchéité de 10 cm x 10 cm dans une boîte de Pétri. Lavez les lamelles en les baignant dans de l’éthanol absolu dans une boîte de Pétri en verre de 35 mm.
- Retirez les lamelles à l’aide d’une pince à épiler du bain d’éthanol et égouttez l’excès de liquide avec du papier de soie. Placez les lamelles sur le film d’étanchéité.
- Absorbez le reste de l’éthanol avec du papier de qualité microscopique. Ajoutez une solution à 0,1 % v/v de poly-D-lysine au centre de la lamelle et étalez-la avec la pointe pour couvrir environ 80 % de sa surface. Fermez la boîte de Pétri et laissez incuber pendant 1 h à 37 °C.
REMARQUE : Pour les lamelles de22 mm 2 , utilisez 200 μL de solution de poly-D-lysine ; pour les lamelles rondes de 12 mm, utiliser 100 μL de solution de poly-D-lysine. - Lavez les lamelles à l’eau ultra-pure trois fois. Utilisez l’aspiration avec un aspirateur pour évacuer l’eau entre chaque lavage. Conserver à 4 °C jusqu’à 1 semaine.
2. Préparation de la solution
- Préparez des solutions mères d’acrylate de sodium (SA) à 38 % (p/p).
- Ajouter lentement 19 g d’AS à 31 ml d’eau exempte de nucléases en remuant.
REMARQUE : Cette solution est très visqueuse ; Faites attention lors du pipetage. - Une fois que l’AS est complètement dissous, conservez-le dans un récipient stérile. Conservez-le à 4 °C et changez-le tous les 6 mois.
REMARQUE : SA montre parfois des signes de contamination par du polyacrylate de sodium selon la marque, ce qui est perceptible lors de la préparation de la solution mère car elle devient jaunâtre et trouble. Utilisez-le avec prudence si tel est le cas.
- Ajouter lentement 19 g d’AS à 31 ml d’eau exempte de nucléases en remuant.
- Préparez une solution mère d’acrylamide à 40 % (p/v) (AA) et une solution mère de N, N'-méthylène bisacrylamide à 2 % (p/v). Dissoudre chaque composé dans de l’eau ultrapure et filtrer avec un filtre à seringue stérile de 0,22 μm. Conserver dans un récipient stérile à 4 °C.
ATTENTION : L’AA et le BIS sont des substances hautement toxiques. Passez sous une hotte et utilisez des éléments de protection appropriés (gants, vêtements de protection, masque et lunettes de sécurité). - Préparez la solution de prévention de la réticulation des protéines (PB) en mélangeant 38 μL d’une solution de formaldéhyde (FA) à 37 % avec 50 μL de la solution mère d’acrylamide à 40 % (étape 2.2) dans 912 μL de solution saline tamponnée au phosphate (PBS ; Tableau 1) pour obtenir une concentration finale de 1,4 % de formaldéhyde et 2 % d’acrylamide.
REMARQUE : Préparez toujours la solution de CP fraîchement et soyez extrêmement précis lors du pipetage. Par exemple, utilisez une pipette P1000 pour prélever 900 μL de PBS et une pipette P20 pour prélever les 12 μL de PBS restants. - Préparer la solution monomère
REMARQUE : N’utilisez pas la solution immédiatement après avoir été préparée ; Conserver à -20 °C au moins 24 h avant d’être utilisé.- Mélanger 500 μL de la solution SA à 38 % (étape 2.1), 250 μL de la solution AA à 40 % et 50 μL de la solution BIS (étape 2.2), ainsi que 100 μL de PBS 10x (tableau 1).
- Aliquote ce volume final de 900 μL dans 10 tubes de microcentrifugation de 1,5 mL contenant chacun 90 μL de solution monomère. Conserver à -20 °C jusqu’à 2 semaines.
REMARQUE : La solution d’AS peut rester sur l’extrémité lors du pipetage car elle est très visqueuse ; Soyez prudent et distribuez tout.
- Préparer la solution dénaturante
- Mélanger 114,3 mL d’une solution de dodécylsulfate de sodium (FDS) de 350 mM préparée dans de l’eau ultrapure avec 10 mL d’une solution de chlorure de sodium 4 M préparée dans de l’eau ultrapure. Ajouter 12 g de Tris en remuant dans un bécher de 250 ml.
ATTENTION : La FDS est très toxique ; Utilisez-le sous une hotte en portant des gants, des vêtements de protection, un masque et des lunettes de sécurité. - Ajustez le pH à 9 avec une solution concentrée d’acide chlorhydrique. Préparer jusqu’à 200 mL avec de l’eau ultrapure et conserver dans un ballon stérile à 4 °C.
- Mélanger 114,3 mL d’une solution de dodécylsulfate de sodium (FDS) de 350 mM préparée dans de l’eau ultrapure avec 10 mL d’une solution de chlorure de sodium 4 M préparée dans de l’eau ultrapure. Ajouter 12 g de Tris en remuant dans un bécher de 250 ml.
- Préparez une solution de persulfate d’ammonium à 10 % (APS) et une solution TEMED à 10 %.
- Dissoudre 0,1 g d’APS dans 1 mL d’eau ultrapure.
- Préparez 1 mL d’une solution TEMED à 10 % dans de l’eau ultrapure.
- Préparez des aliquotes de 100 μL des deux solutions dans des tubes stériles de microcentrifugation de 1,5 mL et conservez-les à -20 °C jusqu’à 1 mois.
- Préparez la solution de paraformaldéhyde.
- Dissoudre 2 g de paraformaldéhyde dans 40 mL de PBS en agitant à 60 °C. Ajouter 1 M de NaOH goutte à goutte jusqu’à ce que la solution passe du blanc à l’incolore.
ATTENTION : Le formaldéhyde est très toxique ; Utilisez-le sous une hotte en portant des gants, des vêtements de protection, un masque et des lunettes de sécurité. - Refroidir la solution à température ambiante et ajuster le pH avec du NaOH à 7,2 dans un volume final de 50 ml. Filtrer avec un filtre à seringue stérile de 0,22 μm et stocker dans un récipient stérile.
- Préparez des aliquotes de 1 mL dans des tubes de microcentrifugation stériles de 1,5 mL et conservez-les à -20 °C.
- Dissoudre 2 g de paraformaldéhyde dans 40 mL de PBS en agitant à 60 °C. Ajouter 1 M de NaOH goutte à goutte jusqu’à ce que la solution passe du blanc à l’incolore.
- Préparez la solution de paraformaldéhyde/glutaraldéhyde.
- Ajouter 0,2 g de paraformaldéhyde dans 3,5 mL d’eau ultrapure et 50 μL de solution de NaOH 16 M. Chauffer la solution à 60 °C pour dissoudre le paraformaldéhyde.
- Refroidir et ajouter 300 μL de glutaraldéhyde à 70 %. Augmentez le volume à 5 mL avec de l’eau ultrapure et enfin à 10 mL avec du PBS.
ATTENTION : Le paraformaldéhyde et le glutaraldéhyde sont très toxiques ; Utilisez-les sous une hotte en portant des gants, des vêtements de protection, un masque et des lunettes de sécurité. - Préparez des aliquotes de 1 mL dans des tubes de microcentrifugation stériles de 1,5 mL et conservez-les à -20 °C.
3. Préparation des cultures de parasites
- Cultivez des épimastigotes de T. cruzi .
- Utiliser une culture axénique dans une fiole T-25 (zone de croissance de 25 cm2 ) et maintenir les cultures dans la phase logarithmique en sous-cultivant toutes les 48 à 72 h dans un milieu de tryptose de perfusion hépatique (LIT) avec 10 % de sérum de veau fœtal (SCF ; Tableau 1).
- Assurez-vous que le capuchon est bien fermé et maintenez le ballon de culture verticalement à 28 °C pour l’incubation. Surveillez la croissance du parasite en comptant les cellules dans une chambre Neubauer lors de chaque sous-culture.
REMARQUE : Pour la souche Dm28c utilisée dans cette étude, la concentration d’épimastigotes dans les cultures en phase logarithmique est entre 1 et 5 x 107 parasites/mL. - Préparer une suspension de 2 x 106 épimastigotes/mL à partir d’une culture en phase logarithmique dans un milieu LIT complété par 10 % de FCS. Centrifuger la suspension à 5 000 x g pendant 10 min à température ambiante (RT). Laver avec du PBS une ou deux fois et remettre en suspension dans 200 μL de PBS.
- Adhérer à la lamelle ronde de 12 mm préalablement enduite de poly-D-lysine (section 1). Incuber à RT pendant 15-20 min. Passez à l’étape de prévention de la réticulation (section 4).
REMARQUE : Alternativement, fixez les épimastigotes avec du méthanol froid pendant 7 min ou une solution de paraformaldéhyde/glutaraldéhyde (étape 2.8) pendant 10 min à RT avant l’adhérence des parasites à la lamelle. Il est possible de stocker les parasites fixés à 4 °C pendant 1 semaine maximum.
- Obtenez les amastigotes à partir de cellules Vero infectées.
- Posez une lamelle ronde stérile de 12 mm au fond d’une plaque de culture tissulaire à 24 puits. Préparez une suspension de 2 x 105 cellules Vero/mL dans le milieu Eagle modifié de Dulbecco (DMEM) complété par 2 % de sérum de veau fœtal (FCS). Grainez 500 μL de suspension par puits.
REMARQUE : Cette étude utilise DMEM avec 2 % de FCS pour permettre aux cellules de se développer plus lentement. - Incuber pendant la nuit (ON ; 12-16 h) à 37 °C et 5 % de CO2 pour assurer la fixation de la cellule.
- Après l’incubation, laver les cellules deux fois avec 500 μL de PBS stérile. Ajouter des trypomastigotes de T. cruzi dans les cellules à une multiplicité d’infection (MOI) de 10 dans 100 μL de DMEM avec 2 % de FCS par puits, ce qui correspond à un million de trypomastigotes par puits. Incuber à 37 °C et 5 % de CO2 pendant 6 h.
- Après l’incubation, rincez deux fois les plaques avec du PBS. Ajouter 500 μL de DMEM complété par 2 % de FCS. À ce stade, passez à l’étape de prévention de la réticulation (section 4).
REMARQUE : Les amastigotes intracytoplasmiques seront visibles au microscope optique inversé 2 jours après l’infection (figure supplémentaire 1). Alternativement, fixez les trypomastigotes avec du méthanol froid pendant 7 min à -20 °C, ou du paraformaldéhyde/glutaraldéhyde pendant 10 min à RT, avant l’adhérence des parasites à la lamelle.
- Posez une lamelle ronde stérile de 12 mm au fond d’une plaque de culture tissulaire à 24 puits. Préparez une suspension de 2 x 105 cellules Vero/mL dans le milieu Eagle modifié de Dulbecco (DMEM) complété par 2 % de sérum de veau fœtal (FCS). Grainez 500 μL de suspension par puits.
- Obtenez des trypomastigotes à partir de cellules Vero infectées.
- Prélever le surnageant d’une monocouche de cellules Vero (30 %-40 % de confluence) infectée par des trypomastigotes (MOI 1:10 ; ON incubation) 4 jours après l’infection.
REMARQUE : Pour une fiole T-25, la concentration initiale de cellules Vero utilisée est de 800 000 cellules, et pour une fiole T-75, elle est de deux millions de cellules. - Déterminer la concentration de trypomastigote à l’aide d’une chambre de Neubauer pour le comptage cellulaire. Centrifugeuse 4 x 106 trypomastigotes à 7 000 x g à RT pendant 10 min.
- Rincer deux fois avec du PBS et remettre en suspension dans 200 μL de PBS. Appliquer sur une lamelle ronde de 12 mm recouverte de poly-D-lysine (section 1). Incuber pendant 10 à 15 min à RT. Procéder à l’étape de prévention de la réticulation (section 4).
REMARQUE : Alternativement, fixez les trypomastigotes avec du méthanol froid pendant 7 min à -20 °C ou du paraformaldéhyde/glutaraldéhyde pendant 10 min à RT avant l’adhérence des parasites à la lamelle.
- Prélever le surnageant d’une monocouche de cellules Vero (30 %-40 % de confluence) infectée par des trypomastigotes (MOI 1:10 ; ON incubation) 4 jours après l’infection.
4. Effectuer la prévention de la réticulation (JOUR 1)
- Immerger la lamelle de 12 mm avec les parasites collés ou les cellules infectées (vers le haut) dans une plaque à 24 puits avec 0,5 mL de solution de PC (étape 2.3) dans chaque puits.
- Remplissez les puits vides avec de l’eau pour réduire l’évaporation. Scellez la plaque avec un film d’étanchéité. Incuber pendant 5 h à 37 °C. Cette étape peut être prolongée jusqu’à une incubation ON à 4 °C.
REMARQUE : Immergez toujours la lamelle dans la solution ; Ne pipetez pas la solution fixatrice sur les lamelles.
5. Réalisation de la gélification de l’échantillon
- Assemblez une chambre humide dans une boîte de Pétri avec un film d’étanchéité sur du papier de soie (Figure 2A). Ajoutez de l’eau dans le papier de soie et laissez incuber à -20 °C pendant 20 min pour qu’il refroidisse. Décongeler une aliquote TEMED et une aliquote APS sur de la glace pendant 20 min (étape 2.6).
REMARQUE : Ne congelez pas et ne décongelez pas l’APS plus de trois fois. - Posez la chambre froide et humide sur de la glace (Figure 2A). Sortez de l’incubateur la plaque à 24 puits préparée dans la section 4. Aspirez la solution de PC à l’aide d’une pipette Pasteur de 3 ml, en laissant un peu de solution, sinon les lamelles seront difficiles à retirer.
- Retirez les lamelles de 12 mm de la solution fixatrice à l’aide d’une pince à épiler et posez-les sur du papier de soie avec les parasites vers le haut.
REMARQUE : Il est utile d’utiliser une aiguille stérile pour soulever la lamelle, puis de la tenir avec la pince à épiler. - À une aliquote de 90 μL de solution monomère (étape 2.4), ajouter 5 μL de TEMED et 5 μL d’APS préalablement décongelés. Mélanger avec un mélangeur vortex pendant pas plus de 2-3 s ; Il n’est pas nécessaire de fermer le tube avec le couvercle.
REMARQUE : Ajoutez toujours TEMED en premier et APS en dernier. L’ajout d’APS d’abord et de TEMED en dernier à la solution monomère accélère le processus de gélification, ne laissant pas le temps de le manipuler. - Versez rapidement une goutte de 35 μL sur le film d’étanchéité de la chambre humide pour chaque lamelle. Ramassez immédiatement la lamelle à l’aide d’une pince à épiler et posez-la sur la goutte (figure 2B) avec les parasites vers le bas.
REMARQUE : Faites un maximum de deux lamelles à la fois ; Il est crucial de ne pas retarder cette étape car la solution polymérise très rapidement. - Incuber la chambre humide pendant 5 min sur de la glace puis pendant 1 h à 37 °C. Allumez un bloc chauffant à 95 °C pour vous assurer que la température est correcte pour l’étape suivante.
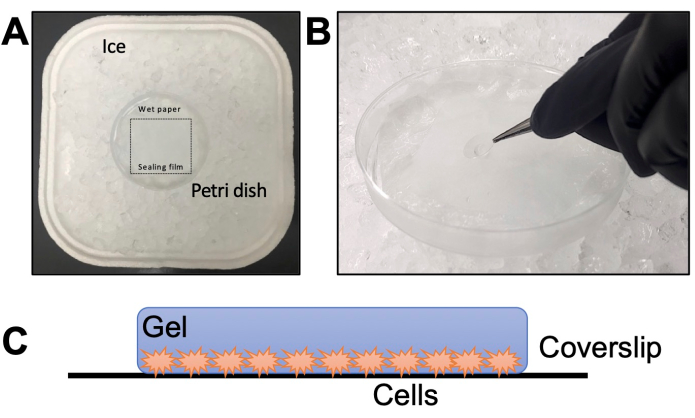
Figure 2 : Détails de l’étape de gélification. (A) Assemblage de la chambre humide. (B) Déposer les lamelles sur la solution monomère avec TEMED et APS pour la gélification. (C) Représentation schématique du gel assemblé pour l’imagerie. Veuillez cliquer ici pour voir une version agrandie de cette figure.
6. Dénaturation des échantillons gélifiés et réalisation de l’expansion isotrope
- Retirer la solution de dénaturation (étape 2.5) à partir de 4 °C. S’il est précipité, mettez-le dans un bain d’eau chaude jusqu’à ce qu’il se dissolve complètement.
- Ajouter 2 mL de la solution dénaturante dans chaque puits d’une plaque à 6 puits. Transférez les lamelles de l’étape 5.3 sur la plaque avec la solution de dénaturation. Incuber pendant 15 minutes à RT en secouant doucement pour que le gel se détache de la lamelle.
- Transvaser délicatement le gel (en le retirant de la lamelle de 12 mm) à l’aide d’une spatule métallique dans un tube de microcentrifugation stérile de 1,5 mL avec 1 mL de solution dénaturante. Utilisez des verrous de capuchon pour fixer les tubes. Incuber pendant 1 h et 30 min à 95 °C dans un bloc chauffant.
REMARQUE : Les gels commencent à se dilater au cours de cette étape ; soyez doux lors du transfert du gel dans le tube de microcentrifugation de 1,5 ml.
ATTENTION : Après l’incubation, le tube contenant le gel est à 95 °C, ce qui peut être dangereux. Utilisez des gants de protection et laissez refroidir les tubes avant de les manipuler pour éviter les brûlures et les projections. - Effectuer le premier tour d’expansion
- Aspirez la solution dénaturante du tube de microcentrifugation de 1,5 mL avec le gel avec une pipette P1000. Transférez le gel du tube de microcentrifugation de 1,5 mL dans une boîte de Pétri avec 10 mL d’eau ultrapure pendant 30 min à l’aide d’une petite spatule.
- Changez l’eau ultrapure à l’aide d’une pipette Pasteur jetable de 3 ml. Incuber ON chez RT.
REMARQUE : Soyez doux avec les gels car, après les 30 premières minutes d’incubation, ils sont devenus fragiles. - Changez une fois de plus l’eau ultrapure à l’aide d’une pipette Pasteur jetable de 3 ml.
REMARQUE : Trois incubations d’eau de 30 minutes chacune sont suffisantes, mais pour des raisons pratiques, il est préférable de laisser la deuxième incubation allumée.
7. Réalisation du marquage par fluorescence des protéines cibles (JOUR 2)
- Prélever l’eau de la boîte de Pétri avec le gel à l’aide d’une pipette Pasteur jetable de 3 ml. Mesurez le diamètre du gel à l’aide d’un pied à coulisse pour calculer la dilatation (entre quatre et cinq fois).
- Laver deux fois avec 10 mL de PBS pendant 15 min. Coupez le gel avec une lame de rasoir en carrés d’environ 10 mm x 10 mm au centre du gel circulaire. Utilisez un carré par condition à tester.
REMARQUE : Le gel rétrécit après les incubations PBS. Les gels peuvent être conservés dans du PBS à 4 °C jusqu’à 1 semaine. - Transvaser chaque carré dans une plaque à 12 puits et incuber avec 500 μL d’anticorps primaire dilué dans 2 % de PBS-albumine sérique bovine (BSA) pendant 2 h et 30 min à 37 °C en agitant.
REMARQUE : L’incubation alternative des anticorps peut être effectuée à 4 °C. Le volume minimum d’anticorps pouvant être utilisé est de 300 μL dans une plaque de 24 puits. En règle générale, utilisez deux fois la concentration d’anticorps utilisée pour l’immunomarquage conventionnel. - Laver trois fois avec 2 ml de PBS avec 0,1 % de polysorbate 20 pendant 10 minutes en agitant dans une assiette à 6 puits.
- Transférez le gel dans une plaque à 12 puits et incubez avec 500 μL d’anticorps secondaires dans du PBS avec DAPI et 10 μg/mL d’ester NHS conjugué au fluorophore souhaité pendant 2 h et 30 min à 37 °C en agitant doucement.
REMARQUE : En règle générale, utilisez deux fois la concentration d’anticorps utilisée pour l’immunomarquage conventionnel. Alternativement, cette incubation peut être effectuée ON à 4 °C. - Laver trois fois avec 2 ml de PBS avec 0,1 % de polysorbate 20 pendant 10 minutes en agitant dans une assiette à 6 puits.
- Transférez le gel dans une boîte de Pétri avec de l’eau ultra-pure. Incuber pendant 30 min. Changez l’eau deux fois avec une pipette Pasteur jetable de 3 ml, comme indiqué à l’étape 6.4.
8. Imagerie et traitement d’images (JOUR 3)
- Prélever l’eau des boîtes de Pétri à l’aide d’une pipette Pasteur jetable de 3 ml et mesurer le diamètre du gel à l’aide d’un pied à coulisse pour calculer le facteur de dilatation.
- Coupez un petit morceau de ~10 mm x 10 mm avec une lame de rasoir et placez-le sur un plat à fond en verre de 35 mm.
REMARQUE : Comme alternative, placez le gel entre une lame et une lamelle (sans poly-D-lysine). La lame doit avoir deux pièces de diapositive plus petites coupées avec un couteau diamanté fixé sur les côtés pour former une chambre de l’épaisseur du gel. - Vérifiez l’orientation à un grossissement de 10x ou 20x. Afin de bien se concentrer, assurez-vous que les parasites font face à la lamelle ; sinon, il faut retourner le gel et vérifier à nouveau (Figure 2C).
- Une fois l’orientation correcte trouvée, séchez le gel restant avec du papier de microscopie. Recouvrez le gel d’une lamelle enrobée de poly-D-lysine.
- Ajoutez une petite goutte d’eau ultrapure au gel pour visualiser avec un microscope confocal.
REMARQUE : Pour assurer une imagerie correcte, il est crucial de maintenir l’orientation du gel tout au long du processus afin que le côté du gel contenant les cellules près de sa surface soit face à la boîte à fond de verre à l’extrémité. Lors de l’utilisation de plats de 35 mm, l’ajout de poly-D-lysine à la lamelle placée dans le fond réduit le déplacement du gel lors de l’acquisition de l’image. - Paramètres d’acquisition d’images
- Imagez les échantillons au microscope confocal (Table of Materials) à l’aide d’un objectif à immersion dans l’huile 63x à ouverture numérique (NA) de 1,4.
- Acquérez des piles Z, la largeur de chaque pas z et un temps d’exposition par pixel, à déterminer empiriquement en fonction de l’échantillon, de l’intensité du signal et de l’optimisation des temps d’acquisition. Utilisez le zoom de balayage pour un grossissement efficace si vous le souhaitez.
- Ouvrez la pile Z à l’aide d’un logiciel de traitement d’images (Table des matériaux). Pour chaque couche, regroupez les images empilées à l’aide de l’option Regrouper le projet Z . Sélectionnez la projection d’intensité maximale.
- Pour fusionner des images, utilisez l’outil Fusionner les couches et sélectionnez la couleur de chaque couche. Ajoutez une barre d’échelle à l’aide de l’outil Barre d’échelle du logiciel de traitement.
REMARQUE : Alternativement, on peut colorer chaque canal comme on le souhaite (voir les exemples dans les résultats représentatifs).
Résultats
Si le protocole a été correctement exécuté (figure 1), les échantillons seront visibles sous la forme d’un gel planaire et translucide qui peut être expansé jusqu’à un facteur de 4 à 4,5 fois dans l’eau (figure 3A). Cette expansion a fourni une résolution effective d’environ 70 nm, qui peut varier en fonction du facteur d’expansion final et du système d’imagerie utilisé. Après le deuxième processus d’...
Discussion
La microscopie à expansion ultrastructurale est une technique qui permet d’obtenir des images à haute résolution d’échantillons biologiques en les élargissant physiquement à plusieurs fois leur taille d’origine. Le protocole U-ExM implique plusieurs étapes critiques qui doivent être exécutées avec soin pour obtenir des résultats optimaux4. Tout d’abord, l’échantillon doit être fixé avec un agent CP et incorporé dans une matrice d’hydroge...
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts à divulguer.
Remerciements
Nous remercions Dolores Campos pour son aide à la culture cellulaire Vero et Romina Manarin pour son aide à la culture de T. cruzi . Ce travail a été soutenu par l’Agencia Nacional de Promoción Científica y Tecnológica, le Ministerio de Ciencia e Innovación Productiva de l’Argentine (PICT2019-0526), le Consejo Nacional de Investigaciones Científicas y Técnicas (PIBAA 1242) et le Conseil de la recherche du Royaume-Uni [MR/P027989/1].
matériels
| Name | Company | Catalog Number | Comments |
| 0.22 micrometers sterile syringe filters PES | Membrane solutions | SFPES030022S | |
| 1 L beaker | Schott Duran | 10005227 | |
| 1.5-mL SPINWIN Micro Centrifuge Tube | Tarson | T38-500010 | |
| 10 mL disposable sterile serynge | NP | 66-32 | |
| 10 mL serological pipette sterile | Jet Biofil | GSP211010 | |
| 12-mm coverslips | Merienfeld GmbH | 01 115 20 | Round coverslips |
| 12-well plates | Jet Biofil | TCP011012 | |
| 22-mm coverslips | Corning | 2845-22 | Square coverslips |
| 24-well plates | Jet Biofil | TCP-011-024 | |
| 250 mL beaker | Schott Duran | C108.1 | |
| 3 mL Pasteur pipette | Deltalab | 200037 | |
| 35-mm glass bottom dishes | Matsunami glass ind | D11130H | |
| 4′,6-Diamidine-2′-phenylindole dihydrochloride | Sigma Aldrich | D9542 | DAPI |
| 5 ml serological pipette sterile | Jet Biofil | GSP010005 | |
| 6-well plates | Sarstedt | 83.3920 | |
| Acrilamide | BioRad | 1610101 | |
| Ammonium persulfate | Sigma Aldrich | A3678-25G | APS |
| ATTO 647 NHS ester | BOC Sciences | F10-0107 | For pan-proteome labelling |
| Biosafty Cabinet | Telstar | Bio II A/P | |
| Bovine Sodium Albumine | Sigma Aldrich | A7906 | BSA |
| CO2 Incubator | Sanyo | MCO-15A | |
| Confocal Microscope | Zeiss | LSM 880 | |
| Disposable Petridish | Tarsons | 460095 | 90 mm diameter |
| DMEM, High Glucose | Thermo Fisher Cientific | 12100046 | Powder |
| Electronic digital caliper | Radar | RADAR-SLIDE-CALIPER | |
| Ethanol Absolute | Supelco | 1,00,98,31,000 | |
| Fetal Calf Serum | Internegocios SA | FCS FRA 500 | Sterile and heat-inactivated |
| Fiji image processing package | ImageJ | doi:10.1038/nmeth.2019 | |
| Formaldehyde 37% | Sigma Aldrich | F8775 | FA |
| Glass Petridish | Marienfeld Superior | PM-3400300 | 60 mm diameter |
| Glucosa D(+) | Cicarelli | 716214 | |
| Glutaraldehyde 70% | Sigma Aldrich | G7776 | |
| Goat anti-Mouse IgG Secondary Antibody Alexa Fluor 555 | Invitrogen | A-21422 | |
| Goat anti-Rabbit IgG Secondary Antibody FICT | Jackson Immunoresearch | 115-095-003 | |
| Graduated cylinder | Nalgene | 3663-1000 | |
| Graduated glass flask | Glassco | GL-274.202.01 | 100 mL |
| Heating Block | IBR | Made in house | |
| Hemin | Frontier Scientific | H651-9 | |
| Hydrochloric acid 36.8-38.0% | Ciccarelli | 918110 | |
| Ice bucket | Corning | 1167U68 | |
| Incubator | Tecno Dalvo | TOC130 | |
| Liver Infusion | Difco | 226920 | |
| Magnetic stirrer and heater | Lab companion | HP-3000 | |
| Metal spatula | SALTTECH | 200MM | |
| Metal tweezers | Marienfeld Superior | PM-6633002 | |
| Methanol absolut | Cicarelli | 897110 | |
| Microcentrifuge tube 1.5 mL | Tarson | 500010-N | |
| Microscopy grade paper KimWipes | Kimtech Science | B0013HT2QW | |
| Milli-Q water sistem | Merk Millipore | IQ-7003 | |
| mouse anti- alpha tubulin clone DM1A | Sigma Aldrich | T9026 | |
| mouse anti-PFR | Purified antibodies | Donated by Dr. Ariel Silber (USP) | |
| N,N´-methylenbisacrilamide | ICN | 193997 | BIS |
| Na2HPO4 | Cicarelli | 834214 | |
| Neubauer chamber | Boeco | BOE 01 | |
| p1000 pipette | Gilson | PIPETMAN P1000 | |
| p1000 pipette tips | Tarson | TAR-521020B | |
| p20 pipette | Gilson | PIPETMAN P20 | |
| p20 pipette tips | Tarson | TAR-527108 | |
| p200 pipette | Gilson | PIPETMAN P200 | |
| p200 pipette tips | Tarson | TAR-521010Y | |
| Paraformaldehyde | Sigma Aldrich | P6148 | PFA |
| pH / ORP / °C meter | HANNA Instruments | HI 2211 | |
| Poly-D-Lysine 0.1% | Sigma Aldrich | P8920 | |
| Potassium Chloride | Cicarelli | 867212 | KCl |
| Razor blade | Printex | BS 2982:1992 | |
| Sealing FIlm "Parafilm M" | Bemis | PM996 | |
| Sodium Acrilate | Sigma Aldrich | 408220-25G | SA |
| Sodium Bicarbonate | Cicarelli | 929211 | NaHCO3 |
| Sodium Chloride | Cicarelli | 750214 | NaCl |
| Sodium Dodecyl Sulfate | BioRad | 1610302 | SDS |
| Sodium Hidroxide | Merk | 1-06498 | NaOH |
| Sorvall ST 16 Centrifuge | Thermo Fisher Scientific | 75004380 | |
| T-25 flasks | Corning | 430639 | |
| TEMED | Invitrogen | 15524-010 | |
| Tissue paper | Elite | ||
| Triptose | Merck | 1106760500 | |
| Tris | BioRad | 1610719 | |
| Tween-20 | Biopack | 2003-07 | Polysorbate 20 |
| Vaccum pump | Silfab | N33-A | |
| Vero cells | ATCC | CRL-1587 | |
| Vortex MIxer | Dragon Lab | MX-S |
Références
- Chen, F., Tillberg, P. W., Boyden, E. S. Expansion microscopy. Science. 347 (6221), 543-548 (2015).
- Chozinski, T. J., et al. Expansion microscopy with conventional antibodies and fluorescent proteins. Nature Methods. 13 (6), 485-488 (2016).
- Ku, T., et al. Multiplexed and scalable super-resolution imaging of three-dimensional protein localization in size-adjustable tissues. Nature Biotechnology. 34 (9), 973-981 (2016).
- Gambarotto, D., et al. Imaging cellular ultrastructures using expansion microscopy (U-ExM). Nature Methods. 16 (1), 71-74 (2018).
- Tillberg, P. W., Chen, F. Expansion microscopy: Scalable and convenient super-resolution microscopy. Annual Review of Cell and Developmental Biology. 35, 683-701 (2019).
- Jurriens, D., van Batenburg, V., Katrukha, E. A., Kapitein, L. C. Mapping the neuronal cytoskeleton using expansion microscopy. Methods in Cell Biology. 161, 105-124 (2021).
- Alonso, V. L. Ultrastructure expansion microscopy (U-ExM) in Trypanosoma cruzi: localization of tubulin isoforms and isotypes. Parasitology Research. 121 (10), 3019-3024 (2022).
- Kalichava, A., Ochsenreiter, T. Ultrastructure expansion microscopy in Trypanosoma brucei. Open Biology. 11 (10), 210132 (2021).
- Amodeo, S., et al. Characterization of the novel mitochondrial genome segregation factor TAP110 in Trypanosoma brucei. Journal of Cell Science. 134 (5), (2021).
- Liffner, B., Absalon, S. Expansion microscopy reveals plasmodium falciparum blood-stage parasites undergo anaphase with a chromatin bridge in the absence of mini-chromosome maintenance complex binding protein. Microorganisms. 9 (11), 2306 (2021).
- Dos Santos Pacheco, N., Soldati-Favre, D. Coupling auxin-inducible degron system with ultrastructure expansion microscopy to accelerate the discovery of gene function in Toxoplasma gondii. Methods in Molecular Biology. 2369, 121-137 (2021).
- Lidani, K. C. F., et al. Chagas disease: From discovery to a worldwide health problem. Frontiers in Public Health. 7, 166 (2019).
- Carrea, A., Diambra, L. Systems biology approach to model the life cycle of Trypanosoma cruzi. PloS One. 11 (1), e0146947 (2016).
- Vidal, J. C., De Souza, W. . Morphological and Functional Aspects of Cytoskeleton of Trypanosomatids. Cytoskeleton - Structure, Dynamics, Function and Disease. , (2017).
- Kunz, T. C., Götz, R., Gao, S., Sauer, M., Kozjak-Pavlovic, V. Using expansion microscopy to visualize and characterize the morphology of mitochondrial cristae. Frontiers in Cell and Developmental Biology. 8, 617 (2020).
- Laporte, M. H., Klena, N., Hamel, V., Guichard, P. Visualizing the native cellular organization by coupling cryofixation with expansion microscopy (Cryo-ExM). Nature Methods. 19 (2), 216-222 (2022).
- Sahabandu, N., et al. Expansion microscopy for the analysis of centrioles and cilia. Journal of Microscopy. 276 (3), 145-159 (2019).
- Wen, G., Leen, V., Rohand, T., Sauer, M., Hofkens, J. Current progress in expansion microscopy: chemical strategies and applications. Chemical Reviews. 123 (6), 3299-3323 (2023).
- Trinks, N., et al. Subdiffraction-resolution fluorescence imaging of immunological synapse formation between NK cells and A. fumigatus by expansion microscopy. Communications Biology. 4 (1), 1151 (2021).
- White, B. M., Kumar, P., Conwell, A. N., Wu, K., Baskin, J. M. Lipid expansion microscopy. Journal of the American Chemical Society. 144 (40), 18212-18217 (2022).
- Steib, E., et al. TissUExM enables quantitative ultrastructural analysis in whole vertebrate embryos by expansion microscopy. Cell Reports Methods. 2 (10), 100311 (2022).
- Shaib, A. H., et al. Visualizing proteins by expansion microscopy. bioRxiv. , (2023).
- M'Saad, O., et al. All-optical visualization of specific molecules in the ultrastructural context of brain tissue. bioRxiv. , (2022).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon