É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Microscopia de Expansão Ultraestrutural em Três Estágios do Ciclo de Vida In Vitro do Trypanosoma cruzi
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Este estudo apresenta um protocolo detalhado para a realização de microscopia de expansão ultraestrutural em três estágios do ciclo de vida in vitro do Trypanosoma cruzi, o patógeno responsável pela doença de Chagas. Incluímos a técnica otimizada para proteínas do citoesqueleto e marcação de pan-proteoma.
Resumo
Descrevemos aqui a aplicação da microscopia de expansão de ultraestrutura (U-ExM) em Trypanosoma cruzi, uma técnica que permite aumentar a resolução espacial de uma célula ou tecido para imagens microscópicas. Isso é feito expandindo fisicamente uma amostra com produtos químicos prontos para uso e equipamentos de laboratório comuns.
A doença de Chagas é um problema de saúde pública generalizado e premente causado pelo T. cruzi. A doença é prevalente na América Latina e tornou-se um problema significativo em regiões não endêmicas devido ao aumento da migração. A transmissão do T. cruzi ocorre por meio de insetos vetores hematófagos pertencentes às famílias Reduviidae e Hemiptera. Após a infecção, as amastigotas de T. cruzi se multiplicam dentro do hospedeiro mamífero e se diferenciam em tripomastigotas, a forma não replicativa da corrente sanguínea. No inseto vetor, os tripomastigotas se transformam em epimastigotas e proliferam por fissão binária. A diferenciação entre os estágios do ciclo de vida requer um extenso rearranjo do citoesqueleto e pode ser recriada em laboratório completamente usando diferentes técnicas de cultura de células.
Descrevemos aqui um protocolo detalhado para a aplicação de U-ExM em três estágios do ciclo de vida in vitro de Trypanosoma cruzi, com foco na otimização da imunolocalização de proteínas do citoesqueleto. Também otimizamos o uso do éster N-hidroxisuccinimida (NHS), um marcador de pan-proteoma que nos permitiu marcar diferentes estruturas do parasita.
Introdução
A microscopia de expansão (ExM) foi descrita pela primeira vez em 2015 por Boyden et al.1. É um protocolo de imagem com o qual um microscópio convencional pode atingir uma resolução espacial abaixo do limite de difração. Essa resolução mais alta é obtida devido a um aumento físico da amostra. Para conseguir isso, moléculas marcadas com fluorescência são reticuladas a um hidrogel, que é posteriormente expandido isotropicamente com água. Como resultado dessa expansão, os sinais são separados quase isotropicamente em todas as três dimensões. Este método emprega produtos químicos de baixo custo e permite uma resolução espacial de aproximadamente 65 nm usando microscópios convencionais (confocais), o que é aproximadamente quatro vezes melhor do que a resolução padrão de um microscópio confocal (aproximadamente 250 nm)1.
O próximo marco, que possibilitou o uso da microscopia de expansão em muitos campos biológicos, foi a adaptação da marcação por imunofluorescência com anticorpos convencionais2. Outra adaptação do protocolo ExM publicado inicialmente é a análise ampliada do proteoma (MAP)3. Este método introduziu o uso de altas concentrações de acrilamida e paraformaldeído antes da imersão da amostra no hidrogel para evitar a reticulação intra e interproteica, o que levou a uma melhor preservação do conteúdo proteico e da arquitetura subcelular das amostras. Este protocolo alternativo foi otimizado para obter maior conservação da ultraestrutura geral de organelas isoladas, utilizando concentrações mais baixas dos agentes fixadores (formaldeído/paraformaldeído e acrilamida); essa abordagem foi denominada microscopia de expansão de ultraestrutura (U-ExM)4.
Para obter ainda mais resolução, a combinação de ExM com técnicas de microscopia de super-resolução, incluindo microscopia de depleção de emissão estimulada ou microscopia de localização de molécula única, também foi relatada para atingir resoluções abaixo de 20 nm5.
O uso de ExM tem sido amplamente relatado nos campos da neurociência e da pesquisa do citoesqueleto6, mas apenas alguns estudos foram realizados em protistas parasitários. Nosso laboratório foi o primeiro a relatar a aplicação de U-ExM em T. cruzi7. O protocolo de fundação é baseado principalmente nos relatórios anteriores do U-ExM em Toxoplasma gondii, Plasmodium ssp. e Trypanosoma brucei 8,9,10,11.
Uma das maiores vantagens do ExM é sua natureza modular, que permite grande flexibilidade para se adaptar a diferentes amostras biológicas. O protocolo pode ser dividido em etapas (como fixação, prevenção de reticulação ou gelificação) que podem ser facilmente ajustadas pelo usuário para atender aos seus requisitos experimentais. Além disso, esse pipeline pode ser modificado para aumentar a compatibilidade com o organismo modelo ou para obter uma resolução específica. Como resultado, o ExM oferece um tremendo potencial para sistemas ópticos avançados e não avançados, garantindo aplicações mais amplas no futuro.
A doença de Chagas, também chamada de tripanossomíase americana, é uma doença endêmica na América Latina causada pelo Trypanosoma cruzi, um protozoário parasita. O ciclo de vida do parasita é complexo e envolve dois estágios de desenvolvimento em mamíferos e dois no inseto hospedeiro (membros da família Triatominidae), que é o vetor biológico dessa doença. A doença de Chagas pertence ao grupo de doenças tropicais negligenciadas listadas pela Organização Mundial da Saúde e representa um importante problema econômico e social na América Latina. Estudos epidemiológicos estimam que 8 milhões de pessoas em todo o mundo vivem com a doença de Chagas e mais de 10.000 mortes por ano. Esses números exemplificam a importância da doença de Chagas como um problema de saúde pública em todo o mundo. A distribuição geográfica da doença de Chagas mudou nas últimas décadas, com muitos indivíduos infectados residindo em grandes áreas urbanas em todo o mundo devido ao aumento das migrações, em oposição às áreas principalmente rurais da América Latina, onde foi originalmente encontrada12.
Os estágios de desenvolvimento do T. cruzi diferem ao longo de seu ciclo de vida, que pode ser replicado completamente in vitro. Os epimastigotas são formas replicativas no inseto vetor e possuem um núcleo esférico na região central do corpo celular e um cinetoplasto em forma de barra (uma estrutura contendo DNA mitocondrial exclusiva dos cinetoplastídeos) na região anterior em relação ao núcleo, com um flagelo livre. Os tripomastigotas são a forma infecciosa, não replicativa, e possuem um núcleo alongado, um cinetoplasto posterior arredondado e um flagelo ligado à membrana plasmática ao longo de todo o comprimento do parasita. As amastigotas são a forma replicativa intracelular; Eles têm um núcleo na região central, um cinetoplasto em forma de bastonete na parte anterior do corpo celular e um flagelo reduzido. A adaptabilidade do parasita a diferentes ambientes é um reflexo dessas variações morfológicas. Vale ressaltar também que esse ciclo de vida envolve divisão simétrica e diferentes estágios de desenvolvimento transicional13. Durante a diferenciação, o citoesqueleto dos tripanossomatídeos desempenha um papel crítico. Essa estrutura é formada por um espartilho de microtúbulos subpeliculares dispostos em uma matriz ordenada de microtúbulos estáveis abaixo da membrana plasmática. Além disso, um bastonete paraflagelar está presente nesses organismos, que é uma estrutura semelhante a uma treliça que corre paralela e está ligada ao axonema flagelar14. A organização precisa do citoesqueleto e as mudanças estruturais nucleares ao longo dos estágios do ciclo celular envolvem mecanismos únicos de regulação gênica específicos para tripanossomatídeos, tornando-os modelos interessantes para estudos de biologia celular.
Dado o pequeno tamanho do T. cruzi e de outros protozoários parasitos, o U-ExM apresenta uma excelente ferramenta para analisar as características estruturais desses importantes patógenos. Como mencionado anteriormente, a aplicabilidade dessa técnica no T. cruzi foi validada pela primeira vez pelo Dr. Alonso7. Este relato detalha um protocolo U-ExM completo, com ênfase na imunolocalização de proteínas do citoesqueleto durante as diferentes fases do ciclo de vida do T. cruzi. Além disso, otimizamos o uso do éster N-hidroxisuccinimida (NHS), um marcador de pan-proteoma que nos permite marcar várias estruturas de parasitas. Além disso, é descrita uma metodologia in vitro para obter os três estágios do parasita.
Protocolo
NOTA: A Figura 1 ilustra o desenho experimental completo.
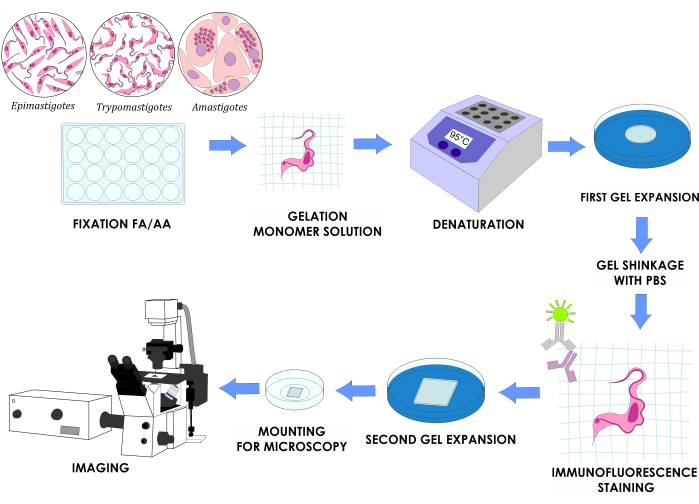
Figura 1: Fluxo de trabalho U-ExM para três estágios do ciclo de vida in vitro do T. cruzi. Clique aqui para ver uma versão maior desta figura.
1. Preparação das lamínulas revestidas com poli-D-lisina
- Coloque um quadrado de 10 cm x 10 cm de filme de vedação em uma placa de Petri. Lavar as lamínulas banhando-as em etanol absoluto numa placa de Petri de vidro de 35 mm.
- Retire as lamínulas com uma pinça do banho de etanol e escorra o excesso de líquido com papel de seda. Coloque as lamínulas sobre a película de vedação.
- Absorva o restante do etanol com papel para microscopia. Adicione uma solução de poli-D-lisina a 0,1% v/v no centro da lamínula e espalhe-a com a ponta para cobrir aproximadamente 80% de sua superfície. Fechar a placa de Petri e incubar durante 1 h a 37 °C.
NOTA: Para lamínulasde 22 mm 2, use 200 μL de solução de poli-D-lisina; para lamínulas redondas de 12 mm, utilizar 100 μL de solução de poli-D-lisina. - Lave as lamínulas com água ultrapura três vezes. Use aspiração com vácuo para remover a água entre cada lavagem. Conservar a 4 °C até 1 semana.
2. Preparação da solução
- Prepare soluções estoque de acrilato de sódio (SA) a 38% (p / p).
- Adicione lentamente 19 g de SA a 31 mL de água livre de nuclease enquanto agita.
NOTA: Esta solução é muito viscosa; Preste atenção ao pipetar. - Assim que o SA estiver completamente dissolvido, armazene-o em um recipiente estéril. Mantenha-o a 4 °C e troque-o a cada 6 meses.
NOTA: A SA às vezes mostra sinais de contaminação com poliacrilato de sódio dependendo da marca, o que é perceptível ao preparar a solução estoque, pois ela fica amarelada e turva. Use-o com cautela se for o caso.
- Adicione lentamente 19 g de SA a 31 mL de água livre de nuclease enquanto agita.
- Preparar uma solução-mãe de acrilamida (AA) a 40% (p/v) e uma solução-mãe de N, N'-metileno bissacrilamida (BIS) a 2%. Dissolva cada composto em água ultrapura e filtre com um filtro de seringa estéril de 0,22 μm. Conservar num recipiente esterilizado a 4 °C.
CUIDADO: AA e BIS são substâncias altamente tóxicas. Trabalhe sob uma hotte e use elementos de proteção adequados (luvas, roupas de proteção, máscara e óculos de segurança). - Prepare a solução de prevenção de reticulação de proteínas (CP) misturando 38 μL de uma solução de formaldeído (FA) a 37% com 50 μL da solução estoque de acrilamida a 40% (etapa 2.2) em 912 μL de solução salina tamponada com fosfato (PBS; Tabela 1) para obter uma concentração final de 1,4% de formaldeído e 2% de acrilamida.
NOTA: Sempre prepare a solução CP na hora e seja extremamente preciso ao pipetar. Por exemplo, use uma pipeta P1000 para receber 900 μL de PBS e uma pipeta P20 para retirar os 12 μL restantes de PBS. - Prepare a solução monomérica
NOTA: Não use a solução imediatamente após ser preparada; Conservar a -20 °C pelo menos 24 h antes de ser utilizado.- Misture 500 μL da solução de SA a 38% (etapa 2.1), 250 μL da solução de AA a 40% e 50 μL da solução de BIS (etapa 2.2), bem como 100 μL de PBS 10x (Tabela 1).
- Alíquota este volume final de 900 μL em 10 tubos de microcentrífuga de 1,5 mL com 90 μL de solução monomérica cada. Conservar a -20 °C até 2 semanas.
NOTA: A solução SA pode permanecer na ponta durante a pipetagem porque é muito viscosa; Tenha cuidado e dispense tudo.
- Prepare a solução desnaturante
- Misture 114,3 mL de uma solução de dodecil sulfato de sódio (SDS) a 350 mM preparada em água ultrapura com 10 mL de uma solução de cloreto de sódio a 4 M preparada em água ultrapura. Adicione 12 g de Tris mexendo num copo de 250 ml.
CUIDADO: SDS é altamente tóxico; Use-o sob um exaustor usando luvas, roupas de proteção, máscara e óculos de segurança. - Ajustar o pH a 9 com uma solução concentrada de ácido clorídrico. Perfazer o volume de 200 ml com água ultrapura e conservar num balão estéril a 4 °C.
- Misture 114,3 mL de uma solução de dodecil sulfato de sódio (SDS) a 350 mM preparada em água ultrapura com 10 mL de uma solução de cloreto de sódio a 4 M preparada em água ultrapura. Adicione 12 g de Tris mexendo num copo de 250 ml.
- Prepare uma solução de persulfato de amônio a 10% (APS) e uma solução TEMED a 10%.
- Dissolva 0,1 g de APS em 1 mL de água ultrapura.
- Prepare 1 mL de uma solução TEMED a 10% em água ultrapura.
- Prepare alíquotas de 100 μL de ambas as soluções em tubos estéreis de microcentrífuga de 1,5 mL e armazene-os a -20 ° C por até 1 mês.
- Preparar a solução de paraformaldeído.
- Dissolva 2 g de paraformaldeído em 40 ml de PBS, agitando a 60 °C. Adicione 1 M NaOH gota a gota até que a solução passe de branca para incolor.
CUIDADO: O formaldeído é altamente tóxico; Use-o sob um exaustor usando luvas, roupas de proteção, máscara e óculos de segurança. - Resfrie a solução à temperatura ambiente e ajuste o pH com NaOH para 7,2 em um volume final de 50 mL. Filtrar com um filtro de seringa estéril de 0,22 μm e conservar num recipiente estéril.
- Preparar alíquotas de 1 ml em tubos de microcentrífuga estéreis de 1,5 ml e conservar a -20 °C.
- Dissolva 2 g de paraformaldeído em 40 ml de PBS, agitando a 60 °C. Adicione 1 M NaOH gota a gota até que a solução passe de branca para incolor.
- Prepare a solução de paraformaldeído/glutaraldeído.
- Adicione 0,2 g de paraformaldeído em 3,5 mL de água ultrapura e 50 μL de solução de NaOH 16 M. Aquecer a solução a 60 °C para dissolver o paraformaldeído.
- Esfrie e adicione 300 μL de glutaraldeído a 70%. Aumente o volume para 5 mL com água ultrapura e, finalmente, para 10 mL com PBS.
CUIDADO: O paraformaldeído e o glutaraldeído são altamente tóxicos; Use-os sob um exaustor usando luvas, roupas de proteção, máscara e óculos de segurança. - Preparar alíquotas de 1 ml em tubos de microcentrífuga estéreis de 1,5 ml e conservar a -20 °C.
3. Preparação das culturas de parasitas
- Cultive epimastigotas de T. cruzi .
- Utilizar uma cultura axénica num balão T-25 (25 cm2 de área de crescimento) e manter as culturas na fase logarítmica subcultivando a cada 48-72 h em meio de triptose para perfusão hepática (LIT) com 10% de soro fetal de vitelo (FCS; Tabela 1).
- Certifique-se de que a tampa está bem fechada e mantenha o frasco de cultura na vertical a 28 °C para incubação. Monitore o crescimento do parasita por contagem de células em uma câmara de Neubauer durante cada subcultura.
NOTA: Para a cepa Dm28c usada neste estudo, a concentração de epimastigotas em culturas de fase logarítmica está entre 1-5 x 107 parasitas/mL. - Prepare uma suspensão de 2 x 106 epimastigotas / mL de uma cultura em fase logarítmica em meio LIT suplementado com 10% de FCS. Centrifugue a suspensão a 5.000 x g por 10 min em temperatura ambiente (RT). Lave com PBS uma ou duas vezes e ressuspenda em 200 μL de PBS.
- Aderir à lamínula redonda de 12 mm previamente revestida com poli-D-lisina (ponto 1). Incube em RT por 15-20 min. Continue para a etapa de prevenção de reticulação (seção 4).
NOTA: Alternativamente, fixe os epimastigotas com metanol frio por 7 min ou solução de paraformaldeído/glutaraldeído (etapa 2.8) por 10 min em RT antes da aderência dos parasitas à lamínula. É possível armazenar os parasitas fixos a 4 °C por até 1 semana.
- Obtenha as amastigotas de células Vero infectadas.
- Coloque uma lamínula redonda estéril de 12 mm no fundo de uma placa de cultura de tecidos de 24 poços. Prepare uma suspensão de 2 x 105 células Vero / mL em meio Eagle modificado de Dulbecco (DMEM) suplementado com 2% de soro fetal de bezerro (FCS). Semear 500 μL da suspensão por alvéolo.
NOTA: Este estudo usa DMEM com 2% FCS para permitir que as células cresçam mais lentamente. - Incubar durante a noite (ON; 12-16 h) a 37 °C e 5% de CO2 para garantir a fixação das células.
- Após a incubação, lave as células duas vezes usando 500 μL de PBS estéril. Adicionar tripomastigotas de T. cruzi nas células a uma multiplicidade de infecção (MOI) de 10 em 100 μL de DMEM com 2% de FCS por poço, correspondendo a um milhão de tripomastigotas por poço. Incubar a 37 °C e 5% de CO2 por 6 h.
- Após a incubação, enxágue as placas duas vezes com PBS. Adicione 500 μL de DMEM suplementado com 2% de FCS. Neste ponto, continue para a etapa de prevenção de reticulação (seção 4).
NOTA: As amastigotas intracitoplasmáticas serão visíveis através de um microscópio óptico invertido 2 dias após a infecção (Figura Suplementar 1). Alternativamente, fixe os tripomastigotas com metanol frio por 7 min a -20 °C, ou paraformaldeído/glutaraldeído por 10 min em RT, antes da aderência dos parasitas à lamínula.
- Coloque uma lamínula redonda estéril de 12 mm no fundo de uma placa de cultura de tecidos de 24 poços. Prepare uma suspensão de 2 x 105 células Vero / mL em meio Eagle modificado de Dulbecco (DMEM) suplementado com 2% de soro fetal de bezerro (FCS). Semear 500 μL da suspensão por alvéolo.
- Obtenha tripomastigotas de células Vero infectadas.
- Colete o sobrenadante de uma monocamada de células Vero (30% -40% de confluência) infectada com tripomastigotas (MOI 1:10; ON incubação) 4 dias após a infecção.
NOTA: Para um frasco T-25, a concentração inicial de células Vero usadas é de 800.000 células, e para um frasco T-75 é de dois milhões de células. - Determinar a concentração de tripomastigotas utilizando uma câmara de Neubauer para a contagem de células. Centrifugar 4 x 106 tripomastigotas a 7.000 x g em RT durante 10 min.
- Enxágue com PBS duas vezes e ressuspenda em 200 μL de PBS. Aplicar sobre uma lamínula redonda de 12 mm revestida com poli-D-lisina (secção 1). Incube por 10-15 min em RT. Prossiga com a etapa de prevenção de reticulação (seção 4).
NOTA: Alternativamente, fixe os tripomastigotas com metanol frio por 7 min a -20 ° C ou paraformaldeído / glutaraldeído por 10 min em RT antes da aderência dos parasitas à lamínula.
- Colete o sobrenadante de uma monocamada de células Vero (30% -40% de confluência) infectada com tripomastigotas (MOI 1:10; ON incubação) 4 dias após a infecção.
4. Realizando a prevenção de reticulação (DIA 1)
- Mergulhe a lamínula de 12 mm com os parasitas aderidos ou células infectadas (voltadas para cima) em uma placa de 24 poços com 0,5 mL de solução de CP (etapa 2.3) em cada poço.
- Encha os poços vazios com água para reduzir a evaporação. Sele a placa com uma película de vedação. Incubar durante 5 h a 37 °C. Esta etapa pode ser estendida até uma incubação ON a 4 °C.
NOTA: Sempre mergulhe a lamínula na solução; Não pipete a solução fixadora sobre as lamínulas.
5. Realização de gelificação da amostra
- Monte uma câmara úmida em uma placa de Petri com um filme de vedação sobre o papel de seda (Figura 2A). Adicione água ao lenço de papel e incube a -20 °C por 20 min para esfriar. Descongele uma alíquota TEMED e APS no gelo por 20 min (etapa 2.6).
NOTA: Não congele e descongele o APS mais de três vezes. - Coloque a câmara fria e úmida no gelo (Figura 2A). Retire da incubadora a placa de 24 poços preparada na seção 4. Aspire a solução de CP com uma pipeta Pasteur de 3 mL, deixando alguma solução, caso contrário, as lamínulas serão difíceis de remover.
- Retirar as lamínulas de 12 mm da solução de fixação com uma pinça e colocá-las sobre papel de seda com os parasitas virados para cima.
NOTA: É útil usar uma agulha estéril para levantar a lamínula e segurá-la com a pinça. - A uma alíquota de 90 μL de solução monomérica (etapa 2.4), adicione 5 μL de TEMED e 5 μL de APS previamente descongelado. Misture com um misturador de vórtice por não mais que 2-3 s; Não é necessário fechar o tubo com a tampa.
NOTA: Sempre adicione TEMED primeiro e APS por último. A adição de APS primeiro e TEMED por último à solução monomérica torna o processo de gelificação mais rápido, não dando tempo para manipulá-lo. - Faça rapidamente uma gota de 35 μL sobre o filme de vedação da câmara úmida para cada lamínula. Pegue imediatamente a lamínula com uma pinça e coloque sobre a gota (Figura 2B) com os parasitas voltados para baixo.
NOTA: Faça no máximo duas lamínulas de cada vez; É crucial não atrasar esta etapa porque a solução polimeriza muito rapidamente. - Incubar a câmara húmida durante 5 min em gelo e depois durante 1 h a 37 °C. Ligue um bloco de aquecimento a 95 °C para garantir a temperatura correta para a próxima etapa.
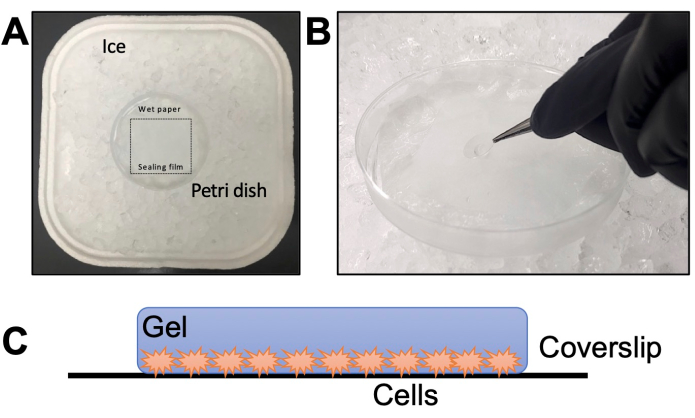
Figura 2: Detalhes da etapa de gelificação. (A) Montagem da câmara úmida. (B) Deixar cair as lamínulas na solução de monômero com TEMED e APS para gelificação. (C) Representação esquemática do gel montado para imagem. Clique aqui para ver uma versão maior desta figura.
6. Desnaturação das amostras gelificadas e realização da expansão isotrópica
- Retirar a solução de desnaturação (passo 2.5) a 4 °C. Se estiver precipitado, coloque-o em banho-maria quente até que se dissolva completamente.
- Adicione 2 mL da solução desnaturante a cada poço de uma placa de 6 poços. Transferir as lamínulas do passo 5.3 para a placa com a solução de desnaturação. Incube por 15 min em RT com agitação suave para que o gel se desprenda da lamínula.
- Transfira cuidadosamente o gel (retirando-o da lamínula de 12 mm) com uma espátula de metal para um tubo de microcentrífuga estéril de 1,5 mL com 1 mL de solução desnaturante. Use travas de tampa para prender os tubos. Incubar durante 1 h e 30 min a 95 °C num bloco de aquecimento.
NOTA: Os géis começam a se expandir durante esta etapa; seja gentil ao transferir o gel para o tubo de microcentrífuga de 1,5 mL.
CUIDADO: Após a incubação, o tubo que contém o gel está a 95 °C, o que pode ser perigoso. Use luvas de proteção e deixe os tubos esfriarem antes de manuseá-los para evitar queimaduras e projeções. - Execute a primeira rodada de expansão
- Aspirar a solução desnaturante do tubo de microcentrífuga de 1,5 ml com o gel com uma pipeta P1000. Transfira o gel do tubo de microcentrífuga de 1,5 mL para uma placa de Petri com 10 mL de água ultrapura por 30 min usando uma pequena espátula.
- Troque a água ultrapura usando uma pipeta Pasteur descartável de 3 mL. Incube ON em RT.
NOTA: Seja gentil com os géis porque, após os primeiros 30 minutos de incubação, eles se tornaram frágeis. - Troque a água ultrapura usando uma pipeta Pasteur descartável de 3 mL mais uma vez.
NOTA: Três incubações de água de 30 min cada são suficientes, mas por praticidade, é melhor deixar a segunda incubação LIGADA.
7. Realização da marcação de fluorescência das proteínas-alvo (DIA 2)
- Retire a água da placa de Petri com o gel com uma pipeta Pasteur descartável de 3 mL. Meça o diâmetro do gel com um paquímetro para calcular a expansão (entre quatro e cinco vezes).
- Lave duas vezes com 10 mL de PBS por 15 min. Corte o gel com uma lâmina de barbear em quadrados de aproximadamente 10 mm x 10 mm no centro do gel circular. Use um quadrado por condição a ser testada.
NOTA: O gel encolhe após as incubações de PBS. Os géis podem ser armazenados em PBS a 4 °C por até 1 semana. - Transfira cada quadrado para uma placa de 12 poços e incube com 500 μL de anticorpo primário diluído em albumina de soro bovino PBS (BSA) a 2% por 2 h e 30 min a 37 ° C com agitação.
NOTA: A incubação de anticorpos alternativos pode ser realizada ON a 4 °C. O volume mínimo de anticorpos que pode ser usado é de 300 μL em uma placa de 24 poços. Como regra geral, use o dobro da concentração de anticorpos usados para imunomarcação convencional. - Lave três vezes com 2 mL de PBS com 0,1% de polissorbato 20 por 10 min agitando em uma placa de 6 poços.
- Transfira o gel para uma placa de 12 poços e incube com 500 μL de anticorpo secundário em PBS com DAPI e 10 μg / mL de éster NHS conjugado ao fluoróforo desejado por 2 h e 30 min a 37 ° C com agitação suave.
NOTA: Como regra geral, use o dobro da concentração de anticorpos usados para imunomarcação convencional. Alternativamente, esta incubação pode ser realizada ON a 4 °C. - Lave três vezes com 2 mL de PBS com 0,1% de polissorbato 20 por 10 min agitando em uma placa de 6 poços.
- Transfira o gel para uma placa de Petri com água ultrapura. Incubar por 30 min. Troque a água duas vezes com uma pipeta Pasteur descartável de 3 mL, como feito na etapa 6.4.
8. Imagem e processamento de imagem (DIA 3)
- Remova a água das placas de Petri com uma pipeta Pasteur descartável de 3 mL e meça o diâmetro do gel com um paquímetro para calcular o fator de expansão.
- Corte um pequeno pedaço de ~10 mm x 10 mm com uma lâmina de barbear e coloque-o em um prato com fundo de vidro de 35 mm.
NOTA: Como alternativa, coloque o gel entre uma lâmina e uma lamínula (sem poli-D-lisina). A lâmina deve ter duas peças menores cortadas com uma faca de diamante presa nas laterais para formar uma câmara com a espessura do gel. - Verifique a orientação com ampliação de 10x ou 20x. Para focar adequadamente, certifique-se de que os parasitas estejam voltados para a lamínula; caso contrário, é preciso virar o gel e verificar novamente (Figura 2C).
- Uma vez encontrada a orientação adequada, seque o gel restante com papel de microscopia. Cubra o gel com uma lamínula revestida de poli-D-lisina.
- Adicione uma pequena gota de água ultrapura ao gel para visualizar com um microscópio confocal.
NOTA: Para garantir uma imagem adequada, é crucial manter a orientação do gel durante todo o processo, de modo que o lado do gel que contém as células próximo à sua superfície fique voltado para o prato com fundo de vidro na extremidade. Ao usar placas de 35 mm, a adição de poli-D-lisina à lamínula colocada na parte inferior reduz o deslocamento do gel durante a aquisição da imagem. - Parâmetros de aquisição de imagem
- Visualize as amostras em um microscópio confocal (Tabela de Materiais) usando uma objetiva de imersão em óleo de 63x 1,4 abertura numérica (NA).
- Adquira pilhas Z, a largura de cada passo z e um tempo de exposição por pixel, a ser determinado empiricamente dependendo da amostra, da intensidade do sinal e da otimização dos tempos de aquisição. Use o zoom de varredura para uma ampliação eficaz, se desejar.
- Abra a pilha Z usando o software de processamento de imagem (Tabela de materiais). Para cada canal, agrupe as imagens empilhadas usando a opção Group Z-project . Selecione a projeção de intensidade máxima.
- Para mesclar imagens, use a ferramenta Mesclar canais e selecione a cor de cada canal. Adicione uma barra de escala usando a ferramenta Barra de escala no software de processamento.
NOTA: Alternativamente, pode-se colorir cada canal conforme desejado (veja exemplos nos resultados representativos).
Resultados
Se o protocolo tiver sido executado corretamente (Figura 1), as amostras serão visíveis como um gel planar e translúcido que pode ser expandido até um fator de 4-4,5x em água (Figura 3A). Essa expansão proporcionou uma resolução efetiva de cerca de 70 nm, que pode variar dependendo do fator de expansão final e do sistema de imagem empregado. Após o segundo processo de expansão e aquisição de imagens em microscópio ...
Discussão
A microscopia de expansão ultraestrutural é uma técnica que permite obter imagens de alta resolução de amostras biológicas, expandindo-as fisicamente para várias vezes seu tamanho original. O protocolo U-ExM envolve várias etapas críticas que devem ser cuidadosamente executadas para alcançar os melhores resultados4. Primeiro, a amostra deve ser fixada com um agente CP e incorporada em uma matriz de hidrogel expansível. O formaldeído presente na soluç?...
Divulgações
Os autores não têm conflitos de interesse a divulgar.
Agradecimentos
Agradecemos a Dolores Campos por auxiliar na cultura de células Vero e Romina Manarin por auxiliar na cultura de T. cruzi . Este trabalho foi apoiado pela Agencia Nacional de Promoción Científica y Tecnológica, Ministerio de Ciencia e Innovación Productiva da Argentina (PICT2019-0526), Consejo Nacional de Investigaciones Científicas y Técnicas (PIBAA 1242) e Research Council United Kingdom [MR / P027989 / 1].
Materiais
| Name | Company | Catalog Number | Comments |
| 0.22 micrometers sterile syringe filters PES | Membrane solutions | SFPES030022S | |
| 1 L beaker | Schott Duran | 10005227 | |
| 1.5-mL SPINWIN Micro Centrifuge Tube | Tarson | T38-500010 | |
| 10 mL disposable sterile serynge | NP | 66-32 | |
| 10 mL serological pipette sterile | Jet Biofil | GSP211010 | |
| 12-mm coverslips | Merienfeld GmbH | 01 115 20 | Round coverslips |
| 12-well plates | Jet Biofil | TCP011012 | |
| 22-mm coverslips | Corning | 2845-22 | Square coverslips |
| 24-well plates | Jet Biofil | TCP-011-024 | |
| 250 mL beaker | Schott Duran | C108.1 | |
| 3 mL Pasteur pipette | Deltalab | 200037 | |
| 35-mm glass bottom dishes | Matsunami glass ind | D11130H | |
| 4′,6-Diamidine-2′-phenylindole dihydrochloride | Sigma Aldrich | D9542 | DAPI |
| 5 ml serological pipette sterile | Jet Biofil | GSP010005 | |
| 6-well plates | Sarstedt | 83.3920 | |
| Acrilamide | BioRad | 1610101 | |
| Ammonium persulfate | Sigma Aldrich | A3678-25G | APS |
| ATTO 647 NHS ester | BOC Sciences | F10-0107 | For pan-proteome labelling |
| Biosafty Cabinet | Telstar | Bio II A/P | |
| Bovine Sodium Albumine | Sigma Aldrich | A7906 | BSA |
| CO2 Incubator | Sanyo | MCO-15A | |
| Confocal Microscope | Zeiss | LSM 880 | |
| Disposable Petridish | Tarsons | 460095 | 90 mm diameter |
| DMEM, High Glucose | Thermo Fisher Cientific | 12100046 | Powder |
| Electronic digital caliper | Radar | RADAR-SLIDE-CALIPER | |
| Ethanol Absolute | Supelco | 1,00,98,31,000 | |
| Fetal Calf Serum | Internegocios SA | FCS FRA 500 | Sterile and heat-inactivated |
| Fiji image processing package | ImageJ | doi:10.1038/nmeth.2019 | |
| Formaldehyde 37% | Sigma Aldrich | F8775 | FA |
| Glass Petridish | Marienfeld Superior | PM-3400300 | 60 mm diameter |
| Glucosa D(+) | Cicarelli | 716214 | |
| Glutaraldehyde 70% | Sigma Aldrich | G7776 | |
| Goat anti-Mouse IgG Secondary Antibody Alexa Fluor 555 | Invitrogen | A-21422 | |
| Goat anti-Rabbit IgG Secondary Antibody FICT | Jackson Immunoresearch | 115-095-003 | |
| Graduated cylinder | Nalgene | 3663-1000 | |
| Graduated glass flask | Glassco | GL-274.202.01 | 100 mL |
| Heating Block | IBR | Made in house | |
| Hemin | Frontier Scientific | H651-9 | |
| Hydrochloric acid 36.8-38.0% | Ciccarelli | 918110 | |
| Ice bucket | Corning | 1167U68 | |
| Incubator | Tecno Dalvo | TOC130 | |
| Liver Infusion | Difco | 226920 | |
| Magnetic stirrer and heater | Lab companion | HP-3000 | |
| Metal spatula | SALTTECH | 200MM | |
| Metal tweezers | Marienfeld Superior | PM-6633002 | |
| Methanol absolut | Cicarelli | 897110 | |
| Microcentrifuge tube 1.5 mL | Tarson | 500010-N | |
| Microscopy grade paper KimWipes | Kimtech Science | B0013HT2QW | |
| Milli-Q water sistem | Merk Millipore | IQ-7003 | |
| mouse anti- alpha tubulin clone DM1A | Sigma Aldrich | T9026 | |
| mouse anti-PFR | Purified antibodies | Donated by Dr. Ariel Silber (USP) | |
| N,N´-methylenbisacrilamide | ICN | 193997 | BIS |
| Na2HPO4 | Cicarelli | 834214 | |
| Neubauer chamber | Boeco | BOE 01 | |
| p1000 pipette | Gilson | PIPETMAN P1000 | |
| p1000 pipette tips | Tarson | TAR-521020B | |
| p20 pipette | Gilson | PIPETMAN P20 | |
| p20 pipette tips | Tarson | TAR-527108 | |
| p200 pipette | Gilson | PIPETMAN P200 | |
| p200 pipette tips | Tarson | TAR-521010Y | |
| Paraformaldehyde | Sigma Aldrich | P6148 | PFA |
| pH / ORP / °C meter | HANNA Instruments | HI 2211 | |
| Poly-D-Lysine 0.1% | Sigma Aldrich | P8920 | |
| Potassium Chloride | Cicarelli | 867212 | KCl |
| Razor blade | Printex | BS 2982:1992 | |
| Sealing FIlm "Parafilm M" | Bemis | PM996 | |
| Sodium Acrilate | Sigma Aldrich | 408220-25G | SA |
| Sodium Bicarbonate | Cicarelli | 929211 | NaHCO3 |
| Sodium Chloride | Cicarelli | 750214 | NaCl |
| Sodium Dodecyl Sulfate | BioRad | 1610302 | SDS |
| Sodium Hidroxide | Merk | 1-06498 | NaOH |
| Sorvall ST 16 Centrifuge | Thermo Fisher Scientific | 75004380 | |
| T-25 flasks | Corning | 430639 | |
| TEMED | Invitrogen | 15524-010 | |
| Tissue paper | Elite | ||
| Triptose | Merck | 1106760500 | |
| Tris | BioRad | 1610719 | |
| Tween-20 | Biopack | 2003-07 | Polysorbate 20 |
| Vaccum pump | Silfab | N33-A | |
| Vero cells | ATCC | CRL-1587 | |
| Vortex MIxer | Dragon Lab | MX-S |
Referências
- Chen, F., Tillberg, P. W., Boyden, E. S. Expansion microscopy. Science. 347 (6221), 543-548 (2015).
- Chozinski, T. J., et al. Expansion microscopy with conventional antibodies and fluorescent proteins. Nature Methods. 13 (6), 485-488 (2016).
- Ku, T., et al. Multiplexed and scalable super-resolution imaging of three-dimensional protein localization in size-adjustable tissues. Nature Biotechnology. 34 (9), 973-981 (2016).
- Gambarotto, D., et al. Imaging cellular ultrastructures using expansion microscopy (U-ExM). Nature Methods. 16 (1), 71-74 (2018).
- Tillberg, P. W., Chen, F. Expansion microscopy: Scalable and convenient super-resolution microscopy. Annual Review of Cell and Developmental Biology. 35, 683-701 (2019).
- Jurriens, D., van Batenburg, V., Katrukha, E. A., Kapitein, L. C. Mapping the neuronal cytoskeleton using expansion microscopy. Methods in Cell Biology. 161, 105-124 (2021).
- Alonso, V. L. Ultrastructure expansion microscopy (U-ExM) in Trypanosoma cruzi: localization of tubulin isoforms and isotypes. Parasitology Research. 121 (10), 3019-3024 (2022).
- Kalichava, A., Ochsenreiter, T. Ultrastructure expansion microscopy in Trypanosoma brucei. Open Biology. 11 (10), 210132 (2021).
- Amodeo, S., et al. Characterization of the novel mitochondrial genome segregation factor TAP110 in Trypanosoma brucei. Journal of Cell Science. 134 (5), (2021).
- Liffner, B., Absalon, S. Expansion microscopy reveals plasmodium falciparum blood-stage parasites undergo anaphase with a chromatin bridge in the absence of mini-chromosome maintenance complex binding protein. Microorganisms. 9 (11), 2306 (2021).
- Dos Santos Pacheco, N., Soldati-Favre, D. Coupling auxin-inducible degron system with ultrastructure expansion microscopy to accelerate the discovery of gene function in Toxoplasma gondii. Methods in Molecular Biology. 2369, 121-137 (2021).
- Lidani, K. C. F., et al. Chagas disease: From discovery to a worldwide health problem. Frontiers in Public Health. 7, 166 (2019).
- Carrea, A., Diambra, L. Systems biology approach to model the life cycle of Trypanosoma cruzi. PloS One. 11 (1), e0146947 (2016).
- Vidal, J. C., De Souza, W. . Morphological and Functional Aspects of Cytoskeleton of Trypanosomatids. Cytoskeleton - Structure, Dynamics, Function and Disease. , (2017).
- Kunz, T. C., Götz, R., Gao, S., Sauer, M., Kozjak-Pavlovic, V. Using expansion microscopy to visualize and characterize the morphology of mitochondrial cristae. Frontiers in Cell and Developmental Biology. 8, 617 (2020).
- Laporte, M. H., Klena, N., Hamel, V., Guichard, P. Visualizing the native cellular organization by coupling cryofixation with expansion microscopy (Cryo-ExM). Nature Methods. 19 (2), 216-222 (2022).
- Sahabandu, N., et al. Expansion microscopy for the analysis of centrioles and cilia. Journal of Microscopy. 276 (3), 145-159 (2019).
- Wen, G., Leen, V., Rohand, T., Sauer, M., Hofkens, J. Current progress in expansion microscopy: chemical strategies and applications. Chemical Reviews. 123 (6), 3299-3323 (2023).
- Trinks, N., et al. Subdiffraction-resolution fluorescence imaging of immunological synapse formation between NK cells and A. fumigatus by expansion microscopy. Communications Biology. 4 (1), 1151 (2021).
- White, B. M., Kumar, P., Conwell, A. N., Wu, K., Baskin, J. M. Lipid expansion microscopy. Journal of the American Chemical Society. 144 (40), 18212-18217 (2022).
- Steib, E., et al. TissUExM enables quantitative ultrastructural analysis in whole vertebrate embryos by expansion microscopy. Cell Reports Methods. 2 (10), 100311 (2022).
- Shaib, A. H., et al. Visualizing proteins by expansion microscopy. bioRxiv. , (2023).
- M'Saad, O., et al. All-optical visualization of specific molecules in the ultrastructural context of brain tissue. bioRxiv. , (2022).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados