Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Mikrokoloniebildender Unit-Assay zur Wirksamkeitsbewertung von Impfstoffen gegen Tuberkulose
In diesem Artikel
Zusammenfassung
Die Bestimmung koloniebildender Einheiten (KBE) ist die Goldstandard-Technik zur Quantifizierung von Bakterien, einschließlich Mycobacterium tuberculosis , die Wochen brauchen kann, um sichtbare Kolonien zu bilden. Hier beschreiben wir eine Mikrokbe für die KBE-Bestimmung mit erhöhter Zeiteffizienz, reduziertem Laborplatz und Reagenzienkosten sowie Skalierbarkeit auf Experimente mit mittlerem und hohem Durchsatz.
Zusammenfassung
Tuberkulose (TB), die weltweit häufigste Todesursache durch einen Infektionserreger, tötete im Jahr 2022 1,6 Millionen Menschen und wurde erst während der Pandemie 2019-2021 von COVID-19 übertroffen. Die Krankheit wird durch das Bakterium Mycobacterium tuberculosis (M.tb) verursacht. Der Mycobacterium bovis-Stamm Bacillus Calmette-Guérin (BCG), der einzige TB-Impfstoff, ist der älteste zugelassene Impfstoff der Welt, der noch verwendet wird. Derzeit befinden sich 12 Impfstoffe in klinischen Studien und Dutzende von Impfstoffen in der präklinischen Entwicklung. Die Methode der Wahl, um die Wirksamkeit von TB-Impfstoffen in präklinischen Studien zu beurteilen, ist die Zählung von Bakterienkolonien durch den Colony-Forming Units (KBE)-Assay. Dieser zeitaufwändige Assay dauert 4 bis 6 Wochen, erfordert viel Platz im Labor und im Inkubator, hat hohe Reagenzienkosten und ist anfällig für Kontaminationen. Hier beschreiben wir eine optimierte Methode zur Koloniezählung, die Mikro-KBE (mCFU), die eine einfache und schnelle Lösung zur Analyse der Wirksamkeit von M.tb-Impfstoffen bietet. Der mCFU-Assay benötigt zehnmal weniger Reagenzien, verkürzt die Inkubationszeit um das Dreifache und dauert 1 bis 2 Wochen, reduziert den Platzbedarf im Labor und die Reagenzienkosten und minimiert die Gesundheits- und Sicherheitsrisiken, die mit der Arbeit mit einer großen Anzahl von M.tb verbunden sind. Um die Wirksamkeit eines Tuberkuloseimpfstoffs zu bewerten, können Proben aus einer Vielzahl von Quellen entnommen werden, einschließlich Gewebe von geimpften Tieren, die mit Mykobakterien infiziert sind. Wir beschreiben auch eine optimierte Methode zur Herstellung einer einzelligen, einheitlichen und qualitativ hochwertigen Mykobakterienkultur für Infektionsstudien. Schließlich schlagen wir vor, dass diese Methoden universell für präklinische Studien zur Bestimmung der Wirksamkeit von Impfstoffen eingesetzt werden sollten, was letztendlich zu einer Zeitverkürzung bei der Entwicklung von Impfstoffen gegen TB führt.
Einleitung
Tuberkulose (TB) ist weltweit die häufigste Todesursache durch einen einzigen Infektionserreger, das Bakterium Mycobacterium tuberculosis (M.tb), das mehr Menschen tötet als jeder andere Krankheitserreger. Im Jahr 2021 war Tuberkulose für 1,6 Millionen Todesfälle verantwortlich und wurde während der Pandemie 2019-2021 von COVID-19 übertroffen1. Darüber hinaus war laut dem globalen TB-Bericht der Weltgesundheitsorganisation von 2022 die COVID-19-Pandemie für einen Anstieg der neuen TB-Fälle verantwortlich. Die WHO berichtet auch von einem starken Rückgang der Zahl der Menschen, bei denen in diesem Zeitraum Tuberkulose diagnostiziert wurde, was die Zahl der Tuberkulosefälle weiter erhöhen könnte1.
Der Bacillus Calmette-Guérin (BCG) ist ein lebend abgeschwächter Stamm des pathogenen Mycobacterium bovis, der vor mehr als 100 Jahren erstmals als Impfstoff eingesetzt wurde. Dies ist der einzige Impfstoff gegen Tuberkulose und der älteste zugelassene Impfstoff der Welt, der noch verwendet wird 2,3. Derzeit befinden sich 12 Impfstoffe in verschiedenen Phasen klinischer Studien4 und Dutzende von Impfstoffen befinden sich in der präklinischen Entwicklung 5,6. Die präklinische Bewertung von Impfstoffen gegen Tuberkulose umfasst die Bewertung der Sicherheit und Immunogenität7, die in verschiedenen Tiermodellen wie Zebrafischen, Mäusen, Meerschweinchen, Kaninchen, Rindern und nichtmenschlichen Primaten erzielt werdenkönnen 8,9,10. Darüber hinaus erfordert die Beurteilung der Fähigkeit eines Impfstoffs, einen Schutz vor einer M.tb-Infektion und/oder -Übertragung zu induzieren, d. h. die Wirksamkeit des Impfstoffs, eine M.tb-Provokation in vivo 5,11. Interessanterweise induziert die BCG-Impfung unspezifische Effekte, die das Überleben anderer bakterieller und viraler Krankheitserreger beeinflussen12,13 durch den Mechanismus der trainierten Immunität14. Um die lebensfähige Bakterienlast in einem infizierten Tier zu quantifizieren, ist die Methode der Wahl die Zählung von Bakterienkolonien durch den Colony-Forming Units (KBE)-Assay 5,15. KBE ist eine Einheit, die die Anzahl der Mikroorganismen (Bakterien oder Pilze) schätzt, die unter bestimmten Wachstumsbedingungen Kolonien bilden. KBE stammen von lebensfähigen und replizierten Mikroorganismen, und die absolute Anzahl der lebenden Mikroorganismen in jeder Kolonie ist schwer abzuschätzen. Es ist unsicher, ob eine Kolonie aus einem oder mehreren Mikroorganismen entstanden ist. Die KBE-Einheit spiegelt diese Unsicherheit wider, so dass eine große Variabilität in Replikaten derselben Probe beobachtet werden kann. Dieser zeitaufwändige Assay erfordert spezialisierte Techniker, die für die Arbeit in einer Einrichtung der Biosicherheitsstufe 3 (BSL3) geschult sind, einen beträchtlichen Labor- und Inkubatorplatz, dauert 4 bis 6 Wochen und ist anfällig für Kontaminationen.
In dieser Studie beschreiben wir eine optimierte Methode zur Koloniezählung, die Mikro-KBE (mCFU), und bieten eine einfache und schnelle Lösung zur Analyse der Ergebnisse 15,16,17,18,19,20. Der mCFU-Assay benötigt zehnmal weniger Reagenzien, verkürzt die Inkubationszeit um das Dreifache und dauert 1 bis 2 Wochen, reduziert den Platzbedarf im Labor und die Reagenzienkosten und minimiert die Gesundheits- und Sicherheitsrisiken, die mit der Arbeit mit einer großen Anzahl von M.tb verbunden sind. Wir schlagen vor, dass diese Methode universell für präklinische Studien zur Bestimmung der Wirksamkeit von Impfstoffen eingesetzt werden sollte, was letztendlich zu einer Zeitverkürzung bei der Entwicklung von Impfstoffen gegen TB führt. Schließlich wurde diese optimierte Methode der KBE-Zählung verwendet, um nicht nur Mykobakterien, sondern auch andere Bakterien wie Escherichia coli und Ralstonia solanacearum21 zu quantifizieren.
Access restricted. Please log in or start a trial to view this content.
Protokoll
HINWEIS: Das hier beschriebene Protokoll gilt für BCG, kann aber auf alle Mykobakterien angewendet werden. BCG kann als Ersatzbakterium für TB-Experimente verwendet werden, wenn BSL3-Einrichtungen nicht verfügbar sind22. Die folgenden Verfahren unter Verwendung von BCG sollten in einem Labor der Biosicherheitsstufe 2 (BSL2) durchgeführt werden und den entsprechenden Biosicherheitsrichtlinien und der guten Laborpraxis für die Manipulation von Mikroorganismen der Gefahrengruppe 2 entsprechen.
1. Vorbereitung der Nährmedien
- Bereiten Sie Middlebrook 7H9-Brühe zu, die mit 10% (v/v) Ölsäure, Albumin, Dextrose und Katalase (OADC) angereichert ist, gemäß den Anweisungen des Lieferanten. Ergänzen Sie die Brühe mit 0,05 % (v/v) Tyloxapol.
HINWEIS: Tyloxapol ist ein nichtionisches flüssiges Polymer, das als Tensid verwendet wurde, um die Bildung von Bakterienklumpen zu verhindern16. - Bereiten Sie Middlebrook 7H10 Feststoffmedium mit 10% (v/v) OADC-Anreicherung gemäß den Anweisungen des Lieferanten vor.
- Verteilen Sie 40 ml Medium pro quadratischer Petrischale (120 mm x 120 mm). Lassen Sie die Platten trocknen, um die Kondensation an der Oberfläche des Agars zu minimieren.
HINWEIS: Diese spezifische Größe der Petrischale ist von grundlegender Bedeutung, um eine direkte Transposation von mindestens 96 Tröpfchen von einer 96-Well-Platte zu ermöglichen. Eine effektive Trocknung der Platten erleichtert später das Auftragen kleiner Tröpfchen der Bakteriensuspension und verhindert die Ausbreitung der Tröpfchen. - Bereiten Sie entweder Roswell Park Memorial Institute Medium (RPMI 1640) oder Dulbecco's Modified Eagle Medium (DMEM) vor, um das Infektionsmedium zu produzieren. In beiden Fällen ergänzen Sie das Medium mit 10 % fötalem Kälberserum, 1 % L-Glutamin und 1 mM Natriumpyruvat. Fügen Sie dem Medium kein Penicillin und Streptomycin hinzu.
2. Probenvorbereitung
- Besorgen Sie sich Proben aus einer Vielzahl von Quellen. Um die KBE zu quantifizieren und die Wirksamkeit eines TB-Impfstoffs zu bewerten, werden in der Regel Proben von geimpftem und ungeimpftem tierischem Gewebe entnommen. Zum Beispiel Mauslunge und Milz11 oder Makakenlunge, thorakale und periphere Lymphknoten, Milz, Leber, Haut, Blut, Knochenmark und bronchoalveoläre Lavagewaschen 23. Alternativ können Proben aus In-vitro-Kulturen von Makrophagen/dendritischen Zellen/Neutrophilen entnommen werden, die mit BCG 18,19,20,24,25,26 infiziert sind.
3. Herstellung von BCG-Kultur
HINWEIS: Bei In-vivo-Studien mit TB-Impfstoffen besteht das Ziel darin, die Wirksamkeit von BCG zu verbessern. Daher werden in der Regel BCG-geimpfte Gruppen als Kontrolle verwendet. BCG-Stämme, die für die Impfung am Menschen verwendet werden, eignen sich ideal für Tests in Tiermodellen. In diesem Fall muss eine BCG-Kultur gemäß den Anweisungen des Lieferanten27 rekonstituiert werden. Eine BCG-Kultur für In-vivo-Studien kann aber auch im eigenen Haus hergestellt werden11. Die Herstellung von einzelligen, einheitlichen und qualitativ hochwertigen BCG-Kulturen für In-vitro-Infektionsprotokolle wurde in mehreren Studiensehr erfolgreich hergestellt 11,16,18,19,20,26,28,29 unter Verwendung des folgenden Protokolls, das auch für Tierversuche verwendet werden kann.
- Kultivieren Sie 50 ml BCG in 7H9-Brühe bei 37 °C unter Rühren bei 200 U/min. Variieren Sie die Lautstärke entsprechend den Bedürfnissen des Experiments.
- Sammeln Sie jeden Tag 8-10 Tage lang 100 μl der Kultur und verdünnen Sie sie, indem Sie 900 μl PBS in eine 1-ml-Küvette geben. Dann wird die optische Dichte der Bakterien gemessen (OD bei λ=600 nm; OD600) in einem Spektralphotometer. Zeichnen Sie aus diesen Werten eine Wachstumskurve. Identifizieren Sie die mittlere logarithmische Phase der Kultur (wenn sich der OD pro Zeiteinheit konsistent verdoppelt).
- Bereiten Sie eine Folgekultur vor und inkubieren Sie, bis die mittlere/späte Wachstumsphase erreicht ist, wie in den Schritten 3.1 und 3.2 beschrieben. Verwenden Sie die im vorherigen Schritt erhaltenen Werte als Leitfaden. Stellen Sie sicher, dass die Kultur nicht die stationäre Wachstumsphase erreicht (wenn sich die OD zu stabilisieren beginnt), um eine qualitativ hochwertige Kultur lebensfähiger Bakterien zu erhalten.
- Sammeln Sie die Kultur in der mittleren/späten Wachstumsphase des Stammes. Bei 3000 x g für 10 min zentrifugieren. Entfernen Sie den Überstand.
- Fügen Sie 10 ml PBS hinzu, um die Bakterien zu waschen. Bei 3000 x g für 10 min zentrifugieren. Entfernen Sie den Überstand.
- Resuspendieren Sie die Bakterien mit 5 ml Infektionsmedium. Legen Sie den Schlauch für 15 Minuten in ein Ultraschallbad, volle Leistung bei 80 Hz.
- Bei 1000 x g 10 min zentrifugieren. Sammeln Sie den Überstand und vermeiden Sie das Pellet, da es reich an Bakterienklumpen ist, die in einer hochwertigen BCG-Kultur vermieden werden sollten, und entsorgen Sie es.
- Messen Sie den Außendurchmesser des Überstandes. Hier entsprechen Kulturen in der exponentiellen Wachstumsphase mit einem OD600 von 0,1 1 x 107 KBE/ml.
HINWEIS: Jedes Labor sollte seine eigenen BCG-Wachstumskurven erstellen, bevor es mit Experimenten beginnt, um eine lineare Regression zwischen OD600 und KBE mit dem Spektralphotometer zu ermitteln. Bitte beachten Sie, dass Spektralphotometer unterschiedliche Lichtwegabstände haben, die die für dieselbe Probe erhaltenen Messwerte variieren können. - Führen Sie einfache Berechnungen durch, um die Anzahl der Bakterien zu bestimmen, die jeder Wirtszellkultur hinzugefügt werden sollen. Die Anzahl der Bakterien pro Wirtszelle ist die Multiplicity of Infection (MOI). Verwenden Sie eine MOI von 10 Bakterien pro Wirtszelle, was die häufigste MOI ist, die für BCG-Infektionsexperimente verwendet wird.
4. Assay für mikrokoloniebildende Einheiten
HINWEIS: Nach Abschluss eines In-vivo - oder In-vitro-Infektionsexperiments kann die Zählung der Bakterien mittels mCFU durchgeführt werden. Für In-vivo-Studien müssen die Proben zunächst in einem Kügelchenschläger oder einem anderen Gewebehomogenisator homogenisiert werden. Für In-vitro-Kulturen von Makrophagen/dendritischen Zellen/Neutrophilen, die mit BCG infiziert sind, müssen die Proben mit einem nichtionischen Detergens (z. B. 0,05%ige Lösung eines nichtionischen, nicht denaturierenden Detergens) lysiert werden.
- Serielle Verdünnungen mit einer 96-Well-Platte: Führen Sie serielle 10-fache Verdünnungen der Lysate in einer sterilen 96-Well-Platte gemäß dem Schema in Abbildung 1A durch. Verteilen Sie die Lysate auf die Reihen A und E. Für jede Platte beträgt die maximale Anzahl von Proben und/oder Replikaten 24.
- Geben Sie 180 μldH2Oin die verbleibenden Vertiefungen, um die serielle Verdünnung durchzuführen.
- Mit einer 12-Kanal-Pipette werden die Lysate in Reihe A resuspendiert und 20 μl in Reihe B überführt (20 μl Lysat + 180 μl dH2O). Gut homogenisieren. Wiederholen Sie diesen Schritt nacheinander für die Reihen B und C, bis die letzte Verdünnung in Reihe D erreicht ist.
HINWEIS: Wir führen in der Regel drei Verdünnungen durch (100, 101, 102, 103), d. h. 4 Reihen der Platte (A-D oder E-H) für jeden Satz von 12 Proben und/oder Replikaten. - Mikrotröpfchenplattierung: Verwenden Sie eine 0,5-10 μl (dünne Spitzen werden bevorzugt) Mehrkanalpipette, um 5 μl von jeder Reihe der 96-Well-Platte auf die quadratische Platte des festen Mediums zu übertragen, wie in Abbildung 1B dargestellt.
- Während Sie die 5-μl-Tröpfchen langsam pipettieren, lassen Sie sie den Agar leicht berühren. Dies trägt dazu bei, den Tropfen von der Spitze in Richtung Agar abzuleiten und die Möglichkeit der Rückhaltung der Flüssigkeit in der Spitze zu verringern.
- Lassen Sie die Tröpfchen trocknen, schließen Sie die Agarplatte und inkubieren Sie sie bei 37 °C, während Sie das Bakterienwachstum überwachen. Optional können Sie die Agarplatten in einem verschlossenen Plastikbeutel inkubieren, um ein Austrocknen der Platten zu verhindern.
- Zählung der Mikrokolonien: Nach ca. 6-10 Tagen Inkubation nach einzelnen Kolonien suchen, die mit bloßem Auge sichtbar sind (Abbildung 2).
- Zählen Sie die Kolonien mit dem Objektiv mit der niedrigsten Vergrößerung (4x oder niedriger) eines inversen optischen Mikroskops oder einer Lupe. Die Zählung sollte in den Verdünnungen durchgeführt werden, in denen die Anzahl der Kolonien kleiner als 300 und höher als 30 ist. Alternativ können Sie eine Kamera verwenden, um ein Bild des Tröpfchens zu machen, um die Kolonien manuell auf dem Computer zu zählen, oder eine Software wie ImageJ verwenden, um die Koloniezählung zu automatisieren.
- Um Zellzahlen in KBE/ml auszudrücken, verwenden Sie die folgende Gleichung:
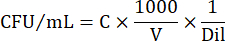
Dabei ist C = Anzahl der gezählten Kolonien, V = Volumen in μl und Dil = Verdünnung, bei der die Kolonien gezählt wurden (100, 101, 102, 103). Wenn z. B. 30 Kolonien in einem 5-μl-Tröpfchen in der Verdünnung 102 gezählt wurden, dann: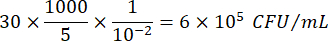
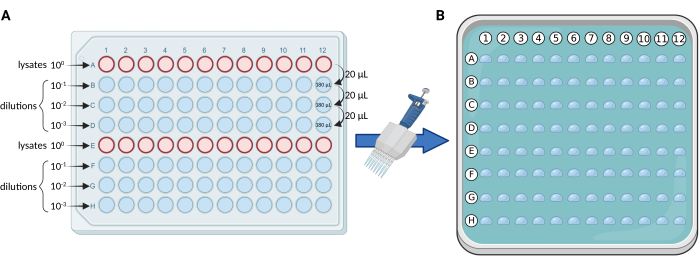
Abbildung 1. Schematische Darstellung des mCFU-Protokolls. (A) Serielle 10-fache Verdünnung der BCG-haltigen Lysate in einer 96-Well-Platte. (B) Quadratische Petrischale, die festes Nährmedium enthält und von 96 Tröpfchen zu je 5 μl überlagert wird. Die Tröpfchen werden mit einer Mehrkanalpipette direkt von der 96-Well-Platte pipettiert. Erstellt mit BioRender.com. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
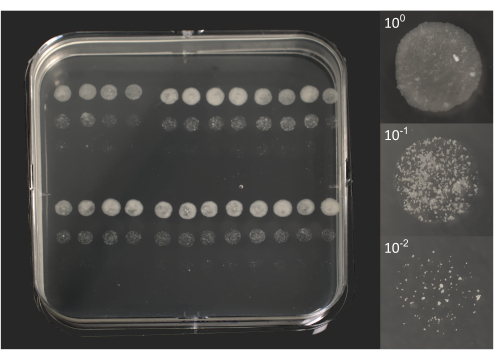
Abbildung 2. Mikrokoloniebildende Einheiten von BCG nach 10 Tagen Inkubation. Links ein Foto einer quadratischen Petrischale, die von 96 Tröpfchen zu je 5 μl überlagert ist, wie zuvor in Abbildung 1B dargestellt. Auf der rechten Seite entsprechen einzelne Fotos von 3 Tröpfchen einem Originallysat (100) und zwei Verdünnungen (101, 102). Die Fotos wurden mit einer DSRL-Kamera aufgenommen, die mit einem 18-55-mm-Zoomobjektiv (Platte) oder einem 105-mm-Makroobjektiv (Tröpfchen) ausgestattet war. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
5. Zählung von Mikrokolonien in Fidschi (BildJ)
HINWEIS: Die mCFU-Methode ermöglicht die Quantifizierung großer Probensätze. Bilder der Tröpfchen können für die posteriore Analyse aufgezeichnet werden, um die Koloniezählung zu erleichtern. Mehrere fotografische Geräte können zu diesem Zweck Bilder mit ausreichender Qualität erzeugen. Dazu gehören Digitalkameras, Webcams, an der Kamera befestigte Mikroskope und Lupen sowie Mobiltelefone. Kostenlose Bildanalyse-Software wie ImageJ bietet die Möglichkeit der manuellen oder automatisierten Koloniezählung in diesen Bildern. Um beide Methoden zu demonstrieren, wird Fiji verwendet, eine Distribution von ImageJ, die mehrere Werkzeuge für die wissenschaftliche Bildanalyse enthält30. Fidschi kann von https://fiji.sc/ heruntergeladen werden.
- Manuelle Zählmethode
- Öffnen Sie das Bild mit der mCFU in Fidschi. Wählen Sie Plugins > analysieren Sie > Zellzähler.
- Wählen Sie im Menü Zellenzähler die Option Initialisieren aus, und wählen Sie dann einen Zähler aus (z. B. Typ 1).
- Fahren Sie fort, indem Sie auf jede Kolonie klicken. Jeder Klick wird auf dem Bild angezeigt und aktualisiert den Zähler (Abbildung 3). Um versehentliche Klicks rückgängig zu machen, wählen Sie Löschen aus.
- Registrieren Sie den Wert, der auf dem Zähler angezeigt wird. Klicken Sie auf die Schaltfläche Zurücksetzen , um die Anzahl zurückzusetzen und ein neues Bild zu öffnen, um weitere Proben zu zählen.
HINWEIS: Weitere Anweisungen zu diesem Plugin finden Sie unter https://imagej.net/plugins/cell-counter.
- Automatisiertes Zählverfahren
- Öffnen Sie das Bild mit der mCFU in Fidschi. Wählen Sie Bild aus, > geben Sie > 8-Bit ein. Dadurch wird das Bild in ein 8-Bit-Graustufenbild konvertiert.
- Wählen Sie das Oval-Werkzeug in der Symbolleiste aus und zeichnen Sie ein Oval um den Bereich mit den Kolonien (Abbildung 4A). Das Oval kann nach dem Zeichnen angepasst werden.
- Wählen Sie "Bearbeiten" > "Außen löschen", um alle Interferenzen aus dem Außenbereich zu entfernen (Abbildung 4B). Wählen Sie Bild > passen Sie > Schwellenwert an.
- Verschieben Sie die Schieberegler im Schwellenwertmenü, bis die Kolonien rot angezeigt werden und Hintergrundgeräusche minimiert werden (Abbildung 4C).
- Wählen Sie Übernehmen aus, und schließen Sie das Fenster mit dem Schwellenwert. Es wird ein Schwarz-Weiß-Bild erzeugt (Abbildung 4D).
- Wählen Sie "Analysieren" > "Partikel analysieren". Geben Sie im Fenster "Partikel analysieren" den Bereich für die Koloniefläche (zwischen 1 und unendlich, gemessen in quadrierten Pixeln) und die Kreisförmigkeit (zwischen 0 und 1, wobei 1 ein perfekter Kreis ist; Abbildung 4E).
- Wählen Sie "Konturen " im Popup-Menü "Einblenden" aus. Aktivieren Sie Ergebnisse anzeigen , um detaillierte Messungen für jede Kolonie im Ergebnisfenster anzuzeigen. Aktivieren Sie Ergebnisse löschen , um alle vorherigen Messungen zu löschen. Aktivieren Sie das Kontrollkästchen Zusammenfassen , um die zusammengefassten Ergebnisse der Messungen anzuzeigen (Abbildung 4E).
- Starten Sie den Analyzer, indem Sie OK auswählen. Es wird ein neues Fenster angezeigt, in dem alle umrissenen Kolonien angezeigt werden, die erkannt und gezählt wurden. Im Ergebnisfenster werden die Details für jede Kolonie angezeigt, und im Fenster mit den zusammengefassten Ergebnissen werden die insgesamt gezählten Kolonien angezeigt (Abbildung 4F).
HINWEIS: Die Einstellungen für Größe und Kreisförmigkeit variieren mit der Auflösung und Vergrößerung des Bildes sowie der Größe und Form der Kolonien. Wiederholen Sie den Vorgang mehrmals, bis die besten Einstellungen gefunden sind, die alle Kolonien erkennen. Weitere Anleitungen zum Plugin "Partikel analysieren" finden Sie unter https://imagej.nih.gov/ij/docs/menus/analyze.html#ap.
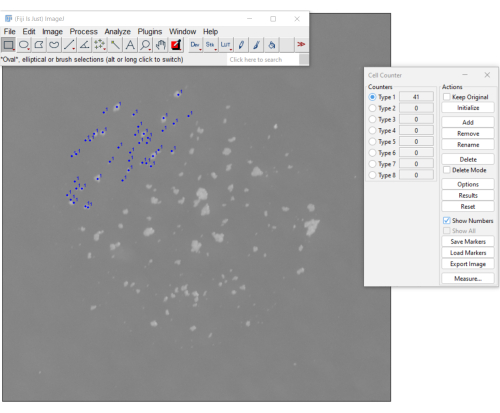
Abbildung 3. Eine manuelle Methode zum Zählen von mCFU mit dem Zellzähler-Plugin der Fiji-Software. Die blauen Punkte zeigen Kolonien an, die der Benutzer bereits angeklickt hat. Das Menü auf der rechten Seite zeigt die Anzahl der bisher gezählten Völker an (die Anzahl beträgt 41). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 4. Eine automatisierte Methode zur Zählung von mCFU mit Fidschi-Software. (A, B) Der Interessenbereich mit den Kolonien wird mit dem ovalen Auswahlwerkzeug ausgewählt, und der äußere Bereich wird mit dem Befehl "Außen löschen" entfernt. (C, D) Ein Schwarz-Weiß-Bild der Kolonien wird mit dem Schwellenwert-Werkzeug erzeugt. (E, F) Die Anzahl der Kolonien wird mit dem Werkzeug "Partikel analysieren" quantifiziert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Access restricted. Please log in or start a trial to view this content.
Ergebnisse
Der hier beschriebene mCFU-Assay erhöht die Menge an Informationen, die aus einer einzelnen Petrischale gewonnen werden können, auf mindestens das 96-fache. Abbildung 5 zeigt einen Vergleich zweier Methoden zur Verabreichung von Medikamenten für die umfunktionierte Verwendung von Saquinavir (SQV)31,32 als wirtsgesteuertes Medikament zur Behandlung von Tuberkulose. In diesem Assay wurden vier verschiedene Stämme von Mycobacter...
Access restricted. Please log in or start a trial to view this content.
Diskussion
Tuberkulose ist ein wichtiges Problem der öffentlichen Gesundheit, das vor allem in Ländern mit niedrigem und mittlerem Einkommen zunehmend an Bedeutung gewinnt. Die Unterbrechung der Gesundheitseinrichtungen zur Diagnose und Behandlung von Tuberkulose während der COVID-19-Pandemie wirkte sich negativ auf die Inzidenz neuer Fälle aus1. Darüber hinaus müssen die multiresistenten und extensiv resistenten M.tb-Stämme und die Koinfektion von M.tb und HIV dringend angegangen we...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
DP und PJGB erklären, dass die Studie ohne kommerzielle oder finanzielle Beziehungen durchgeführt wurde, die als potenzieller Interessenkonflikt ausgelegt werden könnten.
Danksagungen
Diese Arbeit wurde durch interne Mittel der Medizinischen Fakultät der Universidade Católica Portuguesa und externe Mittel der Fundação para a Ciência e a Tecnologia (FCT) im Rahmen der Zuschüsse UIDP/04279/2020, UIDB/04279/2020 und EXPL/SAU-INF/0742/2021 unterstützt.
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| 96-well plates | VWR | 734-2781 | |
| DSLR 15-55 mm lens | Nikon | AF-P DX NIKKOR 18-55mm f/3.5-5.6G VR | |
| DSLR camera | Nikon | D3400 | |
| DSLR macro lens | Sigma | MACRO 105mm F2.8 EX DG OS HSM | |
| Fetal calf serum | Gibco | 10270106 | |
| Fiji Software | https://fiji.sc/ | Fiji is an open-source software supported by several laboratories, institutions, and individuals. All the required plugins are included. | |
| Igepal CA-630 | Sigma-Aldrich | 18896 | |
| L-glutamine | Gibco | 25030-081 | |
| Middlebrook 7H10 | BD | 262710 | |
| Middlebrook 7H9 | BD | 271310 | |
| Multichannel pipette (0.5 - 10 µl) | Gilson | FA10013 | |
| Multichannel pipette (20 - 200 µl) | Gilson | FA10011 | |
| Mycobacterium bovis BCG | American Type Culture Collection | ATCC35734 | strain TMC 1011 [BCG Pasteur] |
| OADC enrichment | BD | 211886 | |
| Phosphate buffered saline (PBS) | NZYTech | MB25201 | |
| RPMI 1640 medium | Gibco | 21875091 | |
| Sodium pyruvate | Gibco | 11360-070 | |
| Spectrophotometer UV-6300PC | VWR | 634-6041 | |
| Square Petri dish 120 x 120 mm | Corning | BP124-05 | |
| Tyloxapol | Sigma-Aldrich | T8761 | |
| Ultrasound bath Elma P 30 H | VWR | 142-0051 |
Referenzen
- World Health Organization. Global Tuberculosis Report 2022. , World Health Organization. Geneva. (2022).
- Bettencourt, P. J. G., Joosten, S. A., Lindestam Arlehamn, C. S., Behr, M. A., Locht, C., Neyrolles, O. 100 years of the Bacillus Calmette-Guérin vaccine. Vaccine. 39 (50), 7221-7222 (2021).
- Bettencourt, P. J. G. The 100th anniversary of bacille Calmette-Guérin (BCG) and the latest vaccines against COVID-19. The International Journal of Tuberculosis and Lung Disease. 25 (8), 611-613 (2021).
- Scriba, T. J., Netea, M. G., Ginsberg, A. M. Key recent advances in TB vaccine development and understanding of protective immune responses against Mycobacterium tuberculosis. Seminars in Immunology. 50, 101431(2020).
- McShane, H., Williams, A. A review of preclinical animal models utilised for TB vaccine evaluation in the context of recent human efficacy data. Tuberculosis. 94 (2), 105-110 (2014).
- Voss, G., et al. Progress and challenges in TB vaccine development. F1000Research. 7, 199(2018).
- Satti, I., McShane, H. Current approaches toward identifying a correlate of immune protection from tuberculosis. Expert Review of Vaccines. 18 (1), 43-59 (2019).
- Young, D. Animal models of tuberculosis. European Journal of Immunology. 39 (8), 2011-2014 (2009).
- Pedroza-Roldán, C., Flores-Valdez, M. A. Recent mouse models and vaccine candidates for preventing chronic/latent tuberculosis infection and its reactivation. Pathogens and disease. 75 (6), (2017).
- Gong, W., Liang, Y., Wu, X. Animal Models of Tuberculosis Vaccine Research: An Important Component in the Fight against Tuberculosis. BioMed Research International. 2020, 1-21 (2020).
- Bettencourt, P., et al. Identification of antigens presented by MHC for vaccines against tuberculosis. NPJ vaccines. 5 (1), 2(2020).
- Moorlag, S. J. C. F. M., Arts, R. J. W., van Crevel, R., Netea, M. G. Non-specific effects of BCG vaccine on viral infections. Clinical Microbiology and Infection. 25 (12), 1473-1478 (2019).
- Wilkie, M., et al. Functional in-vitro evaluation of the non-specific effects of BCG vaccination in a randomised controlled clinical study. Scientific Reports. 12 (1), 7808(2022).
- Netea, M. G., et al. Trained immunity: A program of innate immune memory in health and disease. Science. 352 (6284), aaf1098(2016).
- Bettencourt, P., Pires, D., Carmo, N., Anes, E. Application of Confocal Microscopy for Quantification of Intracellular Mycobacteria in Macrophages. Microscopy: Science, Technology, Applications and Education. 1, 614-621 (2010).
- Bettencourt, P., Carmo, N., Pires, D., Timóteo, P., Anes, E. Mycobacterial infection of macrophages: the effect of the multiplicity of infection. Antimicrobial research: Novel bioknowledge and educational programs. , 651-664 (2017).
- Pires, D., Bettencourt, P., Carmo, N., Niederweis, M., Anes, E. Role of Mycobacterium tuberculosis outer-membrane porins in bacterial survival within macrophages. Drug Discovery Today. 15 (23-24), 1112-1113 (2010).
- Pires, D., et al. Mycobacterium tuberculosis Modulates miR-106b-5p to Control Cathepsin S Expression Resulting in Higher Pathogen Survival and Poor T-Cell Activation. Frontiers in immunology. 8 (DEC), 1819(2017).
- Pires, D., et al. Role of Cathepsins in Mycobacterium tuberculosis Survival in Human Macrophages. Scientific reports. 6 (August), 32247(2016).
- Bettencourt, P., et al. Actin-binding protein regulation by microRNAs as a novel microbial strategy to modulate phagocytosis by host cells: the case of N-Wasp and miR-142-3p. Frontiers in cellular and infection microbiology. 3 (June), 19(2013).
- Bhuyan, S., et al. Microliter spotting and micro-colony observation: A rapid and simple approach for counting bacterial colony forming units. Journal of Microbiological Methods. 207, 106707(2023).
- Jackson, S., McShane, H. Challenges in Developing a Controlled Human Tuberculosis Challenge Model. Current topics in microbiology and immunology. , 1-27 (2022).
- Darrah, P. A., et al. Prevention of tuberculosis in macaques after intravenous BCG immunization. Nature. 577 (7788), 95-102 (2020).
- Madura Larsen, J., et al. BCG stimulated dendritic cells induce an interleukin-10 producing T-cell population with no T helper 1 or T helper 2 bias in vitro. Immunology. 121 (2), 276-282 (2007).
- Bickett, T. E., et al. Characterizing the BCG-Induced Macrophage and Neutrophil Mechanisms for Defense Against Mycobacterium tuberculosis. Frontiers in immunology. 11, 1202(2020).
- Pires, D., et al. Interference of Mycobacterium tuberculosis with the endocytic pathways on macrophages and dendritic cells from healthy donors: role of cathepsins. Drug Discovery Today. 15 (23-24), 1112-1112 (2010).
- Betts, G., et al. Optimising Immunogenicity with Viral Vectors: Mixing MVA and HAdV-5 Expressing the Mycobacterial Antigen Ag85A in a Single Injection. PLoS ONE. 7 (12), e50447(2012).
- Tanner, R., et al. The influence of haemoglobin and iron on in vitro mycobacterial growth inhibition assays. Scientific reports. 7 (1), 43478(2017).
- McNeill, E., et al. Regulation of mycobacterial infection by macrophage Gch1 and tetrahydrobiopterin. Nature communications. 9 (1), 5409(2018).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Pereira, M., Vale, N. Saquinavir: From HIV to COVID-19 and Cancer Treatment. Biomolecules. 12 (7), 944(2022).
- Pires, D., et al. Repurposing Saquinavir for Host-Directed Therapy to Control Mycobacterium Tuberculosis Infection. Frontiers in immunology. 12, 647728(2021).
- Pires, D., et al. Liposomal Delivery of Saquinavir to Macrophages Overcomes Cathepsin Blockade by Mycobacterium tuberculosis and Helps Control the Phagosomal Replicative Niches. International journal of molecular sciences. 24 (2), (2023).
- Maartens, G., Wilkinson, R. J. Tuberculosis. The Lancet. 370 (9604), 2030-2043 (2007).
- Matarazzo, L., Bettencourt, P. J. G. mRNA vaccines: a new opportunity for malaria, tuberculosis and HIV. Frontiers in Immunology. 14, 1172691(2023).
- Young, D., Dye, C. The Development and Impact of Tuberculosis Vaccines. Cell. 124 (4), 683-687 (2006).
- Kommareddi, S., Abramowsky, C. R., Swinehart, G. L., Hrabak, L. Nontuberculous mycobacterial infections: Comparison of the fluorescent auramine-o and Ziehl-Neelsen techniques in tissue diagnosis. Human Pathology. 15 (11), 1085-1089 (1984).
- Sabiiti, W., et al. A Tuberculosis Molecular Bacterial Load Assay (TB-MBLA). Journal of visualized experiments: JoVE. (158), e60460(2020).
- Somoskövi, A., et al. Comparison of Recoveries of Mycobacterium tuberculosis Using the Automated BACTEC MGIT 960 System, the BACTEC 460 TB System, and Löwenstein-Jensen Medium. Journal of Clinical Microbiology. 38 (6), 2395-2397 (2000).
- Tanner, R., et al. The in vitro direct mycobacterial growth inhibition assay (MGIA) for the early evaluation of TB vaccine candidates and assessment of protective immunity: a protocol for non-human primate cells. F1000Research. 10, 257(2021).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten