Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Dosage d’unités formant des microcolonies pour l’évaluation de l’efficacité des vaccins contre la tuberculose
Dans cet article
Résumé
La détermination des unités formant des colonies (UFC) est la technique de référence pour quantifier les bactéries, y compris Mycobacterium tuberculosis qui peut prendre des semaines pour former des colonies visibles. Nous décrivons ici une micro-UFC pour la détermination de l’UFC avec une efficacité accrue en termes de temps, un espace de laboratoire et un coût de réactif réduits, et une évolutivité pour les expériences à moyen et haut débit.
Résumé
La tuberculose, première cause de décès par agent infectieux dans le monde, a tué 1,6 million de personnes en 2022, n’étant dépassée que par la COVID-19 pendant la pandémie de 2019-2021. La maladie est causée par la bactérie Mycobacterium tuberculosis (M.tb). La souche de Mycobacterium bovis Bacillus Calmette-Guérin (BCG), le seul vaccin contre la tuberculose, est le plus ancien vaccin homologué au monde, toujours utilisé. À l’heure actuelle, 12 vaccins font l’objet d’essais cliniques et des dizaines de vaccins sont en cours de développement préclinique. La méthode de choix utilisée pour évaluer l’efficacité des vaccins antituberculeux dans les études précliniques est le dénombrement des colonies bactériennes par le test des unités formant colonies (UFC). Ce test fastidieux prend de 4 à 6 semaines à conclure, nécessite un espace de laboratoire et d’incubateur important, a des coûts de réactifs élevés et est sujet à la contamination. Nous décrivons ici une méthode optimisée pour le dénombrement des colonies, la micro-UFC (mCFU), qui offre une solution simple et rapide pour analyser les résultats d’efficacité du vaccin contre la tubercule anti-M.tb . Le test mCFU nécessite dix fois moins de réactifs, réduit la période d’incubation par trois, prenant 1 à 2 semaines pour conclure, réduit l’espace de laboratoire et le coût des réactifs, et minimise les risques pour la santé et la sécurité associés au travail avec un grand nombre de M.tb. De plus, pour évaluer l’efficacité d’un vaccin antituberculeux, des échantillons peuvent être prélevés à partir de diverses sources, y compris des tissus d’animaux vaccinés infectés par des mycobactéries. Nous décrivons également une méthode optimisée pour produire une culture mycobactérienne unicellulaire, uniforme et de haute qualité pour les études d’infection. Enfin, nous proposons que ces méthodes soient universellement adoptées pour les études précliniques de détermination de l’efficacité des vaccins, ce qui, en fin de compte, conduirait à réduire le temps de développement des vaccins contre la tuberculose.
Introduction
La tuberculose est la principale cause de décès dans le monde causée par un seul agent infectieux, la bactérie Mycobacterium tuberculosis (M.tb), qui tue plus de personnes que tout autre agent pathogène. En 2021, la tuberculose a été responsable de 1,6 million de décès et a été dépassée par la COVID-19 pendant la pandémie de 2019-20211. De plus, selon le rapport mondial sur la tuberculose de 2022 de l’Organisation mondiale de la santé, la pandémie de COVID-19 a été responsable d’une augmentation des nouveaux cas de tuberculose. L’OMS signale également une forte baisse du nombre de personnes diagnostiquées avec la tuberculose au cours de cette période, ce qui pourrait encore augmenter le nombre de cas de tuberculose1.
Le bacille de Calmette-Guérin (BCG) est une souche vivante atténuée de l’agent pathogène Mycobacterium bovis, utilisée pour la première fois comme vaccin il y a plus de 100 ans. C’est le seul vaccin contre la tuberculose et le plus ancien vaccin homologué au monde encore utilisé 2,3. À l’heure actuelle, 12 vaccins sont en cours d’essais cliniques4 et des dizaines de vaccins sont en cours de développement préclinique 5,6. L’évaluation préclinique des vaccins contre la tuberculose comprend l’évaluation de l’innocuité et de l’immunogénicité7, qui peut être obtenue dans divers modèles animaux tels que le poisson-zèbre, la souris, le cochon d’Inde, le lapin, le bétail et les primates non humains 8,9,10. De plus, l’évaluation de la capacité d’un vaccin à induire une protection contre l’infection et/ou la transmission de M.tb, c’est-à-dire l’efficacité du vaccin, nécessite une provocation M.tb in vivo 5,11. Il est intéressant de noter que la vaccination par le BCG induit des effets non spécifiques qui affectent la survie d’autres agents pathogènes bactériens et viraux12,13 par le mécanisme de l’immunité entraînée14. Pour quantifier la charge bactérienne viable chez un animal infecté, la méthode de choix est le dénombrement des colonies bactériennes à l’aide du test des unités formant colonie (UFC) 5,15. L’UFC est une unité qui estime le nombre de micro-organismes (bactéries ou champignons) qui forment des colonies dans des conditions de croissance spécifiques. Les UFC proviennent de micro-organismes viables et réplicatifs, et le nombre absolu de micro-organismes vivants au sein de chaque colonie est difficile à estimer. On ne sait pas avec certitude si une colonie provient d’un ou de plusieurs micro-organismes. L’unité UFC reflète cette incertitude, d’où une grande variabilité peut être observée dans les répétitions d’un même échantillon. Ce test qui prend beaucoup de temps nécessite des techniciens spécialisés formés pour travailler dans une installation de niveau de biosécurité 3 (BSL3), un espace de laboratoire et d’incubateur important, prend de 4 à 6 semaines pour être conclu et est sujet à la contamination.
Dans cette étude, nous décrivons une méthode optimisée pour le dénombrement des colonies, le micro-UFC (mCFU), et proposons une solution simple et rapide pour analyser les résultats 15,16,17,18,19,20. Le test mCFU nécessite dix fois moins de réactifs, réduit la période d’incubation par trois, prenant 1 à 2 semaines pour conclure, réduit l’espace de laboratoire et le coût des réactifs, et minimise les risques pour la santé et la sécurité associés au travail avec un grand nombre de M.tb. Nous proposons que cette méthode soit universellement adoptée pour les études précliniques de détermination de l’efficacité des vaccins, ce qui conduirait à terme à une réduction du temps nécessaire à la mise au point de vaccins contre la tuberculose. Enfin, cette méthode optimisée de dénombrement de l’UFC a été utilisée pour quantifier non seulement les mycobactéries mais aussi d’autres bactéries, telles que Escherichia coli et Ralstonia solanacearum21.
Protocole
REMARQUE : Le protocole décrit ici est pour le BCG mais peut être appliqué à n’importe quelle mycobactérie. Le BCG peut être utilisé comme bactérie de substitution pour les expériences sur la tuberculose lorsque les installations BSL3 ne sont pas disponibles22. Les procédures suivantes utilisant le BCG doivent être effectuées dans un laboratoire de niveau de biosécurité 2 (BSL2) et suivre les directives de biosécurité appropriées et les bonnes pratiques de laboratoire pour la manipulation des micro-organismes du groupe de danger 2.
1. Préparation des milieux de culture
- Préparez le bouillon Middlebrook 7H9 enrichi à 10 % (v/v) d’acide oléique, d’albumine, de dextrose et de catalase (OADC), selon les instructions du fournisseur. Complétez le bouillon avec 0,05 % (v/v) de tyloxapol.
REMARQUE : Tyloxapol est un polymère liquide non ionique qui a été utilisé comme tensioactif pour empêcher la formation de grumeaux bactériens16. - Préparer le milieu solide Middlebrook 7H10 complété par un enrichissement OADC à 10 % (v/v) selon les instructions du fournisseur.
- Répartir 40 mL de boîte de Pétri moyenne par carré (120 mm x 120 mm). Laissez sécher les plaques pour minimiser la condensation à la surface de la gélose.
REMARQUE : Cette taille spécifique de boîte de Pétri est fondamentale pour permettre la transposition directe d’au moins 96 gouttelettes à partir d’une plaque à 96 puits. Un séchage efficace des plaques facilitera plus tard le placage de petites gouttelettes de suspension bactérienne et empêchera les gouttelettes de se propager. - Préparez soit le milieu du Roswell Park Memorial Institute (RPMI 1640), soit le milieu Eagle modifié de Dulbecco (DMEM) pour produire le milieu d’infection. Dans les deux cas, complétez le milieu avec 10 % de sérum de veau fœtal, 1 % de L-glutamine et 1 mM de pyruvate de sodium. N’ajoutez pas de pénicilline et de streptomycine au milieu.
2. Préparation de l’échantillon
- Procurez-vous des échantillons provenant de diverses sources. En règle générale, pour quantifier l’UFC afin d’évaluer l’efficacité d’un vaccin antituberculeux, il faut prélever des échantillons de tissus animaux vaccinés et non vaccinés. Par exemple, les poumons et la ratede souris 11 ou les poumons de macaques, les ganglions lymphatiques thoraciques et périphériques, la rate, le foie, la peau, le sang, la moelle osseuse et le lavage broncho-alvéolaire23. Vous pouvez également obtenir des échantillons provenant de cultures in vitro de macrophages, de cellules dendritiques ou de neutrophiles infectés par le BCG 18,19,20,24,25,26.
3. Production de culture BCG
NOTE : Pour les études in vivo des vaccins antituberculeux, l’objectif est d’améliorer l’efficacité du BCG. Par conséquent, les groupes vaccinés par le BCG sont généralement utilisés comme témoin. Les souches de BCG utilisées pour la vaccination humaine sont idéales pour les tests sur des modèles animaux. Dans ce cas, une culture du BCG doit être reconstituée selon les instructions du fournisseur27. Cependant, une culture BCG pour les études in vivo peut également être produite en interne11. La production d’une culture de BCG unicellulaire, uniforme et de haute qualité pour les protocoles d’infection in vitro a été produite avec beaucoup de succès dans plusieurs études 11,16,18,19,20,26,28,29, en utilisant le protocole suivant, qui peut également être utilisé pour les études de provocation animale.
- Culture 50 mL de BCG dans un bouillon 7H9, à 37 °C, avec agitation à 200 tr/min. Variez le volume en fonction des besoins de l’expérience.
- Chaque jour, pendant 8 à 10 jours, prélever 100 μL de la culture et la diluer en ajoutant 900 μL de PBS dans une cuvette de 1 mL. Procédez ensuite en mesurant la densité optique des bactéries (OD à λ=600 nm ; OD600) dans un spectrophotomètre. Tracez une courbe de croissance à partir de ces valeurs. Identifiez la phase mi-logarithmique de la culture (lorsque la DO double de façon constante par unité de temps).
- Préparer une culture subséquente et incuber jusqu’à ce qu’elle atteigne la phase de croissance logarithmique / tardive comme aux étapes 3.1 et 3.2. Utilisez les valeurs obtenues à l’étape précédente à titre indicatif. Assurez-vous que la culture n’atteint pas la phase de croissance stationnaire (lorsque la DO commence à se stabiliser) pour maintenir une culture de bonne qualité de bactéries viables.
- Récoltez la culture à la phase de croissance des logarithmes au milieu ou à la fin de la phase de croissance. Centrifuger à 3000 x g pendant 10 min. Retirez le surnageant.
- Ajouter 10 mL de PBS pour éliminer les bactéries. Centrifuger à 3000 x g pendant 10 min. Retirez le surnageant.
- Remettre la bactérie en suspension avec 5 mL de milieu d’infection. Placez le tube dans un bain à ultrasons pendant 15 min, à pleine puissance à 80 Hz.
- Centrifuger à 1000 x g pendant 10 min. Récupérez le surnageant en évitant la pastille car elle est riche en amas bactériens qui doivent être évités dans une culture BCG de haute qualité et jetez-la.
- Mesurez la DO du surnageant. Ici, les cultures en phase de croissance exponentielle, avec un OD600 de 0,1, sont équivalentes à 1 x 107 UFC/mL.
NOTE : Chaque laboratoire doit produire ses propres courbes de croissance du BCG avant de commencer les expériences afin d’établir une régression linéaire entre OD600 et CFU à l’aide du spectrophotomètre. Veuillez noter que les spectrophotomètres ont des distances de trajet de la lumière différentes, ce qui peut faire varier les lectures obtenues pour un même échantillon. - Effectuez des calculs simples pour établir le nombre de bactéries à ajouter à chaque culture de cellules hôtes. Le nombre de bactéries par cellule hôte est la multiplicité d’infection (MOI). Utilisez un moment d’inertie de 10 bactéries par cellule hôte, qui est le moment d’inertie le plus couramment utilisé pour les expériences d’infection par le BCG.
4. Essai de l’unité formant des micro-colonies
REMARQUE : Une fois l’expérience d’infection in vivo ou in vitro terminée, le dénombrement des bactéries peut être effectué par mCFU. Pour les études in vivo , les échantillons doivent d’abord être homogénéisés dans un batteur à billes ou un autre homogénéisateur de tissus. Pour les cultures in vitro de macrophages, de cellules dendritiques ou de neutrophiles infectés par le BCG, les échantillons doivent être lysés à l’aide d’un détergent non ionique (p. ex., solution à 0,05 % de détergent non ionique non dénaturant).
- Dilutions en série à l’aide d’une plaque à 96 puits : effectuer des dilutions en série de 10 fois des lysats, dans une plaque stérile à 96 puits selon le schéma de la figure 1A. Répartissez les lysats sur les rangs A et E. Pour chaque plaque, le nombre maximum d’échantillons et/ou de répétitions est de 24.
- Ajouter 180 μL de dH2O dans les puits restants pour effectuer la dilution en série.
- À l’aide d’une pipette à 12 canaux, remettre en suspension les lysats de la rangée A et transférer 20 μL dans la rangée B (lysat de 20 μL + 180 μL dH2O). Bien homogénéiser. Répétez cette étape séquentiellement pour les rangs B et C jusqu’à ce que vous atteigniez la dernière dilution du rang D.
REMARQUE : Nous effectuons généralement trois dilutions (100, 101, 102, 103), utilisant ainsi 4 rangées de la plaque (A-D ou E-H) pour chaque ensemble de 12 échantillons et/ou répétitions. - Placage par microgouttelettes : utiliser une pipette multicanaux de 0,5 à 10 μL (de préférence à pointes fines) pour transférer 5 μL de chaque rangée de la plaque à 96 puits vers la plaque carrée moyenne solide, conformément à la figure 1B.
- Tout en pipetant lentement les gouttelettes de 5 μL, laissez-les toucher légèrement la gélose. Cela aidera à faire décoller la gouttelette de l’embout vers la gélose et à réduire la possibilité de rétention du liquide à l’intérieur de l’embout.
- Laissez sécher les gouttelettes, fermez la plaque de gélose et incubez-la à 37 °C tout en surveillant la croissance bactérienne. Si vous le souhaitez, incubez les plaques de gélose dans un sac en plastique scellé pour éviter qu’elles ne sèchent.
- Dénombrement des microcolonies : après environ 6 à 10 jours d’incubation, vérifier la présence de colonies individuelles, visibles à l’œil nu (figure 2).
- Comptez les colonies à l’aide de l’objectif de grossissement le plus faible (4x ou moins) d’un microscope optique inversé ou d’une loupe. Les dénombrements doivent être effectués dans les dilutions où le nombre de colonies est inférieur à 300 et supérieur à 30. Vous pouvez également utiliser un appareil photo pour prendre une photo de la gouttelette afin de compter manuellement les colonies sur l’ordinateur ou utiliser un logiciel tel qu’ImageJ pour automatiser le comptage des colonies.
- Pour exprimer le nombre de cellules en UFC/mL, utilisez l’équation suivante :
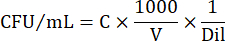
Où C = nombre de colonies dénombrées, V = volume plaqué en μL et Dil = dilution là où les colonies ont été comptées (100, 101, 102, 103). Par exemple, si 30 colonies ont été dénombrées dans une gouttelette de 5 μL en dilution 102, alors :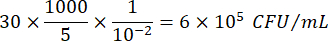
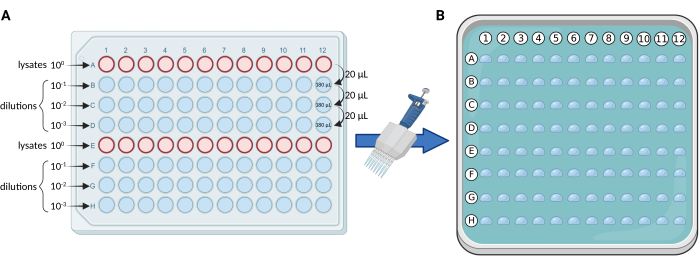
Graphique 1. Représentation schématique du protocole mCFU. (A) Dilutions en série 10 fois des lysats contenant du BCG dans une plaque à 96 puits. (B) Boîte de Pétri carrée contenant un milieu de culture solide et recouverte de 96 gouttelettes de 5 μL chacune. Les gouttelettes sont pipetées directement à partir de la plaque à 96 puits à l’aide d’une pipette multicanaux. Créé avec BioRender.com. Veuillez cliquer ici pour voir une version agrandie de cette figure.
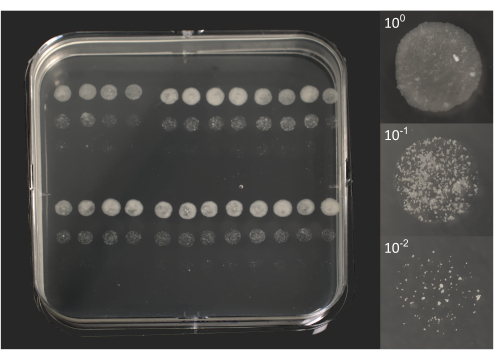
Graphique 2. Unités formant des micro-colonies de BCG après 10 jours d’incubation. À gauche, une photo d’une boîte de Pétri carrée recouverte de 96 gouttelettes de 5 μL chacune, comme le montre la figure 1B. À droite, des photos individuelles de 3 gouttelettes correspondent à un lysat original (100) et à deux dilutions (101, 102). Les photos ont été prises à l’aide d’un appareil photo DSRL équipé d’un objectif zoom 18-55 mm (plaque) ou d’un objectif macro 105 mm (gouttelettes). Veuillez cliquer ici pour voir une version agrandie de cette figure.
5. Comptage des unités formant des micro-colonies aux Fidji (ImageJ)
REMARQUE : La méthode mCFU permet la quantification CFU de grands ensembles d’échantillons. Des photos des gouttelettes peuvent être enregistrées pour une analyse a posteriori afin de faciliter le dénombrement des colonies. Plusieurs appareils photographiques permettent de produire des images d’une qualité suffisante à cet effet. Il s’agit notamment d’appareils photo numériques, de webcams, de microscopes et de loupes fixés à des caméras, ainsi que de téléphones portables. Un logiciel gratuit d’analyse d’images tel qu’ImageJ offre la possibilité de compter manuellement ou automatiquement les colonies dans ces images. Pour démontrer les deux méthodes, Fiji sera utilisé, qui est une distribution d’ImageJ qui regroupe plusieurs outils pour l’analyse scientifique d’images30. Fidji peut être téléchargé à partir de https://fiji.sc/.
- Méthode de comptage manuel
- Ouvrez l’image contenant le mCFU aux Fidji. Sélectionnez Plugins > Analyser > compteur de cellules.
- Dans le menu Compteur de cellules, sélectionnez Initialiser , puis sélectionnez un compteur (par exemple, Type 1).
- Procédez en cliquant sur chaque colonie. Chaque clic sera affiché sur l’image et mettra à jour le compteur (Figure 3). Pour annuler les clics accidentels, sélectionnez Supprimer.
- Enregistrez la valeur affichée sur le compteur. Cliquez sur le bouton Réinitialiser pour réinitialiser le comptage et ouvrir une nouvelle image pour compter des échantillons supplémentaires.
REMARQUE : De plus amples instructions sur ce plugin peuvent être trouvées à https://imagej.net/plugins/cell-counter.
- Méthode de comptage automatisée
- Ouvrez l’image contenant le mCFU aux Fidji. Sélectionnez Image > Tapez > 8 bits. Cela convertira l’image en une image en niveaux de gris 8 bits.
- Sélectionnez l’outil Ovale dans la barre d’outils et dessinez un ovale autour de la zone où se trouvent les colonies (Figure 4A). L’ovale peut être ajusté après avoir été dessiné.
- Sélectionnez Edit > Clear Outside (Modifier effacer l’extérieur) pour supprimer toute interférence de la zone extérieure (Figure 4B). Sélectionnez Image > Ajuster > seuil.
- Déplacez les curseurs dans le menu Seuil jusqu’à ce que les colonies apparaissent en rouge et que le bruit de fond soit réduit au minimum (Figure 4C).
- Sélectionnez Appliquer et quittez la fenêtre de seuil. Une image en noir et blanc est générée (Figure 4D).
- Sélectionnez Analyser > Analyser les particules. Dans la fenêtre d’analyse des particules, spécifiez la plage de la zone de la colonie (entre 1 et l’infini, mesurée en pixels carrés) et de la circularité (entre 0 et 1, où 1 est un cercle parfait ; Graphique 4E).
- Sélectionnez Contours dans le menu contextuel Afficher. Cochez la case Afficher les résultats pour obtenir des mesures détaillées pour chaque colonie dans la fenêtre des résultats. Cochez la case Effacer les résultats pour effacer toutes les mesures précédentes. Cochez la case Summarize (Récapituler ) pour afficher les résultats résumés des mesures (Figure 4E).
- Lancez l’analyseur en sélectionnant OK. Une nouvelle fenêtre apparaît, affichant toutes les colonies délimitées qui ont été détectées et comptées. La fenêtre des résultats affiche les détails de chaque colonie, et la fenêtre des résultats résumés montre le nombre total de colonies dénombrées (Figure 4F).
REMARQUE : Les paramètres de taille et de circularité varient en fonction de la résolution et du grossissement de l’image, ainsi que de la taille et de la forme des colonies. Répétez le processus plusieurs fois jusqu’à ce que les meilleurs paramètres soient trouvés pour détecter toutes les colonies. Vous trouverez de plus amples instructions sur le plugin d’analyse des particules à l’adresse https://imagej.nih.gov/ij/docs/menus/analyze.html#ap.
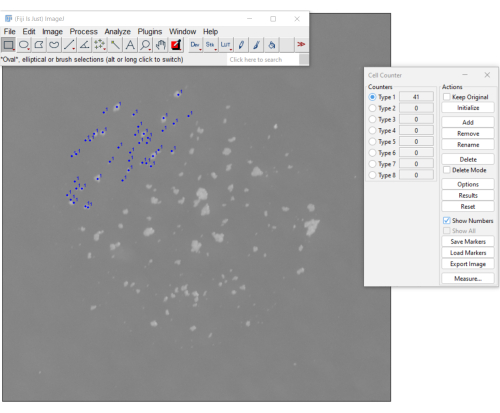
Graphique 3. Une méthode manuelle pour compter les mCFU à l’aide du plug-in de compteur de cellules sur le logiciel Fiji. Les points bleus indiquent les colonies sur lesquelles l’utilisateur a déjà cliqué. Le menu de droite affiche le nombre de colonies dénombrées jusqu’à présent (le décompte est de 41). Veuillez cliquer ici pour voir une version agrandie de cette figure.

Graphique 4. Méthode automatisée de comptage des mCFU à l’aide d’un logiciel fidjien. (A, B) La zone d’intérêt avec les colonies est sélectionnée à l’aide de l’outil de sélection ovale, et la zone extérieure est supprimée à l’aide de la commande Effacer l’extérieur. (C, D) Une image en noir et blanc des colonies est générée à l’aide de l’outil de seuil. (E, F) Le nombre de colonies est quantifié à l’aide de l’outil Analyser les particules. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Résultats
Le test mCFU décrit ici augmente la quantité d’informations pouvant être récupérées à partir d’une seule boîte de Pétri à au moins 96 fois. La figure 5 illustre une comparaison de deux méthodes d’administration de médicaments pour l’utilisation réorientée du saquinavir (SQV)31,32 comme médicament dirigé contre l’hôte pour traiter la tuberculose. Dans ce test, quatre souches différentes de Mycobacterium...
Discussion
La tuberculose est un problème de santé publique important qui prend de plus en plus d’importance, en particulier dans les pays à revenu faible et intermédiaire. La perturbation des établissements de santé pour diagnostiquer et traiter la tuberculose pendant la pandémie de COVID-19 a eu un impact négatif sur l’incidence des nouveaux cas1. De plus, il est urgent de s’attaquer aux souches multirésistantes et très résistantes de M.tb, ainsi qu’à la co-infection de M.t...
Déclarations de divulgation
DP et PJGB déclarent que l’étude a été menée en l’absence de toute relation commerciale ou financière qui pourrait être interprétée comme un conflit d’intérêts potentiel.
Remerciements
Ces travaux ont été financés par des fonds internes de la Faculté de médecine de l’Universidade Católica Portuguesa et des financements externes de la Fundação para a Ciência e a Tecnologia (FCT), dans le cadre des subventions UIDP/04279/2020, UIDB/04279/2020 et EXPL/SAU-INF/0742/2021.
matériels
| Name | Company | Catalog Number | Comments |
| 96-well plates | VWR | 734-2781 | |
| DSLR 15-55 mm lens | Nikon | AF-P DX NIKKOR 18-55mm f/3.5-5.6G VR | |
| DSLR camera | Nikon | D3400 | |
| DSLR macro lens | Sigma | MACRO 105mm F2.8 EX DG OS HSM | |
| Fetal calf serum | Gibco | 10270106 | |
| Fiji Software | https://fiji.sc/ | Fiji is an open-source software supported by several laboratories, institutions, and individuals. All the required plugins are included. | |
| Igepal CA-630 | Sigma-Aldrich | 18896 | |
| L-glutamine | Gibco | 25030-081 | |
| Middlebrook 7H10 | BD | 262710 | |
| Middlebrook 7H9 | BD | 271310 | |
| Multichannel pipette (0.5 - 10 µl) | Gilson | FA10013 | |
| Multichannel pipette (20 - 200 µl) | Gilson | FA10011 | |
| Mycobacterium bovis BCG | American Type Culture Collection | ATCC35734 | strain TMC 1011 [BCG Pasteur] |
| OADC enrichment | BD | 211886 | |
| Phosphate buffered saline (PBS) | NZYTech | MB25201 | |
| RPMI 1640 medium | Gibco | 21875091 | |
| Sodium pyruvate | Gibco | 11360-070 | |
| Spectrophotometer UV-6300PC | VWR | 634-6041 | |
| Square Petri dish 120 x 120 mm | Corning | BP124-05 | |
| Tyloxapol | Sigma-Aldrich | T8761 | |
| Ultrasound bath Elma P 30 H | VWR | 142-0051 |
Références
- World Health Organization. . Global Tuberculosis Report 2022. , (2022).
- Bettencourt, P. J. G., Joosten, S. A., Lindestam Arlehamn, C. S., Behr, M. A., Locht, C., Neyrolles, O. 100 years of the Bacillus Calmette-Guérin vaccine. Vaccine. 39 (50), 7221-7222 (2021).
- Bettencourt, P. J. G. The 100th anniversary of bacille Calmette-Guérin (BCG) and the latest vaccines against COVID-19. The International Journal of Tuberculosis and Lung Disease. 25 (8), 611-613 (2021).
- Scriba, T. J., Netea, M. G., Ginsberg, A. M. Key recent advances in TB vaccine development and understanding of protective immune responses against Mycobacterium tuberculosis. Seminars in Immunology. 50, 101431 (2020).
- McShane, H., Williams, A. A review of preclinical animal models utilised for TB vaccine evaluation in the context of recent human efficacy data. Tuberculosis. 94 (2), 105-110 (2014).
- Voss, G., et al. Progress and challenges in TB vaccine development. F1000Research. 7, 199 (2018).
- Satti, I., McShane, H. Current approaches toward identifying a correlate of immune protection from tuberculosis. Expert Review of Vaccines. 18 (1), 43-59 (2019).
- Young, D. Animal models of tuberculosis. European Journal of Immunology. 39 (8), 2011-2014 (2009).
- Pedroza-Roldán, C., Flores-Valdez, M. A. Recent mouse models and vaccine candidates for preventing chronic/latent tuberculosis infection and its reactivation. Pathogens and disease. 75 (6), (2017).
- Gong, W., Liang, Y., Wu, X. Animal Models of Tuberculosis Vaccine Research: An Important Component in the Fight against Tuberculosis. BioMed Research International. 2020, 1-21 (2020).
- Bettencourt, P., et al. Identification of antigens presented by MHC for vaccines against tuberculosis. NPJ vaccines. 5 (1), 2 (2020).
- Moorlag, S. J. C. F. M., Arts, R. J. W., van Crevel, R., Netea, M. G. Non-specific effects of BCG vaccine on viral infections. Clinical Microbiology and Infection. 25 (12), 1473-1478 (2019).
- Wilkie, M., et al. Functional in-vitro evaluation of the non-specific effects of BCG vaccination in a randomised controlled clinical study. Scientific Reports. 12 (1), 7808 (2022).
- Netea, M. G., et al. Trained immunity: A program of innate immune memory in health and disease. Science. 352 (6284), aaf1098 (2016).
- Bettencourt, P., Pires, D., Carmo, N., Anes, E. Application of Confocal Microscopy for Quantification of Intracellular Mycobacteria in Macrophages. Microscopy: Science, Technology, Applications and Education. 1, 614-621 (2010).
- Bettencourt, P., Carmo, N., Pires, D., Timóteo, P., Anes, E. Mycobacterial infection of macrophages: the effect of the multiplicity of infection. Antimicrobial research: Novel bioknowledge and educational programs. , 651-664 (2017).
- Pires, D., Bettencourt, P., Carmo, N., Niederweis, M., Anes, E. Role of Mycobacterium tuberculosis outer-membrane porins in bacterial survival within macrophages. Drug Discovery Today. 15 (23-24), 1112-1113 (2010).
- Pires, D., et al. Mycobacterium tuberculosis Modulates miR-106b-5p to Control Cathepsin S Expression Resulting in Higher Pathogen Survival and Poor T-Cell Activation. Frontiers in immunology. 8 (DEC), 1819 (2017).
- Pires, D., et al. Role of Cathepsins in Mycobacterium tuberculosis Survival in Human Macrophages. Scientific reports. 6 (August), 32247 (2016).
- Bettencourt, P., et al. Actin-binding protein regulation by microRNAs as a novel microbial strategy to modulate phagocytosis by host cells: the case of N-Wasp and miR-142-3p. Frontiers in cellular and infection microbiology. 3 (June), 19 (2013).
- Bhuyan, S., et al. Microliter spotting and micro-colony observation: A rapid and simple approach for counting bacterial colony forming units. Journal of Microbiological Methods. 207, 106707 (2023).
- Jackson, S., McShane, H. Challenges in Developing a Controlled Human Tuberculosis Challenge Model. Current topics in microbiology and immunology. , 1-27 (2022).
- Darrah, P. A., et al. Prevention of tuberculosis in macaques after intravenous BCG immunization. Nature. 577 (7788), 95-102 (2020).
- Madura Larsen, J., et al. BCG stimulated dendritic cells induce an interleukin-10 producing T-cell population with no T helper 1 or T helper 2 bias in vitro. Immunology. 121 (2), 276-282 (2007).
- Bickett, T. E., et al. Characterizing the BCG-Induced Macrophage and Neutrophil Mechanisms for Defense Against Mycobacterium tuberculosis. Frontiers in immunology. 11, 1202 (2020).
- Pires, D., et al. Interference of Mycobacterium tuberculosis with the endocytic pathways on macrophages and dendritic cells from healthy donors: role of cathepsins. Drug Discovery Today. 15 (23-24), 1112-1112 (2010).
- Betts, G., et al. Optimising Immunogenicity with Viral Vectors: Mixing MVA and HAdV-5 Expressing the Mycobacterial Antigen Ag85A in a Single Injection. PLoS ONE. 7 (12), e50447 (2012).
- Tanner, R., et al. The influence of haemoglobin and iron on in vitro mycobacterial growth inhibition assays. Scientific reports. 7 (1), 43478 (2017).
- McNeill, E., et al. Regulation of mycobacterial infection by macrophage Gch1 and tetrahydrobiopterin. Nature communications. 9 (1), 5409 (2018).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Pereira, M., Vale, N. Saquinavir: From HIV to COVID-19 and Cancer Treatment. Biomolecules. 12 (7), 944 (2022).
- Pires, D., et al. Repurposing Saquinavir for Host-Directed Therapy to Control Mycobacterium Tuberculosis Infection. Frontiers in immunology. 12, 647728 (2021).
- Pires, D., et al. Liposomal Delivery of Saquinavir to Macrophages Overcomes Cathepsin Blockade by Mycobacterium tuberculosis and Helps Control the Phagosomal Replicative Niches. International journal of molecular sciences. 24 (2), (2023).
- Maartens, G., Wilkinson, R. J. Tuberculosis. The Lancet. 370 (9604), 2030-2043 (2007).
- Matarazzo, L., Bettencourt, P. J. G. mRNA vaccines: a new opportunity for malaria, tuberculosis and HIV. Frontiers in Immunology. 14, 1172691 (2023).
- Young, D., Dye, C. The Development and Impact of Tuberculosis Vaccines. Cell. 124 (4), 683-687 (2006).
- Kommareddi, S., Abramowsky, C. R., Swinehart, G. L., Hrabak, L. Nontuberculous mycobacterial infections: Comparison of the fluorescent auramine-o and Ziehl-Neelsen techniques in tissue diagnosis. Human Pathology. 15 (11), 1085-1089 (1984).
- Sabiiti, W., et al. A Tuberculosis Molecular Bacterial Load Assay (TB-MBLA). Journal of visualized experiments: JoVE. (158), e60460 (2020).
- Somoskövi, A., et al. Comparison of Recoveries of Mycobacterium tuberculosis Using the Automated BACTEC MGIT 960 System, the BACTEC 460 TB System, and Löwenstein-Jensen Medium. Journal of Clinical Microbiology. 38 (6), 2395-2397 (2000).
- Tanner, R., et al. The in vitro direct mycobacterial growth inhibition assay (MGIA) for the early evaluation of TB vaccine candidates and assessment of protective immunity: a protocol for non-human primate cells. F1000Research. 10, 257 (2021).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon