É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Ensaio de Unidade Formadora de Microcolônias para Avaliação da Eficácia de Vacinas Contra Tuberculose
Neste Artigo
Resumo
A determinação de unidades formadoras de colônias (UFC) é a técnica padrão-ouro para quantificar bactérias, incluindo o Mycobacterium tuberculosis , que pode levar semanas para formar colônias visíveis. Aqui descrevemos uma micro-UFC para determinação de UFC com maior eficiência de tempo, redução do espaço de laboratório e custo do reagente, e escalabilidade para experimentos de médio e alto rendimento.
Resumo
A tuberculose (TB), principal causa de morte no mundo por um agente infeccioso, matou 1,6 milhão de pessoas em 2022, só sendo superada pela Covid-19 durante a pandemia de 2019-2021. A doença é causada pela bactéria Mycobacterium tuberculosis (M.tb). A cepa de Mycobacterium bovis Bacillus Calmette-Guérin (BCG), única vacina contra a tuberculose, é a mais antiga vacina licenciada no mundo, ainda em uso. Atualmente, há 12 vacinas em testes clínicos e dezenas de vacinas em desenvolvimento pré-clínico. O método de escolha para avaliar a eficácia das vacinas contra TB em estudos pré-clínicos é a enumeração de colônias bacterianas pelo ensaio de unidades formadoras de colônias (UFC). Este ensaio demorado leva de 4 a 6 semanas para ser concluído, requer espaço substancial no laboratório e na incubadora, tem altos custos de reagentes e é propenso a contaminação. Aqui descrevemos um método otimizado para enumeração de colônias, a micro-UFC (mCFU), que oferece uma solução simples e rápida para analisar os resultados de eficácia da vacina M.tb . O ensaio de mCFU requer dez vezes menos reagentes, reduz o período de incubação três vezes, levando de 1 a 2 semanas para ser concluído, reduz o espaço do laboratório e o custo do reagente e minimiza os riscos de saúde e segurança associados ao trabalho com um grande número de M.tb. Além disso, para avaliar a eficácia de uma vacina contra a TB, amostras podem ser obtidas de uma variedade de fontes, incluindo tecidos de animais vacinados infectados com micobactérias. Também descrevemos um método otimizado para produzir uma cultura de micobactérias unicelular, uniforme e de alta qualidade para estudos de infecção. Finalmente, propomos que esses métodos sejam universalmente adotados para estudos pré-clínicos de determinação da eficácia vacinal, levando à redução do tempo no desenvolvimento de vacinas contra TB.
Introdução
A tuberculose (TB) é a principal causa de morte no mundo por um único agente infeccioso, a bactéria Mycobacterium tuberculosis (M.tb), matando mais pessoas do que qualquer outro patógeno. Em 2021, a tuberculose foi responsável por 1,6 milhão de mortes e foi superada pela Covid-19 durante a pandemia1 de 2019-2021. Além disso, de acordo com o relatório global de TB da Organização Mundial da Saúde de 2022, a pandemia de COVID-19 foi responsável por um aumento de novos casos de TB. A OMS também relata grandes quedas no número de pessoas diagnosticadas com TB nesse período, o que poderia aumentar ainda mais o número de casos de TB1.
O Bacillus Calmette-Guérin (BCG) é uma cepa viva atenuada do patogênico Mycobacterium bovis, utilizada pela primeira vez como vacina há mais de 100 anos. Esta é a única vacina contra a TB e é a mais antiga vacina licenciada no mundo ainda em uso 2,3. Atualmente, existem 12 vacinas em diferentes fases de ensaios clínicos4, e dezenas de vacinas estão em desenvolvimento pré-clínico 5,6. A avaliação pré-clínica das vacinas contra TB inclui a avaliação da segurança e imunogenicidade7, que podem ser obtidas em diversos modelos animais, como peixes-zebra, camundongos, cobaias, coelhos, bovinos e primatas não humanos8,9,10. Além disso, avaliar a capacidade de uma vacina em induzir proteção contra a infecção e/ou transmissão da M.tb, ou seja, a eficácia da vacina, requer um desafio M.tb in vivo 5,11. Curiosamente, a vacinação BCG induz efeitos inespecíficos que afetam a sobrevivência de outros patógenos bacterianos e virais12,13 através do mecanismo de imunidade treinada14. Para quantificar a carga bacteriana viável em um animal infectado, o método de escolha é a enumeração de colônias bacterianas através do ensaio de unidades formadoras de colônias (UFC)5,15. A UFC é uma unidade que estima o número de microrganismos (bactérias ou fungos) que formam colônias sob condições específicas de crescimento. As UFCs originam-se de microrganismos viáveis e replicativos, e o número absoluto de microrganismos vivos dentro de cada colônia é difícil de estimar. É incerto se uma colônia se originou de um ou mais microrganismos. A unidade UFC reflete essa incerteza, portanto, uma grande variabilidade pode ser observada em réplicas de uma mesma amostra. Este ensaio demorado requer técnicos especializados treinados para trabalhar em uma instalação de nível de biossegurança 3 (BSL3), espaço substancial de laboratório e incubadora, leva de 4 a 6 semanas para ser concluído e é propenso a contaminação.
Neste estudo, descrevemos um método otimizado para enumeração de colônias, a micro-UFC (mUFC), e oferecemos uma solução simples e rápida para analisar os resultados 15,16,17,18,19,20. O ensaio de mCFU requer dez vezes menos reagentes, reduz o período de incubação três vezes, levando de 1 a 2 semanas para ser concluído, reduz o espaço do laboratório e o custo do reagente e minimiza os riscos de saúde e segurança associados ao trabalho com um grande número de M.tb. Propomos que esse método seja universalmente adotado para estudos pré-clínicos de determinação da eficácia vacinal, levando à redução do tempo no desenvolvimento de vacinas contra TB. Finalmente, este método otimizado de enumeração de UFC tem sido utilizado para quantificar não só micobactérias, mas também outras bactérias, como Escherichia coli e Ralstonia solanacearum21.
Access restricted. Please log in or start a trial to view this content.
Protocolo
NOTA: O protocolo descrito aqui é para BCG, mas pode ser aplicado a qualquer micobactéria. A BCG pode ser usada como bactéria substituta para experimentos de TB quando os recursos de BSL3 não estão disponíveis22. Os procedimentos a seguir usando BCG devem ser realizados em um laboratório de nível de biossegurança 2 (BSL2) e seguir as diretrizes de biossegurança apropriadas e boas práticas de laboratório para a manipulação de microrganismos do grupo de risco 2.
1. Preparação dos meios de cultura
- Preparar caldo Middlebrook 7H9 suplementado com 10% (v/v) de ácido oleico, albumina, dextrose e enriquecimento com catalase (OADC), de acordo com as instruções do fornecedor. Completar o caldo com 0,05% (v/v) de tiloxapol.
NOTA: O tiloxapol é um polímero líquido não iônico que tem sido usado como tensoativo para prevenir a formação de aglomerados bacterianos16. - Preparar meio sólido Middlebrook 7H10 suplementado com 10% (v/v) de enriquecimento OADC de acordo com as instruções do fornecedor.
- Distribuir 40 mL de meio por placa de Petri quadrada (120 mm x 120 mm). Deixe as placas secarem para minimizar a condensação na superfície do ágar.
NOTA: Este tamanho específico da placa de Petri é fundamental para permitir a transposição direta de pelo menos 96 gotículas de uma placa de 96 poços. A secagem eficaz das placas facilitará posteriormente o revestimento de pequenas gotículas de suspensão bacteriana e impedirá que as gotículas se espalhem. - Prepare o Roswell Park Memorial Institute Medium (RPMI 1640) ou o Dulbecco's Modified Eagle Medium (DMEM) para produzir o meio de infecção. Em ambos os casos, suplemente o meio com 10% de soro fetal para bezerros, 1% de L-glutamina e 1 mM de piruvato de sódio. Não adicione penicilina e estreptomicina ao meio.
2. Preparo da amostra
- Obter amostras de uma variedade de fontes. Normalmente, para quantificar UFC para avaliar a eficácia de uma vacina contra a TB, adquira amostras de tecidos animais vacinados e não vacinados. Por exemplo, pulmão e baço de camundongo11 ou pulmão de macaco, linfonodos torácicos e periféricos, baço, fígado, pele, sangue, medula óssea e lavagem do lavado broncoalveolar23. Alternativamente, obter amostras de culturas in vitro de macrófagos/células dendríticas/neutrófilos infectados com BCG 18,19,20,24,25,26.
3. Produção da cultura BCG
NOTA: Para estudos in vivo de vacinas contra a TB, o objetivo é melhorar a eficácia da BCG. Portanto, os grupos vacinados com BCG são geralmente usados como controle. As cepas de BCG utilizadas para vacinação humana são ideais para testes em modelos animais. Nesse caso, uma cultura de BCG deve ser reconstituída de acordo com as instruções do fornecedor27. No entanto, uma cultura de BCG para estudos in vivo também pode ser produzida internamente11. A produção de cultura de BCG unicelular, uniforme e de alta qualidade para protocolos de infecção in vitro tem sido produzida com muito sucesso em vários estudos11,16,18,19,20,26,28,29, utilizando o seguinte protocolo, que também pode ser utilizado para estudos de desafio animal.
- Cultivo de 50 mL de BCG em caldo 7H9, a 37 °C, com agitação a 200 rpm. Varie o volume de acordo com as necessidades do experimento.
- Todos os dias, por 8-10 dias, coletar 100 μL da cultura e diluí-la adicionando 900 μL de PBS em uma cubeta de 1 mL. Em seguida, proceda medindo a densidade óptica das bactérias (OD em λ=600 nm; OD600) em espectrofotômetro. Desenhe uma curva de crescimento a partir desses valores. Identifique a fase de log médio da cultura (quando o DO está dobrando consistentemente por unidade de tempo).
- Preparar uma cultura subsequente e incubar até atingir a fase de crescimento logarítmico médio/tardio, como nos passos 3.1 e 3.2. Use os valores obtidos na etapa anterior como orientação. Certifique-se de que a cultura não atinja a fase estacionária de crescimento (quando o DO começa a estabilizar) para manter uma cultura de boa qualidade de bactérias viáveis.
- Coletar a cultura na fase de crescimento torarítmico médio/tardio. Centrifugar a 3000 x g por 10 min. Remova o sobrenadante.
- Adicione 10 mL de PBS para lavar as bactérias. Centrifugar a 3000 x g por 10 min. Remova o sobrenadante.
- Ressuspender a bactéria com 5 mL de meio de infecção. Coloque o tubo em banho de ultrassom por 15 min, potência máxima a 80 Hz.
- Centrifugar a 1000 x g por 10 min. Coletar o sobrenadante evitando o pellet, pois é rico em aglomerados bacterianos que devem ser evitados em uma cultura de BCG de alta qualidade e descartá-lo.
- Meça o DO do sobrenadante. Aqui, culturas na fase de crescimento exponencial, com OD600 de 0,1, equivalem a 1 x 107 UFC/mL.
NOTA: Cada laboratório deve produzir suas próprias curvas de crescimento de BCG antes de iniciar experimentos para estabelecer uma regressão linear entre OD600 e UFC usando o espectrofotômetro. Por favor, note que os espectrofotômetros têm diferentes distâncias de trajetória de luz, o que pode variar as leituras obtidas para a mesma amostra. - Realizar cálculos simples para estabelecer o número de bactérias a serem adicionadas a cada cultura de células hospedeiras. O número de bactérias por célula hospedeira é a Multiplicidade de Infecção (MOI). Use um MOI de 10 bactérias por célula hospedeira, que é o MOI mais comum usado para experimentos de infecção por BCG.
4. Ensaio da unidade formadora de microcolônias
NOTA: Após a conclusão de um experimento de infecção in vivo ou in vitro , a enumeração de bactérias pode ser realizada por mCFU. Para estudos in vivo , as amostras devem ser primeiro homogeneizadas em um batedor de contas ou outro homogeneizador de tecido. Para culturas in vitro de macrófagos/células dendríticas/neutrófilos infectados com BCG, as amostras devem ser lisadas usando um detergente não iônico (por exemplo, solução a 0,05% de detergente não iônico e não desnaturante).
- Diluições seriadas usando uma placa de 96 poços: realizar diluições seriadas de 10 vezes dos lisados, em uma placa estéril de 96 poços de acordo com o esquema da Figura 1A. Distribua os lisados nas linhas A e E. Para cada placa, o número máximo de amostras e/ou repetições é de 24.
- Adicionar 180 μL de dH2O aos poços restantes para realizar a diluição seriada.
- Usando uma pipeta de 12 canais, ressuspenda os lisados na linha A e transfira 20 μL para a linha B (20 μL lisado + 180 μL dH2O). Homogeneizar bem. Repetir sequencialmente este passo para as linhas B e C até atingir a última diluição na linha D.
OBS: Costumamos realizar três diluições (100, 101, 102, 103), utilizando assim 4 fileiras da placa (A-D ou E-H) para cada conjunto de 12 amostras e/ou repetições. - Microplaqueamento: utilizar uma pipeta multicanal de 0,5-10 μL (pontas finas são preferidas) para transferir 5 μL de cada fileira da placa de 96 poços para a placa quadrada média sólida, de acordo com a Figura 1B.
- Enquanto pipeta lentamente as gotículas de 5 μL, deixe-as tocar ligeiramente o ágar. Isso ajudará a tirar a gotícula da ponta em direção ao ágar e reduzir a possibilidade de retenção do líquido dentro da ponta.
- Deixe as gotículas secarem, feche a placa de ágar e incube-a a 37 °C enquanto monitora o crescimento bacteriano. Opcionalmente, incube as placas de ágar em um saco plástico selado para evitar que as placas sequem.
- Contagem de microcolônias: após aproximadamente 6-10 dias de incubação, verificar se há colônias individuais, visíveis a olho nu (Figura 2).
- Conte as colônias usando a objetiva de ampliação mais baixa (4x ou inferior) de um microscópio óptico invertido ou lupa. As contagens devem ser realizadas nas diluições em que o número de colônias seja inferior a 300 e superior a 30. Como alternativa, use uma câmera para tirar uma foto da gota para contar manualmente as colônias no computador ou use um software como o ImageJ para automatizar a contagem de colônias.
- Para expressar números de células em UFC/mL, use a seguinte equação:
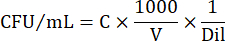
Onde C = número de colônias contadas, V = volume plaqueado em μL e Dil = diluição onde as colônias foram contadas (100, 101, 102, 103). Por exemplo, se 30 colônias foram contadas em uma gota de 5 μL em diluição 102, então: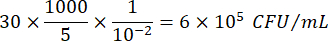
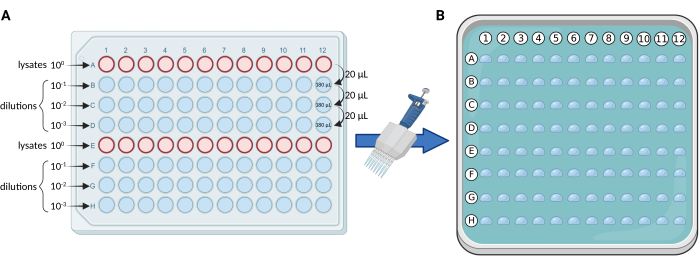
Gráfico 1. Representação esquemática do protocolo mUFC. (A) Diluições seriadas de 10 vezes dos lisados contendo BCG em uma placa de 96 poços. (B) Placa de Petri quadrada contendo meio de cultura sólido e sobreposta por 96 gotículas de 5 μL cada. As gotículas são pipetadas diretamente da placa de 96 poços usando uma pipeta multicanal. Criado com BioRender.com. Clique aqui para ver uma versão maior desta figura.
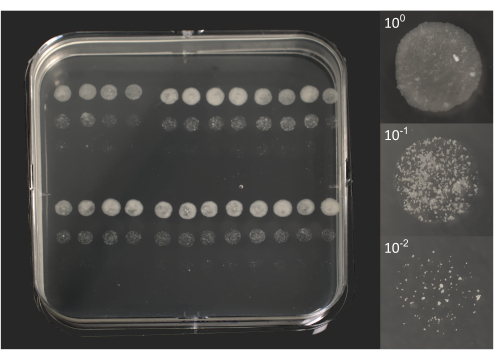
Gráfico 2. Unidades formadoras de microcolônias de BCG após 10 dias de incubação. À esquerda, uma foto de uma placa de Petri quadrada sobreposta por 96 gotículas de 5 μL cada, conforme representado anteriormente na Figura 1B. À direita, fotos individuais de 3 gotículas correspondem a um lisado original (100) e duas diluições (101, 102). As fotos foram tiradas usando uma câmera DSRL equipada com uma lente zoom de 18-55 mm (placa) ou uma lente macro de 105 mm (gotículas). Clique aqui para ver uma versão maior desta figura.
5. Contagem de unidades formadoras de microcolônias em Fiji (ImageJ)
NOTA: O método mUFC permite a quantificação de UFC de grandes conjuntos de amostras. Fotos das gotículas podem ser registradas para posterior análise para facilitar a contagem de colônias. Vários dispositivos fotográficos podem produzir imagens com qualidade suficiente para este fim. Estes incluem câmeras digitais, webcams, microscópios acoplados à câmera e lupas, e telefones celulares. Softwares gratuitos de análise de imagens, como o ImageJ, oferecem a possibilidade de contagem manual ou automatizada de colônias nessas imagens. Para demonstrar ambos os métodos, será utilizado Fiji, que é uma distribuição do ImageJ que empacota várias ferramentas para análise científica de imagens30. Fiji pode ser baixado de https://fiji.sc/.
- Método de contagem manual
- Abra a imagem que contém o mCFU em Fiji. Selecione Plug-ins > Analisar > contador de células.
- No menu Contador de Células, selecione Inicializar e selecione um contador (por exemplo, Tipo 1).
- Prossiga clicando em cada colônia. Cada clique será mostrado na figura e atualizará o contador (Figura 3). Para desfazer cliques acidentais, selecione Excluir.
- Registre o valor exibido no contador. Clique no botão Redefinir para redefinir a contagem e abrir uma nova imagem para contar amostras adicionais.
NOTA: Mais instruções sobre este plugin podem ser encontradas em https://imagej.net/plugins/cell-counter.
- Método de contagem automatizado
- Abra a imagem que contém o mCFU em Fiji. Selecione Imagem > Tipo > 8 bits. Isso converterá a imagem em uma imagem em escala de cinza de 8 bits.
- Selecione a ferramenta Oval na barra de ferramentas e desenhe um oval ao redor da área com as colônias (Figura 4A). O oval pode ser ajustado após ser desenhado.
- Selecione Editar > Limpar lado de fora para remover qualquer interferência da área externa (Figura 4B). Selecione Imagem > Ajustar > limite.
- Mova os controles deslizantes no menu de limite até que as colônias apareçam em vermelho e o ruído de fundo seja minimizado (Figura 4C).
- Selecione Aplicar e saia da janela de limite. Uma imagem em preto e branco é gerada (Figura 4D).
- Selecione Analisar > Analisar partículas. Na janela de análise de partículas, especifique o intervalo para área de colônia (entre 1 e infinito, medido em pixels quadrados) e circularidade (entre 0 e 1, onde 1 é um círculo perfeito; Figura 4E).
- Selecione Contornos no menu pop-up mostrar. Verifique Exibir resultados para obter medições detalhadas para cada colônia na janela de resultados. Marque Limpar resultados para apagar quaisquer medições anteriores. Marque a caixa Resumir para exibir os resultados resumidos das medições (Figura 4E).
- Inicie o analisador selecionando OK. Uma nova janela é exibida, exibindo todas as colônias delineadas que foram detectadas e contadas. A janela de resultados exibe os detalhes de cada colônia e a janela de resultados resumidos mostra o total de colônias contadas (Figura 4F).
NOTA: As configurações de tamanho e circularidade variam de acordo com a resolução e ampliação da imagem e o tamanho e a forma das colônias. Repita o processo várias vezes até encontrar as melhores configurações que detectem todas as colônias. Mais instruções sobre o plugin analyze particles podem ser encontradas em https://imagej.nih.gov/ij/docs/menus/analyze.html#ap.
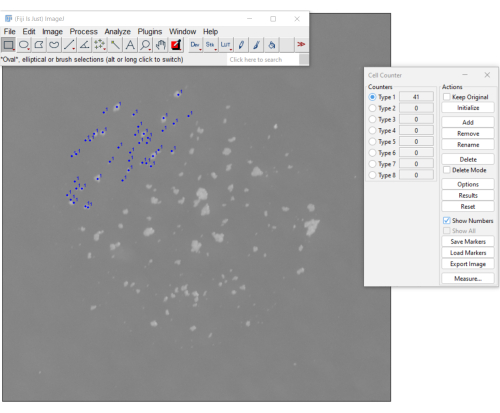
Gráfico 3. Um método manual para contagem de mCFU usando o plugin de contador de células no software Fiji. Os pontos azuis indicam colônias já clicadas pelo usuário. O menu à direita exibe o número de colônias contadas até agora (a contagem é 41). Clique aqui para ver uma versão maior desta figura.

Gráfico 4. Um método automatizado para contagem de mCFU usando o software Fiji. (A, B) A região de interesse com as colônias é selecionada usando a ferramenta de seleção oval e a área externa é removida usando o comando clear outside. (C, D) Uma imagem em preto e branco das colônias é gerada usando a ferramenta de limite. (E, F) O número de colônias é quantificado usando a ferramenta analisar partículas. Clique aqui para ver uma versão maior desta figura.
Access restricted. Please log in or start a trial to view this content.
Resultados
O ensaio de mUFC descrito aqui aumenta a quantidade de informação que pode ser recuperada de uma única placa de Petri para pelo menos 96 vezes. A Figura 5 mostra a comparação de dois métodos de liberação de fármacos para o uso reaproveitado do saquinavir (SQV)31,32 como droga dirigida ao hospedeiro para o tratamento da tuberculose. Neste ensaio, quatro cepas diferentes de Mycobacterium tuberculosis foram usadas para...
Access restricted. Please log in or start a trial to view this content.
Discussão
A TB é um importante problema de saúde pública com importância crescente, particularmente em países de baixa e média renda. A interrupção dos serviços de saúde para diagnosticar e tratar a TB durante a pandemia COVID-19 causou um impacto negativo na incidência de novos casos1. Além disso, as cepas M.tb multidrogas e extensivamente resistentes e a co-infecção de M.tb e HIV devem ser urgentemente abordadas para o controle dessa epidemia 1,...
Access restricted. Please log in or start a trial to view this content.
Divulgações
DP e PJGB declaram que o estudo foi realizado na ausência de quaisquer relações comerciais ou financeiras que pudessem ser interpretadas como um potencial conflito de interesses.
Agradecimentos
Este trabalho foi apoiado por financiamento interno da Faculdade de Medicina da Universidade Católica Portuguesa e financiamento externo da Fundação para a Ciência e a Tecnologia (FCT), no âmbito das bolsas UIDP/04279/2020, UIDB/04279/2020 e EXPL/SAU-INF/0742/2021.
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| 96-well plates | VWR | 734-2781 | |
| DSLR 15-55 mm lens | Nikon | AF-P DX NIKKOR 18-55mm f/3.5-5.6G VR | |
| DSLR camera | Nikon | D3400 | |
| DSLR macro lens | Sigma | MACRO 105mm F2.8 EX DG OS HSM | |
| Fetal calf serum | Gibco | 10270106 | |
| Fiji Software | https://fiji.sc/ | Fiji is an open-source software supported by several laboratories, institutions, and individuals. All the required plugins are included. | |
| Igepal CA-630 | Sigma-Aldrich | 18896 | |
| L-glutamine | Gibco | 25030-081 | |
| Middlebrook 7H10 | BD | 262710 | |
| Middlebrook 7H9 | BD | 271310 | |
| Multichannel pipette (0.5 - 10 µl) | Gilson | FA10013 | |
| Multichannel pipette (20 - 200 µl) | Gilson | FA10011 | |
| Mycobacterium bovis BCG | American Type Culture Collection | ATCC35734 | strain TMC 1011 [BCG Pasteur] |
| OADC enrichment | BD | 211886 | |
| Phosphate buffered saline (PBS) | NZYTech | MB25201 | |
| RPMI 1640 medium | Gibco | 21875091 | |
| Sodium pyruvate | Gibco | 11360-070 | |
| Spectrophotometer UV-6300PC | VWR | 634-6041 | |
| Square Petri dish 120 x 120 mm | Corning | BP124-05 | |
| Tyloxapol | Sigma-Aldrich | T8761 | |
| Ultrasound bath Elma P 30 H | VWR | 142-0051 |
Referências
- World Health Organization. Global Tuberculosis Report 2022. , World Health Organization. Geneva. (2022).
- Bettencourt, P. J. G., Joosten, S. A., Lindestam Arlehamn, C. S., Behr, M. A., Locht, C., Neyrolles, O. 100 years of the Bacillus Calmette-Guérin vaccine. Vaccine. 39 (50), 7221-7222 (2021).
- Bettencourt, P. J. G. The 100th anniversary of bacille Calmette-Guérin (BCG) and the latest vaccines against COVID-19. The International Journal of Tuberculosis and Lung Disease. 25 (8), 611-613 (2021).
- Scriba, T. J., Netea, M. G., Ginsberg, A. M. Key recent advances in TB vaccine development and understanding of protective immune responses against Mycobacterium tuberculosis. Seminars in Immunology. 50, 101431(2020).
- McShane, H., Williams, A. A review of preclinical animal models utilised for TB vaccine evaluation in the context of recent human efficacy data. Tuberculosis. 94 (2), 105-110 (2014).
- Voss, G., et al. Progress and challenges in TB vaccine development. F1000Research. 7, 199(2018).
- Satti, I., McShane, H. Current approaches toward identifying a correlate of immune protection from tuberculosis. Expert Review of Vaccines. 18 (1), 43-59 (2019).
- Young, D. Animal models of tuberculosis. European Journal of Immunology. 39 (8), 2011-2014 (2009).
- Pedroza-Roldán, C., Flores-Valdez, M. A. Recent mouse models and vaccine candidates for preventing chronic/latent tuberculosis infection and its reactivation. Pathogens and disease. 75 (6), (2017).
- Gong, W., Liang, Y., Wu, X. Animal Models of Tuberculosis Vaccine Research: An Important Component in the Fight against Tuberculosis. BioMed Research International. 2020, 1-21 (2020).
- Bettencourt, P., et al. Identification of antigens presented by MHC for vaccines against tuberculosis. NPJ vaccines. 5 (1), 2(2020).
- Moorlag, S. J. C. F. M., Arts, R. J. W., van Crevel, R., Netea, M. G. Non-specific effects of BCG vaccine on viral infections. Clinical Microbiology and Infection. 25 (12), 1473-1478 (2019).
- Wilkie, M., et al. Functional in-vitro evaluation of the non-specific effects of BCG vaccination in a randomised controlled clinical study. Scientific Reports. 12 (1), 7808(2022).
- Netea, M. G., et al. Trained immunity: A program of innate immune memory in health and disease. Science. 352 (6284), aaf1098(2016).
- Bettencourt, P., Pires, D., Carmo, N., Anes, E. Application of Confocal Microscopy for Quantification of Intracellular Mycobacteria in Macrophages. Microscopy: Science, Technology, Applications and Education. 1, 614-621 (2010).
- Bettencourt, P., Carmo, N., Pires, D., Timóteo, P., Anes, E. Mycobacterial infection of macrophages: the effect of the multiplicity of infection. Antimicrobial research: Novel bioknowledge and educational programs. , 651-664 (2017).
- Pires, D., Bettencourt, P., Carmo, N., Niederweis, M., Anes, E. Role of Mycobacterium tuberculosis outer-membrane porins in bacterial survival within macrophages. Drug Discovery Today. 15 (23-24), 1112-1113 (2010).
- Pires, D., et al. Mycobacterium tuberculosis Modulates miR-106b-5p to Control Cathepsin S Expression Resulting in Higher Pathogen Survival and Poor T-Cell Activation. Frontiers in immunology. 8 (DEC), 1819(2017).
- Pires, D., et al. Role of Cathepsins in Mycobacterium tuberculosis Survival in Human Macrophages. Scientific reports. 6 (August), 32247(2016).
- Bettencourt, P., et al. Actin-binding protein regulation by microRNAs as a novel microbial strategy to modulate phagocytosis by host cells: the case of N-Wasp and miR-142-3p. Frontiers in cellular and infection microbiology. 3 (June), 19(2013).
- Bhuyan, S., et al. Microliter spotting and micro-colony observation: A rapid and simple approach for counting bacterial colony forming units. Journal of Microbiological Methods. 207, 106707(2023).
- Jackson, S., McShane, H. Challenges in Developing a Controlled Human Tuberculosis Challenge Model. Current topics in microbiology and immunology. , 1-27 (2022).
- Darrah, P. A., et al. Prevention of tuberculosis in macaques after intravenous BCG immunization. Nature. 577 (7788), 95-102 (2020).
- Madura Larsen, J., et al. BCG stimulated dendritic cells induce an interleukin-10 producing T-cell population with no T helper 1 or T helper 2 bias in vitro. Immunology. 121 (2), 276-282 (2007).
- Bickett, T. E., et al. Characterizing the BCG-Induced Macrophage and Neutrophil Mechanisms for Defense Against Mycobacterium tuberculosis. Frontiers in immunology. 11, 1202(2020).
- Pires, D., et al. Interference of Mycobacterium tuberculosis with the endocytic pathways on macrophages and dendritic cells from healthy donors: role of cathepsins. Drug Discovery Today. 15 (23-24), 1112-1112 (2010).
- Betts, G., et al. Optimising Immunogenicity with Viral Vectors: Mixing MVA and HAdV-5 Expressing the Mycobacterial Antigen Ag85A in a Single Injection. PLoS ONE. 7 (12), e50447(2012).
- Tanner, R., et al. The influence of haemoglobin and iron on in vitro mycobacterial growth inhibition assays. Scientific reports. 7 (1), 43478(2017).
- McNeill, E., et al. Regulation of mycobacterial infection by macrophage Gch1 and tetrahydrobiopterin. Nature communications. 9 (1), 5409(2018).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Pereira, M., Vale, N. Saquinavir: From HIV to COVID-19 and Cancer Treatment. Biomolecules. 12 (7), 944(2022).
- Pires, D., et al. Repurposing Saquinavir for Host-Directed Therapy to Control Mycobacterium Tuberculosis Infection. Frontiers in immunology. 12, 647728(2021).
- Pires, D., et al. Liposomal Delivery of Saquinavir to Macrophages Overcomes Cathepsin Blockade by Mycobacterium tuberculosis and Helps Control the Phagosomal Replicative Niches. International journal of molecular sciences. 24 (2), (2023).
- Maartens, G., Wilkinson, R. J. Tuberculosis. The Lancet. 370 (9604), 2030-2043 (2007).
- Matarazzo, L., Bettencourt, P. J. G. mRNA vaccines: a new opportunity for malaria, tuberculosis and HIV. Frontiers in Immunology. 14, 1172691(2023).
- Young, D., Dye, C. The Development and Impact of Tuberculosis Vaccines. Cell. 124 (4), 683-687 (2006).
- Kommareddi, S., Abramowsky, C. R., Swinehart, G. L., Hrabak, L. Nontuberculous mycobacterial infections: Comparison of the fluorescent auramine-o and Ziehl-Neelsen techniques in tissue diagnosis. Human Pathology. 15 (11), 1085-1089 (1984).
- Sabiiti, W., et al. A Tuberculosis Molecular Bacterial Load Assay (TB-MBLA). Journal of visualized experiments: JoVE. (158), e60460(2020).
- Somoskövi, A., et al. Comparison of Recoveries of Mycobacterium tuberculosis Using the Automated BACTEC MGIT 960 System, the BACTEC 460 TB System, and Löwenstein-Jensen Medium. Journal of Clinical Microbiology. 38 (6), 2395-2397 (2000).
- Tanner, R., et al. The in vitro direct mycobacterial growth inhibition assay (MGIA) for the early evaluation of TB vaccine candidates and assessment of protective immunity: a protocol for non-human primate cells. F1000Research. 10, 257(2021).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados