Method Article
Probenvorbereitung für die In-situ-Kryotomographie von Säugetierzellen
In diesem Artikel
Zusammenfassung
Diese Methode bietet ein zugängliches und flexibles Protokoll für die Herstellung von Elektronenmikroskopie-Gittern (EM) für die zelluläre In-situ-Kryotomographie und die korrelative Licht- und Elektronenmikroskopie (CLEM).
Zusammenfassung
Die zelluläre In-situ-Kryotomographie ist eine leistungsfähige Technik zur Untersuchung komplexer Objekte in ihrem nativen gefroren-hydratisierten zellulären Kontext, was sie für die Zellbiologie und Virologie von hoher Relevanz macht. Das Potenzial der Kombination der Kryotomographie mit anderen Mikroskopiemodalitäten macht sie zu einer perfekten Technik für die integrative und korrelative Bildgebung. Die Probenvorbereitung für die In-situ-Zelltomographie ist jedoch nicht einfach, da sich die Zellen nicht ohne weiteres über das Elektronenmikroskopiegitter anlagern und strecken. Darüber hinaus sind die Gitter selbst zerbrechlich und können bei zu starker Handhabung brechen, was zum Verlust von bildbaren Bereichen führt. Auch die Geometrie von Gewebekulturschalen kann eine Herausforderung darstellen, wenn die Gitter mit einer Pinzette manipuliert werden. Hier beschreiben wir die Tipps und Tricks, um diese (und andere) Herausforderungen zu meistern und qualitativ hochwertige Proben für die zelluläre In-situ-Kryotomographie und die korrelative Bildgebung adhärenter Säugetierzellen vorzubereiten. Mit den kontinuierlichen Fortschritten in der Kryomikroskopie-Technologie ist diese Technik enorm vielversprechend, um unser Verständnis komplexer biologischer Systeme zu verbessern.
Einleitung
Die zelluläre In-situ-Kryotomographie ist eine leistungsfähige Technik, die es ermöglicht, biologisch relevante Strukturen in Zellen ohne chemische Fixierung zu untersuchen. Durch das Anheften von Zellen an EM-Gitter und das Einfrieren der Gitter in einem Kryogen werden Objekte von Interesse in ihren natürlichen zellulären Kontexten eingefroren, ohne dass kristallines Eis aus intrazellulärem Wasser gebildet wird 1,2. Sowohl die chemische Fixierung als auch die Bildung von kristallinem Eis können die Strukturen relevanter Moleküle wie Proteine und Lipide stören und die biologische Genauigkeit der mit diesen Techniken erhaltenen Bilder verringern 3,4. In der Tomographie werden Gitter mit Hilfe der Elektronenmikroskopie in inkrementellen Winkeln abgebildet, und diese Bilder werden dann verwendet, um dreidimensionale Darstellungen des abgebildeten Zielbereichszu konstruieren 5. Die In-situ-Kryotomographie kann zusammen mit anderen Mikroskopietechniken für die integrative und korrelative Bildgebung eingesetzt werden, wie z. B. Kryofluoreszenzbildgebung, Soft-Röntgentomographie und KryoFIB/REM (kryogene fokussierte Ionenstrahl-/Rasterelektronenmikroskopie)6,7,8,9,10,11 . Durch die Integration mehrerer Techniken können mehr Informationen über eine Struktur oder einen Prozess gewonnen werden, als es mit einer einzelnen Mikroskopietechnik möglich wäre.
Trotz aller Vorteile der zellulären In-situ-Kryotomographie kann sich die Probenvorbereitung aus verschiedenen Gründen als Herausforderung erweisen. Aufgrund ihrer Zerbrechlichkeit kann eine gewaltsame Manipulation von Elektronenmikroskopie-Gittern zu Schäden führen, wobei insbesondere die dünne Kohlenstoffschicht empfindlich und reißanfällig ist, wodurch die abbildbare Fläche der Gitter reduziert wird. Elektronenmikroskopie-Gitter sind aufgrund ihrer geringen Größe auch schwer zu manipulieren und neigen dazu, sich von der Oberfläche der Vertiefungen oder Mikroobjektträger zu lösen, die zum Züchten von Zellen verwendet werden. Die Manipulation der Gitter innerhalb der Vertiefungen oder Mikrorutschen kann sich aufgrund der Geometrie dieser als schwierig erweisen. Eine unsachgemäße Vorbereitung der Gitter (z. B. Floating) kann zu einer geringen Zelldichte und einer Verringerung der Anzahl potenzieller Bildgebungsbereiche führen, insbesondere wenn die Zellen nicht dazu neigen, sich an die Gitter selbst anzuheften. Für die direkte zelluläre Kryotomographie müssen sich die Zellen sehr dünn ausbreiten, was aus vielen Gründen gestört werden kann, z. B. bei unsachgemäßen Temperaturen oder unsachgemäßer Handhabung der Gitter.
Durch eine Vielzahl von Optimierungen sollen die in diesem Artikel vorgestellten Techniken diese häufigsten Fallstricke lösen, die bei der Herstellung von elektronenmikroskopischen Gittern für die Kryotomographie auftreten. Die Verwendung einer abgewinkelten 5/15-Pinzette ermöglicht die Manipulation von Gittern innerhalb von Well-Platten oder Mikroobjektträgern. Eine Fibronektinlösung, die vor der Beschichtung auf beide Seiten der Gitter aufgetragen wird, macht schwimmende Gitter unwahrscheinlicher, was vorteilhaft ist, um sicherzustellen, dass die Gitter eine ausreichende Zelldichte aufweisen und dass die Gitter weniger wahrscheinlich durch Manipulation beschädigt werden. Indem wir die Gitter bis kurz vor dem Einfrieren bei 37 °C inkubieren, stellen wir auch sicher, dass die Zellen in einer angenehmen Umgebung gehalten werden, um zu verhindern, dass die Zellen ihre dünnen Ränder zurückziehen. Das Abtupfen der Gitter von der Rückseite verhindert auch eine Beschädigung der Zellen durch mechanische Krafteinwirkung. Insgesamt erhöhen diese Maßnahmen die Erfolgsrate der Probenvorbereitung für zelluläre In-situ-Kryotomographie-Studien und erhöhen die Zugänglichkeit dieses bildgebenden Ansatzes.
Protokoll
1. Vorbereitung des Netzes
HINWEIS: Planen Sie im Versuchsplan insgesamt maximal 8-12 Gitter pro Tauch-Gefrier-Sitzung und 4-5 Gitter pro Vertiefung ein. Darüber hinaus führt dies zu einer sehr langen Tauch-Gefrier-Sitzung, die zu erhöhtem Zellstress, Eiskontamination und Benutzerfehlern führen kann.
- Glimmentladung
- Eine geeignete Anzahl von Gittern wird mit einer perforierten Kohlenstoff-Trägerfolie beladen, die bei 15 mA für 45 s bei 0,39 mBar Atmosphäre in einer Glimmentladungsvorrichtung glühen soll.
- Achte darauf, dass die Kohlenstoffseite des Gitters nach oben zeigt. Wenn Sie fertig sind, geben Sie die Gitter in einen sauberen, mit Filterpapier ausgelegten Behälter (Abbildung 1A1).
HINWEIS: Andere Gitter können verwendet werden, solange das Material nicht giftig für Zellen ist (was Kupfergitter ausschließt). Finder-Raster sind nützlich für korrelative Studien. Auch Gitter aus anderen Materialien sind möglich. Viele Labore haben gute Ergebnisse bei der Züchtung von Zellen auf SiO2-Trägergittern erzielt 8,9.
- Adhärente Behandlung
- Übergeben Sie die Gitter, Fibronektin, PBS, Platten und Pinzetten in eine Biosicherheitswerkbank. Behandeln Sie beide Seiten der Gitter mit 20 μg/ml bovinem Fibronektin, indem Sie zwei großzügig große Punkte einer anhaftenden Lösung auf eine sterile Oberfläche, z. B. den Deckel einer 6-Well-Platte, mikropipettieren. Stellen Sie sicher, dass die Punkte groß genug sind, um das gesamte Gitter zu umhüllen (etwa 100 μl werden empfohlen).
HINWEIS: Vor der Behandlung mit der gewählten adhärenten Lösung besteht hier die Möglichkeit, das Gitter mit Photomikrostrukturierung zu versehen, um die Anhaftung von Zellen an der Mitte von Gitterquadraten zu optimieren. Wenn Sie die Fähigkeit der Zellen, sich an die Balken anzuheften, verringern, erhöht sich die Anzahl der abbildbaren Zellen. Dies ist ideal für Experimente mit KryoFIB-Mahlen. - Verwende eine 5/15-Pinzette, um die Gitter zu manipulieren. Achten Sie darauf, beide Seiten des Gitters in Lösung zu behandeln.
HINWEIS: Es können auch andere Klebstofflösungen verwendet werden (Fibrinogen, Kollagen, andere Bestandteile der extrazellulären Matrix usw.). Hier kommt Fibronektin zum Einsatz, da es bei einer Vielzahl von Zelllinien gute Ergebnisse liefert. (U2OS, HeLa, Vero, Calu-3, Tzm-bl). Die 5/15-Pinzette ist nützlich, da ihre Geometrie eine verbesserte Manövrierfähigkeit um die 6-Well-Platten, 35-mm-Schalen und Mikroobjektträger ermöglicht. Dies führt zu einer geringeren mechanischen Belastung der Gitter und einem intakteren Kohlenstofffilm. Stellen Sie sicher, dass alle erforderlichen Materialien vor Beginn in die Biosicherheitswerkbank gelegt werden, da dies die Möglichkeit einer Kontamination des Gitters verringert.
- Übergeben Sie die Gitter, Fibronektin, PBS, Platten und Pinzetten in eine Biosicherheitswerkbank. Behandeln Sie beide Seiten der Gitter mit 20 μg/ml bovinem Fibronektin, indem Sie zwei großzügig große Punkte einer anhaftenden Lösung auf eine sterile Oberfläche, z. B. den Deckel einer 6-Well-Platte, mikropipettieren. Stellen Sie sicher, dass die Punkte groß genug sind, um das gesamte Gitter zu umhüllen (etwa 100 μl werden empfohlen).
- Anbringen von Gittern
- Nach der Behandlung mit Rinderfibronektin wird das Gitter klebrig. Berühren Sie mit einer 5/15-Pinzette vorsichtig die kohlenstofffreie Oberfläche des Gitters am Boden einer leeren Schale/eines leeren Tellers und öffnen Sie die Pinzette. Das Gitter sollte sich leicht an der neuen Oberfläche befestigen lassen (Abbildung 1A2).
HINWEIS: Versuchen Sie, Gitter nicht direkt in der Mitte oder an den Rändern der Schale/des Tellers zu platzieren. Beim Hinzufügen von Zellen besteht die Tendenz, dass sich die Zelldichte in der Mitte der Platte ansammelt. Dies kann zu Gittern mit übermäßiger Zelldichte führen. Alternativ können hier 3D-gedruckte Gitterhalter verwendet werden, anstatt Zellen direkt an der Oberfläche der Schale/Platte12 anzubringen.
- Nach der Behandlung mit Rinderfibronektin wird das Gitter klebrig. Berühren Sie mit einer 5/15-Pinzette vorsichtig die kohlenstofffreie Oberfläche des Gitters am Boden einer leeren Schale/eines leeren Tellers und öffnen Sie die Pinzette. Das Gitter sollte sich leicht an der neuen Oberfläche befestigen lassen (Abbildung 1A2).
- Inkubation
- Inkubieren Sie die Gitter für 30 Minuten in der Fibronektin-adhärenten Lösung (20 μg/ml). Um sicherzustellen, dass die Gitter während dieses Prozesses nicht trocken werden, mikropipetten Sie alle 15 Minuten 1-2 Tropfen der anhaftenden Lösung direkt auf die Gitter (Abbildung 1A3).
HINWEIS: Die Inkubationszeit variiert je nach verwendeter anhaftender Lösung. Für bovines Fibronektin beträgt die empfohlene Zeit 30 Minuten.
- Inkubieren Sie die Gitter für 30 Minuten in der Fibronektin-adhärenten Lösung (20 μg/ml). Um sicherzustellen, dass die Gitter während dieses Prozesses nicht trocken werden, mikropipetten Sie alle 15 Minuten 1-2 Tropfen der anhaftenden Lösung direkt auf die Gitter (Abbildung 1A3).
- Waschen
- Nach der Inkubationszeit überschüssige adhärente Lösung durch Mikropipettieren entfernen. Waschen Sie die Gitter, indem Sie PBS-Tropfen direkt auf die Gitter pipettieren. Wiederholen Sie dies 2-3 Mal. (Abbildung 1A4).
- Sterilisation
- Verwenden Sie UV-Licht, um die Gitter in der Biosicherheitswerkbank für 1 h zu sterilisieren. Platzieren Sie die Gitter so nah wie möglich an der UV-Quelle, um die Sterilisation zu maximieren (Abbildung 1A5). Um sicherzustellen, dass die Gitter während dieses Prozesses nicht trocken werden, mikropipetten Sie alle 10 Minuten 1-2 Tropfen PBS direkt auf die Gitter.
HINWEIS : Die Schritte 1.4-1.6 können gleichzeitig ausgeführt werden.
- Verwenden Sie UV-Licht, um die Gitter in der Biosicherheitswerkbank für 1 h zu sterilisieren. Platzieren Sie die Gitter so nah wie möglich an der UV-Quelle, um die Sterilisation zu maximieren (Abbildung 1A5). Um sicherzustellen, dass die Gitter während dieses Prozesses nicht trocken werden, mikropipetten Sie alle 10 Minuten 1-2 Tropfen PBS direkt auf die Gitter.
2. Sägitter
- Zellen zählen:
- Trennen und zählen Sie die Zellen mit mechanischen oder enzymatischen Methoden. Bestimmen Sie vor der Zählung die optimale Anzahl von Zellen, die für die Aussaat verwendet werden sollen. Für U2OS, das in einer 6-Well-Platte ausgesät wird, verwenden Sie zwischen 6 x 104 und 1,6 x 105 Zellen pro Well (Abbildung 1B1).
HINWEIS: Die Anzahl der ausgesäten Zellen variiert je nach Zelltyp, der Oberfläche der verwendeten Vertiefung, Schale oder Mikroobjektträger, der Zeit zwischen der Aussaat und dem Einfrieren und dem Versuchsplan. Testen Sie einen Bereich von Zellzahlen, um Bedingungen zu identifizieren, die zum Zeitpunkt des Einfrierens zu 0,25-1 Zellen pro Gitterquadrat führen. Es ist wichtig zu beachten, dass eine hohe Zelldichte die Geschwindigkeit der Wärmeübertragung verringert und den Vitrifikationsprozess beeinträchtigen kann. Um dem entgegenzuwirken, haben einige Labore erfolgreich Zellsiebe eingesetzt, die verhindern, dass sich Zellklumpen bilden13. Schließlich führt dieses Protokoll zur zufälligen Anheftung von Zellen an die Kohlenstoffschicht. Wenn eine gezielte Befestigung erforderlich ist, kann eine Photomikrostrukturierung verwendet werden (siehe Schritt 1.3)14.
- Trennen und zählen Sie die Zellen mit mechanischen oder enzymatischen Methoden. Bestimmen Sie vor der Zählung die optimale Anzahl von Zellen, die für die Aussaat verwendet werden sollen. Für U2OS, das in einer 6-Well-Platte ausgesät wird, verwenden Sie zwischen 6 x 104 und 1,6 x 105 Zellen pro Well (Abbildung 1B1).
- Zugabe von Zellen und Wachstumsmedium : Geben Sie die Zellen über eine Mikropipette in die Vertiefungen und vermeiden Sie dabei Blasen (Abbildung 1B2). In einer 6-Well-Platte wird ein Gesamtwell-Volumen von 1,5-2,0 ml empfohlen.
HINWEIS: Es wird empfohlen, die Gitter vorsichtig zu befeuchten, bevor Sie das gesamte Volumen hinzufügen. Dadurch wird verhindert, dass sich Gitter lösen und in der Lösung schwimmen. Bewegen Sie die Platte vorsichtig von einer Seite zur anderen, um eine homogene Zellverteilung auf der Vertiefung zu ermöglichen. Schwenken Sie die Platte nicht in kreisenden Bewegungen, da dies zu einer übermäßigen Zelldichte in der Mitte der Vertiefung führt. - Inkubation: Inkubieren Sie die Objektträger bei 37 °C für eine angemessene Zeit gemäß dem Versuchsplan, der von den nachgelagerten Anwendungen abhängt. Für dieses Beispiel (Transfektion von HIV-Molekülklonen) wird zwischen 16 und 24 h inkubiert (Abbildung 1B3).
HINWEIS: Längere Inkubationszeiten führen zu mehr Zellteilungszyklen. Passen Sie daher die Startanzahl der Zellen für das Seeding entsprechend an.
3. Transfektion
- Herstellung der Transfektionsmischung:
- Bereiten Sie ein geeignetes kationisches Liposomenreagenz für die Plasmidtransfektion in die ausgewählte Zelllinie vor.
- In diesem Transfektionsbeispiel wird 1 μg Gesamtplasmid-DNA pro Well (in einer 6-Well-Platte) verwendet, die in einem Verhältnis von 1:3 von HIViΔEnv-Plasmid zu psPAX2-Plasmid transfiziert werden soll. Verdünnen Sie für jede Vertiefung 1 μg DNA in 50 μl serumfreies DMEM. Homogenisieren Sie die Mischung durch wiederholtes Mikropipettieren oder sanftes Schwenken der Platte.
- Verdünnen Sie für jede Vertiefung 3 μl Transfektionsreagenz in serumfreiem DMEM und homogenisieren Sie die Mischung dann erneut durch Mikropipettieren oder sanftes Schwenken. Die gesamte verdünnte Transfektionsreagenzmischung wird zu der verdünnten DNA-Mischung gegeben (Schritt 3.1.2) und 15 Minuten lang bei Raumtemperatur (RT) inkubiert (Abbildung 1C1).
HINWEIS: Während diese Mischung inkubiert, ersetzen Sie das Medium in den Vertiefungen durch 1,5 ml frisches DMEM. Achte darauf, dass du die Gitter nicht vom Boden des Brunnens löst.
- Um die Transfektionsmischung aufzutragen, werden 100 μl der Mischung aus Schritt 3.1.3 tropfenweise in jede Vertiefung pipettiert, wobei die Tropfen über den Gittern konzentriert werden, um den Kontakt zu gewährleisten (Abbildung 1C2). Wechseln Sie das Nährmedium 16-24 Stunden nach der Transfektion (Abbildung 1C3).
4. Tiefkühlen
HINWEIS: Bewahren Sie die Zellen bei 37 °C auf, bis sie zum Blotting benötigt werden. Notieren Sie sich außerdem während des gesamten Prozesses die Kohlenstoffseite der Gitter.
- Zelldichte
- Überprüfen Sie die Gitter in einem inversen optischen Mikroskop und notieren Sie die Zelldichte auf jedem Gitter.
HINWEIS: Raster mit weniger als 0,25 Zellen pro Rasterquadrat (oder einer Zelle alle 4 Rasterquadrate) werden als "leer" betrachtet. Gitter mit 0,25-1 Zellen pro Gitterquadrat gelten als "normal". Gitter mit mehr als 1 Zelle pro Gitterquadrat gelten als "voll". - Notieren Sie sich die Zelldichte auf jedem Gitter, um die Löschzeiten später während des Eintauchens wählen zu können. 2-3 ml PBS auf einen leeren Teller/eine leere Schale geben und auf 37 °C erwärmen.
- Überprüfen Sie die Gitter in einem inversen optischen Mikroskop und notieren Sie die Zelldichte auf jedem Gitter.
- Vorbereiten von Fiducials
- Zur Vorbereitung von Gold-Fiducials für nachgeschaltete Kryotomographie-Anwendungen pipettieren Sie 50 μl kolloidale 10-nm-Goldperlenlösung in ein sauberes 1,5-ml-Röhrchen. Geben Sie 2 μl 1 mg/ml BSA in das Röhrchen und homogenisieren Sie es durch wiederholtes Mikropipettieren.
- Zentrifugieren Sie das Röhrchen bei 15.000-20.000 x g für 15 min. Mikropipette, um so viel Überstand wie möglich zu entfernen, ohne das Pellet zu stören.
HINWEIS: Das Pellet wird sehr locker sein. - Geben Sie 50 μl PBS in das Pellet und resuspendieren Sie es. Zentrifugieren Sie das Röhrchen erneut bei 15.000-20.000 x g für 15 min.
- Mit einer 10-μl-Spitze 4 μl des Pellets direkt ansaugen und in ein sauberes 1,5-ml-Röhrchen überführen (Abbildung 1D 1). Verwenden Sie 1 μl des gewaschenen und konzentrierten Goldes pro Gitter.
HINWEIS: Die Art der verwendeten Rahmenmarken hängt von den nachgelagerten Anwendungen ab. Verwenden Sie für die TEM-Tomographie 10-nm-Goldperlen. Für die weiche Röntgentomographie verwenden Sie 200 nm Goldperlen. Verwenden Sie für die Korrelationsmikroskopie fluoreszierende Latexperlen. Fiducials sind normalerweise nicht für Experimente mit KryoFIB-Mahlen erforderlich, da sie durch den Fräsprozess zerstört werden.
- Bekleckernd
- Stellen Sie die Klimakammer auf 95 % Luftfeuchtigkeit und 30 °C ein (Abbildung 1D 1). Wählen Sie mit der 5/15-Pinzette ein Raster aus.
- Waschen Sie die Gitter mit PBS und geben Sie sie in eine Tauchpinzette (Abbildung 1D2). Sobald sie befestigt ist, entfernen Sie die 5/15-Pinzette und schieben Sie die Klemme auf die Tauchpinzette.
- Setzen Sie das Gitter in den Frostfroster ein und halten Sie die Klemme fest (Abbildung 1D3). Fügen Sie 1 μl Gold-Fiducials auf die Rückseite des Rasters hinzu (Abbildung 1D4). Geben Sie 2-3 μl PBS auf die Kohlenstoffseite des Gitters. Verwenden Sie automatisiertes Blotting, um die Rückseite des Gitters abzutupfen und in flüssiges Ethan zu tauchen (Abbildung 1D5).
HINWEIS: Es wird empfohlen, zwischen 3-4 μl Volumen pro Gitter zu haben, um ein optimales Blotting zu erzielen. Die Gitter können nach dem Waschen eine variable Menge an PBS zurückhalten. Passen Sie das zusätzliche PBS-Volumen an, um bereits vorhandene Flüssigkeitsretention im Netz zu berücksichtigen. Es wird empfohlen, die Gold-Fiducials auf der Rückseite des Rasters hinzuzufügen. Die Zugabe auf der Vorderseite kann zu Pools von Gold-Treuhandmetallen in der Nähe der Einfügestelle führen, während die Zugabe von hinten zu einer homogeneren Lösung führt. Die Dauer des Blottings hängt von der Zellzahnheilkunde des Gitters ab. 2 s für leere, 3 s für normale bzw. 4 s für volle Raster. Die Art des verfügbaren Tauchfrosters kann bestimmen, wie das Blotting angegangen wird: Der Leica GP2 Tauchfroster ist derjenige, der in diesem Protokoll verwendet wird und standardmäßig in der Lage ist, automatisiertes einseitiges Blotting durchzuführen. Thermo Fisher Vitrobot führt ein automatisiertes doppelseitiges Blotting durch, wobei die an der Kohlenstoffseite befestigten Zellen oft beschädigt werden. Manuelles Blotting im Vitrobot ist ebenso möglich wie manuelle Schwerkraftkolben.
Ergebnisse
Nach der Kotransfektion von HIViGFPΔEnv und psPAX2 wiesen alle Gitter einen minimalen Riss in der Kohlenstoffschicht auf. Die Gitter wurden 24 Stunden nach der Inkubation mit dem Transfektionsreagenz mittels Phasenlichtmikroskopie und Fluoreszenzmikroskopie abgebildet (Abbildung 2). Zellen sowohl auf den simulierten Gittern als auch auf den co-transfizierten Gittern enthielten lebensfähige Zellen in mehreren Gitterquadraten.
psPAX2 kodiert für alle strukturellen und enzymatischen Proteine von HIV-1 ohne Fluoreszenzmarkierung. HIViGFPΔEnv ähnelt psPAX2, kodiert jedoch für ein GFP-markiertes HIV-Gag-Protein. Beide Plasmide sind ΔEnvelope. Die Kotransfektion führt zu einer nativen Assemblierung und Knospung von fluoreszierenden HIV-1-Partikeln, was dies zu einem großartigen System für CLEM-Studien von HIV unter Bedingungen der Biosicherheitsstufe 1 macht. Die co-transfizierten Gitter zeigten eine Untergruppe von Zellen, die grüne Fluoreszenz aufwiesen, was auf eine erfolgreiche Kotransfektion hindeutet. Keine Zellen auf dem Mock-Grid zeigten Fluoreszenz, was die Kotransfektion mit HIViGFPΔEnv und psPAX2 weiter validiert. Nach der Betrachtung der Gitter mit lichtbasierter Mikroskopie wurden die Gitter eingefroren und in flüssigem Stickstoff langfristig gelagert.
Abbildung 3 zeigt die Ergebnisse von Gittern, die mit der gleichen experimentellen Methode hergestellt wurden, aber leicht unterschiedliche Plasmidkonstrukte verwenden. U2OS-haltige Gitter wurden mit verschiedenen HIV-Klonen (HIVmCherryΔEnv und NL4-3ΔEnvGFP im Verhältnis 1:6) co-transfiziert. Da eine höhere Masse an fluoreszenzmarkierten Plasmiden verwendet wurde, ermöglichten diese Gitter die Beobachtung einer größeren Anzahl transfizierter Zellen, was einen Vorteil bei der Aufnahme von Bildern mit cryoCLEM und cryoET darstellte. Unter Verwendung von cryoCLEM wurden vollständige Gitteratlanten für jedes Gitter mit Hilfe von Kryofluoreszenzmikroskopie erstellt, um die Positionen aller co-transfizierten Zellen aufzuzeichnen. Da die Lage der Zellen bekannt war, wurde eine KryoET durchgeführt. Ein vollständiger Gitteratlas mit geringer Vergrößerung wurde gesammelt und mit dem fluoreszierenden Atlas überlagert, der bei Kryofluoreszenz gesammelt wurde (Abbildung 3A). Kryotomographien wurden an zellulären Stellen gesammelt, um komplizierte Details des viralen Lebenszyklus zu erfassen, einschließlich des Zusammenbaus und der Knospung von HIV aus Zellen (Abbildung 3B).
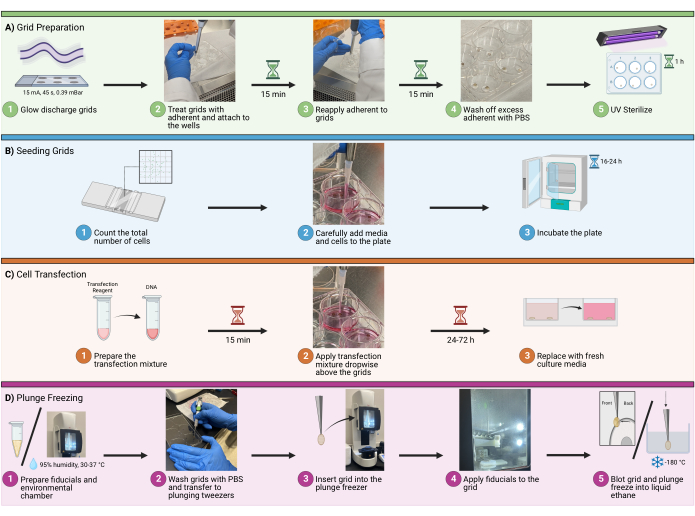
Abbildung 1: Workflow für das Aussading von Zellen in Rastern. Ein Schema, das das gesamte Verfahren zur Aussaat von Zellen auf KryoEM-Gittern darstellt. Der Prozess ist in vier Hauptschritte unterteilt, darunter (A) die Vorbereitung der Gitter in den Wells für die Aussaat, (B) das Hinzufügen der entsprechenden Anzahl von Zellen zu jeder Well, (C) die optionale Transfektion der Zellen für die Fluoreszenzbildgebung und (D) das Einfrieren der Gitter, um die Vitrifikation der Probe zu ermöglichen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2: Kotransfektion von U2OS mit HIViGFPΔEnv und psPAX2. U2OS-Zellen wurden mit GFP-haltigem HIViGFPΔEnv und psPAX2 im Verhältnis 1:3 co-transfiziert. Die Gitter wurden mittels Phasenkontrast- und Fluoreszenzmikroskopie abgebildet. Zellen, von denen gezeigt wird, dass sie eine GFP-Expression aufweisen, deuten auf eine erfolgreiche Kotransfektion hin. Maßstab: 500 μm. Maßstabsleiste (Einsätze): 100 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
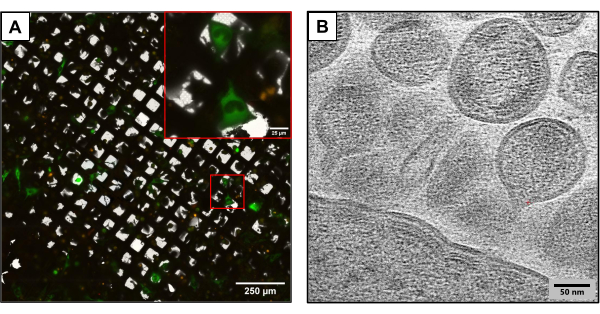
Abbildung 3: Mögliche nachgeschaltete kryobasierte Methoden . (A) Ein KryoCLEM-Bild mit co-transfizierten U2OS-Zellen. Die grün dargestellten Zellen stellen HIV-produzierende Zellen dar und werden zur Messung des Kotransfektionserfolgs verwendet. Rote Punkte stehen für mCherry markierten HIV-1 Gag. Maßstab: 250 μm. Maßstabsbalken (Einschub): 25 μm (B) Ein KryoET-Bild von mehreren HIV-Partikeln, die aus der Plasmamembran von U2OS-Zellen knospen. Maßstab: 50 nm Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
Hier haben wir ein zugängliches, flexibles und reproduzierbares Protokoll für Saatzellen auf elektronenmikroskopischen Gittern für In-situ-Kryoelektronentomographie-Anwendungen bereitgestellt. Diese Methode kann leicht an die Bedürfnisse nachgelagerter Anwendungen und/oder experimenteller Anforderungen angepasst werden. Neben der großen Flexibilität haben wir einen Arbeitsablauf beschrieben, der häufige Fallstricke bei der Gitteraussaat optimiert und reduziert, insbesondere umfangreiche Schäden an der Kohlenstoffschicht, geringe Zelldichte und schlechte strukturelle Integrität von dünnen Zellprojektionen.
Obwohl das hier beschriebene Protokoll mehrere Alternativen bietet, gibt es einige kritische Schritte, die befolgt werden sollten, um die allgemeinen Ergebnisse zu optimieren. Eines der größten Probleme bei der Aussaat von Gitterzellen ist das Ablösen und Schweben von Gittern aus dem Well oder dem Mikroobjektträger. Daher ist es wichtig, das Gitter auf beiden Seiten vollständig mit einer anhaftenden Lösung zu benetzen und ein Austrocknen während der Inkubationszeit zu verhindern. Wenn Sie 3D-gedruckte Gitterhalter verwenden, beachten Sie, dass mehrere Medienwechsel zu diesen Haltern das Potenzial haben, schwimmende Gitter zu erzeugen, da die unter dem Gitter eingeschlossene Luft sie aus dem Halter drücken kann.
Unsere Wahl der Pinzette verbessert auch die Gitterqualität, indem sie eine geometrisch günstige Möglichkeit bietet, die Gitter zu manipulieren, ohne dass das Gitter übermäßig gebogen wird, was die Kohlenstoffschicht beschädigen würde. Die Zellen so lange wie möglich bei 37 °C zu halten, bevor sie eintauchen, reduziert das Zellleiden und verbessert die Anzahl der dünnen, abbildbaren Zellen auf dem Gitter. Schließlich schützt das Blotting von der Goldseite die Zellen vor harten mechanischen Kräften, die zu einer Schädigung empfindlicher Zellstrukturen führen können.
Obwohl sie nicht in diesem Protokoll enthalten ist, hat sich gezeigt, dass die Gitter-Photomikrostrukturierung die Anzahl der abbildbaren Zellen erhöht, indem ihre Befestigung an der Mitte von Gitterquadraten14 optimiert wird. Schließlich wurden kürzlich 3D-gedruckte Gitterhalter verwendet, um Gitterschäden zu reduzieren, indem sie die direkte Manipulation des Gitters einschränken12.
Es ist wichtig zu beachten, dass dieses Protokoll für die Bildgebung von dünnen Kanten und Ausstülpungen von Zellen für die Anwendung der Kryotomographie optimiert ist. Wir empfehlen die Fehlerbehebung bei einer Vielzahl von Bedingungen aus unseren Empfehlungen im Protokoll, um das beste Ergebnis für die nachgelagerten Anwendungen Ihrer Wahl zu finden. Insgesamt bietet dieses Protokoll eine zuverlässige und dennoch vielseitige Methode zur Aussaat von Zellen auf Gittern, die für spezifische Anforderungen optimiert werden können.
Offenlegungen
Die Autoren erklären, dass es keine konkurrierenden Interessen gibt.
Danksagungen
Wir danken dem Mansky-Labor für den Zugang zu Tauchgefriergeräten. Teile dieser Arbeiten wurden in der Charakterisierungseinrichtung der University of Minnesota durchgeführt, die teilweise von der National Science Foundation (NSF) über das Materials Research Science and Engineering Center (MRSEC; Preisnummer DMR-2011401) und der National Neuroscience Curriculum Initiative (NNCI; Fördernummer ECCS-2025124) Programmen. Wir bedanken uns für die Finanzierung durch das Behavior of HIV in Viral Environments Center (B-HIVE; 1U54AI170855-01) und das Duke Center for HIV Structural Biology (DCHSB; U54AI170752) Mitte.
Materialien
| Name | Company | Catalog Number | Comments |
| 10 nm colloidal gold bead solution | Sigma-Aldrich | 741957 | |
| 6 well multidish, 100/CS | Fisher Scientific | FB012927 | |
| Allegra V-15R Benchtop Centrifuge, IVD 120 V 60 Hz | Beckman-Coulter | C63125 | |
| Au G300F1 with R2/2 Quantifoil carbon | Quantifoil | TEM-G300F1-AU | |
| Bovine serum albumin | MilliporeSigma | A9647 | |
| BRAND counting chamber BLAUBRAND Neubauer improved | Sigma-Aldrich | BR717805-1EA | |
| DMi1 Inverted Microscope | Leica | 22A00G119 | |
| Dulbecco's modified eagle's medium - high glucose, no glutamine | Gibco | 11-960-044 | |
| Dumont 5/15 tweezer | Electron Microscopy Sciences | 0103-5/15-PO | |
| EM GP2 | Leica | 587085 | Automated plunge freezer |
| Fetal Bovine Serum | Gibco | A5209 | |
| Fibronectin from bovine plasma, cell culture grade | MilliporeSigma | F1141 | |
| GenJet version II in vitro DNA transfection reagent | SignaGen Laboratories | SL100489 | |
| GlutaMAX I 100x | Fisher Scientific | 35050061 | Media supplement |
| Neslab EX-211 Heating Circulator | Neslab | Out of production | Water bath for media warming |
| Original Portable Pipet-Aid Pipette Controller | Drummond Scientific | 4-000-100 | |
| PBS, pH 7.4 | Gibco | 10010023 | |
| Pelco easyGlow device | Pelco | 91000S | Glow discharge device |
| Penicillin-Streptomycin | Sigma-Aldrich | P0781 | Media supplement |
| Pipetman P1000, 100–1000 µL, Metal Ejector | Gilson | F144059M | |
| Pipetman P2, 0.2–2 µL, Metal Ejector | Gilson | F144054M | |
| Pipetman P20, 2–20 µL, Metal Ejector | Gilson | F144056M | |
| Whatman number 2 filter paper, 55 mm | Whatman | 28455-041 | Blotting paper |
Referenzen
- Zhang, P., Mendonça, L. Analysis of Viruses in the Cellular Context by Electron Tomography. Encyclopedia of Virology. 1, 242-247 (2021).
- Fäßler, F., Dimchev, G., Hodirnau, V. -. V., Wan, W., Schur, F. K. M. Cryo-electron tomography structure of Arp2/3 complex in cells reveals new insights into the branch junction. Nature Communications. 11, 6437 (2020).
- McDowall, A. W., et al. Electron microscopy of frozen hydrated sections of vitreous ice and vitrified biological samples. Journal of Microscopy. 131 (Pt 1), 1-9 (1983).
- Stewart, M., Vigers, G. Electron microscopy of frozen-hydrated biological material. Nature. 319 (6055), 631-636 (1986).
- Gan, L., Jensen, G. J. Electron tomography of cells. Quarterly Reviews of Biophysics. 45 (1), 27-56 (2012).
- Yang, J. E., Larson, M. R., Sibert, B. S., Shrum, S., Wright, E. R. CorRelator: Interactive software for real-time high precision cryo-correlative light and electron microscopy. Journal of Structural Biology. 213 (2), 107709 (2021).
- Mendonça, L., et al. Correlative multi-scale cryo-imaging unveils SARS-CoV-2 assembly and egress. Nature Communications. 12 (1), 4629 (2021).
- Klumpe, S., et al. A modular platform for automated cryo-FIB workflows. eLife. 10, e70506 (2021).
- Wagner, F. R., et al. Preparing samples from whole cells using focused-ion-beam milling for cryo-electron tomography. Nature Protocols. 15 (6), 2041-2070 (2020).
- Klein, S., et al. IFITM3 blocks influenza virus entry by sorting lipids and stabilizing hemifusion. Cell Host & Microbe. 31 (4), 616.e20-633.e20 (2023).
- Shah, P. N. M., et al. Characterization of the rotavirus assembly pathway in situ using cryoelectron tomography. Cell Host & Microbe. 31 (4), 604.e4-615.e4 (2023).
- Fäßler, F., Zens, B., Hauschild, R., Schur, F. K. M. 3D printed cell culture grid holders for improved cellular specimen preparation in cryo-electron microscopy. Journal of Structural Biology. 212 (3), 107633 (2020).
- Hoffmann, P. C., et al. Electron cryo-tomography reveals the subcellular architecture of growing axons in human brain organoids. eLife. 10, e70269 (2021).
- Toro-Nahuelpan, M., et al. Tailoring cryo-electron microscopy grids by photo-micropatterning for in-cell structural studies. Nature Methods. 17 (1), 50-54 (2020).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten