Method Article
Preparo de Amostras para Criotomografia In Situ de Células de Mamíferos
Neste Artigo
Resumo
Este método fornece um protocolo acessível e flexível para a preparação de grades de microscopia eletrônica (ME) para criotomografia celular in situ e microscopia correlativa de luz e eletrônica (CLEM).
Resumo
A criotomografia celular in situ é uma técnica poderosa para estudar objetos complexos em seu contexto celular nativo congelado-hidratado, tornando-a altamente relevante para a biologia celular e virologia. O potencial de combinar a criotomografia com outras modalidades de microscopia a torna uma técnica perfeita para imagens integrativas e correlativas. No entanto, o preparo da amostra para a tomografia celular in situ não é simples, pois as células não se fixam prontamente e se estendem sobre a grade de microscopia eletrônica. Além disso, as próprias grades são frágeis e podem quebrar se manuseadas com muita força, resultando na perda de áreas imagináveis. A geometria das placas de cultura de tecidos também pode representar um desafio ao manipular as grades com pinças. Aqui, descrevemos as dicas e truques para superar esses (e outros) desafios e preparar amostras de boa qualidade para criotomografia celular in situ e imagens correlativas de células aderentes de mamíferos. Com os avanços contínuos na tecnologia de criomicroscopia, esta técnica é uma enorme promessa para o avanço da nossa compreensão de sistemas biológicos complexos.
Introdução
A criotomografia celular in situ é uma técnica poderosa que permite o estudo de estruturas biologicamente relevantes em células sem fixação química. Ao fixar células a grades EM e congelá-las em um criogênio, objetos de interesse são congelados em seus contextos celulares naturais sem a formação de gelo cristalino a partir de água intracelular 1,2. Tanto a fixação química quanto a formação de gelo cristalino podem romper as estruturas de moléculas relevantes, como proteínas e lipídios, reduzindo a precisão biológica das imagens obtidas por essas técnicas 3,4. Na tomografia, grades são imageadas em ângulos incrementais usando microscopia eletrônica, e essas imagens são então usadas para construir representações tridimensionais da região alvofotografada5. A criotomografia in situ pode ser usada em conjunto com outras técnicas de microscopia para imagens integrativas e correlativas, como criofluorescência, tomografia de raios X moles e crioFIB/MEV (criogênico Focused Ion Beam/Scanning Electron Microscopy)6,7,8,9,10,11 . A integração de múltiplas técnicas permite obter mais informações sobre uma estrutura ou processo do que qualquer técnica de microscopia isolada poderia alcançar.
Apesar de todos os benefícios da criotomografia celular in situ , a preparação da amostra pode ser um desafio por uma variedade de razões. Devido à sua fragilidade, a manipulação forçada das grades de microscopia eletrônica pode levar a danos, com a fina camada de carbono em particular sendo delicada e propensa a rasgar, reduzindo a área imaginável das grades. As grades de microscopia eletrônica também são difíceis de manipular devido ao seu pequeno tamanho e são propensas a se desprender da superfície dos poços ou microlâminas usadas para cultivar células. A manipulação das grades dentro dos poços ou microlâminas pode ser difícil devido à geometria destes. A preparação inadequada das grades (por exemplo, permitindo que elas flutuem) pode levar a uma baixa densidade celular e reduzir o número de áreas potenciais de imagem, especialmente quando as células não são propensas a se conectar às próprias grades. Para a criotomografia celular direta, as células devem se espalhar muito finas, o que pode ser interrompido por muitas razões, incluindo temperaturas inadequadas ou manuseio áspero das grades.
Através de uma variedade de otimizações, as técnicas apresentadas neste artigo destinam-se a lidar com essas armadilhas mais comuns que surgem durante a preparação de grades de microscopia eletrônica para criotomografia. O uso de pinças anguladas 5/15 permite a manipulação de grades dentro de placas de poço ou microlâminas. Uma solução de fibronectina aplicada em ambos os lados das grades antes do revestimento torna as grades flutuantes menos prováveis, o que é benéfico para garantir que as grades tenham densidade celular adequada e que as grades tenham menos probabilidade de serem danificadas devido à manipulação. Ao manter as grades incubadas a 37°C até pouco antes do congelamento, também garantimos que as células sejam mantidas em um ambiente confortável para evitar que as células retraiam suas bordas finas. Manchar as grades do lado de trás também evita danos às células pela força mecânica. Em conjunto, essas medidas aumentam a taxa de sucesso do preparo de amostras para estudos de criotomografia celular in situ , aumentando a acessibilidade dessa abordagem de imagem.
Protocolo
1. Preparação da grelha
NOTA: No planejamento experimental, planeje um máximo de 8-12 grades totais por sessão de congelamento de mergulho e 4-5 grades por poço. Mais do que isso, levará a uma sessão de congelamento de mergulho muito longa, o que pode causar aumento do estresse celular, contaminação por gelo e erros do usuário.
- Descarga de brilho
- Carregue um número adequado de grades com um filme de suporte de carbono perfurado para ser descarregado a 15 mA por 45 s a 0,39 mBar atmosfera em um dispositivo de descarga de brilho.
- Certifique-se de que o lado carbono da grade está voltado para cima. Quando terminar, transfira as grades para um recipiente limpo forrado com papel filtro (Figura 1A1).
NOTA: Outras grades podem ser usadas desde que o material não seja tóxico para as células (o que exclui qualquer grade de cobre). As grades do localizador são úteis para estudos correlativos. Grades feitas de outros materiais também são possíveis. Muitos laboratórios têm tido bons resultados cultivando células em cima de grades de suporte de SiO2 8,9.
- Tratamento aderente
- Transfira as grades, fibronectina, PBS, placas e pinças para um gabinete de biossegurança. Trate ambos os lados das grades com 20 μg/mL de fibronectina bovina micropipetando dois pontos de solução aderente de tamanho generoso em uma superfície estéril, como a tampa de uma placa de 6 poços. Certifique-se de que os pontos são grandes o suficiente para envolver toda a grade (cerca de 100 μL é recomendado).
NOTA: Antes do tratamento com a solução aderente selecionada, há a opção de foto-micropadronizar a grade aqui para otimizar a fixação das células no centro dos quadrados da grade. Reduzir a capacidade das células de se ligarem às barras aumentará o número de células imageáveis. Isso é ideal para experimentos envolvendo fresagem crioFIB. - Use uma pinça 5/15 para manipular as grades. Certifique-se de tratar os dois lados da grade em solução.
NOTA: Outras soluções adesivas podem ser utilizadas (fibrinogênio, colágeno, outros componentes da matriz extracelular, etc.). A fibronectina é usada aqui, pois dá bons resultados com uma grande variedade de linhagens celulares. (U2OS, HeLa, Vero, Calu-3, Tzm-bl). A pinça 5/15 é útil, pois sua geometria permite uma melhor manobrabilidade em torno das placas de 6 poços, placas de 35 mm e microcorrediças. Isso resulta em menos estresse mecânico nas grades e um filme de carbono mais intacto. Certifique-se de que todos os materiais necessários sejam colocados no armário de biossegurança antes do início, pois isso reduz a possibilidade de contaminação da rede.
- Transfira as grades, fibronectina, PBS, placas e pinças para um gabinete de biossegurança. Trate ambos os lados das grades com 20 μg/mL de fibronectina bovina micropipetando dois pontos de solução aderente de tamanho generoso em uma superfície estéril, como a tampa de uma placa de 6 poços. Certifique-se de que os pontos são grandes o suficiente para envolver toda a grade (cerca de 100 μL é recomendado).
- Anexando grades
- Uma vez tratada com fibronectina bovina, a grade ficará pegajosa. Usando uma pinça 5/15, toque suavemente a superfície não carbônica da grade no fundo de um prato/prato vazio e abra a pinça. A grade deve ser facilmente fixada à nova superfície (Figura 1A2).
NOTA: Tente evitar colocar grades no centro direto ou nas bordas do prato/prato. Ao adicionar células, há uma tendência para que a densidade celular se acumule no centro da placa. Isso pode resultar em grades com densidade celular excessiva. Alternativamente, os suportes de grade impressos em 3D podem ser usados aqui em vez de fixar células diretamente à superfície do prato/placa12.
- Uma vez tratada com fibronectina bovina, a grade ficará pegajosa. Usando uma pinça 5/15, toque suavemente a superfície não carbônica da grade no fundo de um prato/prato vazio e abra a pinça. A grade deve ser facilmente fixada à nova superfície (Figura 1A2).
- Incubação
- Incubar as grades por 30 min na solução aderente à fibronectina (20 μg/mL). Para garantir que as grades não fiquem secas durante esse processo, a micropipeta 1-2 gotas de solução mais aderente diretamente sobre as grades a cada 15 min (Figura 1A3).
NOTA: O período de incubação irá variar dependendo da solução aderente utilizada. Para fibronectina bovina, o tempo recomendado é de 30 min.
- Incubar as grades por 30 min na solução aderente à fibronectina (20 μg/mL). Para garantir que as grades não fiquem secas durante esse processo, a micropipeta 1-2 gotas de solução mais aderente diretamente sobre as grades a cada 15 min (Figura 1A3).
- Lavagem
- Após o período de incubação, remova o excesso de solução aderente através de micropipetagem. Lave as grades por micropipetagem de gotas de PBS diretamente sobre as grades. Repita isso 2-3 vezes. (Figura 1A4).
- Esterilização
- Use luz UV para esterilizar as grades no armário de biossegurança por 1 h. Coloque as grades o mais próximo possível da fonte UV para maximizar a esterilização (Figura 1A5). Para garantir que as grades não fiquem secas durante esse processo, a micropipeta 1-2 gotas de PBS diretamente sobre as grades a cada 10 min.
Observação : etapas 1.4-1.6 podem ser executadas simultaneamente.
- Use luz UV para esterilizar as grades no armário de biossegurança por 1 h. Coloque as grades o mais próximo possível da fonte UV para maximizar a esterilização (Figura 1A5). Para garantir que as grades não fiquem secas durante esse processo, a micropipeta 1-2 gotas de PBS diretamente sobre as grades a cada 10 min.
2. Grades de semeadura
- Contagem de células:
- Destacar e contar as células usando métodos mecânicos ou enzimáticos. Determine o número ideal de células a serem usadas para semeadura antes da contagem. Para U2OS semeado em uma placa de 6 poços, use entre 6 x 104 a 1,6 x 105 células por poço (Figura 1B1).
OBS: O número de células semeadas variará de acordo com o tipo de célula, a área superficial do poço, placa ou microlâmina utilizada, o tempo entre a semeadura e o congelamento por imersão e o desenho experimental. Teste um intervalo de números de células para identificar condições que resultarão em 0,25-1 células por quadrado de grade no momento do congelamento de imersão. É importante notar que a alta densidade celular reduz a velocidade de transferência de calor e pode afetar o processo de vitrificação. Para combater isso, alguns laboratórios têm encontrado sucesso usando filtros celulares, que atuam para evitar a formação de aglomerados celulares13. Finalmente, este protocolo resulta na fixação aleatória das células à camada de carbono. Se for necessário um acessório direcionado, pode ser utilizada foto-micropadronização (ver passo 1.3)14.
- Destacar e contar as células usando métodos mecânicos ou enzimáticos. Determine o número ideal de células a serem usadas para semeadura antes da contagem. Para U2OS semeado em uma placa de 6 poços, use entre 6 x 104 a 1,6 x 105 células por poço (Figura 1B1).
- Adição de células e meio de crescimento: Adicionar células via micropipeta aos poços, evitando bolhas (Figura 1B2). Em uma placa de 6 poços, recomenda-se um volume total de 1,5-2,0 mL no poço.
NOTA: Recomenda-se molhar cuidadosamente ao redor das grades antes de adicionar todo o volume. Isso ajudará a evitar que as grades se desprendam e flutuem na solução. Mova suavemente a placa de um lado para o outro para facilitar a distribuição homogênea de células no poço. Não gire a placa em um movimento circular, pois isso resultará em alocação excessiva de densidade celular no centro do poço. - Incubação: Incubar as lâminas a 37 °C por um tempo adequado de acordo com o planejamento experimental, que dependerá das aplicações a jusante. Para este exemplo (transfecção de clones moleculares do HIV), incubar entre 16-24 h (Figura 1B3).
NOTA: Tempos de incubação mais longos resultarão em mais ciclos de divisão celular. Portanto, ajuste o número inicial de células para semeadura de acordo.
3. Transfecção
- Preparação da mistura de transfecção:
- Preparar um reagente lipossomal catiônico apropriado para transfecção de plasmídios na linhagem celular selecionada.
- Neste exemplo de transfecção, use 1 μg de DNA plasmidial total por poço (em uma placa de 6 poços) para ser transfectado em uma proporção de 1:3 de plasmídeo HIViΔEnv para plasmídeo psPAX2. Para cada poço, diluir 1 μg de DNA em 50 μL de DMEM livre de soro. Homogeneizar a mistura através de micropipetagem repetida ou redemoinho suave da placa.
- Para cada poço, diluir 3 μL de reagente de transfecção em DMEM sem soro e, em seguida, homogeneizar novamente a mistura por micropipetagem ou turbilhonamento suave. Adicionar toda a mistura diluída de reagentes de transfecção à mistura de ADN diluída (passo 3.1.2) e incubar à temperatura ambiente (TR) durante 15 minutos (figura 1C1).
NOTA: Enquanto esta mistura estiver incubando, substitua o meio nos poços por 1,5 mL de DMEM fresco. Tenha cuidado para não desalojar as grades do fundo do poço.
- Para aplicar a mistura de transfecção, pipetar 100 μL da mistura da etapa 3.1.3 gota a gota para cada poço, concentrando as gotas sobre as grades para garantir o contato (Figura 1C2). Trocar o meio de cultura 16-24 h após a transfecção (Figura 1C3).
4. Congelamento por imersão
NOTA: Mantenha as células a 37 °C até ser necessário para a mancha. Além disso, certifique-se de anotar o lado carbono das grades durante todo o processo.
- Densidade celular
- Verifique as grades em um microscópio óptico invertido e observe as densidades celulares em cada grade.
Observação : grades com menos de 0,25 células por quadrado de grade (ou uma célula a cada 4 quadrados de grade), são consideradas 'vazias'. Grades com 0,25-1 células por quadrado de grade são consideradas 'normais'. Grades com mais de 1 célula por quadrado de grade são consideradas 'cheias'. - Anote a densidade celular em cada grade para permitir a discagem dos tempos de mancha mais tarde durante o congelamento por imersão. Adicionar 2-3 ml de PBS a um prato/prato vazio e aquecer a 37 °C.
- Verifique as grades em um microscópio óptico invertido e observe as densidades celulares em cada grade.
- Preparação de fiduciários
- Para preparar fiduciais de ouro para aplicações de criotomografia a jusante, pipete 50 μL de solução de esferas de ouro coloidal de 10 nm em um tubo limpo de 1,5 mL. Adicionar 2 μL de 1 mg/mL de BSA ao tubo e homogeneizar por micropipetagem repetidamente.
- Centrifugar o tubo a 15.000-20.000 x g por 15 min. Micropipeta para remover o máximo possível do sobrenadante sem perturbar o pellet.
OBS: O pellet estará muito solto. - Adicionar 50 μL de PBS ao pellet e ressuspender. Centrifugar o tubo novamente a 15.000-20.000 x g por 15 min.
- Com uma ponteira de 10 μL, aspirar diretamente 4 μL do pellet e transferi-lo para um tubo limpo de 1,5 mL (Figura 1D1). Use 1 μL do ouro lavado e concentrado por grade.
NOTA: O tipo de fiduciais utilizados depende das aplicações a jusante. Para a tomografia de TEM, utilizar esferas de ouro de 10 nm. Para tomografia de raios X moles, use contas de ouro de 200 nm. Para microscopia de correlação, utilizar esferas fluorescentes de látex. Fiduciais normalmente não são necessários para experimentos envolvendo moagem crioFIB, pois o processo de moagem irá destruí-los.
- Mancha
- Montagem da câmara ambiental a 95% de umidade e 30°C (Figura 1D1). Selecione uma grade usando a pinça 5/15.
- Lave as grades com PBS e transfira-as para uma pinça mergulhadora (Figura 1D2). Uma vez segura, remova a pinça 5/15 e deslize a pinça sobre a pinça mergulhadora.
- Insira a grade no congelador de imersão, mantendo a braçadeira segura (Figura 1D3). Adicione 1 μL de fiduciais de ouro ao lado de trás da grade (Figura 1D4). Adicione 2-3 μL de PBS ao lado carbono da grade. Use o blotting automatizado para manchar o lado traseiro da grade e mergulhar o congelamento em etano líquido (Figura 1D5).
NOTA: Recomenda-se ter entre 3-4 μL de volume por grade para uma mancha ideal. As grades podem reter uma quantidade variável de PBS após a lavagem. Ajuste o volume adicional de PBS para levar em conta a retenção de líquidos pré-existente na rede. Recomenda-se que os fiduciais de ouro sejam adicionados à parte de trás da grade. A adição à frente pode resultar em piscinas de fiduciais de ouro perto do ponto de inserção, enquanto a adição da parte de trás resulta em uma solução mais homogeneizada. A duração do blotting depende da odontologia celular da grade. 2 s para vazio, 3 s para normal e 4 s para redes completas, respectivamente. O tipo de congelador de imersão disponível pode determinar como o blotting é abordado: O congelador de mergulho Leica GP2 é o usado neste protocolo e é capaz de blotting automatizado de face única por padrão. A Thermo Fisher Vitrobot faz blotting automatizado de dupla face, embora isso muitas vezes danifique as células ligadas ao lado do carbono. O blotting manual no Vitrobot também é possível, bem como êmbolos de gravidade manuais.
Resultados
Após a cotransfecção de HIViGFPΔEnv e psPAX2, todas as grades apresentaram ruptura mínima na camada de carbono. As grades foram obtidas por meio de microscopia de fase e fluorescente 24 h após a incubação com o reagente de transfecção (Figura 2). As células nas grades simuladas e nas grades co-transfectadas continham células viáveis em vários quadrados de grade.
psPAX2 codifica para todas as proteínas estruturais e enzimáticas do HIV-1 sem qualquer marcação de fluorescência. HIViGFPΔEnv é semelhante ao psPAX2, mas com códigos para uma proteína HIV Gag marcada com GFP. Ambos os plasmídeos são ΔEnvelope. A cotransfecção resulta em montagem nativa e brotamento de partículas fluorescentes do HIV-1, tornando este um ótimo sistema para estudos CLEM do HIV em condição de Nível de Biossegurança 1. As grades co-transfectadas mostraram um subconjunto de células exibindo fluorescência verde, indicando sucesso na cotransfecção. Nenhuma célula na grade simulada exibiu fluorescência, validando ainda mais a cotransfecção usando HIViGFPΔEnv e psPAX2. Após a visualização das grades usando microscopia baseada em luz, as grades foram congeladas e movidas para armazenamento de longo prazo em nitrogênio líquido.
A Figura 3 mostra os resultados de grades produzidas usando o mesmo método experimental, mas utilizando construções de plasmídeos ligeiramente diferentes. Grades contendo U2OS foram co-transfectadas usando diferentes clones de HIV (HIVmCherryΔEnv e NL4-3ΔEnvGFP na proporção de 1:6). Como foi utilizada uma maior massa de plasmídeos marcados fluorescentemente, essas grades possibilitaram a observação de um maior número de células transfectadas, proporcionando uma vantagem na captura de imagens utilizando crioCLEM e crioET. Usando crioCLEM, atlas de grade completa foram gerados para cada grade usando microscopia de criofluorescência para registrar a localização de todas as células co-transfectadas. Com a localização das células conhecidas, realizou-se o crioTE. Um atlas completo de grade de baixa magnificação foi coletado e sobreposto com o atlas fluorescente coletado em criofluorescência (Figura 3A). Criomogramas foram coletados em sítios celulares capturando detalhes intrincados do ciclo de vida viral, incluindo a montagem e brotamento do HIV a partir das células (Figura 3B).
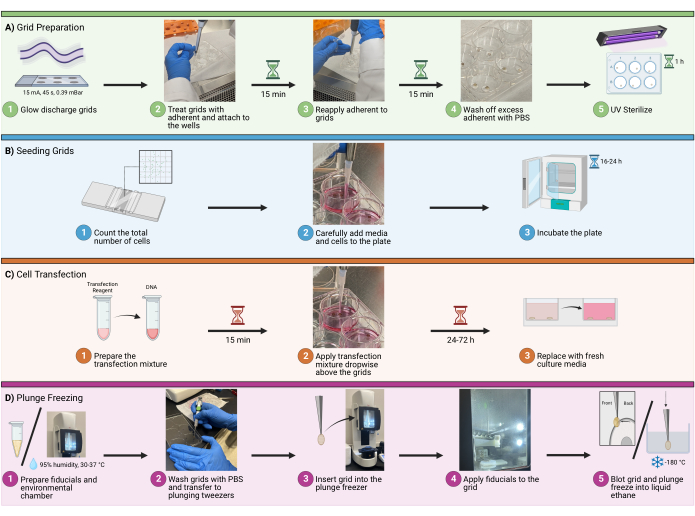
Figura 1: Propagação de células no fluxo de trabalho de grades. Um esquema descrevendo o procedimento geral para semear células em grades crioEM. O processo é dividido em quatro etapas principais, incluindo (A) a preparação de grades nos poços para semeadura, (B) a adição da quantidade adequada de células a cada poço, (C) a transfecção opcional de células para imagens fluorescentes e (D) o congelamento por imersão de grades para permitir a vitrificação da amostra. Clique aqui para ver uma versão maior desta figura.

Figura 2: Cotransfecção de U2OS usando HIViGFPΔEnv e psPAX2. Células U2OS foram co-transfectadas com HIViGFPΔEnv contendo GFP e psPAX2 na proporção de 1:3. As grades foram imageadas por contraste de fase e microscopia de fluorescência. As células que demonstraram ter expressão de GFP indicam cotransfecção bem-sucedida. Barra de escala: 500 μm. Barra de escala (insets): 100 μm. Clique aqui para ver uma versão maior desta figura.
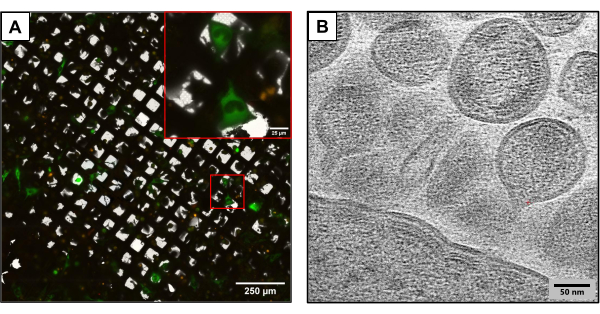
Figura 3: Potenciais métodos criológicos a jusante . (A) Uma imagem crioCLEM com células U2OS co-transfectadas. As células em verde representam as células produtoras de HIV e são usadas para medir o sucesso da cotransfecção. Puncta vermelho representam mCherry marcado HIV-1 Gag. Barra de escala: 250 μm. Barra de escala (inset): 25 μm (B) Uma imagem crioET de múltiplas partículas de HIV brotando da membrana plasmática de células U2OS. Barra de escala: 50 nm Clique aqui para ver uma versão maior desta figura.
Discussão
Aqui, fornecemos um protocolo acessível, flexível e reprodutível para células de sementes em grades de microscopia eletrônica para aplicações em tomografia crioeletrônica in situ . Este método pode ser facilmente adaptado para atender às necessidades de aplicações a jusante e/ou requisitos experimentais. Além da grande flexibilidade, descrevemos um fluxo de trabalho que otimiza e reduz armadilhas comuns na semeadura de grade, notavelmente danos extensos à camada de carbono, baixa densidade celular e baixa integridade estrutural de projeções de células finas.
Embora o protocolo aqui descrito forneça várias alternativas, existem algumas etapas críticas que devem ser seguidas para otimizar os resultados gerais. Um dos maiores problemas com a semeadura de células de grade é o desprendimento e flutuação de grades do poço ou microslide. Portanto, é importante molhar totalmente a grade com uma solução aderente em ambos os lados e evitar que ela seque durante o período de incubação. Se estiver usando suportes de grade impressos em 3D, esteja ciente de que várias mudanças de mídia para esses suportes têm o potencial de produzir grades flutuantes, uma vez que o ar preso sob a grade pode forçá-lo a sair do suporte.
Nossa escolha de pinças também melhora a qualidade da grade na forma de fornecer uma maneira geometricamente favorável de manipular as grades sem flexão extensa da grade que danificaria a camada de carbono. Manter as células a 37 °C pelo maior tempo possível antes de mergulhar reduz o sofrimento celular e melhora o número de células finas na grade. Finalmente, a mancha do lado dourado protegerá as células de forças mecânicas severas que podem levar a danos em estruturas celulares frágeis.
Embora não incluído neste protocolo, a foto-micropadronização em grade mostrou aumentar o número de células imageáveis otimizando sua fixação ao centro dos quadrados da grade14. Finalmente, suportes de grade impressos em 3D foram usados recentemente para reduzir danos à rede, limitando a manipulação direta da grade12.
Pode ser importante notar que este protocolo é otimizado para obtenção de imagens de bordas finas e saliências de células para a aplicação da criotomografia. Sugerimos solucionar uma variedade de condições a partir de nossas recomendações no protocolo para encontrar o melhor resultado para as aplicações a jusante de escolha. No geral, este protocolo fornece um método confiável e versátil de semear células em grades que podem ser ajustadas para necessidades específicas.
Divulgações
Os autores declaram não haver interesses concorrentes.
Agradecimentos
Gostaríamos de agradecer ao laboratório Mansky pelo acesso ao equipamento de congelamento de imersão. Parte deste trabalho foi realizado na instalação de caracterização da Universidade de Minnesota, que recebe apoio parcial da National Science Foundation (NSF) por meio do Materials Research Science and Engineering Center (MRSEC; Prêmio Número DMR-2011401) e a National Neuroscience Curriculum Initiative (NNCI; Prêmio Número ECCS-2025124) programas. Agradecemos o financiamento do Centro de Comportamento do HIV em Ambientes Virais (B-HIVE; 1U54AI170855-01) e do Duke Center for HIV Structural Biology (DCHSB; U54AI170752) centro.
Materiais
| Name | Company | Catalog Number | Comments |
| 10 nm colloidal gold bead solution | Sigma-Aldrich | 741957 | |
| 6 well multidish, 100/CS | Fisher Scientific | FB012927 | |
| Allegra V-15R Benchtop Centrifuge, IVD 120 V 60 Hz | Beckman-Coulter | C63125 | |
| Au G300F1 with R2/2 Quantifoil carbon | Quantifoil | TEM-G300F1-AU | |
| Bovine serum albumin | MilliporeSigma | A9647 | |
| BRAND counting chamber BLAUBRAND Neubauer improved | Sigma-Aldrich | BR717805-1EA | |
| DMi1 Inverted Microscope | Leica | 22A00G119 | |
| Dulbecco's modified eagle's medium - high glucose, no glutamine | Gibco | 11-960-044 | |
| Dumont 5/15 tweezer | Electron Microscopy Sciences | 0103-5/15-PO | |
| EM GP2 | Leica | 587085 | Automated plunge freezer |
| Fetal Bovine Serum | Gibco | A5209 | |
| Fibronectin from bovine plasma, cell culture grade | MilliporeSigma | F1141 | |
| GenJet version II in vitro DNA transfection reagent | SignaGen Laboratories | SL100489 | |
| GlutaMAX I 100x | Fisher Scientific | 35050061 | Media supplement |
| Neslab EX-211 Heating Circulator | Neslab | Out of production | Water bath for media warming |
| Original Portable Pipet-Aid Pipette Controller | Drummond Scientific | 4-000-100 | |
| PBS, pH 7.4 | Gibco | 10010023 | |
| Pelco easyGlow device | Pelco | 91000S | Glow discharge device |
| Penicillin-Streptomycin | Sigma-Aldrich | P0781 | Media supplement |
| Pipetman P1000, 100–1000 µL, Metal Ejector | Gilson | F144059M | |
| Pipetman P2, 0.2–2 µL, Metal Ejector | Gilson | F144054M | |
| Pipetman P20, 2–20 µL, Metal Ejector | Gilson | F144056M | |
| Whatman number 2 filter paper, 55 mm | Whatman | 28455-041 | Blotting paper |
Referências
- Zhang, P., Mendonça, L. Analysis of Viruses in the Cellular Context by Electron Tomography. Encyclopedia of Virology. 1, 242-247 (2021).
- Fäßler, F., Dimchev, G., Hodirnau, V. -. V., Wan, W., Schur, F. K. M. Cryo-electron tomography structure of Arp2/3 complex in cells reveals new insights into the branch junction. Nature Communications. 11, 6437 (2020).
- McDowall, A. W., et al. Electron microscopy of frozen hydrated sections of vitreous ice and vitrified biological samples. Journal of Microscopy. 131 (Pt 1), 1-9 (1983).
- Stewart, M., Vigers, G. Electron microscopy of frozen-hydrated biological material. Nature. 319 (6055), 631-636 (1986).
- Gan, L., Jensen, G. J. Electron tomography of cells. Quarterly Reviews of Biophysics. 45 (1), 27-56 (2012).
- Yang, J. E., Larson, M. R., Sibert, B. S., Shrum, S., Wright, E. R. CorRelator: Interactive software for real-time high precision cryo-correlative light and electron microscopy. Journal of Structural Biology. 213 (2), 107709 (2021).
- Mendonça, L., et al. Correlative multi-scale cryo-imaging unveils SARS-CoV-2 assembly and egress. Nature Communications. 12 (1), 4629 (2021).
- Klumpe, S., et al. A modular platform for automated cryo-FIB workflows. eLife. 10, e70506 (2021).
- Wagner, F. R., et al. Preparing samples from whole cells using focused-ion-beam milling for cryo-electron tomography. Nature Protocols. 15 (6), 2041-2070 (2020).
- Klein, S., et al. IFITM3 blocks influenza virus entry by sorting lipids and stabilizing hemifusion. Cell Host & Microbe. 31 (4), 616.e20-633.e20 (2023).
- Shah, P. N. M., et al. Characterization of the rotavirus assembly pathway in situ using cryoelectron tomography. Cell Host & Microbe. 31 (4), 604.e4-615.e4 (2023).
- Fäßler, F., Zens, B., Hauschild, R., Schur, F. K. M. 3D printed cell culture grid holders for improved cellular specimen preparation in cryo-electron microscopy. Journal of Structural Biology. 212 (3), 107633 (2020).
- Hoffmann, P. C., et al. Electron cryo-tomography reveals the subcellular architecture of growing axons in human brain organoids. eLife. 10, e70269 (2021).
- Toro-Nahuelpan, M., et al. Tailoring cryo-electron microscopy grids by photo-micropatterning for in-cell structural studies. Nature Methods. 17 (1), 50-54 (2020).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados