Method Article
Preparazione del campione per la criotomografia in situ di cellule di mammifero
In questo articolo
Riepilogo
Questo metodo fornisce un protocollo accessibile e flessibile per la preparazione di griglie di microscopia elettronica (EM) per la criotografia cellulare in situ e la microscopia ottica ed elettronica correlata (CLEM).
Abstract
La criotomografia cellulare in situ è una tecnica potente per lo studio di oggetti complessi nel loro contesto cellulare nativo congelato-idratato, il che la rende molto rilevante per la biologia cellulare e la virologia. Il potenziale di combinare la criotomografia con altre modalità di microscopia la rende una tecnica perfetta per l'imaging integrativo e correlativo. Tuttavia, la preparazione del campione per la tomografia cellulare in situ non è semplice, poiché le cellule non si attaccano e si allungano facilmente sulla griglia di microscopia elettronica. Inoltre, le griglie stesse sono fragili e possono rompersi se maneggiate con troppa forza, con conseguente perdita di aree visualizzabili. Anche la geometria delle piastre di coltura tissutale può rappresentare una sfida quando si manipolano le griglie con le pinzette. Qui, descriviamo i suggerimenti e i trucchi per superare queste (e altre) sfide e preparare campioni di buona qualità per la criotomografia cellulare in situ e l'imaging correlato di cellule di mammifero aderenti. Con i continui progressi nella tecnologia della criomicroscopia, questa tecnica è estremamente promettente per far progredire la nostra comprensione dei sistemi biologici complessi.
Introduzione
La criotomografia cellulare in situ è una tecnica potente che consente lo studio di strutture biologicamente rilevanti nelle cellule senza fissazione chimica. Attaccando le cellule alle griglie EM e congelando le griglie in un criogene, gli oggetti di interesse vengono congelati nei loro contesti cellulari naturali senza la formazione di ghiaccio cristallino dall'acqua intracellulare 1,2. Sia la fissazione chimica che la formazione di ghiaccio cristallino possono alterare le strutture di molecole rilevanti, come proteine e lipidi, riducendo l'accuratezza biologica delle immagini ottenute utilizzando queste tecniche 3,4. In tomografia, le griglie vengono visualizzate ad angoli incrementali utilizzando la microscopia elettronica e queste immagini vengono quindi utilizzate per costruire rappresentazioni tridimensionali della regione target di cui è stata eseguita l'immagine5. La criotomografia in situ può essere utilizzata insieme ad altre tecniche di microscopia per l'imaging integrativo e correlativo, come l'imaging a criofluorescenza, la tomografia a raggi X molli e la crioFIB/SEM (criogenic Focused Ion Beam/Scanning Electron Microscopy)6,7,8,9,10,11 . L'integrazione di più tecniche consente di ottenere più informazioni su una struttura o un processo rispetto a qualsiasi singola tecnica di microscopia.
Nonostante tutti i vantaggi della criotomiografia cellulare in situ , la preparazione del campione può rivelarsi impegnativa per una serie di motivi. A causa della loro fragilità, la manipolazione forzata delle griglie di microscopia elettronica può portare a danni, in particolare il sottile strato di carbonio che è delicato e soggetto a strappi, riducendo l'area visualizzabile delle griglie. Le griglie per microscopia elettronica sono anche difficili da manipolare a causa delle loro piccole dimensioni e tendono a staccarsi dalla superficie dei pozzetti o dei microvetrini utilizzati per far crescere le cellule. La manipolazione delle griglie all'interno dei pozzetti o dei microvetrini può rivelarsi difficile a causa della geometria di questi. Una preparazione impropria delle griglie (ad esempio, lasciandole galleggiare) può portare a una bassa densità cellulare e alla riduzione del numero di potenziali aree di imaging, soprattutto quando le cellule non sono inclini ad attaccarsi alle griglie stesse. Per la criotografia cellulare diretta, le cellule devono diffondersi molto sottili, il che può essere interrotto per molte ragioni, tra cui temperature improprie o manipolazione brusca delle griglie.
Attraverso una varietà di ottimizzazioni, le tecniche presentate in questo articolo hanno lo scopo di gestire queste insidie più comuni che sorgono durante la preparazione di griglie di microscopia elettronica per la criotomografia. L'uso di pinzette angolate 5/15 consente la manipolazione di griglie all'interno di piastre a pozzetti o microvetrini. Una soluzione di fibronectina applicata su entrambi i lati delle griglie prima della placcatura rende meno probabili le griglie flottanti, il che è utile per garantire che le griglie abbiano un'adeguata densità cellulare e che le griglie abbiano meno probabilità di essere danneggiate a causa della manipolazione. Mantenendo le griglie incubate a 37°C fino a poco prima del surgelazione, ci assicuriamo anche che le cellule siano mantenute in un ambiente confortevole per evitare che le cellule ritraggano i loro bordi sottili. Tamponando le griglie dal lato posteriore si evitano anche danni alle celle causati dalla forza meccanica. Complessivamente, queste misure aumentano il tasso di successo della preparazione del campione per gli studi di criotografia cellulare in situ , aumentando l'accessibilità di questo approccio di imaging.
Protocollo
1. Preparazione della griglia
NOTA: Nel disegno sperimentale, pianificare un massimo di 8-12 griglie totali per sessione di surgelazione e 4-5 griglie per pozzetto. Inoltre, ciò porterà a una sessione di congelamento a tuffo molto lunga, che può causare un aumento dello stress cellulare, contaminazione da ghiaccio ed errori dell'utente.
- Scarica luminosa
- Caricare un numero appropriato di griglie con una pellicola di supporto in carbonio perforato da scaricare a incandescenza a 15 mA per 45 s a 0,39 mBar atmosfera in un dispositivo di scarica a incandescenza.
- Assicurati che il lato in carbonio della griglia sia rivolto verso l'alto. Al termine, trasferite le griglie in un contenitore pulito foderato con carta da filtro (Figura 1A 1).
NOTA: È possibile utilizzare altre griglie purché il materiale non sia tossico per le celle (il che esclude qualsiasi griglia in rame). Le griglie del finder sono utili per gli studi correlativi. Sono possibili anche griglie realizzate con altri materiali. Molti laboratori hanno ottenuto buoni risultati facendo crescere le cellule sopra le griglie di supporto SiO2 8,9.
- Trattamento aderente
- Trasferire le griglie, la fibronectina, il PBS, le piastre e le pinzette in una cabina di biosicurezza. Trattare entrambi i lati delle griglie con fibronectina bovina da 20 μg/mL micropipettando due punti di soluzione aderente di dimensioni generose su una superficie sterile, come il coperchio di una piastra a 6 pozzetti. Assicurarsi che i punti siano abbastanza grandi da avvolgere l'intera griglia (si consiglia circa 100 μL).
NOTA: Prima del trattamento con la soluzione aderente selezionata, c'è la possibilità di foto-micropatternare la griglia qui per ottimizzare l'attaccamento delle cellule al centro dei quadrati della griglia. Riducendo la capacità delle celle di attaccarsi alle barre aumenterà il numero di celle visualizzabili. Questo è l'ideale per gli esperimenti che coinvolgono la fresatura crioFIB. - Usa 5/15 pinzette per manipolare le griglie. Assicurarsi di trattare entrambi i lati della griglia in soluzione.
NOTA: Possono essere utilizzate altre soluzioni adesive (fibrinogeno, collagene, altri componenti della matrice extracellulare, ecc.). La fibronectina viene utilizzata qui, in quanto dà buoni risultati con un'ampia varietà di linee cellulari. (U2OS, HeLa, Vero, Calu-3, Tzm-bl). La pinzetta 5/15 è utile in quanto la sua geometria consente una migliore manovrabilità intorno alle piastre a 6 pozzetti, alle piastre da 35 mm e ai micro vetrini. Ciò si traduce in una minore sollecitazione meccanica sulle griglie e in un film di carbonio più intatto. Assicurarsi che tutti i materiali necessari siano collocati nell'armadio di biosicurezza prima di iniziare, in quanto ciò riduce la possibilità di contaminazione della rete.
- Trasferire le griglie, la fibronectina, il PBS, le piastre e le pinzette in una cabina di biosicurezza. Trattare entrambi i lati delle griglie con fibronectina bovina da 20 μg/mL micropipettando due punti di soluzione aderente di dimensioni generose su una superficie sterile, come il coperchio di una piastra a 6 pozzetti. Assicurarsi che i punti siano abbastanza grandi da avvolgere l'intera griglia (si consiglia circa 100 μL).
- Fissaggio di griglie
- Una volta trattata con fibronectina bovina, la griglia diventerà appiccicosa. Usando una pinzetta da 5/15, tocca delicatamente la superficie non in carbonio della griglia sul fondo di un piatto/piatto vuoto e apri le pinzette. La griglia dovrebbe fissarsi facilmente alla nuova superficie (Figura 1A2).
NOTA: Cercare di evitare di posizionare le griglie al centro diretto o ai bordi del piatto/piatto. Quando si aggiungono cellule, c'è la tendenza ad accumulare densità cellulare al centro della piastra. Ciò può portare a griglie con un'eccessiva densità cellulare. In alternativa, è possibile utilizzare i supporti per griglie stampati in 3D invece di attaccare le celle direttamente alla superficie del piatto/piatto12.
- Una volta trattata con fibronectina bovina, la griglia diventerà appiccicosa. Usando una pinzetta da 5/15, tocca delicatamente la superficie non in carbonio della griglia sul fondo di un piatto/piatto vuoto e apri le pinzette. La griglia dovrebbe fissarsi facilmente alla nuova superficie (Figura 1A2).
- Incubazione
- Incubare le griglie per 30 minuti nella soluzione aderente alla fibronectina (20 μg/mL). Per garantire che le griglie non si asciughino durante questo processo, micropipettare 1-2 gocce di soluzione più aderente direttamente sopra le griglie ogni 15 minuti (Figura 1A3).
NOTA: Il periodo di incubazione varia a seconda della soluzione aderente utilizzata. Per la fibronectina bovina, il tempo consigliato è di 30 min.
- Incubare le griglie per 30 minuti nella soluzione aderente alla fibronectina (20 μg/mL). Per garantire che le griglie non si asciughino durante questo processo, micropipettare 1-2 gocce di soluzione più aderente direttamente sopra le griglie ogni 15 minuti (Figura 1A3).
- Lavaggio
- Dopo il periodo di incubazione, rimuovere la soluzione aderente in eccesso mediante micropipettaggio. Lavare le griglie micropipettando gocce di PBS direttamente sopra le griglie. Ripeti l'operazione 2-3 volte. (Figura 1A4).
- Sterilizzazione
- Utilizzare la luce UV per sterilizzare le griglie nella cabina di biosicurezza per 1 ora. Posizionare le griglie il più vicino possibile alla sorgente UV per massimizzare la sterilizzazione (Figura 1A5). Per garantire che le griglie non si asciughino durante questo processo, micropipetta 1-2 gocce di PBS direttamente sopra le griglie ogni 10 minuti.
NOTA : I passaggi 1.4-1.6 possono essere eseguiti contemporaneamente.
- Utilizzare la luce UV per sterilizzare le griglie nella cabina di biosicurezza per 1 ora. Posizionare le griglie il più vicino possibile alla sorgente UV per massimizzare la sterilizzazione (Figura 1A5). Per garantire che le griglie non si asciughino durante questo processo, micropipetta 1-2 gocce di PBS direttamente sopra le griglie ogni 10 minuti.
2. Griglie di semina
- Conteggio celle:
- Staccare e contare le cellule utilizzando metodi meccanici o enzimatici. Determinare il numero ottimale di celle da utilizzare per la semina prima del conteggio. Per U2OS seminato in una piastra a 6 pozzetti, utilizzare un numero di cellule compreso tra 6 x 104 e 1,6 x 105 per pozzetto (Figura 1B1).
NOTA: Il numero di cellule seminate varia in base al tipo di cellula, alla superficie del pozzetto, del piatto o del microvetrino utilizzato, al tempo che intercorre tra la semina e il congelamento a tuffo e al disegno sperimentale. Testare un intervallo di numeri di celle per identificare le condizioni che si tradurranno in 0,25-1 celle per quadrato della griglia al momento del congelamento a tuffo. È importante notare che un'elevata densità cellulare riduce la velocità di trasferimento del calore e può influire sul processo di vetrificazione. Per combattere questo, alcuni laboratori hanno avuto successo utilizzando filtri cellulari, che agiscono per prevenire la formazione di grumi cellulari13. Infine, questo protocollo si traduce nell'attaccamento casuale delle cellule allo strato di carbonio. Se è necessario un attacco mirato, è possibile utilizzare il foto-micropatterning (vedere il punto 1.3)14.
- Staccare e contare le cellule utilizzando metodi meccanici o enzimatici. Determinare il numero ottimale di celle da utilizzare per la semina prima del conteggio. Per U2OS seminato in una piastra a 6 pozzetti, utilizzare un numero di cellule compreso tra 6 x 104 e 1,6 x 105 per pozzetto (Figura 1B1).
- Aggiunta di cellule e terreno di coltura: aggiungere le cellule tramite micropipetta ai pozzetti, evitando la formazione di bolle (Figura 1B2). In una piastra a 6 pozzetti, si consiglia un volume totale del pozzetto di 1,5-2,0 mL.
NOTA: Si consiglia di bagnare accuratamente intorno alle griglie prima di aggiungere l'intero volume. Ciò contribuirà a evitare che le griglie si stacchino e fluttuino nella soluzione. Muovere delicatamente la piastra da un lato all'altro per facilitare una distribuzione omogenea delle cellule sul pozzetto. Non far roteare la piastra con un movimento circolare, poiché ciò comporterebbe un'eccessiva allocazione della densità cellulare al centro del pozzetto. - Incubazione: Incubare i vetrini a 37 °C per un tempo adeguato secondo il disegno sperimentale, che dipenderà dalle applicazioni a valle. Per questo esempio (trasfezione di cloni molecolari di HIV), incubare tra le 16 e le 24 ore (Figura 1B3).
NOTA: Tempi di incubazione più lunghi si tradurranno in più cicli di divisione cellulare. Pertanto, regolare di conseguenza il numero iniziale di celle per la semina.
3. Trasfezione
- Preparazione della miscela di trasfezione:
- Preparare un reagente liposomico cationico appropriato per la trasfezione del plasmide nella linea cellulare selezionata.
- In questo esempio di trasfezione, utilizzare 1 μg di DNA plasmidico totale per pozzetto (in una piastra a 6 pozzetti) da trasfettare con un rapporto 1:3 tra plasmide HIViΔEnv e plasmide psPAX2. Per ogni pozzetto, diluire 1 μg di DNA in 50 μL di DMEM esente da siero. Omogeneizzare la miscela mediante micropipettaggio ripetuto o rotazione delicata della piastra.
- Per ogni pozzetto, diluire 3 μL di reagente di trasfezione in DMEM privo di siero, quindi omogeneizzare nuovamente la miscela mediante micropipettaggio o leggera agitazione. Aggiungere l'intera miscela di reagenti di trasfezione diluita alla miscela di DNA diluito (fase 3.1.2) e incubare a temperatura ambiente (RT) per 15 minuti (Figura 1C1).
NOTA: Durante l'incubazione di questa miscela, sostituire il terreno nei pozzetti con 1,5 mL di DMEM fresco. Fare attenzione a non rimuovere le griglie dal fondo del pozzo.
- Per applicare la miscela di trasfezione, pipettare 100 μL della miscela dal punto 3.1.3 goccia a goccia a ciascun pozzetto, concentrando le gocce sulle griglie per garantire il contatto (Figura 1C2). Cambiare il terreno di coltura 16-24 ore dopo la trasfezione (Figura 1C3).
4. Surgelazione a immersione
NOTA: Mantenere le cellule a 37 °C fino al momento del blotting. Inoltre, assicurati di prendere nota del lato carbonio delle griglie durante tutto il processo.
- Densità cellulare
- Controllare le griglie in un microscopio ottico invertito e annotare le densità delle cellule su ciascuna griglia.
NOTA: le griglie con meno di 0,25 celle per quadrato della griglia (o una cella ogni 4 quadrati della griglia) sono considerate "vuote". Le griglie con 0,25-1 celle per quadrato della griglia sono considerate "normali". Le griglie con più di 1 cella per quadrato della griglia sono considerate "piene". - Prendere nota della densità delle celle su ciascuna griglia per consentire la composizione dei tempi di tamponamento in un secondo momento durante il surgelazione. Aggiungere 2-3 ml di PBS in un piatto vuoto e scaldare a 37 °C.
- Controllare le griglie in un microscopio ottico invertito e annotare le densità delle cellule su ciascuna griglia.
- Preparazione dei fiduciali
- Per preparare i fiduciali d'oro per le applicazioni di criotomografia a valle, pipettare 50 μL di soluzione di microsfere d'oro colloidale da 10 nm in una provetta pulita da 1,5 mL. Aggiungere 2 μL di 1 mg/mL di BSA alla provetta e omogeneizzare mediante micropipettaggio ripetuto.
- Centrifugare la provetta a 15.000-20.000 x g per 15 min. Micropipetta per rimuovere il più possibile il surnatante senza disturbare il pellet.
NOTA: Il pellet sarà molto sciolto. - Aggiungere 50 μL di PBS al pellet e risospendere. Centrifugare nuovamente la provetta a 15.000-20.000 x g per 15 min.
- Utilizzando una punta da 10 μL, aspirare direttamente 4 μL di pellet e trasferirlo in una provetta pulita da 1,5 mL (Figura 1D1). Utilizzare 1 μL di oro lavato e concentrato per griglia.
NOTA: Il tipo di fiduciali utilizzati dipende dalle applicazioni a valle. Per la tomografia TEM, utilizzare perline d'oro da 10 nm. Per la tomografia a raggi X molli, utilizzare perline d'oro da 200 nm. Per la microscopia di correlazione, utilizzare perline di lattice fluorescenti. I fiduciali non sono normalmente necessari per gli esperimenti che coinvolgono la fresatura crioFIB, poiché il processo di macinazione li distruggerà.
- Blotting
- Impostare la camera climatica al 95% di umidità e a 30 °C (Figura 1D1). Selezionare una griglia utilizzando le pinzette 5/15.
- Lavate le griglie con PBS e trasferitele su una pinzetta a tuffo (Figura 1D2). Una volta fissate, rimuovere le pinzette 5/15 e far scorrere il morsetto sulle pinzette a immersione.
- Inserire la griglia nel congelatore a immersione, mantenendo il clamp saldamente (Figura 1D3). Aggiungere 1 μL di fiduciali d'oro sul lato posteriore della griglia (Figura 1D4). Aggiungere 2-3 μL di PBS sul lato carbonio della griglia. Utilizzare il tamponamento automatico per tamponare il lato posteriore della griglia e immergere il congelamento nell'etano liquido (Figura 1D5).
NOTA: Si consiglia di avere tra 3-4 μL di volume per griglia per un blotting ottimale. Le griglie possono trattenere una quantità variabile di PBS dopo il lavaggio. Regolare il volume aggiunto di PBS per tenere conto della ritenzione di liquidi preesistente sulla griglia. Si consiglia di aggiungere i fiduciali dorati sul retro della griglia. L'aggiunta nella parte anteriore può portare a pool di fiduciali d'oro vicino al punto di inserimento, mentre l'aggiunta dalla parte posteriore si traduce in una soluzione più omogeneizzata. La durata del blotting dipende dall'odontoiatria cellulare della griglia. 2 s per le griglie vuote, 3 s per le normali e 4 s per le griglie piene, rispettivamente. Il tipo di congelatore a immersione disponibile può determinare il modo in cui viene affrontato il blotting: il congelatore a immersione Leica GP2 è quello utilizzato in questo protocollo ed è in grado di eseguire automaticamente il blotting su un lato per impostazione predefinita. Thermo Fisher Vitrobot esegue il blotting bifacciale automatizzato, anche se questo spesso danneggia le celle attaccate al lato in carbonio. È possibile anche il tamponamento manuale nel Vitrobot, così come gli stantuffi manuali a gravità.
Risultati
A seguito della cotrasfezione di HIViGFPΔEnv e psPAX2, tutte le griglie presentavano una lacerazione minima nello strato di carbonio. Le griglie sono state riprese utilizzando la microscopia a luce di fase e la microscopia a luce fluorescente 24 ore dopo l'incubazione con il reagente di trasfezione (Figura 2). Le celle sia sulle griglie fittizie che sulle griglie co-trasfettate contenevano celle vitali in più quadrati della griglia.
psPAX2 codifica per tutte le proteine strutturali ed enzimatiche dell'HIV-1 senza alcuna marcatura di fluorescenza. HIViGFPΔEnv è simile a psPAX2 ma con codici per una proteina HIV Gag marcata con GFP. Entrambi i plasmidi sono ΔEnvelope. La cotrasfezione si traduce in un assemblaggio e in una gemmazione simili a quelli nativi di particelle fluorescenti di HIV-1, rendendo questo un ottimo sistema per gli studi CLEM sull'HIV in condizioni di biosicurezza di livello 1. Le griglie co-trasfettate hanno mostrato un sottoinsieme di cellule che mostravano fluorescenza verde, indicando una cotrasfezione riuscita. Nessuna cellula sulla griglia simulata ha mostrato fluorescenza, convalidando ulteriormente la cotrasfezione utilizzando HIViGFPΔEnv e psPAX2. Dopo aver osservato le griglie utilizzando la microscopia basata sulla luce, le griglie sono state congelate per immersione e spostate per la conservazione a lungo termine in azoto liquido.
La Figura 3 illustra i risultati delle griglie prodotte utilizzando lo stesso metodo sperimentale, ma utilizzando costrutti plasmidici leggermente diversi. Le griglie contenenti U2OS sono state co-trasfettate utilizzando diversi cloni di HIV (HIVmCherryΔEnv e NL4-3ΔEnvGFP con un rapporto 1:6). Poiché è stata utilizzata una massa maggiore di plasmidi marcati con fluorescenza, queste griglie hanno permesso l'osservazione di un numero maggiore di cellule trasfettate, fornendo un vantaggio durante l'acquisizione di immagini utilizzando cryoCLEM e cryoET. Utilizzando cryoCLEM, sono stati generati atlanti a griglia completi per ciascuna griglia utilizzando la microscopia a criofluorescenza per registrare le posizioni di tutte le cellule co-trasfettate. Una volta note le posizioni delle cellule, è stata eseguita la crioET. Un atlante a griglia a basso ingrandimento completo è stato raccolto e sovrapposto all'atlante fluorescente raccolto alla criofluorescenza (Figura 3A). I criotomogrammi sono stati raccolti nei siti cellulari catturando dettagli intricati del ciclo di vita virale, tra cui l'assemblaggio e la gemmazione dell'HIV dalle cellule (Figura 3B).
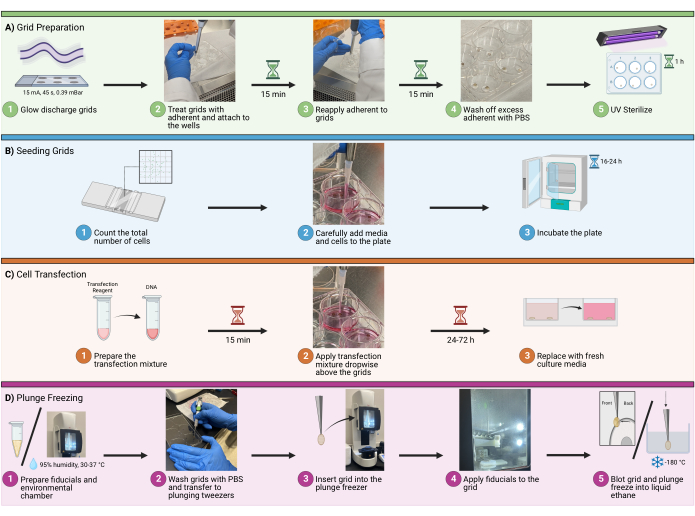
Figura 1: Flusso di lavoro di seeding delle celle sulle griglie. Uno schema che illustra l'intera procedura per seminare le cellule su griglie crioEM. Il processo è suddiviso in quattro fasi principali, tra cui (A) la preparazione delle griglie nei pozzetti per la semina, (B) l'aggiunta della quantità appropriata di cellule a ciascun pozzetto, (C) la trasfezione opzionale delle cellule per l'imaging fluorescente e (D) il congelamento a tuffo delle griglie per consentire la vitrificazione del campione. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 2: Cotrasfezione di U2OS utilizzando HIViGFPΔEnv e psPAX2. Le cellule U2OS sono state co-trasfettate con HIViGFPΔEnv e psPAX2 contenenti GFP in un rapporto 1:3. Le griglie sono state visualizzate mediante microscopia a contrasto di fase e a fluorescenza. Le cellule che hanno dimostrato di avere un'espressione di GFP indicano una cotrasfezione riuscita. Barra graduata: 500 μm. Barra della scala (inserti): 100 μm. Fare clic qui per visualizzare una versione più grande di questa figura.
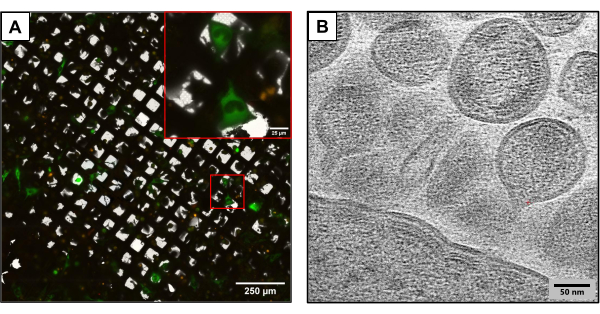
Figura 3: Potenziali metodi criogenici a valle . (A) Un'immagine cryoCLEM con cellule U2OS co-trasfettate. Le cellule in verde rappresentano le cellule produttrici di HIV e vengono utilizzate per misurare il successo della cotrasfezione. I punti rossi rappresentano mCherry etichettato HIV-1 Gag. Barra graduata: 250 μm. Barra della scala (riquadro): 25 μm (B) Un'immagine crioET di più particelle di HIV che germogliano dalla membrana plasmatica delle cellule U2OS. Barra della scala: 50 nm Fare clic qui per visualizzare una versione più grande di questa figura.
Discussione
Qui, abbiamo fornito un protocollo accessibile, flessibile e riproducibile per seminare cellule su griglie di microscopia elettronica per applicazioni di tomografia crioelettronica in situ . Questo metodo può essere facilmente adattato per soddisfare le esigenze delle applicazioni a valle e/o i requisiti sperimentali. Oltre alla grande flessibilità, abbiamo descritto un flusso di lavoro che ottimizza e riduce le insidie comuni nella semina della griglia, in particolare danni estesi allo strato di carbonio, bassa densità cellulare e scarsa integrità strutturale delle proiezioni di celle sottili.
Sebbene il protocollo qui descritto fornisca diverse alternative, ci sono alcuni passaggi critici che dovrebbero essere seguiti per ottimizzare i risultati generali. Uno dei maggiori problemi con la semina delle cellule a griglia è il distacco e il galleggiamento delle griglie dal pozzo o dal microscivolo. Pertanto, è importante bagnare completamente la griglia con una soluzione aderente su entrambi i lati ed evitare che si asciughi durante il periodo di incubazione. Se si utilizzano supporti per griglie stampati in 3D, tenere presente che più cambi di supporti a questi supporti hanno il potenziale per produrre griglie galleggianti poiché l'aria intrappolata sotto la griglia può costringerla a uscire dal supporto.
La nostra scelta di pinzette migliora anche la qualità della griglia in quanto fornisce un modo geometricamente favorevole di manipolare le griglie senza piegature estese della griglia che danneggerebbero lo strato di carbonio. Mantenere le celle a 37 °C il più a lungo possibile prima di immergersi riduce la sofferenza cellulare e migliora il numero di cellule sottili visualizzabili sulla griglia. Infine, l'asciugatura dal lato dell'oro proteggerà le cellule da forze meccaniche dure che potrebbero portare a danni alle fragili strutture cellulari.
Sebbene non sia incluso in questo protocollo, è stato dimostrato che il foto-micropatterning della griglia aumenta il numero di cellule visualizzabili ottimizzando il loro attacco al centro dei quadrati della griglia14. Infine, i supporti per griglie stampati in 3D sono stati recentemente utilizzati per ridurre i danni alla griglia limitando la manipolazione diretta della griglia12.
Può essere importante notare che questo protocollo è ottimizzato per l'imaging di bordi sottili e sporgenze dalle cellule per l'applicazione della criotografia. Suggeriamo di risolvere una serie di condizioni dalle nostre raccomandazioni nel protocollo per trovare il miglior risultato per le applicazioni a valle di scelta. Nel complesso, questo protocollo fornisce un metodo affidabile ma versatile per seminare le cellule su griglie che possono essere modificate per esigenze specifiche.
Divulgazioni
Gli autori dichiarano di non avere interessi contrastanti.
Riconoscimenti
Ringraziamo il laboratorio Mansky per l'accesso alle attrezzature per la surgelazione. Parte di questo lavoro è stato svolto nella struttura di caratterizzazione dell'Università del Minnesota, che riceve un supporto parziale dalla National Science Foundation (NSF) attraverso il Materials Research Science and Engineering Center (MRSEC; Numero di premio DMR-2011401) e la National Neuroscience Curriculum Initiative (NNCI; ECCS-2025124). Vorremmo ringraziare i finanziamenti del centro Behavior of HIV in Viral Environments (B-HIVE; 1U54AI170855-01) e del Duke Center for HIV Structural Biology (DCHSB; U54AI170752) centro.
Materiali
| Name | Company | Catalog Number | Comments |
| 10 nm colloidal gold bead solution | Sigma-Aldrich | 741957 | |
| 6 well multidish, 100/CS | Fisher Scientific | FB012927 | |
| Allegra V-15R Benchtop Centrifuge, IVD 120 V 60 Hz | Beckman-Coulter | C63125 | |
| Au G300F1 with R2/2 Quantifoil carbon | Quantifoil | TEM-G300F1-AU | |
| Bovine serum albumin | MilliporeSigma | A9647 | |
| BRAND counting chamber BLAUBRAND Neubauer improved | Sigma-Aldrich | BR717805-1EA | |
| DMi1 Inverted Microscope | Leica | 22A00G119 | |
| Dulbecco's modified eagle's medium - high glucose, no glutamine | Gibco | 11-960-044 | |
| Dumont 5/15 tweezer | Electron Microscopy Sciences | 0103-5/15-PO | |
| EM GP2 | Leica | 587085 | Automated plunge freezer |
| Fetal Bovine Serum | Gibco | A5209 | |
| Fibronectin from bovine plasma, cell culture grade | MilliporeSigma | F1141 | |
| GenJet version II in vitro DNA transfection reagent | SignaGen Laboratories | SL100489 | |
| GlutaMAX I 100x | Fisher Scientific | 35050061 | Media supplement |
| Neslab EX-211 Heating Circulator | Neslab | Out of production | Water bath for media warming |
| Original Portable Pipet-Aid Pipette Controller | Drummond Scientific | 4-000-100 | |
| PBS, pH 7.4 | Gibco | 10010023 | |
| Pelco easyGlow device | Pelco | 91000S | Glow discharge device |
| Penicillin-Streptomycin | Sigma-Aldrich | P0781 | Media supplement |
| Pipetman P1000, 100–1000 µL, Metal Ejector | Gilson | F144059M | |
| Pipetman P2, 0.2–2 µL, Metal Ejector | Gilson | F144054M | |
| Pipetman P20, 2–20 µL, Metal Ejector | Gilson | F144056M | |
| Whatman number 2 filter paper, 55 mm | Whatman | 28455-041 | Blotting paper |
Riferimenti
- Zhang, P., Mendonça, L. Analysis of Viruses in the Cellular Context by Electron Tomography. Encyclopedia of Virology. 1, Elsevier, Academic Press. 242-247 (2021).
- Fäßler, F., Dimchev, G., Hodirnau, V. -V., Wan, W., Schur, F. K. M. Cryo-electron tomography structure of Arp2/3 complex in cells reveals new insights into the branch junction. Nature Communications. 11, 6437(2020).
- McDowall, A. W., et al. Electron microscopy of frozen hydrated sections of vitreous ice and vitrified biological samples. Journal of Microscopy. 131 (Pt 1), 1-9 (1983).
- Stewart, M., Vigers, G. Electron microscopy of frozen-hydrated biological material. Nature. 319 (6055), 631-636 (1986).
- Gan, L., Jensen, G. J. Electron tomography of cells. Quarterly Reviews of Biophysics. 45 (1), 27-56 (2012).
- Yang, J. E., Larson, M. R., Sibert, B. S., Shrum, S., Wright, E. R. CorRelator: Interactive software for real-time high precision cryo-correlative light and electron microscopy. Journal of Structural Biology. 213 (2), 107709(2021).
- Mendonça, L., et al. Correlative multi-scale cryo-imaging unveils SARS-CoV-2 assembly and egress. Nature Communications. 12 (1), 4629(2021).
- Klumpe, S., et al. A modular platform for automated cryo-FIB workflows. eLife. 10, e70506(2021).
- Wagner, F. R., et al. Preparing samples from whole cells using focused-ion-beam milling for cryo-electron tomography. Nature Protocols. 15 (6), 2041-2070 (2020).
- Klein, S., et al. IFITM3 blocks influenza virus entry by sorting lipids and stabilizing hemifusion. Cell Host & Microbe. 31 (4), 616.e20-633.e20 (2023).
- Shah, P. N. M., et al. Characterization of the rotavirus assembly pathway in situ using cryoelectron tomography. Cell Host & Microbe. 31 (4), 604.e4-615.e4 (2023).
- Fäßler, F., Zens, B., Hauschild, R., Schur, F. K. M. 3D printed cell culture grid holders for improved cellular specimen preparation in cryo-electron microscopy. Journal of Structural Biology. 212 (3), 107633(2020).
- Hoffmann, P. C., et al. Electron cryo-tomography reveals the subcellular architecture of growing axons in human brain organoids. eLife. 10, e70269(2021).
- Toro-Nahuelpan, M., et al. Tailoring cryo-electron microscopy grids by photo-micropatterning for in-cell structural studies. Nature Methods. 17 (1), 50-54 (2020).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon