Method Article
Schnelle glykoqualitative Bewertung rekombinanter Proteine mit einem vollautomatischen System
In diesem Artikel
Zusammenfassung
Zuvor wurde ein System zur automatisierten und schnellen Analyse von Glykanen auf Proteinen entwickelt. In diesem Artikel wird das Protokoll für die "glykoqualitative Analyse" vorgestellt, das für ein breites Spektrum von Anwendern optimiert ist, z. B. für diejenigen, die sich mit der Analyse von Glykanstrukturen in Biopharmazeutika und anderen Glykokonjugatmaterialien beschäftigen.
Zusammenfassung
Die Proteinglykosylierung, eine kritische posttranslationale Modifikation, beeinflusst die Stabilität, Wirksamkeit und Immunogenität rekombinanter Proteine, einschließlich Biopharmazeutika. Glykanstrukturen weisen eine signifikante Heterogenität auf, die je nach Produktionszelltyp, Kulturbedingungen und Reinigungsmethoden variiert. Daher ist die Überwachung und Bewertung der Glykanstrukturen rekombinanter Proteine von entscheidender Bedeutung, insbesondere in der biopharmazeutischen Produktion. Der Lektin-Microarray, eine zur Massenspektrometrie komplementäre Technik, zeichnet sich durch eine hohe Empfindlichkeit und Benutzerfreundlichkeit aus. In der Regel dauert es jedoch mehr als einen Tag, bis Ergebnisse erzielt werden. Um es an die nicht-glykowissenschaftliche Forschung oder die Entwicklung von Arzneimittelprozessen anzupassen, ist eine automatisierte Alternative mit hohem Durchsatz erforderlich. Aus diesem Grund wurde das weltweit erste vollautomatische Lektin-basierte Glykan-Profiling-System entwickelt, das das Technologiekonzept "Bead Array in a Single Tip (BIST)" verwendet. Dieses System ermöglicht die Aufbereitung und Lagerung von lektinimmobilisierten Kügelchen in Einheiten von 1.000 Stück, mit anpassbaren parallelen Einfügeaufträgen für verschiedene Zwecke. In diesem Artikel wird ein praktisches Protokoll für die Forschung mit "glykoqualifizierten" rekombinanten Proteinen vorgestellt. Nach dem Test ihrer Reaktivität gegen 12 Polyacrylamid-Glykan-Konjugate wurden 15 Lektine ausgewählt, um die Vielseitigkeit des Systems zu erhöhen. Darüber hinaus wurde der Probenmarkierungsprozess durch die Umstellung von Cy3 auf Biotin optimiert, wodurch die Gesamtverarbeitungszeit um 30 Minuten reduziert wurde. Zur sofortigen Datenqualifizierung werden Lektin-Bindungssignale als Dotcode auf dem oberen Monitor angezeigt. Die Zuverlässigkeit des Systems wurde durch tägliche Reproduzierbarkeitstests, Wiederholbarkeitstests und Langzeitlagertests mit einem Variationskoeffizienten von <10 % bestätigt. Dieser benutzerfreundliche und schnelle Glyko-Analysator hat potenzielle Anwendungen in der Qualitätsüberwachung von endogenen Glykoproteinen für die Bewertung und Validierung von Biomarkern. Diese Methode erleichtert die Analyse für diejenigen, die neu in der Glykowissenschaft sind, und erweitert damit ihren praktischen Nutzen.
Einleitung
Die Proteinglykosylierung ist eine entscheidende posttranslationale Modifikation, die in Biopharmazeutika untersucht werden muss. Die Glykanprofile von Proteinen können je nach Kulturbedingungen, Reinigungsprozessen und Wirtszellen variieren1. Einfache Instrumente sind erforderlich, um die Glykosylierung innerhalb der Bioprozess-Pipeline zu qualifizieren. Es wird geschätzt, dass über 50% der sezernierten Proteine und Membranproteine in vivo mit mehreren Glykanen modifiziert sind, die sich je nach Zelllinie, Entwicklungsstadium und Krankheitsstatus, wie z. B. dem Auftreten von Malignität, ändern2. Die Überwachung von Glykanprofilen birgt ein erhebliches Potenzial für die Identifizierung einzigartiger diagnostischer Marker und Wirkstoffziele. Automatisierte Instrumente, die in der Lage sind, große Probengrößen schnell zu messen, sind sehr gefragt, um eine solche aberrante Glykosylierung von Hunderten von Patientenproben in der Forschungspipeline zu verifizieren und zu validieren.
Die Microarray-Technologie wurde in die Glykomik integriert, um das Glykanprofiling von Glykoproteinenzu bewerten 3. Bei dieser Methode werden mehrere Lektine, die Glykan-bindende Proteine sind, auf einer Oberfläche wie einem Objektträger immobilisiert. Diese interaktionsbasierte Glykoanalysetechnologie erfordert keine vorherige Freisetzung von Glykanen aus Kernproteinen, was den Prozess für Forscher, die neu in der Glykotechnologie sind, vereinfacht. Trotz seiner weit verbreiteten Verwendung wurde für industrielle Anwendungen wie die Bioproduktion ein automatisiertes System benötigt, das in der Lage ist, Glykane für eine größere Anzahl von Analysezielen schnell und einfach zu überwachen. Um dieses Problem zu lösen, wurde bereits über ein automatisiertes Glykan-Profiling-System berichtet, das auf einem einzigartigen Konzept namens "Bead Array in a single tip" (BIST) basiert und ursprünglich für die Genotypisierung entwickelt wurde. Dieses System vereinfacht den Prozess mit einem Ein-Box-Typ mit automatischem Durchsatz4. Unter Verwendung von Spitzen, in denen verschiedene Lektin-fixierte Kügelchen parallel angeordnet sind 4,5, wurde ein Verfahren zur Analyse von Glykanstrukturen, die in Glykoproteinen modifiziert sind, etabliert und als GlycoBIST bezeichnet (im Folgenden als "automatisches Glykan-Profiling-System" bezeichnet) (Abbildung 1A). Lektine können auf 1.000 Kügelchen fixiert und getrocknet werden, um die Aktivität ein Jahr lang aufrechtzuerhalten, sowohl vor als auch nach dem Einpacken einer Spitze. Sobald Spitzen und Kartuschen, die Reagenzien wie HRP-markierte Anti-Streptavidin-Antikörper (SA-HRP) enthalten, in das Messgerät des Prototyps (ein automatisiertes Reaktionsmessgerät, siehe Materialtabelle) eingesetzt werden, fungiert die Spitze als Autopipette. Ein Chemilumineszenz-Detektionsscanner an der Innenseite des Instruments quantifiziert die Signale von acht Spitzen gleichzeitig. Die quantitativen Daten dieser acht Spitzen werden kompakt und gleichzeitig als Punktcodes auf dem Touchscreen des Geräts angezeigt, um eine schnelle Bestätigung der Messergebnisse zu ermöglichen. Darüber hinaus wird der Wert, der als Maximum des gemessenen Peaks dargestellt wird, als Rohdaten vom Instrument transportiert und ermöglicht die grafische Darstellung durch einzelne Forscher (Abbildung 1A, unteres Bild).
In diesem Artikel beschreiben die Autoren eine verbesserte Methode zur Biotin-Markierung von Proteinen, die die Verarbeitungszeit auf 30 Minuten reduziert. Zielproteine werden im Voraus biotinyliert und mit SA-HRP nachgewiesen (Abbildung 1B). Eine Standard-GlycoBIST-Spitze (spezialisierte Spitze für automatisches Glykan-Profiling) mit 15 ausgewählten Lektinen wurde konstruiert, um das vielseitige Glykoprotein-Glykan-Profiling für analytische Vollständigkeit zu erreichen.
Protokoll
Die Einzelheiten zu den Reagenzien und den für diese Studie verwendeten Geräten sind in der Materialtabelle aufgeführt.
1. Vorbereitung der spezialisierten Spitze für die automatische Erstellung von Glykanprofilen
- Bringen Sie die getrockneten, lektinfixierten Kügelchen, die bei 4 °C in einem verschlossenen Beutel gelagert werden, auf die Werkbank und lassen Sie sie wieder auf Raumtemperatur (~23 °C) zurückkehren.
HINWEIS: Dies verhindert Kondensation auf den Perlen, die deren Qualität beeinträchtigen kann. Über diese Methode zur Herstellung von Lektin-Fixierkügelchen wurde bereits berichtet5. - Legen Sie ein antistatisches Tuch auf den Schreibtisch und platzieren Sie den tragbaren Entladungsverstärker mit Blick auf den Arbeitsbereich. Dadurch können nachträgliche Arbeiten auf dem Prüfstand mit antistatischem Schutz durchgeführt werden (Abbildung 2A).
- Führen Sie die Perlen mit einer antistatischen Pinzette so in die leere Spitze ein, dass zwei Abstandsperlen zwischen den Lektin-Fixierperlen platziert werden (Abbildung 1A).
- Drücken Sie die Oberseite der Spitze vorsichtig mit einer Zange zusammen, um die Öffnung zu schließen, und achten Sie darauf, die Spitze nicht zu schneiden, nachdem alle Perlen gefüllt sind (Abbildung 2B).
HINWEIS: Dies dient dazu, die Perlen in der Spitze zu sichern. Es wird empfohlen, die Zange so zu gestalten, dass ein Durchtrennen der Spitze vermieden wird (Abbildung 2C). - Richte die Spitze mit der Hand aus, wenn sie verbogen ist.
- Bewahren Sie die vorbereitete Spitze bei 4 °C in einem verschlossenen Beutel mit einem Trockenmittel auf, bis sie benötigt wird.
HINWEIS: Bevor Sie die Spitze verwenden, sollte sie auf Raumtemperatur gebracht werden, bevor Sie den versiegelten Beutel öffnen und herausnehmen. Die Feuchtigkeit reduziert die Reaktivität der Lektine drastisch.
2. Vorbereitung der Analyten
- Verpacken Sie das Biotinylierungsreagenz für die Routineanalyse.
HINWEIS: Für die Routineanalytik ist es hilfreich, das Biotinylierungsreagenz (Biotin-(AC5)2 Sulfo-Osu, siehe Materialtabelle) in kleinen Portionen wie folgt vorzuverpacken, um eine schnelle Analyse zu ermöglichen. Es wird empfohlen, das Biotinylierungsreagenz zu trocknen und zu lagern, da sein Succinimidylester im Allgemeinen anfällig für Hydrolyse ist.- Das Biotinylierungsreagenz wird in entionisiertem Wasser gelöst.
- 10 μg des Reagenzes mit einer Pipette in Röhrchen abfüllen.
- Trocknen Sie die Röhrchen mit Biotinylierungsreagenz in einem gekühlten Vakuumkonzentrator und lagern Sie sie mit einem Trockenmittel bei 4 °C in einem lichtdichten Beutel bis zur Verwendung.
- Die Analytlösung (200 ng Äquivalent Protein) wird in das Röhrchen gegeben, das das in Schritt 2.1 hergestellte getrocknete Biotinylierungsreagenz enthält.
HINWEIS: Achten Sie auf die Pufferzusammensetzung, die zum Auflösen und Verdünnen der Analyten verwendet wird, abhängig von den Eigenschaften des verwendeten Biotinylierungsreagenzes. Vermeiden Sie beispielsweise die Verwendung von primären Aminreagenzien als Puffer für die Aminkopplung. Dadurch wird eine Verringerung der Biotinylierungseffizienz der Analyten verhindert. - Gut mischen, indem Sie es vortexen, dann in einer Tischzentrifuge nach unten schleudern (1000 x g für 5 s bei Raumtemperatur), um die Flüssigkeit auf den Deckel auf den Boden des Röhrchens fallen zu lassen.
- Inkubieren Sie die Röhrchen 1 h lang bei Raumtemperatur, abgeschirmt vor Licht.
- Verdünnen Sie die biotinylierten Proben 10-fach im Sondierungspuffer (1 % Triton X-100, 0,5 M Glycin, 1 mM CaCl2 und 1 mM MnCl2 in TBS), um alle nicht umgesetzten Biotinylierungsreagenzien zu inaktivieren.
HINWEIS: Bei Reaktionen durch Aminkopplung wird das nicht umgesetzte Biotinylierungsreagenz durch die Reaktion mit einem Puffer verbraucht, der ein primäres Aminreagenz wie Tris in TBS enthält, das die Biotinylierung von Lektinen und anderen Substanzen, die bei der nachfolgenden Messung auf den Kügelchen immobilisiert sind, verhindert. - Bei Raumtemperatur, lichtgeschützt, 2 h inkubieren.
- Die Biotinylierungseffizienz der Analyten ist durch Western Blot zu beurteilen, wobei bei Bedarf HRP-markiertes Streptavidin als Nachweissonde verwendet wird. Bitte beachten Sie die Literatur zu Western-Blotting-Methoden6.
3. Vorbereitung der Reagenzien für die Glykanprofilierung mit dem automatisierten Reaktionsmessgerät
- Verdünnen Sie das HRP-konjugierte Streptavidin mit der Blockierungslösung im Verhältnis 1:3000.
- Mischen Sie mit gleichen Volumina von Substrat A und Substrat B (siehe Materialtabelle). Verwenden Sie 160 μl gemischtes Substrat pro Probe.
- Geben Sie die Blockierungslösung, den Waschpuffer (0,1 % Triton X-100/TBS), TBS, HRP-konjugierte Streptavidin-Lösung und jedes Substrat in die dafür vorgesehenen Vertiefungen einer Kartusche mit 10 Flüssigkeitsbehältern.
HINWEIS: Das empfohlene Volumen und die Platzierung der Lösungen zum Befüllen jeder Vertiefung der Reaktionspatrone werden im Folgenden beschrieben. Die Lösungen und die Anordnung der Vertiefungen können jedoch im automatisierten Reaktionsmessgerät im Vorfeld entsprechend geändert werden.- 0,2 ml HRP-markiertes Streptavidin zugeben, das in Schritt 3.1 hergestellt wurde. mit einer Pipette zur Vertiefung #2. Die Vertiefung wird von der Vorderseite der Patrone aus als #1 bezeichnet.
- Geben Sie 0,2 ml Blockierungslösung mit einer Pipette in Vertiefung #3.
- Geben Sie 1 ml TBS mit einer Pipette in die Vertiefungen #4 und #6.
- Geben Sie mit einer Pipette 1 ml Waschpuffer in die Vertiefungen #7 und #8.
- Geben Sie 0,16 mL der Substratmischung mit einer Pipette in Vertiefung #10.
- Vergewissern Sie sich, dass die Wells #1, #5 und #9 leer sind.
- Geben Sie die in Schritt 2 vorbereiteten Analyten in ein 2 ml-Mikroröhrchen mit niedrigem Adsorptionsgrad und Schraubverschluss.
- Öffnen Sie die Fronttür des automatisierten Reaktionsmessgeräts.
- Die in Schritt 3.3 vorbereiteten Flüssigkeitsbehälter, die in 1 vorbereiteten Spitzen und die in Schritt 3.4 vorbereiteten Analyten werden wie folgt in die dafür vorgesehenen Positionen des automatischen Reaktionsmessgeräts (Abbildung 2D) gebracht. Jede Messung wird in einer einzigen vertikalen Linienrichtung durchgeführt.
- Platzieren Sie eine in Schritt 1 vorbereitete Spitze in jedem Loch an der Position "a" gelbes Symbol in Abbildung 2D.
HINWEIS: Auch hier gilt: Bringen Sie die Spitzen im Kühlschrank auf Raumtemperatur, bevor Sie den versiegelten Beutel öffnen. Lektine verlieren durch Feuchtigkeit dramatisch ihr Signal. - Entfernen Sie den Deckel des Röhrchens, das den in Schritt 3.4 hergestellten Analyten enthält, und führen Sie ihn in jede Öffnung an Position "b" auf ähnliche Weise ein.
- Setzen Sie die Kartusche mit der in Schritt 3.3 vorbereiteten Flüssigkeit ein und achten Sie darauf, dass sie nicht in Position "c" verschüttet wird.
- Entfernen Sie die gesamte "Abfallbox" in Abbildung 2D , wenn sie voll ist, werfen Sie den Inhalt in den Mülleimer, und bringen Sie die "Abfallbox" wieder an ihre ursprüngliche Position.
HINWEIS: Die reagierten Spitzen werden nach der Messung automatisch in der "Abfallbox" gesammelt.
- Platzieren Sie eine in Schritt 1 vorbereitete Spitze in jedem Loch an der Position "a" gelbes Symbol in Abbildung 2D.
4. Glykan-Profiling mit dem automatisierten Reaktionsmessgerät
- Schalten Sie den Netzschalter an der Seite des Hauptgeräts ein.
- Berühren Sie die auf dem Bildschirm angezeigte Schaltfläche "Diagnose starten" (Abbildung 3A). Das Selbstdiagnoseprogramm startet automatisch. Nach einer Weile ist das Selbstdiagnoseprogramm abgeschlossen, und der HOME-Bildschirm wird angezeigt (Abbildung 3B).
HINWEIS: Die folgenden Vorgänge können durch Berühren der entsprechenden Symbole ausgeführt werden. "Assay", um die Messung zu starten. "Wartung", um einen Systemfehler zu bestätigen. "Verlauf", um vergangene Messdaten anzuzeigen. - Berühren Sie "Assay", das auf dem HOME-Bildschirm angezeigt wird, um das Protokoll auszuwählen (Abbildung 3B).
- Wählen Sie den "BAssaySTD" der Assay-Methode aus (Abbildung 3C) und drücken Sie dann unten rechts auf "Weiter".
- Geben Sie bei Bedarf den Probennamen (Abbildung 3D) für jedes Muster ein, und tippen Sie dann unten rechts auf "Weiter".
- Geben Sie bei Bedarf den Namen der Spitze (Abbildung 3E) ein, der für jede Probe verwendet wird, und tippen Sie dann unten rechts auf "Weiter".
- Bestätigen Sie die Hinweise auf der rechten Seite (Abbildung 3F), und wenn es kein Problem gibt, berühren Sie "Start" unten rechts, um die Messung durchzuführen.
- Bestätigen Sie, dass der Bildschirm in den laufenden Modus wechselt (Abbildung 3G) und warten Sie, bis der Pfeil "Abschließen" hervorgehoben ist. Berühren Sie bei Bedarf "Pause", um den laufenden Modus zu stoppen.
HINWEIS: Die von dem automatischen Reaktionsmessgerät automatisch gesteuerten Vorgänge sind wie folgt: (1) Auto-Mikropipettendüsen im Inneren des Instruments können maximal acht Spitzen gleichzeitig anbringen (siehe gelbes Symbol "a" in Abbildung 2D); (2) Die Lösung in dem Röhrchen, das den Analyten enthält, 5 Minuten lang wiederholt aspirieren/ablassen; (3) In die Blockierungslösung überführen und 5 Minuten lang aspirieren/entladen; (4) Auf TBS übertragen und 2 Minuten lang aspirieren/entladen; (5) In die HRP-markierte Streptavidin-Lösung überführen und 5 min aspirieren/entleeren; (6) In den Waschpuffer umfüllen und 7 Minuten lang absaugen/entladen; (7) Auf TBS übertragen und 2 Minuten lang aspirieren/entladen; (8) In die Substratmischung überführen und sofort aspirieren; (9) Scannen Sie die Chemilumineszenz von acht Spitzen gleichzeitig auf der Rückseite des Geräts, um Spitzendaten zu erhalten. (10) Legen Sie gebrauchte Spitzen in die Abfallbox; (11) Die Düse kehrt in die Ausgangsposition zurück. - Vergewissern Sie sich, dass der Bildschirm in den Ergebnismodus gewechselt hat (Abbildung 3H), wenn die Messung abgeschlossen ist. Die acht Dotcodes werden gleichzeitig auf dem Bildschirm angezeigt, um eine einfache Überprüfung zu ermöglichen.
HINWEIS: Berühren Sie den "Verlauf" unten links, um das Scan-Diagramm (Abbildung 3I,J), das Balkendiagramm (Abbildung 3K,L), die Tabelle (Abbildung 3M) und die Details (Abbildung 3N) als Ergebnis auf dem Bildschirm anzuzeigen. Zeigen Sie die Ergebnisse aus anderen Lanes in der Dropdown-Liste "Lane" an. - Stecken Sie einen USB-Speicher in den USB-Anschluss an der Seite des Hauptgeräts und berühren Sie "Exportieren" unten rechts im Ergebnismodus, um die Daten aller Lanes einzeln auf dem USB-Speicher zu speichern.
- Exportieren Sie die Rohdaten (Abbildung 3M) als CSV-Datei über einen USB-Anschluss für die weitere Datenanalyse und individuelle grafische Darstellung in Excel. Der "Wert" der Tabelle gibt die Signalintensität jedes Lektins an. Die Intensitätswerte stellen den Maximalwert der Peakdaten (Abbildung 3I,J) im Scan der reagierten Spitzen dar.
HINWEIS: Der Schwellenwert für die Peak-Extraktion kann eingestellt werden. Abhängig von den Analyten kann das Signal im Spacerbead-Abschnitt insgesamt höher sein, wobei in diesem Fall der Wert im Spacer-Abschnitt als Hintergrund subtrahiert wird.
Ergebnisse
Zunächst wird die Signalverteilung für jedes Lektin gezeigt, um die Eigenschaften des Lektinbead-Arrays zu verstehen. Es wurde eine Bibliothek mit lektinfixierten Kügelchen (je 1.000 Kügelchen) erstellt. Fünfzehn Perlen wurden zufällig aus den 1.000 ausgewählt und zu einer einzigen Spitze hinzugefügt. Ein typisches Diagramm ist am unteren Rand von Abbildung 1A dargestellt und stellt fast die gleiche Signalintensität dar, die bei den 15 Kügelchen beobachtet wurde, die das gleiche Lektin immobilisieren. Dieses Messverfahren kann angewendet werden, um die Herstellung von handgefertigten Lektinkügelchen vor dem Einsatz von Glykanprofilen zu qualifizieren. So werden beispielsweise sechs Spitzen für die gleichzeitige Messung verwendet, um den Variationskoeffizienten (CV) aus den Signalen auf den 90 Kügelchen zu berechnen. Ein typisches Beispiel ist in Tabelle 1 dargestellt. Als die Lektin-fixierte Kügelchenbibliothek um 28 verschiedene Lektine erweitert wurde, zeigten 25 von 28 Lektinen eine hohe Reproduzierbarkeit mit einem CV von weniger als 10 %.
Als nächstes werden die Daten für ein Lektinbead-Array mit 15 verschiedenen Lektinen gezeigt. Die in Tabelle 2 gezeigten 15 Lektine wurden für das Lektinbead-Array ausgewählt. Zusätzlich wurden 12 in Tabelle 3 aufgeführte Polyacrylamid (PAA)-Mono/Di/Trisaccharide gemessen. Nach der Beurteilung der Messzuverlässigkeit anhand einer Dotcoding-Anzeige auf dem Bildschirm wurden die Rohdaten aus dem automatisierten Reaktionsmessgerät exportiert (siehe Protokollschritt 4.11) und in Excel ein Diagramm mit den Reaktionsspitzenwerten an jedem Saccharid erstellt (Abbildung 4A). Diese Werte wurden dann für die Hauptkomponentenanalyse verwendet, um die Korrelation zwischen den ausgewählten 15 Lektinen und den umgesetzten Sacchariden zu visualisieren (Abbildung 4B,C). Dies führte zu einem deutlichen Unterschied im Bindungsmuster jedes Lektins im Vergleich zu den 12 PAA-Zuckern, die in Abbildung 4A gezeigt sind. Hier wird gezeigt, dass sich die Validität jedes Signals auf integrierte vorherige Listen von Lektin- und Saccharidspezifitätenbezieht 7,8 (siehe auch diese Spezifitäten in Tabelle 2). Da AAL an terminale α-Fuc, Sia-Lex und Lex bindet, bestätigte die automatische Glykananalyse die Erkennung von Fucose-haltigen Molekülen durch AAL (Abbildung 4A). SSA und MAL (siehe Tabelle 2 für die vollständigen Namen der Lektine und die angegebenen Abkürzungen) erkannten Siaα2-6Gal/GalNAc bzw. Siaα2-3Gal. MAL im Bead-Array kann auch an 3'-O-Sulfo-Galβ3GalNAc und LacNAc binden. Dies steht im Einklang mit früheren Ergebnissen, die mit maschinellem Lernenanalysiert wurden 7, in denen MAL das 3'-O-Sulfo-Gal dominiert, während LacNAc nicht dominant ist9. RCA120, ein Gal-erkennendes Lektin, reagierte stark mit terminalem β-Gal, einschließlich Lac/LacNAc, und durfte an Siaα2-6Lac, aber nicht an Siaα2-3Lac binden. ECA hatte eine viel höhere Reaktivität mit LacNAc als mit Lac. WFA wies eine breitere Spezifität auf, die nicht nur α/β-GalNAc, sondern auch terminale β-Gal erkannte, wie berichtetwurde 7,10. GSL II, das GlcNAc und ein galaktosyliertes tri/tetra-Antennen-N-Glykane erkennt, bindete an keine Saccharide mit Ausnahme eines einzigen GlcNAc unter den präparierten Analyten. GRFT, das hohe N-Glykane vom Mannose-Typ erkennt, reagierte mit α-Man. ABA, Jacalin und MAH wurden im Allgemeinen zum Nachweis von O-Glykanen verwendet. Da ABA die Galβ1-3GalNAcα-Thr/Ser (T)- und Sialyl-T-Strukturen bevorzugt, reagierte ABA im Bead-Array stark mit 3'-O-Sulfo-Galβ1-3GalNAc und war schwach an α-GalNAc gebunden. ABA erkannte auch β-GlcNAc, was mit den früheren Berichten über die Bindung an agalactosylierte N-Glykane übereinstimmt 7. Jacalin hat eine relativ breitere Spezifität und reagierte daher mit α-Mannose, β-GalNAc, βGal, α-GalNAc, 3'-O-sulfo-Galβ1-3GalNAc. MAH erkannte spezifisch 3'-O-Sulfat-Galβ1,3GalNAc, wie zuvor berichtet7. Die restlichen vier Lektine, NPA, LCA, PHA-L und PHA-E, erkannten die innere Struktur von N-Glykan, das nicht in den hergestellten PAA-Sacchariden enthalten war, und wiesen daher keine Affinität zu allen Analyten auf.
Anhand der erhaltenen Daten kann die Ähnlichkeit der Lektine, die zur Bindung an jedes Saccharid beitragen, geklärt werden, indem sie in einem Hauptkomponentenanalysediagramm dargestellt werden (Abbildung 4B). Alle Saccharide, die zur Varianz in Abbildung 4B beitrugen, wurden durch Eigenvektor dargestellt, eine Methode der statistischen Analyse, bei der das Beitragsverhältnis durch einen Vektor dargestellt wird (Abbildung 4C). Bemerkenswert ist hierbei, dass die umgesetzten Saccharide unverzerrt zu jedem Lektin beitragen. Lektine zeigen keine Spezifität für nur eine Art von Saccharid. Zum Beispiel wird das Vorhandensein von Glykanen vom Komplextyp beim Menschen durch die teilweise Erkennung einiger Glykane innerhalb von Glykanen vom Komplextyp bestimmt. Jacalin, das O-Glykane erkennt (siehe die Spezifitäten in Tabelle 2), kann die 3'suTF-Struktur und α-GalNAc unter den in dieser Studie verwendeten Sacchariden erkennen, und daher wurden die 3'suTF und α-GalNAc gegen die Positionsrichtung von Jacalin (brauner Punkt) aufgetragen, die in Abbildung 4B dargestellt ist. WFA kann α/β-GalNAc unter den in dieser Studie verwendeten Sacchariden erkennen; Daher wurde α/β-GalNAc in Abbildung 4C in der gleichen Richtung wie der in Abbildung 4B dargestellte WFA dargestellt. Im Gegensatz dazu wird α-GalNAc zwischen WFA und Jacalin aufgetragen, da es sich um eine Saccharidstruktur handelt, an die Jacalin leicht binden kann. Alle 11 Saccharide, die in dieser Studie dargestellt wurden, stimmen mit der Spezifität (Saccharidbindung) der zuvor berichteten Lektine überein 7,8, was darauf hindeutet, dass die vorgeschlagene Methode eine zuverlässige Messmethode ist. Darüber hinaus sind die Eigenvektoren, die jedem Saccharid entsprechen, dispergiert, was dafür spricht, dass die 15 Lektine mit minimaler Verzerrung ausgewählt wurden, um eine umfassende Abdeckung in der Analyse zu gewährleisten. Folglich wurde die mit diesen 15 Lektinen aufgefüllte Spitze als Standard-GlycoBIST-Spitze definiert (Tabelle 2).
Im praktischeren Beispiel wurden gereinigte Proteinprodukte der Spitzenmessung unterzogen (Abbildung 5). Rinderthyreoglobulin, das eine N-Glykosylierungsstelle aufweist und komplexe, hybride und hohe Mannose-N-Glykane 11 enthält, wurde analysiert. Die Standardspitze zeigte eine erhöhte Reaktivität bestimmter Lektine gegenüber den Sacchariden8 (siehe die Besonderheiten in Tabelle 2): PHA-E, das die Teilung von GlcNAc erkennt; GRFT, Erkennung von N-Glykanen vom Mannose-Typ; SSA, Anerkennung von Siaα2-6Gal/GalNAc; RCA120, erkennt Laktose und LacNAc; und AAL, das Fucose erkennt. Diese Glykanstrukturen sind in komplexen, hybriden und N-Glykanen vom hohen Mannosetyp vorhanden, was darauf hindeutet, dass die N-Glykanstruktur von Thyreoglobulin11 effektiv bewertet werden könnte. Darüber hinaus zeigte Thyreoglobulin, das mit Sialidase A, einem Enzym, das Sialinsäure verdaut, behandelt wurde, eine verminderte Reaktivität mit SSA, das Sialinsäure erkennt, und eine erhöhte Reaktivität mit RCA120, WFA und ECA, die nach der Entfernung von Sialinsäuren leichter erkannt werden. Das Vorhandensein von O-Glykanstrukturen in Rinderthyreoglobulin wurde bisher nicht berichtet. Erwartungsgemäß wurde O-Glykan in Thyreoglobulin mit oder ohne Sialidase-Behandlung in der automatischen Glykananalyse nicht erkannt.
Ein Reproduzierbarkeitstest innerhalb des Laufs mit den 15 ausgewählten Lektinen wird gezeigt, um die Robustheit der Messung zu verstehen (Tabelle 4). Für diesen Test wurden sieben Standardspitzen für Analyten und eine zusätzliche Spitze für die Negativkontrolle vorbereitet, wobei alle acht Spitzen gleichzeitig gemessen wurden. Dieser Vorgang wurde an einem einzigen Tag dreimal wiederholt. "Mix Analytes", eine Kombination von Analyten, die dazu führte, signifikante Signale auf allen Lektinen zu erhalten, wurden für die Qualitätskontrolle des Beads-Arrays vorbereitet. Die Mix-Analyten wurden durch geeignete Mischung von Sialidase-verdautem Erythropoietin (EPO), Sialidase- und Galactosidase-verdautem EPO, humanem IgA, Matrix-Metalloproteinase 3 (MMP3) und Thyreoglobulin formuliert. Jedes Glykoprotein (200 ng) wurde mit 10 μg Biotinylierungsreagenz in PBS markiert, das Triton X-100 enthielt. Falls erforderlich, wurden Sialidase- und Galactosidase-Behandlungen wie in der Anleitung beschrieben durchgeführt. Nach der Verdauung wurden die Produkte in einem Hitzeblock bei 75 °C für 10 min inkubiert, um das Enzym zu inaktivieren. Die entsprechenden Mengen an biotinylierten Glykoproteinen (3 ng EPO, 8 ng humanes IgA, 5 ng MMP3 und 15 ng Thyreoglobulin) wurden unmittelbar vor der Analyse gemischt. Die Messergebnisse zeigten, dass der maximale CV-Wert 13,5 % und der durchschnittliche CV für die 15 Lektine 8,2 % betrug, was eine hohe Reproduzierbarkeit zeigt.
Darüber hinaus wird gezeigt, dass ein täglicher Reproduzierbarkeitstest die Schwankungen aufgrund des Messdatums versteht (Tabelle 5). Standardspitzen und Mix-Analyten wurden im Voraus vorbereitet, und die Messungen von sieben Spitzen wurden täglich an fünf aufeinanderfolgenden Tagen wiederholt. Die Ergebnisse zeigten, dass die meisten Lektine einen CV von weniger als 10% aufwiesen. Einige Lektine, wie z. B. LCA und ECA, wiesen jedoch höhere CVs auf. Die durchschnittliche CV der 15 Lektine pro Zyklus betrug bis zu 7,7% und die durchschnittlichen CV-Werte über 5 Tage lagen bei weniger als 10%, was auf eine hohe Reproduzierbarkeit hindeutet. Es wurde festgestellt, dass Jacalin aufgrund der Selbstverdauung von MMP3 im Mix-Analyten eine geringe zeitliche Reproduzierbarkeit zeigte. Daher sollte ein geeigneterer Analyt gefunden werden, um MMP3 zu ersetzen.
Um die Stabilität von Lektin-fixierten Kügelchen zu verstehen, wird ein Langzeitstabilitätstest mit diesen getrockneten lektinfixierten Kügelchen gezeigt (Tabelle 6). Standardspitzen, Assay-Reagenzien und Mischanalyten wurden im Voraus vorbereitet, und die Messungen wurden nach 12-monatiger Lagerung durchgeführt. Die oben genannten fünf Arten von biotinylierten Analyten wurden einzeln gelagert und vor der Messung gemischt. Die Ergebnisse zeigten, dass der durchschnittliche CV für die 15 Lektine weniger als 10% betrug, selbst bis zu 12 Monate nach der Trockenlagerung. Dies deutet auf die Machbarkeit schneller und präziser Messungen hin, indem mehrere Spitzen (bis zu 1.000) aus derselben Charge gleichzeitig gelagert werden.
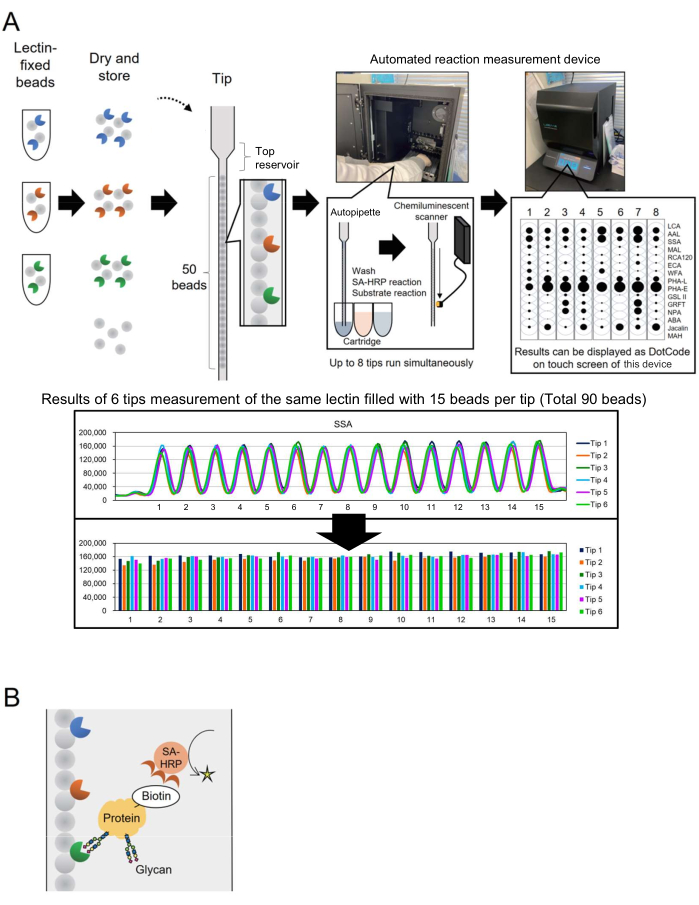
Abbildung 1: Schemata des "Bead-Arrays in einer einzigen Spitze"-Instrument und Analysemethode. (A) Schema des automatischen Glykan-Profiling-Systems. Lektinfixierte Kügelchen können getrocknet und in Spitzen aufbewahrt werden. Wenn Spitzen und Kartuschen, die Reagenzien wie HRP-markierte Anti-Streptavidin-Antikörper (SA-HRP) enthalten, in das Messgerät (automatisiertes Reaktionsmessgerät) gesetzt und aktiviert werden, fungiert die Spitze als Autopipette. Der Chemilumineszenz-Detektionsscanner an der Rückseite des Instruments quantifiziert Signale von acht Spitzen gleichzeitig. Quantitative Daten werden als Punktcodes auf dem Touchscreen des Geräts angezeigt, um eine schnelle Bestätigung der Messergebnisse zu ermöglichen. Der untere Bereich zeigt die Ergebnisse der Messung von 90 Kügelchen mit dem gleichen Lektin. Die Messergebnisse werden vom Gerät transportiert und vom einzelnen Forscher grafisch dargestellt. (B) Schema der in diesem Versuch verwendeten Nachweismethode. Die Zielproteine werden präbiotinyliert und mit SA-HRP nachgewiesen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2: Bilder von Materialien und Aufbau für die automatische Glykanprofilierung mit dem automatisierten Reaktionsmessgerät. (A) Darstellung von Lektin-fixierenden Kügelchen, die in eine Spitze gepackt werden. Nehmen Sie die Kügelchen mit einer antistatischen Pinzette auf die Antistatikmatte und füllen Sie die Spitze mit den Perlen. (B) Kneifen Sie die Spitze mit einer Zange ein. Ein leichter oberer Teil der oberen Perle wird zusammengedrückt. (C) Eine Zange zum Crimpen der Spitzen. Der Kunststoff (gelber Kreis) wird eingeklemmt, um zu verhindern, dass sie vollständig abgeschnitten werden. (D) Anordnung der in der automatisierten Reaktionsmesseinrichtung verwendeten Materialien. A: Spitzen, B: Röhrchen mit Analyten und C: Kartuschen mit Flüssigkeit sollten eingesetzt werden. In der Abfallbox werden die reagierten Spitzen nach der Messung gesammelt. Die Box kann entnommen werden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
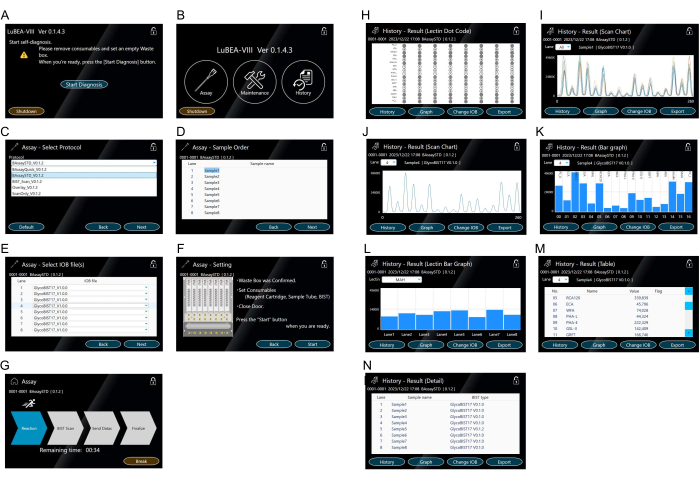
Abbildung 3: Touchscreen für die Maschinenmanipulation. (A) Das Selbstdiagnoseprogramm wird automatisch gestartet, wenn das Gerät eingeschaltet wird. (B) Auf dem HOME-Bildschirm werden "Assay", "Wartung" und "Verlauf" angezeigt. (C-G) Betrieb des "Assay"-Modus. Auswahl der Assay-Methode (C). Mehrere Protokolle können im Voraus registriert werden. Eingabe der Reihenfolge der Probe (Analyt) (D). Auswahl des Typs der Spitze (E). Bestätigung der Einstellungen der Assay-Methode (F) und Starten des Assays über den Bildschirm "Assay starten" (G). (H-N) Darstellung des Ergebnisses in "History". Anzeige des Lektin-Dotcodes (H) nach Abschluss des Assays oder im "Verlauf"-Modus. Auf dem Bildschirm wird für alle Proben (I) oder für jede Probe (J) ein Scan-Diagramm mit 15 Lektinen angezeigt. Das Scandiagramm wird als Balkendiagramm (K) angezeigt. Balkendiagramme für andere Fahrspuren können aus der Dropdown-Liste "Bahn" ausgewählt werden. Balkendiagramm, das acht Spuren von Proben pro Lektin (L) vergleicht. Andere Lektine können aus der Dropdown-Liste "Lektin" ausgewählt werden. Tabelle mit quantitativen Werten (M). Aufzeichnung des ausgeführten Messverfahrens (N). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
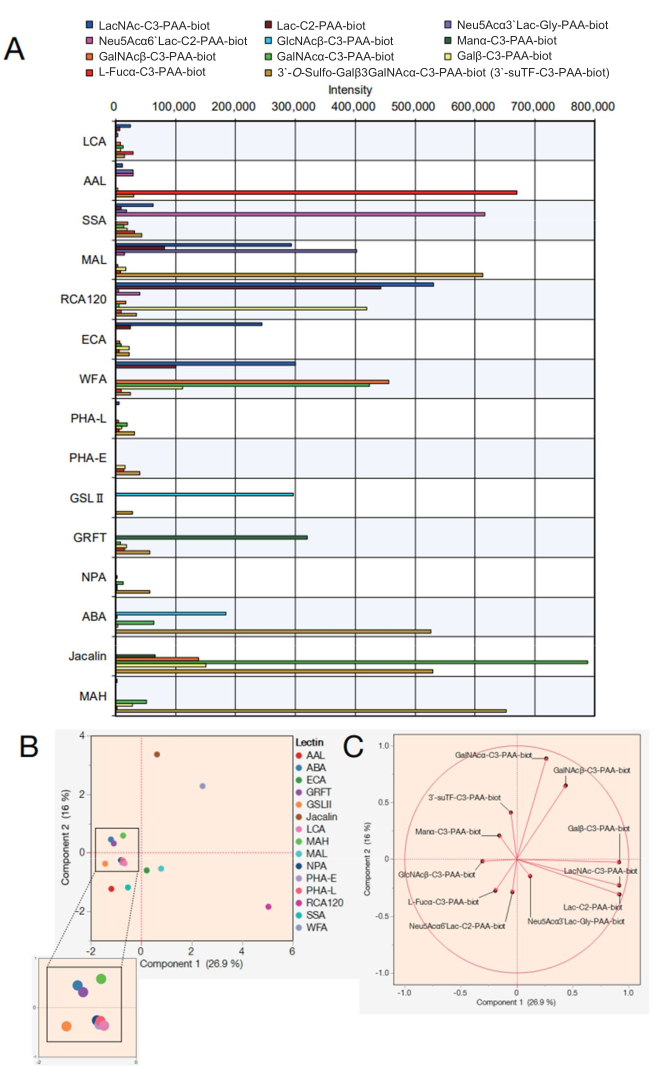
Abbildung 4: Bewertung der Reaktivität mit PAA-Sacchariden im automatischen Glykan-Profiling-System. (A) Ein Diagramm, das die Reaktivität von Lektinen mit verschiedenen PAA-Sacchariden zeigt, wurde um negative Kontrollwerte bereinigt. (B) Analyse der Hauptkomponenten auf der Grundlage der Daten in (A). (C) Eigenvektoren aus der Analyse in (B). Für die Teile (B) und (C) wurde eine kompatible Analysesoftware verwendet. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
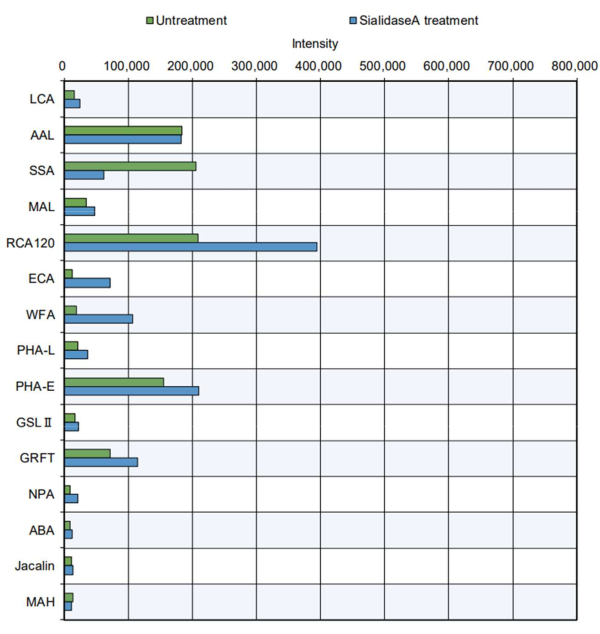
Abbildung 5: Nachweis der GlycoBIST-Reaktivität anhand von gereinigtem bovinem Thyreoglobulinprotein. Die Probenvorbereitung ohne Behandlung wurde unter den gleichen Aufschlussbedingungen wie der Puffer allein durchgeführt, ohne Zugabe von Sialidase A. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Tabelle 1: Wiederholbarkeitstest mit lektinfixierten Kügelchen. Die Wiederholbarkeit der Reaktivität für 28 Lektine wurde untersucht. Mit jeder Spitze wurden Analyten umgesetzt. EPO: Erythropoietin, PSA: Prostata-spezifisches Antigen, Tf: Transferrin, M2BP: Mac-2-Bindungsprotein, hIgG: humanes IgG, MMP3: Matrix-Metalloproteinase 3, (Sia+): Sialidase A-verdaute Analyten, (Sia+, Gal+): Sialidase A und Galactosidase-verdaute Analyten, Ab: Antikörper. DSA: Datura stramonium Agglutinin, HypninA2: Hypnea japonica Agglutinin, WGA: Weizenkeim-Agglutinin, UDA: Urtica dioica Agglutinin, BPL: Bauhinia purpurea Lektin, Orysata: Oryza sativa Lektin, LSL-N: Laetiporus sulphureus Lektin N-terminale Domäne, SNA: Sambucus nigra Lektin, BanLec: Bananenlektin, MPA: Maclura pomifera Agglutinin, TxLC-I: Tulipa gesneriana Agglutinin, AOL: Aspergillus oryzae Lektin, ACG: Agrocybe cylindracea Galectin. CV: Variationskoeffizient. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Tabelle 2: Liste der Lektine, die die Standardspitze für die automatische Glykanprofilierung bilden. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Tabelle 3: PAA-Saccharide, die als Standards für das automatische Glykan-Profiling-System verwendet werden. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Tabelle 4: Ergebnisse des Reproduzierbarkeitstests innerhalb eines Laufs. CV: Variationskoeffizient. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Tabelle 5: Ergebnisse des Reproduzierbarkeitstests zwischen den Tagen. CV: Variationskoeffizient. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Tabelle 6: Ergebnisse des Langzeitstabilitätstests. CV: Variationskoeffizient. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Diskussion
In dieser Studie wurde eine schnelle Evaluierungstechnik für die Glykosylierung unter Verwendung der "Bead Array in a Single Tip"-Technologie entwickelt. In der vorliegenden Studie wurde eine Standard-GlycoBIST-Spitze eingeführt, die sowohl für Glykowissenschaftler als auch für Nicht-Glykowissenschaftler entwickelt wurde, um eine routinemäßige, umfassende Glykosylierungsbewertung zu ermöglichen. Das Lektin-Microarray, das typischerweise 20-100 Lektine12,13 verwendet, wurde in großem Umfang in Evaluierungen eingesetzt. Unter Berücksichtigung der überlappenden Spezifitäten einiger Lektine auf dem Microarray und der relativ begrenzten Vielfalt an Glykoformen auf einem Zielglykoprotein im Vergleich zur Glykomik roher klinischer Proben wurde jedoch erwartet, dass 15 Lektine für eine vereinfachte Bewertung des fokussierten Glykanprofilings ausreichen würden.
Der entscheidende Schritt im Protokoll besteht darin, die mit Lektin immobilisierten Kügelchen oder Spitzen, die mit diesen Kügelchen gefüllt sind, nach der Lagerung wieder auf Raumtemperatur zu bringen, bevor sie für Messungen verwendet werden. Insbesondere wurde beobachtet, dass Kondensation die Signale schwächt; Daher wird empfohlen, den Aufbewahrungsbeutel erst zu öffnen, wenn er wieder Raumtemperatur erreicht hat. Für die Lagerung von Biotinylierungsreagenzien ist es ebenfalls unerlässlich, vor der Lagerung vollständig zu trocknen. Ein unzureichender Trocknungsprozess verringert die Effizienz der Biotinylierung zum Protein aufgrund der Hydrolyse des Biotinylierungsreagenzes14.
Im Hinblick auf Modifikationen und Fehlerbehebung wurde eine Methode zur Reduzierung der Variabilität untersucht, insbesondere in Bezug auf die Messmethode in der automatisierten Reaktionsmesseinrichtung. Insbesondere wenn das Puffervolumen in dem für die Analyse verwendeten Reservoir 150 μl beträgt, gelangen Blasen in die Spitze, was zu einer verringerten Reaktionseffizienz und einer großen Variation der Werte führt. Daher wird empfohlen, mindestens 200 μl Puffer in das Reservoir zu geben.
Auch die Menge an Flüssigkeit, die die Spitzen beim automatischen Pipettieren füllt, ist ein wichtiger Faktor. Die Spitzen können die Flüssigkeit nicht vollständig ausstoßen, wenn sie zum nächsten Reaktionsschritt übergeht. Daher wird in nicht geringer Menge der Puffer aus dem vorherigen Schritt in den nächsten Schritt übertragen, und der vorherige Puffer verbleibt im oberen Bereich der eingefügten Lösung, wenn der neue Puffer aspiriert wird. Daher sollten das Substrat und andere Lösungen ausreichend angesaugt werden, um den oberen Behälter zu füllen (siehe Abbildung 1A). Angesichts der Irreversibilität von Antikörperreaktionen auf dem Gerät ist es wichtig, bei der Ersteinrichtung Vorsicht walten zu lassen und Fehler zu vermeiden.
Eine Einschränkung des Verfahrens besteht darin, dass es auf lösliche Proteine beschränkt ist, die aus Serum- und Kulturüberständen gewonnen werden. Bei der derzeitigen Methode ist Lektin nicht kovalent mit dem Kügelchen vernetzt, so dass es nicht möglich ist, Proben zu analysieren, die hohe Konzentrationen von Tensiden enthalten, wie z. B. Zell- und Gewebeextrakte. Folglich sind zukünftige Verbesserungen erforderlich, um diese Einschränkungen zu beheben.
Die Bedeutung des Lektinperlenarrays liegt in der Austauschbarkeit und Erweiterbarkeit der auf den Kügelchen fixierten Lektinspezies. Bei einer groß angelegten Differentialanalyse (>1.000 Proben) mit dem automatisierten Glykan-Profiling-System können Benutzer beispielsweise eine 15-Lektin-Aufstellung in der Spitze auf der Grundlage eines vorläufigen Lektin-Microarray-basierten Glykanprofils des Zielglykoproteins (<100 Proben) anpassen. Darüber hinaus ermöglicht die hohe Stabilität der lektinfixierten Kügelchen ein sofortiges Experimentieren von der ersten Spitzenkonstruktion bis zur Messung mit Routineverfahren. Nicht nur die 25 von 28 Lektinen, die eine hohe Zuverlässigkeit gezeigt haben (Tabelle 1), sondern alle Lektine, die für den Benutzer von Interesse sind, können verwendet werden, um ein benutzerdefiniertes automatisiertes Glykanprofilierungssystem für seine spezifischen Experimente zu erstellen, nachdem der oben erwähnte routinemäßige Zuverlässigkeitstest durchgeführt wurde. Dieser Ansatz führte zur Entwicklung eines möglichen 120-Lektin-Bead-Arrays mit acht verschiedenen Arten von Spitzen für eine erweiterte Messung in Verbindung mit dem Standard-Glykan-Profiling-System.
Eine frühere Studie konzentrierte sich auf die Etablierung eines Nachweisverfahrens für Cy3-markierte Glykoproteine in Anlehnung an den Lektin-Microarray15. Diese Methode war aufgrund der Fluoreszenzinterferenz auf 13 Lektine parallel in der Spitze beschränkt. Die aktuelle Methode, bei der HRP-Detektionsmarkierungen verwendet werden, nimmt 15 Lektin-fixierte Kügelchen und zwei Kontrollkügelchen (positiv und negativ) in einer Spitze auf. Darüber hinaus hat die Biotinylierung von Proben den Analyseprozess verkürzt.
Diese Analysemethode kann nicht nur in der akademischen Forschung, sondern auch in der medizinischen und pharmazeutischen Forschung, in der Lebensmittel- und Kosmetikindustrie und in anderen Industriebereichen angewendet werden. Obwohl Lektin-Microarray- und Glykoproteomik-Technologien gut etabliert sind, bleibt der Ansatz dieser Studie aufgrund seiner vollständigen Automatisierung und der Fähigkeit zur schnellen Analyse mit weniger Schritten von Bedeutung. In Zukunft wird diese Analyse zu einer quantitativen Methode erweitert, indem das Signal mit bestimmten Glykoproteinen normalisiert wird, wobei Antikörper-fixierte Kügelchen neben Lektin-fixierten Kügelchen in einer Spitze verwendet werden.
Offenlegungen
AO, TO, NT, HS, KK, MA, SY, TM, KN, OS und KS sind Mitarbeiter von Precision System Science Co., Ltd.
Danksagungen
Diese Arbeit wurde durch das Adaptable and Seamless Technology Transfer Program through Target-driven R&D (A-STEP) unterstützt, das von der Japan Science and Technology Agency (JST) unter der Fördernummer JPMJTR204A und teilweise von der Japan Society for the Promotion of Science (JSPS) KAKENHI Fördernummer 23H02680 an die AK finanziert wurde. Wir danken Editage (www.editage.jp) für die Bearbeitung der englischen Sprache.
Materialien
| Name | Company | Catalog Number | Comments |
| 0.2 mL 8 PCR tubes + flat cap | FastGene | FG-0028FC/SE | For dispensing and storage of biotin labeling reagents |
| 2.0 mL SC Micro Tube protein LB | SARSTEDT AG&Co. KG | 72.694.600 | For loading samples in LuBEA-VIII |
| Analysis software | SAS Institute Inc. | JMP 17 | |
| Anti-Static Ionizer. 110 volts | Plas-Labs | 800-AS/SPI | Use while inserting beads into BIST Tip. |
| Antistatic tweezers NK2A | RUBIS | 9-5681-01 | Use while inserting beads into BIST Tip. |
| Automated reaction measurement device | Precision System Science Co., Ltd. | LuBEA | All data in this study were obtained using LuBEA-VIII. While LuBEA is commercialized, LuBEA-VIII is currently in the development phase. The website for product details of LuBEA and LuBEA-VIII can be found at the following URL (https://www.pss.co.jp/english/technology/apit/bist02.html). |
| Biotin-(AC5)2 Sulfo-Osu | Dojindo | 341-06801 | As biotinilation reagent |
| BIST spacer bead- V-11 | Precision System Science Co., Ltd. | F4938000 | Diameter: 1 mm |
| BIST tip and sheath | Precision System Science Co., Ltd. | F4930-000 | Fill this empty tip with beads. |
| Calcium chloride | Wako | 039-00475 | For preparing buffer solutions |
| Can Get Signal Solution 2 | TOYOBO | NKB-301 | This blocking reagent is good for this method |
| CentriVap Benchtop Vacuum Concentrators | Labconco | 7810010 | For dry of biotinilation reagent. The substitution provided by other companies is possible. |
| Clarity Western ECL Substrate | Bio-Rad | 1705060 | For measuring the samples by LuVEA VIII |
| Cold trap CentriVap series | Labconco | 7460040 | For dry of biotinilation reagent. The substitution provided by other companies is possible. |
| Glycine | Wako | 077-00735 | For preparing buffer solutions |
| Manganese(II) Chloride Tetrahydrate | Wako | 139-00722 | For preparing buffer solutions |
| Milli-Q reference | MERCK | ZIQ7000T0C | As deionazed water for dilution of reagents and buffers |
| PBS | Wako | 162-19321 | For preparing buffer solutions |
| Peroxidase Streptavidin | Jackson | 016-030-084 | This antibody is good for GlycoBIST analysis. It is useful for the detection of the western blotting, although substitutions can be provided by other companies. |
| Probe bead | Precision System Science Co., Ltd. | FP4936000 | For preparing lectin-fixed beads. See Shimazaki, H. et al., Current Protocols in Protein Science, 2020 in reference section for detail. |
| Protein LoBind Tubes 1.5 mL | Eppendorf | 0030108442 | Use while preparing the samples such as biotinylation. |
| Silica gel (for desiccan) | Kanto chemical | 37039-02 | Use while storing the GlycoBIST tip and dried biotinylation reagent. |
| Static electricity removal sheet M | TRUSCO NAKAYAMA | SD5050 | Use while inserting beads into BIST Tip. |
| SV Cartridge II | Daido Chemical Industry | ED041 | For measuring the samples by LuVEA VIII |
| Table Microcentrifuge-CAPSULEFUGE | TOMY | PMC-060 | For spin-down of the mixture in each process |
| Triton X-100 | Nacalai tesque | 35501-15 | For preparing buffer solutions |
Referenzen
- Butler, M., Reichl, U. Animal cell expression systems. Adv Biochem Eng Biotechnol. 175, 1-36 (2021).
- Silva, M. L. S. Chapter One - Lectin biosensors in cancer glycan biomarker detection. Adv Clin Chem. 93, 1-61 (2019).
- Hiono, T., Nagai-Okatani, C., Kuno, A. 4.07 - Application of glycan-related microarrays. Comprehensive Glycoscience (Second Edition). , 134-148 (2021).
- Shimazaki, H., et al. Lectin bead array in a single tip facilitates fully automatic glycoprotein profiling). Anal Chem. 91 (17), 11162-11169 (2019).
- Shimazaki, H., et al. GlycoBIST: A System for automatic glycan profiling of a target protein using milli-bead array in a tip. Curr Protoc Protein Sci. 99 (1), e103(2020).
- Mahmood, T., Yang, P. C. Western blot: Technique, theory, and trouble shooting. N Am J Med Sci. 4 (9), 429-434 (2012).
- Bojar, D., et al. A useful guide to lectin binding: machine-learning directed annotation of 57 unique lectin specificities. ACS Chem Biol. 17 (11), 2993-3012 (2022).
- Nagai-Okatani, C., et al. 2.0: An integrated visualization for lectin microarray-based mouse tissue glycome mapping data with lectin histochemistry. J Proteome Res. 20 (4), 2069-2075 (2021).
- Geisler, C., Jarvis, D. L. Effective glycoanalysis with Maackia amurensis lectins requires a clear understanding of their binding specificities. Glycobiology. 21 (8), 988-993 (2011).
- Noro, E., et al. N -glycan structures of Wisteria floribunda agglutinin-positive Mac2 binding protein in the serum of patients with liver fibrosis. Glycobiology. 31 (10), 1268-1278 (2021).
- Rawitch, A. B., Pollock, H. G., Yang, S. X. Thyroglobulin glycosylation: Location and nature of the N-Linked oligosaccharide units in bovine thyroglobulin. Arch Biochem Biophys. 300 (1), 271-279 (1993).
- Dang, K., Zhang, W., Jiang, S., Lin, X., Qian, A. Application of lectin microarrays for biomarker discovery. Chemistry Open. 9 (3), 285-300 (2020).
- Chen, S., Qin, R., Mahal, L. K. Sweet systems: Technologies for glycomic analysis and their integration into systems biology. Crit Rev Biochem Mol Biol. 56 (3), 301-320 (2021).
- Dwivedi-Agnihotri, H., Srivastava, A., Shukla, A. K. Reversible biotinylation of purified proteins for measuring protein-protein interactions. Methods Enzymol. 633, 281-294 (2020).
- Shimazaki, H., et al. Auto-lectin dotcoding by two octopuses: Rapid analysis of fluorescence-labeled glycoproteins by an 8-channel fully-automatic bead array scanner with a rolling-circle detector. Anal Chem. 95 (32), 11868-11873 (2023).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten