Method Article
Valutazione rapida glico-qualitativa delle proteine ricombinanti utilizzando un sistema completamente automatizzato
In questo articolo
Riepilogo
In precedenza era stato sviluppato un sistema per l'analisi automatizzata e rapida dei glicani sulle proteine. Questo articolo presenta il protocollo per l'"analisi glico-qualitativa", ottimizzato per un ampio spettro di utenti, come quelli impegnati nell'analisi delle strutture dei glicani nei biofarmaci e in altri materiali glicoconiugati.
Abstract
La glicosilazione delle proteine, una modifica post-traduzionale critica, influenza la stabilità, l'efficacia e l'immunogenicità delle proteine ricombinanti, compresi i biofarmaci. Le strutture dei glicani mostrano una significativa eterogeneità, che varia in base ai tipi di cellule di produzione, alle condizioni di coltura e ai metodi di purificazione. Di conseguenza, il monitoraggio e la valutazione delle strutture dei glicani delle proteine ricombinanti è vitale, in particolare nella produzione biofarmaceutica. Il microarray di lectine, tecnica complementare alla spettrometria di massa, vanta un'elevata sensibilità e facilità d'uso. Tuttavia, in genere richiede più di un giorno per produrre risultati. Per adattarlo alla ricerca non sulla scienza del glico o allo sviluppo di processi di prodotti farmaceutici, è necessaria un'alternativa automatizzata e ad alto rendimento. Pertanto, è stato sviluppato il primo sistema di profilazione dei glicani completamente automatizzato al mondo a base di lectina, utilizzando il concetto di tecnologia "bead array in a single tip (BIST)". Questo sistema consente la preparazione e lo stoccaggio di perle immobilizzate con lectina in unità da 1.000, con ordini di inserimento paralleli personalizzabili per vari scopi. Questo articolo presenta un protocollo pratico per la ricerca che coinvolge proteine ricombinanti "glico-qualificate". Dopo aver testato la loro reattività contro 12 coniugati poliacrilammide-glicano, sono state selezionate 15 lectine per aumentare la versatilità del sistema. Inoltre, il processo di etichettatura dei campioni è stato ottimizzato passando da Cy3 alla biotina, riducendo il tempo di elaborazione complessivo di 30 minuti. Per una qualificazione immediata dei dati, i segnali di legame delle lectine vengono visualizzati come un codice punto sul monitor superiore. L'affidabilità del sistema è stata confermata attraverso test di riproducibilità giornalieri, test di ripetibilità e test di conservazione a lungo termine, con un coefficiente di variazione del <10%. Questo glicoanalizzatore rapido e facile da usare ha potenziali applicazioni nel monitoraggio della qualità delle glicoproteine endogene per la valutazione e la convalida dei biomarcatori. Questo metodo facilita l'analisi per coloro che sono nuovi alla scienza del glico, ampliando così la sua utilità pratica.
Introduzione
La glicosilazione delle proteine è una modifica post-traduzionale cruciale che deve essere valutata nei biofarmaci. I profili dei glicani delle proteine possono variare in base alle condizioni di coltura, ai processi di purificazione e alle cellule ospiti1. Sono necessari strumenti semplici per qualificare la glicosilazione all'interno della pipeline del bioprocesso. Si stima che oltre il 50% delle proteine secrete e di membrana in vivo siano modificate con glicani multipli, che cambiano in base al lignaggio cellulare, allo stadio di sviluppo e allo stato della malattia, come l'insorgenza della malignità2. Il monitoraggio dei profili dei glicani ha un potenziale significativo per l'identificazione di marcatori diagnostici unici e bersagli farmacologici. Gli strumenti automatizzati in grado di misurare rapidamente campioni di grandi dimensioni sono molto richiesti per verificare e convalidare tale glicosilazione aberrante da centinaia di campioni di pazienti nella pipeline di scoperta.
La tecnologia dei microarray è stata incorporata nella glicomica per valutare il profilo dei glicani delle glicoproteine3. In questo metodo, diverse lectine, che sono proteine leganti i glicani, vengono immobilizzate su una superficie come un vetrino. Questa tecnologia di glicoanalisi basata sull'interazione non richiede il rilascio anticipato di glicani dalle proteine del nucleo, semplificando il processo per i ricercatori che non conoscono la glicotecnologia. Nonostante il suo uso diffuso, per applicazioni industriali come la bioproduzione era necessario un sistema automatizzato in grado di monitorare rapidamente e facilmente i glicani per un numero maggiore di target di analisi. Per risolvere questo problema, è stato precedentemente segnalato un sistema automatizzato di profilazione dei glicani basato su un concetto unico chiamato "array di perline in una singola punta" (BIST), inizialmente sviluppato per la genotipizzazione. Questo sistema semplifica il processo con un auto-instrument ad alta produttività di tipo one-box4. Utilizzando punte in cui varie perle fissate con lectina sono disposte in parallelo 4,5, è stato messo a punto un metodo per analizzare le strutture dei glicani modificate nelle glicoproteine e denominato GlycoBIST (di seguito denominato "sistema automatico di profilazione dei glicani") (Figura 1A). Le lectine possono essere fissate su 1.000 perline ed essiccate per mantenere l'attività per un anno, sia prima che dopo l'imballaggio in una punta. Una volta che i puntali e le cartucce contenenti reagenti come l'anticorpo anti-streptavidina marcato con HRP (SA-HRP) sono stati inseriti nello strumento prototipo di misurazione (un dispositivo di misurazione automatica della reazione, vedere la Tabella dei materiali), il puntale funziona come un'autopipetta. Uno scanner di rilevamento a chemiluminescenza nella parte posteriore interna dello strumento quantifica i segnali di otto punte contemporaneamente. I dati quantitativi di queste otto punte vengono visualizzati in modo compatto e simultaneo come codici punto sul touchscreen dello strumento per una rapida conferma dei risultati della misurazione. Inoltre, il valore rappresentato come il massimo del picco misurato viene trasportato dallo strumento come dati grezzi e consente la rappresentazione grafica da parte dei singoli ricercatori (Figura 1A, pannello inferiore).
In questo articolo, gli autori descrivono un metodo migliorato per marcare le proteine della biotina, riducendo il tempo di elaborazione a 30 minuti. Le proteine bersaglio sono biotinilate in anticipo e rilevate da SA-HRP (Figura 1B). Per ottenere la versatile profilazione dei glicani per la completezza analitica, è stata costruita una punta GlycoBIST standard (punta specializzata per la profilazione automatica dei glicani) con 15 lectine selezionate.
Protocollo
I dettagli dei reagenti e delle attrezzature utilizzate per questo studio sono elencati nella Tabella dei Materiali.
1. Preparazione della punta specializzata per la profilazione automatica dei glicani
- Portare le perle essiccate fissate con lectina, conservate a 4 °C in un sacchetto sigillato, sul banco e lasciarle tornare a temperatura ambiente (~23 °C).
NOTA: In questo modo si evita la formazione di condensa sulle perline, che può diminuirne la qualità. Questo metodo di preparazione delle perle di fissaggio delle lectine è stato precedentemente riportato5. - Posizionare un panno antistatico sulla scrivania e posizionare l'eliminatore di elettricità statica portatile rivolto verso l'area di lavoro. Ciò consente di eseguire lavori successivi sul banco con protezione antistatica (Figura 2A).
- Inserire le perline nella punta vuota utilizzando una pinzetta antistatica in modo che due perline distanziatrici siano posizionate tra le perle di fissaggio della lectina (Figura 1A).
- Schiacciare delicatamente la parte superiore della punta con una tronchestra per chiudere l'apertura, facendo attenzione a non tagliare la punta dopo che tutte le perline sono state riempite (Figura 2B).
NOTA: Questo serve per fissare le perline all'interno della punta. Si consiglia di realizzare la tronchessina per evitare di tagliare la punta (Figura 2C). - Raddrizzare la punta a mano se è piegata.
- Conservare la punta preparata a 4 °C in un sacchetto sigillato con un essiccante fino al momento del bisogno.
NOTA: Prima di utilizzare la punta, deve essere portata a temperatura ambiente prima di aprire il sacchetto sigillato ed estrarlo. L'umidità riduce drasticamente la reattività delle lectine.
2. Preparazione degli analiti
- Preconfezionare il reagente di biotinilazione per l'analisi di routine.
NOTA: Per le analisi di routine, è utile preconfezionare il reagente di biotinilazione (Biotina-(AC5)2 Sulfo-Osu, vedere la Tabella dei materiali) in piccole porzioni come segue per un'analisi rapida. Si raccomanda di essiccare e conservare il reagente di biotinilazione, poiché il suo estere succinimidil è generalmente suscettibile all'idrolisi.- Sciogliere il reagente di biotinilazione in acqua deionizzata.
- Erogare 10 μg di reagente nelle provette utilizzando una pipetta.
- Asciugare le provette con il reagente di biotinilazione in un concentratore sottovuoto refrigerato e conservarle con un essiccante a 4 °C in un sacchetto resistente alla luce fino al momento dell'uso.
- Aggiungere la soluzione dell'analita (200 ng eq. di proteine) alla provetta contenente il reagente di biotinilazione essiccato preparato al punto 2.1.
NOTA: Prestare attenzione alla composizione del tampone utilizzata per sciogliere e diluire gli analiti, a seconda delle proprietà del reagente di biotinilazione utilizzato. Ad esempio, evitare di utilizzare reagenti amminici primari come tamponi per l'accoppiamento ammina. Ciò impedirà una riduzione dell'efficienza di biotinilazione degli analiti. - Mescolare bene con il vortice, quindi centrifugare verso il basso (1000 x g per 5 s a temperatura ambiente) in una centrifuga da tavolo per far cadere il liquido sul coperchio fino al fondo della provetta.
- Incubare le provette per 1 ora a temperatura ambiente, al riparo dalla luce.
- Diluire i campioni biotinilati 10 volte nel tampone di sonda (1% Triton X-100, 0,5 M Glycine, 1 mM CaCl2 e 1 mM MnCl2 in TBS) per inattivare eventuali reagenti di biotinilazione non reagiti.
NOTA: Nel caso di reazioni per accoppiamento amminico, il reagente di biotinilazione non reagito viene consumato dalla reazione con un tampone contenente un reagente amminico primario come Tris in TBS, che impedisce la biotinilazione delle lectine e di altre sostanze immobilizzate sulle perle nella misurazione successiva. - Incubare a temperatura ambiente, al riparo dalla luce, per 2 ore.
- Valutare l'efficienza della biotinilazione degli analiti attraverso il Western blotting, utilizzando la streptavidina marcata con HRP come sonda di rilevamento, se necessario. Si prega di fare riferimento alla letteratura per i metodi di Western blotting6.
3. Preparazione dei reagenti per la profilazione dei glicani con il dispositivo di misurazione automatica della reazione
- Diluire la streptavidina coniugata con HRP con la soluzione bloccante in rapporto 1:3000.
- Miscelare con volumi uguali di substrato A e substrato B (vedere la tabella dei materiali). Utilizzare 160 μl di substrato misto per campione.
- Erogare la soluzione bloccante, il tampone di lavaggio (0,1% Triton X-100/TBS), TBS, la soluzione di streptavidina coniugata HRP e ciascun substrato nei pozzetti designati di una cartuccia con 10 serbatoi di liquido.
NOTA: Il volume consigliato e il posizionamento delle soluzioni per riempire ciascun pozzetto della cartuccia di reazione sono descritti di seguito. Tuttavia, le soluzioni e la disposizione dei pozzetti possono essere modificate in anticipo nel dispositivo di misurazione automatica della reazione, a seconda dei casi.- Aggiungere 0,2 mL di streptavidina marcata con HRP preparata al punto 3.1. al pozzo #2 usando una pipetta. Il pozzetto è chiamato #1 dalla parte anteriore della cartuccia.
- Aggiungere 0,2 mL di soluzione bloccante al pozzetto #3 utilizzando una pipetta.
- Aggiungere 1 mL di TBS ai pozzetti #4 e #6 utilizzando una pipetta.
- Aggiungere 1 mL di tampone di lavaggio ai pozzetti #7 e #8 utilizzando una pipetta.
- Aggiungere 0,16 mL di miscela di substrato al pozzetto #10 utilizzando una pipetta.
- Verificare che i pozzetti #1, #5 e #9 siano vuoti.
- Aggiungere gli analiti preparati nella fase 2 a una microprovetta con tappo a vite a basso contenuto proteico da 2 mL.
- Aprire lo sportello anteriore del dispositivo di misurazione automatica della reazione.
- Posizionare i serbatoi di liquido preparati al punto 3.3, i puntali preparati al punto 1 e gli analiti preparati al punto 3.4 nelle posizioni designate del dispositivo di misurazione automatica della reazione (Figura 2D) come segue. Ogni misurazione viene eseguita in un'unica direzione di linea verticale.
- Posizionare una punta preparata al punto 1 in ciascun foro in corrispondenza del simbolo giallo della posizione "a" nella Figura 2D.
NOTA: Anche in questo caso, se refrigerato, portare le punte a temperatura ambiente prima di aprire il sacchetto sigillato. Le lectine perdono drasticamente il loro segnale a causa dell'umidità. - Rimuovere il coperchio della provetta contenente l'analita preparato al punto 3.4 e inserirlo in ciascun foro in posizione "b" in modo analogo.
- Inserire la cartuccia contenente il liquido preparato al punto 3.3, facendo attenzione a non rovesciarla in posizione "c".
- Rimuovere l'intero "contenitore dei rifiuti" nella Figura 2D quando è pieno, gettare il contenuto nel cestino della spazzatura e riportare il "contenitore dei rifiuti" nella sua posizione originale.
NOTA: Le punte reagite vengono raccolte automaticamente nella "scatola dei rifiuti" dopo la misurazione.
- Posizionare una punta preparata al punto 1 in ciascun foro in corrispondenza del simbolo giallo della posizione "a" nella Figura 2D.
4. Profilazione dei glicani con il dispositivo di misurazione automatica della reazione
- Accendere l'interruttore di alimentazione sul lato dell'unità principale.
- Toccare il pulsante "Avvia diagnosi" visualizzato sullo schermo (Figura 3A). Il programma di autodiagnosi si avvia automaticamente. Dopo un po' di tempo, il programma di autodiagnosi è completato e viene visualizzata la schermata HOME (Figura 3B).
NOTA: Le seguenti operazioni possono essere eseguite toccando le rispettive icone. "Saggio" per avviare la misurazione. "Manutenzione" per confermare eventuali errori di sistema. "Cronologia" per visualizzare i dati di misurazione passati. - Toccare "Assay" visualizzato nella schermata HOME per selezionare il protocollo (Figura 3B).
- Selezionare il "BAssaySTD" del metodo di analisi (Figura 3C) e quindi premere "Avanti" in basso a destra.
- Immettere il nome del campione (Figura 3D) per ciascun campione, se necessario, quindi toccare "Avanti" in basso a destra.
- Immettere il nome della punta (Figura 3E) utilizzata per ciascun campione, se necessario, quindi toccare "Avanti" in basso a destra.
- Confermare le note a destra (Figura 3F) e, se non ci sono problemi, toccare "Start" in basso a destra per la misurazione.
- Confermare l'interruttore dello schermo sulla modalità di funzionamento (Figura 3G) e attendere fino a quando non viene evidenziata la freccia "finalizza". Se necessario, toccare "Interrompi" per interrompere la modalità di marcia.
NOTA: Le operazioni controllate automaticamente dal dispositivo di misurazione automatica della reazione sono le seguenti: (1) gli ugelli per micropipette automatiche all'interno dello strumento attaccano un massimo di otto puntali contemporaneamente (vedere il simbolo "a" giallo nella Figura 2D); (2) Aspirare/scaricare ripetutamente la soluzione nella provetta contenente l'analita per 5 minuti; (3) Trasferire nella soluzione bloccante e aspirare/scaricare per 5 minuti; (4) Trasferimento a TBS e aspirazione/scarico per 2 minuti; (5) Trasferire nella soluzione di streptavidina marcata con HRP e aspirare/scaricare 5 minuti; (6) Trasferire nel tampone di lavaggio e aspirare/scaricare per 7 minuti; (7) Trasferimento a TBS e aspirazione/scarico per 2 minuti; (8) Trasferire nella miscela di substrato e aspirare immediatamente; (9) Scansionare la chemiluminescenza di otto punte contemporaneamente sul retro del dispositivo per ottenere i dati di picco; (10) Versare i puntali usati nel contenitore dei rifiuti; (11) L'ugello ritorna nella posizione iniziale. - Verificare che la schermata sia passata alla modalità risultato (Figura 3H) al termine della misurazione. Gli otto codici punto vengono visualizzati contemporaneamente sullo schermo per una facile verifica.
NOTA: Toccare la "Cronologia" in basso a sinistra per visualizzare il grafico di scansione (Figura 3I, J), il grafico a barre (Figura 3K, L), la tabella (Figura 3M) e i dettagli (Figura 3N) come risultato sullo schermo. Visualizza i risultati di altre corsie dall'elenco a discesa "Corsia". - Inserire una memoria USB nella porta USB sul lato dell'unità principale e toccare "Esporta" in basso a destra della modalità risultato per salvare i dati di tutte le corsie singolarmente nella memoria USB.
- Esporta i dati grezzi (Figura 3M) come file CSV utilizzando una porta USB per un'ulteriore analisi dei dati e grafici individuali in Excel. Il "valore" della tabella indica l'intensità del segnale di ciascuna lectina. I valori di intensità rappresentano il valore massimo dei dati di picco (Figura 3I,J) nella scansione delle punte reagite.
NOTA: È possibile impostare la soglia di estrazione di picco. A seconda degli analiti, il segnale nella porzione del distanziatore può essere complessivamente più alto, nel qual caso il valore nella porzione del distanziatore viene sottratto come sfondo.
Risultati
Innanzitutto, viene mostrata la distribuzione del segnale per ciascuna lectina per comprendere le caratteristiche dell'array di perline di lectina. È stata preparata una libreria di perline fissate con lectina (1.000 perline ciascuna). Quindici perline sono state scelte a caso tra le 1.000 e aggiunte a una singola punta. Un grafico tipico è mostrato nella parte inferiore della Figura 1A, che rappresenta quasi la stessa intensità del segnale osservata tra le 15 perle che immobilizzano la stessa lectina. Questa procedura di misurazione può essere applicata per qualificare la produzione di perle di lectina fatte a mano prima dell'uso della profilazione dei glicani. Ad esempio, per la misurazione simultanea vengono utilizzate sei punte per calcolare il coefficiente di variazione (CV) dai segnali sulle 90 perline. Un esempio tipico è mostrato nella Tabella 1. Quando la libreria di perline fissate con lectina è stata ampliata per ospitare 28 diverse lectine, 25 su 28 lectine hanno dimostrato un'elevata riproducibilità con un CV inferiore al 10%.
Successivamente, vengono mostrati i dati per un array di perline di lectina con 15 diverse lectine. Le 15 lectine mostrate nella Tabella 2 sono state selezionate per l'array di perline di lectine. Inoltre, sono stati misurati 12 mono/di/trisaccaridi di poliacrilammide (PAA) elencati nella Tabella 3. Dopo aver valutato l'affidabilità della misurazione utilizzando un display di codifica a punti sullo schermo, i dati grezzi sono stati esportati dal dispositivo di misurazione automatica della reazione (vedere il passaggio 4.11 del protocollo) ed è stato creato un grafico in Excel utilizzando i valori di picco della reazione in ciascun saccaride (Figura 4A). Questi valori sono stati poi utilizzati per l'analisi delle componenti principali per visualizzare la correlazione tra le 15 lectine selezionate e i saccaridi reagiti (Figura 4B, C). Ciò ha comportato una netta differenza nel modello di legame di ciascuna lectina rispetto ai 12 zuccheri PAA mostrati nella Figura 4A. In questo caso, si dimostra che la validità di ciascun segnale si riferisce a precedenti elenchi integrati di specificità di lectine e saccaridi 7,8 (fare riferimento anche a queste specificità nella Tabella 2). Poiché l'AAL si lega al α-Fuc terminale, Sia-Lex e Lex, l'analisi automatica dei glicani ha confermato il riconoscimento da parte dell'AAL delle molecole contenenti fucosio (Figura 4A). SSA e MAL (vedere la Tabella 2 per i nomi completi delle lectine e le abbreviazioni date) hanno riconosciuto rispettivamente Siaα2-6Gal/GalNAc e Siaα2-3Gal. Il MAL nell'array di perline può anche legarsi a 3'-O-sulfo-Galβ3GalNAc e LacNAc. Ciò è coerente con i risultati precedenti analizzati utilizzando l'apprendimento automatico7, in cui MAL preferisce il 3'-O-sulfo-Gal in modo dominante, mentre LacNAc non è dominante9. RCA120, una lectina che riconosce il Gal, ha reagito fortemente con il β-Gal terminale, incluso Lac/LacNAc, ed è stato permesso di legarsi a Siaα2-6Lac ma non a Siaα2-3Lac. L'ECA ha avuto una reattività con LacNAc molto più elevata di Lac. La WFA aveva una specificità più ampia, che riconosceva non solo α/β-GalNAc ma anche β-Gal terminale, come riportato 7,10. GSL II, che riconosce GlcNAc e un N-glicani galattosilati tri/tetra-antennari, non si è legato a nessun saccaride ad eccezione di un singolo GlcNAc tra gli analiti preparati. La GRFT, che riconosce alti N-glicani di tipo mannosio, ha reagito con α-Man. ABA, Jacalin e MAH sono stati generalmente utilizzati per rilevare gli O-glicani. Poiché l'ABA preferisce le strutture Galβ1-3GalNAcα-Thr/Ser (T) e sialil-T, l'ABA nell'array di perline ha reagito fortemente con il 3'-O-sulfo-Galβ1-3GalNAc e si è debolmente legato al α-GalNAc. L'ABA ha anche riconosciuto il β-GlcNAc, in linea con i precedenti rapporti sul legame con gli N-glicani agalactosilati7. La jacalina ha una specificità relativamente più ampia e quindi ha reagito con α-mannosio, β-GalNAc, βGal, α-GalNAc, 3'-O-sulfo-Galβ1-3GalNAc. Il MAH ha riconosciuto specificamente il 3'-O-solfato-Galβ1,3GalNAc come precedentemente riportato7. Le quattro lectine residue, NPA, LCA, PHA-L e PHA-E, riconoscevano la struttura interna dell'N-glicano non incluso nei saccaridi PAA preparati e quindi non avevano affinità con tutti gli analiti.
Utilizzando i dati ottenuti, la somiglianza delle lectine che contribuiscono al legame con ciascun saccaride può essere chiarita tracciandole su un grafico di analisi delle componenti principali (Figura 4B). Tutti i saccaridi che hanno contribuito alla varianza nella Figura 4B sono stati rappresentati da autovettori, un metodo di analisi statistica in cui il rapporto di contribuzione è rappresentato da un vettore (Figura 4C). È interessante notare che i saccaridi reagiti contribuiscono a ciascuna lectina senza pregiudizi. Le lectine non mostrano specificità per un solo tipo di saccaride. Ad esempio, la presenza di glicani di tipo complesso è determinata dal riconoscimento parziale di alcuni glicani all'interno di glicani di tipo complesso nell'uomo. Jacalin, che riconosce gli O-glicani (vedi le specificità nella Tabella 2), può riconoscere la struttura 3'suTF e α-GalNAc tra i saccaridi utilizzati in questo studio, e quindi, il 3'suTF e il α-GalNAc sono stati tracciati (Figura 4C) rispetto alla direzione di posizione di Jacalin (punto marrone) tracciata nella Figura 4B. WFA è in grado di riconoscere α/β-GalNAc tra i saccaridi utilizzati in questo studio; pertanto, il α/β-GalNAc nella Figura 4C è stato tracciato nella stessa direzione del WFA tracciato nella Figura 4B. Al contrario, α-GalNAc è tracciato tra WFA e Jacalin perché è una struttura saccaridica a cui Jacalin è in grado di legarsi facilmente. Tutti gli 11 saccaridi tracciati in questo studio sono coerenti con la specificità (legame saccaridico) delle lectine riportata in precedenza 7,8, indicando che il metodo proposto è un metodo di misurazione affidabile. Inoltre, gli autovettori corrispondenti a ciascun saccaride sono dispersi, supportando il fatto che le 15 lectine sono state selezionate con una distorsione minima per garantire una copertura completa nell'analisi. Di conseguenza, la punta rifornita con queste 15 lectine è stata definita come la punta GlycoBIST standard (Tabella 2).
Nell'esempio più pratico, i prodotti proteici purificati sono stati sottoposti alla misurazione della punta (Figura 5). È stata analizzata la tireoglobulina bovina, che ha un sito di N-glicosilazione e contiene N-glicani complessi, ibridi e ad alto contenuto di mannosio11. La punta standard indicava un aumento della reattività di alcune lectine ai saccaridi8 (fare riferimento alle specificità nella Tabella 2): PHA-E, che riconosce la bisezione di GlcNAc; GRFT, che riconosce gli N-glicani di tipo mannosio; SSA, che riconosce Siaα2-6Gal/GalNAc; RCA120, che riconosce il lattosio e il LacNAc; e AAL, che riconosce il fucosio. Queste strutture dei glicani sono presenti in N-glicani complessi, ibridi e ad alto contenuto di mannosio, suggerendo che la struttura N-glicani della tireoglobulina11 potrebbe essere valutata efficacemente. Inoltre, la tireoglobulina trattata con sialidasi A, un enzima che digerisce l'acido sialico, ha mostrato una diminuzione della reattività con SSA, che riconosce l'acido sialico, e un aumento della reattività con RCA120, WFA ed ECA, che sono più facilmente riconoscibili dopo la rimozione degli acidi sialici. La presenza di strutture O-glicani nella tireoglobulina bovina non è stata precedentemente riportata. Come previsto, non c'è stato alcun riconoscimento di O-glicano nella tireoglobulina con o senza trattamento con sialidasi nell'analisi automatica dei glicani.
È stato dimostrato che un test di riproducibilità all'interno della corsa utilizzando le 15 lectine selezionate comprende la robustezza della misurazione (Tabella 4). Per questo test, sono stati preparati sette puntali standard per gli analiti e un puntale aggiuntivo per il controllo negativo, e tutti e otto i puntali sono stati misurati contemporaneamente. Questa procedura è stata ripetuta tre volte in un solo giorno. Gli "analiti di miscelazione", una combinazione di analiti che ha portato all'ottenimento di segnali significativi su tutte le lectine, sono stati preparati per il controllo di qualità dell'array di sfere. Gli analiti Mix sono stati formulati mescolando opportunamente eritropoietina digerita con sialidasi (EPO), EPO digerita con sialidasi e galattosidasi, IgA umana, metalloproteinasi 3 della matrice (MMP3) e tireoglobulina. Ogni glicoproteina (200 ng) è stata marcata con 10 μg di reagente di biotinilazione in PBS contenente Triton X-100. Se necessario, i trattamenti con sialidasi e galattosidasi sono stati eseguiti come descritto nelle istruzioni. Dopo la digestione, i prodotti sono stati incubati in un blocco termico a 75 °C per 10 minuti per inattivare l'enzima. Le quantità appropriate di glicoproteine biotinilate (3 ng di EPO, 8 ng di IgA umane, 5 ng di MMP3 e 15 ng di tireoglobulina) sono state miscelate immediatamente prima dell'analisi. I risultati della misurazione hanno indicato che il valore CV massimo era del 13,5% e il CV medio per le 15 lectine era dell'8,2%, dimostrando un'elevata riproducibilità.
Inoltre, è stato dimostrato che un test di riproducibilità giornaliero comprende le variazioni dovute alla data di misurazione (Tabella 5). I puntali standard e gli analiti Mix sono stati preparati in anticipo e le misurazioni di sette puntali sono state ripetute ogni giorno per cinque giorni consecutivi. I risultati hanno mostrato che la maggior parte delle lectine aveva un CV inferiore al 10%. Tuttavia, alcune lectine, come LCA ed ECA, hanno mostrato CV più elevate. Il CV medio delle 15 lectine per ciclo è stato fino al 7,7% e i valori CV medi su 5 giorni sono stati inferiori al 10%, il che significa un'elevata riproducibilità. È stato notato che la jacalina, a causa dell'autodigestione di MMP3 nell'analita Mix, ha dimostrato una bassa riproducibilità temporale. Pertanto, è necessario identificare un analita più adatto per sostituire MMP3.
Per comprendere la stabilità delle perle fissate con lectina, viene mostrato un test di stabilità a lungo termine utilizzando queste perle fissate con lectina essiccate (Tabella 6). I puntali standard, i reagenti per saggi e gli analiti per miscele sono stati preparati in anticipo e le misurazioni sono state eseguite dopo 12 mesi di conservazione. I suddetti cinque tipi di analiti biotinilati sono stati conservati singolarmente e miscelati prima della misurazione. I risultati hanno mostrato che il CV medio per le 15 lectine era inferiore al 10%, anche fino a 12 mesi dopo lo stoccaggio a secco. Ciò suggerisce la fattibilità di misurazioni rapide e precise memorizzando contemporaneamente numerose punte (fino a 1.000) dallo stesso lotto.
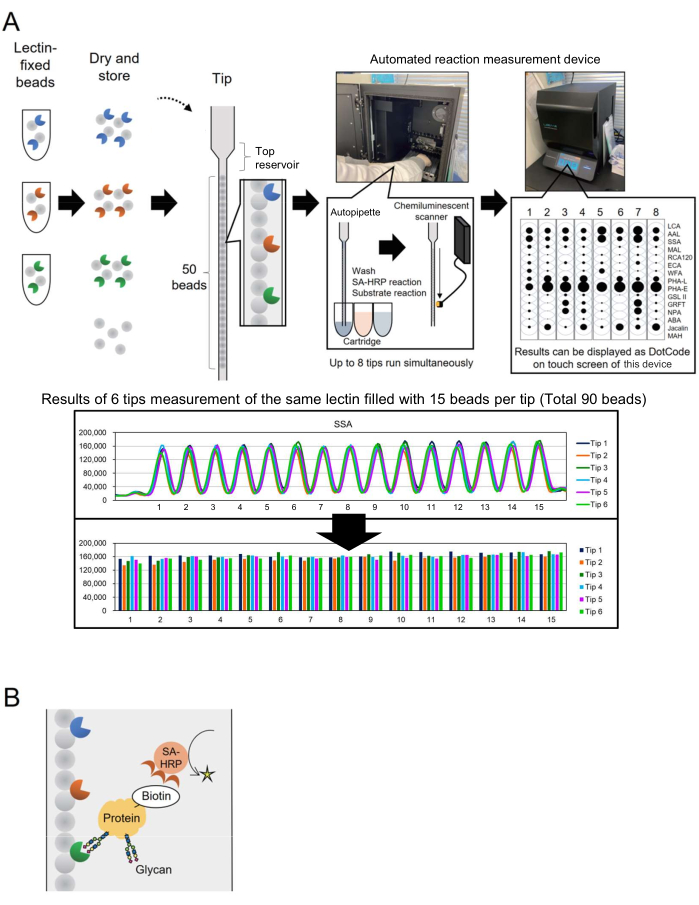
Figura 1: Schemi dello strumento "bead array in a single tip" e metodo di analisi. (A) Schema del sistema automatico di profilazione dei glicani. Le perle fissate con lectina possono essere essiccate e conservate in punte. Quando i puntali e le cartucce contenenti reagenti come l'anticorpo anti-streptavidina marcato con HRP (SA-HRP) vengono inseriti nello strumento di misura (dispositivo di misurazione automatica della reazione) e attivati, il puntale funziona come un'autopipetta. Lo scanner di rilevamento della chemiluminescenza sul retro dello strumento quantifica i segnali provenienti da otto punte contemporaneamente. I dati quantitativi vengono visualizzati come codici punto sul touchscreen dello strumento per una rapida conferma dei risultati della misurazione. La sezione inferiore mostra i risultati della misurazione di 90 perle con la stessa lectina. I risultati delle misurazioni vengono trasportati dallo strumento e rappresentati graficamente dal singolo ricercatore. (B) Schema del metodo di rilevamento utilizzato in questo esperimento. Le proteine bersaglio sono pre-biotinilate e rilevate utilizzando SA-HRP. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 2: Immagini dei materiali e configurazione per la profilazione automatica dei glicani con il dispositivo di misurazione automatica della reazione. (A) Rappresentazione di perle che fissano la lectina inserite in una punta. Raccogliere le perline con una pinzetta antistatica sul tappetino antistatico e riempire la punta con le perline. (B) Pizzicare la punta con una tronchesina. Una leggera parte superiore della perlina superiore viene schiacciata. (C) Una pinza per crimpare le punte. La plastica (cerchio giallo) viene pizzicata per evitare che si stacchino completamente. (D) Disposizione dei materiali utilizzati nel dispositivo automatico di misurazione della reazione. A: I puntali, B: la provetta contenente analiti e C: le cartucce contenenti liquido devono essere inserite in posizione, rispettivamente. Nella scatola dei rifiuti, le punte reagite vengono raccolte dopo la misurazione. La scatola può essere rimossa. Clicca qui per visualizzare una versione più grande di questa figura.
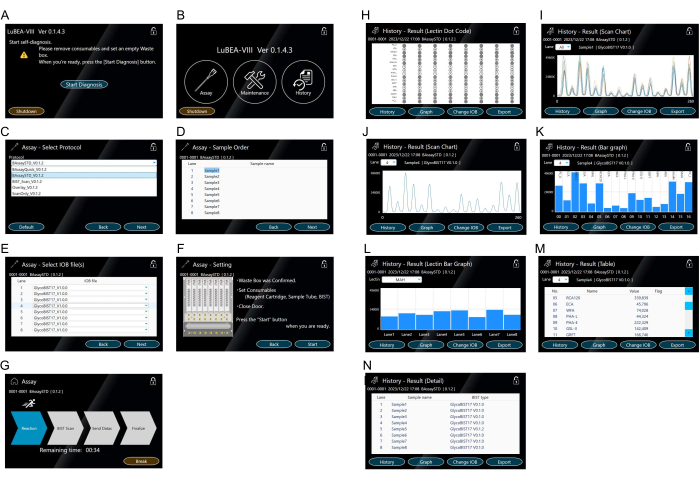
Figura 3: Touch screen per la manipolazione della macchina. (A) Il programma di autodiagnosi si avvia automaticamente all'accensione dell'unità. (B) La schermata HOME visualizza "Saggio", "Manutenzione" e "Cronologia". (C-G) Funzionamento della modalità "Analisi". Selezione del metodo di saggio (C). È possibile registrare in anticipo più protocolli. Inserimento dell'ordine del campione (analita) (D). Selezione del tipo di punta (E). Conferma delle impostazioni del metodo di analisi (F) e avvio del test dalla schermata "Avvia analisi" (G). (H-N) Rappresentazione del risultato nella "Storia". Codice punto lectina (H) visualizzato dopo il completamento del test o dalla modalità "Cronologia". Sullo schermo viene visualizzato un grafico di scansione di 15 lectine per tutti i campioni (I) o per ciascun campione (J). Il grafico di scansione viene visualizzato come grafico a barre (K). I grafici a barre per altre corsie possono essere selezionati dall'elenco a discesa "Corsia". Grafico a barre che confronta otto corsie di campioni per lectina (L). Altre lectine possono essere selezionate dall'elenco a discesa "Lectina". Tabella che mostra i valori quantitativi (M). Registrazione del metodo di misurazione eseguito (N). Clicca qui per visualizzare una versione più grande di questa figura.
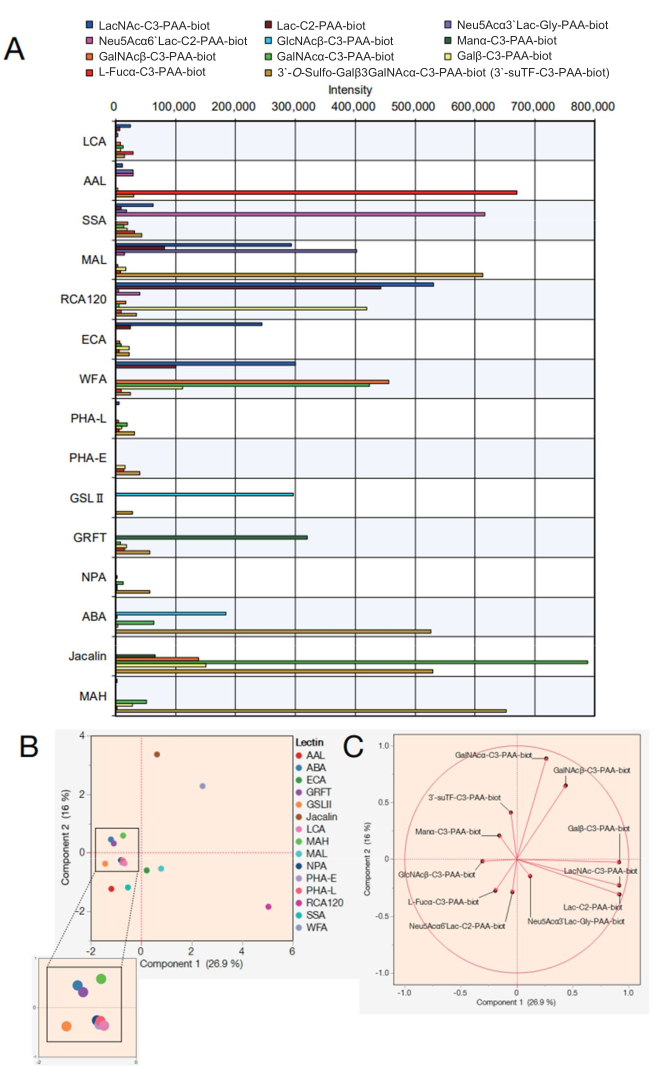
Figura 4: Valutazione della reattività con i saccaridi PAA nel sistema automatico di profilazione dei glicani. (A) Un grafico che illustra la reattività delle lectine con vari saccaridi PAA è stato aggiustato per i valori di controllo negativi. (B) Analisi delle componenti principali basata sui dati di cui al punto (A). (C) Autovettori dall'analisi in (B). Per le parti (B) e (C) è stato utilizzato un software di analisi compatibile. Clicca qui per visualizzare una versione più grande di questa figura.
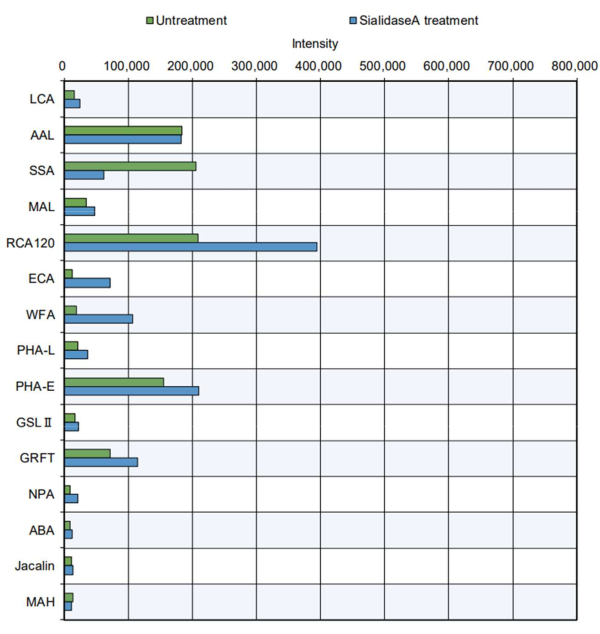
Figura 5: Verifica della reattività di GlycoBIST utilizzando la proteina tireoglobulina bovina purificata. La preparazione del campione senza trattamento è stata condotta nelle stesse condizioni di digestione del solo tampone, escludendo l'aggiunta di sialidasi A. Fare clic qui per visualizzare una versione più grande di questa figura.
Tabella 1: Test di ripetibilità con perle fissate con lectina. È stata valutata la ripetibilità della reattività per 28 lectine. Gli analiti sono stati fatti reagire ad ogni punta. EPO: eritropoietina, PSA: antigene prostatico specifico, Tf: transferrina, M2BP: proteina legante Mac-2, hIgG: IgG umane, MMP3: metalloproteinasi 3 della matrice, (Sia+): analiti digeriti con sialidasi A, (Sia+, Gal+): analiti con sialidasi A e galattosidasi digeriti, Ab: anticorpo. DSA: Datura stramonium agglutinin, HypninA2: Hypnea japonica agglutinin, WGA: agglutinina germe di grano, UDA: Urtica dioica agglutinina, BPL: Bauhinia purpurea lectina, Orysata: Oryza sativa lectina, LSL-N: Laetiporus sulphureus lectina dominio N-terminale, SNA: Sambucus nigra lectina, BanLec: lectina banana, MPA: Maclura pomifera agglutinina, TxLC-I: Tulipa gesneriana agglutinina, AOL: Aspergillus oryzae lectina, ACG: Agrocybe cylindracea galectina. CV: coefficiente di variazione. Clicca qui per scaricare questa tabella.
Tabella 2: Elenco delle lectine che costituiscono la punta standard per la profilazione automatica dei glicani. Clicca qui per scaricare questa tabella.
Tabella 3: Saccaridi PAA utilizzati come standard per il sistema automatico di profilazione dei glicani. Clicca qui per scaricare questa tabella.
Tabella 4: Risultati del test di riproducibilità all'interno della corsa. CV: coefficiente di variazione. Clicca qui per scaricare questa tabella.
Tabella 5: Risultati del test di riproducibilità tra un giorno e l'altro. CV: coefficiente di variazione. Clicca qui per scaricare questa tabella.
Tabella 6: Risultati della prova di stabilità a lungo termine. CV: coefficiente di variazione. Clicca qui per scaricare questa tabella.
Discussione
In questo studio, è stata sviluppata una tecnica di valutazione rapida per la glicosilazione che impiega la tecnologia "bead array in a single tip". Il presente studio ha introdotto una punta GlycoBIST standard, progettata sia per i ricercatori di glicoscienze che per quelli non glicoscientifici, per facilitare la valutazione completa e di routine della glicosilazione. Il microarray di lectine, che impiega tipicamente 20-100 lectine12,13, è stato ampiamente utilizzato nelle valutazioni. Tuttavia, considerando le specificità sovrapposte di alcune lectine sul microarray e la varietà relativamente limitata di glicoforme su una glicoproteina bersaglio rispetto ai glicomici di campioni clinici grezzi, si prevedeva che 15 lectine sarebbero state sufficienti per una valutazione semplificata del profilo dei glicani focalizzati.
La fase critica del protocollo consiste nel consentire alle perle immobilizzate con lectina o alle punte riempite con tali perle di tornare a temperatura ambiente dopo la conservazione prima di utilizzarle per le misurazioni. In particolare, è stato osservato che la condensa indebolisce i segnali; Pertanto, si consiglia di non aprire mai la custodia fino a quando non torna a temperatura ambiente. Per la conservazione dei reagenti di biotinilazione, è inoltre essenziale asciugare completamente prima dello stoccaggio. Un processo di essiccazione insufficiente riduce l'efficienza della biotinilazione in proteine a causa dell'idrolisi del reagente di biotinilazione14.
Per quanto riguarda le modifiche e la risoluzione dei problemi, è stato esplorato un metodo per ridurre la variabilità, in particolare per quanto riguarda il metodo di misurazione nel dispositivo di misurazione automatica della reazione. In particolare, quando il volume del tampone nel serbatoio utilizzato per l'analisi è di 150 μL, le bolle entrano nella punta, portando a una riduzione dell'efficienza di reazione e determinando una grande variazione dei valori. Pertanto, si raccomanda di inserire almeno 200 μL di tampone nel serbatoio.
Anche la quantità di liquido che riempie i puntali durante il pipettaggio automatico è un fattore importante. Le punte non possono espellere completamente il liquido mentre si sposta verso la fase di reazione successiva. Pertanto, in quantità non trascurabile, il tampone della fase precedente viene riportato nella fase successiva e il tampone precedente rimane nell'area superiore della soluzione inserita quando il nuovo tampone viene aspirato. Pertanto, il substrato e le altre soluzioni devono essere sufficientemente aspirati per riempire il serbatoio superiore (vedere la Figura 1A). Data l'irreversibilità delle reazioni anticorpali sullo strumento, è fondamentale prestare attenzione ed evitare errori durante la configurazione iniziale.
Una limitazione del metodo è che è limitato alle proteine solubili derivate dal siero e dai surnatanti di coltura. Nel metodo attuale, la lectina non è reticolata in modo covalente alla perla, rendendola incapace di analizzare campioni contenenti alte concentrazioni di tensioattivi, come estratti di cellule e tessuti. Di conseguenza, sono necessari miglioramenti futuri per affrontare queste limitazioni.
Il significato dell'array di perline di lectina risiede nell'intercambiabilità e nell'espandibilità delle specie di lectina fissate sulle perline. Ad esempio, nell'analisi differenziale su larga scala (>1.000 campioni) utilizzando il sistema automatizzato di profilazione dei glicani, gli utenti possono personalizzare una gamma di 15 lectine nella punta in base alla profilazione preliminare dei glicani basata su microarray di lectine della glicoproteina target (<100 campioni). Inoltre, l'elevata stabilità delle perle fissate con lectina consente una sperimentazione immediata, dalla progettazione iniziale della punta alla misurazione con procedure di routine. Non solo le 25 delle 28 lectine che hanno dimostrato un'elevata affidabilità (Tabella 1), ma tutte le lectine di interesse per l'utente possono essere utilizzate per creare un sistema di profilazione dei glicani automatizzato personalizzato per i loro esperimenti specifici, seguendo il suddetto test di affidabilità di routine. Questo approccio ha portato alla progettazione di un possibile array di perline da 120 lectine di otto diversi tipi di punte per una misurazione estesa in combinazione con il sistema standard di profilazione dei glicani.
Uno studio precedente si è concentrato sulla definizione di una procedura di rilevamento per le glicoproteine marcate con Cy3 in linea con il microarray di lectina15. Questo metodo era limitato a 13 lectine in parallelo nella punta a causa dell'interferenza della fluorescenza. Il metodo attuale, che utilizza etichette di rilevamento HRP, ospita 15 perle fissate con lectina e due perle di controllo (positivo e negativo) in una punta. Inoltre, la biotinilazione dei campioni ha abbreviato il processo di analisi.
Questo metodo analitico può essere applicato non solo alla ricerca accademica, ma anche alla ricerca medica e farmaceutica, alimentare, cosmetica e altri campi industriali. Sebbene le tecnologie di microarray di lectine e glicoproteomica siano ben consolidate, l'approccio di questo studio rimane significativo per la sua completa automazione e capacità di analisi rapida con meno passaggi. In futuro, verranno apportati miglioramenti a questa analisi a un metodo quantitativo normalizzando il segnale utilizzando determinate glicoproteine, impiegando perle fissate con anticorpi insieme a perle fissate con lectina in una punta.
Divulgazioni
AO, TO, NT, HS, KK, MA, SY, TM, KN, OS e KS sono dipendenti di Precision System Science Co., Ltd.
Riconoscimenti
Questo lavoro è stato sostenuto dal programma di trasferimento tecnologico adattabile e senza soluzione di continuità attraverso la ricerca e lo sviluppo mirati (A-STEP), finanziato dall'Agenzia giapponese per la scienza e la tecnologia (JST), nell'ambito della sovvenzione numero JPMJTR204A e in parte dalla Japan Society for the Promotion of Science (JSPS) KAKENHI Grant Number 23H02680 ad AK. Ringraziamo Editage (www.editage.jp) per la modifica della lingua inglese.
Materiali
| Name | Company | Catalog Number | Comments |
| 0.2 mL 8 PCR tubes + flat cap | FastGene | FG-0028FC/SE | For dispensing and storage of biotin labeling reagents |
| 2.0 mL SC Micro Tube protein LB | SARSTEDT AG&Co. KG | 72.694.600 | For loading samples in LuBEA-VIII |
| Analysis software | SAS Institute Inc. | JMP 17 | |
| Anti-Static Ionizer. 110 volts | Plas-Labs | 800-AS/SPI | Use while inserting beads into BIST Tip. |
| Antistatic tweezers NK2A | RUBIS | 9-5681-01 | Use while inserting beads into BIST Tip. |
| Automated reaction measurement device | Precision System Science Co., Ltd. | LuBEA | All data in this study were obtained using LuBEA-VIII. While LuBEA is commercialized, LuBEA-VIII is currently in the development phase. The website for product details of LuBEA and LuBEA-VIII can be found at the following URL (https://www.pss.co.jp/english/technology/apit/bist02.html). |
| Biotin-(AC5)2 Sulfo-Osu | Dojindo | 341-06801 | As biotinilation reagent |
| BIST spacer bead- V-11 | Precision System Science Co., Ltd. | F4938000 | Diameter: 1 mm |
| BIST tip and sheath | Precision System Science Co., Ltd. | F4930-000 | Fill this empty tip with beads. |
| Calcium chloride | Wako | 039-00475 | For preparing buffer solutions |
| Can Get Signal Solution 2 | TOYOBO | NKB-301 | This blocking reagent is good for this method |
| CentriVap Benchtop Vacuum Concentrators | Labconco | 7810010 | For dry of biotinilation reagent. The substitution provided by other companies is possible. |
| Clarity Western ECL Substrate | Bio-Rad | 1705060 | For measuring the samples by LuVEA VIII |
| Cold trap CentriVap series | Labconco | 7460040 | For dry of biotinilation reagent. The substitution provided by other companies is possible. |
| Glycine | Wako | 077-00735 | For preparing buffer solutions |
| Manganese(II) Chloride Tetrahydrate | Wako | 139-00722 | For preparing buffer solutions |
| Milli-Q reference | MERCK | ZIQ7000T0C | As deionazed water for dilution of reagents and buffers |
| PBS | Wako | 162-19321 | For preparing buffer solutions |
| Peroxidase Streptavidin | Jackson | 016-030-084 | This antibody is good for GlycoBIST analysis. It is useful for the detection of the western blotting, although substitutions can be provided by other companies. |
| Probe bead | Precision System Science Co., Ltd. | FP4936000 | For preparing lectin-fixed beads. See Shimazaki, H. et al., Current Protocols in Protein Science, 2020 in reference section for detail. |
| Protein LoBind Tubes 1.5 mL | Eppendorf | 0030108442 | Use while preparing the samples such as biotinylation. |
| Silica gel (for desiccan) | Kanto chemical | 37039-02 | Use while storing the GlycoBIST tip and dried biotinylation reagent. |
| Static electricity removal sheet M | TRUSCO NAKAYAMA | SD5050 | Use while inserting beads into BIST Tip. |
| SV Cartridge II | Daido Chemical Industry | ED041 | For measuring the samples by LuVEA VIII |
| Table Microcentrifuge-CAPSULEFUGE | TOMY | PMC-060 | For spin-down of the mixture in each process |
| Triton X-100 | Nacalai tesque | 35501-15 | For preparing buffer solutions |
Riferimenti
- Butler, M., Reichl, U. Animal cell expression systems. Adv Biochem Eng Biotechnol. 175, 1-36 (2021).
- Silva, M. L. S. Chapter One - Lectin biosensors in cancer glycan biomarker detection. Adv Clin Chem. 93, 1-61 (2019).
- Hiono, T., Nagai-Okatani, C., Kuno, A. 4.07 - Application of glycan-related microarrays. Comprehensive Glycoscience (Second Edition). , 134-148 (2021).
- Shimazaki, H., et al. Lectin bead array in a single tip facilitates fully automatic glycoprotein profiling). Anal Chem. 91 (17), 11162-11169 (2019).
- Shimazaki, H., et al. GlycoBIST: A System for automatic glycan profiling of a target protein using milli-bead array in a tip. Curr Protoc Protein Sci. 99 (1), e103(2020).
- Mahmood, T., Yang, P. C. Western blot: Technique, theory, and trouble shooting. N Am J Med Sci. 4 (9), 429-434 (2012).
- Bojar, D., et al. A useful guide to lectin binding: machine-learning directed annotation of 57 unique lectin specificities. ACS Chem Biol. 17 (11), 2993-3012 (2022).
- Nagai-Okatani, C., et al. 2.0: An integrated visualization for lectin microarray-based mouse tissue glycome mapping data with lectin histochemistry. J Proteome Res. 20 (4), 2069-2075 (2021).
- Geisler, C., Jarvis, D. L. Effective glycoanalysis with Maackia amurensis lectins requires a clear understanding of their binding specificities. Glycobiology. 21 (8), 988-993 (2011).
- Noro, E., et al. N -glycan structures of Wisteria floribunda agglutinin-positive Mac2 binding protein in the serum of patients with liver fibrosis. Glycobiology. 31 (10), 1268-1278 (2021).
- Rawitch, A. B., Pollock, H. G., Yang, S. X. Thyroglobulin glycosylation: Location and nature of the N-Linked oligosaccharide units in bovine thyroglobulin. Arch Biochem Biophys. 300 (1), 271-279 (1993).
- Dang, K., Zhang, W., Jiang, S., Lin, X., Qian, A. Application of lectin microarrays for biomarker discovery. Chemistry Open. 9 (3), 285-300 (2020).
- Chen, S., Qin, R., Mahal, L. K. Sweet systems: Technologies for glycomic analysis and their integration into systems biology. Crit Rev Biochem Mol Biol. 56 (3), 301-320 (2021).
- Dwivedi-Agnihotri, H., Srivastava, A., Shukla, A. K. Reversible biotinylation of purified proteins for measuring protein-protein interactions. Methods Enzymol. 633, 281-294 (2020).
- Shimazaki, H., et al. Auto-lectin dotcoding by two octopuses: Rapid analysis of fluorescence-labeled glycoproteins by an 8-channel fully-automatic bead array scanner with a rolling-circle detector. Anal Chem. 95 (32), 11868-11873 (2023).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon