Method Article
Évaluation glyco-qualitative rapide de protéines recombinantes à l’aide d’un système entièrement automatisé
Dans cet article
Résumé
Un système d’analyse automatisée et rapide des glycanes sur les protéines a déjà été développé. Cet article présente le protocole d'« analyse glyco-qualitative », optimisé pour un large éventail d’utilisateurs, tels que ceux engagés dans l’analyse des structures glycanes dans les produits biopharmaceutiques et autres matériaux glycoconjugués.
Résumé
La glycosylation des protéines, une modification post-traductionnelle critique, influence la stabilité, l’efficacité et l’immunogénicité des protéines recombinantes, y compris les produits biopharmaceutiques. Les structures des glycanes présentent une hétérogénéité significative, variant selon les types de cellules de production, les conditions de culture et les méthodes de purification. Par conséquent, la surveillance et l’évaluation des structures glycanes des protéines recombinantes sont vitales, en particulier dans la production biopharmaceutique. La puce à lectine, technique complémentaire à la spectrométrie de masse, est d’une grande sensibilité et d’une grande facilité d’utilisation. Cependant, il faut généralement plus d’une journée pour donner des résultats. Pour l’adapter à la recherche non glycoscientifique ou au développement de procédés de produits pharmaceutiques, une alternative automatisée à haut débit est nécessaire. Par conséquent, le premier système de profilage des glycanes à base de lectine entièrement automatisé au monde a été développé, en utilisant le concept technologique « bead array in a single tip (BIST) ». Ce système permet la préparation et le stockage de billes immobilisées par lectine par unités de 1 000, avec des ordres d’insertion parallèles personnalisables à diverses fins. Cet article présente un protocole pratique pour la recherche impliquant des protéines recombinantes « glyco-qualifiées ». Après avoir testé leur réactivité contre 12 conjugués polyacrylamide-glycane, 15 lectines ont été sélectionnées pour augmenter la polyvalence du système. De plus, le processus d’étiquetage des échantillons a été optimisé en passant de Cy3 à la biotine, ce qui a permis de réduire le temps de traitement global de 30 minutes. Pour une qualification immédiate des données, les signaux de liaison aux lectines sont affichés sous forme de dotcode sur le moniteur supérieur. La fiabilité du système a été confirmée par des tests de reproductibilité au jour le jour, des tests de répétabilité et des tests de stockage à long terme, avec un coefficient de variation de <10 %. Ce glyco-analyseur convivial et rapide a des applications potentielles dans le suivi de la qualité des glycoprotéines endogènes pour l’évaluation et la validation des biomarqueurs. Cette méthode facilite l’analyse pour ceux qui découvrent la glycoscience, élargissant ainsi son utilité pratique.
Introduction
La glycosylation des protéines est une modification post-traductionnelle cruciale qui doit être évaluée dans les produits biopharmaceutiques. Les profils glycanes des protéines peuvent varier en fonction des conditions de culture, des processus de purification et des cellules hôtes1. Des instruments simples sont nécessaires pour qualifier la glycosylation dans le pipeline de bioprocédés. On estime que plus de 50 % des protéines sécrétées et membranaires in vivo sont modifiées par de multiples glycanes, qui changent en fonction de la lignée cellulaire, du stade de développement et de l’état de la maladie, comme l’apparition de la malignité2. La surveillance des profils glycanes présente un potentiel important pour identifier des marqueurs diagnostiques uniques et des cibles médicamenteuses. Les instruments automatisés capables de mesurer rapidement des échantillons à grande échelle sont très demandés pour vérifier et valider une glycosylation aussi aberrante à partir de centaines d’échantillons de patients en cours de découverte.
La technologie des microréseaux a été incorporée à la glycomique pour évaluer le profilage glycane des glycoprotéines3. Dans cette méthode, plusieurs lectines, qui sont des protéines de liaison aux glycanes, sont immobilisées sur une surface telle qu’une lame de verre. Cette technologie de glyco-analyse basée sur l’interaction ne nécessite pas de libérer au préalable des glycanes des protéines centrales, ce qui simplifie le processus pour les chercheurs novices en glycotechnologie. Malgré son utilisation généralisée, pour des applications industrielles telles que la bioproduction, un système automatisé capable de surveiller rapidement et facilement les glycanes pour un plus grand nombre de cibles d’analyse était nécessaire. Pour remédier à ce problème, un système automatisé de profilage des glycanes basé sur un concept unique appelé « réseau de billes en une seule pointe » (BIST), initialement développé pour le génotypage, a déjà été signalé. Ce système simplifie le processus grâce à un auto-instrument à haut débit de type one-box4. À l’aide d’embouts dans lesquels diverses billes fixées à la lectine sont disposées en parallèle 4,5, une méthode d’analyse des structures glycanes modifiées dans les glycoprotéines a été établie et nommée GlycoBIST (ci-après dénommé « système de profilage automatique des glycanes ») (Figure 1A). Les lectines peuvent être fixées sur 1 000 billes et séchées pour maintenir leur activité pendant un an, avant et après l’emballage d’une pointe. Une fois que les pointes et les cartouches contenant des réactifs tels que l’anticorps anti-streptavidine marqué HRP (SA-HRP) sont placées dans le prototype d’instrument de mesure (un appareil de mesure de réaction automatisé, voir Tableau des matériaux), la pointe fonctionne comme une autopipette. Un scanner de détection de chimiluminescence à l’arrière intérieur de l’instrument quantifie les signaux de huit pointes simultanément. Les données quantitatives de ces huit pointes sont affichées de manière compacte et simultanée sous forme de codes points sur l’écran tactile de l’instrument pour une confirmation rapide des résultats de mesure. De plus, la valeur représentée par le maximum du pic mesuré est transportée de l’instrument sous forme de données brutes, et elle permet aux chercheurs individuels de représenter graphiquement (Figure 1A, panneau inférieur).
Dans cet article, les auteurs décrivent une méthode améliorée de marquage des protéines à la biotine, réduisant le temps de traitement à 30 minutes. Les protéines cibles sont biotinylées à l’avance et détectées par SA-HRP (Figure 1B). Une pointe GlycoBIST standard (pointe spécialisée pour le profilage automatique des glycanes) avec 15 lectines sélectionnées a été construite pour obtenir le profilage polyvalent des glycoprotéines glycanes pour une exhaustivité analytique.
Protocole
Les détails des réactifs et de l’équipement utilisés pour cette étude sont énumérés dans la table des matériaux.
1. Préparation de l’embout spécialisé pour le profilage automatique des glycanes
- Apportez les billes séchées fixées à la lectine, stockées à 4 °C dans un sac scellé, sur la paillasse et laissez-les revenir à température ambiante (~23 °C).
REMARQUE : Cela empêche la condensation sur les billes, ce qui peut diminuer leur qualité. Cette méthode de préparation des billes fixatrices de lectine a déjà été signalée5. - Placez un chiffon antistatique sur le bureau et placez l’éliminateur d’électricité statique portable face à l’espace de travail. Cela permet d’effectuer des travaux ultérieurs sur le banc avec protection antistatique (Figure 2A).
- Insérez les billes dans l’extrémité vide à l’aide d’une pince à épiler antistatique de sorte que deux billes d’espacement soient placées entre les billes de fixation des lectines (Figure 1A).
- Pressez doucement le haut de la pointe avec une pince pour fermer l’ouverture, en prenant soin de ne pas couper la pointe une fois que toutes les perles ont été remplies (Figure 2B).
REMARQUE : Il s’agit de fixer les perles à l’intérieur de la pointe. Il est recommandé de fabriquer la pince pour éviter de couper la pointe (figure 2C). - Redressez la pointe à la main si elle est pliée.
- Conservez l’embout préparé à 4 °C dans un sac scellé avec un déshydratant jusqu’à ce que vous en ayez besoin.
REMARQUE : Avant d’utiliser l’embout, il doit être amené à température ambiante avant d’ouvrir le sac scellé et de le retirer. L’humidité réduit drastiquement la réactivité des lectines.
2. Préparation des analytes
- Préemballez le réactif de biotinylation pour l’analyse de routine.
REMARQUE : Pour une analyse de routine, il est utile de pré-emballer le réactif de biotinylation (Biotin-(AC5)2 Sulfo-Osu, voir le tableau des matériaux) en petites portions comme suit pour une analyse rapide. Il est recommandé de sécher et de stocker le réactif de biotinylation, car son ester succinimidylique est généralement sensible à l’hydrolyse.- Dissoudre le réactif de biotinylation dans de l’eau désionisée.
- Versez 10 μg de réactif dans des tubes à l’aide d’une pipette.
- Séchez les tubes avec le réactif de biotinylation dans un concentrateur sous vide réfrigéré et stockez-les avec un dessiccant à 4 °C dans un sac étanche à la lumière jusqu’à utilisation.
- Ajouter la solution d’analyte (200 ng eq. de protéines) dans le tube contenant le réactif de biotinylation séché préparé à l’étape 2.1.
REMARQUE : Soyez conscient de la composition tampon utilisée pour dissoudre et diluer les analytes, en fonction des propriétés du réactif de biotinylation utilisé. Par exemple, évitez d’utiliser des réactifs d’amines primaires comme tampons pour le couplage des amines. Cela permettra d’éviter une réduction de l’efficacité de biotinylation des analytes. - Bien mélanger par vortex, puis tourner (1000 x g pendant 5 s à température ambiante) dans une centrifugeuse de table pour déposer le liquide sur le couvercle au fond du tube.
- Incuber les tubes pendant 1 h à température ambiante, à l’abri de la lumière.
- Diluez les échantillons biotinylés 10 fois dans le tampon de sonde (1 % de Triton X-100, 0,5 M de glycine, 1 mM de CaCl2 et 1 mM de MnCl2 dans le TBS) pour inactiver tous les réactifs de biotinylation n’ayant pas réagi.
REMARQUE : Dans le cas de réactions par couplage d’amines, le réactif de biotinylation n’ayant pas réagi est consommé par la réaction avec un tampon contenant un réactif d’amine primaire tel que le Tris dans le TBS, ce qui empêche la biotinylation des lectines et d’autres substances immobilisées sur les billes lors de la mesure ultérieure. - Incuber à température ambiante, à l’abri de la lumière, pendant 2 h.
- Évaluer l’efficacité de la biotinylation des analytes par transfert Western, en utilisant la streptavidine marquée HRP comme sonde de détection si nécessaire. Veuillez vous référer à la littérature pour les méthodes de transfert Western6.
3. Préparation des réactifs pour le profilage des glycanes avec l’appareil de mesure de réaction automatisé
- Diluer la streptavidine conjuguée à la HRP avec la solution bloquante dans un rapport de 1:3000.
- Mélanger avec des volumes égaux de substrat A et de substrat B (voir tableau des matériaux). Utiliser 160 μL de substrat mélangé par échantillon.
- Versez la solution de blocage, le tampon de lavage (0,1 % de Triton X-100/TBS), le TBS, la solution de streptavidine conjuguée à la HRP et chaque substrat dans les puits désignés d’une cartouche avec 10 réservoirs de liquide.
REMARQUE : Le volume et l’emplacement recommandés des solutions pour remplir chaque puits de la cartouche de réaction sont décrits ci-dessous. Cependant, les solutions et la disposition des puits peuvent être modifiées à l’avance dans l’appareil de mesure de réaction automatisé.- Ajouter 0,2 mL de streptavidine marquée au HRP préparée à l’étape 3.1. au puits #2 à l’aide d’une pipette. Le puits est nommé #1 à l’avant de la cartouche.
- Ajouter 0,2 mL de solution de blocage dans le puits #3 à l’aide d’une pipette.
- Ajouter 1 mL de TBS dans les puits #4 et #6 à l’aide d’une pipette.
- Ajouter 1 mL de tampon de lavage dans les puits #7 et #8 à l’aide d’une pipette.
- Ajouter 0,16 mL du mélange de substrat dans le puits #10 à l’aide d’une pipette.
- Vérifiez que les puits #1, #5 et #9 sont vides.
- Ajouter les analytes préparés à l’étape 2 dans un microtube à bouchon à vis à faible adsorbant de 2 mL.
- Ouvrez la porte avant de l’appareil de mesure de réaction automatisé.
- Placer les réservoirs de liquide préparés à l’étape 3.3, les pointes préparées à l’étape 1 et les analytes préparés à l’étape 3.4 aux positions désignées de l’appareil de mesure automatique de la réaction (figure 2D) comme suit. Chaque mesure est effectuée dans une seule direction de ligne verticale.
- Placez une pointe préparée à l’étape 1 dans chaque trou à la position « a » symbole jaune sur la figure 2D.
REMARQUE : Encore une fois, s’il est réfrigéré, amenez les pointes à température ambiante avant d’ouvrir le sac scellé. Les lectines perdent considérablement leur signal à cause de l’humidité. - Retirez le couvercle du tube contenant l’analyte préparé à l’étape 3.4 et insérez-le dans chaque trou à la position « b » de la même manière.
- Insérez la cartouche contenant le liquide préparé à l’étape 3.3, en faisant attention de ne pas le renverser en position « c ».
- Retirez toute la « boîte à déchets » de la figure 2D lorsqu’elle est pleine, videz le contenu dans la poubelle et remettez la « boîte à déchets » dans sa position d’origine.
REMARQUE : Les pointes ayant réagi sont automatiquement collectées dans la « boîte à déchets » après la mesure.
- Placez une pointe préparée à l’étape 1 dans chaque trou à la position « a » symbole jaune sur la figure 2D.
4. Profilage des glycanes avec l’appareil de mesure de réaction automatisé
- Allumez l’interrupteur d’alimentation sur le côté de l’unité principale.
- Appuyez sur le bouton « Démarrer le diagnostic » affiché à l’écran (Figure 3A). Le programme d’autodiagnostic démarre automatiquement. Au bout d’un certain temps, le programme d’autodiagnostic est terminé et l’écran HOME s’affiche (Figure 3B).
REMARQUE : Les opérations suivantes peuvent être effectuées en touchant les icônes respectives. « Assay » pour commencer la mesure. « Maintenance » pour confirmer toute erreur du système. « Historique » pour afficher les données de mesure passées. - Appuyez sur « Test » affiché sur l’écran d’accueil pour sélectionner le protocole (Figure 3B).
- Sélectionnez le « BAssaySTD » de la méthode de dosage (Figure 3C), puis appuyez sur « Suivant » en bas à droite.
- Entrez le nom de l’échantillon (Figure 3D) pour chaque échantillon si nécessaire, puis appuyez sur « Suivant » en bas à droite.
- Entrez le nom de l’embout (Figure 3E) utilisé pour chaque échantillon si nécessaire, puis appuyez sur « Suivant » en bas à droite.
- Confirmez les notes à droite (Figure 3F) et, s’il n’y a pas de problème, appuyez sur « Start » en bas à droite pour la mesure.
- Confirmez le passage de l’écran en mode de fonctionnement (Figure 3G) et attendez que la flèche « finaliser » soit mise en surbrillance. Si nécessaire, appuyez sur « Pause » pour arrêter le mode de fonctionnement.
REMARQUE : Les opérations contrôlées automatiquement par le dispositif de mesure de réaction automatisé sont les suivantes : (1) Les buses auto-micropipettes à l’intérieur de l’instrument fixent un maximum de huit pointes simultanément (voir le symbole jaune « a » sur la figure 2D) ; (2) Aspirer/décharger à plusieurs reprises la solution dans le tube contenant l’analyte pendant 5 min ; (3) Transférer dans la solution de blocage et aspirer/décharger pendant 5 min ; (4) Transfert au TBS et aspiration/décharge pendant 2 min ; (5) Transfert dans la solution de streptavidine marquée HRP et aspiration/décharge 5 min ; (6) Transférer dans le tampon de lavage et aspirer/décharger pendant 7 min ; (7) Transfert au TBS et aspiration/décharge pendant 2 min ; (8) Transférer dans le mélange de substrat et aspirer immédiatement ; (9) Scanner la chimiluminescence de huit pointes simultanément à l’arrière de l’appareil pour obtenir des données de pic ; (10) Déposez les pointes usagées dans la boîte à déchets ; (11) La buse revient à la position initiale. - Vérifiez que l’écran est passé en mode résultat (Figure 3H) une fois la mesure terminée. Les huit dotcodes sont affichés simultanément à l’écran pour une vérification facile.
REMARQUE : Appuyez sur « Historique » en bas à gauche pour afficher le graphique de numérisation (Figure 3I, J), le graphique à barres (Figure 3K, L), le tableau (Figure 3M) et les détails (Figure 3N) comme résultat à l’écran. Affichez les résultats des autres voies à partir de la liste déroulante « Lane ». - Insérez une clé USB dans le port USB sur le côté de l’unité principale et appuyez sur « Exporter » en bas à droite du mode de résultat pour enregistrer les données de toutes les voies individuellement dans la mémoire USB.
- Exportez les données brutes (Figure 3M) sous forme de fichier CSV à l’aide d’un port USB pour une analyse plus approfondie des données et la création de graphiques individuels dans Excel. La « valeur » du tableau indique l’intensité du signal de chaque lectine. Les valeurs d’intensité représentent la valeur maximale des données de crête (Figure 3I,J) dans le balayage des pointes réagies.
REMARQUE : Le seuil d’extraction de crête peut être défini. Selon les analytes, le signal dans la partie de la perle d’espacement peut être globalement plus élevé, auquel cas la valeur de la partie d’espacement est soustraite en tant qu’arrière-plan.
Résultats
Tout d’abord, la distribution du signal est montrée pour chaque lectine afin de comprendre les caractéristiques du réseau de billes de lectine. Une bibliothèque de billes fixées par des lectines (1 000 perles chacune) a été préparée. Quinze perles ont été choisies au hasard parmi les 1 000 et ajoutées à une seule pointe. Un graphique typique est présenté au bas de la figure 1A, représentant presque la même intensité de signal observée parmi les 15 billes immobilisant la même lectine. Cette procédure de mesure peut être appliquée pour qualifier la production artisanale de billes de lectine avant l’utilisation du profilage des glycanes. Par exemple, six pointes sont utilisées pour la mesure simultanée afin de calculer le coefficient de variation (CV) à partir des signaux sur les 90 billes. Le tableau 1 en donne un exemple typique. Lorsque la bibliothèque de billes fixées aux lectines a été élargie pour accueillir 28 lectines différentes, 25 des 28 lectines ont démontré une reproductibilité élevée avec un CV inférieur à 10 %.
Ensuite, les données d’un réseau de billes de lectines avec 15 lectines différentes sont présentées. Les 15 lectines présentées dans le tableau 2 ont été sélectionnées pour le tableau des billes de lectines. De plus, 12 mono/di/trisaccharides de polyacrylamide (PAA) énumérés dans le tableau 3 ont été mesurés. Après avoir évalué la fiabilité de la mesure à l’aide d’un affichage en dotcoding à l’écran, les données brutes ont été exportées à partir de l’appareil de mesure de réaction automatisé (voir l’étape 4.11 du protocole), et un graphique a été créé dans Excel à l’aide des valeurs des pics de réaction à chaque saccharide (figure 4A). Ces valeurs ont ensuite été utilisées pour l’analyse en composantes principales afin de visualiser la corrélation entre les 15 lectines sélectionnées et les saccharides ayant réagi (Figure 4B,C). Cela a entraîné une différence nette dans le modèle de liaison de chaque lectine par rapport aux 12 sucres PAA illustrés à la figure 4A. Ici, la validité de chaque signal est démontrée par rapport aux listes précédentes intégrées de spécificités de lectine et de saccharide 7,8 (voir également ces spécificités dans le tableau 2). Comme AAL se lie au α-Fuc terminal, au Sia-Lex et au Lex, l’analyse automatique des glycanes a confirmé la reconnaissance par AAL des molécules contenant du fucose (Figure 4A). SSA et MAL (voir le tableau 2 pour les noms complets des lectines et les abréviations données) ont reconnu Siaα2-6Gal/GalNAc et Siaα2-3Gal, respectivement. Les MAL dans le réseau de billes peuvent également se lier à 3'-O-sulfo-Galβ3GalNAc et LacNAc. Ceci est cohérent avec les résultats précédents analysés à l’aide de l’apprentissage automatique7, dans lesquels MAL préfère le 3'-O-sulfo-Gal de manière dominante, tandis que LacNAc n’est pas dominant9. RCA120, une lectine reconnaissant le Gal, a fortement réagi avec le β-Gal terminal, y compris Lac/LacNAc, et a été autorisé à se lier à Siaα2-6Lac mais pas à Siaα2-3Lac. La réactivité de la CEA avec LacNAc était beaucoup plus élevée que celle de la Lac. Le WFA avait une spécificité plus large, qui reconnaissait non seulement le α/β-GalNAc, mais aussi le β-Gal terminal, comme indiqué 7,10. GSL II, qui reconnaît la GlcNAc et un N-glycanes tri/tétra-antennaire galactosylé, ne s’est lié à aucun saccharide, à l’exception d’un seul GlcNAc parmi les analytes préparés. Le GRFT, qui reconnaît les N-glycanes de type mannose à haute teneur en mangas, a réagi avec le α-Man. L’ABA, le Jacalin et le MAH ont généralement été utilisés pour détecter les O-glycanes. Comme l’ABA préfère les structures Galβ1-3GalNAcα-Thr/Ser (T) et sialyl-T, l’ABA dans le réseau de billes a fortement réagi avec 3'-O-sulfo-Galβ1-3GalNAc et s’est faiblement liée à α-GalNAc. L’ABA a également reconnu β-GlcNAc, conformément aux rapports précédents sur la liaison aux N-glycanes 7 agalactosylés. Jacalin a une spécificité relativement plus large et a donc réagi avec le α-mannose, le β-GalNAc, le βGal, le α-GalNAc, le 3'-O-sulfo-Galβ1-3GalNAc. Le MAH a spécifiquement reconnu le 3'-O-sulfate-Galβ1,3GalNAc comme indiqué précédemment7. Les quatre lectines résiduelles, NPA, LCA, PHA-L et PHA-E, reconnaissaient la structure interne du N-glycane non inclus dans les saccharides PAA préparés et n’avaient donc aucune affinité avec tous les analytes.
À l’aide des données obtenues, la similitude des lectines contribuant à la liaison à chaque saccharide peut être clarifiée en les traçant sur un graphique d’analyse en composantes principales (Figure 4B). Tous les saccharides qui ont contribué à la variance de la figure 4B ont été représentés par un vecteur propre, une méthode d’analyse statistique dans laquelle le rapport de contribution est représenté par un vecteur (figure 4C). Il est à noter ici que les saccharides réactifs contribuent à chaque lectine sans biais. Les lectines ne présentent pas de spécificité pour un seul type de saccharide. Par exemple, la présence de glycanes de type complexe est déterminée par la reconnaissance partielle de certains glycanes au sein de glycanes de type complexe chez l’homme. Jacalin, qui reconnaît les O-glycanes (voir les spécificités dans le tableau 2), peut reconnaître la structure 3'suTF et α-GalNAc parmi les saccharides utilisés dans cette étude, et donc, le 3'suTF et le α-GalNAc ont été tracés (Figure 4C) en fonction de la direction de position de Jacalin (point brun) tracé dans la Figure 4B. WFA peut reconnaître α/β-GalNAc parmi les saccharides utilisés dans cette étude ; par conséquent, α/β-GalNAc de la figure 4C a été tracé dans la même direction que la WFA tracée de la figure 4B. En revanche, α-GalNAc est tracé entre WFA et Jacalin parce qu’il s’agit d’une structure saccharide à laquelle Jacalin est capable de se lier facilement. Les 11 saccharides représentés dans cette étude sont cohérents avec la spécificité (liaison aux saccharides) des lectines signalée précédemment 7,8, ce qui indique que la méthode proposée est une méthode de mesure fiable. De plus, les vecteurs propres correspondant à chaque saccharide sont dispersés, ce qui confirme que les 15 lectines ont été sélectionnées avec un biais minimal pour assurer une couverture complète de l’analyse. Par conséquent, l’embout réapprovisionné avec ces 15 lectines a été défini comme l’embout GlycoBIST standard (tableau 2).
Dans l’exemple le plus pratique, les produits protéiques purifiés ont été soumis à la mesure de la pointe (Figure 5). La thyroglobuline bovine, qui a un site de N-glycosylation et contient des N-glycanes11 complexes, hybrides et de type mannose élevé, a été analysée. L’embout standard indiquait une réactivité accrue de certaines lectines aux saccharides8 (voir les spécificités dans le tableau 2) : PHA-E, qui reconnaît la GlcNAc coupante en deux ; GRFT, reconnaissant les N-glycanes de type mannose ; SSA, reconnaissant Siaα2-6Gal/GalNAc ; RCA120, reconnaissant le lactose et le LacNAc ; et AAL, reconnaissant fucose. Ces structures glycanes sont présentes dans les N-glycanes complexes, hybrides et à haute teneur en mannose, ce qui suggère que la structure N-glycane de la thyroglobuline11 pourrait être évaluée efficacement. De plus, la thyroglobuline traitée avec la sialidase A, une enzyme qui digère l’acide sialique, a montré une réactivité réduite avec l’acide sialique, et une réactivité accrue avec RCA120, WFA et ECA, qui sont plus facilement reconnues après l’élimination des acides sialiques. La présence de structures O-glycanes dans la thyroglobuline bovine n’a pas été signalée auparavant. Comme prévu, il n’y a pas eu de reconnaissance de l’O-glycane dans la thyroglobuline avec ou sans traitement par sialidase dans l’analyse automatique des glycanes.
Un test de reproductibilité en cours d’exécution utilisant les 15 lectines sélectionnées permet de comprendre la robustesse de la mesure (tableau 4). Pour ce test, sept pointes standard pour les analytes et une pointe supplémentaire pour le contrôle négatif ont été préparées, et les huit pointes ont été mesurées simultanément. Cette procédure a été répétée trois fois en une seule journée. Des « analytes mixtes », une combinaison d’analytes qui a permis d’obtenir des signaux significatifs sur toutes les lectines, ont été préparés pour le contrôle de la qualité du réseau de billes. Les analytes mélangés ont été formulés en mélangeant de manière appropriée de l’érythropoïétine (EPO) digérée par la sialidase, de l’EPO digérée par la sialidase et la galactosidase, des IgA humaines, de la métalloprotéinase matricielle 3 (MMP3) et de la thyroglobuline. Chaque glycoprotéine (200 ng) a été marquée avec 10 μg de réactif de biotinylation dans du PBS contenant du Triton X-100. Si nécessaire, des traitements à la sialidase et à la galactosidase ont été effectués comme décrit dans les instructions. Après la digestion, les produits ont été incubés dans un bloc chauffant à 75 °C pendant 10 min pour inactiver l’enzyme. Les quantités appropriées de glycoprotéines biotinylées (3 ng d’EPO, 8 ng d’IgA humaines, 5 ng de MMP3 et 15 ng de thyroglobuline) ont été mélangées immédiatement avant l’analyse. Les résultats de mesure ont indiqué que la valeur CV maximale était de 13,5 % et que le CV moyen pour les 15 lectines était de 8,2 %, démontrant une reproductibilité élevée.
De plus, il est démontré qu’un test de reproductibilité au jour le jour permet de comprendre les variations dues à la date de mesure (tableau 5). Les pointes standard et les analytes mélangés ont été préparées à l’avance, et les mesures de sept pointes ont été répétées quotidiennement pendant cinq jours consécutifs. Les résultats ont montré que la plupart des lectines avaient un CV inférieur à 10%. Cependant, certaines lectines, telles que l’ACV et l’ECA, présentaient des CV plus élevés. Le CV moyen des 15 lectines par cycle était de 7,7 %, et les valeurs moyennes du CV sur 5 jours étaient inférieures à 10 %, ce qui signifie une reproductibilité élevée. Il a été noté que Jacalin, en raison de l’autodigestion de MMP3 dans l’analyte mixte, a démontré une faible reproductibilité temporelle. Par conséquent, un analyte plus approprié devrait être identifié pour remplacer le MMP3.
Pour comprendre la stabilité des billes fixées aux lectines, un test de stabilité à long terme est présenté à l’aide de ces billes séchées fixées aux lectines (tableau 6). Les pointes standard, les réactifs de dosage et les analytes mixtes ont été préparés à l’avance, et les mesures ont été effectuées après 12 mois de stockage. Les cinq types d’analytes biotinylés susmentionnés ont été stockés individuellement et mélangés avant la mesure. Les résultats ont montré que le CV moyen pour les 15 lectines était inférieur à 10%, même jusqu’à 12 mois après le stockage à sec. Cela suggère la possibilité de mesures rapides et précises en stockant de nombreuses pointes (jusqu’à 1 000) du même lot à la fois.
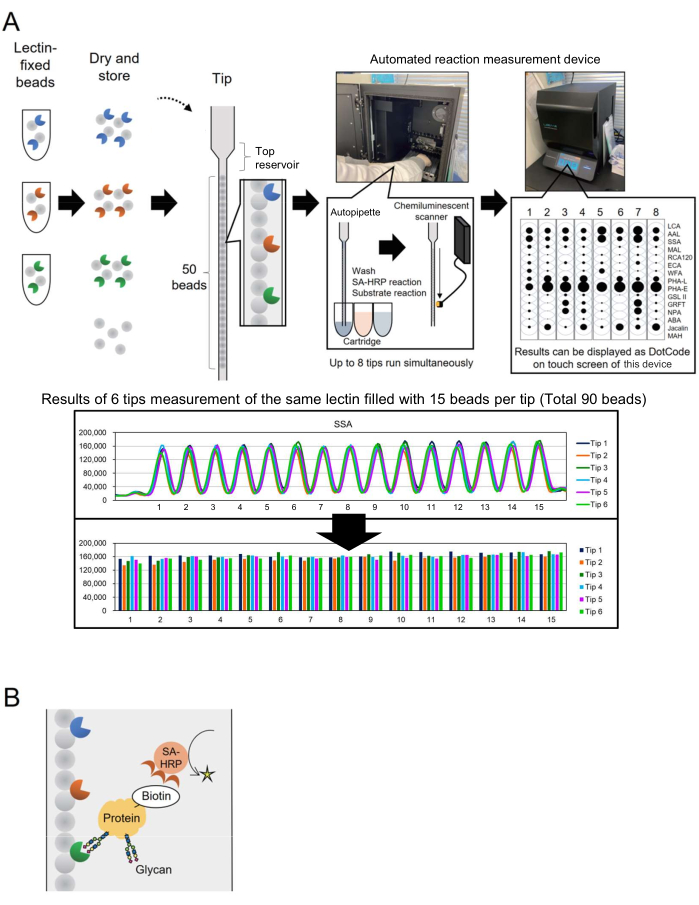
Figure 1 : Schémas de l’instrument et de la méthode d’analyse « réseau de billes dans une seule pointe ». (A) Schéma du système de profilage automatique des glycanes. Les billes fixées à la lectine peuvent être séchées et stockées dans des pointes. Lorsque des pointes et des cartouches contenant des réactifs tels que l’anticorps anti-streptavidine marqué HRP (SA-HRP) sont placées dans l’instrument de mesure (appareil de mesure de réaction automatisé) et activées, la pointe fonctionne comme une autopipette. Le scanner de détection de chimiluminescence situé à l’arrière de l’instrument quantifie simultanément les signaux provenant de huit pointes. Les données quantitatives sont affichées sous forme de codes points sur l’écran tactile de l’instrument pour une confirmation rapide des résultats de mesure. La partie inférieure montre les résultats de la mesure de 90 billes avec la même lectine. Les résultats de mesure sont transportés à partir de l’instrument et représentés graphiquement par le chercheur individuel. (B) Schéma de la méthode de détection utilisée dans cette expérience. Les protéines cibles sont pré-biotinylées et détectées à l’aide de SA-HRP. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 2 : Images des matériaux et configuration pour le profilage automatique des glycanes avec le dispositif de mesure de réaction automatisé. (A) Représentation de billes fixatrices de lectines emballées dans une pointe. Ramasser les billes avec une pince à épiler antistatique sur le tapis antistatique et remplir la pointe avec les billes. (B) Pincer la pointe avec une pince. Une légère partie supérieure de la perle supérieure est comprimée. (C) Une pince pour sertir les pointes. Le plastique (cercle jaune) est pincé pour éviter qu’ils ne se coupent complètement. (D) Disposition des matériaux utilisés dans le dispositif automatisé de mesure de la réaction. A : Embouts, B : Tube contenant des analytes, et C : Des cartouches contenant du liquide doivent être insérées en place, respectivement. Dans la boîte à déchets, les pointes ayant réagi sont collectées après la mesure. La boîte peut être retirée. Veuillez cliquer ici pour voir une version agrandie de cette figure.
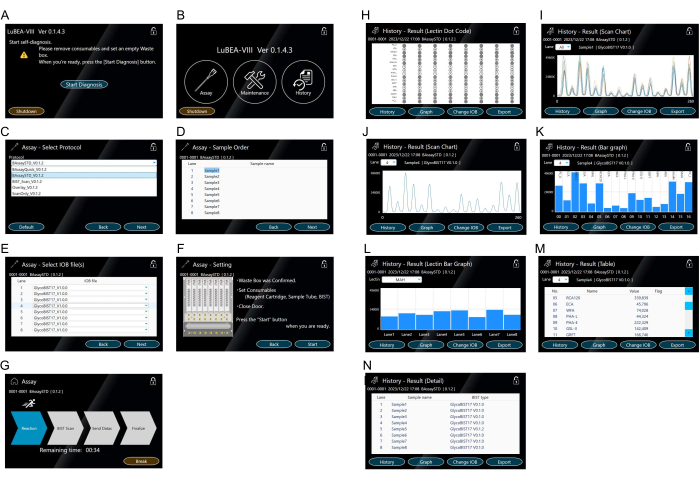
Figure 3 : Écran tactile pour la manipulation de la machine. (A) Le programme d’autodiagnostic démarre automatiquement lorsque l’appareil est sous tension. (B) L’écran d’accueil affiche « Test », « Maintenance » et « Historique ». (C-G) Fonctionnement du mode « Assay ». Sélection de la méthode de dosage (C). Plusieurs protocoles peuvent être enregistrés à l’avance. Entrée de l’ordre de l’échantillon (analyte) (D). Sélection du type de pointe (E). Confirmation des paramètres de la méthode d’essai (F) et démarrage de l’essai à partir de l’écran « Démarrer l’essai » (G). (H-N) Représentation du résultat dans « l’Histoire ». Le dotcode de lectine (H) s’affiche après la fin du test ou à partir du mode « Historique ». Un tableau de balayage de 15 lectines s’affiche à l’écran pour tous les échantillons (I) ou pour chaque échantillon (J). Le graphique de balayage s’affiche sous la forme d’un graphique à barres (K). Les graphiques à barres pour d’autres voies peuvent être sélectionnés dans la liste déroulante « Voies ». Graphique à barres comparant huit voies d’échantillons par lectine (L). D’autres lectines peuvent être sélectionnées dans la liste déroulante « Lectines ». Tableau des valeurs quantitatives (M). Enregistrement de la méthode de mesure exécutée (N). Veuillez cliquer ici pour voir une version agrandie de cette figure.
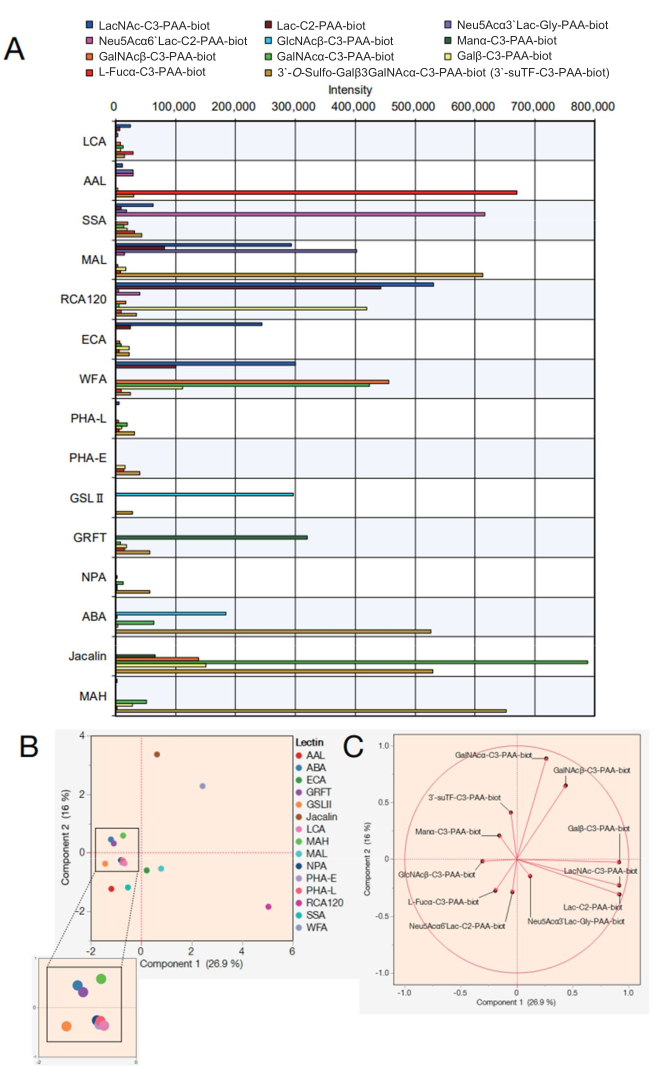
Figure 4 : Évaluation de la réactivité avec les saccharides PAA dans le système de profilage automatique des glycanes. (A) Un graphique illustrant la réactivité des lectines avec divers saccharides PAA a été ajusté pour les valeurs de contrôle négatives. (B) Analyse en composantes principales basée sur les données de (A). (C) Vecteurs propres de l’analyse en (B). Un logiciel d’analyse compatible a été utilisé pour les pièces (B) et (C). Veuillez cliquer ici pour voir une version agrandie de cette figure.
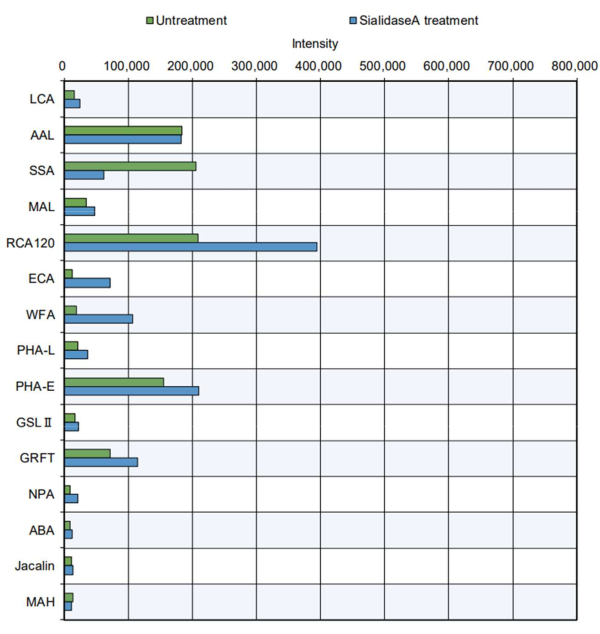
Figure 5 : Vérification de la réactivité de GlycoBIST à l’aide de protéines de thyroglobuline bovine purifiées. La préparation de l’échantillon sans traitement a été effectuée dans les mêmes conditions de digestion que le tampon seul, à l’exclusion de l’ajout de sialidase A. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Tableau 1 : Test de répétabilité avec des billes fixées à la lectine. La répétabilité de la réactivité pour 28 lectines a été évaluée. Les analytes ont réagi à chaque pointe. EPO : érythropoïétine, PSA : antigène spécifique de la prostate, Tf : transferrine, M2BP : protéine de liaison Mac-2, hIgG : IgG humaines, MMP3 : métalloprotéinase matricielle 3, (Sia+) : analytes digérés par la sialidase A, (Sia+, Gal+) : analytes digérés par la sialidase A et la galactosidase, Ab : anticorps. DSA : Datura stramonium agglutininin, HypninA2 : Hypnea japonica agglutinine, WGA : agglutinine de germe de blé, UDA : Urtica dioica agglutinine, BPL : Bauhinia purpurea lectine, Orysata : Oryza sativa lectine, LSL-N : Laetiporus sulphureus lectine N-terminale, SNA : Sambucus nigra lectine, BanTec : lectine de banane, MPA : Maclura pomifera agglutinine, TxLC-I : Tulipa gesneriana agglutinine, AOL : Aspergillus oryzae lectine, ACG : Agrocybe cylindracea galectine. CV : coefficient de variation. Veuillez cliquer ici pour télécharger ce tableau.
Tableau 2 : Liste des lectines constituant l’embout standard pour le profilage automatique des glycanes. Veuillez cliquer ici pour télécharger ce tableau.
Tableau 3 : Saccharides PAA utilisés comme étalons pour le système de profilage automatique des glycanes. Veuillez cliquer ici pour télécharger ce tableau.
Tableau 4 : Résultats de l’essai de reproductibilité à l’intérieur de la course. CV : coefficient de variation. Veuillez cliquer ici pour télécharger ce tableau.
Tableau 5 : Résultats de l’essai de reproductibilité entre les jours. CV : coefficient de variation. Veuillez cliquer ici pour télécharger ce tableau.
Tableau 6 : Résultats de l’essai de stabilité à long terme. CV : coefficient de variation. Veuillez cliquer ici pour télécharger ce tableau.
Discussion
Dans cette étude, une technique d’évaluation rapide de la glycosylation utilisant la technologie « bead array in a single tip » a été développée. La présente étude a introduit un embout GlycoBIST standard, conçu pour les chercheurs en glycoscience et non en glycoscience, afin de faciliter l’évaluation complète et régulière de la glycosylation. La puce à lectines, qui emploie généralement de 20 à 100 lectines12,13, a été largement utilisée dans les évaluations. Cependant, compte tenu du chevauchement des spécificités de certaines lectines sur la puce et de la variété relativement limitée de glycoformes sur une glycoprotéine cible par rapport à la glycomique d’échantillons cliniques bruts, il était prévu que 15 lectines suffiraient pour une évaluation simplifiée du profilage des glycanes ciblés.
L’étape critique du protocole consiste à permettre aux billes immobilisées par la lectine ou aux pointes remplies de ces billes de revenir à température ambiante après le stockage avant de les utiliser pour les mesures. En particulier, il a été observé que la condensation affaiblit les signaux ; Par conséquent, il est conseillé de ne jamais ouvrir le sac de rangement tant qu’il n’est pas revenu à température ambiante. Pour le stockage des réactifs de biotinylation, il est également essentiel de sécher complètement avant de le stocker. Un processus de séchage insuffisant diminue l’efficacité de la biotinylation en protéines en raison de l’hydrolyse du réactif de biotinylation14.
En ce qui concerne les modifications et le dépannage, une méthode a été explorée pour réduire la variabilité, en particulier en ce qui concerne la méthode de mesure dans le dispositif de mesure de réaction automatisé. En particulier, lorsque le volume de tampon dans le réservoir utilisé pour l’analyse est de 150 μL, des bulles pénètrent dans la pointe, entraînant une réduction de l’efficacité de réaction et entraînant une grande variation des valeurs. Par conséquent, il est recommandé de placer au moins 200 μL de tampon dans le réservoir.
La quantité de liquide qui remplit les pointes lors du pipetage automatique est également un facteur important. Les pointes ne peuvent pas expulser complètement le liquide lorsqu’il passe à l’étape de réaction suivante. Par conséquent, en grande quantité, la mémoire tampon de l’étape précédente est transférée à l’étape suivante, et la mémoire tampon précédente reste dans la partie supérieure de la solution insérée lorsque la nouvelle mémoire tampon est aspirée. Par conséquent, le substrat et les autres solutions doivent être suffisamment aspirés pour remplir le réservoir supérieur (voir figure 1A). Étant donné l’irréversibilité des réactions d’anticorps sur l’instrument, il est crucial de faire preuve de prudence et d’éviter les erreurs lors de la configuration initiale.
L’une des limites de la méthode est qu’elle est limitée aux protéines solubles dérivées de surnageants sériques et de culture. Dans la méthode actuelle, la lectine n’est pas réticulée de manière covalente à la bille, ce qui la rend incapable d’analyser des échantillons contenant de fortes concentrations de tensioactifs, tels que des extraits de cellules et de tissus. Par conséquent, des améliorations futures sont nécessaires pour remédier à ces limites.
L’importance de la matrice de perles de lectine réside dans l’interchangeabilité et l’expansibilité des espèces de lectines fixées sur les billes. Par exemple, dans le cadre d’une analyse différentielle à grande échelle (>1 000 échantillons) à l’aide du système automatisé de profilage des glycanes, les utilisateurs peuvent personnaliser une gamme de 15 lectines dans la pointe en fonction d’un profilage préliminaire des glycanes de la glycoprotéine cible basé sur une puce à lectines (<100 échantillons). De plus, la grande stabilité des billes fixées aux lectines permet une expérimentation immédiate, de la conception initiale de la pointe à la mesure avec des procédures de routine. Non seulement les 25 des 28 lectines qui ont démontré une grande fiabilité (tableau 1), mais toutes les lectines d’intérêt pour l’utilisateur peuvent être utilisées pour créer un système automatisé de profilage des glycanes personnalisé pour leurs expériences spécifiques, à la suite du test de fiabilité de routine susmentionné. Cette approche a conduit à la conception d’un réseau possible de 120 billes de lectine de huit types différents d’embouts pour une mesure étendue en conjonction avec le système standard de profilage des glycanes.
Une étude précédente s’est concentrée sur l’établissement d’une procédure de détection des glycoprotéines marquées par Cy3 en ligne avec la puce à lectine15. Cette méthode a été limitée à 13 lectines en parallèle dans la pointe en raison de l’interférence de fluorescence. La méthode actuelle, qui utilise des étiquettes de détection HRP, intègre 15 billes fixées à la lectine et deux billes de contrôle (positive et négative) dans une pointe. De plus, la biotinylation des échantillons a raccourci le processus d’analyse.
Cette méthode analytique peut être appliquée non seulement à la recherche universitaire, mais aussi à la recherche médicale et pharmaceutique, à l’alimentation, aux cosmétiques et à d’autres domaines industriels. Bien que les technologies de microréseaux de lectines et de glycoprotéomique soient bien établies, l’approche de cette étude reste importante en raison de son automatisation complète et de sa capacité d’analyse rapide avec moins d’étapes. À l’avenir, des améliorations seront apportées à cette analyse à une méthode quantitative en normalisant le signal à l’aide de certaines glycoprotéines, en utilisant des billes fixées par des anticorps et des billes fixées par la lectine dans une pointe.
Déclarations de divulgation
AO, TO, NT, HS, KK, MA, SY, TM, KN, OS et KS sont des employés de Precision System Science Co., Ltd.
Remerciements
Ce travail a été soutenu par le Programme de transfert de technologie adaptable et sans couture par le biais de la R&D axée sur les objectifs (A-STEP), financé par l’Agence japonaise pour la science et la technologie (JST), dans le cadre de la subvention numéro JPMJTR204A et en partie par la Société japonaise pour la promotion de la science (JSPS) KAKENHI numéro de subvention 23H02680 à AK. Nous tenons à remercier Editage (www.editage.jp) pour l’édition de la langue anglaise.
matériels
| Name | Company | Catalog Number | Comments |
| 0.2 mL 8 PCR tubes + flat cap | FastGene | FG-0028FC/SE | For dispensing and storage of biotin labeling reagents |
| 2.0 mL SC Micro Tube protein LB | SARSTEDT AG&Co. KG | 72.694.600 | For loading samples in LuBEA-VIII |
| Analysis software | SAS Institute Inc. | JMP 17 | |
| Anti-Static Ionizer. 110 volts | Plas-Labs | 800-AS/SPI | Use while inserting beads into BIST Tip. |
| Antistatic tweezers NK2A | RUBIS | 9-5681-01 | Use while inserting beads into BIST Tip. |
| Automated reaction measurement device | Precision System Science Co., Ltd. | LuBEA | All data in this study were obtained using LuBEA-VIII. While LuBEA is commercialized, LuBEA-VIII is currently in the development phase. The website for product details of LuBEA and LuBEA-VIII can be found at the following URL (https://www.pss.co.jp/english/technology/apit/bist02.html). |
| Biotin-(AC5)2 Sulfo-Osu | Dojindo | 341-06801 | As biotinilation reagent |
| BIST spacer bead- V-11 | Precision System Science Co., Ltd. | F4938000 | Diameter: 1 mm |
| BIST tip and sheath | Precision System Science Co., Ltd. | F4930-000 | Fill this empty tip with beads. |
| Calcium chloride | Wako | 039-00475 | For preparing buffer solutions |
| Can Get Signal Solution 2 | TOYOBO | NKB-301 | This blocking reagent is good for this method |
| CentriVap Benchtop Vacuum Concentrators | Labconco | 7810010 | For dry of biotinilation reagent. The substitution provided by other companies is possible. |
| Clarity Western ECL Substrate | Bio-Rad | 1705060 | For measuring the samples by LuVEA VIII |
| Cold trap CentriVap series | Labconco | 7460040 | For dry of biotinilation reagent. The substitution provided by other companies is possible. |
| Glycine | Wako | 077-00735 | For preparing buffer solutions |
| Manganese(II) Chloride Tetrahydrate | Wako | 139-00722 | For preparing buffer solutions |
| Milli-Q reference | MERCK | ZIQ7000T0C | As deionazed water for dilution of reagents and buffers |
| PBS | Wako | 162-19321 | For preparing buffer solutions |
| Peroxidase Streptavidin | Jackson | 016-030-084 | This antibody is good for GlycoBIST analysis. It is useful for the detection of the western blotting, although substitutions can be provided by other companies. |
| Probe bead | Precision System Science Co., Ltd. | FP4936000 | For preparing lectin-fixed beads. See Shimazaki, H. et al., Current Protocols in Protein Science, 2020 in reference section for detail. |
| Protein LoBind Tubes 1.5 mL | Eppendorf | 0030108442 | Use while preparing the samples such as biotinylation. |
| Silica gel (for desiccan) | Kanto chemical | 37039-02 | Use while storing the GlycoBIST tip and dried biotinylation reagent. |
| Static electricity removal sheet M | TRUSCO NAKAYAMA | SD5050 | Use while inserting beads into BIST Tip. |
| SV Cartridge II | Daido Chemical Industry | ED041 | For measuring the samples by LuVEA VIII |
| Table Microcentrifuge-CAPSULEFUGE | TOMY | PMC-060 | For spin-down of the mixture in each process |
| Triton X-100 | Nacalai tesque | 35501-15 | For preparing buffer solutions |
Références
- Butler, M., Reichl, U. Animal cell expression systems. Adv Biochem Eng Biotechnol. 175, 1-36 (2021).
- Silva, M. L. S. Chapter One - Lectin biosensors in cancer glycan biomarker detection. Adv Clin Chem. 93, 1-61 (2019).
- Hiono, T., Nagai-Okatani, C., Kuno, A. 4.07 - Application of glycan-related microarrays. Comprehensive Glycoscience (Second Edition). , 134-148 (2021).
- Shimazaki, H., et al. Lectin bead array in a single tip facilitates fully automatic glycoprotein profiling). Anal Chem. 91 (17), 11162-11169 (2019).
- Shimazaki, H., et al. GlycoBIST: A System for automatic glycan profiling of a target protein using milli-bead array in a tip. Curr Protoc Protein Sci. 99 (1), e103(2020).
- Mahmood, T., Yang, P. C. Western blot: Technique, theory, and trouble shooting. N Am J Med Sci. 4 (9), 429-434 (2012).
- Bojar, D., et al. A useful guide to lectin binding: machine-learning directed annotation of 57 unique lectin specificities. ACS Chem Biol. 17 (11), 2993-3012 (2022).
- Nagai-Okatani, C., et al. 2.0: An integrated visualization for lectin microarray-based mouse tissue glycome mapping data with lectin histochemistry. J Proteome Res. 20 (4), 2069-2075 (2021).
- Geisler, C., Jarvis, D. L. Effective glycoanalysis with Maackia amurensis lectins requires a clear understanding of their binding specificities. Glycobiology. 21 (8), 988-993 (2011).
- Noro, E., et al. N -glycan structures of Wisteria floribunda agglutinin-positive Mac2 binding protein in the serum of patients with liver fibrosis. Glycobiology. 31 (10), 1268-1278 (2021).
- Rawitch, A. B., Pollock, H. G., Yang, S. X. Thyroglobulin glycosylation: Location and nature of the N-Linked oligosaccharide units in bovine thyroglobulin. Arch Biochem Biophys. 300 (1), 271-279 (1993).
- Dang, K., Zhang, W., Jiang, S., Lin, X., Qian, A. Application of lectin microarrays for biomarker discovery. Chemistry Open. 9 (3), 285-300 (2020).
- Chen, S., Qin, R., Mahal, L. K. Sweet systems: Technologies for glycomic analysis and their integration into systems biology. Crit Rev Biochem Mol Biol. 56 (3), 301-320 (2021).
- Dwivedi-Agnihotri, H., Srivastava, A., Shukla, A. K. Reversible biotinylation of purified proteins for measuring protein-protein interactions. Methods Enzymol. 633, 281-294 (2020).
- Shimazaki, H., et al. Auto-lectin dotcoding by two octopuses: Rapid analysis of fluorescence-labeled glycoproteins by an 8-channel fully-automatic bead array scanner with a rolling-circle detector. Anal Chem. 95 (32), 11868-11873 (2023).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon