Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Umfassende Charakterisierung der Gewebemineralisierung in einem ex vivo Modell
In diesem Artikel
Zusammenfassung
Das vorgeschlagene Protokoll beinhaltet einen globalen Ansatz zur Bewertung der Knochenbildung im Rahmen der Knochenregeneration mit Hilfe multimodaler Analysen. Ziel ist es, qualitative und quantitative Informationen über die Knochenneubildung bereitzustellen und so die Strenge und Validität grundlegender und präklinischer Untersuchungen zu verbessern.
Zusammenfassung
Die umfassende Charakterisierung der Gewebemineralisierung im Rahmen der Knochenregeneration stellt angesichts der zahlreichen Modalitäten, die derzeit für die Analyse zur Verfügung stehen, eine große Herausforderung dar. In dieser Arbeit schlagen wir einen Workflow für eine umfassende Bewertung der Knochenneubildung anhand eines relevanten großtierischen ossären ex vivo Explantats vor. In einem explantierten Femurkopf des Schafes wird ein Knochendefekt (Durchmesser = 3,75 mm; Tiefe = 5,0 mm) erzeugt und mit einem makroporösen Knochenersatzmaterial injiziert, das mit einem pro-osteogenen Wachstumsfaktor (bone morphogenetic protein 2 - BMP2) beladen ist. Anschließend wird das Explantat für einen Zeitraum von 28 Tagen in Kultur gehalten, was eine zelluläre Besiedlung und die anschließende Knochenbildung ermöglicht. Um die Qualität und Struktur des neu mineralisierten Gewebes zu bewerten, werden die folgenden aufeinander aufbauenden Methoden eingeführt: (i) Charakterisierung und hochauflösende 3D-Bilder des gesamten Explantats mittels Mikro-CT, gefolgt von Deep-Learning-Bildanalysen, um die Unterscheidung von mineralisiertem Gewebe zu verbessern; ii) Nano-Indentation zur Bestimmung der mechanischen Eigenschaften des neu gebildeten Gewebes; (iii) histologische Untersuchungen wie Hämatoxylin/Eosin/Safran (HES), Goldner-Trichrom und Movat-Pentachrom zur qualitativen Beurteilung des mineralisierten Gewebes, insbesondere im Hinblick auf die Visualisierung der Osteoidbarriere und des Vorhandenseins von Knochenzellen; (iv) Rückstreuende Rasterelektronenmikroskopie (REM)-Kartierung mit interner Referenz zur Quantifizierung des Mineralisierungsgrades und zur Bereitstellung detaillierter Einblicke in die Oberflächenmorphologie, die Mineralzusammensetzung und die Knochen-Biomaterial-Grenzfläche; (v) Raman-Spektroskopie zur Charakterisierung der molekularen Zusammensetzung der mineralisierten Matrix und zur Erlangung von Einblicken in die Persistenz von BMP2 im Zement durch den Nachweis von Peptidbindungen. Diese multimodale Analyse bietet eine effektive Beurteilung des neu gebildeten Knochens und umfassende qualitative und quantitative Einblicke in mineralisierte Gewebe. Durch die Standardisierung dieser Protokolle wollen wir Vergleiche zwischen Studien erleichtern und die Validität und Zuverlässigkeit der Forschungsergebnisse verbessern.
Einleitung
Knochendefekte, sei es durch Traumata, Tumorresektionen, angeborene Anomalien oder Infektionen, stellen eine große Herausforderung für die regenerative Medizin dar. Diese Veränderungen beeinträchtigen die strukturelle Integrität des Skelettsystems, was zu Beschwerden, Funktionsbeeinträchtigungen und einer Verringerung der Lebensqualität der Patienten führt.
Um diese Herausforderungen zu meistern, wurden innovative Knochenreparaturstrategien entwickelt, die sich auf die Verbesserung der Osteogenese und der Regeneration des Knochengewebes konzentrieren. Zu diesen Ansätzen gehört die Verwendung von implantierbaren, injizierbaren oder 3D-druckbaren Knochenersatzmaterialien, die natürlichen Ursprungs (z. B. biobasierte Makromoleküle, tierisches Hydroxylapatit) oder synthetisch (z. B. Biobrillen, Calciumphosphate) sein können1. Um ihre geringe inhärente Fähigkeit, die Knochenregeneration zu steuern und zu stimulieren, zu verbessern, können Knochenersatzmaterialien mit osteoinduktiven Faktoren wie knochenmorphogenetischen Proteinen (BMPs) beladen werden, um die osteogene Differenzierung von Vorläuferzellen zu fördern und die Knochenbildung zu verbessern2.
Die Knochenbildung basiert auf der anfänglichen Bildung einer Kollagenmatrix, die dann durch Hydroxylapatitkristalle mineralisiert wird und dadurch die Knochenstruktur stärkt3. Dieser Prozess verleiht dem Knochen eine spezifische Steifigkeit und Festigkeit. Die Qualität des mineralisierten Gewebes wird in hohem Maße durch seine mikrostrukturellen Eigenschaften und den Mineralisierungsgradbestimmt 4. Diese Eigenschaft spielt eine entscheidende Rolle für die Knochenheilung und die Funktionalität des regenerierten Knochens5. Die Charakterisierung der Knochenmineralisierung bleibt jedoch aufgrund der inhärenten Variabilität in multivariaten Studien eine schwierige Aufgabe 6,7,8.
Darüber hinaus werden in der Regel erste Bewertungen der Biokompatibilität, Zytokompatibilität und des Differenzierungspotenzials von Knochenersatzmaterialien in vitro durchgeführt. Methodische Unterschiede behindern jedoch einen nahtlosen Vergleich der Ergebnisse. Darüber hinaus erfassen diese In-vitro-Studien die multizellulären Wechselwirkungen und den komplexen Dialog zwischen Zellpopulationen, einschließlich Knochenmarkzellen, die für die Regulierung des Knochenregenerationsprozesses unerlässlich sind, nicht vollständig9. Dieser Mangel an genauer Darstellung der Mikroumgebung des Knochens kann die Genauigkeit nachfolgender präklinischer Studien beeinträchtigen10.
Obwohl In-vivo-Bewertungen eine genauere Darstellung physiologischer Zusammenhänge bieten, sind sie durch ethische, logistische und finanzielle Überlegungen eingeschränkt. Folglich spielen Ex-vivo-Evaluierungen eine zentrale Rolle als Schnittstelle zwischen In-vitro- und In-vivo-Studien und dienen als notwendiger Zwischenschritt, bevor sie zu Experimenten an lebenden Probanden übergehen 11,12,13.
In diesem Zusammenhang ist die Implementierung umfassender Charakterisierungsmethoden erforderlich, um die Qualität des regenerierten Knochengewebes zu beurteilen und die Relevanz der Strategie sicherzustellen, bevor zu einem präklinischen Modell übergegangen wird. Daher schlagen wir ein Protokoll vor, das auf der Analyse eines Explantatmodells unter Verwendung von Kniegelenksgewebe von Schafen basiert. Bei dieser innovativen Methode wird BMP2-beladener Zement in die Explantate implantiert und eine detaillierte Analyse der Gewebemineralisierung nach 28-tägiger Kultur durchgeführt.
Die in dieser Studie verwendeten technischen Ansätze sind vielfältig und komplementär und bieten zusammen einen umfassenden Ansatz zur Bewertung der Qualität von regeneriertem Knochengewebe (Abbildung 1). Die hochauflösende Mikro-CT-Bildgebung ermöglicht eine detaillierte 3D-Visualisierung der Knochenstruktur und liefert wertvolle Einblicke in die Mineraldichte, Morphologie und Integrität des neu gebildeten Gewebes. Diese Technik ist entscheidend für die Beurteilung der Wirksamkeit der Knochenregeneration und die Überwachung des Fortschreitens der Mineralisierung im Laufe der Zeit. Die Nanoindentation ist ein präziser Ansatz, um die mechanischen Eigenschaften des Gewebes, wie z. B. seine Härte und Festigkeit, zu bestimmen. Durch die Messung der Reaktion des Materials auf eine ausgeübte Kraft auf einer nanometrischen Skala ermöglicht diese Methode die Beurteilung der Robustheit und Qualität des mineralisierten Gewebes. Histologische Untersuchungen mit gängigen Färbungen wie Hämatoxylin/Eosin/Safran (HES), Goldner-Trichrom und Movat-Pentachrom liefern unschätzbare Einblicke in die Gewebestruktur und -zusammensetzung. Diese Färbungen ermöglichen die Differenzierung der verschiedenen Gewebebestandteile, einschließlich Zellen, extrazellulärer Matrix und mineralischer Ablagerungen, und ermöglichen so eine umfassende qualitative Beurteilung des Knochenregenerationsprozesses. Das Backscatter-Rasterelektronenmikroskopie-Mapping (REM) bietet eine hochauflösende Visualisierung der Oberfläche der Proben, die eine detaillierte Analyse des Mineralisierungsgrades der Knochenmatrix sowie der Grenzflächen zwischen dem implantierten Material und dem Wirtsgewebe ermöglicht. Schließlich gibt die Raman-Spektroskopie Aufschluss über die molekulare Zusammensetzung des Gewebes, insbesondere durch die Identifizierung bestimmter Bestandteile wie Proteine, Lipide und Mineralien. Dieser Ansatz ermöglicht die Charakterisierung der mineralisierten Matrix und den Nachweis von Wachstumsfaktoren wie BMP2 und liefert damit entscheidende Informationen über die Persistenz von pro-osteogenen Reizen im Regenerationsmedium.
Mit einem multidisziplinären Ansatz, der verschiedene Analysetechniken integriert, zielt unsere Studie darauf ab, eine gründliche und umfassende Bewertung der Qualität von regeneriertem Knochengewebe zu liefern und so eine solide Grundlage für die Bewertung von Knochenersatzmaterialien und deren potenzielle klinische Anwendung zu schaffen.
Access restricted. Please log in or start a trial to view this content.
Protokoll
Diese Studie wurde von einer Ethik- und Tierschutzkommission und von der französischen nationalen Veterinär- und Lebensmittelbehörde unter der Nummer G44171 genehmigt.
1. Präparation und Kultivierung von osteochondralen Explantaten
- Ernten Sie Proben von Schafspediten von frisch eingeschläferten Tieren in einer aseptischen Umgebung. Positionieren Sie das Schaf in Rückenlage und rasieren Sie das linke Hinterglied. Bereiten Sie sich vor, indem Sie das Kniegelenk mit Alkohol desinfizieren. Verwenden Sie eine laterale parapatellare Arthrotomie, um das vordere und hintere Kreuzband freizulegen, gefolgt von einer Patellaluxation, um die femorale Trochlea freizulegen.
- Nach der Gewebeentnahme wird mit einem osteochondralen Autotransplantat-Transfersystem ein Defekt mit einem Durchmesser von 4,75 mm im lateralen Kondylus erzeugt.
- Erzeugen Sie mit einem orthopädischen Hammer einen Defekt in 10 mm Tiefe und entnehmen Sie das Explantat aus dem Kondylus. Anschließend wird ein zweiter Defekt mit einem Durchmesser von 8 mm durchgeführt, der um den ersten zentriert ist, unter Verwendung eines osteochondralen Autotransplantat-Transfersystems. Daraus ergeben sich osteochondrale Explantate mit den Maßen 8 mm x 10 mm, die einen zentralen Defekt von 4,75 mm x 10,0 mm enthalten.
- Falls es aus logistischen Gründen erforderlich ist, transportieren Sie die Explantate in Hank's Balanced Salt Solution (hBSS), ergänzt mit 1% Penicillin-Streptomycin, und lagern Sie sie bei 4 °C.
- Spülen Sie die Explantate 3x mit phosphatgepufferter Kochsalzlösung (PBS) und injizieren Sie dann den 4,75 mm x 10,0 mm großen Defekt mit einem Calciumphosphatzement, der mit 40 μg/ml BMP2 ergänzt wird. Bereiten Sie den Zement wie unten beschrieben vor.
- Herstellung von α-Tricalciumphosphat (α-TCP) durch Wärmebehandlung von apatitischen Tricalciumphosphatstäben (isostatische Presse bei 120 MPa) bei 1364 °C für mindestens 12 Stunden, gefolgt von Luftabschreckung14.
- Mahlen Sie die α-TCP-Stäbe in absolutem Ethanol mit einer Planeten-Mühlenkugel 2x für 5 min bei 500 U/min, um ein feines Pulver von 13,1 ± 1,7 μm herzustellen (gemessen mittels Laserbeugung, wie zuvorbeschrieben 14).
- Sterilisieren Sie das α-TCP-Pulver, indem Sie es 45 Minuten lang bei 180 °C trocken erhitzen. Bereiten Sie den Calciumphosphat-Zement vor, indem Sie α-TCP-Pulver mit einer 2,5%igen (w/v) Na2HPO4 0,22 μm filtrierten Lösung (Flüssig-Pulver-Verhältnis von 0,35) für 1 min mischen.
- Geben Sie den Zementkleber vor der Injektion mit einer 18-G-Nadel in eine 3-ml-Spritze.
- Nach 10 Minuten bei Raumtemperatur (RT) werden die Explantate in einen 25 cm² großen Kolben überführt und vorsichtig 10 ml vollständiges Kulturmedium hinzugefügt, bestehend aus Dulbecco's Modified Eagle Medium (DMEM) hoher Glukose, ergänzt mit 10 % fötalem Rinderserum und 1 % Penicillin-Streptomycin.
- Bewahren Sie die Explantate bis zu 28 Tage in einem 37 °C heißen Inkubator mit 5 % CO2 auf und wechseln Sie das gesamte Kulturmedium alle 2 Tage unter einer Laminar-Flow-Haube.
- Spülen Sie die Explantate 3x mit PBS aus. Füllen Sie sie in 50-ml-Röhrchen um, fügen Sie 20 ml 4% Paraformaldehyd (PFA) hinzu und halten Sie sie 3 Tage lang bei 4 °C. Spülen Sie dann die Explantate mit PBS und lagern Sie sie in 70%igem Ethanol, bevor Sie sie für nachfolgende Analysen verwenden.
2. Mikro-CT-Analyse
- Führen Sie Mikrotomographie-Analysen mit einem Mikro-CT-Röntgenbild durch. Erfassen Sie Röntgenprojektionen mit einer Auflösung von 10,7 μm, mit einer Belichtungszeit von 1200 ms und einem 1-mm-Aluminiumfilter (80 kV und 125 μA). Durchschnittlich drei Bilder pro 0,45°-Rotationsinkrement, um das Signal-Rausch-Verhältnis zu verbessern.
- Rekonstruieren Sie dreidimensionale (3D) Bilder mit der Rekonstruktionssoftware des Herstellers nach einer X/Y-Ausrichtung mit einem Referenzscan mit den folgenden Parametern - Glättung: 0, Ringartefakt: 3, Strahlhärtung: 35%. Bereinigen Sie die Bildstapel mit einer Bildverarbeitungssoftware und beobachten Sie mit zweidimensionalen (2D) und 3D-Visualisierungswerkzeugen.
3. Deep-Learning-Bildanalysen
- Führen Sie die Bildsegmentierung mit spezieller, spezialisierter Software durch. Verwenden Sie den integrierten Segmentierungsassistenten, um ein Deep-Learning-Modell für die Unterscheidung von Knochen und Zement zu trainieren.
- Wählen Sie aus den rekonstruierten Mikro-CT-Bildern eine repräsentative Zone (d. h. einen Rahmen) aus, die Knochen, Zement und Hintergrund enthält. Segmentieren Sie dieses erste Bild manuell mit einfachen (z. B. Schwellenwert) oder leistungsstarken Werkzeugen (z. B. Ausbreitung), die von der Software bereitgestellt werden.
- Generieren Sie ein Deep-Learning-Modell auf der Registerkarte Modell und wählen Sie im Dropdown-Menü die Option 3D-Unet-Routine aus. Definieren Sie seine experimentellen Parameter in Adgleichung mit den zu analysierenden Bildern, indem Sie mit der rechten Maustaste auf das generierte Modell klicken. hier: Tiefe von 5, Patch-Größe 32 x 32, Adadetla-Algorithmus, Schrittzahl von 0,25, Datenerweiterung x10.
- Verwenden Sie den segmentierten Rahmen, um die Deep-Learning-Routine zu trainieren (klicken Sie auf die Schaltfläche "Trainieren"), die sich aufgrund ihrer schnellen und genauen Bildsegmentierungsfunktionen als relevant erwiesenhat 3,15. Definieren Sie nach Abschluss des Trainings einen zweiten Arbeitsbereich (Rahmen) und segmentieren Sie automatisch mit der Vorhersagefunktion.
- Wenden Sie bei Bedarf eine manuelle Korrektur an, um genauere Trainingsdaten zu generieren. Trainieren Sie wieder Routine. Wiederholen Sie den Vorgang, bis ein zufriedenstellendes Ergebnis erzielt wird. Die Anzahl der Datenerweiterungen nimmt mit zunehmender Anzahl der Trainingsframes schrittweise ab.
HINWEIS: Der DICE-Index, der während des Modelltrainings bereitgestellt wird, gibt einen Hinweis auf die Genauigkeit des Modells (im Vergleich zu den Eingabeinformationen), ist jedoch nicht ausreichend. Die Bestätigung der Relevanz des Modells sollte durch die Validierung der automatisch segmentierten Daten durch 3 unabhängige erfahrene Forscher erfolgen.
- Wenden Sie bei Bedarf eine manuelle Korrektur an, um genauere Trainingsdaten zu generieren. Trainieren Sie wieder Routine. Wiederholen Sie den Vorgang, bis ein zufriedenstellendes Ergebnis erzielt wird. Die Anzahl der Datenerweiterungen nimmt mit zunehmender Anzahl der Trainingsframes schrittweise ab.
- Veröffentlichen Sie das Modell, indem Sie auf die Schaltfläche Exportieren klicken, und wenden Sie es auf den gesamten Mikro-CT-Datensatz an, indem Sie auf Segment > Exportiertes Modell > Segment-Volldatensatz klicken.
4. Einbettung
- Dehydrieren Sie die Knochenexplantate, indem Sie sie in ein 40-ml-Glasfläschchen mit 25 ml Dehydratisierungslösung geben, die aus 70 % Aceton und 30 % Xylol besteht. Stellen Sie das Glasfläschchen für 1 h bei RT auf ein rotierendes Rad. Wiederholen Sie diesen Schritt 3x.
- Ersetzen Sie die Dehydratisierungslösung durch 25 mL Xylol und stellen Sie das Glasfläschchen für 1 h bei RT auf das rotierende Rad.
- Ersetzen Sie die Xylollösung durch 25 ml Methylmethacrylat, angereichert (MMA) mit 10 % Benzoylperoxid (BPO) und 10 % Dibutylphthalat (DBP). Lege es für 1 h bei RT auf das rotierende Rad und wiederhole diesen Schritt 2x.
- Ersetzen Sie die MMA-Lösung durch 25 ml MMA, angereichert mit 10 % BPO, 10 % DBP und 450 μl N, N-Dimethylanilin, verdünnt 1:20 in Propan-2-ol. Stellen Sie das Glasfläschchen über Nacht bei -20 °C auf. Die Lösung wird gelblich.
- Legen Sie das Explantat in eine mittelgroße Einbettform und gießen Sie MMA-BPO-DBP-N, N-Anilin in die Form. Stellen Sie die Form in eine Kunststoffbox und belüften Sie sie 5 Minuten lang mit Stickstoff. Verschließen Sie die Box hermetisch und stellen Sie sie für 48 h bei 4 °C für die MMA-Polymerisation und -Härtung auf.
- Entfernen Sie das Harz, das das Explantat enthält, aus der Form und halten Sie es bei 4 °C, bis es für die anschließende Analyse verarbeitet wird.
5. Rasterelektronenmikroskopie (REM) - quantitative Rückstreuelektronenbildgebung (qBEI)
- Schneiden Sie den pMMA-Block mit dem Explantat mit einer Diamantsäge entlang seiner Längsachse ab. Bewahren Sie die erste Hälfte des Blocks für die histologische Analyse auf. Schneiden Sie die andere Hälfte weiter ab, um einen 1,5 mm dicken Abschnitt zu erhalten. Führen Sie den Schnitt bei 3000 U/min mit einer Geschwindigkeit von 3 mm/min durch.
- Schleifen Sie den Abschnitt mit Siliziumkarbidpapier mit aufsteigenden Zahlen, die der niedrigeren Korngröße entsprechen: SiC 320 für 10 s, SiC 1000 für 15 s, SiC 2000 für 30 s und SiC 4000 für 30 s.
- Polieren Sie den Abschnitt mit Diamantpaste und speziellen Kleidungsstücken: Poliertuch mit 3 μm Mol B3 Diamantlösung für 1 min und Vliestuch mit 1 μm Nap B1 Diamantlösung für 1 min.
- Spülen Sie den Abschnitt unter doppelt destilliertem Wasser ab und reinigen Sie Diamantpartikel mit einem Wattestäbchen. Trocknen Sie den Abschnitt unter Stickstoffgas.
- Beschichten Sie den dicken Abschnitt mit Carbon (10 nm Kohlenstofffilmstärke) und montieren Sie ihn mit silberner Farbe auf einen Aluminiumstummel. Mache eine silberne Farbbrücke zwischen dem oberen Abschnitt und dem Stummel, um die Evakuierung der Elektronenladung zum Boden zu ermöglichen.
- Platzieren Sie den Stummel auf dem REM-Tisch. Führen Sie neben der Probe einen Kontrollstutzen ein, der aus einem Faradayschen Becher mit Kohlenstoff-, Aluminium- und Silikonstandards besteht, in die REM-Kammer. Dies wird verwendet, um den Elektronenstrahl zu kalibrieren und die Graustufen in Prozentsätze von Ca umzuwandeln. Schließen Sie die REM-Kammer und vakuumieren Sie sie.
- Schalten Sie den Elektronenstrahl ein und passen Sie die REM-Parameter so an, dass sie im Modus mit rückgestreuten Elektronen ausgeführt werden. Bei rückgestreuten Bildern beträgt der Grauwert des Kohlenstoffstandards 25, des Aluminiumstandards 225 und des Siliziumstandards 253. Als Anhaltspunkt sind die REM-Parameter auf dem verwendeten System die Beschleunigungsspannung 20 keV, der mit dem Faradayschen Becher gemessene Sondenstrom: 250 pA, der Arbeitsabstand: 15 mm, die Blende: 30 μm, die Bildauflösung: 1024 x 768 Pixel, das Vakuum: < 4,10-4 Pa, die Verweilzeit von 100 μs/Pixel, die Helligkeit ~38 und der Kontrast ~72.
- Wenn das REM mit den Standards kalibriert ist, nehmen Sie Bilder der Probe im Rückstreuelektronenmodus auf.
- Verwenden Sie das rückgestreute Elektronenbild der Probe, um die Graustufen in Kalziumgehalt umzuwandeln, wie in Roschger et al.16 beschrieben. Führen Sie dies mit einer beliebigen Bildanalysesoftware durch.
- Plotten Sie die Verteilung des Kalziumgehalts. Die Verteilung des Kalziumgehalts zeigt eine Gaußsche Verteilung. Drei Hauptparameter von Interesse werden berechnet:Ca-Mittelwert als mittlerer Kalziumgehalt auf dem Bild,Ca-Peak als die häufigste Kalziumkonzentration auf dem Bild undCa-Breite als volle Breite bei halber Maximumverteilung des Kalziumgehalts.
HINWEIS: Die Reproduzierbarkeit von qBEi wurde untersucht, indem die gleiche Region of Interest an 5 aufeinanderfolgenden Tagen (eine Aufnahme pro Tag) abgebildet wurde. Das bedeutet, dass der Elektronenstrahl zwischen den Sitzungen ein- und ausgeschaltet wurde. Der Fehlerprozentsatz inCa-Mittelwert,Ca-Peak und Cawidth wurde auf 0,5 %, 0,7 % bzw. 1,2 % geschätzt. Der Kalziumgehalt wurde auch mittels energiedispersiver Röntgenanalyse (EDS) untersucht. Es wurde ein sehr guter linearer Zusammenhang zwischen demCa-Mittelwert und dem von EDS geschätzten Calciumgehalt mit einem R²-Wert von 0,99 festgestellt, ähnlich den Daten von Roschger et al. zuvor17.
6. Histologie
- Für jede Probe werden 7 μm-Schnitte mit einem Hartgewebemikrotom und einer Wolframklinge 18,19 geschnitten. Färben Sie Abschnitte mit Goldner Trichrome, HES und MOVAT mit einem automatischen Färbesystem.
- Zur Entplastifizierung die Proben nacheinander 5 min in Aceton einweichen und 2x wiederholen. Spülen Sie die Proben 5 Minuten lang in destilliertem Wasser und wiederholen Sie dies 2x.
- Führen Sie die Goldner Trichrome-Färbung wie unten beschrieben durch.
- Legen Sie die Proben für 20 Minuten in Weigert Eisen-Hämatoxylin. Mit Leitungswasser waschen. Tauchen Sie die Proben 30 s-1 min lang in 0,5 % sauren Alkohol. 20 Min. mit Leitungswasser waschen.
- Die Proben werden 5 Minuten lang in Ponceau/Fuchsinsäure/Azophloxin-Lösung gelegt. 1 min in 1%ige Essigsäure tauchen.
- Mit Phosphomolybdsäure/Orange G-Lösung 20 min einfärben. 1 min in 1%ige Essigsäure tauchen.
- Mit schneller grüner Lösung 15 min bei Raumtemperatur oder 8 min bei 60 °C beizen. Gründlich mit Leitungswasser abspülen; 3x wiederholen.
- Führen Sie die Movat Pentachrome-Färbung wie unten beschrieben durch.
- Alcian Blue Färbung: Proben 30 Minuten lang in Alcian Blue Lösung tauchen. Spülen Sie die Proben 5 Minuten lang mit Leitungswasser ab. Legen Sie die Proben für 60 Minuten in alkalisches Ethanol. Spülen Sie die Proben 10 Minuten lang in Leitungswasser. Führen Sie eine abschließende Spülung mit destilliertem Wasser durch.
- Weigert Hämatoxylin-Färbung: Proben für 20-30 min in Weigert's Ferric Hämatoxylin legen. Spülen Sie die Proben 15 Minuten lang mit Leitungswasser ab. Erneut mit destilliertem Wasser abspülen.
- Färbung mit Crocein Brillant/Fuchsinsäure: Tauchen Sie die Proben für 10 min in eine Lösung aus Crocein Brillant/Sauerfuchsin. Mit 0,5 % Essigsäure abspülen. Tauchen Sie die Proben 20 Minuten lang in eine 5%ige Phosphowolframsäurelösung. Mit 0,5%iger Essigsäure 2 Min. spülen. Führen Sie drei aufeinanderfolgende Spülgänge mit 100 % Ethanol durch, die jeweils 5 Minuten dauern.
- Färben mit Safran du Gatinais: Proben für 15 Minuten in Safran du Gatinais legen. Führen Sie eine abschließende Spülung mit Leitungswasser durch.
- Führen Sie die HES-Färbung wie unten beschrieben durch.
- Färbung mit Weigert-Hämatoxylin: Tauchen Sie die Proben für 30 min in Weigert-Hämatoxylin. 2 Min. mit Leitungswasser abspülen.
- Entfärbung: Tauchen Sie die Proben 10 s lang in Salzsäurealkohol. 2 Min. mit Leitungswasser abspülen.
- Neutralisation: Tauchen Sie die Proben 1 Minute lang in eine Lösung aus Lithiumcarbonat. 2 Min. mit Leitungswasser abspülen. Führen Sie eine abschließende Spülung mit destilliertem Wasser für 1 Minute durch.
- Färbung mit Eosin-Erythrosin: Legen Sie die Proben für 3 Minuten in eine Lösung von Eosin-Erythrosin. 10 s mit Leitungswasser spülen.
- Dehydratisierung: Tauchen Sie die Proben 15 s lang in 95%igen Alkohol. 15 s lang auf 100% Alkohol umfüllen. Wiederholen Sie das Eintauchen in 100% Alkohol für 30 s.
- Abschließende Färbung mit alkoholischem Safranin: Tauchen Sie die Proben 10 Minuten lang in alkoholisches Safranin. Führen Sie eine abschließende Spülung mit Leitungswasser durch.
- Führen Sie die Folienmontage wie unten beschrieben durch.
- Spülen Sie die Proben mit 95 % Ethanol, dann mit 100 % Ethanol, dann mit Methylcyclohexan und wiederholen Sie dies 3x für jeden Schritt.
- Montieren Sie die Proben mit einem Montagesatz und fügen Sie ein Deckglas hinzu. Scannen Sie die Bilder mit einem digitalen Diascanner.
7. Raman-Mikrospektroskopie
- Verwenden Sie den gleichen Abschnitt, der für die Raman-Analyse verwendet wird wie das REM. Schleifen und polieren Sie den Abschnitt kurz, wie in den Schritten 5.2 bis 5.3 beschrieben, um die für das REM hinzugefügte dünne Kohlenstoffschicht zu entfernen, die die Wärmeableitung aus der Probe beeinträchtigt.
- Legen Sie den Schnitt auf den Tisch des Raman-Mikrospektrometers. Kalibrieren Sie die Wellenzahl, um die Genauigkeit sicherzustellen und den Laser vor der Messung auszurichten. Da solche Verfahren für jedes Instrument spezifisch sind, werden sie in diesem Artikel nicht beschrieben.
- Lokalisieren Sie interessante Bereiche im Gewebe, die analysiert werden müssen. Positionieren Sie die zu analysierende Zone in der Mitte des Videowerkzeugs und erfassen Sie Raman-Spektren mit folgenden Parametern: 785 nm Laser bei 30 mW, Integrationszeit: 20 s 3 mal wiederholt, Spektralbereich: 350-1800 cm-1, Gitter 1200 Linien/mm.
- Sammeln Sie 10 Spektren des Einbettungsharzes mit den gleichen Raman-Einstellungen und mitteln Sie diese. Verwenden Sie dies, um den Harzbeitrag in Schritt 7.6 zu subtrahieren.
- Bearbeiten Sie die Spektren wie folgt vor, um den Beitrag von Fluoreszenz, Rauschen und Harz zu entfernen: Basislinienkorrektur mit einer Polynomanpassung fünfter Ordnung, Savitzky-Golay-Filter mit einem Grad von 4 und einer Fenstergröße zwischen 17 und 21, digitale Harzsubtraktion unter Verwendung des ~812 cm-1-Peaks des Harzes. Eine solche Vorverarbeitung kann mit verschiedenen Softwares durchgeführt werden, die für die Datenanalyse und -visualisierung entwickelt wurden.
- Analysieren Sie die vorverarbeiteten Spektren weiter, um wertvolle Parameter zu extrahieren, indem Sie die Spitzenintensität der interessierenden Schwingung mit der Spitzenintensität der Referenzschwingung rationieren. Verwenden Sie für die Knochenanalyse z. B. die folgenden Parameter, um sie zu berechnen.
- Mineral-zu-Matrix-Verhältnisse: Teilen Sie die Spitzenintensität der v1PO4-Schwingungen bei ~960 cm-1 durch die Spitzenintensität der Schwingungsmoden Amid I (~1668 cm-1), Amid III (~1250 cm-1), CH2-wag (~1450 cm-1) oder die Summe aus Prolin (~854 cm-1) und Hydroxyprolin (Hyp, ~872 cm-1). Der v2PO4, der sich bei ~430 cm-1 befindet, oder der v4PO4, der sich bei ~600 cm-1 befindet, kann ebenfalls verwendet und mit dem Amide III Vibration20 abgestimmt werden. Diese Parameter stellen den Mineralisierungsgrad der organischen Phase der Knochenmatrix dar.
- Karbonat/Phosphat: Teilen Sie die Spitzenintensität des v1CO3, das sich bei ~1070 cm-1 befindet, durch das v1PO4. Dies stellt die Menge der Typ-B-Karbonatsubstitution im Apatitgitter dar. Hyp/Pro, repräsentiert den Gehalt an Hydroxyprolin, ~1375 cm-1/Amide III, repräsentiert den Proteoglykangehalt, ~1150 cm-1/CH2-wag und ~1495 cm-1/CH2-wag, repräsentiert die Menge der fortgeschrittenen Glykationsendprodukte Carboxymethylysin und Pentosidin, 1670 cm-1/1690 cm-1 und 1670 cm-1/1640 cm-1 stellen die ungeordnete Sekundärstruktur des Kollagens, auch Kollagenreife genannt, bzw. geordnete Strukturen in Form von α-Helix im Typ-I-Kollagen dar.
- Bewerten Sie die Reproduzierbarkeit von Raman-Messungen, indem Sie denselben interessierenden Bereich in 5 aufeinanderfolgenden Aufnahmen (eine Aufnahme pro Tag) untersuchen. Für jeden analysierten Parameter lag die Varianz unter 0,2 % des Parameterwertes, was eine gute Reproduzierbarkeit unterstützt. Die Spezifität jedes untersuchten Peaks wurde in der Praxis ausführlich diskutiert, und ein Überblick wird in der jüngsten Übersichtsarbeit von Unal21 gegeben.
8. Nanoindentation
HINWEIS: Aufgrund der destruktiven Natur der Nanoindentation wird sie in der Regel am Ende der Probenanalyseroutine durchgeführt. Das Nanoindentationssystem, das wir besitzen, ist mit einem pyramidenförmigen Berkovitch-Diamanteindringkörper ausgestattet. Es gibt jedoch mehrere Eindringkörperformen, und für Knochen- oder Biomaterialproben wurde in der Literatur kein Konsens festgestellt.
- Hydratisieren Sie die eingebettete Knochenprobe 16 Stunden lang in Kochsalzlösung bei RT, bevor Sie den Nanoindentationstest durchführen.
- Kalibrieren Sie das Nanoindentationssystem mit Quarzglas und zeichnen Sie den resultierenden Indentationsmodul auf. Für Quarzglas beträgt der Eindringmodul etwa 72 GPa bei einem Poisson-Koeffizienten von 0,17.
- Sobald das Nanoindentationssystem kalibriert ist, platzieren Sie die für die Raman-Analysen verwendete Probe in das optische System des Nanoindentationsgeräts, um die Stellen zu identifizieren, an denen die Nanoindentation durchgeführt wird.
HINWEIS: Diese Modalität variiert von Gerät zu Gerät; Wir gehen nicht detailliert darauf ein, wie das Nanoindentationssystem und die Software funktionieren, sondern geben allgemeine Ratschläge zu den verschiedenen Schritten, die durchgeführt werden sollten. - Sobald der Ort der Nanoindentation ausgewählt ist, bewegen Sie die Probe unter das Indentationsgerät und führen Sie die Indentation durch. Führen Sie die Nanoindentation bei einer konstanten Tiefe von 900 nm mit einer Lade-/Entladegeschwindigkeit von 40 mm/min und einer Pause von 15 s zwischen Be- und Entladen durch. Legen Sie den Poisson-Koeffizienten für Knochengewebe auf 0,3 fest.
- Holen Sie sich die verschiedenen Parameter aus den Nanoindentationskurven und berechnen Sie sie gemäß der Theorie von Oliver und Pharr22.
- Der Eindringmodul (EIT) leitet sich aus den bekannten Eigenschaften der Eindringspitze und dem reduzierten Modul ab und kombiniert den lokalen Elastizitätsmodul der Probe. Berechnen Sie den Einzug wie folgt:
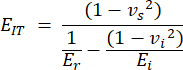
wobei vs, vi, Ei und Er den Poisson-Koeffizienten der Probe darstellen, der für Knochen mit 0,3 angenommen wird, der Poisson-Koeffizient des Diamant-Eindringkörpers, der mit 0,07 angenommen wird, der Elastizitätsmodul des Diamant-Eindringkörpers, festgelegt auf 1140 GPa, und der reduzierte Modul, berechnet wie folgt: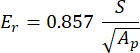
Dabei steht S für die Neigung des Entladesegments und Ap für die projizierte Fläche und wird wie folgt berechnet: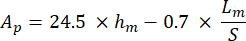
- Härte (HIT): entspricht der Widerstandsfähigkeit des Knochens gegen die Entstehung und Ausbreitung von Rissen. Mit anderen Worten, es ist ein Indikator für die Zähigkeit der Knochen. Berechnen SieH IT als:

- Eindringkriechgeschwindigkeit (CIT): Dies spiegelt die Knochenverformung im Laufe der Zeit bei konstanter Belastung wider, die auf die viskoelastischen Eigenschaften der Probe hinweist, berechnet als:
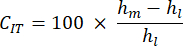
- Maximale Belastung (Lm): Dies entspricht der entgegengesetzten Belastung, die notwendig ist, um in die angegebene Tiefe in die mineralisierte Knochenmatrix einzudringen.
- Eindringarbeiten (Wplast): Dies entspricht der Fläche unter der Last-/Entlastungsverformungskurve. Es handelt sich um eine direkte Reflexion der abgegebenen Energie, um eine plastische Verformung zu induzieren.
HINWEIS: Die Reproduzierbarkeit der Nanoindentation ist aufgrund der destruktiven Natur dieser Methode an Knochenproben schwieriger zu beurteilen. Nichtsdestotrotz haben wir die Reproduzierbarkeit der Standard- und einheitlichen Quarzglasprobe abgeschätzt. Für alle oben genannten Parameter wurde die Varianz auf unter 0,26 % geschätzt (5 aufeinanderfolgende Messungen an 5 verschiedenen Tagen), was eine sehr gute Reproduzierbarkeit unterstützt. Auf der anderen Seite ist es aufgrund der anisotropen Natur von Knochenproben schwieriger, ihre Reproduzierbarkeit zu beurteilen.
- Der Eindringmodul (EIT) leitet sich aus den bekannten Eigenschaften der Eindringspitze und dem reduzierten Modul ab und kombiniert den lokalen Elastizitätsmodul der Probe. Berechnen Sie den Einzug wie folgt:
Access restricted. Please log in or start a trial to view this content.
Ergebnisse
Ein Mikro-CT-Bild des Explantats ist in Abbildung 2 dargestellt. Die manuelle Segmentierung kann den Knochen nicht optimal vom Zement trennen, der im Zentralkanal vorhanden ist, indem die globale Schwellenwertierung verwendet wird. Um die Erkennung von trabekulärem Knochen und Zement zu verbessern, schlagen wir den Einsatz von Deep Learning vor. Deep Learning ist leistungsstark für die Erkennung von Biomaterialeigenschaften und hilft, die Trennung zwischen...
Access restricted. Please log in or start a trial to view this content.
Diskussion
Die Reparatur von Knochendefekten ist eine große Herausforderung in der regenerativen Medizin, um die Beweglichkeit wiederherzustellen, Schmerzen zu lindern und die Lebensqualität der Betroffenen zu verbessern. Die Verwendung von Explantatmodellen bietet eine Reihe von Vorteilen gegenüber in vivo Studien zur Untersuchung der Knochendefektreparatur. Neben ethischen Überlegungen ermöglicht dieses Modell auch die rigorose Kontrolle der Versuchsbedingungen und die Reduzierung d...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Die Autoren haben keine Interessenkonflikte offenzulegen.
Danksagungen
Wir danken den technischen Einrichtungen, die an der Entnahme und Verarbeitung der Proben beteiligt sind, darunter SC3M (SFR François Bonamy (UMS 016), Universität Nantes), SFR ICAT (Universität Angers), BIO3, HiMolA und SC4BIO. Das Inserm UMR_S 1229 RMeS wird durch Zuschüsse der französischen Regierung über die Einrichtungen Inserm, Nantes Université, Univ Angers und Oniris VetAgroBio unterstützt. CL ist auch HTL Biotechnology dankbar.
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| 0.20 filters | VWR | 28145-501 | |
| 18 G needle (1,2x40 mm) | Sterican | 4665120 | |
| 3 mL syringe | HENKE-JECT | 8300005762 | |
| 37% hydrochloric acid | VWR | 1.00317.1000 | |
| Acetic acid (glacial) | Sigma | A6283 | |
| Acetone | VWR | 20063-365 | |
| Alcian Blue 8GX | VWR | 361186 | |
| Ammonium hydroxide | VWR | 318612 | |
| Apatitic tricalcium phosphate | Centre for Biomedical and Healthcare Engineering (Mines Saint Etienne, France) | TV26U | |
| Azophloxine | Sigma | 210633 | |
| Benzoyl peroxide | Sigma | 8.01641.0250 | |
| BMP2 | Medtronic | InductOs 1.5 mg/mL | |
| Brillant crocein | Aldrich | 2107507 | |
| CTVox | Bruker | - | |
| DataViewer | Skyscan | - | |
| Diamond blade | Struers | MOD13 | |
| Diamond saw | Struers | Accutom-50 | |
| DiaPro Mol B3 diamond solution | Struers | 40600379 | |
| DiaPro Nap B1 diamond solution | Struers | 40600373 | |
| Dibasic sodium phosphate (Na2HPO4) | Sigma | 102404598 | |
| Dibutyl Phtalate | Chimie-Plus Laboratoires | 28656 | |
| DragonFly software | ORS | 2022.1.0.1231. | |
| Dulbecco's Modified Eagle Medium (DMEM) high glucose, GlutaMAX(TM), pyruvate | ThermoFisher Scientific | 31966-021 | |
| Eosine Y- Surgipath | Sigma | 1002830105 | |
| Erythrosin B | Sigma | 102141057 | |
| Ethanol absolute | VWR | 20820362 | |
| Eukitt | Dutscher | 6.00.01.0003.06.01.01 | |
| Falcon 50 mL | Sarstedt | 62.547.254 | |
| Ferric chloride hexahydrate (FeCl3, 6H2O) | Merck | 1.03943.0250 | |
| Fetal Bovine Serum (FBS) | Eurobio | CVFSVF00 | |
| Fuchsine acid | Merck | 1.05231.0025 | |
| Hank's Balanced Salt Solution (HBSS) | Biosera | MS01NG100J | |
| Hematoxylin | Sigma | 86.118.9 | |
| Isostatic press | Nova Suisse | Pmax 1500 bars | |
| Laser diffraction granulometry | Malvern | Mastersizer 3000 | |
| Light green | Prolabo | 28947135 | |
| Lithium carbonate | Sigma | A13149 | |
| MD-Mol polishing cloth | Struers | 40500077 | |
| Methylcyclohexane | VWR | 8.06147.1000 | |
| Methylcyclohexane | VWR | 8.06147.1000 | |
| Methylcyclohexane | VWR | 8.06147.1000 | |
| Methylmethacrylate | Sigma | 8.00590.2500 | |
| Micro-CT, micro-scanner | Bruker | Skyscan 1272 | |
| Monobasic sodium phosphate (NAH2PO4) | Sigma | 71496 | |
| Mortar | Fritsch | Pulverisette 6 | |
| N,N, Dimethylanilin | Sigma | 803060 | |
| Nanoindentation station | Anton Paar | NHT2 | |
| ND-Nap polishing cloth | Struers | 40500080 | |
| OATS Osteochondral Autograft Transfer System Set, 4,75 mm | Arthrex | AR-1981-04S | |
| OATS Osteochondral Autograft Transfer System Set, 8 mm | Arthrex | AR-1981-08S | |
| Orange G | Ral | M15 | |
| Paraformaldehyde (PFA) | Sigma | P6148 | |
| Peel-a-way disposable embbedding moulds | Polysciences, Inc | 18646C-1 | |
| Penicillin/Streptomycin (P/S) | ThermoFisher Scientific | 15140122 | |
| Phosphate Buffered Saline (PBS) | ThermoFisher Scientific | 10010023 | |
| Phosphomolybdic acid | Sigma | 221856-100 g | |
| Phosphotungstic acid | Aldrich | 12863-5 | |
| Polishing machine | Sturers | Dap V | |
| Poupinel | MEMMERT | TV26U | |
| Raman microspectrometer | Renishaw | InVia Qontor | |
| Safran du Gâtinais | Labonord | 11507737 | |
| Scanning electron microscope | Carl Zeiss | Evo LS 10 | |
| SEM | Zeiss | Carl Zeiss Evo LS10 | |
| SiC foils/Grinding papers | Struers | 40400008 (#320), 40400011 (#1000), 40400122 (#2000), 40400182 (#4000) | |
| Silver paint | Electron microscopy sciences | 12686-15 | |
| Standard stub with Faraday cup, carbon, aluminium and silicon standards | Micro-Analysis Consultants Ltd | 8602 | |
| T25 flask | Corning | 430639 | |
| Xylene | VWR | 28975.325 | |
| Xylidine Ponceau | Aldrich | 19.976-1 |
Referenzen
- Feroz, S., Cathro, P., Ivanovski, S., Muhammad, N. Biomimetic bone grafts and substitutes: A review of recent advancements and applications. Biomed Eng Adv. 6, 100107(2023).
- Tsuji, K., et al. BMP2 activity, although dispensable for bone formation, is required for the initiation of fracture healing. Nat Genet. 38 (12), 1424-1429 (2006).
- Hadjidakis, D. J., Androulakis, I. I. Boneremodeling. Ann N Y Acad Sci. 1092, 385-396 (2006).
- Boivin, G., Meunier, P. J. The degree of mineralization of bone tissue measured by computerized quantitative contact microradiography. Calcified Tiss Int. 70 (6), 503-511 (2002).
- Boivin, G., et al. Influence of remodeling on the mineralization of bone tissue. Osteoporo Int. 20 (6), 1023-1026 (2009).
- Zanghellini, B., et al. Multimodal analysis and comparison of stoichiometric and structural characteristics of parosteal and conventional osteosarcoma with massive sclerosis in human bone. J Str Biol. 216 (3), 108106(2024).
- Trento, G., et al. formation around two titanium implant surfaces placed in bone defects with and without a bone substitute material: A histological, histomorphometric, and micro-computed tomography evaluation. Clin Implant Dent Relat Res. 22 (2), 177-185 (2020).
- Palmquist, A. A multiscale analytical approach to evaluate osseointegration. J Mater Sci Mater Med. 29 (5), 60(2018).
- Budán, F., et al. Novel radiomics evaluation of bone formation utilizing multimodal (SPECT/X-ray CT) in vivo imaging. PLoS ONE. 13 (9), e0204423(2018).
- Hulsart-Billström, G., et al. A surprisingly poor correlation between in vitro and in vivo testing of biomaterials for bone regeneration: results of a multicentre analysis. Eur Cells Mater. 31, 312-322 (2016).
- Cramer, E. E. A., Ito, K., Hofmann, S. Ex vivo bone models and their potential in preclinical evaluation. Curr Osteoporo Rep. 19 (1), 75-87 (2021).
- Chan, M. E., et al. A trabecular bone explant model of osteocyte-osteoblast co-culture for bone mechanobiology. Cell Mol Bioeng. 2 (3), 405-415 (2009).
- Doke, S. K., Dhawale, S. C. Alternatives to animal testing: A review. Saudi Pharma J. 23 (3), 223-229 (2015).
- Moussi, H., et al. New formulation of injectable and degradable calcium phosphate/silanized hyaluronic acid composite foam: Investigation in a rabbit model of long bone defect. SSRN. , (2024).
- Paré, A., et al. Standardized and axially vascularized calcium phosphate-based implants for segmental mandibular defects: A promising proof of concept. Acta Biomater. 154, 626-640 (2022).
- Roschger, P., Fratzl, P., Eschberger, J., Klaushofer, K. Validation of quantitative backscattered electron imaging for the measurement of mineral density distribution in human bone biopsies. Bone. 23 (4), 319-326 (1998).
- Roschger, P., Plenk, H., Klaushofer, K., Eschberger, J. A new scanning electron microscopy approach to the quantification of bone mineral distribution: backscattered electron image grey-levels correlated to calcium K alpha-line intensities. Scanning Micro. 9 (1), 75-86 (1995).
- Porter, A., et al. Quick and inexpensive paraffin-embedding method for dynamic bone formation analyses. Sci Rep. 7, 42505(2017).
- Goldschlager, T., Abdelkader, A., Kerr, J., Boundy, I., Jenkin, G. Undecalcified bone preparation for histology, histomorphometry and fluorochrome analysis. J Vis Exp. (35), e1707(2010).
- Kazanci, M., Roschger, P., Paschalis, E. P., Klaushofer, K., Fratzl, P. Bone osteonal tissues by Raman spectral mapping: Orientation-composition. J Str Biol. 156 (3), 489-496 (2006).
- Unal, M., Ahmed, R., Mahadevan-Jansen, A., Nyman, J. S. Compositional assessment of bone by Raman spectroscopy. Analyst. 146 (24), 7464-7490 (2021).
- Oliver, W. C., Pharr, G. M. An improved technique for determining hardness and elastic modulus using load and displacement sensing indentation experiments. J Mater Res. 7 (6), 1564-1583 (1992).
- Proffen, B. L., McElfresh, M., Fleming, B. C., Murray, M. M. A comparative anatomical study of the human knee and six animal species. Knee. 19 (4), 493-499 (2012).
- Allen, M. J., Houlton, J. E. F., Adams, S. B., Rushton, N. The surgical anatomy of the stifle joint in Sheep. Vet Surg. 27 (6), 596-605 (1998).
- Kleuskens, M. W. A., van Donkelaar, C. C., Kock, L. M., Janssen, R. P. A., Ito, K. An ex vivo human osteochondral culture model. J Ortho Res. 39 (4), 871-879 (2021).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten