Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Caractérisation complète de la minéralisation tissulaire dans un modèle ex vivo
Dans cet article
Résumé
Le protocole proposé implique une approche globale pour évaluer la formation osseuse dans le contexte de la régénération osseuse à l’aide d’analyses multimodales. Il vise à fournir des informations qualitatives et quantitatives sur la formation de nouveaux os, en améliorant la rigueur et la validité des investigations fondamentales et précliniques.
Résumé
La caractérisation extensive de la minéralisation tissulaire dans le contexte de la régénération osseuse représente un défi important, compte tenu des nombreuses modalités actuellement disponibles pour l’analyse. Ici, nous proposons un flux de travail pour une évaluation complète de la formation de nouveaux os à l’aide d’un explant ex vivo osseux de grand animal pertinent. Un défaut osseux (diamètre = 3,75 mm ; profondeur = 5,0 mm) est créé dans une tête fémorale de mouton explantée et injecté avec un substitut osseux macroporeux chargé d’un facteur de croissance pro-ostéogénique (protéine morphogénétique osseuse 2 - BMP2). Par la suite, l’explant est maintenu en culture pendant une période de 28 jours, ce qui permet la colonisation cellulaire et la formation osseuse ultérieure. Pour évaluer la qualité et la structure des tissus nouvellement minéralisés, les méthodes successives suivantes sont mises en place : (i) Caractérisation et images 3D haute résolution de l’ensemble de l’explant à l’aide de micro-CT, suivies d’analyses d’images d’apprentissage profond pour améliorer la discrimination des tissus minéralisés ; (ii) Nano-indentation pour déterminer les propriétés mécaniques du tissu nouvellement formé ; (iii) Examens histologiques, tels que l’hématoxyline/éosine/safran (HES), le trichrome de Goldner et le pentachrome de Movat, afin de fournir une évaluation qualitative du tissu minéralisé, notamment en ce qui concerne la visualisation de la barrière ostéoïde et la présence de cellules osseuses ; (iv) cartographie par microscopie électronique à balayage (MEB) à rétrodiffusion avec référence interne pour quantifier le degré de minéralisation et fournir des informations détaillées sur la morphologie de surface, la composition minérale et l’interface os-biomatériau ; (v) Spectroscopie Raman pour caractériser la composition moléculaire de la matrice minéralisée et fournir des informations sur la persistance de BMP2 dans le ciment grâce à la détection de liaisons peptidiques. Cette analyse multimodale fournira une évaluation efficace de l’os nouvellement formé et des informations qualitatives et quantitatives complètes sur les tissus minéralisés. Grâce à la normalisation de ces protocoles, nous visons à faciliter les comparaisons entre les études et à améliorer la validité et la fiabilité des résultats de recherche.
Introduction
Les défauts osseux, qu’ils soient causés par un traumatisme, une résection tumorale, des anomalies congénitales ou une infection, représentent un défi majeur pour la médecine régénérative. Ces altérations compromettent l’intégrité structurelle du système squelettique, entraînant une gêne, une déficience fonctionnelle et une réduction de la qualité de vie des patients.
Pour surmonter ces défis, des stratégies innovantes de réparation osseuse ont vu le jour, en mettant l’accent sur l’amélioration de l’ostéogenèse et de la régénération du tissu osseux. Ces approches incluent l’utilisation de substituts osseux implantables, injectables ou imprimables en 3D, qui peuvent être d’origine naturelle (par exemple, macromolécules bio-sourcées, hydroxyapatite d’origine animale) ou synthétique (par exemple, bioverres, phosphates de calcium)1. Pour améliorer leur faible capacité inhérente à guider et à stimuler la régénération osseuse, les substituts osseux peuvent être chargés de facteurs ostéoinducteurs, tels que les protéines morphogénétiques osseuses (BMP), pour favoriser la différenciation ostéogénique des cellules progénitrices et améliorer la formation osseuse2.
La formation osseuse repose sur la formation initiale d’une matrice de collagène, qui est ensuite minéralisée par des cristaux d’hydroxyapatite, renforçant ainsi la structure osseuse3. Ce processus confère une rigidité et une résistance spécifiques à l’os. La qualité du tissu minéralisé est intimement régie par ses attributs microstructuraux et son degré de minéralisation4. Cette qualité joue un rôle central dans la cicatrisation osseuse et la fonctionnalité de l’os régénéré5. Cependant, la caractérisation de la minéralisation osseuse demeure une tâche difficile en raison de la variabilité inhérente aux études multivariées 6,7,8.
De plus, les évaluations initiales de la biocompatibilité, de la cytocompatibilité et du potentiel de différenciation des substituts de greffe osseuse sont généralement effectuées in vitro. Cependant, les disparités méthodologiques empêchent la comparaison transparente des résultats. De plus, ces études in vitro ne capturent pas pleinement les interactions multicellulaires et le dialogue complexe entre les populations cellulaires, y compris les cellules de la moelle osseuse, qui sont essentielles à la régulation du processus de régénération osseuse9. Ce manque de représentation précise du microenvironnement osseux peut compromettre la précision des études précliniques ultérieures10.
Bien que les évaluations in vivo fournissent une représentation plus précise des contextes physiologiques, elles sont limitées par des considérations éthiques, logistiques et financières. Par conséquent, les évaluations ex vivo jouent un rôle pivot en tant qu’interface entre les études in vitro et in vivo, servant d’étape intermédiaire nécessaire avant de passer à des expériences sur des sujets vivants 11,12,13.
Dans ce contexte, la mise en œuvre de méthodologies de caractérisation complètes est nécessaire pour évaluer la qualité du tissu osseux régénéré et s’assurer de la pertinence de la stratégie avant de passer à un modèle préclinique. Par conséquent, nous proposons un protocole basé sur l’analyse d’un modèle d’explant utilisant le tissu articulaire du genou de mouton. Cette méthodologie innovante consiste à implanter du ciment chargé en BMP2 dans les explants et à effectuer une analyse détaillée de la minéralisation tissulaire après 28 jours de culture.
Les approches techniques employées dans cette étude sont diverses et complémentaires, fournissant collectivement une approche globale pour évaluer la qualité du tissu osseux régénéré (Figure 1). L’imagerie micro-CT à haute résolution permet une visualisation 3D détaillée de la structure osseuse, fournissant des informations précieuses sur la densité minérale, la morphologie et l’intégrité du tissu nouvellement formé. Cette technique est cruciale pour évaluer l’efficacité de la régénération osseuse et suivre la progression de la minéralisation dans le temps. La nanoindentation est une approche précise pour déterminer les propriétés mécaniques du tissu, telles que sa dureté et sa résistance. En mesurant la réponse du matériau à une force appliquée à l’échelle nanométrique, cette méthode permet d’évaluer la robustesse et la qualité du tissu minéralisé. Les examens histologiques utilisant des colorants courants tels que l’hématoxyline/éosine/safran (HES), le trichrome de Goldner et le pentachrome de Movat fournissent des informations inestimables sur la structure et la composition des tissus. Ces colorations permettent de différencier les différents composants tissulaires, y compris les cellules, la matrice extracellulaire et les dépôts minéraux, permettant ainsi une évaluation qualitative complète du processus de régénération osseuse. La cartographie par microscopie électronique à balayage (MEB) à rétrodiffusion offre une visualisation à haute résolution de la surface des échantillons, permettant une analyse détaillée du degré de minéralisation de la matrice osseuse, ainsi que des interfaces entre le matériau implanté et le tissu hôte. Enfin, la spectroscopie Raman fournit des informations sur la composition moléculaire du tissu, notamment grâce à l’identification de composants spécifiques tels que les protéines, les lipides et les minéraux. Cette approche permet de caractériser la matrice minéralisée et de détecter des facteurs de croissance tels que BMP2, fournissant ainsi des informations cruciales sur la persistance des stimuli pro-ostéogéniques dans le milieu de régénération.
En utilisant une approche multidisciplinaire, intégrant diverses techniques analytiques, notre étude vise à fournir une évaluation approfondie et exhaustive de la qualité du tissu osseux régénéré, fournissant ainsi une base solide pour l’évaluation des substituts de greffe osseuse et leur application clinique potentielle.
Access restricted. Please log in or start a trial to view this content.
Protocole
Cette étude a été approuvée par une commission d’éthique et de bien-être animal et par l’Administration nationale vétérinaire et alimentaire sous le numéro G44171.
1. Préparation et culture d’explants ostéochondrales
- Récoltez des spécimens d’étuis d’animaux fraîchement euthanasiés dans un environnement aseptique. Positionnez le mouton en position couchée et rasez le membre postérieur gauche. Préparez en désinfectant avec de l’alcool autour de l’articulation du genou. Utiliser l’arthrotomie parapatellaire latérale pour exposer les ligaments croisés antérieur et postérieur, suivie d’une luxation rotulienne pour révéler la trochlée fémorale.
- Après le prélèvement tissulaire, créez un défaut de 4,75 mm de diamètre dans le condyle latéral à l’aide d’un système d’autogreffe ostéochondrale.
- À l’aide d’un marteau orthopédique, créez un défaut de 10 mm de profondeur et récupérez l’explant du condyle. Par la suite, effectuez un deuxième défaut de 8 mm de diamètre, centré autour du premier, à l’aide d’un système d’autogreffe ostéochondrale. Cela donne des explants ostéochondrales mesurant 8 mm x 10 mm, contenant un défaut central de 4,75 mm x 10,0 mm.
- Si nécessaire pour des raisons logistiques, transportez les explants dans la solution saline équilibrée de Hank (hBSS) complétée par 1 % de pénicilline-streptomycine et conservez-les à 4 °C.
- Rincer les explants 3 fois avec une solution saline tamponnée au phosphate (PBS), puis injecter le défaut de 4,75 mm x 10,0 mm avec un ciment de phosphate de calcium complété par 40 μg/mL de BMP2. Préparez le ciment comme décrit ci-dessous.
- Produire du phosphate α-tricalcique (α-TCP) par traitement thermique de bâtonnets de phosphate tricalcique apatitique (presse isostatique à 120 MPa) à 1364 °C pendant au moins 12 h, suivi d’une trempe à l’air14.
- Broyer les tiges de α-TCP dans de l’éthanol absolu avec une bille de broyeur planétaire 2x pendant 5 min à 500 tr/min pour produire une poudre fine de 13,1 ± 1,7 μm (mesurée par diffraction laser, comme décrit précédemment14).
- Stérilisez la poudre de α-TCP en la chauffant à sec à 180 °C pendant 45 min. Préparez le ciment de phosphate de calcium en mélangeant la poudre de α-TCP avec une solution filtrée à 2,5 % (p/v) de Na2HPO4 0,22 μm (rapport liquide/poudre de 0,35) pendant 1 min.
- Chargez la pâte de ciment dans une seringue de 3 ml avant l’injection à l’aide d’une aiguille de 18 G.
- Après 10 min à température ambiante (RT), transvaser les explants dans une fiole de 25 cm² et ajouter délicatement 10 ml de milieu de culture complet composé de Dulbecco’s Modified Eagle Medium (DMEM) à haute teneur en glucose complété par 10 % de sérum de bovin fœtal et 1 % de pénicilline-streptomycine.
- Conservez les explants jusqu’à 28 jours dans un incubateur à 37 °C avec 5% de CO2 et changez le milieu de culture complet tous les 2 jours sous une hotte à flux laminaire.
- Rincez les explants 3 fois avec du PBS. Transférez-les dans des tubes de 50 ml, ajoutez 20 ml de paraformaldéhyde (PFA) à 4 % et conservez-les à 4 °C pendant 3 jours. Ensuite, rincez les explants avec du PBS et stockez-les dans de l’éthanol à 70 % avant les analyses ultérieures.
2. Analyse par micro-tomodensitométrie
- Effectuer des analyses de microtomographie à l’aide d’une radiographie micro-CT. Acquérir des projections de rayons X à une résolution de 10,7 μm, avec un temps d’exposition de 1200 ms et un filtre en aluminium de 1 mm (80 kV et 125 μA). Faites la moyenne de trois images pour chaque incrément de rotation de 0,45° afin d’améliorer le rapport signal/bruit.
- Reconstruisez des images tridimensionnelles (3D) à l’aide du logiciel de reconstruction du fabricant après un alignement X/Y avec un balayage de référence, avec les paramètres suivants - Lissage : 0, Artefact annulaire : 3, Durcissement du faisceau : 35%. Nettoyez les piles d’images avec un logiciel de traitement d’images et observez avec des outils de visualisation bidimensionnelle (2D) et 3D.
3. Analyses d’images par apprentissage profond
- Effectuez la segmentation des images à l’aide d’un logiciel spécialisé dédié. Utilisez l’assistant de segmentation intégré pour entraîner un modèle d’apprentissage profond pour la discrimination des os et du ciment.
- Sélectionnez une zone représentative (c’est-à-dire une image) contenant de l’os, du ciment et l’arrière-plan à partir des images micro-CT reconstruites. Segmentez cette première image manuellement à l’aide d’outils simples (par exemple, seuil) ou puissants (par exemple, propagation) fournis par le logiciel.
- Générez un modèle de Deep Learning dans l’onglet Modèle et sélectionnez Routine 3D-Unet dans le menu déroulant. Définir ses paramètres expérimentaux en adéquation avec les images à analyser par un clic droit sur le modèle généré ; ici : profondeur de 5, taille du patch 32 x 32, algorithme Adadetla, rapport de foulée de 0,25, augmentation des données x10.
- Utilisez le cadre segmenté pour entraîner la routine d’apprentissage profond (cliquez sur le bouton Entraîner), qui est reconnue comme pertinente pour ses capacités de segmentation d’image rapides et précises 3,15. Une fois l’entraînement terminé, définissez une deuxième zone de travail (cadre) et segmentez automatiquement à l’aide de la fonction predict.
- Appliquez une correction manuelle si nécessaire pour générer des données d’entraînement plus précises. Entraînez-vous à nouveau avec la routine. Répétez le processus jusqu’à ce qu’un résultat satisfaisant soit obtenu ; Le nombre d’augmentations de données diminue progressivement à mesure que le nombre de cadres d’entraînement augmente.
REMARQUE : L’indice DICE fourni lors de l’entraînement du modèle donne une indication de la précision du modèle (par rapport aux informations d’entrée) mais n’est pas suffisant. La confirmation de la pertinence du modèle devrait être effectuée par la validation des données segmentées automatiquement par 3 chercheurs indépendants et chevronnés.
- Appliquez une correction manuelle si nécessaire pour générer des données d’entraînement plus précises. Entraînez-vous à nouveau avec la routine. Répétez le processus jusqu’à ce qu’un résultat satisfaisant soit obtenu ; Le nombre d’augmentations de données diminue progressivement à mesure que le nombre de cadres d’entraînement augmente.
- Publiez le modèle en cliquant sur le bouton Exporter et appliquez-le à l’ensemble du jeu de données micro-CT en cliquant sur Segment > Modèle exporté > Segment complet du jeu de données.
4. Intégration
- Déshydratez les explants osseux en les plaçant dans un flacon en verre de 40 mL contenant 25 mL de solution de déshydratation composée de 70 % d’acétone et de 30 % de xylène. Placez le flacon en verre sur une roue tournante pendant 1 h à RT. Répétez cette étape 3 fois.
- Remplacer la solution de déshydratation par 25 mL de xylène et placer le flacon en verre sur la roue rotative pendant 1 h à RT.
- Remplacer la solution de xylène par 25 mL de méthacrylate de méthyle enrichi (MMA) avec 10 % de peroxyde de benzoyle (BPO) et 10 % de phtalate de dibutyle (DBP). Placez-le sur la roue tournante pendant 1 h à RT. Répétez cette étape 2 fois.
- Remplacer la solution de MMA par 25 mL de MMA enrichi de 10 % de BPO, de 10 % de DBP et de 450 μL de N, N diméthylaniline diluée 1:20 dans du propan-2-ol. Placez le flacon en verre à -20 °C pendant la nuit. La solution devient jaunâtre.
- Placez l’explant dans un moule d’enrobage de taille moyenne et versez le MMA-BPO-DBP-N, N aniline dans le moule. Placez le moule dans une boîte en plastique et aérez avec un flux d’azote pendant 5 min. Fermez hermétiquement la boîte et placez-la à 4 °C pendant 48 h pour la polymérisation et le durcissement du MMA.
- Retirez la résine contenant l’explant du moule et maintenez-la à 4 °C jusqu’à ce qu’elle soit traitée pour une analyse ultérieure.
5. Microscopie électronique à balayage (MEB) - imagerie électronique quantitative rétrodiffusée (qBEI)
- Coupez le bloc pMMA contenant l’explant à l’aide d’une scie diamantée le long de son axe long. Conservez la première moitié du bloc pour l’analyse histologique. Coupez l’autre moitié pour obtenir une section de 1,5 mm d’épaisseur. Effectuez la coupe à 3000 tr/min avec une vitesse de 3 mm/min.
- Meulez la section avec du papier carbure de silicium avec des nombres croissants correspondant à la granulométrie inférieure : SiC 320 pendant 10 s, SiC 1000 pendant 15 s, SiC 2000 pendant 30 s et SiC 4000 pendant 30 s.
- Polir la section avec de la pâte diamantée et des vêtements spécifiques : chiffon de polissage avec 3 μm de solution de diamant Mol B3 pendant 1 min et chiffon polaire avec 1 μm de solution de diamant Nap B1 pendant 1 min.
- Rincez la section sous de l’eau distillée deux fois et nettoyez les particules de diamant avec un coton-tige. Séchez la section sous azote gazeux.
- Du carbone enduit la section épaisse (épaisseur du film de carbone de 10 nm) et montez-la sur un talon d’aluminium avec de la peinture argentée. Faites un pont de peinture argentée entre le haut de la section et le talon pour permettre l’évacuation de la charge d’électrons vers le sol.
- Placez le talon sur la platine SEM. À côté de l’échantillon, insérez un talon de contrôle composé d’une coupelle de Faraday avec des étalons de carbone, d’aluminium et de silicium dans la chambre MEB. Cela sera utilisé pour calibrer le faisceau d’électrons et convertir les niveaux de gris en pourcentages de Ca. Fermez la chambre MEB et faites le vide.
- Activez le faisceau d’électrons et ajustez les paramètres MEB pour qu’il fonctionne en mode électrons rétrodiffusés. Sur les images rétrodiffusées, le niveau de gris de la norme carbone est de 25, celui de l’aluminium est de 225 et celui du silicium est de 253. À titre indicatif, sur le système utilisé, les paramètres MEB sont la tension d’accélération 20 keV, le courant de sonde mesuré avec la coupelle de Faraday : 250 pA, la distance de travail : 15 mm, l’ouverture : 30 μm, la résolution d’image : 1024 x 768 pixels, le vide : < 4,10-4 Pa, le temps de séjour de 100 μs/pixel, la luminosité ~38 et le contraste ~72.
- Lorsque le MEB est étalonné avec les étalons, acquérir des images de l’échantillon en mode électron rétrodiffusé.
- Utilisez l’image électronique rétrodiffusée de l’échantillon pour convertir les niveaux de gris en teneur en calcium, comme décrit dans Roschger et al.16. Pour ce faire, utilisez n’importe quel logiciel d’analyse d’images.
- Tracez la distribution de la teneur en calcium. La distribution de la teneur en calcium montre une distribution gaussienne. Trois paramètres principaux d’intérêt sont calculés : lamoyenne Ca, comme la teneur moyenne en calcium sur l’image, lepic Ca comme la concentration de calcium la plus fréquente rencontrée sur l’image et la largeur Ca comme lalargeur totale à la moitié de la distribution de la teneur en calcium.
REMARQUE : La reproductibilité de qBEi a été étudiée en imageant la même région d’intérêt pendant 5 jours consécutifs (une acquisition par jour). Cela signifie que le faisceau d’électrons a été activé et désactivé entre les sessions. Le pourcentage d’erreur dans lamoyenne Ca, lepic Ca et la largeur Cawidth a été estimé à 0,5 %, 0,7 % et 1,2 %, respectivement. La teneur en calcium a également été étudiée par analyse par rayons X à dispersion d’énergie (EDS). Une très bonne relation linéaire a été établie entre lamoyenne Ca et la teneur en calcium estimée par EDS avec une valeur R² de 0,99, similaire aux données obtenues par Roschger et al. précédemment17.
6. Histologie
- Pour chaque échantillon, coupez des sections de 7 μm à l’aide d’un microtome à tissu dur avec une lame en tungstène 18,19. Sections de teinture avec coloration trichrome Goldner, HES et MOVAT à l’aide d’un système de coloration automatique.
- Pour la déplastification, trempez successivement les échantillons dans de l’acétone pendant 5 min et répétez l’opération 2x. Rincez les échantillons à l’eau distillée pendant 5 minutes et répétez l’opération 2 fois.
- Effectuez la coloration trichrome Goldner comme décrit ci-dessous.
- Placer les échantillons dans de l’hématoxyline ferrique Weigert pendant 20 min. Laver à l’eau du robinet. Immerger les échantillons dans de l’alcool acide à 0,5 % pendant 30 s-1 min. Laver à l’eau du robinet pendant 20 min.
- Placer les échantillons dans une solution de Ponceau/acide fuchsine/azophloxine pendant 5 min. Immerger dans de l’acide acétique à 1 % pendant 1 min.
- Colorer avec une solution d’acide phosphomolybdique/Orange G pendant 20 min. Immerger dans de l’acide acétique à 1 % pendant 1 min.
- Colorer avec une solution verte rapide pendant 15 min à température ambiante ou 8 min à 60 °C. Rincez abondamment à l’eau du robinet ; Répétez 3 fois.
- Effectuez la coloration Movat Pentachrome comme décrit ci-dessous.
- Coloration au bleu d’Alcian : immerger les échantillons dans la solution de bleu d’Alcian pendant 30 min. Rincer les échantillons à l’eau du robinet pendant 5 min. Placer les échantillons dans de l’éthanol alcalin pendant 60 min. Rincer les échantillons à l’eau du robinet pendant 10 min. Effectuez un dernier rinçage à l’eau distillée.
- Coloration à l’hématoxyline de Weigert : placer les échantillons dans l’hématoxyline ferrique de Weigert pendant 20 à 30 min. Rincer les échantillons à l’eau du robinet pendant 15 min. Rincez à nouveau à l’eau distillée.
- Coloration à l’acide Crocein Brilliant/fuchsine : immerger les échantillons dans une solution de Crocein Brilliant/Acid Fuchsin pendant 10 min. Rincer avec de l’acide acétique à 0,5 %. Immerger les échantillons dans une solution d’acide phosphotungstique à 5 % pendant 20 min. Rincer avec de l’acide acétique à 0,5 % pendant 2 min. Effectuez trois rinçages successifs à 100 % d’éthanol, d’une durée de 5 min chacun.
- Coloration au Safran du Gatinais : placer les échantillons dans le Safran du Gatinais pendant 15 min. Effectuez un dernier rinçage à l’eau du robinet.
- Effectuez la coloration HES comme décrit ci-dessous.
- Coloration à l’hématoxyline de Weigert : immerger les échantillons dans l’hématoxyline de Weigert pendant 30 min. Rincer à l’eau du robinet pendant 2 min.
- Décoloration : tremper les échantillons dans de l’alcool à l’acide chlorhydrique pendant 10 s. Rincer à l’eau du robinet pendant 2 min.
- Neutralisation : immerger les échantillons dans une solution de carbonate de lithium pendant 1 min. Rincer à l’eau du robinet pendant 2 min. Effectuez un dernier rinçage à l’eau distillée pendant 1 min.
- Coloration à l’éosine-érythrosine : placer les échantillons dans une solution d’éosine-érythrosine pendant 3 min. Rincer à l’eau du robinet pendant 10 s.
- Déshydratation : Immerger les échantillons dans de l’alcool à 95 % pendant 15 s. Transfert dans 100% d’alcool pendant 15 s. Répétez l’immersion dans de l’alcool à 100 % pendant 30 s.
- Coloration finale au safranin alcoolique : immerger les échantillons dans du safranin alcoolique pendant 10 min. Effectuez un dernier rinçage à l’eau du robinet.
- Effectuez le montage de la glissière comme décrit ci-dessous.
- Rincez les échantillons avec de l’éthanol à 95 %, puis de l’éthanol à 100 %, puis du méthylcyclohexane et répétez cette opération 3 fois pour chaque étape.
- Montez les échantillons à l’aide d’un kit de montage et ajoutez-y une lamelle. Numérisez les images à l’aide d’un scanner de diapositives numérique.
7. Microspectroscopie Raman
- Utilisez la même section que celle utilisée pour l’analyse Raman que celle du MEB. Broyez et polissez brièvement la section comme décrit aux étapes 5.2 et 5.3 pour enlever la fine couche de carbone ajoutée pour le MEB qui nuit à la dissipation de la chaleur de l’échantillon.
- Placez la section sur la platine du microspectromètre Raman. Calibrez le numéro d’onde pour assurer la précision et alignez le laser avant la mesure. Ces procédures étant propres à chaque instrument, elles ne seront pas décrites dans cet article.
- Localisez les régions d’intérêt dans le tissu qui doivent être analysées. Positionnez la zone à analyser au centre de l’outil vidéo et collectez les spectres Raman avec les paramètres suivants : laser 785 nm utilisé à 30 mW, temps d’intégration : 20 s répété 3 fois, plage spectrale : 350-1800 cm-1, réseau 1200 lignes/mm.
- Collectez 10 spectres de la résine d’enrobage avec les mêmes paramètres Raman et faites-en la moyenne. Utilisez-le pour soustraire la contribution de la résine à l’étape 7.6.
- Pré-traitez les spectres comme suit pour éliminer la contribution de la fluorescence, du bruit et de la résine : correction de base avec un ajustement polynomial à cinq ordres, filtre Savitzky-Golay avec un degré de 4 et une taille de fenêtre comprise entre 17 et 21, soustraction numérique de résine en utilisant le pic de ~812 cm-1 de la résine. Un tel prétraitement peut être effectué à l’aide de différents logiciels conçus pour l’analyse et la visualisation des données.
- Analysez davantage les spectres prétraités pour en extraire des paramètres précieux en rationnant l’intensité maximale de la vibration d’intérêt avec l’intensité maximale de la vibration de référence. Pour l’analyse osseuse, par exemple, utilisez les paramètres suivants pour le calculer.
- Rapports minéral/matrice : divisez l’intensité maximale des vibrations de v1PO4 à ~960 cm-1 par l’intensité maximale des modes vibratoires Amide I (~1668 cm-1), amide III (~1250 cm-1), CH2-wag (~1450 cm-1) ou la somme de la proline (~854 cm-1) et de l’hydroxyproline (Hyp, ~872 cm-1). Le v2PO4, situé à ~430 cm-1, ou le v4PO4, situé à ~600 cm-1, peuvent également être utilisés et rapportés avec la vibration Amide III20. Ces paramètres représentent le degré de minéralisation de la phase organique de la matrice osseuse.
- Carbonate/Phosphate : divisez l’intensité maximale du v1CO3, situé à ~1070 cm-1, par le v1PO4. Cela représente la quantité de substitution de carbonate de type B dans le réseau d’apatite. Hyp/Pro, représente la teneur en hydroxyproline, ~1375 cm-1/Amide III, représente la teneur en protéoglycanes, ~1150 cm-1/CH2-wag et ~1495 cm-1/CH2-wag, représente la quantité des produits finaux de glycation avancée carboxyméthylysine et pentosidine, 1670 cm-1/1690 cm-1 et 1670 cm-1/1640 cm-1 représentent la structure secondaire désordonnée du collagène, également connue sous le nom de maturité du collagène, et les structures ordonnées sous forme d’hélice α dans le collagène de type I, respectivement.
- Evaluez la reproductibilité des mesures Raman en examinant la même région d’intérêt dans 5 acquisitions consécutives (une acquisition par jour). Pour chaque paramètre analysé, la variance était inférieure à 0,2 % de la valeur du paramètre, ce qui favorise une bonne reproductibilité. La spécificité de chaque pic étudié a été largement discutée sur le terrain, et une vue d’ensemble est présentée dans la récente revue d’Unal21.
8. Nanoindentation
REMARQUE : En raison de la nature destructrice de la nanoindentation, elle est généralement effectuée à la fin de la routine d’analyse de l’échantillon. Le système de nanoindentation que nous possédons est équipé d’un pénétrateur pyramidal en diamant Berkovitch. Cependant, il existe plusieurs formes de pénétrateur, et aucun consensus dans la littérature n’a été déterminé pour les échantillons d’os ou de biomatériaux.
- Hydrater l’échantillon d’os incrusté dans une solution saline pendant 16 h à RT avant le test de nanoindentation.
- Calibrez le système de nanoindentation à l’aide de silice fondue et enregistrez le module d’indentation résultant. Pour la silice fondue, le module d’indentation est d’environ 72 GPa avec un coefficient de Poisson de 0,17.
- Une fois le système de nanoindentation calibré, placez l’échantillon utilisé pour les analyses Raman dans le système optique du dispositif de nanoindentation pour indiquer les endroits où la nanoindentation sera effectuée.
REMARQUE : Cette modalité varie d’un équipement à l’autre ; Nous ne détaillons pas le fonctionnement du système de nanoindentation et du logiciel, mais donnons des conseils généraux sur les différentes étapes à effectuer. - Une fois l’emplacement de la nanoindentation choisi, déplacez l’échantillon sous le dispositif d’indentation et effectuez l’indentation. Effectuez une nanoindentation à une profondeur constante de 900 nm, avec une vitesse de chargement/déchargement de 40 mm/min et une pause de 15 s entre le chargement et le déchargement. Réglez le coefficient de Poisson pour le tissu osseux à 0,3.
- Récupérez les différents paramètres des courbes de nanoindentation et calculez-les selon la théorie d’Oliver et Pharr22.
- Le module d’indentation (EIT) est dérivé des propriétés connues de la pointe d’indentation et du module réduit et combine le module d’élasticité local de l’éprouvette. Calculez l’indentation comme suit :
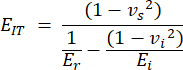
Avec vs, vi, Ei et Er représentant le coefficient de Poisson de l’échantillon, supposé être de 0,3 pour l’os, le coefficient de Poisson du pénétrateur diamant, supposé être de 0,07, le module d’élasticité du pénétrateur diamanté, fixé à 1140 GPa et le module réduit, calculé comme suit :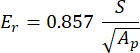
Avec S représentant la pente du segment de déchargement et Ap étant la surface projetée et calculée comme suit :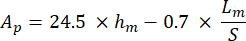
- Dureté (HIT) : correspondant à la résistance de l’os à l’initiation et à la propagation des fissures. En d’autres termes, il s’agit d’un indicateur de la résistance des os. Calculez HIT comme suit :

- Taux de fluage par indentation (CIT) : Il reflète la déformation osseuse au fil du temps à charge constante, indicatif des propriétés viscoélastiques de l’échantillon, calculé comme suit :
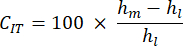
- Charge maximale (Lm) : Elle correspond à la charge opposée nécessaire pour pénétrer à la profondeur indiquée dans la matrice osseuse minéralisée.
- Travaux d’indentation (Wplast) : Correspond à la zone sous la courbe de déformation charge/décharge. Il s’agit d’un reflet direct de l’énergie dissipée pour induire une déformation plastique.
REMARQUE : La reproductibilité de la nanoindentation est plus difficile à évaluer sur des échantillons d’os en raison de la nature destructrice de cette méthodologie. Néanmoins, nous avons estimé la reproductibilité de l’échantillon de silice fondue standard et uniforme. Pour tous les paramètres ci-dessus, la variance a été estimée en dessous de 0,26 % (5 mesures consécutives sur 5 jours distincts), ce qui soutient une très bonne reproductibilité. D’autre part, en raison de la nature anisotrope des échantillons osseux, il est plus difficile d’apprécier leur reproductibilité.
- Le module d’indentation (EIT) est dérivé des propriétés connues de la pointe d’indentation et du module réduit et combine le module d’élasticité local de l’éprouvette. Calculez l’indentation comme suit :
Access restricted. Please log in or start a trial to view this content.
Résultats
Une image micro-CT de l’explant est présentée à la figure 2. La segmentation manuelle ne permet pas de séparer de manière optimale l’os du ciment, présent dans le canal central, à l’aide du seuillage global. Pour améliorer la reconnaissance de l’os trabéculaire et du ciment, nous proposons d’utiliser l’apprentissage profond. L’apprentissage profond est puissant pour reconnaître les caractéristiques des biomatériaux et contribue à a...
Access restricted. Please log in or start a trial to view this content.
Discussion
La réparation des défauts osseux est un défi majeur en médecine régénérative pour restaurer la mobilité, réduire la douleur et améliorer la qualité de vie des personnes touchées. L’utilisation de modèles d’explants offre un certain nombre d’avantages par rapport aux études in vivo pour l’étude de la réparation des défauts osseux. Outre les considérations éthiques, ce modèle permet un contrôle rigoureux des conditions expérimentales et la réduction ...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts à divulguer.
Remerciements
Nous tenons à remercier les structures techniques impliquées dans la collecte et le traitement des échantillons, notamment SC3M (SFR François Bonamy (UMS 016), Université de Nantes), SFR ICAT (Université d’Angers), BIO3, HiMolA et SC4BIO. L’Inserm UMR_S 1229 RMeS est soutenu par des subventions de l’État français à travers l’Inserm, Nantes Université, l’Université d’Angers et les établissements Oniris VetAgroBio. CL est également reconnaissant envers HTL Biotechnology.
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| 0.20 filters | VWR | 28145-501 | |
| 18 G needle (1,2x40 mm) | Sterican | 4665120 | |
| 3 mL syringe | HENKE-JECT | 8300005762 | |
| 37% hydrochloric acid | VWR | 1.00317.1000 | |
| Acetic acid (glacial) | Sigma | A6283 | |
| Acetone | VWR | 20063-365 | |
| Alcian Blue 8GX | VWR | 361186 | |
| Ammonium hydroxide | VWR | 318612 | |
| Apatitic tricalcium phosphate | Centre for Biomedical and Healthcare Engineering (Mines Saint Etienne, France) | TV26U | |
| Azophloxine | Sigma | 210633 | |
| Benzoyl peroxide | Sigma | 8.01641.0250 | |
| BMP2 | Medtronic | InductOs 1.5 mg/mL | |
| Brillant crocein | Aldrich | 2107507 | |
| CTVox | Bruker | - | |
| DataViewer | Skyscan | - | |
| Diamond blade | Struers | MOD13 | |
| Diamond saw | Struers | Accutom-50 | |
| DiaPro Mol B3 diamond solution | Struers | 40600379 | |
| DiaPro Nap B1 diamond solution | Struers | 40600373 | |
| Dibasic sodium phosphate (Na2HPO4) | Sigma | 102404598 | |
| Dibutyl Phtalate | Chimie-Plus Laboratoires | 28656 | |
| DragonFly software | ORS | 2022.1.0.1231. | |
| Dulbecco's Modified Eagle Medium (DMEM) high glucose, GlutaMAX(TM), pyruvate | ThermoFisher Scientific | 31966-021 | |
| Eosine Y- Surgipath | Sigma | 1002830105 | |
| Erythrosin B | Sigma | 102141057 | |
| Ethanol absolute | VWR | 20820362 | |
| Eukitt | Dutscher | 6.00.01.0003.06.01.01 | |
| Falcon 50 mL | Sarstedt | 62.547.254 | |
| Ferric chloride hexahydrate (FeCl3, 6H2O) | Merck | 1.03943.0250 | |
| Fetal Bovine Serum (FBS) | Eurobio | CVFSVF00 | |
| Fuchsine acid | Merck | 1.05231.0025 | |
| Hank's Balanced Salt Solution (HBSS) | Biosera | MS01NG100J | |
| Hematoxylin | Sigma | 86.118.9 | |
| Isostatic press | Nova Suisse | Pmax 1500 bars | |
| Laser diffraction granulometry | Malvern | Mastersizer 3000 | |
| Light green | Prolabo | 28947135 | |
| Lithium carbonate | Sigma | A13149 | |
| MD-Mol polishing cloth | Struers | 40500077 | |
| Methylcyclohexane | VWR | 8.06147.1000 | |
| Methylcyclohexane | VWR | 8.06147.1000 | |
| Methylcyclohexane | VWR | 8.06147.1000 | |
| Methylmethacrylate | Sigma | 8.00590.2500 | |
| Micro-CT, micro-scanner | Bruker | Skyscan 1272 | |
| Monobasic sodium phosphate (NAH2PO4) | Sigma | 71496 | |
| Mortar | Fritsch | Pulverisette 6 | |
| N,N, Dimethylanilin | Sigma | 803060 | |
| Nanoindentation station | Anton Paar | NHT2 | |
| ND-Nap polishing cloth | Struers | 40500080 | |
| OATS Osteochondral Autograft Transfer System Set, 4,75 mm | Arthrex | AR-1981-04S | |
| OATS Osteochondral Autograft Transfer System Set, 8 mm | Arthrex | AR-1981-08S | |
| Orange G | Ral | M15 | |
| Paraformaldehyde (PFA) | Sigma | P6148 | |
| Peel-a-way disposable embbedding moulds | Polysciences, Inc | 18646C-1 | |
| Penicillin/Streptomycin (P/S) | ThermoFisher Scientific | 15140122 | |
| Phosphate Buffered Saline (PBS) | ThermoFisher Scientific | 10010023 | |
| Phosphomolybdic acid | Sigma | 221856-100 g | |
| Phosphotungstic acid | Aldrich | 12863-5 | |
| Polishing machine | Sturers | Dap V | |
| Poupinel | MEMMERT | TV26U | |
| Raman microspectrometer | Renishaw | InVia Qontor | |
| Safran du Gâtinais | Labonord | 11507737 | |
| Scanning electron microscope | Carl Zeiss | Evo LS 10 | |
| SEM | Zeiss | Carl Zeiss Evo LS10 | |
| SiC foils/Grinding papers | Struers | 40400008 (#320), 40400011 (#1000), 40400122 (#2000), 40400182 (#4000) | |
| Silver paint | Electron microscopy sciences | 12686-15 | |
| Standard stub with Faraday cup, carbon, aluminium and silicon standards | Micro-Analysis Consultants Ltd | 8602 | |
| T25 flask | Corning | 430639 | |
| Xylene | VWR | 28975.325 | |
| Xylidine Ponceau | Aldrich | 19.976-1 |
Références
- Feroz, S., Cathro, P., Ivanovski, S., Muhammad, N. Biomimetic bone grafts and substitutes: A review of recent advancements and applications. Biomed Eng Adv. 6, 100107(2023).
- Tsuji, K., et al. BMP2 activity, although dispensable for bone formation, is required for the initiation of fracture healing. Nat Genet. 38 (12), 1424-1429 (2006).
- Hadjidakis, D. J., Androulakis, I. I. Boneremodeling. Ann N Y Acad Sci. 1092, 385-396 (2006).
- Boivin, G., Meunier, P. J. The degree of mineralization of bone tissue measured by computerized quantitative contact microradiography. Calcified Tiss Int. 70 (6), 503-511 (2002).
- Boivin, G., et al. Influence of remodeling on the mineralization of bone tissue. Osteoporo Int. 20 (6), 1023-1026 (2009).
- Zanghellini, B., et al. Multimodal analysis and comparison of stoichiometric and structural characteristics of parosteal and conventional osteosarcoma with massive sclerosis in human bone. J Str Biol. 216 (3), 108106(2024).
- Trento, G., et al. formation around two titanium implant surfaces placed in bone defects with and without a bone substitute material: A histological, histomorphometric, and micro-computed tomography evaluation. Clin Implant Dent Relat Res. 22 (2), 177-185 (2020).
- Palmquist, A. A multiscale analytical approach to evaluate osseointegration. J Mater Sci Mater Med. 29 (5), 60(2018).
- Budán, F., et al. Novel radiomics evaluation of bone formation utilizing multimodal (SPECT/X-ray CT) in vivo imaging. PLoS ONE. 13 (9), e0204423(2018).
- Hulsart-Billström, G., et al. A surprisingly poor correlation between in vitro and in vivo testing of biomaterials for bone regeneration: results of a multicentre analysis. Eur Cells Mater. 31, 312-322 (2016).
- Cramer, E. E. A., Ito, K., Hofmann, S. Ex vivo bone models and their potential in preclinical evaluation. Curr Osteoporo Rep. 19 (1), 75-87 (2021).
- Chan, M. E., et al. A trabecular bone explant model of osteocyte-osteoblast co-culture for bone mechanobiology. Cell Mol Bioeng. 2 (3), 405-415 (2009).
- Doke, S. K., Dhawale, S. C. Alternatives to animal testing: A review. Saudi Pharma J. 23 (3), 223-229 (2015).
- Moussi, H., et al. New formulation of injectable and degradable calcium phosphate/silanized hyaluronic acid composite foam: Investigation in a rabbit model of long bone defect. SSRN. , (2024).
- Paré, A., et al. Standardized and axially vascularized calcium phosphate-based implants for segmental mandibular defects: A promising proof of concept. Acta Biomater. 154, 626-640 (2022).
- Roschger, P., Fratzl, P., Eschberger, J., Klaushofer, K. Validation of quantitative backscattered electron imaging for the measurement of mineral density distribution in human bone biopsies. Bone. 23 (4), 319-326 (1998).
- Roschger, P., Plenk, H., Klaushofer, K., Eschberger, J. A new scanning electron microscopy approach to the quantification of bone mineral distribution: backscattered electron image grey-levels correlated to calcium K alpha-line intensities. Scanning Micro. 9 (1), 75-86 (1995).
- Porter, A., et al. Quick and inexpensive paraffin-embedding method for dynamic bone formation analyses. Sci Rep. 7, 42505(2017).
- Goldschlager, T., Abdelkader, A., Kerr, J., Boundy, I., Jenkin, G. Undecalcified bone preparation for histology, histomorphometry and fluorochrome analysis. J Vis Exp. (35), e1707(2010).
- Kazanci, M., Roschger, P., Paschalis, E. P., Klaushofer, K., Fratzl, P. Bone osteonal tissues by Raman spectral mapping: Orientation-composition. J Str Biol. 156 (3), 489-496 (2006).
- Unal, M., Ahmed, R., Mahadevan-Jansen, A., Nyman, J. S. Compositional assessment of bone by Raman spectroscopy. Analyst. 146 (24), 7464-7490 (2021).
- Oliver, W. C., Pharr, G. M. An improved technique for determining hardness and elastic modulus using load and displacement sensing indentation experiments. J Mater Res. 7 (6), 1564-1583 (1992).
- Proffen, B. L., McElfresh, M., Fleming, B. C., Murray, M. M. A comparative anatomical study of the human knee and six animal species. Knee. 19 (4), 493-499 (2012).
- Allen, M. J., Houlton, J. E. F., Adams, S. B., Rushton, N. The surgical anatomy of the stifle joint in Sheep. Vet Surg. 27 (6), 596-605 (1998).
- Kleuskens, M. W. A., van Donkelaar, C. C., Kock, L. M., Janssen, R. P. A., Ito, K. An ex vivo human osteochondral culture model. J Ortho Res. 39 (4), 871-879 (2021).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon