È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Caratterizzazione completa della mineralizzazione tissutale in un modello ex vivo
In questo articolo
Riepilogo
Il protocollo proposto prevede un approccio globale per valutare la formazione ossea nel contesto della rigenerazione ossea utilizzando analisi multimodali. Ha lo scopo di fornire informazioni qualitative e quantitative sulla formazione di nuovo osso, migliorando il rigore e la validità delle indagini di base e precliniche.
Abstract
L'ampia caratterizzazione della mineralizzazione tissutale nel contesto della rigenerazione ossea rappresenta una sfida significativa, date le numerose modalità attualmente disponibili per l'analisi. Qui, proponiamo un flusso di lavoro per una valutazione completa della nuova formazione ossea utilizzando un espiante osseo ex vivo di grandi dimensioni di grandi dimensioni. Un difetto osseo (diametro = 3,75 mm; profondità = 5,0 mm) viene creato in una testa femorale di pecora espiantata e iniettato con un sostituto osseo macroporoso caricato con un fattore di crescita pro-osteogenico (proteina morfogenetica ossea 2 - BMP2). Successivamente, l'espianto viene mantenuto in coltura per un periodo di 28 giorni, permettendo la colonizzazione cellulare e la successiva formazione ossea. Per valutare la qualità e la struttura del tessuto appena mineralizzato, vengono messi a punto i seguenti metodi: (i) Caratterizzazione e immagini 3D ad alta risoluzione dell'intero espianto mediante micro-CT, seguite da analisi di immagini con deep learning per migliorare la discriminazione dei tessuti mineralizzati; (ii) Nano-indentazione per determinare le proprietà meccaniche del tessuto di nuova formazione; (iii) Esami istologici, come l'ematossilina/eosina/zafferano (HES), il tricromo di Goldner e il pentacromo di Movat per fornire una valutazione qualitativa del tessuto mineralizzato, in particolare per quanto riguarda la visualizzazione della barriera osteoide e la presenza di cellule ossee; (iv) Mappatura al microscopio elettronico a scansione (SEM) a retrodiffusione con riferimento interno per quantificare il grado di mineralizzazione e fornire informazioni dettagliate sulla morfologia della superficie, sulla composizione minerale e sull'interfaccia osso-biomateriale; (v) Spettroscopia Raman per caratterizzare la composizione molecolare della matrice mineralizzata e per fornire informazioni sulla persistenza di BMP2 all'interno del cemento attraverso la rilevazione di legami peptidici. Questa analisi multimodale fornirà una valutazione efficace dell'osso di nuova formazione e una visione qualitativa e quantitativa completa dei tessuti mineralizzati. Attraverso la standardizzazione di questi protocolli, miriamo a facilitare i confronti tra studi e a migliorare la validità e l'affidabilità dei risultati della ricerca.
Introduzione
I difetti ossei, siano essi causati da traumi, resezione tumorale, anomalie congenite o infezioni, rappresentano una sfida importante per la medicina rigenerativa. Queste alterazioni compromettono l'integrità strutturale dell'apparato scheletrico, portando a disagio, compromissione funzionale e riduzione della qualità di vita dei pazienti.
Per superare queste sfide, sono emerse strategie innovative di riparazione ossea, con particolare attenzione al miglioramento dell'osteogenesi e della rigenerazione del tessuto osseo. Questi approcci includono l'uso di sostituti ossei impiantabili, iniettabili o stampabili in 3D, che possono essere di origine naturale (ad esempio, macromolecole di origine biologica, idrossiapatite di origine animale) o sintetici (ad esempio, bioocchiali, fosfati di calcio)1. Per migliorare la loro bassa capacità intrinseca di guidare e stimolare la rigenerazione ossea, i sostituti ossei possono essere caricati con fattori osteoinduttivi, come le proteine morfogenetiche ossee (BMP), per promuovere la differenziazione osteogenica delle cellule progenitrici e migliorare la formazione ossea2.
La formazione ossea si basa sulla formazione iniziale di una matrice di collagene, che viene poi mineralizzata da cristalli di idrossiapatite, rafforzando così la struttura ossea3. Questo processo conferisce rigidità e resistenza specifiche all'osso. La qualità del tessuto mineralizzato è governata in modo intricato dalle sue caratteristiche microstrutturali e dal grado di mineralizzazione4. Questa qualità svolge un ruolo fondamentale nella guarigione ossea e nella funzionalità dell'osso rigenerato5. Tuttavia, la caratterizzazione della mineralizzazione ossea rimane un compito impegnativo a causa della variabilità intrinseca negli studi multivariati 6,7,8.
Inoltre, le valutazioni iniziali della biocompatibilità, della citocompatibilità e del potenziale di differenziazione dei sostituti dell'innesto osseo sono tipicamente condotte in vitro. Tuttavia, le disparità metodologiche impediscono un confronto senza soluzione di continuità dei risultati. Inoltre, questi studi in vitro non catturano completamente le interazioni multicellulari e il complesso dialogo tra le popolazioni cellulari, comprese le cellule del midollo osseo, che sono essenziali per regolare il processo di rigenerazione ossea9. Questa mancanza di rappresentazione accurata del microambiente osseo può compromettere l'accuratezza dei successivi studi preclinici10.
Sebbene le valutazioni in vivo forniscano una rappresentazione più accurata dei contesti fisiologici, sono vincolate da considerazioni etiche, logistiche e finanziarie. Di conseguenza, le valutazioni ex vivo svolgono un ruolo fondamentale come interfaccia tra gli studi in vitro e in vivo, fungendo da passaggio intermedio necessario prima di passare agli esperimenti su soggetti viventi 11,12,13.
In questo contesto, l'implementazione di metodologie di caratterizzazione complete è necessaria per valutare la qualità del tessuto osseo rigenerato e per garantire la pertinenza della strategia prima di passare a un modello preclinico. Di conseguenza, proponiamo un protocollo basato sull'analisi di un modello di espianto utilizzando tessuto articolare del ginocchio di pecora. Questa metodologia innovativa prevede l'impianto di cemento caricato con BMP2 negli espianti e l'esecuzione di un'analisi dettagliata della mineralizzazione dei tessuti dopo 28 giorni di coltura.
Gli approcci tecnici impiegati in questo studio sono diversi e complementari e forniscono collettivamente un approccio completo alla valutazione della qualità del tessuto osseo rigenerato (Figura 1). L'imaging micro-CT ad alta risoluzione consente una visualizzazione 3D dettagliata della struttura ossea, fornendo preziose informazioni sulla densità minerale, la morfologia e l'integrità del tessuto appena formato. Questa tecnica è fondamentale per valutare l'efficacia della rigenerazione ossea e monitorare la progressione della mineralizzazione nel tempo. La nanoindentazione è un approccio preciso per determinare le proprietà meccaniche del tessuto, come la durezza e la resistenza. Misurando la risposta del materiale a una forza applicata su scala nanometrica, questo metodo consente di valutare la robustezza e la qualità del tessuto mineralizzato. Gli esami istologici che utilizzano coloranti comuni come ematossilina/eosina/zafferano (HES), il tricromo di Goldner e il pentacroma di Movat forniscono informazioni preziose sulla struttura e la composizione dei tessuti. Queste colorazioni consentono la differenziazione dei vari componenti tissutali, tra cui cellule, matrice extracellulare e depositi minerali, consentendo così una valutazione qualitativa completa del processo di rigenerazione ossea. La mappatura al microscopio elettronico a scansione backscatter (SEM) offre una visualizzazione ad alta risoluzione della superficie dei campioni, consentendo un'analisi dettagliata del grado di mineralizzazione della matrice ossea, nonché delle interfacce tra il materiale impiantato e il tessuto ospite. Infine, la spettroscopia Raman fornisce informazioni riguardanti la composizione molecolare del tessuto, in particolare attraverso l'identificazione di componenti specifici come proteine, lipidi e minerali. Questo approccio consente la caratterizzazione della matrice mineralizzata e la rilevazione di fattori di crescita come BMP2, fornendo così informazioni cruciali sulla persistenza di stimoli pro-osteogenici nel mezzo di rigenerazione.
Utilizzando un approccio multidisciplinare, che integra varie tecniche analitiche, il nostro studio mira a fornire una valutazione approfondita e completa della qualità del tessuto osseo rigenerato, fornendo così una solida base per la valutazione dei sostituti dell'innesto osseo e della loro potenziale applicazione clinica.
Access restricted. Please log in or start a trial to view this content.
Protocollo
Questo studio è stato approvato da un comitato per l'etica e il benessere degli animali e dall'Amministrazione nazionale francese per la veterinaria e l'alimentazione con il numero G44171.
1. Preparazione e coltura di espianti osteocondrali
- Raccogli esemplari di pecore da animali appena soppressi in un ambiente asettico. Posiziona la pecora in posizione supina e rade l'arto posteriore sinistro. Prepararsi disinfettando con alcool intorno all'articolazione del ginocchio. Utilizzare l'artrotomia pararotulea laterale per esporre i legamenti crociati anteriore e posteriore, seguita dalla lussazione rotulea per rivelare la troclea femorale.
- Dopo il prelievo di tessuto, creare un difetto di 4,75 mm di diametro nel condilo laterale utilizzando un sistema di trasferimento autotrapianto osteocondrale.
- Utilizzando un martello ortopedico, creare un difetto di 10 mm di profondità e recuperare l'espiante dal condilo. Successivamente, eseguire un secondo difetto di 8 mm di diametro, centrato attorno al primo, utilizzando un sistema di trasferimento autoinnesto osteocondrale. Si ottengono espianti osteocondrali di 8 mm x 10 mm, contenenti un difetto centrale di 4,75 mm x 10,0 mm.
- Se necessario per motivi logistici, trasportare gli espianti nella soluzione salina bilanciata di Hank (hBSS) integrata con l'1% di penicillina-streptomicina e conservare a 4 °C.
- Sciacquare gli espianti 3 volte con soluzione salina tamponata con fosfato (PBS), quindi iniettare il difetto di 4,75 mm x 10,0 mm con un cemento fosfato di calcio integrato con 40 μg/mL di BMP2. Preparare il cemento come descritto di seguito.
- Produrre fosfato α-tricalcico (α-TCP) mediante trattamento termico di barre di fosfato tricalcico apatitico (pressa isostatica a 120 MPa) a 1364 °C per un minimo di 12 ore, seguito da tempra in aria14.
- Macinare le barre α-TCP in etanolo assoluto con una sfera di mulino planetario 2 volte per 5 minuti a 500 giri/min per produrre una polvere fine di 13,1 ± 1,7 μm (misurata utilizzando la diffrazione laser, come descritto in precedenza14).
- Sterilizzare la polvere di α-TCP riscaldandola a secco a 180 °C per 45 minuti. Preparare il cemento fosfato di calcio mescolando la polvere di α-TCP con una soluzione filtrata al 2,5% (p/v) di Na2HPO4 0,22 μm (rapporto liquido/polvere di 0,35) per 1 minuto.
- Caricare la pasta di cemento in una siringa da 3 ml prima dell'iniezione utilizzando un ago da 18 G.
- Dopo 10 minuti a temperatura ambiente (RT), trasferire gli espianti in un pallone da 25 cm² e aggiungere delicatamente 10 ml di terreno di coltura completo costituito da Dulbecco's Modified Eagle Medium (DMEM) ad alto contenuto di glucosio integrato con il 10% di siero fetale bovino e l'1% di penicillina-streptomicina.
- Mantenere gli espianti per un massimo di 28 giorni in un incubatore a 37 °C con CO2 al 5% e sostituire l'intero terreno di coltura ogni 2 giorni sotto una cappa a flusso laminare.
- Sciacquare gli espianti 3 volte con PBS. Trasferirli in provette da 50 mL, aggiungere 20 mL di paraformaldeide (PFA) al 4% e mantenerli a 4 °C per 3 giorni. Quindi, sciacquare gli espianti con PBS e conservarli in etanolo al 70% prima delle analisi successive.
2. Analisi Micro-CT
- Condurre analisi di micro-tomografia utilizzando una radiografia micro-CT. Acquisisci proiezioni di raggi X con risoluzione di 10,7 μm, con un tempo di esposizione di 1200 ms e un filtro in alluminio da 1 mm (80 kV e 125 μA). Calcola la media di tre immagini per ogni incremento di rotazione di 0,45° al fine di migliorare il rapporto segnale/rumore.
- Ricostruisci immagini tridimensionali (3D) utilizzando il software di ricostruzione del produttore dopo un allineamento X/Y con una scansione di riferimento, con i seguenti parametri - Levigatura: 0, Artefatto dell'anello: 3, Indurimento del fascio: 35%. Pulisci le pile di immagini con il software di elaborazione delle immagini e osserva con strumenti di visualizzazione bidimensionali (2D) e 3D.
3. Analisi delle immagini con deep learning
- Conduci la segmentazione delle immagini utilizzando un software specializzato dedicato. Utilizza la procedura guidata di segmentazione integrata per addestrare un modello di deep learning per la discriminazione di ossa e cemento.
- Selezionare una zona rappresentativa (ad esempio, il telaio) contenente osso, cemento e sfondo dalle immagini micro-CT ricostruite. Segmenta manualmente questo primo fotogramma utilizzando strumenti semplici (ad esempio, soglia) o potenti (ad esempio, la propagazione) forniti dal software.
- Genera un modello di deep learning nella scheda Modello e seleziona Routine 3D-Unet nel menu a discesa. Definire i suoi parametri sperimentali in allineamento con le immagini da analizzare con un clic destro sul modello generato; qui: profondità di 5, dimensione della patch 32 x 32, algoritmo Adadetla, rapporto di falcata di 0,25, aumento dei dati x10.
- Utilizza il fotogramma segmentato per addestrare la routine di deep learning (fai clic sul pulsante Addestra), che è riconosciuta per le sue capacità di segmentazione delle immagini rapide e accurate 3,15. Una volta completata la formazione, definire una seconda zona di lavoro (frame) e segmentare automaticamente utilizzando la funzione di previsione.
- Se necessario, applicare la correzione manuale per generare dati di training più accurati. Allenati di nuovo alla routine. Ripetere il processo fino ad ottenere un risultato soddisfacente; Il numero di incrementi di dati diminuisce progressivamente all'aumentare del numero di frame di training.
NOTA: L'indice DICE fornito durante l'addestramento del modello fornisce un'indicazione dell'accuratezza del modello (rispetto alle informazioni di input) ma non è sufficiente. La conferma della pertinenza del modello dovrebbe essere effettuata mediante la convalida dei dati segmentati automaticamente da 3 ricercatori esperti indipendenti.
- Se necessario, applicare la correzione manuale per generare dati di training più accurati. Allenati di nuovo alla routine. Ripetere il processo fino ad ottenere un risultato soddisfacente; Il numero di incrementi di dati diminuisce progressivamente all'aumentare del numero di frame di training.
- Pubblica il modello cliccando sul pulsante Esporta e applicalo all'intero dataset micro -CT cliccando su Segmenta > Modello Esportato > Segmenta Set di Dati Completo.
4. Incorporamento
- Disidratare gli espianti ossei mettendoli in un flaconcino di vetro da 40 mL contenente 25 mL di soluzione disidratante composta per il 70% da acetone e per il 30% da xilene. Posizionare la fiala di vetro su una ruota rotante per 1 ora a RT. Ripetere questo passaggio 3 volte.
- Sostituire la soluzione di disidratazione con 25 ml di xilene e posizionare il flaconcino di vetro sulla ruota rotante per 1 ora a RT.
- Sostituire la soluzione di xilene con 25 mL di metilmetacrilato arricchito (MMA) con il 10% di perossido di benzoile (BPO) e il 10% di dibutilftalato (DBP). Posizionarlo sulla rotella rotante per 1 ora a RT. Ripetere questo passaggio 2 volte.
- Sostituire la soluzione di MMA con 25 mL di MMA arricchiti con il 10% di BPO, il 10% di DBP e 450 μL di N, N dimetilanilina diluita 1:20 in propan-2-olo. Posizionare il flaconcino di vetro a -20 °C per una notte. La soluzione diventa giallastra.
- Posizionare l'espiante in uno stampo da incasso di medie dimensioni e versare l'anilina MMA-BPO-DBP-N, N nello stampo. Mettere lo stampo in una scatola di plastica e ventilare con flusso di azoto per 5 minuti. Chiudere ermeticamente la scatola e posizionarla a 4 °C per 48 h per la polimerizzazione e l'indurimento dell'MMA.
- Rimuovere la resina contenente l'espianto dallo stampo e mantenerla a 4 °C fino alla lavorazione per la successiva analisi.
5. Microscopia elettronica a scansione (SEM) - imaging elettronico retrodiffuso quantitativo (qBEI)
- Tagliare il blocco di pMMA contenente l'espiante con una sega diamantata lungo il suo asse lungo. Conservare la prima metà del blocco per l'analisi istologica. Tagliare ulteriormente l'altra metà per generare una sezione spessa 1,5 mm. Eseguire il taglio a 3000 giri/min con una velocità di 3 mm/min.
- Macinare la sezione con carta al carburo di silicio con numeri crescenti corrispondenti alla granulometria inferiore: SiC 320 per 10 s, SiC 1000 per 15 s, SiC 2000 per 30 s e SiC 4000 per 30 s.
- Lucidare la sezione con pasta diamantata e capi specifici: panno lucidante con soluzione diamantata Mol B3 da 3 μm per 1 min e panno in pile con soluzione diamantata Nap B1 da 1 μm per 1 min.
- Sciacquare la sezione sotto acqua distillata doppia e pulire le particelle di diamante con un batuffolo di cotone. Asciugare la sezione sotto azoto gassoso.
- Rivestire in carbonio la sezione spessa (10 nm di spessore del film di carbonio) e montarla su un tronchetto di alluminio con vernice argento. Crea un ponte di vernice argentata tra la parte superiore della sezione e il troncone per consentire l'evacuazione della carica di elettroni a terra.
- Posizionare lo stub sul livello SEM. Accanto al campione, inserire nella camera SEM uno stub di controllo composto da una coppa di Faraday con standard di carbonio, alluminio e silicio. Questo servirà per calibrare il fascio di elettroni e convertire i livelli di grigio in percentuali di Ca. Chiudere la camera SEM e il vuoto.
- Accendi il fascio di elettroni e regola i parametri SEM in modo che funzionino in modalità elettronica retrodiffusa. Nelle immagini retrodiffuse, il livello di grigio dello standard di carbonio è 25, lo standard di alluminio è 225 e lo standard di silicio è 253. A titolo indicativo, sul sistema utilizzato, i parametri SEM sono tensione di accelerazione 20 keV, corrente della sonda misurata con la tazza di Faraday: 250 pA, distanza di lavoro: 15 mm, apertura: 30 μm, risoluzione dell'immagine: 1024 x 768 pixel, vuoto: < 4,10-4 Pa, tempo di permanenza di 100 μs/pixel, luminosità ~38 e contrasto ~72.
- Quando il SEM è calibrato con gli standard, acquisire immagini del campione in modalità elettronica retrodiffusa.
- Utilizzare l'immagine elettronica retrodiffusa del campione per convertire i livelli di grigio in contenuto di calcio, come descritto in Roschger et al.16. Eseguire questa operazione utilizzando qualsiasi software di analisi delle immagini.
- Tracciare la distribuzione del contenuto di calcio. La distribuzione del contenuto di calcio mostra una distribuzione gaussiana. Vengono calcolati tre parametri principali di interesse: Camedia, come il contenuto medio di calcio nell'immagine, Capicco come la concentrazione di calcio più frequente incontrata nell'immagine e Cawidth come l'intera larghezza a metà del massimo della distribuzione del contenuto di calcio.
NOTA: La riproducibilità di qBEi è stata studiata mediante imaging della stessa regione di interesse per 5 giorni consecutivi (un'acquisizione al giorno). Ciò significa che il fascio di elettroni è stato acceso e spento tra una sessione e l'altra. La percentuale di errore nellamedia del Ca, nelpicco del Ca e nella Cawidth è stata stimata rispettivamente allo 0,5%, 0,7% e 1,2%. Il contenuto di calcio è stato studiato anche mediante analisi a raggi X a dispersione di energia (EDS). Un'ottima relazione lineare è stata stabilita tra il Camedio e il contenuto di calcio stimato dall'EDS con un valore di R² di 0,99, simile ai dati ottenuti da Roschger et al. precedentemente17.
6. Istologia
- Per ogni campione, tagliare sezioni da 7 μm utilizzando un microtomo per tessuti duri con una lama di tungsteno 18,19. Sezioni di colorazione con colorante Goldner trichrome, HES e MOVAT utilizzando un sistema di colorazione automatico.
- Per la deplastificazione, immergere i campioni in una successione di acetone per 5 minuti e ripetere 2 volte. Sciacquare i campioni in acqua distillata per 5 minuti e ripetere 2 volte.
- Eseguire la colorazione Goldner Trichrome come descritto di seguito.
- Posizionare i campioni nell'ematossilina ferrica Weigert per 20 minuti. Lavare con acqua di rubinetto. Immergere i campioni in alcol acido allo 0,5% per 30 s-1 min. Lavare con acqua di rubinetto per 20 min.
- Porre i campioni in una soluzione di Ponceau/acido fucssino/azophlossina per 5 minuti. Immergere in acido acetico all'1% per 1 minuto.
- Colorare con soluzione di acido fosfomolibdico/Arancia G per 20 min. Immergere in acido acetico all'1% per 1 minuto.
- Colorare con soluzione fast green per 15 min a temperatura ambiente o 8 min a 60 °C. Sciacquare abbondantemente con acqua di rubinetto; Ripeti 3 volte.
- Eseguire la colorazione Movat Pentachrome come descritto di seguito.
- Colorazione Alcian Blue: immergere i campioni nella soluzione Alcian Blue per 30 min. Sciacquare i campioni in acqua di rubinetto per 5 minuti. Porre i campioni in etanolo alcalino per 60 minuti. Sciacquare i campioni in acqua di rubinetto per 10 minuti. Eseguire un risciacquo finale con acqua distillata.
- Colorazione con ematossilina di Weigert: posizionare i campioni nell'ematossilina ferrica di Weigert per 20-30 minuti. Sciacquare i campioni in acqua di rubinetto per 15 minuti. Risciacquare nuovamente con acqua distillata.
- Colorazione con Crocein Brilliant/acido fucsina: immergere i campioni in una soluzione di Crocein Brilliant/Acid Fuchsin per 10 min. Risciacquare con acido acetico allo 0,5%. Immergere i campioni in una soluzione di acido fosfotungstico al 5% per 20 minuti. Risciacquare con acido acetico allo 0,5% per 2 minuti. Eseguire tre risciacqui successivi con etanolo al 100%, ciascuno della durata di 5 minuti.
- Colorazione con Safran du Gatinais: posizionare i campioni in Safran du Gatinais per 15 min. Eseguire un risciacquo finale con acqua di rubinetto.
- Eseguire la colorazione HES come descritto di seguito.
- Colorazione con ematossilina di Weigert: immergere i campioni nell'ematossilina di Weigert per 30 minuti. Risciacquare con acqua di rubinetto per 2 min.
- Decolorazione: immergere i campioni in alcool di acido cloridrico per 10 s. Risciacquare con acqua di rubinetto per 2 min.
- Neutralizzazione: immergere i campioni in una soluzione di carbonato di litio per 1 min. Risciacquare con acqua di rubinetto per 2 min. Eseguire un risciacquo finale con acqua distillata per 1 minuto.
- Colorazione con Eosina-Eritrosina: porre i campioni in una soluzione di Eosina-Eritrosina per 3 minuti. Sciacquare con acqua di rubinetto per 10 s.
- Disidratazione: Immergere i campioni in alcol al 95% per 15 s. Trasferire al 100% di alcol per 15 s. Ripetere l'immersione in alcool al 100% per 30 s.
- Colorazione finale con Safranina Alcolica: immergere i campioni in safranina alcolica per 10 min. Eseguire un risciacquo finale con acqua di rubinetto.
- Eseguire il montaggio della slitta come descritto di seguito.
- Sciacquare i campioni con etanolo al 95%, quindi etanolo al 100%, quindi metilcicloesano e ripetere l'operazione 3 volte per ogni passaggio.
- Montare i campioni utilizzando un kit di montaggio e aggiungere un vetrino coprioggetti. Scansiona le immagini utilizzando uno scanner digitale per diapositive.
7. Microspettroscopia Raman
- Utilizzare la stessa sezione utilizzata per l'analisi Raman come SEM. Macinare e lucidare brevemente la sezione come descritto nei passaggi 5.2 - 5.3 per rimuovere il sottile strato di carbonio aggiunto per il SEM che compromette la dissipazione del calore dal campione.
- Posizionare la sezione sul tavolino del microspettrometro Raman. Calibrare il numero d'onda per garantire la precisione e allineare il laser prima della misurazione. Poiché tali procedure sono specifiche per ogni strumento, non saranno descritte in questo articolo.
- Individua le regioni di interesse nel tessuto che devono essere analizzate. Posizionare la zona da analizzare al centro dello strumento video e raccogliere gli spettri Raman con i seguenti parametri: laser da 785 nm utilizzato a 30 mW, tempo di integrazione: 20 s ripetuto 3 volte, intervallo spettrale: 350-1800 cm-1, reticolo 1200 linee/mm.
- Raccogli 10 spettri della resina da incasso con le stesse impostazioni Raman e fai la media. Utilizzare questa opzione per sottrarre il contributo di resina nel passaggio 7.6.
- Pre-elabora gli spettri come segue per rimuovere il contributo di fluorescenza, rumore e resina: correzione della linea di base con un adattamento polinomiale a cinque ordini, filtro Savitzky-Golay con un grado di 4 e una dimensione della finestra compresa tra 17 e 21, sottrazione digitale della resina utilizzando il picco di ~812 cm-1 della resina. Tale pre-elaborazione può essere eseguita con diversi software progettati per l'analisi e la visualizzazione dei dati.
- Analizza ulteriormente gli spettri pre-elaborati per estrarre parametri preziosi razionando l'intensità di picco della vibrazione di interesse con l'intensità di picco della vibrazione di riferimento. Per l'analisi ossea, ad esempio, utilizzare i seguenti parametri per calcolarla.
- Rapporti minerale/matrice: dividere l'intensità di picco delle vibrazioni v1PO4 a ~960 cm-1 per l'intensità di picco delle modalità vibrazionali Ammide I (~1668 cm-1), Ammide III (~1250 cm-1), CH2-wag (~1450 cm-1) o la somma dei modi vibrazionali prolina (~854 cm-1) e idrossiprolina (Hyp, ~872 cm-1). Il v2PO4, situato a ~430 cm-1, o il v4PO4, situato a ~600 cm-1, possono anche essere utilizzati e razionati con l'Amide III vibration20. Questi parametri rappresentano il grado di mineralizzazione della fase organica della matrice ossea.
- Carbonato/Fosfato: dividere l'intensità di picco del v1CO3, situato a ~1070 cm-1, per il v1PO4. Questo rappresenta la quantità di sostituzione del carbonato di tipo B nel reticolo di apatite. Hyp/Pro, rappresenta il contenuto di idrossiprolina, ~1375 cm-1/Amide III, rappresenta il contenuto di proteoglicani, ~1150 cm-1/CH2-wag e ~1495 cm-1/CH2-wag, rappresenta la quantità dei prodotti finali della glicazione avanzata carbossimetilisina e pentosidina, 1670 cm-1/1690 cm-1 e 1670 cm-1/1640 cm-1 rappresentano rispettivamente la struttura secondaria disordinata del collagene, nota anche come maturità del collagene, e le strutture ordinate sotto forma di α-elica nel collagene di tipo I.
- Valutare la riproducibilità delle misure Raman esaminando la stessa regione di interesse in 5 acquisizioni consecutive (un'acquisizione al giorno). Per ogni parametro analizzato, la varianza era inferiore allo 0,2% del valore del parametro, supportando una buona riproducibilità. La specificità di ciascun picco studiato è stata ampiamente discussa sul campo e una panoramica è presentata nella recente revisione di Unal21.
8. Nanoindentazione
NOTA: A causa della natura distruttiva della nanoindentazione, di solito viene eseguita alla fine della routine di analisi del campione. Il sistema di nanoindentazione che possediamo è dotato di un penetratore piramidale in diamante Berkovitch. Tuttavia, esistono diverse forme di penetratore e non è stato determinato alcun consenso in letteratura per campioni di osso o biomateriale.
- Idratare il campione osseo incorporato in soluzione salina per 16 ore a RT prima del test di nanoindentazione.
- Calibrare il sistema di nanoindentazione utilizzando silice fusa e registrare il modulo di indentazione risultante. Per la silice fusa, il modulo di indentazione è di circa 72 GPa utilizzando un coefficiente di Poisson di 0,17.
- Una volta calibrato il sistema di nanoindentazione, posizionare il campione utilizzato per le analisi Raman nel sistema ottico del dispositivo di nanoindentazione per indicare le posizioni in cui verrà eseguita la nanoindentazione.
NOTA: Questa modalità varia da un'apparecchiatura all'altra; Non dettagliamo come funzionano il sistema di nanoindentazione e il software, ma diamo consigli generali sui diversi passaggi che dovrebbero essere eseguiti. - Una volta scelta la posizione della nanoindentazione, spostare il campione sotto il dispositivo di indentazione ed eseguire l'indentazione. Eseguire la nanoindentazione a una profondità costante di 900 nm, con una velocità di carico/scarico di 40 mm/min e una pausa di 15 s tra il carico e lo scarico. Impostare il coefficiente di Poisson per il tessuto osseo a 0,3.
- Recupera i diversi parametri dalle curve di nanoindentazione e calcolali secondo la teoria22 di Oliver e Pharr.
- Il modulo di indentazione (EIT) è derivato dalle proprietà note della punta di indentazione e del modulo ridotto e combina il modulo elastico locale del provino. Calcola l'indentazione come:
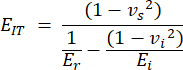
Con vs, vi, Ei eE r che rappresentano il coefficiente di Poisson del campione, assunto essere 0,3 per l'osso, il coefficiente di Poisson del penetratore di diamante, assunto pari a 0,07, il modulo elastico del penetratore di diamante, fissato a 1140 GPa e il modulo ridotto, calcolato come: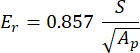
Dove S rappresenta la pendenza del segmento di scarico e Ap è l'area proiettata e calcolata come: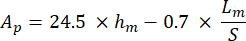
- Durezza (HIT): corrispondente alla resistenza dell'osso all'innesco e alla propagazione delle cricche. In altre parole, è un indicatore della tenacità ossea. Calcola HIT come:

- Velocità di scorrimento dell'indentazione (CIT): Riflette la deformazione ossea nel tempo a carico costante, indicativa delle proprietà viscoelastiche del campione, calcolata come:
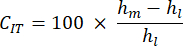
- Carico massimo (Lm): corrisponde al carico opposto necessario per penetrare la profondità indicata nella matrice ossea mineralizzata.
- Lavoro di indentazione (Wplast): corrisponde all'area sotto la curva di deformazione carico/scarico. È una riflessione diretta dell'energia dissipata per indurre la deformazione plastica.
NOTA: La riproducibilità della nanoindentazione è più difficile da valutare su campioni ossei a causa della natura distruttiva di questa metodologia. Tuttavia, abbiamo stimato la riproducibilità del campione di silice fusa standard e uniforme. Per tutti i parametri di cui sopra, la varianza è stata stimata al di sotto dello 0,26% (5 misure consecutive in 5 giorni separati), supportando un'ottima riproducibilità. D'altra parte, a causa della natura anisotropa dei campioni ossei, è più difficile apprezzarne la riproducibilità.
- Il modulo di indentazione (EIT) è derivato dalle proprietà note della punta di indentazione e del modulo ridotto e combina il modulo elastico locale del provino. Calcola l'indentazione come:
Access restricted. Please log in or start a trial to view this content.
Risultati
Un'immagine micro-CT dell'espianto è mostrata nella Figura 2. Utilizzando la segmentazione manuale non è possibile separare in modo ottimale l'osso dal cemento, presente nel canale centrale, utilizzando la soglia globale. Per migliorare il riconoscimento dell'osso trabecolare e del cemento, proponiamo di utilizzare il deep learning. Il deep learning è potente per riconoscere le caratteristiche dei biomateriali e aiuta a migliorare la separazione tra osso ...
Access restricted. Please log in or start a trial to view this content.
Discussione
La riparazione dei difetti ossei è una delle principali sfide della medicina rigenerativa per ripristinare la mobilità, ridurre il dolore e migliorare la qualità della vita delle persone colpite. L'uso di modelli di espianti offre una serie di vantaggi rispetto agli studi in vivo per lo studio della riparazione dei difetti ossei. Oltre alle considerazioni etiche, questo modello consente il controllo rigoroso delle condizioni sperimentali e la riduzione della variabilità biol...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Gli autori non hanno conflitti di interesse da rivelare.
Riconoscimenti
Vogliamo ringraziare le strutture tecniche coinvolte nella raccolta e nell'elaborazione dei campioni, tra cui SC3M (SFR François Bonamy (UMS 016), Università di Nantes), SFR ICAT (Università di Angers), BIO3, HiMolA e SC4BIO. L'Inserm UMR_S 1229 RMeS è sostenuto da sovvenzioni del governo francese attraverso le istituzioni Inserm, Nantes Université, Univ Angers e Oniris VetAgroBio. CL è anche grata a HTL Biotechnology.
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| 0.20 filters | VWR | 28145-501 | |
| 18 G needle (1,2x40 mm) | Sterican | 4665120 | |
| 3 mL syringe | HENKE-JECT | 8300005762 | |
| 37% hydrochloric acid | VWR | 1.00317.1000 | |
| Acetic acid (glacial) | Sigma | A6283 | |
| Acetone | VWR | 20063-365 | |
| Alcian Blue 8GX | VWR | 361186 | |
| Ammonium hydroxide | VWR | 318612 | |
| Apatitic tricalcium phosphate | Centre for Biomedical and Healthcare Engineering (Mines Saint Etienne, France) | TV26U | |
| Azophloxine | Sigma | 210633 | |
| Benzoyl peroxide | Sigma | 8.01641.0250 | |
| BMP2 | Medtronic | InductOs 1.5 mg/mL | |
| Brillant crocein | Aldrich | 2107507 | |
| CTVox | Bruker | - | |
| DataViewer | Skyscan | - | |
| Diamond blade | Struers | MOD13 | |
| Diamond saw | Struers | Accutom-50 | |
| DiaPro Mol B3 diamond solution | Struers | 40600379 | |
| DiaPro Nap B1 diamond solution | Struers | 40600373 | |
| Dibasic sodium phosphate (Na2HPO4) | Sigma | 102404598 | |
| Dibutyl Phtalate | Chimie-Plus Laboratoires | 28656 | |
| DragonFly software | ORS | 2022.1.0.1231. | |
| Dulbecco's Modified Eagle Medium (DMEM) high glucose, GlutaMAX(TM), pyruvate | ThermoFisher Scientific | 31966-021 | |
| Eosine Y- Surgipath | Sigma | 1002830105 | |
| Erythrosin B | Sigma | 102141057 | |
| Ethanol absolute | VWR | 20820362 | |
| Eukitt | Dutscher | 6.00.01.0003.06.01.01 | |
| Falcon 50 mL | Sarstedt | 62.547.254 | |
| Ferric chloride hexahydrate (FeCl3, 6H2O) | Merck | 1.03943.0250 | |
| Fetal Bovine Serum (FBS) | Eurobio | CVFSVF00 | |
| Fuchsine acid | Merck | 1.05231.0025 | |
| Hank's Balanced Salt Solution (HBSS) | Biosera | MS01NG100J | |
| Hematoxylin | Sigma | 86.118.9 | |
| Isostatic press | Nova Suisse | Pmax 1500 bars | |
| Laser diffraction granulometry | Malvern | Mastersizer 3000 | |
| Light green | Prolabo | 28947135 | |
| Lithium carbonate | Sigma | A13149 | |
| MD-Mol polishing cloth | Struers | 40500077 | |
| Methylcyclohexane | VWR | 8.06147.1000 | |
| Methylcyclohexane | VWR | 8.06147.1000 | |
| Methylcyclohexane | VWR | 8.06147.1000 | |
| Methylmethacrylate | Sigma | 8.00590.2500 | |
| Micro-CT, micro-scanner | Bruker | Skyscan 1272 | |
| Monobasic sodium phosphate (NAH2PO4) | Sigma | 71496 | |
| Mortar | Fritsch | Pulverisette 6 | |
| N,N, Dimethylanilin | Sigma | 803060 | |
| Nanoindentation station | Anton Paar | NHT2 | |
| ND-Nap polishing cloth | Struers | 40500080 | |
| OATS Osteochondral Autograft Transfer System Set, 4,75 mm | Arthrex | AR-1981-04S | |
| OATS Osteochondral Autograft Transfer System Set, 8 mm | Arthrex | AR-1981-08S | |
| Orange G | Ral | M15 | |
| Paraformaldehyde (PFA) | Sigma | P6148 | |
| Peel-a-way disposable embbedding moulds | Polysciences, Inc | 18646C-1 | |
| Penicillin/Streptomycin (P/S) | ThermoFisher Scientific | 15140122 | |
| Phosphate Buffered Saline (PBS) | ThermoFisher Scientific | 10010023 | |
| Phosphomolybdic acid | Sigma | 221856-100 g | |
| Phosphotungstic acid | Aldrich | 12863-5 | |
| Polishing machine | Sturers | Dap V | |
| Poupinel | MEMMERT | TV26U | |
| Raman microspectrometer | Renishaw | InVia Qontor | |
| Safran du Gâtinais | Labonord | 11507737 | |
| Scanning electron microscope | Carl Zeiss | Evo LS 10 | |
| SEM | Zeiss | Carl Zeiss Evo LS10 | |
| SiC foils/Grinding papers | Struers | 40400008 (#320), 40400011 (#1000), 40400122 (#2000), 40400182 (#4000) | |
| Silver paint | Electron microscopy sciences | 12686-15 | |
| Standard stub with Faraday cup, carbon, aluminium and silicon standards | Micro-Analysis Consultants Ltd | 8602 | |
| T25 flask | Corning | 430639 | |
| Xylene | VWR | 28975.325 | |
| Xylidine Ponceau | Aldrich | 19.976-1 |
Riferimenti
- Feroz, S., Cathro, P., Ivanovski, S., Muhammad, N. Biomimetic bone grafts and substitutes: A review of recent advancements and applications. Biomed Eng Adv. 6, 100107(2023).
- Tsuji, K., et al. BMP2 activity, although dispensable for bone formation, is required for the initiation of fracture healing. Nat Genet. 38 (12), 1424-1429 (2006).
- Hadjidakis, D. J., Androulakis, I. I. Boneremodeling. Ann N Y Acad Sci. 1092, 385-396 (2006).
- Boivin, G., Meunier, P. J. The degree of mineralization of bone tissue measured by computerized quantitative contact microradiography. Calcified Tiss Int. 70 (6), 503-511 (2002).
- Boivin, G., et al. Influence of remodeling on the mineralization of bone tissue. Osteoporo Int. 20 (6), 1023-1026 (2009).
- Zanghellini, B., et al. Multimodal analysis and comparison of stoichiometric and structural characteristics of parosteal and conventional osteosarcoma with massive sclerosis in human bone. J Str Biol. 216 (3), 108106(2024).
- Trento, G., et al. formation around two titanium implant surfaces placed in bone defects with and without a bone substitute material: A histological, histomorphometric, and micro-computed tomography evaluation. Clin Implant Dent Relat Res. 22 (2), 177-185 (2020).
- Palmquist, A. A multiscale analytical approach to evaluate osseointegration. J Mater Sci Mater Med. 29 (5), 60(2018).
- Budán, F., et al. Novel radiomics evaluation of bone formation utilizing multimodal (SPECT/X-ray CT) in vivo imaging. PLoS ONE. 13 (9), e0204423(2018).
- Hulsart-Billström, G., et al. A surprisingly poor correlation between in vitro and in vivo testing of biomaterials for bone regeneration: results of a multicentre analysis. Eur Cells Mater. 31, 312-322 (2016).
- Cramer, E. E. A., Ito, K., Hofmann, S. Ex vivo bone models and their potential in preclinical evaluation. Curr Osteoporo Rep. 19 (1), 75-87 (2021).
- Chan, M. E., et al. A trabecular bone explant model of osteocyte-osteoblast co-culture for bone mechanobiology. Cell Mol Bioeng. 2 (3), 405-415 (2009).
- Doke, S. K., Dhawale, S. C. Alternatives to animal testing: A review. Saudi Pharma J. 23 (3), 223-229 (2015).
- Moussi, H., et al. New formulation of injectable and degradable calcium phosphate/silanized hyaluronic acid composite foam: Investigation in a rabbit model of long bone defect. SSRN. , (2024).
- Paré, A., et al. Standardized and axially vascularized calcium phosphate-based implants for segmental mandibular defects: A promising proof of concept. Acta Biomater. 154, 626-640 (2022).
- Roschger, P., Fratzl, P., Eschberger, J., Klaushofer, K. Validation of quantitative backscattered electron imaging for the measurement of mineral density distribution in human bone biopsies. Bone. 23 (4), 319-326 (1998).
- Roschger, P., Plenk, H., Klaushofer, K., Eschberger, J. A new scanning electron microscopy approach to the quantification of bone mineral distribution: backscattered electron image grey-levels correlated to calcium K alpha-line intensities. Scanning Micro. 9 (1), 75-86 (1995).
- Porter, A., et al. Quick and inexpensive paraffin-embedding method for dynamic bone formation analyses. Sci Rep. 7, 42505(2017).
- Goldschlager, T., Abdelkader, A., Kerr, J., Boundy, I., Jenkin, G. Undecalcified bone preparation for histology, histomorphometry and fluorochrome analysis. J Vis Exp. (35), e1707(2010).
- Kazanci, M., Roschger, P., Paschalis, E. P., Klaushofer, K., Fratzl, P. Bone osteonal tissues by Raman spectral mapping: Orientation-composition. J Str Biol. 156 (3), 489-496 (2006).
- Unal, M., Ahmed, R., Mahadevan-Jansen, A., Nyman, J. S. Compositional assessment of bone by Raman spectroscopy. Analyst. 146 (24), 7464-7490 (2021).
- Oliver, W. C., Pharr, G. M. An improved technique for determining hardness and elastic modulus using load and displacement sensing indentation experiments. J Mater Res. 7 (6), 1564-1583 (1992).
- Proffen, B. L., McElfresh, M., Fleming, B. C., Murray, M. M. A comparative anatomical study of the human knee and six animal species. Knee. 19 (4), 493-499 (2012).
- Allen, M. J., Houlton, J. E. F., Adams, S. B., Rushton, N. The surgical anatomy of the stifle joint in Sheep. Vet Surg. 27 (6), 596-605 (1998).
- Kleuskens, M. W. A., van Donkelaar, C. C., Kock, L. M., Janssen, R. P. A., Ito, K. An ex vivo human osteochondral culture model. J Ortho Res. 39 (4), 871-879 (2021).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon