Method Article
Lokalisierung funktionsspezifischer Ziele für die transkranielle Magnetstimulation in Abwesenheit von Navigationsgeräten
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
In dieser Arbeit wird beschrieben, wie funktionsspezifische Ziele für repetitive transkranielle Magnetstimulationseingriffe oder -behandlungen lokalisiert werden können, wenn keine Navigationsgeräte verfügbar sind.
Zusammenfassung
Die repetitive transkranielle Magnetstimulation (rTMS) ist eine nicht-invasive Technik, die die neuronale Aktivität im Gehirn moduliert. Studien haben gezeigt, dass rTMS die neuronale Plastizität regulieren kann, die Reorganisation neuronaler Netzwerke fördert und bei neuropsychiatrischen Erkrankungen wie Schlaganfällen weit verbreitet ist. Obwohl einige Studien darauf hindeuten, dass rTMS bei der Schlaganfallrehabilitation helfen kann, bleibt ihre Wirksamkeit ungewiss, möglicherweise aufgrund von Einschränkungen in der traditionellen Lokalisation des handmotorischen Hotspots.
Der handmotorische Hotspot wird durch motorisch evozierte Potentiale (MEPs) bestimmt, die die Leitfähigkeit der kortikospinalen oder pyramidalen Bahn widerspiegeln und eine nicht-willkürliche Bewegung darstellen. Im Gegensatz dazu definieren funktionelle Magnetresonanztomographie (fMRT)-Aktivierungspunkte aus einer motorischen Aufgabe funktionsspezifische Ziele, die sowohl die Wahrnehmung als auch die motorische Ausführung betreffen und willkürliche Bewegungen darstellen. Auf dieser Grundlage schlagen wir das Konzept der funktionsspezifischen Ziele vor – Ziele, die durch bildgebende Verfahren des Gehirns identifiziert werden, die auf bestimmte Funktionen abzielen. Funktionsspezifische Ziele weisen eine stärkere und umfangreichere funktionelle Konnektivität mit Gehirnregionen auf, die mit der motorischen Kognition in Verbindung stehen, und bieten möglicherweise effektivere regulatorische Effekte als die Hotspots.
Wir haben die modulatorischen Effekte von funktionsspezifischen Zielen in früheren Studien untersucht und validiert. Institutionen ohne Navigationsgeräte sind jedoch nicht in der Lage, diese funktionsspezifischen Ziele zu nutzen. Aus diesem Grund haben wir eine nicht-navigierte Lokalisierungsmethode für funktionsspezifische Ziele entwickelt, die speziell für die Definition und Lokalisierung von rTMS-Zielen in der ipsilateralen Hemisphäre nach einem Schlaganfall entwickelt wurde und die Herausforderungen adressiert, mit denen Institutionen konfrontiert sind, denen es an Navigationsgeräten mangelt, wenn funktionsspezifische zielgerichtete rTMS angewendet werden.
Einleitung
Die repetitive transkranielle Magnetstimulation (rTMS) ist eine nicht-invasive Neuromodulationstechnik, die die Gehirnaktivität regulieren kann und bei der Behandlung neuropsychiatrischer Störungen, wie z. B. bei der Rehabilitation von handmotorischen Funktionsstörungen bei Schlaganfallpatienten, weit verbreitet ist. Einige Studien haben gezeigt, dass rTMS therapeutische Wirkungen auf die Folgen eines Schlaganfalls hat 1,2,3, aber seine Wirksamkeit bleibt ungewiss. Ein Hauptgrund für diese Unsicherheit ist die Schwierigkeit, genaue Stimulationsziele zu identifizieren. TMS-Studien, die auf die motorische Funktion abzielen, stützen sich häufig auf das International 10-20 Elektroenzephalogramm-System zur Lokalisierung, wobei C3/C4 als Stimulationsziele verwendet werden, oder sie verwenden individualisierte Ziele, wie z. B. den handmotorischen Hotspot. Diese Methoden können jedoch die von TMS betroffenen kortikalen Bereiche nicht genau bestimmen. Die funktionelle Magnetresonanztomographie (fMRT)-gesteuerte, zielgerichtete rTMS wird häufig bei der Behandlung von Depressionen eingesetzt.
Unsere frühere Forschung untersuchte auch seine Anwendung bei der Behandlung des Tourette-Syndroms durch Stimulation des ergänzenden motorischen Bereichs4, aber es wurde noch nicht auf das primäre motorische Areal (M1) angewendet. Für die rTMS unterscheidet sich M1 von anderen Hirnregionen, da es den handmotorischen Hotspot enthält. Die durch TMS induzierten Muskelkontraktionen stellen unwillkürliche Bewegungen dar, die die Top-Down-Leitung durch die kortikospinalen oder pyramidalen Bahnen widerspiegeln. Im Gegensatz dazu sind die durch fMRT definierten Aktivierungsspitzenvoxel während Fingerklopfaufgaben funktionell stärker mit Gehirnregionen verbunden, die an der motorischen Kognition beteiligt sind, was willkürliche Bewegungen darstellt5. Daher kann bei der Behandlung von Bewegungsstörungen die Verwendung der durch die fMRT definierten aufgabenbezogenen "Aktivierung" als funktionsspezifische Ziele zu verbesserten therapeutischen Ergebnissen führen 5,6. In unserer früheren Arbeit haben wir die Aktivierungsmuster des Gehirns zwischen der visuell geführten Aufgabe und der selbstinitiierten Aufgabe mittels fMRT verglichen und festgestellt, dass die selbstinitiierte Aufgabe stärker mit den Anforderungen des aktiven Rehabilitationstrainings übereinstimmt6. Wir bestätigten dieses Ergebnis, indem wir eine Teilmenge von Daten aus der ursprünglichen Studie erneut analysierten (Abbildung 1).
Um bestimmte Bereiche der Gehirnfunktion genau anzusprechen, sind präzise Navigationswerkzeuge erforderlich. Die derzeitigen Systeme sind jedoch nicht nur umständlich zu bedienen und in ihrer Funktionalität eingeschränkt, sondern die am Kopf montierten Kalibratoren bleiben während der Eingriffe oft nicht stabil, neigen zu Verschiebungen und sind teuer -- manchmal kosten sie bis zu einer Million chinesischer Yuan (CNY), etwa 140.000 US-Dollar (USD). Laut einer Umfrage über das Nutzungsverhalten unter den Mitgliedsinstitutionen des Precision Medicine Consortium for Imaging-Guided Transcranial Magnetic Stimulation Therapy (PRECISE) haben diese Nachteile dazu geführt, dass Navigationstechnologien trotz ihrer potenziellen Vorteile in weniger als 5 % der TMS-Forschung und klinischen Praxis in China eingesetzt werden. Noch wichtiger ist jedoch, dass sich diese Systeme nur auf die "Lokalisierung" von Stimulationsstellen konzentrieren, ohne sich mit der kritischen Frage der "Definition" des Ziels, d.h. der Auswahl des am besten geeigneten Bereichs für die Stimulation, zu befassen. Angesichts der hohen Kosten, der betrieblichen Komplexität und des Zeitaufwands ist dies der Grund, warum diese Geräte noch keine breite klinische Akzeptanz erreicht haben.
Um der Herausforderung zu begegnen, funktionsspezifische Ziele ohne Navigationsgeräte zu verwenden, haben wir die Methode der nicht-navigierten, zielgerichteten rTMS untersucht. Mit Hilfe der fMRT identifizierten wir funktionsspezifische Ziele im motorischen Kortex und projizierten sie auf die Kopfhautoberfläche, was eine Zieldefinition und -lokalisierung ohne Navigationsgeräte ermöglichte7. Während die nicht-navigierte rTMS keine Echtzeitüberwachung während des gesamten Prozesses bietet, löst sie die Probleme der Präzision bei der Ziellokalisierung unter klinischen Bedingungen, wenn keine Navigationsgeräte verfügbar sind. In diesem Beitrag wird die allgemeine Begründung der Studie erläutert und der gesamte experimentelle Prozess skizziert, wobei der Schwerpunkt auf dem Vergleich der Auswirkungen funktionsspezifischer Ziele auf die Gehirnfunktion sowohl unter navigierten als auch unter nicht-navigierten Bedingungen liegt. Um die Machbarkeit einer funktionsspezifischen zielgerichteten rTMS zu überprüfen, wurden in der aktuellen Studie nur gesunde Personen eingeschlossen.
Protokoll
Diese Arbeit wurde von der Ethikkommission der Chengdu Sport University genehmigt, und alle Teilnehmer haben eine schriftliche Einverständniserklärung abgegeben (Abbildung 2). Dieses Protokoll beschreibt nicht-navigierte und navigierte funktionsspezifische zielgerichtete rTMS.
1. Rekrutierung von Teilnehmern
- Rekrutieren Sie 10 gesunde erwachsene Rechtshänder (22 bis 29 Jahre, davon 5 Frauen und 5 Männer; Durchschnittsalter 24 ± 2 Jahre). Ausschluss eines Teilnehmers aufgrund einer Kopfbewegung von mehr als 2,5 mm in der Translation oder 2,5 ° in der Drehung. Beziehen Sie schließlich 9 Teilnehmer in die statistische Analyse ein.
- Einschlusskriterien
- Rekrutieren Sie Teilnehmer im Alter von 18 bis 30 Jahren, die Rechtshänder sind, sowohl MRT- als auch TMS-Sicherheitsuntersuchungen bestehen und keine Epilepsie oder andere neurologische oder psychiatrische Erkrankungen in der Vorgeschichte haben.
- Stellen Sie sicher, dass die Teilnehmer zusätzliche Kriterien erfüllen, wie z. B. keine Kontraindikationen für MRT-Scans, keine Hirnverletzungen oder schwere Herzerkrankungen in der Vorgeschichte und dass sie derzeit keine Antiepileptika oder Antikoagulanzien einnehmen.
- Rekrutieren Sie Teilnehmer, die keine Bewusstseinsstörungen, keine Metallgegenstände in ihrem Körper (wie Herzschrittmacher, Zahnimplantate aus Metall oder Intrauterinpessar), keine schwere Klaustrophobie oder Schwangerschaft haben und deren Sehvermögen entweder normal oder normal korrigiert ist.
- Einschlusskriterien
- Kriterien für den Ausschluss von Daten nach dem Experiment
- Schließen Sie Daten von Teilnehmern aus, die das Experiment nicht abschließen können oder deren Kopfbewegung während der fMRT-Untersuchung 2,5 mm Translation oder 2,5° Drehung überschreitet.
- Richtlinien für Teilnehmer vor dem Experiment
- Stellen Sie sicher, dass alle Teilnehmer die Einwilligungserklärung unterschrieben haben, in der der Zweck der Forschung, die experimentellen Verfahren sowie mögliche Nebenwirkungen und Risiken erläutert werden.
- Durchführung von Sicherheitsscreenings für die Teilnehmer.
- Erläutern Sie die experimentellen Verfahren und Vorsichtsmaßnahmen, um eine reibungslose Durchführung des Experiments zu gewährleisten.
- Raten Sie den Teilnehmern, vor dem Experiment auf Alkohol, Kaffee oder kräftige Bewegung zu verzichten.
- Erinnern Sie die Teilnehmer daran, ausreichend Schlaf zu bekommen und nicht lange aufzubleiben.
2. fMRT-Datenerfassung
HINWEIS: Alle Teilnehmer werden im Zentrum für Magnetresonanz-Hirn-Bildgebung auf dem Qingshuihe-Campus der Universität für elektronische Wissenschaft und Technologie in China mit einem 3T GE MR750-Scanner einer MRT-Untersuchung unterzogen. Jede Scan-Sitzung umfasst ein T1-gewichtetes Strukturbild, eine 8-minütige fMRT im Ruhezustand (RS-fMRT) und eine 4-minütige Task-fMRT. Die Teilnehmer erhalten zwei rTMS-Interventionen: eine mit Navigation und eine ohne, mit einem Intervall von 1 Woche zwischen den Sitzungen, um Resteffekte zu eliminieren. Führen Sie vor und nach jedem Eingriff MRT-Scans durch, insgesamt vier Scans.
HINWEIS: Gleichen Sie die Reihenfolge der navigierten und nicht navigierten Bedingungen zwischen den Teilnehmern aus.
- Scan-Verfahren
- Passen Sie den Teilnehmern vor dem Betreten des MRT-Raums Ohrstöpsel an, um den Lärm zu reduzieren und sicherzustellen, dass alle Metallgegenstände entfernt werden.
- Erläutern Sie die Aufgaben, die die Teilnehmer während des Scans ausführen müssen.
- Stellen Sie sicher, dass die Teilnehmer in Rückenlage auf dem Scanbett liegen und ihre Köpfe mit Schaumstoffpolstern sicher fixiert sind, um Kopfbewegungen zu minimieren.
- Weisen Sie die Teilnehmer während des RS-fMRT-Scans an, die Augen zu schließen, absichtliches Denken zu vermeiden und wach zu bleiben, um ein Einschlafen zu verhindern.
- Exportieren Sie die Bilder manuell auf das angegebene Netzlaufwerk oder ein externes Speichergerät.
- Scan-Parameter
- Verwenden Sie die folgenden RS-fMRT-Scanparameter: Wiederholzeit (TR) = 2.000 ms, Echozeit (TE) = 30 ms, Flip-Winkel (FA) = 90°, Sichtfeld (FOV) = 220 mm × 220 mm, Matrix = 64 x 64, Schichtdicke/-spalt = 3,4 mm/0 mm, mit insgesamt 41 Schichten, die das gesamte Gehirn abdecken, und 240 gesammelten Zeitpunkten.
- Verwenden Sie die folgenden T1-gewichteten Parameter für das Scannen von Strukturbildern: Spoiled Gradient Recalled Echo (SPRG)-Sequenz, sagittaler Scan TR/TE = 8,2 ms/2,98 ms, FA = 8°, FOV = 256 mm x 256 mm, Matrix = 256 x 256, Schichtdicke/-spalt = 1 mm/0 mm, mit 166 Schichten, die das gesamte Gehirn abdecken.
- Verwenden Sie task-fMRT-Scan-Parameter, die mit denen der RS-fMRT identisch sind, mit der Ausnahme, dass nur 120 Zeitpunkte erfasst werden.
- Details zur Aufgabenausführung
- Positionieren Sie die Teilnehmer mit den Handflächen nach oben und halten Sie eine Knopfbox fest.
- Legen Sie Handtücher zwischen die Köpfe der Teilnehmer und die Spule, um ihre Köpfe zu stabilisieren und Bewegungen zu minimieren.
- Blockgestaltung, selbstinitiierte Aufgabe (4 min): Wenn ein Bild eines "+" auf dem Bildschirm erscheint, bitten Sie die Teilnehmer, sich auszuruhen. Wenn das Bild einer Uhr auf dem Bildschirm erscheint, weisen Sie den Teilnehmer an, den Knopf alle 2 Sekunden mit dem rechten Daumen zu drücken und die Zeit selbst zu messen (Ergänzende Abbildung S1).
3. Messung der Ruhemotorschwelle (RMT)
HINWEIS: Verwenden Sie die Oberflächen-Elektromyographie (EMG), um die Amplitude des motorisch evozierten Potentials (MEP) des Musculus abductor pollicis brevis (APB) aufzuzeichnen, indem Sie eine 70-mm-Achterspule verwenden, die an den Magstim Super Rapid2-Stimulator angeschlossen ist, um die RMT mit Einzelpulsstimulation zu messen.
- Entfernen Sie vor dem Test alle Metallgegenstände, um Störungen zu vermeiden und die Sicherheit zu gewährleisten.
- Lassen Sie die Teilnehmer auf einem Stuhl sitzen und sich vollkommen entspannen.
- Tragen Sie ein Peeling und 75% Alkohol auf die Hände der Teilnehmer auf.
- Platzieren Sie Silber/Silberchlorid (Ag/AgCl) Oberflächenelektroden auf dem Muskelbauch.
- Positionieren Sie die Referenzelektrode auf dem Metacarpophalangealgelenk und stellen Sie sicher, dass der Abstand zwischen den Elektroden zwischen 20 mm und 30 mm liegt.
HINWEIS: Relevante Parameter: Für die Messungen Elektroden mit einem Durchmesser von 9 mm verwenden. Das EMG-Signal des APB-Muskels wird 1.000-fach verstärkt, zwischen 20 Hz und 2,5 kHz bandpassgefiltert und dann über eine mikrodigitale Schnittstelle mit einer Abtastrate von 5 kHz digitalisiert. Die Daten werden dann auf einem Computer gespeichert und auf einem Bildschirm angezeigt. - Laden Sie das T1-Strukturbild der Person. Platzieren Sie die Spule über dem kontralateralen primären motorischen Bereich, genauer gesagt am "mittleren Knie" des zentralen Sulcus, auch bekannt als "Handknopf", der den Handbereich im primären motorischen Kortex darstellt.
HINWEIS: Bestätigen Sie die Muskelentspannung sowohl visuell als auch über die EMG-Überwachung. - Bewegen Sie die Spule in Schritten von 0,5 cm um den "Handknopf".
- Positionieren Sie den Griff in einem Winkel von 45° zur mittleren Sagittalebene, um den MEP zu messen.
- Beginnen Sie mit einer unterschwelligen Stimulationsintensität und erhöhen Sie diese jedes Mal um 5 % der maximalen Reizleistung. Wenn die Spitze-Spitze-Amplitude des MEP 50 μV überschreitet, verringern Sie die Stimulationsintensität schrittweise um 1 % der maximalen Leistung.
- Zeichnen Sie die minimale Stimulationsintensität auf, die mindestens fünf MEPs größer oder gleich 50 μV in 10 aufeinanderfolgenden Einzelpulsstimulationen als RMT hervorruft, wobei diese Stelle als Hotspot gekennzeichnet ist. Wenn nach sechs Stimulationen kein Hotspot bestimmt werden kann, bewegen Sie die Spule an die nächste Stelle.
4. Individualisierte, funktionsspezifische, zielgerichtete rTMS
- Definieren Sie das individualisierte funktionsspezifische Ziel.
- Klicken Sie nach dem Öffnen der Vorverarbeitungssoftware auf DPARSF 5.4 und wählen Sie dann DPARSF Advanced Edition , um die Aufgabenzustandsdaten mit den spezifischen Parametern vorzuverarbeiten, die in der Zusatzdatei 1 gezeigt werden. Führen Sie Slice-Timing- und Kopfbewegungskorrekturen durch. Registrieren Sie die Funktionsbilder zu Strukturbildern und wenden Sie eine räumliche Glättung mit einer vollen Breite bei halbem Maximum (FWHM) von 6 mm an.
HINWEIS: Passen Sie die spezifischen Parameter je nach Gerätemodell oder Scanaufgabe an. - Öffnen Sie SPM12 und klicken Sie auf Coregister Estimate. Wählen Sie als Referenzbild die Datei mit dem Namen "sub*crop_1.nii" aus dem Ordner T1Img aus. Wählen Sie als Quellbild die Datei "mean*.nii" aus dem Ordner RealignParameter aus. Wählen Sie für das andere Bild die Datei "ra*.nii" aus dem Ordner FunImgAR aus.
HINWEIS: Verwenden Sie die funktionale Bilddatei, die nach der Bewegungskorrektur und der Slice-Timing-Korrektur generiert wurde, als "Anderes Bild". Je nach Forschungsziel können alternative Dateien ausgewählt werden. - Klicken Sie auf Segment | Volumes und wählen Sie die Datei mit dem Namen "sub*crop_1.nii" aus dem Ordner T1Img aus. Wählen Sie für Verformungsfelder die Option Invers + Vorwärts aus, und klicken Sie dann auf Ausführen. Wiederholen Sie diesen Vorgang, um die Datei "sub*.nii" aus dem Ordner T1Img zu segmentieren.
HINWEIS: Segmentieren Sie "sub*crop_1.nii", um den Aktivierungspunkt der einzelnen Aufgaben zu berechnen. Segmentieren Sie "sub*.nii", um die Standard-Leerzeichenmaske in individuellen Raum umzuwandeln. - Klicken Sie auf Glätten, wählen Sie die Dateien "ra*.nii" aus dem FunImgAR-Ordner für die Option Image to Smooth aus und geben Sie 6 6 6 in das Feld FWHM ein.
- Führen Sie eine First-Level-Analyse durch, um individuelle Aktivierungskarten zu erhalten und das Peak-Voxel der Aktivierung als Stimulationsziel zu identifizieren. Führen Sie die folgenden drei Schritte ein:
- Erstellen Sie einen neuen Ordner mit dem Namen "indiv_act" und klicken Sie auf 1. Ebene angeben. Wählen Sie im Feld Verzeichnis den Ordner "indiv_act" aus, klicken Sie auf Einheiten für Design, wählen Sie Scans und geben Sie 2 für das Interscan-Intervall ein. Wählen Sie im Abschnitt "Data & Design " unter "Scans" die Dateien "sra*.nii" aus. Legen Sie im Abschnitt Bedingung den Namen auf tap (benutzerdefinierter Name) fest, geben Sie 0 30 60 90 für den Beginn ein, und legen Sie die Dauer auf 15 fest. Klicken Sie auf Mehrere Regressoren und wählen Sie die Datei "rp_a*.txt" aus den RealignParameters aus.
HINWEIS: Geben Sie die Informationen zu Beginn und Dauer entsprechend dem tatsächlichen Versuchsplan ein. - Schätzung: Wählen Sie unter "SPM.mat auswählen" die Datei "SPM.mat" aus dem Ordner "indiv_act" aus und generieren Sie die Aktivierungskarte der einzelnen Aufgaben, "spmT_0001".
- Klicken Sie auf Ergebnisse, wählen Sie die Datei "SPM.mat" aus dem Ordner "indiv_act" aus, aktivieren Sie den t-Kontrast und klicken Sie auf Neuen Kontrast definieren. Geben Sie einen benutzerdefinierten Namen in das Namensfeld ein, geben Sie 1 0 in das Kontrastfeld ein, klicken Sie auf Senden | OK | Fertig. Wählen Sie unter Maskierung anwenden die Option Keine aus. Wählen Sie unter P-Wertanpassung zur Steuerung die Option Keine mit dem Wert 0,001 aus. Legen Sie den Schwellenwert & extend auf 0 fest.
- Erstellen Sie einen neuen Ordner mit dem Namen "indiv_act" und klicken Sie auf 1. Ebene angeben. Wählen Sie im Feld Verzeichnis den Ordner "indiv_act" aus, klicken Sie auf Einheiten für Design, wählen Sie Scans und geben Sie 2 für das Interscan-Intervall ein. Wählen Sie im Abschnitt "Data & Design " unter "Scans" die Dateien "sra*.nii" aus. Legen Sie im Abschnitt Bedingung den Namen auf tap (benutzerdefinierter Name) fest, geben Sie 0 30 60 90 für den Beginn ein, und legen Sie die Dauer auf 15 fest. Klicken Sie auf Mehrere Regressoren und wählen Sie die Datei "rp_a*.txt" aus den RealignParameters aus.
- Klicken Sie auf Normalisieren (Schreiben) | Daten. Wählen Sie unter Verformungsfelder die Datei "iy_Crop_1" aus dem Ordner T1Img aus. Wählen Sie für Bild zum Schreiben die M1-Maske für die Gehirnregion aus. Geben Sie die einzelnen Begrenzungsrahmen- und Voxelgrößen ein.
HINWEIS: Geben Sie den Begrenzungsrahmen und die Voxelgrößen basierend auf den spezifischen Eigenschaften der Daten ein. - Klicken Sie auf Coregister (Reslice) und wählen Sie dann spmT_0001 aus dem Ordner "indiv_act" für Image Defining Space aus. Wählen Sie für Image to Reslice die Datei "w*.nii" aus, die im Schritt 4.1.6 generiert wurde.
- Berechnen Sie die Aktivierungsspitze für einzelne Aufgaben: Führen Sie in MATLAB den Sortierungs-Positivcode aus. Wählen Sie für InputName1 den Pfad der Datei "rw*.nii" aus, die in Schritt 4.1.7 generiert wurde. Wählen Sie für InputName2 den Pfad der Datei "spmT_0001" aus dem Ordner "indiv_act" aus. Wählen Sie für InputName3 den Pfad des Ausgabeordners aus. Die erste X-Koordinate mit einem negativen Wert (die linke Hemisphäre) in den sortierten Ergebnissen ist die Spitze der individuellen Aufgabenaktivierung. Notieren Sie sich die Koordinaten dieses Punktes.
- Klicken Sie nach dem Öffnen der Vorverarbeitungssoftware auf DPARSF 5.4 und wählen Sie dann DPARSF Advanced Edition , um die Aufgabenzustandsdaten mit den spezifischen Parametern vorzuverarbeiten, die in der Zusatzdatei 1 gezeigt werden. Führen Sie Slice-Timing- und Kopfbewegungskorrekturen durch. Registrieren Sie die Funktionsbilder zu Strukturbildern und wenden Sie eine räumliche Glättung mit einer vollen Breite bei halbem Maximum (FWHM) von 6 mm an.
- Suchen Sie das individualisierte funktionsspezifische Ziel (navigiert).
- Bestimmen Sie die Ausgangsintensität des Stimulators basierend auf der RMT des Teilnehmers.
- Verwenden Sie ein rahmenloses stereotaktisches optisches Tracking-Neuronavigationssystem, bei dem der Teilnehmer bequem sitzt und einen am Kopf montierten Kalibrator trägt.
- Klicken Sie auf die Option Anatomie : Importieren Sie die T1-gewichteten Strukturbilder des Teilnehmers in das Navigationssystem für die Kopfmodellierung.
- Klicken Sie auf die Option Rekonstruktion : Rekonstruieren Sie die Haut auf dem Bild.
- Klicken Sie auf die Option Orientierungspunkte : Verwenden Sie das Lokalisierungswerkzeug , um vier Orientierungspunkte (die Nase, die Nasenspitze und die präaurikulären Punkte auf beiden Seiten) auf dem Kopf zu markieren.
- Klicken Sie auf die Option Ziel : Identifizieren und etablieren Sie die Zielbahn in der Gehirnregion. Lokalisieren Sie das Stimulationsziel auf den Einzelbildern des Teilnehmers. Verschieben Sie nach der Positionierung das Ziel, um es am Fadenkreuz auszurichten. Vollständige TMS-Lokalisierung.
HINWEIS: Machen Sie die Spule tangential zur Kopfhaut und richten Sie den Stimulationsfokus auf das Ziel aus.
- Suchen Sie das individualisierte funktionsspezifische Ziel (nicht navigiert).
HINWEIS: Der gesamte Code für die Lokalisierung von Kopfhautzielen wird in Ergänzende Akte 2.- Verwenden Sie SPM12, um die Standard-Gehirnvorlage des Montreal Neurological Institute (MNI) (mni_icbm152_t1_tal_nlin_asym_09c.nii, befindet sich im Vorlagenordner von DPABI) zu segmentieren und die Standard-Kopfhautmaske zu erhalten. Die einzelnen Schritte lauten wie folgt:
- Öffnen Sie SPM12, klicken Sie auf fMRI und wählen Sie dann Segment aus dem Popup-Menü aus. Klicken Sie in der Parameterschnittstelle auf die Schaltfläche Volumes , wählen Sie die Standard-Brain-Template-Datei (d. h. die MNI-Brain-Vorlage) aus der Option Volumes aus und klicken Sie dann auf Deformationsfelder , um Inverse + Forward auszuwählen.
- Umreißen der inneren und äußeren Ränder der Standardskalp: Führen Sie in MATLAB die Codekanten aus. Wählen Sie im Popup-Fenster das Bild c5.nii aus, klicken Sie auf Fertig und generieren Sie die Datei "c5_edges.nii".
- Skizzieren Sie das äußerste Kantenbild der Standardskalp: Führen Sie in MATLAB den outer_edge Code aus. Wählen Sie in der Popup-Oberfläche die Datei c5_edges.nii aus und klicken Sie auf Fertig , um die Datei "c5_outer_edge.nii" zu generieren, die die Kopfhautgrenze im Standardraum darstellt.
- Verwenden Sie SPM12, um den Standard-Kopfhautrand wieder in einen individuellen Raum zu verwandeln. Klicken Sie in der Menüoberfläche auf Normalisieren (Schreiben) und dann in der Parameteroberfläche auf Daten. Wählen Sie unter Verformungsfelder die Datei iy_sub*.nii aus dem Ordner T1Img aus. Wählen Sie c5_outer_edge.nii für Zu schreibende Bilder aus und geben Sie den individuellen Begrenzungsrahmen und die Voxelgrößen ein.
- Kortikalkoordinaten in Kopfhautkoordinaten konvertieren: Öffnen Sie den TransCortex2Scalp-Code in MATLAB und führen Sie die erste Zeile aus. Geben Sie in der Popup-Oberfläche die Koordinaten der einzelnen Aufgabenaktivierungspunkte ein und wählen Sie die Datei wc5_outer_edge.nii aus. Notieren Sie die Koordinaten der Kopfhaut.
- Öffnen Sie DPABI_Viewer, klicken Sie auf Unterlage und wählen Sie das individuelle T1-Strukturbild aus. Lokalisieren und notieren Sie die Koordinaten der vier Orientierungspunkte: die linke und rechte Ohrspitze, die Nasion und das Inion.
- Definieren Sie den Ursprung der Kopfhaut: Öffnen Sie den Schnittpunktcode in MATLAB. Geben Sie im Editor die Koordinaten der vier Orientierungspunkte an den angegebenen Positionen ein. Führen Sie den Code aus, um die Schnittpunktkoordinaten der Linie zu berechnen, die die linken und rechten Ohrspitzen mit der Linie verbindet, die das Nasen- und das Inion verbindet, und notieren Sie dann die Koordinaten.
- Verschieben Sie den Schnittpunkt entlang der Z-Achse auf die Kopfhaut: Öffnen Sie den Ursprungscode in MATLAB. Geben Sie die Koordinaten des Schnittpunkts an der Position Punkt H definieren im Editor ein. Führen Sie den Code aus und wählen Sie dann die Datei wc5_outer_edge.nii im Popup-Fenster aus, um die Ursprungskoordinaten O der Kopfhaut abzurufen.
- Zeichnen Sie eine Linie, die die beiden Ohrspitzen verbindet, um die X-Achse zu definieren, und eine Linie, die das Nasenlicht und den äußeren Hinterhauptsvorsprung verbindet, um die Y-Achse zu definieren. Die Achse senkrecht zu beiden definiert die Z-Achse. Die zweidimensionale Ebene, die von der XY-Achse gebildet wird, ist die XY-Ebene.
- Berechnen Sie die tatsächliche Entfernung vom Skalp-Ursprung O zu jedem Punkt: Führen Sie den Entfernungscode aus, wählen Sie die wc5_outer_edge-Datei in der Popup-Oberfläche aus und folgen Sie im Befehlsfenster den Anweisungen, um den Skalp-Ursprung, das Skalp-Ziel und die vier Orientierungspunkte einzugeben.
HINWEIS: "Jeder Punkt" bezieht sich auf die vier Orientierungspunkte der Kopfhaut in Schritt 4.3.4 und das Kopfhautziel. Mit diesem Code kann jeweils nur der Bogenabstand zwischen einem Punkt und einem anderen berechnet werden. Um den Abstand zwischen einem anderen Punktpaar zu berechnen, müssen Sie den Code erneut ausführen. - Berechnen Sie den Winkel zwischen der Linie, die das Scalp-Ziel mit dem Scalp-Ursprung verbindet, und der X-Achse in der XY-Ebene: Öffnen Sie den Code calculate_angle_X_axis und führen Sie die erste Zeile aus. Geben Sie im Befehlsfenster die Koordinaten des Ursprungs der Kopfhaut und des Stimulationsziels ein, wenn Sie dazu aufgefordert werden.
- Verwenden Sie das Ziellineal (wie in Abbildung 3 gezeigt), um die entsprechende weiche Linealposition basierend auf der in den vorherigen Schritten berechneten Entfernung und dem Winkel zu fixieren. Markieren Sie die Kopfhaut mit einem abwaschbaren Stift. Schließen Sie die Lokalisierung des Kopfhautstimulationsziels ab (Abbildung 4).
- Verwenden Sie SPM12, um die Standard-Gehirnvorlage des Montreal Neurological Institute (MNI) (mni_icbm152_t1_tal_nlin_asym_09c.nii, befindet sich im Vorlagenordner von DPABI) zu segmentieren und die Standard-Kopfhautmaske zu erhalten. Die einzelnen Schritte lauten wie folgt:
- rTMS
- Wählen Sie den Wiederholungsmodus, um die Stimulationsparameter einzustellen, einschließlich Stimulationsintensität, Frequenz (10 Hz), Dauer (3 s), Anzahl der Impulse in jedem Zug (30 Impulse), Wartezeit (12 s), Anzahl der Züge (60 Züge) und Gesamtzahl der pro Tag abgegebenen Impulse (1.800 Impulse).
- Speichern Sie die Sitzung und drücken Sie die Run-Taste, um die Stimulation zu starten.
HINWEIS: Die Stimulationsintensität wird entsprechend der RMT des Teilnehmers eingestellt, die in dieser Studie auf 100% RMT eingestellt ist. - Lassen Sie den Teilnehmer innerhalb einer halben Stunde nach Beendigung der Stimulation einer weiteren MRT-Untersuchung unterziehen, wobei die gleiche Scansequenz wie vor der Stimulation verwendet wird.
5. Nachweis des modulatorischen rTMS-Effekts (Verarbeitung und Analyse von MRT-Daten)
HINWEIS: Verwenden Sie eine Vorverarbeitungssoftware, um die RS-fMRT-Datenvorverarbeitung durchzuführen, die die folgenden Schritte umfasst:
- Entfernen Sie die ersten 10 Zeitpunkte, um ein Signalgleichgewicht zu erreichen und den Teilnehmern die Anpassung an das Scannerrauschen zu ermöglichen.
- Korrigieren Sie die Erfassungszeitverzögerung zwischen den Slices.
- Führen Sie eine Korrektur der Kopfbewegung durch.
HINWEIS: Je nach den spezifischen Studienanforderungen können unterschiedliche Grenzwerte für die Kopfbewegung festgelegt werden. - Normalisieren Sie die Funktionsbilder mithilfe der EPI-Vorlage in den MNI-Raum.
- Regression von störenden Signalen, einschließlich derjenigen aus der weißen Substanz, der Zerebrospinalflüssigkeit und sechs Kopfbewegungsparametern.
- Entfernen Sie lineare Trends.
- Wenden Sie eine Bandpassfilterung (0,01-0,1 Hz) an.
- Führen Sie eine räumliche Glättung mit einem Gaußschen Kern mit einer FWHM von 6 mm durch.
- Berechnen Sie Gehirnaktivitätsmetriken nach der Vorverarbeitung, einschließlich der Amplitude der niederfrequenten Fluktuation (ALFF) und der funktionellen Konnektivität (FC). Berechnen Sie die Unterschiede in den lokalen Hirnaktivitätsmetriken (ALFF und FC) zwischen prä-rTMS und post-rTMS sowohl unter navigierten als auch unter nicht-navigierten Bedingungen und führen Sie gepaarte t-Tests auf den Differenzkarten durch (GRF-Korrektur, Voxel p < 0,001, Cluster p < 0,05).
Ergebnisse
Die Ergebnisse des gepaarten t-Tests und der bidirektionalen ANOVA zeigten, dass es keine signifikanten Unterschiede in den Veränderungen von ALFF oder FC vor und nach rTMS sowohl unter navigierten als auch unter nicht-navigierten Bedingungen gab (GRF-Korrektur, Voxel p < 0,001, Cluster p < 0,05). Es wurden keine signifikanten Unterschiede zwischen den Navigations- und Nicht-Navigationsbedingungen beobachtet. Dieses Ergebnis stimmt mit unseren Erwartungen überein und deutet darauf hin, dass unsere Nicht-Navigationsmethode keinen wesentlichen Nachteil gegenüber der Navigationsmethode hat. Um zu vermeiden, dass unbewiesene Behauptungen aufgestellt werden, dass es keine signifikanten Unterschiede gibt, präsentieren wir hier die Ein-Stichproben-t-Test-Karten für beide rTMS-Bedingungen (unkorrigiert, Voxel p < 0,05) (Abbildung 5). Diese Ergebnisse überstehen keine Art von Mehrfachvergleichsanpassung, wie z. B. FDR- oder GRF-Korrektur. Um die Äquivalenz von Veränderungen der Gehirnfunktion zu beurteilen, die durch die nicht-navigierte und die navigierte Methode induziert wurden, wurde eine Power-Analyse mit Cohens d durchgeführt. Die Ergebnisse zeigten, dass der Cohen-d-Wert für ALFF 0,22 betrug, während der Cohen-d-Wert für FC 0,56 betrug.
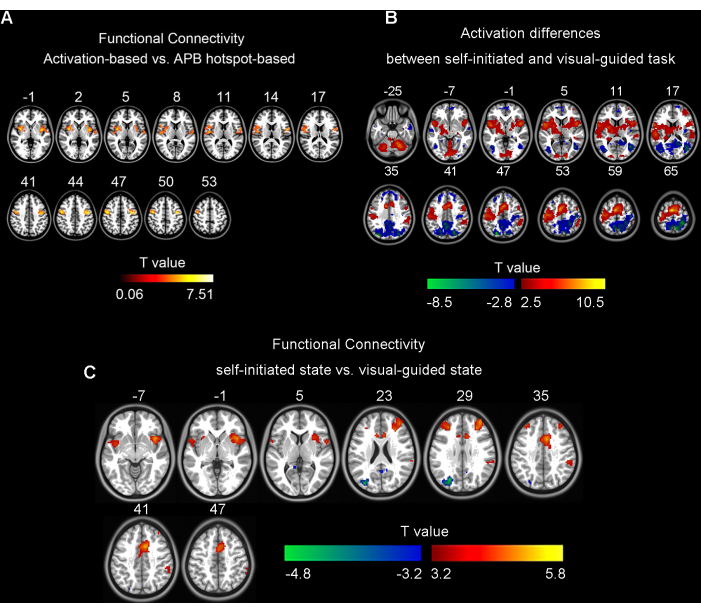
Abbildung 1: Ergebnisse für gepaarte t-Tests. (A) Die Unterschiede zwischen aktivierungsbasierter und APB-Hotspot-basierter funktionaler Konnektivität (GRF-Korrektur, Einzelvoxel p < 0,001, Clusterebene p < 0,05). (B) Die Unterschiede in der Gehirnaktivierung zwischen selbstinitiierten und visuell gesteuerten Fingerklopfaufgaben bei 25 Teilnehmern (FDR-Korrektur, Frage < 0,05). (C) Die Unterschiede zwischen selbstinitiierter und visuell geführter Zustandsaktivierungs-basierter funktionaler Konnektivität bei 35 Teilnehmern (GRF-Korrektur, Einzelvoxel p < 0,001, Cluster p < 0,05). Abbildung 1A wurde von Wang et al. (2020)5 übernommen; Abbildung 1B,C wurden erstellt, indem eine andere Teilmenge von Daten aus Wang et al. (2023)6 extrahiert wurde. Abkürzungen: APB = Abductor Pollicis Brevis; GRF = Gaußsches Zufallsfeld; FDR = Falschentdeckungsrate. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
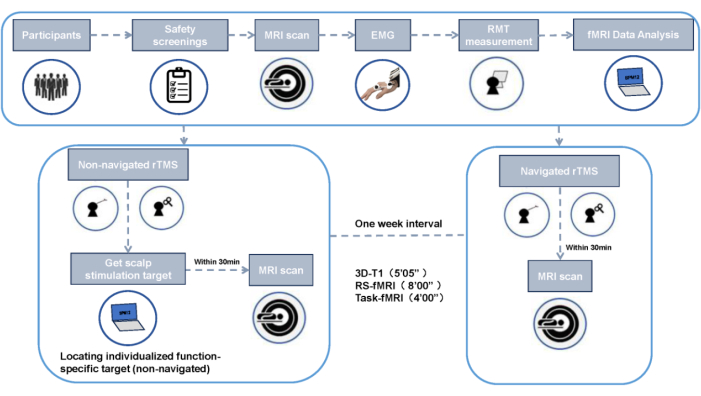
Abbildung 2: Flussdiagramm des experimentellen Designs. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
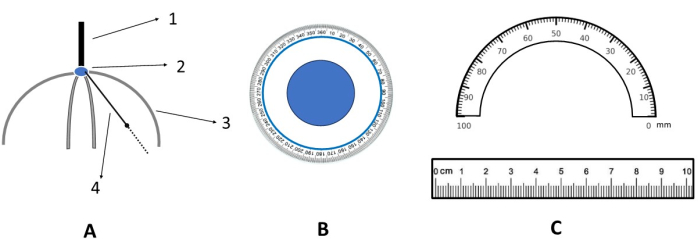
Abbildung 3: Schematische Darstellung des Targeting-Lineals. (A) Vorderansicht des Targeting-Lineals. 1. Handhaben; 2. Ankerpunkt der Kopfhaut (d. h. der Ursprung der Kopfhaut in der XY-Ebene); 3. Starres Messlineal (Acrylmaterial); 4. Drehbares und flexibles Messlineal (Silikonmaterial). (B) Vergrößerte Ansicht des Ankerpunkts der Kopfhaut (d. h. eine vergrößerte Ansicht von 2 in A). (C) Vergrößerte Ansicht des flexiblen Messlineals (d. h. vergrößerte Ansichten von 3 und 4 in A). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 4: Umwandlung des funktionsspezifischen kortikalen Ziels in das funktionsspezifische Kopfhautziel. Der rote Punkt stellt das funktionsspezifische kortikale Ziel dar, der grüne Punkt stellt das funktionsspezifische Kopfhautziel dar und der blaue Punkt zeigt den Ursprung des 2D-Koordinatensystems auf der Kopfhaut an. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
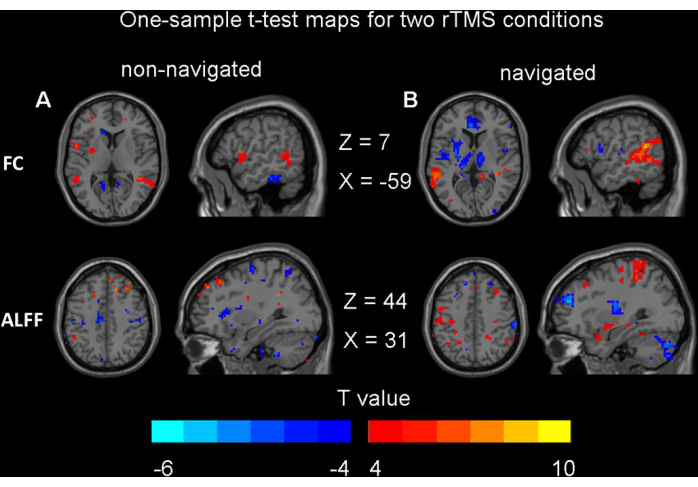
Abbildung 5: Ergebnisse für t-Tests bei einer Stichprobe. (A) Nicht navigierte rTMS-modulatorische Effekte auf die Gehirnfunktion (p < 0,05, unkorrigiert). (B) Navigierte modulatorische rTMS-Effekte auf die Gehirnfunktion (p < 0,05, unkorrigiert). Abkürzungen: FC = funktionale Konnektivität; ALFF = Amplitude der niederfrequenten Fluktuation; rTMS = repetitive transkranielle Magnetstimulation. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergänzende Datei 1: Parameter, die in der DPARSF Advanced Edition verwendet werden, wie in Protokollabschnitt 4.1.1 erwähnt. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Datei 2: Der Zip-Ordner mit dem in dieser Studie verwendeten MATLAB-Code. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Abbildung S1: Selbstinitiierte Fingerklopfaufgabe. Die Aufgabe bestand aus acht Blöcken mit einer Dauer von jeweils 30 s, was zu einer Gesamtlänge von 4 Minuten führte. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
In dieser Studie schlagen wir das Konzept der funktionsspezifischen Ziele vor, d. h. Gehirnregionen, die mit spezifischen Funktionen assoziiert sind, die durch bildgebende Verfahren identifiziert wurden. Inspiriert von früheren Studien 8,9,10 haben wir ein neues Toolkit 7,11,12 zur Lokalisierung von Kopfhautzielen entwickelt, die funktionsspezifischen kortikalen Bereichen entsprechen und eine funktionsspezifische gezielte rTMS ermöglichen, ohne dass Navigationsgeräte erforderlich sind. Im Vergleich zur Stimulation mit Navigationsgeräten wurden keine signifikanten Unterschiede in den Auswirkungen auf die Gehirnfunktion beobachtet. Dies deutet darauf hin, dass unser Verfahren in bestimmten Fällen eine individualisierte, funktionsspezifische, zielgerichtete rTMS erreichen kann, ohne dass teure Navigationsgeräte erforderlich sind.
Wesentliche Schritte im Versuchsprotokoll
Um die Genauigkeit der nicht navigierten rTMS-Lokalisierung zu gewährleisten, muss der Bediener die Skala auf dem Ziellineal an den Landmarken für das linke und rechte Ohr, dem Nasion und dem Inion ausrichten. Die Waage sollte fest gegen die Kopfhautoberfläche gedrückt werden, um Messfehler durch die Haardicke zu minimieren. Dieser Prozess ist entscheidend für die Verbesserung der Lokalisierungsgenauigkeit und die Gewährleistung einer präzisen Ausrichtung der Stimulationsstelle.
Verbesserungen der experimentellen Methode und mögliche technische Probleme
Da es sich bei dieser Methode um eine weiterentwickelte Version einer zuvor entwickelten Technik11 handelt, wurden bisher keine verbesserungswürdigen Bereiche identifiziert. Was mögliche technische Probleme betrifft, so können individuelle Unterschiede in der Schädelform bei einigen Teilnehmern zu weniger ausgeprägten Hinterhauptsprotuberanzen führen, was zu Lokalisationsfehlern führen kann. In solchen Fällen kann die Hinterhauptsprotuberanz weggelassen werden, und andere Landmarken (wie die Markierung des linken und rechten Ohrs und des Nasen) können zur Lokalisierung verwendet werden, ohne die Genauigkeit zu beeinträchtigen, da die Redundanz bereits in der Entwicklungsphase berücksichtigt wurde.
Einschränkungen der nicht-navigierten rTMS-Methode
Der Hauptunterschied zur navigierten rTMS besteht darin, dass die relative Entfernung und Richtung der Spule zum Stimulationsziel nicht in Echtzeit überwacht werden kann. Doch selbst mit navigiertem rTMS erfordert die Echtzeitüberwachung immer noch erfahrene Bediener, die manuelle Anpassungen vornehmen.
Bedeutung der experimentellen Methode im Vergleich zu bestehenden Methoden
Im Vergleich zu Navigationsgeräten erfordert unsere Methode keine langwierige Positionierung oder Gerätekalibrierung. Stattdessen geben die Nutzer einfach MRT-Daten in das Code-Skript ein und berechnen dann per Code die entsprechenden Entfernungen, woraufhin die Positionierung mit einem Messwerkzeug schnell abgeschlossen ist. Nach unseren Erfahrungen spart diese Methode mindestens 15 Minuten im Vergleich zu den aufwendigen Abläufen bei der Navigation. Navigationsgeräte erfordern in der Regel teure Hardware und spezielle Schulungen, während unsere Methode nur MRT-Bilder und Standardberechnungen benötigt, um eine schnelle, bequeme und präzise Lokalisierung zu erreichen, wodurch sowohl die Vorlaufkosten als auch die betriebliche Komplexität erheblich reduziert werden.
Was die Kosten betrifft, so wurde unserem Messgerät ein Erfindungspatent (ZL202411874788.9)12. erteilt, das zum Schutz des geistigen Eigentums beiträgt, aber die Produktionskosten nicht wesentlich erhöht. Die 3D-Modellierung ist derzeit im Gange, und wir werden bald in der Lage sein, das Tool für unsere klinischen Mitarbeiter in 3D zu drucken. Kostenüberlegungen wurden von Anfang an in die Entwurfsphase integriert. Für Nicht-Kollaborateure, die das Tool erwerben möchten, beträgt der Preis nur 500 CNY (ca. 70 USD), der trotz des Patentschutzes erschwinglich bleibt.
Bedeutung und Einsatzmöglichkeiten der Methode in spezifischen Forschungsfeldern
Die Intervention und Behandlung von rTMS hat in den letzten Jahren sowohl in der Forschung als auch im klinischen Bereich immer mehr an Popularität gewonnen. Wie bei allen Therapieverfahren geht die Entwicklung in Richtung präziser, individualisierter Behandlungen, die auf bestimmte Funktionen abzielen. Navigationssysteme und -geräte sind jedoch teuer, und die meisten Krankenhäuser in China haben derzeit keinen Zugang zu solchen Geräten. Diese Methode adressiert das Problem der individualisierten, funktionsspezifischen, zielgerichteten rTMS, ohne dass eine Navigation erforderlich ist. Es projiziert kortikale Zielkoordinaten auf die Kopfhaut und verwendet ein Werkzeug, um die Koordinaten auf der Kopfhautoberfläche zu markieren. Die fMRT-basierte kortikale Targeting-Methode, die bei diesem Ansatz verwendet wird, ist identisch mit den fMRT-Zielkoordinaten, die international von Navigationssystemen und -geräten verwendet werden. Obwohl es den relativen Abstand und die Richtung zwischen der Spule und dem Stimulationsziel nicht in Echtzeit überwachen kann, bietet es dennoch Vorteile gegenüber aktuellen klinischen "Blind Targeting"-Methoden (z. B. die Verwendung von anatomischen Landmarken auf der Hautoberfläche oder die Auswahl des handmotorischen Hotspots). Diese Methode dient als Übergangsansatz zwischen präziser Echtzeit-Navigation und "Blind Targeting". Für klinische Einrichtungen ohne Navigationssysteme und -geräte kann es praktische klinische Probleme lösen. Diese Methode wird die fMRT-gesteuerte TMS-Präzisionsbehandlung erheblich fördern, was zur Entdeckung effektiverer Stimulationsziele und zur Verbesserung der Wirksamkeit von Behandlungen für verschiedene neurologische und psychiatrische Erkrankungen führt.
Offenlegungen
Die Autoren haben keine Interessenkonflikte anzugeben.
Danksagungen
Diese Studie wurde vom Sichuan Province Science and Technology Support Program (Nr. 2024ZYD0189) unterstützt. Die Autoren danken dem PREcision medicine Consortium for Imaging-guided transcranial magnetic Stimulation thErapy (PRECISE) für die professionelle Beratung.
Materialien
| Name | Company | Catalog Number | Comments |
| Brainsight Neuronavigation system | Rogue Research Inc. | KITBSF0104 | |
| DPABI_V7.0 toolkit | DeepBrain | for RS-fMRI and task-based fMRI data analysis | |
| Magstim Rapid2 | The MAGSTIM Company Limited | 3012-00 | |
| SPM12 (7771) | Wellcome Centre for Human Neuroimaging | for RS-fMRI and task-based fMRI data analysis | |
| The Brainsight 2 channel electromyography acquisition device | Rogue Research Inc. | NTBX001001 |
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten