Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
La investigación de los tejidos y órganos específicos de las respuestas fitocromo usando FACS asistida por células de tipo Perfiles de expresión específica en Arabidopsis thaliana
En este artículo
Resumen
La base molecular de las respuestas específicas de espacio-fitocromo está siendo investigado el uso de plantas transgénicas que presentan los tejidos y órganos específicos deficiencias fitocromo. El aislamiento de células específicas exhibir agotamiento inducido cromóforo fitocromo por fluorescencia de células activadas clasificación seguido por análisis de microarrays se está utilizando para identificar genes implicados en el espacio-las respuestas específicas de fitocromo.
Resumen
Media luz una serie de procesos de desarrollo y de adaptación a través del ciclo de vida de una planta. Las plantas utilizan la luz que absorben las moléculas llamadas fotorreceptores para percibir y adaptarse a la luz. El rojo / rojo lejano que absorbe la luz fotorreceptores fitocromo se han estudiado ampliamente. Fitocromos existir como una familia de proteínas con diferentes funciones y la superposición de todos los sistemas de plantas superiores en las que se han estudiado
Protocolo
1. Crecimiento de las Plantas
- Confirmó UAS-GAL4 BVR X-GFP línea de trampas potenciador aislado como se describe 4 (Para un resumen ver fig. 1) y las líneas de tipo salvaje o de los padres se siembran en el suelo, es decir, ~ 2000 semillas esterilizadas por línea.
- Las plantas son cultivadas durante 5 semanas en el suelo bajo una iluminación blanca de 100 μmolm -2 s -1 a 22 ° C y 70% de humedad.
2. Hoja de aislamiento de protoplastos (adaptado de Denecke Vitale y 9)
- Para aislar protoplastos, preparar TEX buffer. Para 1 litro de amortiguación TEX, pese a cabo los siguientes componentes: 3,1 g Gamborg de sales B5, 0,5 g de 2 - (N-morfolino) etanosulfónico (MES, 2,56 mM), 0,75 cloruro de calcio dihidrato (CaCl 2 .2 H 2 O, 6.75 mM), 0,25 g de nitrato de amonio (NH 4 NO 3, 3,12 mM), 136,9 g de sacarosa (0,4 M). Disolver los componentes por completo en ~ 900 ml de agua desionizada, destilada (DDC 2 O) y llevar el pH a 5.7 con 1 M KOH. Alcanzar un volumen final de 1 litro y esterilizar con un filtro de 0,2 micras tapa de la botella-filtro conectado a una bomba de vacío.
- Recoger las hojas verdes y saludables de las plantas (~ 250 ml de hojas muy apretadas en un vaso de precipitados) y enjuague con ~ 40 ml ddH2O 4 veces, seguido de lavado dos veces con agua estéril ddH 2 O. El uso de un bisturí # 20, las hojas cortadas en tiras finas y dividir el tejido en partes iguales en dos tubos de 50 ml de plástico estéril. Prepare 1x mezcla de digestión de la hoja mediante la adición de 5 ml de alícuota de solución madre 10 veces la hoja de la digestión (digestión 10x hoja de solución de reserva: disolver 2% w / v Macerozyme R-10, 4% w / v celulasa "Onozuka" R-10 en TEX buffer, filtro de esterilización con filtro de 0,2 micras y la congelación de alícuotas de 5 ml a -80 ° C) y 45 ml de tampón fresco TEX. Añadir ~ 25 ml de la mezcla de digestión 1x hoja por cada 50 ml tubo para cubrir todo el tejido de la hoja.
- Vacío infiltrado en el tejido de la hoja en la mezcla de digestión en tubos abiertos durante 1 hora a temperatura ambiente usando un secador de vacío conectado a una bomba de agua, seguida por 3 horas de incubación a temperatura ambiente en un eje de balancín con agitación suave. Tubos tapados con tejido de la hoja en la mezcla de digestión 1x hoja se mantienen envueltas en papel de aluminio durante el proceso para prevenir la exposición a la luz.
- Después de 3 horas, aumentar la velocidad del eje de balancín de ~ 2 minutos para liberar protoplastos. Filtrar la suspensión de protoplastos crudo a través de dos capas de gasa estéril para eliminar los residuos y recoger el filtrado en un vaso de vidrio estéril. Filtrar el filtrado a través de una malla de nylon de 100 micras estéril en una placa de Petri estéril. Recoge el flujo a través y la transferencia a un nuevo tubo estéril de 50 ml. El uso de 15-20 ml de tampón fresco TEX para lavar la placa de Petri estéril y recoger los protoplastos de adherirse a la superficie.
- Centrífuga del flujo mediante el uso de un rotor basculantes en 100xg a 10 ° C durante 15 minutos (6 de aceleración, desaceleración 0).
- Quitar ~ 25 - 30 ml del líquido por debajo de la capa de protoplasma flotante, que contiene los desechos residuales y pellets, con una estéril 9 "pipeta Pasteur de vidrio conectado a una bomba peristáltica sin remover la capa flotante de protoplastos y dejando a ~ 10 a 15 ml de volumen.
- Añadir nuevo buffer TEX a un volumen final de 40 ml, mientras que suavemente resuspensión de los protoplastos.
- Repita los pasos 2,5 a 2,7 dos veces para eliminar los desechos celulares tanto como sea posible. El tiempo de centrifugación se reduce a 10 minutos de la primera repetición y a 5 minutos en la repetición final.
- Aspirar protoplastos flotante con un corte, una pipeta de transferencia estéril ml en un tubo de 15 ml nuevos. Proceder directamente a su clasificación o almacenamiento en la oscuridad durante un máximo de 2 horas a 4 ° C hasta su ordenación.
3. Protoplastos clasificación por la selección de células activadas por fluorescencia (FACS)
- Antes de ordenar, analizar protoplastos por microscopía de escaneo láser confocal (CLSM) utilizando un láser de 488 nm para la excitación para confirmar la integridad de protoplastos y la presencia de la fluorescencia de GFP en la piscina de protoplastos. Los residuos es necesario un mínimo para evitar la obstrucción de la boquilla de clasificación FACS.
- Ordenar aislado protoplastos en TEX buffer mediante FACS (BD FACSVantage SE, BD Biosciences) utilizando una boquilla de 200 micras en una cabeza de tipo macro en las tasas de eventos entre 6.000 y 15.000, con un sistema de presión de alrededor de 9 psi a raíz de un protocolo de adaptación 10.
- De tipo salvaje GFP no protoplastos se utilizan para determinar los umbrales de autofluorescencia.
- Para recoger las buenas prácticas agrarias-positivas protoplastos, células usando un tipo refrigerado por aire con láser de argón (Spectra Physics modelo 177, Newport Corporation, Irvine, CA), operado a 100 mW en una línea de 488-nm argón para identificar la fluorescencia de GFP con una banda 530/30 filtro de paso.
- Tras la recogida de GFP-positivas protoplastos, examinar protoplastos ordenados por CLSM como se detalla en el paso 3.1.
- Extraer el ARN total a partir de protoplastos ordenados mediante un Qiagen RNeasy Plant Mini Kit. cDNA se puede preparar para la hibridación como se describe en Affymetrix GeneChip Expresión Manual de Análisis Técnico y luego hibridizado a ATH1 Arabidopsis arrays Affymetrix todo el genoma.
Resultados representante
Se detectó un número significativo de las buenas prácticas agrarias de células positivas en el canal de GFP por FACS (Fig. 2). En los ensayos de optimización de uso de las buenas prácticas agrarias potenciador trampa de los padres, la línea constitutivamente expresan GFP, J0571, muestra ~ 17% a 24% positivas GFP-protoplastos, mientras que la línea con la expresión vascular y cutánea, J1071, había ~ 1,4% GFP-positivas protoplastos (Tabla 1). Clasificación de 3 x 500 l (alrededor de 1,5 ml de la suspensión total de protoplastos) de J0571 protoplastos durante 1 hora dio ~ 100.000 GFP-positivas protoplastos. Clasificación de 3 x 500 l (alrededor de 1,5 ml de suspensión de protoplastos total) de J1071 protoplastos durante 1,5 horas dieron positivo ~ 3.000 GFP-protoplastos. Confocal de imágenes indica un rendimiento muy alto de protoplastos de ambas muestras J0571 J1071 y antes de la clasificación se llevó a cabo (Fig. 3C y 3E), y las fracciones contenidas ordenados única luz fluorescente GFP-protoplastos (Fig. 3G y datos no presentados). Este hallazgo confirma que intacta positivas GFP-protoplastos pueden ser ordenados a través de la FACS. Los protoplastos no las buenas prácticas agrarias también pueden ser ordenados y recogidos en un canal separado. Los protoplastos no GFP servir como un control ideal negativo para posteriores análisis de microarrays para detectar los cambios específicos en la expresión génica en la reducción de los niveles de holophytochrome. La extracción de RNA a partir de protoplastos aislados (1 ml) de ARN produce la cantidad suficiente para la detección de fluorospectrometry (Fig. 4). ARN aislado se evaluó mediante un instrumento NanoDrop (NanoDrop 1000, Thermo Scientific) y se cuantifica por NanoDrop 3.7.1 software de cuantificación del ARN. Los rendimientos de ARN a partir de pre-ordenados C24 de tipo salvaje protoplastos o FACS-ordenados, GFP-positivas protoplastos trampa potenciador superado el mínimo de 20 ng necesarios para su uso en el ARN de etiquetado para los ensayos de microarrays (Tabla 2).
| Potenciador trampa Línea | % De GFP-positivas protoplastos | Número de protoplastos GFP ordenados |
| J0571 | 17,18% 24,06% ~ | 26400 ~ 36000 |
| J1071 | ~ 1,43% | 1000 ~ 1300 |
Tabla 1. Porcentaje de GFP-positivas protoplastos a partir de dos líneas de trampa potenciador antes de la clasificación y el número de GFP-positivas protoplastos recolectados mediante la ejecución de 500 l de suspensión de protoplastos a través del Clasificador de fluorescencia de células activadas (FACSVantage, BD).
| Planta de línea | ARN rendimiento (ng) |
| C24 PESO | 12,486.5 |
| J0571 | 60 |
| J1071 | 71.5 |
Tabla 2. Rendimiento de aislamiento de ARN a partir de protoplastos. Se aisló el ARN de pre-ordenados C24 de tipo salvaje o ordenados GFP-positivas protoplastos a partir de dos líneas de trampa potenciador y cuantificados.
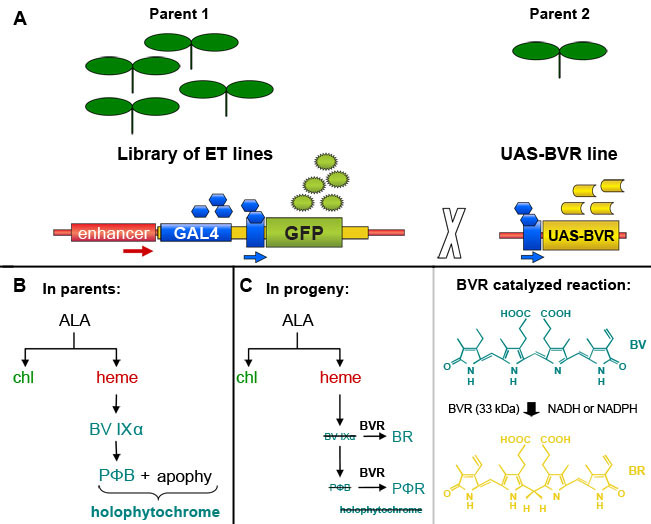
Figura 1. GAL4 potenciador trampa basada en la inducción de la biliverdina reductasa (BVR) la expresión en plantas transgénicas de Arabidopsis thaliana. (A). Un individuo seleccionado de una biblioteca de GAL4 basada en líneas de trampa potenciador, que contienen un gen GFP GAL4 sensible marcador, se puede cruzar con una línea que contiene un gen diana GAL4 sensible de inducir la expresión del gen diana en GAL4 que contienen las células marcadas por la fluorescencia de GFP. Sobre la base de una figura del Dr. Jim Haseloff (http://www.plantsci.cam.ac.uk/Haseloff/geneControl/GAL4Frame.html). (B) Producción de fitocromo cromóforo, phytochromobilin (PΦB) y holophytochrome en las líneas parentales. (C) A la izquierda, la reducción de la biliverdina IXα (BV IXα) y PΦB por la biliverdina reductasa (BVR) la actividad de BR y PΦR, respectivamente. Resultados BVR actividad en el agotamiento de los PΦB y conduce a una reducción en la producción de fotoactivos holophytochrome. Derecha, la reacción catalizada por la BVR se muestra.
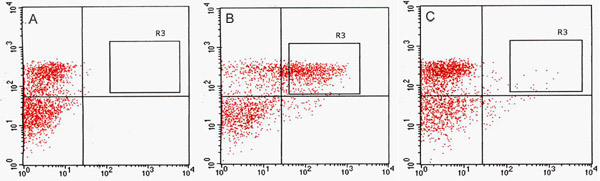
Figura 2. Fluorescencia clasificación de células activadas (FACS) representa la adquisición de puntos. Comparación de la célula de protoplastos de clasificación de C24 de tipo salvaje (A), J0571 (B) y J1071 (C). (A) Adquisición de diagrama de puntos de no-GFP fluorescentes C24 de tipo salvaje protoplastos excitado por un láser de 488 nm de argón y se utiliza para determinar el umbral de autofluorescencia. (B) y (C) representa la adquisición de puntos muestran las proporciones de los protoplastos de las buenas prácticas agrarias que son positivos en respuesta a la excitación de un láser de 488 nm. R3 puertas de la clasificación en B y C delimitar los objetivos de las buenas prácticas agrarias-positivas que fueron ordenados por clasificador de células activadas por fluorescencia (FACSVantage, BD) y se recoge. Canal rojo indica valores de autofluorescencia de clorofila a partir de protoplastos y el canal verde indica que los valores de la fluorescencia de GFP.

Figura 3. Microscopía confocal de protoplastos de plantas utilizadas para la clasificación de la célula. Láser confocal de imágenes escaneadas de protoplastos antes (A, C, E) y después (G) la clasificación a través de clasificador de células activadas por fluorescencia (FACS). B, D, F y H son imágenes DIC. Protoplastos de C24 de tipo salvaje (A, B), J1071 (C, D) y J0571 (E, F, G, H) se muestran. Imágenes C, E y G se combinan imágenes de fluorescencia de GFP (BP 505 nm - 575 nm) y autofluorescencia (LP 650 nm) obtenidos a partir de la excitación con un láser de 488 nm. A través de H están en la media de 4 exploraciones de petróleo en 63x. Bar = 10 micras.
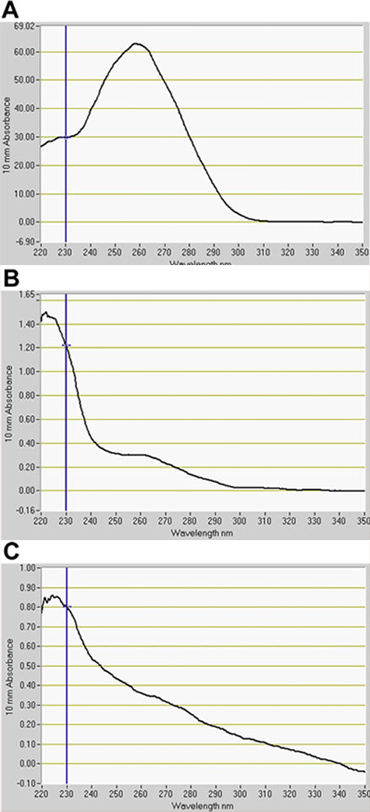
Figura 4. Cuantificación de ARN aislado a partir de protoplastos por fluorospectrometry. ARN extraído de (A) tipo C24 salvajes, (B) J0571, J1071 y (C). A indica ARN para la pre-ordenados de tipo salvaje protoplastos. B y C indican ARN de GFP-positivas protoplastos ordenados por la selección de células activadas por fluorescencia (FACS). Software de cuantificación del ARN, NanoDrop 3.7.1, (NanoDrop 1000, Thermo Scientific).
Discusión
Perfiles de expresión génica mediante microarrays (1) ha indicado que más del 30% de los genes en plantas de Arabidopsis se regula la luz 11 y (2) ha identificado un vasto grupo de genes que codifican proteínas de transducción de señales de luz que participan en la cascada de señalización fitocromo 12, 13 . Estos experimentos sugieren que la luz induce cambios rápidos y de largo plazo en la expresión génica. Cada grupo de fitocromos puede controlar sólo un subconjunto de respuestas de d...
Agradecimientos
El trabajo en el laboratorio de Montgomery sobre las respuestas de fitocromo en las plantas es apoyada por la National Science Foundation (subvención no. MCB-0919100 de BLM) y Ciencias Químicas, Geociencias y Ciencias Biológicas de la División de la Oficina de Ciencias Básicas de Energía, Oficina de Ciencia, EE.UU. Departamento de Energía (concesión no. FG02 DE 91ER20021 a BLM). Damos las gracias a Melissa Whitaker para la asistencia técnica durante el rodaje y la lectura crítica del manuscrito, Stephanie Costigan de asistencia experimental, el Dr. Luis Rey de asistencia con el desarrollo y la optimización de fluorescencia de células activadas clasificación protocolos para la clasificación de protoplastos de Arabidopsis y Marco Dra. Melinda ayuda con confocal microscopía. Damos las gracias a Marlene Cameron para asistencia en el diseño gráfico y de aves Karen por su asistencia editorial.
Materiales
| Name | Company | Catalog Number | Comments |
| Anti-BVR antibody | QED Bioscience Inc. | 56257-100 | |
| Cellulase “Onozuka” R-10 | SERVA Electrophoresis | MSPC 0930 | |
| Gamborg’s B5 basal salt mixture | Sigma-Aldrich | G5768 | |
| Macerozyme R-10 | SERVA Electrophoresis | PTC 001 | |
| MES, low moisture content | Sigma-Aldrich | M3671 | |
| Murashige and Skoog salts | Caisson Laboratories | 74904 | |
| Phytablend | Caisson Laboratories | 28302 | |
| RNeasy Plant Minikit | Qiagen | 16419 |
Referencias
- Franklin, K. A., Quail, P. H. Phytochrome functions in Arabidopsis development. J. Exp. Bot. 61, 11-24 (2010).
- Montgomery, B. L. Right place, right time: Spatiotemporal light regulation of plant growth and development. Plant Signal Behav. 3, 1053-1060 (2008).
- Laplaze, L. GAL4-GFP enhancer trap lines for genetic manipulation of lateral root development in Arabidopsis thaliana. J. Exp. Bot. 56, 2433-2442 (2005).
- Costigan, S., Warnasooriya, S. N., Montgomery, B. L. Root-localized phytochrome chromophore synthesis is required for tissue-specific photoregulation of root elongation and impacts sensitivity to jasmonic acid in Arabidopsis thaliana. , .
- Lagarias, D. M., Crepeau, M. W., Maines, M. D., Lagarias, J. C. Regulation of photomorphogenesis by expression of mammalian biliverdin reductase in transgenic Arabidopsis plants. Plant Cell. , 675-688 (1997).
- Montgomery, B. L., Yeh, K. C., Crepeau, M. W., Lagarias, J. C. Modification of distinct aspects of photomorphogenesis via targeted expression of mammalian biliverdin reductase in transgenic Arabidopsis plants. Plant Physiol. 121, 629-639 (1999).
- Warnasooriya, S. N., Montgomery, B. L. Detection of spatial-specific phytochrome responses using targeted expression of biliverdin reductase in Arabidopsis. Plant Physiol. 149, 424-433 (2009).
- Warnasooriya, S. N., Porter, K. J., Montgomery, B. L. Light-dependent anthocyanin accumulation and phytochromes in Arabidopsis thaliana. , .
- Denecke, J., Vitale, A. The use of protoplasts to study protein synthesis and transport by the plant endomembrane system. Methods Cell Biol. 50, 335-348 (1995).
- Birnbaum, K. Cell type-specific expression profiling in plants via cell sorting of protoplasts from fluorescent reporter lines. Nat. Methods. 2, 615-619 (2005).
- Ma, L. Light control of Arabidopsis development entails coordinated regulation of genome expression and cellular pathways. Plant Cell. 13, 2589-2607 (2001).
- Chen, M., Chory, J., Fankhauser, C. Light signal transduction in higher plants. Annu. Rev. Genet. 38, 87-117 (2004).
- Ulm, R., &, N. a. g. y., F, . Signalling and gene regulation in response to ultraviolet light. Curr. Opin. Plant Biol. 8, 477-482 (2005).
- Ma, L. Organ-specific expression of Arabidopsis genome during development. Plant Physiol. 138, 80-91 (2005).
- Neff, M. M., Fankhauser, C., &, C. h. o. r. y., J, . Light: an indicator of time and place. Genes Dev. 14, 257-271 (2000).
- Birnbaum, K. A gene expression map of the Arabidopsis root. Science. 302, 1956-1960 (2003).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados