É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Investigando Tecido e órgãos específicos Responses Fitocromo usando FACS assistida Célula tipo Profiling expressão específica no Arabidopsis thaliana
Neste Artigo
Resumo
A base molecular do espaço-respostas específicas fitocromo está sendo investigado o uso de plantas transgênicas que apresentam tecidos e órgãos específicos deficiências fitocromo. O isolamento de células específicas exibindo esgotamento cromóforo fitocromo induzida por fluorescência-Activated Celular classificação seguido por análises de microarray está sendo utilizada para identificar genes envolvidos em respostas espaciais específicos fitocromo.
Resumo
Media luz uma série de processos de desenvolvimento e de adaptação ao longo do ciclo de vida de uma planta. Plantas utilizam a luz de absorção de moléculas chamadas fotorreceptores de sentir e adaptar-se a luz. O vermelho / vermelho-extremo de absorção de luz fotorreceptores fitocromo têm sido estudadas extensivamente. Fitocromos existir como uma família de proteínas com funções distintas e sobrepostas em todos os sistemas de plantas superiores em que foram estudados
Protocolo
1. Crescimento da Planta
- Confirmado UAS-BVR X GAL4-GFP linha armadilha enhancer isoladas como descrito 4 (Para um resumo ver Fig. 1.) E as linhas do tipo selvagem ou parental são semeadas em solo, ou seja, ~ 2000 sementes esterilizadas por linha.
- As plantas são cultivadas por 5 semanas em solo sob iluminação branca de 100 μmolm -2 s -1 a 22 ° C e umidade de 70%.
2. Folha de protoplastos de isolamento (adaptado de Denecke Vitale e 9)
- Para isolar protoplastos, prepare TEX buffer. Para um buffer de litros TEX, pesar os seguintes componentes: 3,1 g sais Gamborg do B5, 0,5 g 2 - (N-morfolino) etanosulfónico (MES, 2,56 mM), 0,75 g cloreto de cálcio dihidratado (CaCl 2 .2 H 2 O, 6,75 mM), 0,25 g nitrato de amónio (NH 4 NO 3, 3,12 mM), 136,9 g de sacarose (0,4). Dissolve componentes completamente em ~ 900 ml de água, deionizada (DDH 2 O) e trazer o pH para 5,7 com 1 M KOH. Trazer um volume final de 1 litro e filtro de esterilizar com uma garrafa de 0,2 m top-filtro ligado a uma bomba de vácuo.
- Colete verde, folhas sadias de plantas (~ 250 ml de folhas ligeiramente comprimido em um copo) e enxaguar com ~ 40 ml DDH 2 O 4 vezes, seguido de lavagem duas vezes com estéril DDH 2 O. Usando um bisturi # 20, as folhas cortadas em tiras finas e dividir o tecido igualmente em dois tubos de 50 ml de plástico estéril. Prepare 1x mistura de digestão de folhas, adicionando 5 ml de alíquota 10x solução estoque folha digestão (10x folha solução estoque digestão: dissolver 2% w / v Macerozyme R-10, 4% w / v celulase "Onozuka" R-10 em TEX buffer, filtro de esterilizar com 0,2 m de filtro e congelar alíquotas 5 ml a -80 ° C) a 45 ml de tampão TEX fresco. Adicionar ~ 25 ml de 1x mix de folhas digestão por 50 ml tubo para cobrir todo o tecido da folha.
- Vácuo infiltrar tecido foliar em mistura de digestão em tubos abertos para 1 hora em temperatura ambiente usando um exsicador de vácuo conectada a uma bomba de água, seguido de 3 horas de incubação a temperatura ambiente em um rocker, agitando suavemente. Tubos tampados com tecido foliar no mix de folhas 1x digestão são mantidos embrulhados em papel alumínio durante este processo para evitar a exposição à luz.
- Após 3 horas, aumentar a velocidade rocker por aproximadamente 2 minutos para liberação de protoplastos. Filtrar a suspensão de protoplastos bruto por meio de duas camadas de pano de queijo estéril para remover detritos e recolher o filtrado em um copo de vidro esterilizado. Filtrar o filtrado através de uma malha de nylon estéril 100 mícrons em uma placa de Petri estéril. Recolher o fluxo através de e transferi-lo para um tubo novo ml estéril 50. Use cerca de 15-20 ml de tampão TEX fresca para lavar a placa de Petri estéril e coletar qualquer protoplastos aderindo à superfície.
- Centrifugar o fluxo através da utilização de um rotor balde swing em 100xg a 10 ° C por 15 min (6 Aceleração, Desaceleração 0).
- Remover ~ 25 - 30 ml do líquido abaixo da camada flutuante de protoplastos, que contém os restos residuais e peletizada, com uma estéril 9 "de vidro Pasteur pipeta conectado a uma bomba peristáltica, sem perturbar a camada flutuante de protoplastos e deixando ~ 10-15 ml de volume.
- Adicionar tampão TEX fresco para um volume final de 40 ml, enquanto delicadamente ressuspender a protoplastos.
- Repita os passos 2,5-2,7 duas vezes para remover o máximo possível os restos celulares. O tempo de centrifugação é reduzida para 10 minutos na primeira repetição e cinco minutos na repetição final.
- Aspirar protoplastos flutuante com um corte, uma pipeta de transferência estéril ml em um tubo ml novo 15. Prosseguir diretamente para a classificação ou armazenar no escuro por até 2 horas a 4 ° C até a classificação.
3. Classificando de protoplastos por Fluorescência-activated cell sorting (FACS)
- Antes da separação, examine protoplastos por Microscopia Confocal Laser Scanning (CLSM) usando um laser de 488 nm para excitação para confirmar a integridade de protoplastos ea presença de fluorescência da GFP na piscina de protoplastos. Detritos mínima é necessária para evitar o entupimento do bico de classificação FACS.
- Ordenar protoplastos isolados em TEX tampão via FACS (BD FACSVantage SE, BD Biosciences), utilizando um bico 200 mícrons em uma cabeça tipo macro na taxa de eventos entre 6.000 e 15.000, com um sistema de pressão de cerca de 9 psi na sequência de um protocolo adaptado 10.
- Tipo selvagem não GFP-protoplastos são usados para determinar os limiares autofluorescência.
- Para coletar GFP positivas protoplastos, células de ordenação usando um ar-refrigerado laser de argônio (Spectra Physics Modelo 177 de Newport Corporation, Irvine, CA) operado com 100 mW em uma linha de argônio 488-nm para identificar GFP fluorescência usando uma banda 530/30 filtro passa.
- Após a coleta de GFP-positivos protoplastos, examine protoplastos classificadas por CLSM conforme detalhado no passo 3.1.
- Extrato de RNA total a partir de protoplastos classificados usando um Qiagen Usina RNeasy Kit Mini. cDNA pode então ser preparado para hibridação, conforme descrito no Affymetrix GeneChip ExprManual de Análise Técnica e ession então hibridizado com ATH1 Arabidopsis Affymetrix arrays genoma inteiro.
Resultados representante
Detectamos um número significativo de células positivas para GFP-no canal de GFP por FACS (Fig. 2). Em ensaios de otimização usando GFP enhancer trap-pais, o constitutivamente expressando GFP-line, J0571, exibido ~ 17% a 24% GFP positivas protoplastos, enquanto que a linha com a expressão vascular e cutânea, J1071, tinha ~ 1,4% GFP positivas protoplastos (Tabela 1). Classificação de 3 x 500 mL (~ total de 1,5 ml de suspensão de protoplastos) de J0571 protoplastos por 1 h ~ GFP deu positivo 100.000 protoplastos. Classificação de 3 x 500 mL (~ 1,5 ml de suspensão de protoplastos total) de J1071 protoplastos por 1,5 h deu ~ 3000 GFP positivas protoplastos. Imagens confocal indicaram um rendimento muito elevado de protoplastos para amostras tanto J0571 e J1071 antes da separação foi realizada (Fig. 3C e 3E), e as frações classificadas continha apenas brilhante fluorescente GFP-protoplastos (Fig. 3G e dados não mostrados). Este achado confirma que intacta GFP positivas protoplastos podem ser classificados através FACS. Os protoplastos não GFP também podem ser classificados e recolhidos em um canal separado. Os protoplastos não GFP servir como um controle ideal negativo para análises posteriores microarray para detectar as alterações na expressão de genes específicos na redução dos níveis de holophytochrome. A extração de RNA a partir de protoplastos isolados (1 ml) produz RNA em quantidade suficiente para a detecção por fluorospectrometry (Fig. 4). Isolado RNA foi avaliada através de um instrumento NanoDrop (NanoDrop 1000, Thermo Scientific) e quantificados por NanoDrop software quantificação 3.7.1 RNA. Produz RNA a partir de pré-classificados C24 do tipo selvagem ou protoplastos FACS-classificados, GFP-positivos protoplastos armadilha enhancer excedeu o ng 20 mínimos necessários para utilização em RNA-rotulagem ensaios para microarray (Tabela 2).
| Enhancer Linha Armadilha | % Da GFP positivas protoplastos | Número de protoplastos GFP classificadas |
| J0571 | 17,18% ~ 24,06% | 26400 ~ 36000 |
| J1071 | ~ 1,43% | 1000 ~ 1300 |
Tabela 1. Percentagem de GFP-positivos protoplastos de duas linhas enhancer trap antes da separação e do número de GFP-positivos protoplastos coletados, executando 500 mL da suspensão de protoplastos através da fluorescência celular Sorter Ativado (FACSVantage, BD).
| Linha de plantas | RNA rendimento (ng) |
| C24 WT | 12486,5 |
| J0571 | 60 |
| J1071 | 71,5 |
Tabela 2. Rendimento de RNA isolamento de protoplastos. RNA foi isolado a partir de pré-classificados C24 do tipo selvagem ou classificados GFP positivas protoplastos a partir de duas linhas armadilha enhancer e quantificados.
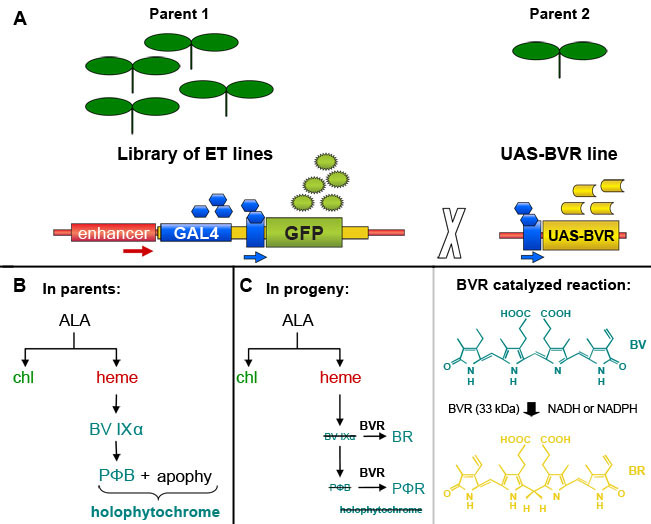
Figura 1. GAL4 enhancer-trap baseada indução de biliverdina redutase (BVR) expressão em plantas transgênicas de Arabidopsis thaliana. (A). Um indivíduo selecionado de uma biblioteca de GAL4 baseado linhas enhancer trap, que contêm um GAL4-responsive gene marcador GFP, pode ser cruzada com uma linha contendo um gene-alvo GAL4 de resposta para induzir a expressão do gene alvo em GAL4 contendo células marcadas GFP por fluorescência. Baseado em uma figura do Dr. Jim Haseloff (http://www.plantsci.cam.ac.uk/Haseloff/geneControl/GAL4Frame.html). (B) Produção de fitocromo cromóforo, phytochromobilin (PΦB) e holophytochrome em linhas pai. (C) esquerda, redução da biliverdina IXα (BV IXα) e pela biliverdina redutase PΦB (BVR) atividade a BR e PΦR, respectivamente. Atividade resulta em depleção de BVR PΦB e leva a uma redução na produção de fotoativo holophytochrome. Direita, a reação catalisada pela BVR é mostrado.
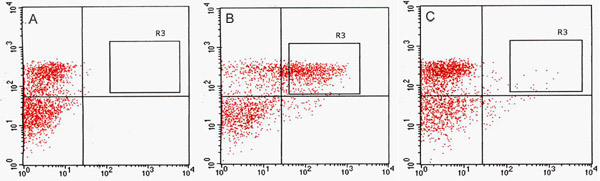
Figura 2. Separação de células de fluorescência Ativado (FACS) parcelas dot aquisição. Comparação de células de protoplastos de classificação para C24 do tipo selvagem (A), J0571 (B) e J1071 (C). (A) gráfico de pontos Aquisição de não-fluorescente GFP C24 do tipo selvagem protoplastos animado por um 488-nm laser de argônio e usado para determinar o limiar autofluorescência. (B) e (C) parcelas dot aquisição mostram proporções de protoplastos, que são GFP positivas em resposta à excitação por um laser 488 nm. R3 portões classificação em B e C delimitar as metas GFP-positivos que foram ordenados por Fluorescência-Activated Sorter Cell (FACSVantage, BD) e recolhidos. Canal vermelho indica valores de autofluorescência de clorofila a partir de protoplastos e canal verde indica valores para GFP fluorescência.

Figura 3. Microscopia confocal de protoplastos de plantas utilizadas na separação de células. Confocal a laser imagens digitalizadas antes de protoplastos (A, C, E) e depois (G) a classificação através de fluorescência-Activated Sorter Cell (FACS). B, D, F e H são imagens DIC. Protoplastos de C24 do tipo selvagem (A, B), J1071 (C, D) e J0571 (E, F, G, H) são mostrados. Imagens C, E e G são mescladas imagens de fluorescência da GFP (BP 505 nm - 575 nm) e autofluorescência (LP 650 nm) obtidos a partir de excitação com um laser 488 nm. A a H são média de 4 scans sob óleo 63x. Bar = 10 mM.
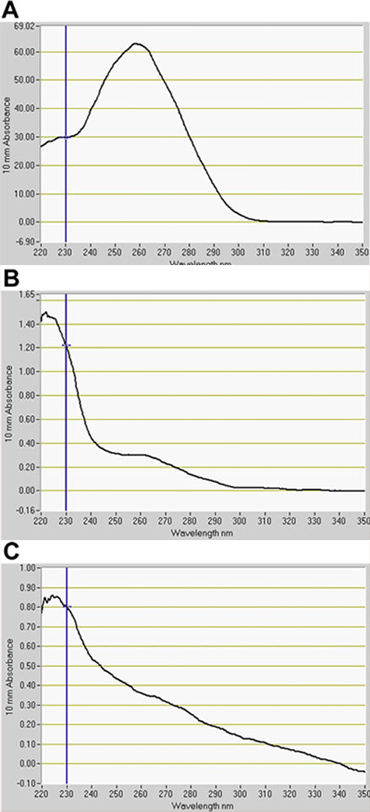
Figura 4. Quantificação do RNA isolado de protoplastos por fluorospectrometry. RNA extraído de (A) do tipo C24 selvagem, (B) J0571, e (C) J1071. A indica RNA para a pré-classificadas do tipo selvagem protoplastos. B e C indicam RNA de GFP-positivos protoplastos classificadas por Fluorescência-activated cell sorting (FACS). RNA software de quantificação, NanoDrop 3.7.1, (NanoDrop 1000, Thermo Scientific).
Discussão
Perfil de expressão gênica através de microarrays (1) indicou que mais de 30% dos genes em Arabidopsis mudas são leves regulamentados e 11 (2) identificou um vasto conjunto de genes que codificam proteínas de transdução de sinal de luz envolvidos na cascata de sinalização fitocromo 12, 13 . Tais experimentos sugerem que a luz induz a mudanças rápidas e de longo prazo na expressão genética. Cada pool de fitocromos pode controlar apenas um subconjunto de respostas de desenvolvimento e de...
Agradecimentos
Trabalho no laboratório Montgomery nas respostas fitocromo em plantas é apoiado pela National Science Foundation (Grant no. MCB-0919100 para BLM) e Ciências Químicas, Geociências e Biociências Divisão do Escritório de ciências básicas da energia, o Office of Science, EUA Departamento de energia (não concedem. FG02 DE 91ER20021 a BLM). Agradecemos a Melissa Whitaker de assistência técnica durante as filmagens e leitura crítica do manuscrito, Stephanie Costigan de assistência experimental, Dr. Luís Rei de assistência ao desenvolvimento e otimização de fluorescência-Activated Celular classificação protocolos para a classificação de protoplastos Arabidopsis e Frame Dr. Melinda para assistência com confocal microscopia. Agradecemos a Marlene Cameron para a assistência de design gráfico e Bird Karen de assistência editorial.
Materiais
| Name | Company | Catalog Number | Comments |
| Anti-BVR antibody | QED Bioscience Inc. | 56257-100 | |
| Cellulase “Onozuka” R-10 | SERVA Electrophoresis | MSPC 0930 | |
| Gamborg’s B5 basal salt mixture | Sigma-Aldrich | G5768 | |
| Macerozyme R-10 | SERVA Electrophoresis | PTC 001 | |
| MES, low moisture content | Sigma-Aldrich | M3671 | |
| Murashige and Skoog salts | Caisson Laboratories | 74904 | |
| Phytablend | Caisson Laboratories | 28302 | |
| RNeasy Plant Minikit | Qiagen | 16419 |
Referências
- Franklin, K. A., Quail, P. H. Phytochrome functions in Arabidopsis development. J. Exp. Bot. 61, 11-24 (2010).
- Montgomery, B. L. Right place, right time: Spatiotemporal light regulation of plant growth and development. Plant Signal Behav. 3, 1053-1060 (2008).
- Laplaze, L. GAL4-GFP enhancer trap lines for genetic manipulation of lateral root development in Arabidopsis thaliana. J. Exp. Bot. 56, 2433-2442 (2005).
- Costigan, S., Warnasooriya, S. N., Montgomery, B. L. Root-localized phytochrome chromophore synthesis is required for tissue-specific photoregulation of root elongation and impacts sensitivity to jasmonic acid in Arabidopsis thaliana. , .
- Lagarias, D. M., Crepeau, M. W., Maines, M. D., Lagarias, J. C. Regulation of photomorphogenesis by expression of mammalian biliverdin reductase in transgenic Arabidopsis plants. Plant Cell. , 675-688 (1997).
- Montgomery, B. L., Yeh, K. C., Crepeau, M. W., Lagarias, J. C. Modification of distinct aspects of photomorphogenesis via targeted expression of mammalian biliverdin reductase in transgenic Arabidopsis plants. Plant Physiol. 121, 629-639 (1999).
- Warnasooriya, S. N., Montgomery, B. L. Detection of spatial-specific phytochrome responses using targeted expression of biliverdin reductase in Arabidopsis. Plant Physiol. 149, 424-433 (2009).
- Warnasooriya, S. N., Porter, K. J., Montgomery, B. L. Light-dependent anthocyanin accumulation and phytochromes in Arabidopsis thaliana. , .
- Denecke, J., Vitale, A. The use of protoplasts to study protein synthesis and transport by the plant endomembrane system. Methods Cell Biol. 50, 335-348 (1995).
- Birnbaum, K. Cell type-specific expression profiling in plants via cell sorting of protoplasts from fluorescent reporter lines. Nat. Methods. 2, 615-619 (2005).
- Ma, L. Light control of Arabidopsis development entails coordinated regulation of genome expression and cellular pathways. Plant Cell. 13, 2589-2607 (2001).
- Chen, M., Chory, J., Fankhauser, C. Light signal transduction in higher plants. Annu. Rev. Genet. 38, 87-117 (2004).
- Ulm, R., &, N. a. g. y., F, . Signalling and gene regulation in response to ultraviolet light. Curr. Opin. Plant Biol. 8, 477-482 (2005).
- Ma, L. Organ-specific expression of Arabidopsis genome during development. Plant Physiol. 138, 80-91 (2005).
- Neff, M. M., Fankhauser, C., &, C. h. o. r. y., J, . Light: an indicator of time and place. Genes Dev. 14, 257-271 (2000).
- Birnbaum, K. A gene expression map of the Arabidopsis root. Science. 302, 1956-1960 (2003).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados