Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Étude des tissus et des organes spécifiques Réponses phytochrome utilisant FACS assistée type cellulaire profilage expression spécifique dans Arabidopsis thaliana
Dans cet article
Résumé
La base moléculaire de l'espace des réponses spécifiques phytochrome est étudiée en utilisant des plantes transgéniques qui présentent des tissus et des organes spécifiques déficiences phytochrome. L'isolement des cellules spécifiques présentant induite par la déplétion chromophore phytochrome Fluorescence-Activated tri cellulaire suivie par des analyses microarray est utilisée pour identifier les gènes impliqués dans les réponses phytochrome spatiale spécifique.
Résumé
Médie lumière un éventail de processus de développement et d'adaptation tout au long du cycle de vie d'une plante. Les plantes utilisent la lumière d'absorption des molécules appelées photorécepteurs de détecter et de s'adapter à la lumière. Le rouge / rouge lointain absorbant la lumière photorécepteurs phytochrome ont été largement étudiés. Phytochromes exister comme une famille de protéines ayant des fonctions distinctes et qui se chevauchent dans tous les systèmes de plantes supérieures dans lesquelles ils ont été étudiés
Protocole
1. Croissance des végétaux
- Confirmé UAS-GAL4 BVR X-GFP ligne de trappe enhancer isolée comme décrit 4 (Résumé, voir fig. 1) et les lignes de type sauvage ou parental sont semées sur le sol, soit ~ 2000 graines stérilisées par ligne.
- Les plantes sont cultivées pendant 5 semaines sur le sol sous un éclairage blanc de 100 μmolm -2 s -1 à 22 ° C et une humidité de 70%.
2. Feuille de protoplastes Isolement (adapté de Denecke et Vitale 9)
- Pour isoler des protoplastes, préparer TEX tampon. Pour 1 litre TEX tampon, pesez les éléments suivants: 3.1 sels g B5 de Gamborg, 0,5 g 2 - (N-morpholino) éthanesulfonique (MES, 2,56 mM), 0,75 dihydraté g de chlorure de calcium (CaCl 2 .2 H 2 O, 6,75 mM), 0,25 g de nitrate d'ammonium (NH 4 NO 3, 3,12 mM), 136,9 g de saccharose (0,4 M). Dissoudre les composants complètement en ~ 900 ml d'déminéralisée, eau distillée (FD 2 O) et amener le pH à 5,7 avec une M KOH. Amener le volume final à 1 litre et stériliser par filtration avec une bouteille de 0,2 um-dessus du filtre relié à une pompe à vide.
- Recueillir vert, feuilles saines à partir de plantes (~ 250 ml de feuilles tassées dans un bécher) et rincer avec ~ 40 ml ddH 2 O 4 fois, suivi d'un rinçage à deux reprises avec deux stériles O. ddH L'utilisation d'un bistouri n ° 20, les feuilles coupées en fines lanières et diviser les tissus également en deux tubes de 50 ml en plastique stérile. Préparer 1x mélange de digestion des feuilles en ajoutant 5 ml de solution aliquote de la feuille d'actions 10x digestion (10x solution stock de feuilles de digestion: dissoudre 2% p / v Macerozyme R-10, 4% p / v cellulase "Onozuka" R-10 dans TEX tampon, stériliser par filtration avec filtre de 0,2 um et congeler des aliquotes de 5 ml à -80 ° C) à 45 ml TEX tampon frais. Ajouter ~ 25 ml de mélange de la digestion 1x feuille par tube de 50 ml de couvrir tous les tissus de la feuille.
- Vide d'infiltrer les tissus foliaires au mélange de digestion dans des tubes ouverts pendant 1 heure à température ambiante en utilisant un dessiccateur à vide reliée à une pompe d'eau suivie de 3 heures d'incubation à température ambiante sur une bascule en agitant doucement. Tubes plafonné aux tissus de la feuille dans le mélange de feuilles de digestion 1x sont conservées enveloppées dans du papier aluminium pendant ce processus pour prévenir l'exposition à la lumière.
- Après 3 heures, augmenter la vitesse de bascule pour ~ 2 minutes pour libérer protoplastes. Filtrer la suspension de protoplastes brut par deux couches de toile à fromage stérile pour enlever les débris et recueillir le filtrat dans un bécher de verre stérile. Filtrer le filtrat à travers une maille de nylon de 100 um stérile dans une boîte de Pétri stérile. Recueillir le débit à travers et le transférer sur un nouveau tube stérile de 50 ml. Utilisez environ 15-20 ml de tampon frais TEX pour laver les boîtes de Pétri stériles et de recueillir toute protoplastes adhérant à la surface.
- Centrifuger le débit à travers l'aide d'un rotor à godets swing au 100xg à 10 ° C pendant 15 min (accélération 6, la décélération 0).
- Retirer ~ 25 - 30 ml de liquide sous la couche de protoplastes flottant, qui contient des débris résiduels et granulés, avec une solution stérile 9 "pipette Pasteur en verre relié à une pompe péristaltique sans déranger la couche de protoplastes flottant et laissant ~ 10 - volume de 15 ml.
- Ajouter frais de tampon TEX à un volume final de 40 ml tout en douceur remise en suspension des protoplastes.
- Répétez les étapes 2.5 à 2.7 deux fois pour enlever autant de débris cellulaires que possible. Le temps de centrifugation est réduit à 10 minutes dans la première répétition et à 5 minutes dans la répétition finale.
- Aspirer protoplastes flottant avec une coupe, une pipette de transfert stérile ml dans un nouveau tube de 15 ml. Passez directement au tri ou de stocker dans l'obscurité pendant 2 heures à 4 ° C jusqu'à ce que le tri.
3. Tri protoplastes par le tri de cellules à fluorescence (FACS)
- Avant le tri, d'examiner les protoplastes par microscopie confocale à balayage laser (CLSM) en utilisant un laser 488-nm pour l'excitation de confirmer l'intégrité des protoplastes et la présence de fluorescence de la GFP dans la piscine de protoplastes. Les débris minimale est nécessaire pour éviter le colmatage de la buse de tri FACS.
- Trier protoplastes isolés dans TEX tampon par FACS (BD FACSVantage SE, BD Biosciences) en utilisant une buse de 200 um sur une tête tri macro au taux d'événements entre 6000 et 15000, avec une pression du système d'environ 9 psi suite d'un protocole adapté 10.
- Wild-type non-GFP protoplastes sont utilisés pour déterminer les seuils d'autofluorescence.
- Pour collecter des GFP-positives protoplastes, les cellules de tri en utilisant un refroidissement par air au laser argon (Spectra Physics Modèle 177, Newport Corporation, Irvine, CA) a fonctionné à 100 mW sur une ligne de 488 nm d'argon pour identifier fluorescence de la GFP en utilisant une bande 530/30 Filtre passe.
- Après la collecte de la GFP-positives protoplastes, examiner protoplastes triés par CLSM comme détaillé dans l'étape 3.1.
- Extrait d'ARN total à partir de protoplastes triés en utilisant un Qiagen RNeasy Mini Kit végétales. ADNc peuvent alors être préparés à l'hybridation comme décrit dans Affymetrix GeneChip ExprManuel d'analyse ession Technique puis hybridées à ATH1 Arabidopsis Affymetrix génome entier.
Les résultats représentatifs
Nous avons détecté un nombre important de cellules positives pour la GFP dans le canal de la GFP par FACS (Fig. 2). Dans des essais d'optimisation utilisant la GFP enhancer-trap les parents, les constitutivement exprimant la GFP en ligne, J0571, affiché ~ 17% à 24% GFP-positives protoplastes, alors que la ligne avec une expression vasculaire et cutanée, J1071, avait ~ 1,4% GFP-positives protoplastes (Tableau 1). Tri de 3 x 500 pi (~ 1,5 ml au total de suspension de protoplastes) de J0571 protoplastes pendant 1 h ~ a donné 100 000 GFP-positives protoplastes. Tri de 3 x 500 pi (~ 1,5 ml de suspension de protoplastes total) de J1071 protoplastes pendant 1,5 h ~ 3000 a donné GFP-positives protoplastes. Images confocale indique un rendement très élevé de protoplastes pour les échantillons à la fois J0571 et J1071 avant le tri a été effectué (Fig. 3C et 3E), et les fractions triées ne contenait que brillante fluorescente GFP-protoplastes (Fig. 3G et données non présentées). Cette constatation confirme que intacte GFP-positives protoplastes peuvent être triées par FACS. Les protoplastes non-GFP peuvent également être triés et collectés dans un canal séparé. Les protoplastes non-GFP servir un contrôle idéal négatif pour des analyses ultérieures puces pour détecter les changements spécifiques dans l'expression des gènes lors de la réduction des niveaux holophytochrome. L'extraction d'ARN à partir de protoplastes isolés (1 ml) d'ARN rendements en quantité suffisante pour la détection par fluorospectrometry (fig. 4). ARN isolé a été évaluée en utilisant un instrument NanoDrop (NanoDrop 1000, Thermo Scientific) et quantifiés par NanoDrop 3.7.1 logiciel de quantification d'ARN. Rendements d'ARN à partir de pré-triés C24 de type sauvage protoplastes ou FACS-triés, GFP-positives protoplastes piège enhancer dépassé le minimum de 20 ng nécessaires pour une utilisation dans l'ARN-étiquetage des dosages pour biopuces (tableau 2).
| Enhancer Piège Ligne | % De la GFP-positives protoplastes | Nombre de protoplastes GFP triés |
| J0571 | 17,18% 24,06% ~ | 26400 ~ 36000 |
| J1071 | ~ 1,43% | 1000 ~ 1300 |
Tableau 1. Pourcentage de la GFP-positives protoplastes à partir de deux lignes de trappe Enhancer avant le tri et le nombre de GFP-positives protoplastes collectées en exécutant 500 pl de suspension de protoplastes à travers le trieur de cellules activé par fluorescence (FACSVantage, BD).
| Ligne des végétaux | ARN rendement (ng) |
| C24 WT | 12486.5 |
| J0571 | 60 |
| J1071 | 71,5 |
Tableau 2. Rendement de l'isolement d'ARN à partir de protoplastes. L'ARN a été isolé à partir de pré-triés C24 de type sauvage ou triés GFP-positives protoplastes à partir de deux lignes de trappe enhancer et quantifiés.
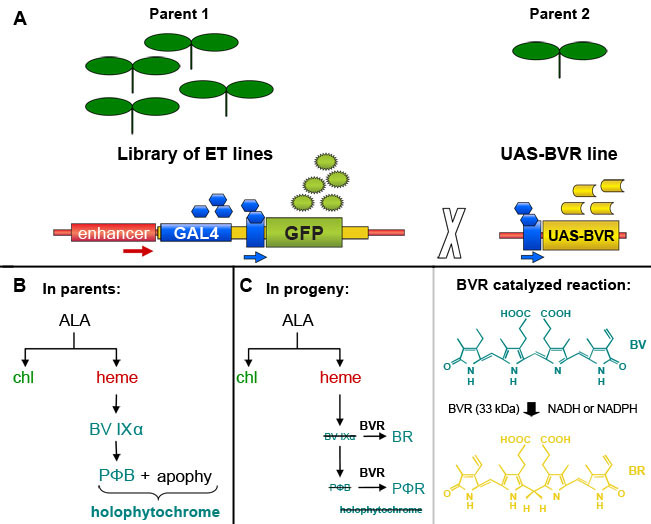
Figure 1. GAL4 enhancer-trap à base d'induction de biliverdine réductase (BVR) expression dans des plantes transgéniques d'Arabidopsis thaliana. (A). Une personne choisie parmi une bibliothèque de base de GAL4 lignes de trappe enhancer, qui contiennent un gène GAL4-réactive GFP marqueur, peut être traversé par une ligne contenant un gène cible GAL4-réactive pour induire l'expression du gène cible dans GAL4 contenant les cellules marquées par fluorescence de la GFP. Basé sur un personnage du Dr Jim Haseloff (http://www.plantsci.cam.ac.uk/Haseloff/geneControl/GAL4Frame.html). (B) Production de phytochrome chromophore, phytochromobilin (PΦB) et holophytochrome de lignées parentales. (C) A gauche, la réduction de la biliverdine IXα (BV IXα) et par la biliverdine réductase PΦB (BVR) des activités à BR et PΦR, respectivement. Résultats de l'activité dans le BVR épuisement des PΦB et conduit à une réduction de la production de photoactifs holophytochrome. A droite, la réaction catalysée par BVR est montré.
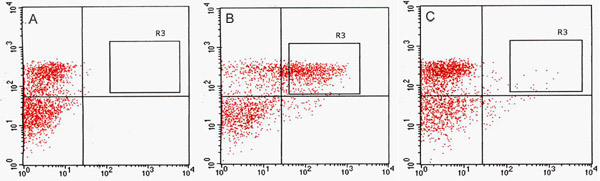
Figure 2. Fluorescence tri cellulaire (FACS) parcelles de points d'acquisition. Comparaison des cellules protoplastes de tri pour C24 de type sauvage (A), J0571 (B) et J1071 (C). (A) dot plot Acquisition de non-GFP fluorescente C24 de type sauvage protoplastes excité par un laser 488-nm argon et utilisé pour déterminer le seuil d'autofluorescence. (B) et (C) l'acquisition des parcelles de points montrent des proportions de protoplastes qui sont GFP-positives en réponse à une excitation par un laser 488-nm. R3 portes de tri dans B et C délimiter les cibles GFP-positives qui ont été triés par trieur de cellules activées par fluorescence (FACSVantage, BD) et collectées. Canal rouge indique valeurs pour autofluorescence chlorophylle à partir de protoplastes et canal vert indique les valeurs pour fluorescence de la GFP.

Figure 3. Microscopie confocale des protoplastes de plantes utilisées pour le tri cellulaire. Confocale laser, les images numérisées avant de protoplastes (A, C, E) et après (G) le tri par trieur de cellules activées par fluorescence (FACS). B, D, F et H sont des images DIC. Protoplastes de C24 de type sauvage (A, B), J1071 (C, D) et J0571 (E, F, G, H) sont représentés. Images C, E et G sont fusionnés images de fluorescence de la GFP (BP 505 nm - 575 nm) et autofluorescence (LP 650 nm) obtenus à partir d'excitation avec un laser 488-nm. A à H sont en moyenne de 4 scans dans l'huile 63x. Barre = 10 um.
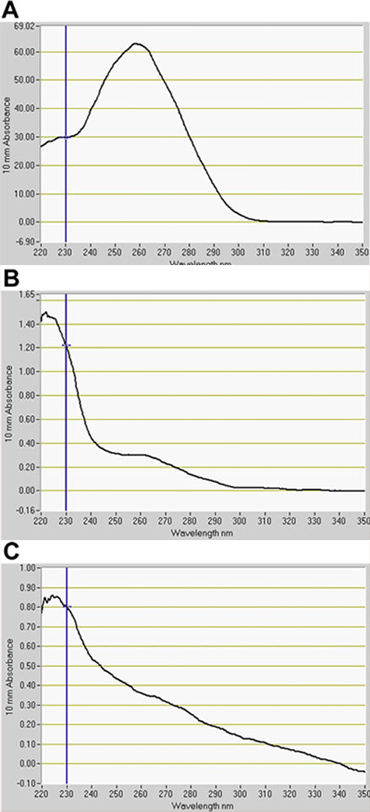
Figure 4. Quantification de l'ARN isolé à partir de protoplastes par fluorospectrometry. L'ARN extrait à partir de (A) C24 de type sauvage, (B) J0571, et (C) J1071. A indique l'ARN pré-triés de type sauvage protoplastes. B et C indiquent ARN à partir de la GFP-positives protoplastes triés par tri cellulaire activé par fluorescence (FACS). Un logiciel de quantification d'ARN, NanoDrop 3.7.1, (NanoDrop 1000, Thermo Scientific).
Discussion
Profilage de l'expression de gènes par puces à ADN (1) a indiqué que plus de 30% des gènes dans des plants d'Arabidopsis sont réglementés et de lumière 11 (2) a identifié un vaste groupe de gènes codant des protéines de transduction du signal lumineux impliqués dans la cascade de signalisation phytochrome 12, 13 . De telles expériences suggèrent que la lumière induit des changements rapides et à long terme dans l'expression des gènes. Chaque pool de phytochromes peut con...
Remerciements
Le travail dans le laboratoire de Montgomery sur les réponses phytochrome dans les plantes est soutenue par la National Science Foundation (subvention aucune. MCB-0919100 au BLM) et les sciences chimiques, sciences de la terre et les sciences biologiques, Bureau des sciences fondamentales de l'énergie, Bureau des sciences, Département américain de Energie (subvention aucune. DE FG02 91ER20021 au BLM). Nous tenons à remercier Melissa Whitaker pour l'assistance technique pendant le tournage et la lecture critique du manuscrit, Stéphanie Costigan à l'aide d'expérimentation, le Dr Louis roi de l'aide pour développer et optimiser Fluorescence-Activated tri cellulaire protocoles pour le tri des protoplastes d'Arabidopsis et le Dr Melinda cadre de l'aide pour confocale microscopie. Nous tenons à remercier Marlene Cameron pour l'assistance de conception graphique et Bird Karen pour son assistance éditoriale.
matériels
| Name | Company | Catalog Number | Comments |
| Anti-BVR antibody | QED Bioscience Inc. | 56257-100 | |
| Cellulase “Onozuka” R-10 | SERVA Electrophoresis | MSPC 0930 | |
| Gamborg’s B5 basal salt mixture | Sigma-Aldrich | G5768 | |
| Macerozyme R-10 | SERVA Electrophoresis | PTC 001 | |
| MES, low moisture content | Sigma-Aldrich | M3671 | |
| Murashige and Skoog salts | Caisson Laboratories | 74904 | |
| Phytablend | Caisson Laboratories | 28302 | |
| RNeasy Plant Minikit | Qiagen | 16419 |
Références
- Franklin, K. A., Quail, P. H. Phytochrome functions in Arabidopsis development. J. Exp. Bot. 61, 11-24 (2010).
- Montgomery, B. L. Right place, right time: Spatiotemporal light regulation of plant growth and development. Plant Signal Behav. 3, 1053-1060 (2008).
- Laplaze, L. GAL4-GFP enhancer trap lines for genetic manipulation of lateral root development in Arabidopsis thaliana. J. Exp. Bot. 56, 2433-2442 (2005).
- Costigan, S., Warnasooriya, S. N., Montgomery, B. L. Root-localized phytochrome chromophore synthesis is required for tissue-specific photoregulation of root elongation and impacts sensitivity to jasmonic acid in Arabidopsis thaliana. , .
- Lagarias, D. M., Crepeau, M. W., Maines, M. D., Lagarias, J. C. Regulation of photomorphogenesis by expression of mammalian biliverdin reductase in transgenic Arabidopsis plants. Plant Cell. , 675-688 (1997).
- Montgomery, B. L., Yeh, K. C., Crepeau, M. W., Lagarias, J. C. Modification of distinct aspects of photomorphogenesis via targeted expression of mammalian biliverdin reductase in transgenic Arabidopsis plants. Plant Physiol. 121, 629-639 (1999).
- Warnasooriya, S. N., Montgomery, B. L. Detection of spatial-specific phytochrome responses using targeted expression of biliverdin reductase in Arabidopsis. Plant Physiol. 149, 424-433 (2009).
- Warnasooriya, S. N., Porter, K. J., Montgomery, B. L. Light-dependent anthocyanin accumulation and phytochromes in Arabidopsis thaliana. , .
- Denecke, J., Vitale, A. The use of protoplasts to study protein synthesis and transport by the plant endomembrane system. Methods Cell Biol. 50, 335-348 (1995).
- Birnbaum, K. Cell type-specific expression profiling in plants via cell sorting of protoplasts from fluorescent reporter lines. Nat. Methods. 2, 615-619 (2005).
- Ma, L. Light control of Arabidopsis development entails coordinated regulation of genome expression and cellular pathways. Plant Cell. 13, 2589-2607 (2001).
- Chen, M., Chory, J., Fankhauser, C. Light signal transduction in higher plants. Annu. Rev. Genet. 38, 87-117 (2004).
- Ulm, R., &, N. a. g. y., F, . Signalling and gene regulation in response to ultraviolet light. Curr. Opin. Plant Biol. 8, 477-482 (2005).
- Ma, L. Organ-specific expression of Arabidopsis genome during development. Plant Physiol. 138, 80-91 (2005).
- Neff, M. M., Fankhauser, C., &, C. h. o. r. y., J, . Light: an indicator of time and place. Genes Dev. 14, 257-271 (2000).
- Birnbaum, K. A gene expression map of the Arabidopsis root. Science. 302, 1956-1960 (2003).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon