È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Indagare tessuti e risposte fitocromo organo-specifici utilizzando FACS-Cell-assistita di tipo profili specifici di espressione in Arabidopsis thaliana
In questo articolo
Riepilogo
Le basi molecolari delle risposte fitocromo spazio-specifica è indagato con le piante transgeniche che presentano tessuti e carenze fitocromo organo-specifiche. L'isolamento di cellule specifiche esporre indotte esaurimento fitocromo cromoforo da Fluorescence-Activated cella Ordinamento seguita da analisi di microarray viene utilizzata per identificare i geni coinvolti nello spazio-risposte specifiche fitocromo.
Abstract
Media luce una serie di processi evolutivi e adattativi per tutto il ciclo di vita di una pianta. Le piante utilizzano la luce di assorbimento delle molecole chiamate fotorecettori di senso e di adattarsi alla luce. Il rosso / lontano-rosso che assorbono la luce fotorecettori fitocromo sono stati studiati ampiamente. Fitocromi esistere come una famiglia di proteine con funzioni diverse e sovrapposte in tutti i sistemi vegetali superiori in cui sono stati studiati
Protocollo
1. Crescita delle piante
- Confermato UAS-BVR GAL4 X-GFP linea trappola enhancer isolato come descritto 4 (per sintesi vedi fig. 1) e le linee di wild-type o parentale vengono seminate sul terreno, ovvero ~ 2000 semi sterilizzati per riga.
- Le piante sono coltivate per 5 settimane in terra sotto illuminazione bianca di 100 μmolm -2 s -1 a 22 ° C e 70% di umidità.
2. Foglia di protoplasti di isolamento (adattato da Denecke e Vitale 9)
- Per isolare protoplasti, preparare TEX buffer. Per buffer di 1 litro TEX, pesare i seguenti componenti: 3,1 g Gamborg di sali B5, 0,5 g di 2 - (N-morfolino) etano acido (MES, 2,56 mM), 0,75 diidrato g di calcio cloruro (CaCl 2 .2 H 2 O, 6,75 mM), 0,25 g di nitrato di ammonio (NH 4 NO 3, 3.12 mM), 136,9 g di saccarosio (0.4M). Sciogliere i componenti completamente in ~ 900 ml di acqua deionizzata, acqua distillata (DDH 2 O) e portare il pH a 5,7 con 1 M KOH. Portare il volume finale a 1 litro e sterilizzare con filtro da 0,2 micron per bottiglie con filtro collegato ad una pompa a vuoto.
- Raccogliere verde, foglie di piante sane (~ 250 ml di foglie poco compresso in un bicchiere) e sciacquare con ~ 40 ml DDH 2 O 4 volte, seguita da risciacquo per due volte con sterili DDH 2 O. Usando un bisturi # 20, foglie tagliate a striscioline e si dividono equamente in due tessuti 50 ml, tubi di plastica sterile. Preparare 1x miscela digestione foglia aggiungendo 5 ml aliquota di 10x soluzione madre digestione foglia (10x digestione soluzione foglia magazzino: sciogliere 2% w / v Macerozyme R-10, 4% w / v Cellulase "Onozuka" R-10 nel TEX buffer, sterilizzare con filtro 0,2 micron filtro e blocco 5 aliquote ml a -80 ° C) a 45 ml di tampone fresco TEX. Aggiungi ~ 25 ml di 1x mix digestione foglia per tubo da 50 ml per coprire tutto il tessuto fogliare.
- Vuoto infiltrarsi nel tessuto di foglie miscela digestione in tubi aperti per 1 ora a temperatura ambiente utilizzando un essiccatore a vuoto collegata ad una pompa d'acqua seguita da 3-ore di incubazione a temperatura ambiente su una sedia a dondolo agitando delicatamente. Tubi ricoperto con foglia in tessuto misto 1x digestione foglia sono tenuti avvolti in carta stagnola durante questo processo per evitare l'esposizione alla luce.
- Dopo 3 ore, aumentare la velocità rocker per circa 2 minuti per rilasciare protoplasti. Filtrare la sospensione greggio protoplasti attraverso due strati di telo sterile per rimuovere i detriti e raccogliere il filtrato in un bicchiere di vetro sterile. Filtrare il filtrato attraverso una sterile da 100 micron mesh di nylon in una piastra di Petri sterile. Raccogliere il flusso attraverso e trasferirlo ad un nuovo sterile tubo da 50 ml. Usa circa 15-20 ml di tampone fresco TEX a lavare i piastra di Petri sterile e raccogliere qualsiasi protoplasti aderire alla superficie.
- Centrifugare il flusso attraverso usando un rotore a rotazione 100xg a 10 ° C per 15 minuti (6 accelerazione, decelerazione 0).
- Rimuovere ~ 25 - 30 ml di liquido al di sotto dello strato protoplasti fluttuante, che contiene residui e pellet, con una sterile 9 "pipetta Pasteur di vetro collegata ad una pompa peristaltica senza disturbare il livello fluttuante protoplasti e lasciando ~ 10-15 ml.
- Aggiungi nuovo tampone TEX per un volume finale di 40 ml, mentre delicatamente risospendere l'protoplasti.
- Ripetere i passaggi 2,5-2,7 due volte per rimuovere il più possibile di detriti cellulari. Il tempo di centrifugazione è ridotto a 10 minuti nella prima ripetizione ed a 5 minuti nella ripetizione finale.
- Aspirare protoplasti galleggiante con un taglio, 1 pipetta sterile trasferire ml in un nuovo tubo 15 ml. Procedere direttamente alla cernita o conservare al buio per un massimo di 2 ore a 4 ° C fino ordinamento.
3. Ordinamento protoplasti da Fluorescence-Activated ordinamento Cell (FACS)
- Prima di smistamento, esaminare protoplasti da microscopia confocale a scansione laser (CLSM) utilizzando un laser a 488-nm per l'eccitazione per confermare l'integrità protoplasti e la presenza di fluorescenza GFP in piscina protoplasti. Detriti minimo è necessario per evitare l'intasamento l'ordinamento FACS ugello.
- Protoplasti sorta isolato in TEX tampone tramite FACS (BD FACSVantage SE, BD Biosciences) con un 200-micron ugello su una testa tipo macro a tassi di eventi tra 6.000 e 15.000, con una pressione del sistema di circa 9 psi a seguito di un adeguato protocollo 10.
- Wild-type non GFP protoplasti vengono utilizzati per determinare le soglie di autofluorescenza.
- Per raccogliere GFP-positive protoplasti, le cellule utilizzando un tipo raffreddato ad aria, argon laser (Spectra Physics Modello 177, Newport Corporation, Irvine, CA), gestito a 100 mW a 488-nm linea argon per identificare GFP fluorescenza mediante una band 530/30 filtro passa.
- Dopo la raccolta di GFP-positive protoplasti, esaminare protoplasti ordinati per CLSM come descritto al punto 3.1.
- Estrarre l'RNA totale da protoplasti ordinati utilizzando un impianto di Qiagen RNeasy Mini kit. cDNA possono quindi essere pronti per l'ibridazione, come descritto nella Affymetrix GeneChip Expressione manuale analisi tecnica e poi ibridato a ATH1 Arabidopsis array Affymetrix intero genoma.
Rappresentante Risultati
Abbiamo rilevato un numero significativo di cellule GFP-positive nel canale GFP da FACS (Fig. 2). Nei test di ottimizzazione usando GFP enhancer-trap genitori, la GFP che esprimono costitutivamente linea, J0571, visualizzati ~ 17% al 24% GFP-positive protoplasti, mentre la linea con l'espressione vascolare e cutaneo, J1071, aveva ~ 1,4% GFP-positive protoplasti (Tabella 1). Ordinamento di 3 x 500 microlitri (~ 1,5 ml di sospensione totale di protoplasti) di J0571 protoplasti per 1 ora ha dato ~ 100.000 GFP-positive protoplasti. Ordinamento di 3 x 500 microlitri (~ 1,5 ml sospensione di protoplasti totale) di J1071 protoplasti per 1,5 h dato ~ 3.000 GFP-positive protoplasti. Immagini confocale indicato un rendimento molto elevato di protoplasti per campioni sia J0571 e J1071 prima di ordinare è stata effettuata (Fig. 3C e 3E), e le frazioni ordinati conteneva solo luminoso fluorescente GFP-protoplasti (Fig. 3G e dati non riportati). Questa scoperta conferma che intatti GFP-positive protoplasti possono essere ordinati tramite FACS. La non-GFP protoplasti possono anche essere ordinati e raccolti in un canale separato. La non-GFP protoplasti servire come controllo negativo ideale per analisi microarray successive per rilevare i cambiamenti specifici nell'espressione genica con la riduzione dei livelli holophytochrome. Estrazione di RNA da protoplasti isolati (1 ml) RNA produce in quantità sufficiente per il rilevamento da parte fluorospectrometry (Fig. 4). Isolato l'RNA è stata valutata utilizzando uno strumento NanoDrop (NanoDrop 1000, Thermo Scientific) e quantificato NanoDrop 3.7.1 del software quantificazione RNA. Rendimenti RNA da pre-ordinati C24 wild-type protoplasti o FACS-ordinati, GFP-positive protoplasti trappola enhancer superato il minimo di 20 ng necessaria per l'utilizzo in RNA di etichettatura per i test microarray (Tabella 2).
| Enhancer Trappola di linea | % Di GFP-positive protoplasti | Numero di protoplasti GFP ordinati |
| J0571 | 17,18% ~ 24,06% | 26.400 ~ 36.000 |
| J1071 | ~ 1,43% | 1000 ~ 1300 |
Tabella 1. Percentuale di GFP-positive protoplasti da due linee trappola potenziatore prima selezione e il numero di GFP-positive protoplasti raccolti eseguendo 500 microlitri di sospensione di protoplasti attraverso l'Ordine fluorescenza cellulare attivato (FACSVantage, BD).
| Impianti di linea | RNA rendimento (ng) |
| C24 WT | 12486,5 |
| J0571 | 60 |
| J1071 | 71,5 |
Tabella 2. Resa dall'isolamento RNA da protoplasti. L'RNA è stato isolato da pre-ordinati C24 wild-type o ordinati GFP-positive protoplasti da due linee trappola potenziatore e quantificati.
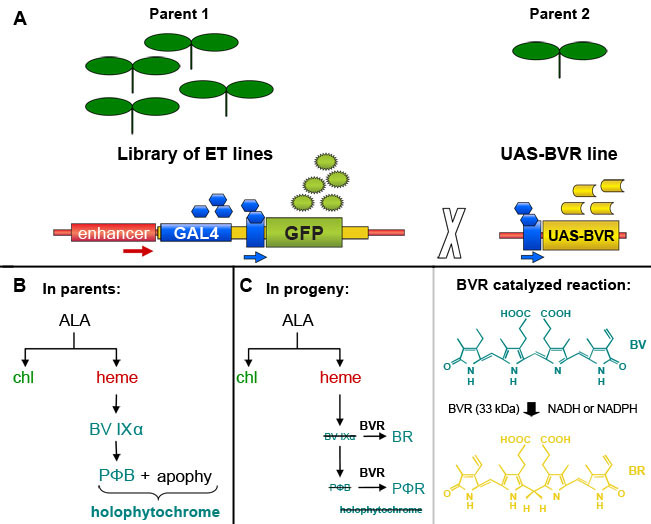
Figura 1. GAL4 enhancer-trap a base di induzione della biliverdina reduttasi (BVR) espressione in piante transgeniche di Arabidopsis thaliana. (A). Un individuo selezionato da una libreria di GAL4 trappola a base di linee enhancer, che contengono un GAL4-reattiva gene marcatore GFP, può essere attraversata con una linea contenente un GAL4-reattiva gene bersaglio di indurre l'espressione del gene bersaglio in GAL4 contenenti cellule segnato fluorescenza di GFP. Sulla base di una figura del Dr. Jim Haseloff (http://www.plantsci.cam.ac.uk/Haseloff/geneControl/GAL4Frame.html). (B) Produzione di fitocromo cromoforo, phytochromobilin (PΦB) e holophytochrome in linee parentali. (C) Sinistra, Riduzione della biliverdina IXα (BV IXα) e PΦB da biliverdina reduttasi (BVR) attività di BR e PΦR, rispettivamente. Risultati dell'attività BVR a esaurimento delle PΦB e porta ad una riduzione nella produzione di fotoattivi holophytochrome. A destra, la reazione catalizzata da BVR è mostrato.
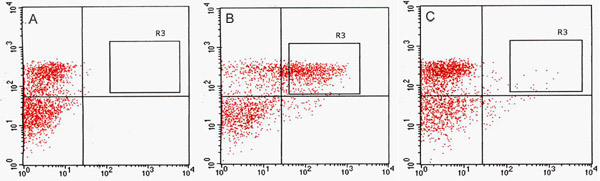
Figura 2. Fluorescenza separazione delle cellule attivate (FACS) appezzamenti punto di acquisizione. Confronto di celle protoplasti di ordinamento per C24 di tipo selvatico (A), J0571 (B) e J1071 (C). (A) Acquisizione di dot plot non-C24 fluorescente GFP wild-type protoplasti eccitati da un 488-nm argon laser e utilizzato per determinare la soglia autofluorescenza. (B) e (C) appezzamenti dot acquisizione mostrano proporzioni di protoplasti che sono GFP positive in risposta ad eccitazione di un 488-nm laser. R3 cancelli ordinamento in B e C delimitano la GFP-positive obiettivi che sono stati ordinati per Fluorescence-Activated cell sorter (FACSVantage, BD) e raccolti. Canale rosso indica vALORI per autofluorescenza clorofilla da protoplasti e il canale verde indica valori di fluorescenza GFP.

Figura 3. Microscopia confocale di protoplasti pianta usata per l'ordinamento delle cellule. Confocale a scansione laser immagini di protoplasti prima (A, C, E) e dopo (G) ordinamento tramite fluorescenza-Activated cell sorter (FACS). B, D, F e H sono immagini DIC. Protoplasti di C24 wild-type (A, B), J1071 (C, D) e J0571 (E, F, G, H) sono mostrati. Immagini C, E e G si fondono immagini di fluorescenza GFP (BP 505 nm - 575 nm) e autofluorescenza (LP 650 nm) ottenuti da eccitazione con un 488-nm laser. Da A ad H sono nella media di 4 scansioni sott'olio 63x. Bar = 10 micron.
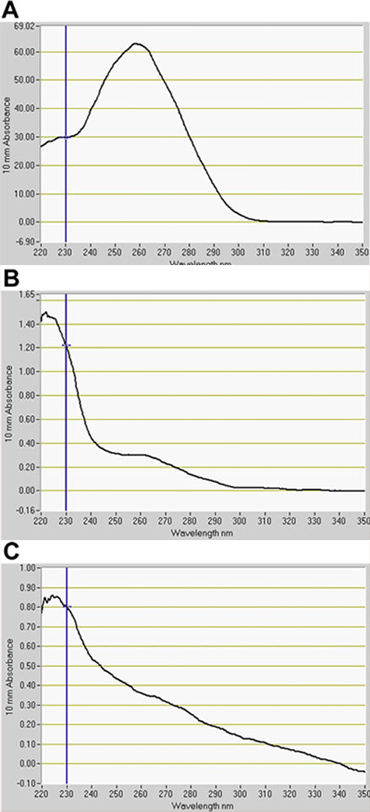
Figura 4. Quantificazione di RNA isolati da protoplasti da fluorospectrometry. RNA estratto da (A) tipo C24 selvatico, (B) J0571, e (C) J1071. A indica RNA per la pre-ordinati wild-type protoplasti. B e C indicare RNA da GFP-positive protoplasti ordinati per Fluorescence-Activated ordinamento Cell (FACS). RNA software quantificazione, NanoDrop 3.7.1, (NanoDrop 1000, Thermo Scientific).
Discussione
Profili di espressione genica attraverso microarray (1) ha indicato che oltre il 30% dei geni in piantine di Arabidopsis sono regolati luce 11 e (2) ha identificato un vasto gruppo di geni che codificano le proteine luce di trasduzione del segnale coinvolte nella cascata di segnalazione fitocromo 12, 13 . Tali esperimenti suggeriscono che la luce induce cambiamenti rapidi e lungo termine espressione genica. Ogni pool di fitocromi può controllare solo un sottoinsieme di risposte di sviluppo e...
Riconoscimenti
Lavoro in laboratorio Montgomery sulle risposte fitocromo nelle piante è sostenuta dalla National Science Foundation (non concede. MCB-0919100 per BLM) e Scienze Chimiche, Scienze geologiche e Bioscienze Divisione, Ufficio di Basic Sciences Energy, Office of Science, US Department of energia (non concedere alcuna. DE FG02 91ER20021 a BLM). Ringraziamo Melissa Whitaker per l'assistenza tecnica durante le riprese e la lettura critica del manoscritto, Stephanie Costigan sperimentale per l'assistenza, il Dott. Luigi Re di assistenza sviluppare e ottimizzare le Fluorescence-Activated cella ordinamento protocolli per l'ordinamento di protoplasti di Arabidopsis e il Dr. Telaio Melinda per assistenza confocale microscopia. Ringraziamo Marlene Cameron per l'assistenza di progettazione grafica e Bird Karen per l'assistenza editoriale.
Materiali
| Name | Company | Catalog Number | Comments |
| Anti-BVR antibody | QED Bioscience Inc. | 56257-100 | |
| Cellulase “Onozuka” R-10 | SERVA Electrophoresis | MSPC 0930 | |
| Gamborg’s B5 basal salt mixture | Sigma-Aldrich | G5768 | |
| Macerozyme R-10 | SERVA Electrophoresis | PTC 001 | |
| MES, low moisture content | Sigma-Aldrich | M3671 | |
| Murashige and Skoog salts | Caisson Laboratories | 74904 | |
| Phytablend | Caisson Laboratories | 28302 | |
| RNeasy Plant Minikit | Qiagen | 16419 |
Riferimenti
- Franklin, K. A., Quail, P. H. Phytochrome functions in Arabidopsis development. J. Exp. Bot. 61, 11-24 (2010).
- Montgomery, B. L. Right place, right time: Spatiotemporal light regulation of plant growth and development. Plant Signal Behav. 3, 1053-1060 (2008).
- Laplaze, L. GAL4-GFP enhancer trap lines for genetic manipulation of lateral root development in Arabidopsis thaliana. J. Exp. Bot. 56, 2433-2442 (2005).
- Costigan, S., Warnasooriya, S. N., Montgomery, B. L. Root-localized phytochrome chromophore synthesis is required for tissue-specific photoregulation of root elongation and impacts sensitivity to jasmonic acid in Arabidopsis thaliana. , .
- Lagarias, D. M., Crepeau, M. W., Maines, M. D., Lagarias, J. C. Regulation of photomorphogenesis by expression of mammalian biliverdin reductase in transgenic Arabidopsis plants. Plant Cell. , 675-688 (1997).
- Montgomery, B. L., Yeh, K. C., Crepeau, M. W., Lagarias, J. C. Modification of distinct aspects of photomorphogenesis via targeted expression of mammalian biliverdin reductase in transgenic Arabidopsis plants. Plant Physiol. 121, 629-639 (1999).
- Warnasooriya, S. N., Montgomery, B. L. Detection of spatial-specific phytochrome responses using targeted expression of biliverdin reductase in Arabidopsis. Plant Physiol. 149, 424-433 (2009).
- Warnasooriya, S. N., Porter, K. J., Montgomery, B. L. Light-dependent anthocyanin accumulation and phytochromes in Arabidopsis thaliana. , .
- Denecke, J., Vitale, A. The use of protoplasts to study protein synthesis and transport by the plant endomembrane system. Methods Cell Biol. 50, 335-348 (1995).
- Birnbaum, K. Cell type-specific expression profiling in plants via cell sorting of protoplasts from fluorescent reporter lines. Nat. Methods. 2, 615-619 (2005).
- Ma, L. Light control of Arabidopsis development entails coordinated regulation of genome expression and cellular pathways. Plant Cell. 13, 2589-2607 (2001).
- Chen, M., Chory, J., Fankhauser, C. Light signal transduction in higher plants. Annu. Rev. Genet. 38, 87-117 (2004).
- Ulm, R., &, N. a. g. y., F, . Signalling and gene regulation in response to ultraviolet light. Curr. Opin. Plant Biol. 8, 477-482 (2005).
- Ma, L. Organ-specific expression of Arabidopsis genome during development. Plant Physiol. 138, 80-91 (2005).
- Neff, M. M., Fankhauser, C., &, C. h. o. r. y., J, . Light: an indicator of time and place. Genes Dev. 14, 257-271 (2000).
- Birnbaum, K. A gene expression map of the Arabidopsis root. Science. 302, 1956-1960 (2003).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon