Method Article
Mutagénesis y funcional protocolos de selección de la evolución dirigida de proteínas en E. coli
En este artículo
Resumen
Aquí se demuestra un protocolo sencillo para crear una biblioteca de mutantes al azar de una secuencia objetivo determinado. Se muestra cómo este método, que se realiza en vivo en Escherichia coli, se pueden acoplar con la selección funcional para desarrollar nuevas actividades enzimáticas.
Resumen
La generación eficiente de la diversidad genética representa una valiosa herramienta molecular que puede ser utilizada para etiquetar la síntesis de ADN, para crear los patrones moleculares, o para desarrollar las proteínas en el laboratorio. A continuación, presentamos un protocolo que permite la generación de gran tamaño (> 10 11) bibliotecas de mutantes en una secuencia objetivo determinado. Este método se basa en la replicación de un plásmido ColE1 codificación de la secuencia deseada por una variante de baja fidelidad de la DNA polimerasa I (LF-Pol I). El objetivo del plásmido se transforma en una cepa de E. mutador coli y siembra en medio sólido, que oscilan entre un 0,2 y un mutaciones / kb, dependiendo de la localización del gen objetivo. Mayores frecuencias de mutación se logran mediante la iteración de este proceso de mutagénesis. En comparación con otros métodos de mutagénesis, el protocolo se destaca por su sencillez, como la clonación o la PCR no están involucrados. Por lo tanto, nuestro método es ideal para el etiquetado de las mutaciones de los plásmidos u otras plantillas que Pol o para explorar una gran parte del espacio de secuencias de la evolución de las actividades que no están presentes en la meta original. El estricto control espacial que la PCR o al azar oligonucleótidos basados en ofrecer métodos también se puede lograr a través de la posterior clonación de las secciones específicas de la biblioteca. Aquí les ofrecemos los protocolos que muestra cómo crear una biblioteca de mutantes al azar y la forma de establecer las drogas basadas en las selecciones de E. coli para identificar mutantes presentan nuevas actividades bioquímicas.
Protocolo
I. Generación de una biblioteca de mutantes al azar
Pol I interviene en la iniciación de la replicación del plásmido ColE1 (revisado en (1-3). Nuestro método de mutagénesis se basa en la colocación de una secuencia diana en un plásmido ColE1 junto con el origen o la replicación y propagación en células que expresan de baja fidelidad ADN polimerasa I (LF-Pol I). LF-Pol I es una polimerasa de ADN mutante que codifica tres mutaciones que disminuyen la fidelidad de la replicación, es decir, I709N (en el tema A), A759R (en el tema B) y D424A (inactivación de corrección) 4,5 . LF-Pol I se expresa en una cepa de E. coli, JS200, que tiene un alelo sensible a la temperatura de Pol I (polA12) (6) a fin de que LF-Pol I se convierte en la actividad predominante en el 37 º C. La replicación de la secuencia diana en polA12 células bajo condiciones restrictivas resultados en la generación de una biblioteca de mutantes al azar. Mutagénesis es más eficiente en cultivos saturados 4. Por razones que aún no están claros, la mutagénesis no es continua, es decir, la frecuencia de la mutación no se incrementa linealmente con el número de las generaciones, una vez que el cultivo alcance la saturación, incluso si las células se les permite ampliar aún más en el nuevo medio. Por lo tanto, aumentando aún más la carga de la mutación de la biblioteca requiere iterativo rondas de mutagénesis y la recuperación del plásmido. A continuación presentamos los protocolos de LF-Pol I mutagénesis. Tenga en cuenta que los protocolos que aquí se presenta se han simplificado considerablemente en relación con nuestro hijo de 4 Descripción original con el fin de facilitar la repetición del proceso para el logro de la carga de la mutación deseada (Fig. 1).
Materiales
- Células: JS200 recA718 polA12 (ts) uvrA155 trpE65 lon-11 sula
- JS200 WT-Pol I: JS200 células que expresan el tipo salvaje (WT) Pol I
- JS200 LF-Pol I: JS200 expresar baja fidelidad (LF) Pol I
- Cepa de lectura: JS200 WT-Pol I o (de complementación) de una cepa que carecen de una actividad específica
- Objetivo plásmido
- Plásmido que contiene un origen de replicación ColE1 con el objetivo de genes clonados en
1. Antes de Mutagénesis: Preparación de la electro-competentes JS200 LF-Pol células I
- Elija una sola JS200 LF-Pol I colonia de una placa de LB crecido a 30 ° C (condiciones permisivas) durante la noche que contiene la selección antibiótico apropiado para el rodamiento plásmido LF-Pol I (0.03mg / ml de cloranfenicol) y el lugar de la colonia en una prueba tubo que contiene 8 ml de LB con cloranfenicol. Mantener el cultivo a 30 ° C y agitación a 250rpm durante la noche.
- Por la mañana, expandir la cultura mediante el vertido de los 8 ml de JS200 LF-Pol I en un frasco con 400 ml de LB con cloranfenicol. Deje que la cultura crezca a 30 ° C mientras se agita a 250rpm hasta que alcanza un 600 A de 0.4 hasta 0.7 (por lo general 3-4h).
- Una vez en un 600 A de 0.4-0.7, enfriar la cultura en hielo durante 15 minutos.
- Transferir la cultura de frío a un recipiente adecuado para su centrifugación. Pellet células por centrifugación a 4 ° C. Retirar el sobrenadante y agregar 10 ml de agua estéril y frío (en hielo) de glicerol al 10% en el recipiente y volver a suspender las células mediante una pipeta serológica.
- Transferencia de las células suspendidas de nuevo en un tubo cónico de 50 ml. Llene el tubo cónico hasta la marca de 45 ml con 10% de glicerol y centrifugar durante 15 minutos a 4 ° C y 4000rpm.
- Retirar el sobrenadante, añadir ml, una vez más 10 de 10% de glicerol en el tubo cónico, y volver a suspender las células mediante una pipeta serológica o normal. Una vez más, llenar el tubo cónico de la marca de 45 ml con 10% de glicerol y se centrifuga durante 15 minutos a 4 ° C y 4000rpm. Repita este proceso dos veces más para eliminar todos los restos de sales.
- Después de la vuelta final, volver a suspender el pellet de células en partes iguales el 10% de glicerol (es decir, volver a suspender 2 ml de células con 2 ml de glicerol al 10%).
- Alícuota de entre 100 L y 500 L de la suspensión de células en tubos de almacenamiento de varios. De congelación rápida de las células en hielo seco y luego almacenarlos a -80 ° C.
- Antes de utilizar las células para la electro-competentes transformaciones, descongelar las células lentamente sobre el hielo.
2. Mutagénesis: La transformación de la meta del plásmido
- Pipeta de 40 ml de la electro-competentes JS200 LF-Pol células I y entre 30 250 ng del ADN diana plásmido en una cubeta de electroporación de 2 mm brecha.
Nota # 1: Un ColE1 GFP plásmido que contiene puede ser llevado a través de la mutagénesis en paralelo con el objetivo de genes como un control. Después de la terminación de la etapa de lectura, las buenas prácticas agrarias pueden ser chapada en placas de agar LB y se visualizan para la mutagénesis. Las colonias que aparecen oscuras o contienen poca inactivación de las mutaciones. - Pulso de la mezcla en el electroporador a 1800V. Compruebe la constante de tiempo (T C) para garantizar unas condiciones uniformes de electroporación para cada muestra, lo ideal es T C = 5.6 seg.
- Recuperar la mezcla de células / ADN en 1 ml de caldo de LB durante 40 minutos a 37 ° C (rescondiciones constrictiva) con agitación a 250 rpm.
- Placa 50 l de la cultura de la recuperación en un plato de agar LB Petri de 100 mm de pre-calentado a 37 ° C que contiene tanto el cloranfenicol y la concentración apropiada de un antibiótico para seleccionar el objetivo plásmido.
Nota: N º 2: Tratar de placa de las células en un "jardín cerca de la" concentración. Un "jardín cerca de la" concentración se define como un número distinto, pero no numerable de las colonias (> 1000 colonias / plato de 100 mm). La dilución plateado, en su caso, dependerá de la electro-competentes las células. Si las células no son muy electro-competente y un "césped cerca de" no se puede lograr mediante siembra de la cultura de la recuperación "limpio" y después centrifugar la cultura de la recuperación de 2 minutos a 4000rpm, se vierte el sobrenadante, resuspender las células en 50 l de caldo de LB y la placa de la cultura. - Incubar los platos y Petri (s) durante la noche a 37 ° C.
3. Mutagénesis: la recuperación de plásmido
- Al día siguiente, lavar los platos Petri con pipeta 2 ml de caldo de LB en las células. Transferencia de las colonias bacterianas de la LB agar placas de Petri con el caldo de LB por "fregar" los de la placa con una varilla de vidrio esterilizado en forma de triángulo. Añadir 1 ml de caldo de LB en primer lugar, recoger la lavada y repita el procedimiento con la segunda ml de caldo de LB.
- Aislar el ADN del plásmido del lavado de placas. (Este plásmido de ADN que constituye la biblioteca).
Nota 3: El lavado de recogida de la placa LB puede ser demasiado denso para mini-prep en su totalidad. Si este es el caso, mini-prep el monto máximo asignado por su mini-kit de preparación (por lo general, esto implicaba diluir su lavado de OD = 1 y preparando ~ 3 ml de la cultura diluida) o la escala hasta un máximo de preparación
4. Iteración
- Restringir 1 g de ADN plásmido aislado con una enzima de restricción que linealiza la I LF-Pol plásmido, pero no corta el objetivo plásmido (Suplementario Fig. 1).
- Limpieza de la digestión de restricción con un kit de purificación de ADN.
Nota 4: Este paso elimina todo rastro de la enzima de restricción y su buffer. Este paso es esencial para mantener la baja concentración de sal para su posterior electro-competentes transformaciones. - Volver a transformar de 30 250 ng de la parte posterior de la biblioteca de destino-plásmido restringido a un espacio ventilado JS200 LF-Pol I células para poner la biblioteca a través de las siguientes rondas de mutagénesis.
Nota # 5: Alineación de la Pol I plásmido con una enzima de restricción que asegura que sólo el objetivo de plásmido se transforma. - Repetir los puntos 2 y 3.
5. Lectura
- Restringir el ADN aislado del plásmido con una enzima de restricción (s) que se alinea tanto el objetivo del plásmido y la Pol I del plásmido. Ejecutar el resumen en un 1% en gel de agarosa para asegurar la cantidad y la calidad de los plásmidos. La restricción ~ 400ng del ADN plásmido aislado suele ser suficiente para su análisis.
- Restringir 1 g de ADN plásmido aislado con una enzima de restricción que linealiza la I LF-Pol plásmido, pero no corta el objetivo plásmido.
- Limpieza de la digestión de restricción con un kit de purificación de ADN.
- Transformar la biblioteca de destino restringido plásmido en una cepa de lectura para caracterizar las mutaciones.
II. Pantalla mutante y Análisis rasgo utilizando placas de gradiente de crecimiento.
Con el fin de ilustrar cómo la gran diversidad genética presente en nuestras bibliotecas puede ser acoplado a una selección funcional, aquí le ofrecemos un protocolo de drogas basado en selecciones funcionales in vivo en E. coli. Este método se basa en el crecimiento a lo largo de un gradiente de drogas en agar sólido. Esto permite la caracterización simultánea de múltiples muestras (hasta 12) en un rango de concentraciones, proporcionando un rango dinámico más amplio que un tratamiento farmacológico solo. Otra ventaja es que la lectura no lineal de este ensayo buffers diferencias moderadas en la viabilidad, dentro de un rango de 2 veces. Por lo tanto, este ensayo de citotoxicidad de resistencia es un medio sólido y rápido para seleccionar bibliotecas de mutantes y para determinar el perfil fenotípico de mutantes individuales. Fig. 2 muestra un ejemplo de cada uno de estos usos: el panel A muestra la selección de mutantes individuales de una desmetilasa humanos oxidativo ABH2 biblioteca. Las colonias que crecen por encima del umbral WT son seleccionados para una mayor protección contra la citotoxicidad causada por el agente de metilación metano sulfonato de metilo (MMS) 7. El panel B muestra un ejemplo de gradientes utilizados para la caracterización de clones individuales. El nivel de resistencia a la tercera generación cefotaxima antibiótico de cefalosporina se muestra en un gradiente de agar para WT β-lactamasas y por dos mutantes de espectro extendido, R164H y E104K R164S G267R 4. Tenga en cuenta que dependiendo de la intensidad de los efectos observados, más que una sola placa puede ser necesario para la cuantificación adecuada: mientras que el gradiente de 0.4mg/ml permite la comparación directa con el control de los clones, el nivel de resistencia de los mutantes más poderosos sólo se puede se realiza mediante una mayor concentracióntración de cefotaxima (4mg/mL).
Materiales
- Equipo
- 100x100x15mm cuadrados Petri (Fisher Ciencia # 0875711A)
- Plato de Petri de 100 mm ronda
- 25x75x1mm vidrio portaobjetos del microscopio (Fischer Ciencia # 1255015)
- Tubo de 50 ml graduado
- Medios de comunicación
- Agar LB: derretido y equilibrada en un baño de agua a 56 ° C
Nota 6: la temperatura de los medios de comunicación pueden afectar a la estabilidad y por lo tanto la actividad de la droga o un compuesto que se examina. - Soft Agar: derretido y equilibrada en un baño de agua a 42 ° C
- Agar LB: derretido y equilibrada en un baño de agua a 56 ° C
1. La construcción de la pendiente
- Marca de diez carriles, de manera uniforme a través de uno de los bordes de la parte inferior de la plaza una placa de Petri.
- Coloque el plato en una forma que el borde inferior marcada inclinación se eleva de 7 mm;
una espesa hierba u otro objeto plano se puede utilizar como apoyo para elevar el plato. Verter 25 ml de agua caliente (~ 56 ° C) agar LB mezcla bien con una concentración adecuada del agente de selección en el plato inclinado. Esta es la capa inferior de la pendiente. Asegúrese de que el agar LB uniformemente cubre la placa de Petri de tal manera que el extremo elevada, marcada del plato contiene ~ 1 mm de agar LB y la parte baja contiene ~ 8 mm de agar LB. Luego permitir agar para establecer durante 10-15 minutos.
Nota N º 7: Para la selección de los agentes hidrofóbicos, el 0,1% de surfactante (antiespumante B emulsión) se pueden añadir al agar LB para facilitar la suspensión y la distribución uniforme de la droga. Añadir surfactante para el agar LB estéril caliente con agitación vigorosa antes de la adición del agente de selección. El surfactante debe suspender como una niebla fina de pequeñas gotas, gotas grandes indican los medios de comunicación está muy caliente y puede inhibir la distribución uniforme de la prueba de la droga. - Después de los primeros 25 ml de agar LB se ha endurecido, el plato se mueve a una superficie plana. A continuación, 25 ml de agar LB sin el agente de selección se vierte a la superposición de la superficie de agar LB primero. Esta es la capa superior de la pendiente. Asegúrese de que el agar LB cubre toda la superficie de la capa inferior. Cubrir con la tapa ladeada para la ventilación, y permitir que el agar para establecer durante 10-15 minutos.
Nota N º 8: Sea consciente de los peligros potenciales de aerosol y la volatilización del compuesto de ensayo, vierta los gradientes en una campana de seguridad química o biológica si se lo prescribe los requisitos de seguridad química.
Nota N º 9: platos de gradiente debe ser utilizado dentro de 4 horas para mantener el gradiente de concentración de la droga o sustancia de ensayo.
2. Sello de la transferencia de bacterias
- Agar blando deben equilibrarse a 42 ° C. Transferencia de 2 ml de agar blando líquido en la tapa o el fondo de un plato de 100 mm ronda Petri. Pipetear 40 ml del cultivo bacteriano en el agar suave y luego mezclar con balanceo de la placa.
Nota N º 10: La etapa de crecimiento de un cultivo de bacterias puede afectar su respuesta a un fármaco o compuesto de ensayo. Culturas en la fase de registro o de cultivos de una noche en la fase estacionaria debe ser utilizado con la consistencia de resultados uniformes. Densidad de las células también pueden sesgar los resultados relativos, por lo tanto todas las culturas deben ser diluidas a haber igualado A 600 valores de densidad. Cultivos de una noche debe ser diluido para tener un A 600 de densidad inferior a 1,0 - Cubra el borde largo de la lámina de vidrio con la mezcla de agar blando. Entonces, alineando el borde recubierto de la diapositiva con la marca inferior (de menor a mayor concentración) en el plato de gradiente, toque en la diapositiva a la superficie del agar. Un toque suave es suficiente para transferir una cinta de agar blando en la superficie de gradiente. La diapositiva se deje a un lado para la limpieza y la reutilización.
- Repita este proceso, para el resto de las muestras de bacterias. Las muestras de referencia deben ser incluidos en cada plato gradiente gradientes si varios están en ejecución.
- Incubar la parte superior plato gradiente de un día para otro a 37 ° C. Tiempos y temperaturas de incubación puede variar para la lectura de diferentes cepas de bacterias, pero la noche a 37 ° C suele ser suficiente para el crecimiento visible.
3. Imágenes y el análisis del crecimiento
- Imágenes: Después de un crecimiento durante la noche, los gradientes pueden ser fotografiado directamente o fijan y se tiñen con una solución de 0,2 mg / ml naranja de acridina en EtOH al 95%, para mejorar el contraste. La placa se incuba a temperatura ambiente durante 5 minutos, en la solución colorante, luego se lava con EtOH al 95% y luego reflejado en una caja de luz UV.
Nota N º 11: Tenga cuidado de no lavar las colonias de la placa de roca, sino más bien la solución de tinción y se lava sobre el plato y quitar las soluciones de las esquinas. - Para el análisis de fenotipo de cada uno de los plásmidos mutantes, la distancia de crecimiento contra el gradiente de concentración se normaliza en un estándar en cada gradiente. Estos valores relativos se puede comparar a través de gradientes.
Nota N º 12: Dependiendo de la naturaleza del efecto citotóxico, un borde filoso o un borde más difuso se puede observar (comparar por ejemplo los paneles A y B en la Fig. 2). En el caso de bordes difusos, es aconsejable medir el borde of continuo crecimiento, como las colonias individuales tienden a mostrar una mayor variabilidad. - Para la selección de la biblioteca, las colonias individuales que crecen en concentraciones superiores a las de control de tipo salvaje padres son aislados y secuenciados para identificar las mutaciones que contribuyen a una mayor protección.
III. Los resultados representativos:
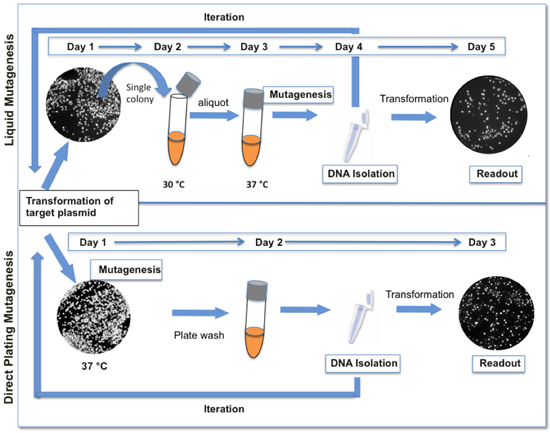
Figura 1. Comparación entre los protocolos de mutagénesis líquido y directo recubrimiento. El protocolo de placas mutagénesis dirigida se presenta aquí (abajo) es más rápido y requiere menos pasos que el protocolo original mutagénesis líquido (arriba). Al GFP se utiliza como un periodista, las variaciones en la fluorescencia son indicativos de la diversidad genética presente en la biblioteca. Por lo general, un ciclo de los resultados de mutagénesis en las colonias de 12-18% con niveles sensiblemente menor de la fluorescencia.
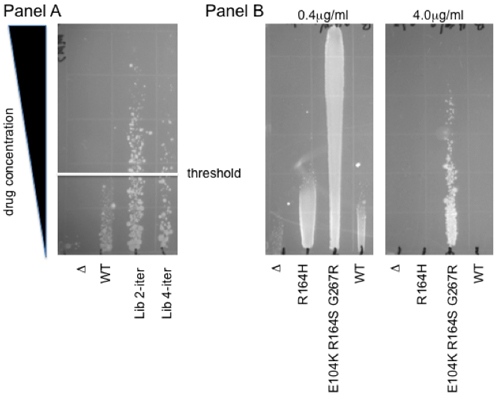
Figura 2. Ensayos de resistencia a la gradiente. El panel A de selección para una mayor resistencia a metil-metanosulfonato (MMS). Bibliotecas plásmido del ser humano desmetilasa oxidativo ABH2 fueron seleccionados para una mayor resistencia a MMS. Dos bibliotecas como representantes de dos y cuatro iteraciones del protocolo de mutagénesis se muestran en comparación con el tipo de padres silvestres (WT) y el vector vacío (Δ). La línea blanca representa el umbral por encima del individuo que las colonias mutantes aisladas para el análisis fenotípico más. Grupo B de protección cefotaxima, indicativo de β-lactamasas de espectro extendido la actividad. R164H y R164H E104K G267R, dos mutantes de beta-lactamasa identificados previamente siguiente LF- Pol mutagénesis que junto a la selección de aztreonam 4, se muestran en una 0.4μg/mL y un gradiente de 4μg/mL cefotaxima. Tenga en cuenta que la naturaleza de tipo β-lactamasas de la enzima no confiere protección en relación con las células que expresan un vector vacío, Δ. Por lo tanto, estos mutantes representan la evolución de una nueva actividad bioquímica de 8,9.
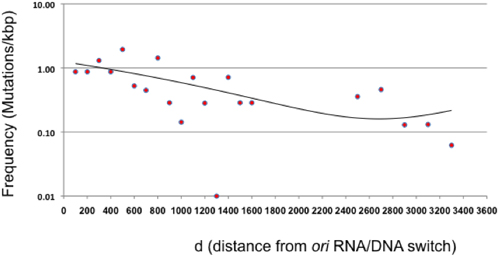
Figura 3. La frecuencia de mutación en función de la distancia de ori (d). La frecuencia de mutación después de un solo ciclo de mutagénesis siembra directa se muestra a intervalos de 100 pb en relación con el cambio del ADN / ARN del origen de replicación ColE1. El área entre 1600 y 2400 no está representado por β-lactamasas no representa un blanco neutral. Los puntos más allá de β-lactamasas representan intervalos de 200 pb debido a la frecuencia general de baja de la mutación en esta área. La tendencia (ecuación binomial, con un R 2 = 0,41) se muestra como una línea.

Complementaria la figura 1. LF Pol I-Pol I que contiene el plásmido. Una secuencia (en formato FASTA). Secuencia de la información para la determinación de la enzima de restricción adecuada (s) a utilizar al linealizar el plásmido que Pol ya sea para la iteración (generación de una etapa de mutación al azar de la biblioteca 4) o de lectura (paso 5). B Características generales y mapa de restricción del plásmido. El localización del origen de la replicación del pSC101, el marcador de resistencia a cloranfenicol (CAT) y LF-Pol gen que se presentan. La ubicación de sitios de restricción solo se indica también.
| Biblioteca | La siembra directa | Líquido (1 día) | Líquido (3 días) | |
| Las mutaciones (#) | 95 | 40 | 142 | |
| Clones secuenciados | 288 | 96 | 190 | |
| Cobertura Total (pb) | 182000 | 102000 | 213000 | |
| Frecuencia de mutación (x10 3 pb) | 0.52 | 0.39 | 0.67 | |
| Frecuencia d <1000 (x10 3 pb) | 0.92 | 0.41 | 0.70 |
Tabla 1. Las frecuencias de mutación frecuencias (expresado como N º de mutaciones / pb) para d <1000, es decir, dentro de los 1000 pb al lado del interruptor de ADN / ARN, de tres protocolos de mutagénesis:. Siembra directa, la saturación de líquido (1 día), y hypersaturation líquido ( 3 días).
| La siembra directa | Líquido | ||
| Las mutaciones (#) | 95 | 182 | |
| Espectro (%) | |||
| A a la G | A a la G | 6.3 | 19.2 |
| T de C | 4.2 | 3.8 | |
| C y T | G a A | 27.4 | 13.7 |
| C y T | 35.8 | 35.2 | |
| A T | A T | 5.3 | 8.2 |
| T A | 5.3 | 7.1 | |
| T a G | T a G | 1.1 | 1.1 |
| A a C | 0.0 | 2.2 | |
| G a T | G a T | 1.1 | 2.7 |
| C a A | 2.1 | 2.7 | |
| C y G | C y G | 2.1 | 1.1 |
| G a C | 5.3 | 2.7 | |
| Indeles | Ins | 3.2 | 0.0 |
| Del | 1.1 | 0.0 | |
| A a N | 11.6 | 29.7 | |
| G a N | 33.7 | 19.2 | |
| T a N | 10.5 | 12.1 | |
| C y N | 40.0 | 39.0 | |
| Ts | 73.7 | 72.0 | |
| Televisión | 22.1 | 28.0 | |
| Indeles | 4.2 | 0 |
Tabla 2. Métricas de la mutación de la siembra directa y la mutagénesis líquido. La tabla presenta el número de mutaciones observadas (en número) y el espectro de mutaciones (en%) después de un solo ciclo de mutagénesis. El espectro se divide en pares complementarios, por los cambios de nucleótidos, y por el tipo de mutación.
Discusión
En este artículo se presenta un protocolo de mutagénesis que permite la generación de grandes bibliotecas de mutantes al azar sin la necesidad de que la clonación o la PCR. Este método se basa en propenso a errores de replicación de un plásmido que codifica una secuencia de interés. En teoría, las mutaciones se debe en gran parte limitada a los 100 a 300 pb situado inmediatamente aguas abajo del interruptor de ADN / ARN, el tamaño de la cadena líder intermedios producidos por Pol I 2,10. Hemos encontrado que en las condiciones se presentan en este protocolo, Pol mutaciones que ocurren en todo el plásmido, a pesar de la disminución en la frecuencia como la distancia a partir del plásmido aumenta ori (Figura 3). Este hallazgo implica que la transición o "switch" de Pol I Pol III durante la replicación del plásmido ColE1 es mucho más gradual que se informó anteriormente, por lo menos bajo condiciones experimentales dos, y está de acuerdo con estudios anteriores que sugiere una redundancia funcional entre I y Pol Pol III 11.
El protocolo original describe mutagénesis en cultivos líquidos (4). Este protocolo rendimientos 0,41 mutaciones / kb para d <1000, es decir, dentro de los 1000 pb al lado del ADN / ARN interruptor (Tabla 1). Hypersaturation dejando el cultivo líquido en el agitador durante 3 días sin la adición de nuevos medios de comunicación aumenta la frecuencia de mutación de 0,70 mutaciones / kb, pero los resultados en el rendimiento de plásmido muy pobres (menos del 1% en comparación con el día 1; datos no mostrados) ( Tabla 1). Aquí se presenta un protocolo simplificado basado en placas directamente la transformación de la meta del plásmido en agar sólido a 37 ° C (Figura 1). Este procedimiento produce la mayor frecuencia de mutación / ciclo de mutagénesis (0,92 mutaciones / kb para d <1,000) y facilita la iteración. Cambiar las condiciones de cultivo para los medios sólidos también afecta a la mutación del espectro (Tabla 2). Mutagénesis siembra directa reduce la marcada asimetría entre los pares de bases complementarias sustituciones visto en cultivo líquido (compare C → T vs G → A y A → G vs T → C). Por otro lado, la siembra directa produce más indeles (de menos de 0,5% a 4%) y disminuyó el número de mutaciones A → G por tres veces, dando lugar a una sobrerrepresentación de G / C mutaciones (74%). En general, creemos que la mayor simplicidad y una mayor eficiencia del protocolo de siembra directa que aquí se presenta mayor que los beneficios de la mutación del espectro un poco más equilibrada producida por mutagénesis líquido.
Dentro del objetivo de plásmido, las mutaciones generadas por nuestro método no se limitan a la secuencia de destino deseado. Sin embargo, la evolución de la novela de las actividades bioquímicas debe depender de las mutaciones en el gen de interés, ya que representa un salto cualitativo en lugar de un cambio cuantitativo. Por lo tanto, la combinación de nuestra mutagénesis plásmido con la detección de mutantes en las placas de gradiente aprovecha los puntos fuertes de nuestro sistema (las grandes bibliotecas, y la disponibilidad de la selección) para el desarrollo de nuevas propiedades bioquímicas. Otras aplicaciones de la mutagénesis al azar, tales como la optimización de las actuales actividades enzimáticas o asignaron al azar a las áreas específicas de un gen requieren clonación siguientes mutagénesis aleatoria. En este caso, tener la biblioteca en un plásmido en comparación con un producto de amplificación por PCR mejora la eficiencia de la clonación, al facilitar la amplificación y restricción.
En resumen, hemos demostrado un protocolo sencillo para crear una biblioteca de mutantes al azar de un gen diana, ya que destaca por su sencillez y por la diversidad de las bibliotecas generado. Se muestra cómo este método puede ser, junto con las selecciones funcionales para la evolución eficiente de las nuevas actividades bioquímicas. Además, nuestro in vivo generados por las bibliotecas pueden ser fácilmente clonadas, lo que permite mutagénesis sitio-específica o la optimización de las actividades existentes.
Divulgaciones
Agradecimientos
Este trabajo ha sido financiado por el premio K08 CA116429-04 en MC y por una beca de la Conquista del Cáncer Ahora (preocupación) base 8501 #
Materiales
| Name | Company | Catalog Number | Comments |
| carbenicillin | Cellgro | 46100R6 | 0.1mg/ml final |
| tetracycline | Fisher Scientific | BP7640 | 0.05mg/ml final |
| chloramphenicol | Genlantis | M120100 | 0.03mg/ml final |
| LB Agar Miller | Fisher Scientific | BP1425 | |
| LB Broth Miller | Difco Laboratories | 244620 | |
| Soft Agar | Difco Laboratories | 214580 | Made in house |
| Acridine Orange | Sigma-Aldrich | A38401-1 | |
| Antifoam B emulsion | Sigma-Aldrich | A5757 | |
| Glycerol | Acros Organics | 332030025 | |
| 100x100x15mm sq dish | Fisher Scientific | 0875711A | |
| 100mm rnd dish | Fisher Scientific | 0875712A | |
| Microscope slide 25x75x1mm | Gold Seal | 3048 | |
| Electroporator 2510 | Eppendorf | ||
| Electroporator 2510 | Eppendorf | ||
| 2mm gap tubes | Molecular BioProducts | 5520 | |
| Zippy mini prep kit | Zymo Research Corp. | D4020 |
Referencias
- Camps, M. Modulation of ColE1-like plasmid replication for recombinant gene expression. Recent Pat DNA Gene Seq. 4, 58-73 (2010).
- Itoh, T., Tomizawa, J. Initiation of replication of plasmid ColE1 DNA by RNA polymerase, ribonuclease H, and DNA polymerase I. Cold Spring Harb Symp Quant Biol. 43 Pt 1, 409-417 (1979).
- Polisky, B. ColE1 replication control circuitry: sense from antisense. Cell. 55, 929-932 (1988).
- Camps, M., Naukkarinen, J., Johnson, B. P., Loeb, L. A. Targeted gene evolution in Escherichia coli using a highly error-prone DNA polymerase I. Proc Natl Acad Sci U S A. 100, 9727-9732 (2003).
- Shinkai, A., Loeb, L. A. In vivo mutagenesis by Escherichia coli DNA polymerase I. Ile(709) in motif A functions in base selection. J Biol Chem. 276, 46759-46764 (2001).
- Uyemura, D., Lehman, I. R. Biochemical characterization of mutant forms of DNA polymerase I from Escherichia coli. I. The polA12 mutation. J Biol Chem. 251, 4078-4084 (1976).
- Sedgwick, B., Robins, P., Lindahl, T. Direct removal of alkylation damage from DNA by AlkB and related DNA dioxygenases. Methods Enzymol. 408, 108-120 (2006).
- Aharoni, A., Gaidukov, L., Khersonsky, O., Mc, Q. G. S., Roodveldt, C., Tawfik, D. S. The 'evolvability' of promiscuous protein functions. Nat Genet. 37, 73-76 (2005).
- Elena, S. F., Lenski, R. E. Evolution experiments with microorganisms: the dynamics and genetic bases of adaptation. Nat Rev Genet. 4, 457-469 (2003).
- Itoh, T., Tomizawa, J. FoFormation of an RNA primer for initiation of replication of ColE1 DNA by ribonuclease H. Proc Natl Acad Sci U S A. 77, 2450-2454 (1980).
- Bryan, S. K., Moses, R. E. Sufficiency of the Klenow fragment for survival of polC(Ts) pcbA1 Escherichia coli at 43 degrees. C. J Bacteriol. 170, 456-458 (1988).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados