Method Article
Mutagênese e Funcional Protocolos de Seleção para a Evolução Dirigida de proteínas em E. coli
Neste Artigo
Resumo
Aqui demonstramos um protocolo simples para criar uma biblioteca aleatória mutante para uma seqüência alvo. Nós mostramos como esse método, que é realizado in vivo em Escherichia coli, pode ser acoplado com seleções funcional para evoluir novas atividades enzimáticas.
Resumo
A geração eficiente de diversidade genética representa uma ferramenta valiosa molecular que pode ser usado para rotular a síntese de DNA, para criar assinaturas única molecular, ou a evoluir proteínas no laboratório. Aqui, apresentamos um protocolo que permite a geração de grandes (> 10 11) bibliotecas mutante para uma seqüência alvo. Este método é baseado na replicação de um plasmídeo codificando ColE1 a seqüência desejada por uma variante de baixa fidelidade da DNA polimerase I (LF-Pol I). O alvo do plasmídeo é transformada em uma cepa de E. mutator coli e semeadas em meios sólidos, rendendo entre 0,2 e 1 mutações / kb, dependendo da localização do gene alvo. Freqüências mais altas de mutação são alcançados por iteração deste processo de mutagênese. Em comparação com métodos alternativos de mutagênese, nosso protocolo destaca-se pela sua simplicidade, como nenhuma clonagem ou PCR estão envolvidos. Assim, nosso método é ideal para a rotulagem de mutação de plasmídeos ou modelos I, com Pol ou para explorar grandes seções de espaço de seqüência para a evolução das actividades não presentes no alvo original. O rígido controle espacial que PCR ou randomizado oligonucleotídeos baseados em oferecer métodos também podem ser alcançados por meio da clonagem posterior de seções específicas da biblioteca. Aqui nós fornecemos protocolos mostrando como criar uma biblioteca de mutantes aleatórios e como estabelecer droga baseada em seleções em E. coli para identificar mutantes exibindo novas atividades bioquímicas.
Protocolo
I. Geração de uma Biblioteca Aleatório Mutant
Pol I iniciação media de ColE1 replicação do plasmídeo (revisto em (1-3). Nosso método de mutagênese se baseia em colocar uma seqüência alvo de um plasmídeo adjacentes ColE1 a origem ou a replicação e propagação em células que expressam baixa fidelidade DNA polimerase I (LF-Pol I). LF-Pol I é uma DNA polimerase mutante eu codificação de três mutações que diminuem a fidelidade da replicação, ou seja, I709N (em motif A), A759R (em motivo B) e D424A (inativação de revisão) 4,5 . LF-Pol I é expressa em uma cepa de E. coli, JS200, que tem um alelo sensíveis à temperatura da Pol I (polA12) (6), para que LF-Pol I torna-se a atividade predominante a 37 ° C. A replicação do seqüência alvo em polA12 células sob condições restritivas resultados na geração de uma biblioteca aleatória mutante. Mutagênese é mais eficiente em culturas saturadas 4. Por razões que ainda não estão claros, mutagênese não é contínua, ou seja, a freqüência de mutação não aumenta linearmente com o número das gerações uma vez que a cultura atinge a saturação, mesmo se as células estão autorizados a expandir ainda mais na mídia fresco. Portanto, aumentando ainda mais a carga de mutação da biblioteca exige iterativo rodadas de mutagênese e recuperação plasmídeo. Aqui nós fornecemos protocolos para LF-Pol mutagênese I. Note-se que os protocolos aqui apresentados foram bastante simplificadas em relação a nossos 4 descrição original, a fim de facilitar a iteração do processo para atingir a carga mutação desejada (Fig. 1).
Materiais
- Células: JS200 recA718 polA12 (ts) uvrA155 trpE65 lon-11 sula
- JS200 WT-Pol I: JS200 células que expressam tipo selvagem (WT) Pol I
- JS200 LF-Pol I: JS200 expressando fidelidade baixa (LF) Pol I
- Tensão de leitura: JS200 WT-Pol I ou (para complementação) uma cepa falta de uma atividade específica
- Alvo Plasmid
- Plasmídeo contendo uma origem de replicação ColE1 com o gene alvo clonado em
1. Antes de Mutagênese: Preparação de electro-JS200 células competentes LF-Pol I
- Escolher um único JS200 LF-Pol colônia I de uma placa de LB crescido a 30 ° C (condições permissivas) durante a noite contendo a seleção apropriada de antibióticos para o rolamento plasmídeo LF-Pol I (0,03 mg / mL cloranfenicol) e coloque a colônia em um teste tubo contendo 8 mL de LB com cloranfenicol. Crescer a cultura a 30 ° C e agitação a 250rpm durante a noite.
- Na parte da manhã, expandir a cultura, derramando o mL 8 de JS200 LF-Pol I em um frasco contendo 400 mL LB com cloranfenicol. Deixe crescer a cultura a 30 ° C agitando a 250rpm até atingir um 600 A de ,4-0,7 (normalmente 3-4h).
- Uma vez em um 600 A de 0,4-0,7, chill a cultura no gelo molhado por 15 minutos.
- Transferir a cultura refrigerados para um recipiente adequado para a centrifugação. Pellet células por centrifugação a 4 ° C. Deitar fora o sobrenadante, e depois adicionar 10 mL de glicerol estéril e refrigerados (em gelo molhado) 10% para o recipiente e volte a suspender as células com uma pipeta sorológica.
- Transferência das células re-suspensas em tubo cônico de 50 mL. Encha o tubo cônico até a marca de 45 mL de glicerol 10% e, em seguida, centrifugar por 15 minutos a 4 ° C e 4000rpm.
- Deitar fora o sobrenadante, adicionar 10 mL uma vez mais, de glicerol 10% para o tubo cônico, e re-suspender as células com uma pipeta sorológica ou normal. Mais uma vez, encher o tubo cônico de 45 mL da marca com 10% de glicerol e centrifugar por 15 minutos a 4 ° C e 4000rpm. Repita esse processo mais duas vezes para remover todos os vestígios de sais.
- Após a rodada final, re-suspender o pellet de células em partes iguais o glicerol 10% (ie re-suspensão 2 mL de células com 2 mL de glicerol 10%).
- Alíquota entre 100 mL e 500 mL da suspensão de células em tubos de armazenamento diversos. Quick-congelar as células em gelo seco e, em seguida, armazená-los a -80 ° C.
- Antes de usar as células para eletro-competentes transformações, descongelar as células lentamente em gelo.
2. Mutagênese: Transformando o alvo plasmídeo
- Pipetar 40 mL do electro-JS200 células competentes LF-Pol I e entre 30-250ng do DNA alvo plasmídeo em uma cubeta de eletroporação gap 2mm.
Nota # 1: A ColE1 plasmídeo contendo GFP pode ser realizada através de mutagênese em paralelo com o gene alvo, um controle. Após a conclusão da etapa de leitura, GFP pode ser semeadas em placas de ágar LB e visualizados por mutagênese. Colônias que estão escuras ou dim conter mutações inativadoras. - Pulso a mistura no electroporator a 1800V. Verificar a constante de tempo (T C) para garantir condições de eletroporação uniforme para cada amostra; idealmente T C = 5-6 seg.
- Recuperar a mistura de células / DNA em 1 mL de caldo LB por 40 minutos a 37 ° C (rescondições trictive), agitando a 250 rpm.
- ML placa 50 da cultura de recuperação em um prato de agar LB 100 milímetros Petri pré-aquecido a 37 ° C contendo tanto cloranfenicol e uma concentração adequada de um antibiótico para selecionar o alvo plasmídeo.
Nota: # 2: Procure a placa de células em um "perto do gramado" concentração. A "perto do gramado" concentração é definido como um número distinto, mas incontável de colônias (> 1000 colônias / prato 100mm). A diluição banhado, se houver, vai depender de como eletro-competentes as células são. Se as células não são muito electro-competente e um "gramado próximo" não é alcançável por plaqueamento da cultura de recuperação "puro", em seguida, centrifugar a cultura de recuperação de 2 minutos a 4000rpm, despeje o sobrenadante, ressuspender as células em 50 mL de caldo LB e placa de cultura. - Incubar a dishe Petri (s) durante a noite a 37 ° C.
3. Mutagênese: recuperação Plasmid
- No dia seguinte, lave os pratos de Petri por pipetagem 2 mL de caldo LB sobre as células. Transferir as colônias de bactérias a partir do agar LB pratos de Petri para o caldo LB por "lavagem"-los fora da placa com uma vareta de vidro esterilizado em forma de triângulo. Adicionar 1 mL de caldo LB primeiro, recolher a lavagem e repetir o procedimento com a segunda mL de caldo LB.
- Isolar o DNA do plasmídeo da lavagem de placas. (Este DNA plasmídeo constitui a biblioteca).
Nota # 3: A lavagem coletados a partir da placa LB pode ser muito denso para mini-prep na sua totalidade. Se este for o caso, mini-prep máximo o valor atribuído pelo seu kit mini-prep (tipicamente, isto envolveu diluindo sua lavagem para OD = 1 e preparando ~ 3 mL da cultura diluída) ou escala de até um maxi-prep
4. Iteração
- Restringir 1μg do DNA isolado do plasmídeo com uma enzima de restrição que lineariza o LF-Pol I plasmídeo, mas não cortar seu alvo plasmídeo (Recurso Fig. 1).
- Limpar a digerir restrição usando um kit de purificação de DNA.
Note # 4: Esta etapa remove todos os traços da enzima de restrição e seu buffer. Este passo é essencial para manter a baixa concentração de sal para posterior eletro-competentes transformações. - Re-transform 30-250ng do restrito volta biblioteca-alvo plasmídeo em fresco JS200 LF-Pol eu células para colocar a biblioteca através de rondas subsequentes de mutagênese.
Nota 5: linearização da Pol I plasmídeo usando uma enzima de restrição garante que somente o alvo plasmídeo se transforma. - Repita as seções 2 e 3.
5. Leitura
- Restringir o DNA isolado do plasmídeo com uma enzima de restrição (s) que lineariza alvo tanto do plasmídeo e da Pol I plasmídeo. Executar o digest em um gel de agarose 1% para garantir a quantidade ea qualidade dos plasmídeos. Restringindo ~ 400ng do DNA isolado do plasmídeo é geralmente suficiente para análise.
- Restringir 1μg do DNA isolado do plasmídeo com uma enzima de restrição que lineariza o LF-Pol I plasmídeo, mas não cortar seu alvo plasmídeo.
- Limpar a digerir restrição usando um kit de purificação de DNA.
- Transformar a biblioteca de destino restrito plasmídeo em uma cepa de leitura para caracterizar as mutações.
II. Tela mutante e Análise de Traço Usando placas de crescimento Gradiente.
A fim de ilustrar como a grande diversidade genética presente em nossas bibliotecas podem ser acoplados a uma seleção funcional, aqui nós fornecer um protocolo de drogas baseado em seleções funcional in vivo em E. coli. Este método baseia-se crescimento ao longo de um gradiente de droga em ágar sólido. Isto permite a caracterização simultânea de múltiplas amostras (até 12) em uma faixa de concentrações, proporcionando uma maior gama dinâmica do que um tratamento medicamentoso único. Outra vantagem é que a leitura não-linear deste ensaio buffers diferenças moderadas da viabilidade, dentro de uma faixa de 2 vezes. Assim, este ensaio de citotoxicidade resistência fornece um meio robusto e rápido para selecionar mutantes e bibliotecas para determinar o perfil fenotípico de mutantes individual. Fig. 2 mostra um exemplo de cada um desses usos: Um painel de seleção de mutantes mostra individual de um oxidativo demetilase humana ABH2 biblioteca. Colônias que crescem acima do limiar WT são selecionados para maior proteção contra a citotoxicidade causada pela metilação de metano sulfonato de agente de metila (MMS) 7. Painel B mostra um exemplo de gradientes utilizados para a caracterização clone individual. O nível de resistência à terceira geração de cefalosporinas com antibióticos cefotaxima é mostrado em um gradiente de agar para WT β-lactamase e por duas espectro estendido-mutantes, R164H e R164S E104K G267R 4. Note que, dependendo da força dos efeitos observados, mais do que uma única placa pode ser necessário para a quantificação adequada: enquanto o gradiente 0.4mg/ml permite a comparação direta com o controle de clones, o nível de resistência do mutante mais poderosa só pode ser estabelecida usando uma maior concentraçãoconcentração de cefotaxima (4mg/ml).
Materiais
- Equipamento
- 100x100x15mm quadrados placas de Petri (Fisher Sci # 0875711A)
- 100 milímetros de Petri rodada
- 25x75x1mm lâmina de vidro (Fischer Sci # 1255015)
- 50 tubo de ml
- Mídia
- Agar LB: derretido e equilibrada em banho-maria a 56 ° C
Nota 6: temperatura da mídia pode afetar a estabilidade e, portanto, a atividade da droga ou composto sendo examinados. - Agar Soft: derretido e equilibrada em banho-maria a 42 ° C
- Agar LB: derretido e equilibrada em banho-maria a 56 ° C
1. Construção do gradiente
- Marca de dez faixas, espaçados uniformemente ao longo de uma borda da parte inferior da placa de Petri quadrados.
- Coloque o prato sobre uma inclinação tal que a borda inferior é elevado marcado sete milímetros;
uma grama grossa ou outro objeto plano pode ser usado como apoio para elevar o prato. Despeje 25 ml de água morna (~ 56 ° C) agar LB bem misturada com uma concentração adequada do agente selecionando no prato inclinado. Esta é a camada inferior do gradiente. Verifique se o agar LB uniformemente casacos placa de Petri de tal forma que ao final, marcada elevados do prato contém ~ 1mm de agar LB ea parte baixa contém ~ 8mm de agar LB. Em seguida, permitir agar para definir por 10-15 minutos.
Nota 7: Para selecionar agentes hidrofóbicos, surfactante 0,1% (antiespumante B emulsão) podem ser adicionados ao ágar LB para facilitar a suspensão e distribuição uniforme da droga. Adicionar surfactante para o agar LB morna estéril com agitação vigorosa antes da adição do agente de seleção. O surfactante deve suspender como uma névoa fina de pequenas gotículas, gotículas grandes indicam os meios de comunicação é muito quente e pode inibir a distribuição uniforme da droga teste. - Após a primeira de 25 mL de agar LB tem endurecido, o prato é movido para uma superfície plana. Em seguida, 25 mL de agar LB sem o agente seleção é derramado para cobrir a superfície de ágar primeiro LB. Esta é a camada superior do gradiente. Verifique se o agar LB cobre toda a superfície da camada inferior. Cubra com askew tampa para ventilação e permitir agar para definir por 10-15 minutos.
Nota 8: Esteja ciente dos riscos potenciais de aerossóis e volatilização do composto de ensaio, deitar gradientes em uma capa de segurança química ou biológica se prescrito pelos requisitos de segurança química.
Note # 9: pratos Gradiente deve ser utilizado dentro 4h para preservar o gradiente de concentração do medicamento ou substância de prova.
2. Selo de transferência de bactérias
- Ágar mole deve ser equilibrado a 42 ° C. Transferir 2 mL de ágar mole líquido na tampa ou fundo de um prato de Petri 100 milímetros rodada. Pipetar 40 mL da cultura bacteriana no agar macio e então misture balançando a placa.
Note # 10: O estágio de crescimento de uma cultura bacteriana pode afetar sua resposta a uma droga ou substância de prova. Culturas em fase log ou culturas durante a noite em fase estacionária deve ser usado com consistência para resultados uniformes. Densidade celular também pode distorcer os resultados relativos, assim, todas as culturas devem ser diluídas de ter combinado A 600 valores de densidade. Culturas devem ser diluídas durante a noite para ter um A 600 de densidade inferior a 1,0 - Brasão beira da longa da lâmina de vidro com a mistura de agar macio. Então, alinhando a borda revestida do slide com a marca inferior (de baixo para alta concentração) no prato gradiente, toque no slide para a superfície do ágar. Um toque suave é suficiente para transferir uma fita do ágar mole à superfície gradiente. A lâmina é reservada para limpeza e reutilização.
- Repita esse processo, para o restante das amostras bacterianas. Amostras de referência devem ser incluídos em cada prato, se gradiente gradientes múltiplos estão sendo executados.
- Incubar a top prato gradiente baixo durante a noite a 37 ° C. Tempos e temperaturas de incubação pode variar de leitura diferentes cepas de bactérias, mas durante a noite a 37 ° C é geralmente suficiente para o crescimento visível.
3. Analisando imagens e Crescimento
- Imagem: Depois de um crescimento durante a noite, gradientes podem ser observados directamente ou fixadas e coradas com uma solução de 0,2 mg / mL de acridina laranja em EtOH 95%, para melhorar o contraste. A placa é incubada a temperatura ambiente por 5 min, na solução de coloração, em seguida, lavado com EtOH 95% e, em seguida, com imagens sobre uma caixa de luz UV.
Note # 11: Tome cuidado para não lavar as colônias da placa de rocha, mas sim a solução de coloração e lava sobre a placa e remova as soluções dos cantos. - Para a análise de fenótipo de cada um dos plasmídeos mutante, a distância de crescimento contra o gradiente de concentração é normalizado para um padrão em cada gradiente. Estes valores relativos podem então ser comparados entre gradientes.
Note # 12: Dependendo da natureza do efeito citotóxico, uma borda afiada ou uma borda mais difusa pode ser observada (compare por exemplo Painéis A e B na Figura 2). No caso de bordas difusas, é aconselhável para medir o limitef crescimento contínuo, como colônias individuais tendem a mostrar maior variabilidade. - Para a seleção de biblioteca, colônias individuais que crescem em concentrações mais elevadas do que o controle tipo parental selvagem estão isolados e seqüenciados para identificar as mutações que contribuem para maior proteção.
III. Resultados representativos:
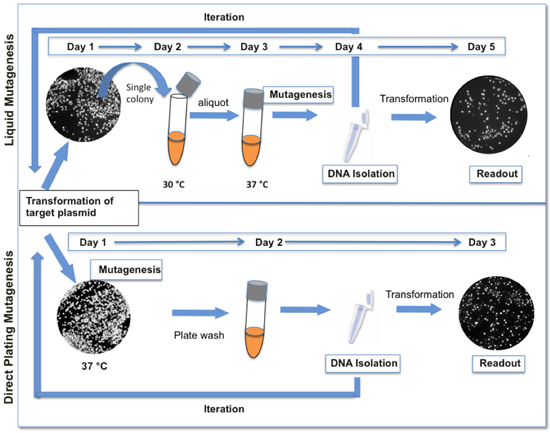
Figura 1. Comparação entre protocolos de mutagênese líquido e direta em placas. O protocolo de mutagênese direta plating apresentado aqui (em baixo) é mais rápido e exige menos etapas do que o nosso protocolo de mutagênese originais líquido (superior). Quando GFP é utilizada como um repórter, as variações de fluorescência são indicativos da diversidade genética presente na biblioteca. Tipicamente, um ciclo de resultados mutagênese em colônias 12-18% com níveis significativamente diminuídos de fluorescência.
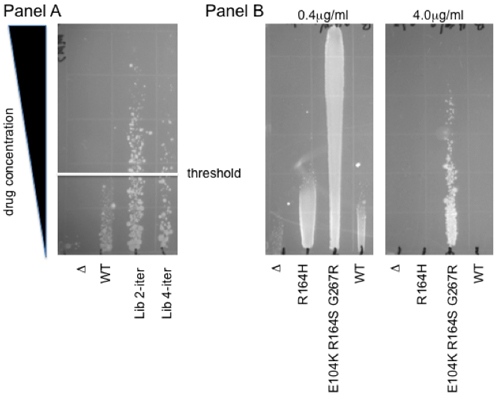
Figura 2. Resistência ensaios gradiente. Um painel de seleção para aumento da resistência ao Metil-metanossulfonato (MMS). Bibliotecas Plasmid do ser humano oxidativo demetilase ABH2 foram selecionados para aumento da resistência ao MMS. Duas bibliotecas que representem dois e quatro iterações do protocolo de mutagênese são mostrados em relação ao tipo parental selvagem (WT) eo vetor vazio (Δ). A linha branca representa o limiar acima do qual individuais colônias mutantes foram isolados para análise fenotípica mais. Painel B proteção Cefotaxima, indicativo de espectro estendido atividade β-lactamase. R164H e E104K R164H G267R, dois mutantes de beta-lactamase previamente identificados seguintes LF- Pol mutagênese eu acoplado ao aztreonam seleção 4, são mostrados em uma 0.4μg/mL e um gradiente cefotaxima 4μg/mL. Note que o tipo selvagem da enzima β-lactamase não confere proteção em relação a células que expressam um vetor vazio, Δ. Portanto, esses mutantes representam a evolução de uma nova atividade bioquímica 8,9.
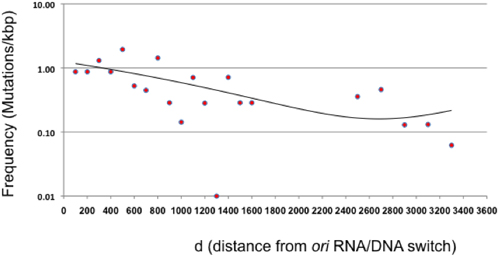
Figura 3. Freqüência de mutação em função da distância da ori (d). A frequência de mutação após um único ciclo de mutagênese plaqueamento direto é mostrado para intervalos de 100 pb em relação ao interruptor de RNA / DNA da origem ColE1 de replicação. A área entre 1600 e 2400 não é representado por β-lactamase não representa um alvo neutro. Os pontos além β-lactamase intervalos representam 200 bp, dada a baixa freqüência de mutação nesta área. A tendência (equação binomial, com um R 2 = 0,41) é mostrado como uma linha.

Figura suplementares 1. Pol LF Pol I-I contendo plasmídeo. A seqüência (formato FASTA). Informações de seqüência para a determinação da enzima de restrição adequada (s) para usar quando linearização da Pol I plasmídeo ou para a iteração (geração de um passo aleatório mutação biblioteca 4) ou de leitura (passo 5). B características gerais e mapa de restrição do plasmídeo. A localização da origem de replicação pSC101, o marcador de resistência cloranfenicol (CAT) e LF-Pol gene que são apresentados. A localização de sítios de restrição único é indicado também.
| Biblioteca | Plating direta | Líquido (1 dia) | Líquido (3 dias) | |
| Mutações (#) | 95 | 40 | 142 | |
| Clones seqüenciados | 288 | 96 | 190 | |
| Cobertura total (bp) | 182000 | 102000 | 213000 | |
| Mutação freq (x10 3 bp) | 0,52 | 0,39 | 0,67 | |
| Freq d <1000 (x10 3 bp) | 0,92 | 0,41 | 0,70 |
Tabela 1. Freqüências de mutação Frequências (expresso em n º de mutações / bp) para d <1000, ou seja, dentro da bp 1000 ao lado do interruptor de RNA / DNA, por três protocolos mutagênese:. Plaqueamento direto, saturação de líquido (1 dia), e hypersaturation líquido ( 3 dias).
| Plating direta | Líquido | ||
| Mutações (#) | 95 | 182 | |
| Espectro (%) | |||
| A a G | A a G | 6,3 | 19,2 |
| T para C | 4,2 | 3,8 | |
| C para T | G a A | 27,4 | 13,7 |
| C para T | 35,8 | 35,2 | |
| A a T | A a T | 5,3 | 8,2 |
| T a A | 5,3 | 7,1 | |
| T para G | T para G | 1,1 | 1,1 |
| A para C | 0,0 | 2,2 | |
| G para T | G para T | 1,1 | 2,7 |
| C para A | 2,1 | 2,7 | |
| C para G | C para G | 2,1 | 1,1 |
| G para C | 5,3 | 2,7 | |
| Indels | Ins | 3,2 | 0,0 |
| Del | 1,1 | 0,0 | |
| A a N | 11,6 | 29,7 | |
| G a N | 33,7 | 19,2 | |
| T para N | 10,5 | 12,1 | |
| C para N | 40,0 | 39,0 | |
| Ts | 73,7 | 72,0 | |
| TV | 22,1 | 28,0 | |
| Indels | 4,2 | 0 |
Tabela 2. Métricas de mutação para plaqueamento direto e mutagênese líquido. O quadro apresenta o número de mutações observadas (em número) eo espectro de mutação (em%) após um único ciclo de mutagênese. O espectro é dividido em pares complementares, por mudanças de nucleotídeos, e pelo tipo de mutação.
Discussão
Este artigo apresenta um protocolo de mutagênese que permite a geração de grandes bibliotecas de mutantes aleatórios sem a necessidade de clonagem ou PCR. Este método baseia-se propenso a erros de replicação de um plasmídeo uma seqüência de interesse. Em teoria, as mutações devem ser em grande parte restrita à 100-300 pb, localizado imediatamente a jusante do interruptor de RNA / DNA, o tamanho do líder vertente intermediários produzidos por Pol I 2,10. Descobrimos que nas condições apresentadas neste protocolo, mutações Pol I ocorrem em todo o plasmídeo, embora diminuindo em freqüência como a distância entre os aumentos plasmídeo ori (Figura 3). Este achado implica que a transição ou "switch" de Pol I Pol III ColE1 durante a replicação do plasmídeo é muito mais gradual do que o anteriormente reportado, pelo menos, nas nossas condições experimentais 2, e concorda com estudos anteriores sugerindo uma redundância funcional entre I e Pol Pol III 11.
Nosso protocolo original descrito mutagênese em culturas de líquido (4). Este protocolo produz mutações 0,41 / kb para d <1000, ou seja, dentro da bp 1000 ao lado do RNA / DNA switch (Tabela 1). Hypersaturation deixando a cultura líquido no shaker durante 3 dias sem a adição de qualquer mídia fresco aumenta a freqüência de mutação para 0,70 mutações / kb, mas resulta em muito pobre rendimento plasmídeo (menos de 1% em relação ao dia 1; dados não mostrados) ( Tabela 1). Aqui apresentamos um protocolo simplificado baseado diretamente plating a transformação do plasmídeo em ágar-alvo sólido a 37 ° C (Figura 1). Este procedimento produz a maior freqüência de mutação / ciclo de mutagênese (0,92 mutações / kb para d <1,000) e facilita muito a iteração. Mudanças nas condições de cultura para meios sólidos também afeta o espectro de mutação (Tabela 2). Mutagênese plaqueamento direto reduz a marcada assimetria entre substituições de bases complementares par visto na cultura líquida (compare C → T vs G → A e A → G vs T → C). Por outro lado, plaqueamento direto produzido indels mais (de menos de 0,5% a 4%) e diminuiu o número de mutações A → G em 3 vezes, levando a uma super-representação de G / C mutações (74%). No geral, acreditamos que a maior simplicidade e maior eficiência do protocolo de plaqueamento direto aqui apresentados superam os benefícios do espectro de mutação um pouco mais equilibrada produzidos por mutagênese líquido.
Dentro da meta plasmídeo, as mutações geradas pelo nosso método não se restringem à seqüência alvo desejado. No entanto, a evolução do romance de atividades bioquímicas deve ser dependente de mutações no gene alvo, uma vez que representa uma mais qualitativa do que uma mudança quantitativa. Assim, combinando a nossa mutagênese plasmídeo com a triagem de mutantes em placas gradiente capitaliza os pontos fortes do nosso sistema (grandes bibliotecas e disponibilidade de seleção) para a evolução de novas propriedades bioquímicas. Outras aplicações de mutagênese aleatória como a otimização existentes atividades enzimáticas ou randomizing áreas específicas de um gene exigem clonagem seguintes mutagênese aleatória. Neste caso, tendo a biblioteca em um plasmídeo, por oposição a um produto de amplificação PCR melhora a eficiência da clonagem, facilitando a amplificação e restrição.
Em suma, nós demonstramos um protocolo simples para criar uma biblioteca aleatória mutante de um gene-alvo, dado que se destaca por sua simplicidade e pela diversidade das bibliotecas geradas. Nós mostramos como esse método pode ser acoplado com seleções funcionais para a evolução eficiente de novas atividades bioquímicas. Além disso, nossa in vivo gerado bibliotecas podem ser facilmente clonados, permitindo site-specific mutagênese ou otimização das atividades existentes.
Divulgações
Agradecimentos
Este trabalho tem sido apoiado por K08 prêmio CA116429-04 para MC e por uma concessão do Cancer Conquer Now (preocupação) # fundação 8501
Materiais
| Name | Company | Catalog Number | Comments |
| carbenicillin | Cellgro | 46100R6 | 0.1mg/ml final |
| tetracycline | Fisher Scientific | BP7640 | 0.05mg/ml final |
| chloramphenicol | Genlantis | M120100 | 0.03mg/ml final |
| LB Agar Miller | Fisher Scientific | BP1425 | |
| LB Broth Miller | Difco Laboratories | 244620 | |
| Soft Agar | Difco Laboratories | 214580 | Made in house |
| Acridine Orange | Sigma-Aldrich | A38401-1 | |
| Antifoam B emulsion | Sigma-Aldrich | A5757 | |
| Glycerol | Acros Organics | 332030025 | |
| 100x100x15mm sq dish | Fisher Scientific | 0875711A | |
| 100mm rnd dish | Fisher Scientific | 0875712A | |
| Microscope slide 25x75x1mm | Gold Seal | 3048 | |
| Electroporator 2510 | Eppendorf | ||
| Electroporator 2510 | Eppendorf | ||
| 2mm gap tubes | Molecular BioProducts | 5520 | |
| Zippy mini prep kit | Zymo Research Corp. | D4020 |
Referências
- Camps, M. Modulation of ColE1-like plasmid replication for recombinant gene expression. Recent Pat DNA Gene Seq. 4, 58-73 (2010).
- Itoh, T., Tomizawa, J. Initiation of replication of plasmid ColE1 DNA by RNA polymerase, ribonuclease H, and DNA polymerase I. Cold Spring Harb Symp Quant Biol. 43 Pt 1, 409-417 (1979).
- Polisky, B. ColE1 replication control circuitry: sense from antisense. Cell. 55, 929-932 (1988).
- Camps, M., Naukkarinen, J., Johnson, B. P., Loeb, L. A. Targeted gene evolution in Escherichia coli using a highly error-prone DNA polymerase I. Proc Natl Acad Sci U S A. 100, 9727-9732 (2003).
- Shinkai, A., Loeb, L. A. In vivo mutagenesis by Escherichia coli DNA polymerase I. Ile(709) in motif A functions in base selection. J Biol Chem. 276, 46759-46764 (2001).
- Uyemura, D., Lehman, I. R. Biochemical characterization of mutant forms of DNA polymerase I from Escherichia coli. I. The polA12 mutation. J Biol Chem. 251, 4078-4084 (1976).
- Sedgwick, B., Robins, P., Lindahl, T. Direct removal of alkylation damage from DNA by AlkB and related DNA dioxygenases. Methods Enzymol. 408, 108-120 (2006).
- Aharoni, A., Gaidukov, L., Khersonsky, O., Mc, Q. G. S., Roodveldt, C., Tawfik, D. S. The 'evolvability' of promiscuous protein functions. Nat Genet. 37, 73-76 (2005).
- Elena, S. F., Lenski, R. E. Evolution experiments with microorganisms: the dynamics and genetic bases of adaptation. Nat Rev Genet. 4, 457-469 (2003).
- Itoh, T., Tomizawa, J. FoFormation of an RNA primer for initiation of replication of ColE1 DNA by ribonuclease H. Proc Natl Acad Sci U S A. 77, 2450-2454 (1980).
- Bryan, S. K., Moses, R. E. Sufficiency of the Klenow fragment for survival of polC(Ts) pcbA1 Escherichia coli at 43 degrees. C. J Bacteriol. 170, 456-458 (1988).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados