Method Article
Protocoles de sélection mutagenèse et fonctionnelles pour l'évolution dirigée de protéines dans les E. coli
Dans cet article
Résumé
Ici nous démontrons un protocole simple de créer une bibliothèque aléatoire mutant pour une séquence cible donnée. Nous montrons comment cette méthode, qui est réalisée in vivo chez Escherichia coli, peuvent être couplées avec des sélections fonctionnelles d'évoluer de nouvelles activités enzymatiques.
Résumé
La génération efficace de la diversité génétique représente un outil précieux moléculaire qui peut être utilisé pour étiqueter synthèse de l'ADN, afin de créer des signatures moléculaires uniques, ou de faire évoluer les protéines dans le laboratoire. Ici, nous présentons un protocole qui permet la génération de grande taille (> 10 11) banques de mutants pour une séquence cible donnée. Cette méthode est basée sur la réplication d'un plasmide d'encodage ColE1 la séquence désirée par une variante basse-fidélité de l'ADN polymérase I (Pol LF-I). Le plasmide cible est transformé en une souche de E. mutator coli et étalées sur un milieu solide, ce qui donne entre 0,2 et 1 mutations / ko, en fonction de la localisation du gène cible. Fréquences de mutation plus élevés sont obtenus par itération de ce processus de mutagenèse. Comparé à d'autres méthodes de mutagenèse, notre protocole se distingue par sa simplicité, comme le clonage ou la PCR ne sont impliqués. Ainsi, notre méthode est idéale pour l'étiquetage mutationnel de plasmides ou autres modèles, je Pol ou à explorer une grande partie de l'espace de séquence pour l'évolution des activités non présentes dans la cible originale. Le contrôle serré spatiale que la PCR ou randomisé à base d'oligonucléotides offrent des méthodes peuvent aussi être atteints par clonage ultérieur des sections spécifiques de la bibliothèque. Ici nous fournir des protocoles montrant comment créer une bibliothèque aléatoire de mutants et comment établir des médicaments à base des sélections dans E. coli afin d'identifier des mutants présentant de nouvelles activités biochimiques.
Protocole
Génération d'une bibliothèque I. aléatoire Mutant
Pol I médie l'initiation de la réplication plasmidique ColE1 (examiné dans (1-3). Notre méthode de mutagenèse est basé sur le placement d'une séquence cible dans un plasmide ColE1 adjacentes à l'origine ou la réplication et la propager dans les cellules exprimant l'ADN polymérase basse-fidélité, je (LF-Pol I). LF-Pol I est une ADN polymérase I mutant codant trois mutations qui diminuent la fidélité de la réplication, à savoir I709N (dans le motif A), A759R (dans le motif B) et D424A (inactivation relecture) 4,5 . LF-Pol I est exprimé dans une souche de E. coli, JS200, qui a un allèle sensibles à la température de Pol I (polA12) (6) de telle sorte que la LF-Pol I devient l'activité prédominante à 37 ° C. La réplication de l' séquence cible dans polA12 cellules sous des conditions restrictives des résultats dans la génération aléatoire d'une bibliothèque de mutants. mutagenèse est plus efficace dans les cultures saturées 4. Pour des raisons encore obscures, la mutagenèse n'est pas continu, c'est à dire la fréquence des mutations n'augmente pas linéairement avec le nombre des générations fois la culture atteint la saturation, même si les cellules sont autorisés à développer davantage dans des milieux frais. Par conséquent, à accroître encore la charge de mutation de la bibliothèque nécessite séries itératives de la mutagenèse et la récupération de plasmide. Ici nous fournir des protocoles pour LF-Pol mutagenèse j'ai. Notez que les protocoles présentés ici ont été considérablement simplifiées par rapport à nos 4 description originale afin de faciliter l'itération du processus pour parvenir à la charge mutation désirée (figure 1).
Matériaux
- Cellules: JS200 recA718 polA12 (ts) uvrA155 trpE65 lon-11 sula
- JS200 WT-Pol I: JS200 cellules exprimant le type sauvage (WT) Pol I
- JS200 LF-Pol I: JS200 exprimer basse fidélité (LF) Pol I
- Souche Lecture: JS200 WT-Pol I ou (pour la complémentation) une souche manque une activité spécifique
- Plasmide cible
- Plasmide contenant une origine de réplication ColE1 avec le gène cible clonée dans
1. Avant de mutagenèse: Préparation des électro-compétentes JS200 cellules I LF-Pol
- Choisissez un seul JS200 LF-Pol colonie J'ai partir d'une plaque LB cultivées à 30 ° C (conditions permissives) pendant la nuit contenant une sélection appropriée des antibiotiques pour le plasmide portant LF-Pol I (0,03 mg / ml de chloramphénicol) et le lieu de la colonie dans un test tube contenant 8 ml de LB avec le chloramphénicol. Cultiver la culture à 30 ° C et en agitant à 250rpm pendant la nuit.
- Dans la matinée, développez la culture en versant le ml 8 de JS200 LF-Pol-je dans un flacon contenant 400 ml LB avec du chloramphénicol. Laissez pousser la culture à 30 ° C tout en agitant à 250rpm jusqu'à ce qu'il atteigne une 600 A de 0.4 à 0,7 (typiquement 3-4h).
- Une fois à une 600 A de 0,4 à 0,7, chill la culture sur de la glace pendant 15 minutes.
- Transfert de la culture réfrigéré à un récipient approprié pour la centrifugation. Cellules par centrifugation à 4 ° C. Décanter le surnageant, puis ajoutez 10 ml de solution stérile et réfrigéré (sur la glace mouillée) de glycérol 10% sur le conteneur et remettre en suspension les cellules à l'aide d'une pipette sérologique.
- Transférer les cellules remises en suspension dans un tube conique de 50 ml. Remplir le tube conique jusqu'à la marque 45 ml avec du glycérol 10%, puis centrifuger pendant 15 minutes à 4 ° C et 4000rpm.
- Décanter le surnageant, ajouter 10 ml une fois de plus de 10% de glycérol dans le tube conique, et remettre en suspension les cellules à l'aide d'une pipette sérologique ou normale. Encore une fois, remplir le tube conique à la marque 45 ml avec du glycérol 10% et centrifuger pendant 15 minutes à 4 ° C et 4000rpm. Répétez ce processus deux fois pour éliminer toutes traces de sels.
- Après l'essorage final, re-suspendre le culot de cellules à parts égales de glycérol 10% (soit de remettre en suspension de 2 mL de cellules avec 2 ml de glycérol 10%).
- Aliquoter entre 100 et 500 uL uL des cellules en suspension dans des tubes de stockage de plusieurs. Quick-geler les cellules sur la glace sèche, puis les stocker à -80 ° C.
- Avant d'utiliser les cellules électro-compétentes pour les transformations, décongeler les cellules lentement sur la glace.
2. Mutagenèse: Transformer le plasmide cible
- Pipeter 40 uL de la JS200 cellules électro-compétentes j'ai LF-Pol et entre 30-250ng de l'ADN cible plasmide dans une cuvette d'électroporation 2mm écart.
Note # 1: Un plasmide contenant ColE1 GFP peut être réalisée par mutagenèse en parallèle avec le gène cible de contrôle. Après achèvement de l'étape de lecture, la GFP peut être étalé sur des plaques d'agar LB et visualisées pour la mutagénèse. Les colonies qui apparaissent sombres ou mal éclairés contiennent des mutations inactivant. - Pulse le mélange dans le électroporateur à 1800V. Vérifiez la constante de temps (T C) pour assurer des conditions uniformes d'électroporation pour chaque échantillon, idéalement T C = 5-6 sec.
- Récupérer le mélange de cellules / ADN dans 1 ml de bouillon LB pendant 40 minutes à 37 ° C (resconditions trictive) agitation à 250 rpm.
- Plaque 50 ul de la culture de récupération sur un plat d'agar LB 100mm Petri pré-chauffé à 37 ° C contenant à la fois le chloramphénicol et une concentration appropriée d'un antibiotique pour la sélection de plasmide cible.
Note: # 2: Viser à la plaque les cellules à une "pelouse près" la concentration. Un «pelouse près" la concentration est définie comme un nombre incalculable distinctes mais des colonies (> 1000 colonies / plat 100mm). La dilution plaqué, le cas échéant, dépendra de la façon électro-compétentes, les cellules sont. Si les cellules ne sont pas très électro-compétentes et une «pelouse près» n'est pas réalisable en étalant la culture de récupération "soignée", puis centrifuger à la culture de récupération de 2 minutes à 4000rpm, éliminer le surnageant, remettre en suspension les cellules dans 50 ul de bouillon LB et la plaque de la culture. - Incuber les dishe Petri (s) nuit à 37 ° C.
3. Mutagenèse: récupération plasmidique
- Le lendemain, lavez les boîtes de Petri par pipetage 2 ml de bouillon LB sur les cellules. Transférer les colonies bactériennes à partir des plats de Pétri LB agar au bouillon LB par "frotter" les hors la plaque avec une tige de verre en forme de triangle stérilisé. Ajouter 1 ml de bouillon LB abord, recueillir les laver et répéter la procédure avec le second ml de bouillon LB.
- Isoler l'ADN plasmidique de la laver la plaque. (Ce qui constitue l'ADN plasmidique de la bibliothèque).
Note 3: Le lavage recueillies auprès de la plaque LB peut être trop dense pour un mini-prep dans son intégralité. Si c'est le cas, mini-préparation du montant maximum alloué par votre kit de mini-préparation (généralement, il s'agissait de diluer votre lavage à OD = 1 et apprêter ~ 3 mL de la culture diluée) ou l'échelle, jusqu'à un maxi-prep
4. Itération
- Restreindre 1 pg de l'ADN plasmidique isolé avec une enzyme de restriction qui linéarise le LF-Pol I plasmide, mais ne coupez pas votre plasmide cible (supplémentaire Fig 1).
- Nettoyer la digestion de restriction en utilisant un kit de purification d'ADN.
Note n ° 4: Cette étape élimine toutes les traces de l'enzyme de restriction et de sa mémoire tampon. Cette étape est essentielle pour maintenir la concentration en sel faible pour subséquentes électro-compétentes transformations. - Re-transformer 30-250ng de l'arrière-bibliothèque cible plasmidique restreint dans de nouveaux JS200 LF-Pol I cellules de mettre la bibliothèque à travers des séries ultérieures de mutagénèse.
Note n ° 5: la linéarisation du plasmide Pol I en utilisant une enzyme de restriction ne garantit la plasmide cible se transforme. - Répétez les sections 2 et 3.
5. Lecture
- Restreindre l'ADN plasmidique isolé avec une enzyme de restriction (s) qui linéarise la fois la cible plasmide et le Pol I plasmide. Exécutez le condensé sur un gel d'agarose 1% afin d'assurer la quantité et la qualité des plasmides. Limiter ~ 400ng de l'ADN plasmidique isolé est généralement suffisant pour l'analyse.
- Restreindre 1 pg de l'ADN plasmidique isolé avec une enzyme de restriction qui linéarise le LF-Pol I plasmide, mais ne coupez pas votre plasmide cible.
- Nettoyer la digestion de restriction en utilisant un kit de purification d'ADN.
- Transformez la bibliothèque cible restreinte plasmide dans une souche lecture pour caractériser les mutations.
II. Ecran Mutant et analyse en utilisant des plaques de croissance Trait dégradé.
Afin d'illustrer la façon dont la grande diversité génétique présente dans nos bibliothèques peuvent être couplés à une sélection fonctionnelle, ici nous fournir un protocole pour les médicaments à base sélections fonctionnelle in vivo dans E. coli. Cette méthode est basée sur la croissance le long d'un gradient de drogue sur gélose solide. Cela permet la caractérisation simultanée de plusieurs échantillons (jusqu'à 12) sur une plage de concentrations, fournissant une gamme dynamique plus large qu'un traitement médicamenteux seul. Un autre avantage est que la lecture non linéaire de ce dosage tampons de légères différences dans la viabilité, dans une fourchette de 2 fois. Ainsi, ce test de cytotoxicité de résistance fournit un moyen robuste et rapide pour sélectionner les bibliothèques de mutants et de déterminer le profil phénotypique des mutants individuels. Fig. 2 montre un exemple de chacune de ces utilisations: panneau montre une sélection de mutants individuels à partir d'un oxydant déméthylase humaine ABH2 bibliothèque. Colonies de croissance au-dessus du seuil de WT sont sélectionnés pour une protection accrue contre la cytotoxicité causée par le méthanesulfonate de méthyle agent de méthylation (MMS) 7. Groupe B montre un exemple de gradients utilisés pour la caractérisation des clones individuels. Le niveau de résistance à la troisième génération céfotaxime antibiotique céphalosporine est montré sur une pente de gélose pour WT β-lactamase et pour deux à spectre étendu mutants, R164H et E104K R164S G267R 4. Notez que selon la force des effets observés, plus que d'une seule plaque peut être nécessaire pour la quantification adéquate: tandis que le gradient 0.4mg/ml permet une comparaison directe avec les clones de contrôle, le niveau de résistance du mutant plus puissants ne peuvent être établie en utilisant une concentration plus élevéetration de céfotaxime (4mg/mL).
Matériaux
- Équipement
- 100x100x15mm carrés boîtes de Pétri (Fisher Sci # 0875711A)
- 100mm plat rond de Petri
- Glisser 25x75x1mm microscope en verre (Fischer # 1255015 Sci)
- Tube de 50 ml graduée
- Médias
- LB agar: fondue et équilibrée dans un bain d'eau à 56 ° C
Note n ° 6: la température des médias peuvent affecter la stabilité et donc l'activité de la drogue ou d'un composé à l'étude. - Agar mou: fondue et équilibrée dans un bain d'eau à 42 ° C
- LB agar: fondue et équilibrée dans un bain d'eau à 56 ° C
1. Construction de la pente
- Mark dix voies, espacés uniformément sur un bord du fond de l'assiette carrée de Pétri.
- Placez le plat sur une telle que le bord inférieur marqué est surélevée 7mm incliné;
une herbe épaisse ou un autre objet plat peut être utilisé comme support pour élever le plat. Verser 25 ml d'eau tiède (~ 56 ° C) LB agar bien mélangé avec une concentration appropriée de l'agent de sélection dans le plat incliné. Ceci est la couche inférieure de la pente. Assurez-vous que l'agar LB enrobe uniformément la boîte de Pétri de telle sorte que l'élévation, à la fin marquée du plat contient ~ 1mm d'agar LB et la partie abaissée contient ~ 8mm d'agar LB. Puis permettra de mettre en agar pendant 10-15 minutes.
Note n ° 7: Pour les agents hydrophobes sélection, tensioactif de 0,1% (antimousse B émulsion) peut être ajouté à la gélose LB pour faciliter la suspension et la distribution uniforme de la drogue. Ajouter tensioactif à l'agar LB stérile chaude avec agitation vigoureuse avant l'ajout de l'agent de sélection. L'agent tensioactif devrait suspendre comme une brume fine de petites gouttelettes, de grosses gouttelettes indiquent les médias est trop chaude et peut inhiber la distribution uniforme du médicament testé. - Après les premiers 25 ml de gélose LB a durci, le plat est déplacé vers une surface plane. Ensuite, 25 ml d'agar LB sans l'agent de sélection est versé à la superposition de la surface LB agar premier. Ceci est la couche supérieure de la pente. Assurez-vous que l'agar LB couvre toute la surface de la couche inférieure. Couvrez avec le couvercle de travers pour la ventilation, et permettent de mettre en agar pendant 10-15 minutes.
Note n ° 8: Soyez conscient des dangers potentiels d'aérosol et la volatilisation du composé à tester; versez dégradés dans une hotte de sécurité chimique ou biologique s'il est prescrit par les exigences de la sécurité chimique.
Note n ° 9: plats dégradé doit être utilisé dans 4h pour préserver le gradient de la drogue ou la concentration du composé test.
2. Cachet transfert de bactéries
- Agar mou doit être équilibrée à 42 ° C. Transfert 2 mL de gélose molle liquide dans le couvercle ou le fond d'une boîte de Pétri 100mm ronde. Pipeter 40 ul de la culture bactérienne dans l'agar mou et mélanger en faisant basculer la plaque.
Note n ° 10: Le stade de croissance d'une culture bactérienne peut influer sur sa réaction à un médicament ou un composé d'essai. Cultures en phase journal ou cultures de la nuit dans la phase stationnaire doit être utilisé avec cohérence pour des résultats uniformes. La densité cellulaire peut aussi fausser les résultats relatifs, donc toutes les cultures doivent être dilués à avons identifié A 600 valeurs de densité. Cultures de la nuit doit être dilué pour avoir un A 600 densité inférieure à 1,0 - Enduire le bord long de la lame de microscope en verre avec le mélange agar mou. Puis, en alignant le bord revêtement de la lame avec la marque en bas (de faible à forte concentration) sur le plat de gradient, toucher la lame à la surface de l'agar-agar. Un toucher doux est suffisant pour transférer un ruban de la gélose molle à la surface du gradient. La lame est ensuite mis de côté pour le nettoyage et la réutilisation.
- Répétez ce processus, pour le reste des échantillons bactériens. Les échantillons de référence doit être inclus sur chaque plat, si gradient gradients multiples sont en cours d'exécution.
- Incuber le sommet plat de gradient du jour au lendemain à 37 ° C. Temps et températures d'incubation peut varier pour une lecture différente des souches bactériennes, mais nuit à 37 ° C est généralement suffisante pour la croissance visible.
3. Imagerie et analyse de la croissance
- Imagerie: Après une croissance durant la nuit, les gradients peuvent être directement imagés ou fixées et colorées avec une solution de 0,2 mg / ml orange d'acridine dans EtOH 95%, pour améliorer le contraste. La plaque est incubée à température ambiante pendant 5 min, dans la solution de coloration, puis lavés avec de l'EtOH 95% et ensuite imagé sur une boîte à lumière UV.
Note n ° 11: Prendre soin de ne pas rincer les colonies de la plaque de pierre, mais plutôt la solution de coloration et de lave sur la plaque et retirez les solutions de tous les coins. - Pour l'analyse du phénotype de l'individu plasmides mutants, la distance de croissance contre le gradient de concentration est normalisée à une norme sur chaque gradient. Ces valeurs relatives peuvent ensuite être comparés entre les gradients.
Note n ° 12: Selon la nature de l'effet cytotoxique, une arête vive ou d'un bord plus diffuses peuvent être observées (comparer par exemple des panneaux A et B dans la figure 2). Dans le cas des bords diffus, il est conseillé de mesurer la pointe of croissance continue, comme les colonies individuelles ont tendance à montrer une variabilité accrue. - Pour la sélection des bibliothèques, des colonies individuelles qui poussent à des concentrations supérieures à la commande de type sauvage parentale sont isolés et séquencés pour identifier les mutations qui contribuent à une protection accrue.
III. Les résultats représentatifs:
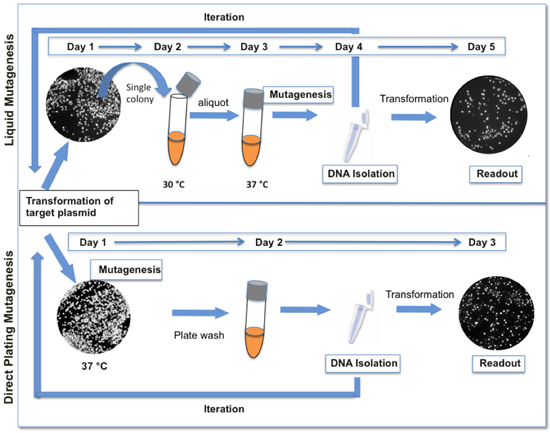
Figure 1. Comparaison entre les liquides et les protocoles de mutagenèse directe placage. Le protocole ensemencement direct mutagenèse présentés ici (en bas) est plus rapide et nécessite moins d'étapes que notre protocole de mutagenèse originale liquide (en haut). Lorsque la GFP est utilisée en tant que journaliste, les variations de fluorescence sont indicatifs de la diversité génétique présente dans la bibliothèque. Typiquement, un cycle de mutagenèse des résultats dans des colonies de 12-18% avec des niveaux sensiblement diminué de fluorescence.
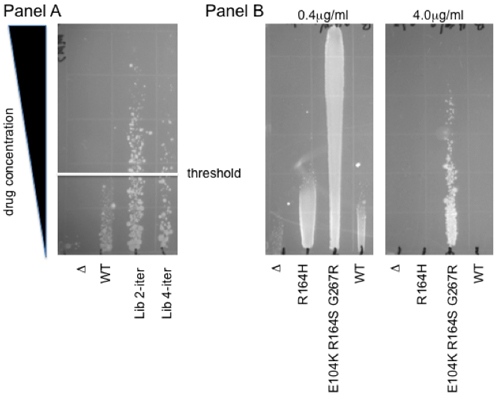
Figure 2. Tests de résistance dégradé. Groupe A de sélection pour une résistance accrue à Methyl-méthanesulfonate (MMS). Bibliothèques de plasmides de l'humain déméthylase oxydatif ABH2 ont été sélectionnées pour une résistance accrue aux MMS. Deux de ces bibliothèques représentent deux et quatre itérations du protocole de mutagenèse sont présentés en comparaison avec le type parental sauvage (WT) et le vecteur vide (Δ). La ligne blanche représente le seuil à partir duquel chaque colonies mutantes ont été isolés pour l'analyse phénotypique plus loin. Panel B céfotaxime protection, indicatif de spectre étendu β-lactamase activité. R164H et E104K R164H G267R, deux mutants de beta-lactamase précédemment identifiés suivants LF- Pol I mutagenèse couplée à la sélection aztréonam 4, sont présentées sur une 0.4μg/mL et un gradient 4μg/mL céfotaxime. Notez que le type sauvage β-lactamase enzyme confère pas de protection par rapport aux cellules exprimant un vecteur vide, Δ. Par conséquent, ces mutants représentent l'évolution d'une nouvelle activité biochimique 8,9.
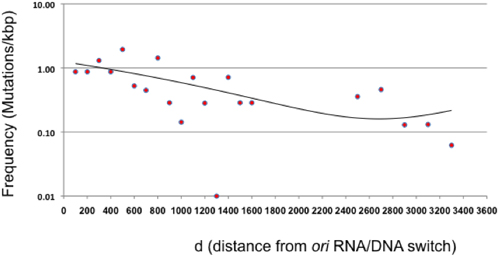
Figure 3. Fréquence de mutation en fonction de la distance par Ori (d). La fréquence de mutation après un seul cycle de mutagenèse ensemencement direct est indiqué pour des intervalles de 100 pb par rapport à l'interrupteur ARN / ADN de l'origine de réplication ColE1. La région entre 1600 et 2400 n'est pas représenté parce β-lactamase ne représente pas une cible neutre. Les points au-delà β-lactamase représentent des intervalles de 200 pb étant donné la fréquence de la mutation globale faible dans ce domaine. La tendance (équation binôme, avec un R 2 = 0,41) est représentée par une ligne.

Figure 1 supplémentaire. Pol I-LF contenant Pol I plasmide. Une séquence (format fasta). Information sur la séquence pour déterminer l'enzyme de restriction appropriée (s) à utiliser lors de la linéarisation du plasmide Pol I soit pour l'itération (génération d'une étape bibliothèque mutation aléatoire 4) ou de lecture (étape 5). B Caractéristiques générales et carte de restriction du plasmide. Le emplacement de l'origine pSC101 de réplication, le marqueur de résistance au chloramphénicol (CAT) et LF-Pol gène I sont présentés. La localisation des sites de restriction unique est indiqué aussi bien.
| Bibliothèque | Ensemencement direct | Liquid (1 jour) | Liquid (3 jours) | |
| Mutations (#) | 95 | 40 | 142 | |
| Clones séquencés | 288 | 96 | 190 | |
| Couverture totale (pb) | 182000 | 102000 | 213000 | |
| Mutation freq (x10 3 pb) | 0,52 | 0,39 | 0,67 | |
| Freq d <1000 (x10 3 pb) | 0,92 | 0,41 | 0,70 |
Tableau 1. Fréquences de mutation Fréquences (exprimée en nombre de mutations / pb) pour d <1000, c'est à dire au sein du BP 1000 à côté de l'interrupteur ARN / ADN, pour trois protocoles mutagenèse:. Ensemencement direct, la saturation de liquide (1 jour), et hypersaturation liquide ( 3 jours).
| Ensemencement direct | Liquid | ||
| Mutations (#) | 95 | 182 | |
| Spectre (%) | |||
| A à G | A à G | 6.3 | 19,2 |
| T à C | 4.2 | 3.8 | |
| C à T | G à A | 27,4 | 13,7 |
| C à T | 35,8 | 35,2 | |
| A à T | A à T | 5.3 | 8.2 |
| T à A | 5.3 | 7.1 | |
| T à G | T à G | 1.1 | 1.1 |
| A à C | 0.0 | 2.2 | |
| G à T | G à T | 1.1 | 2.7 |
| C à A | 2.1 | 2.7 | |
| C à G | C à G | 2.1 | 1.1 |
| G à C | 5.3 | 2.7 | |
| Indels | Ins | 3.2 | 0.0 |
| Del | 1.1 | 0.0 | |
| A à N | 11,6 | 29,7 | |
| G à N | 33,7 | 19,2 | |
| T à N | 10,5 | 12,1 | |
| C à N | 40,0 | 39,0 | |
| Ts | 73,7 | 72,0 | |
| Tv | 22,1 | 28,0 | |
| Indels | 4.2 | 0 |
Tableau 2. Métriques de mutation pour l'ensemencement direct et la mutagénèse liquide. Le tableau présente le nombre de mutations observées (en nombre) et le spectre de mutation (en%) après un seul cycle de mutagenèse. Le spectre est ventilé par paires complémentaires, par des changements de nucléotides, et par le type de mutation.
Discussion
Cet article présente un protocole de mutagenèse qui permet la génération aléatoire des grandes banques de mutants sans la nécessité pour le clonage ou la PCR. Cette méthode est basée sur erreurs de réplication d'un plasmide codant pour une séquence d'intérêt. En théorie, les mutations devraient être largement limités aux 100-300 pb située immédiatement en aval de l'interrupteur de l'ARN / ADN, la taille de leader brin intermédiaire produite par Pol I 2,10. Nous avons constaté que dans les conditions présentées dans le présent protocole, Pol mutations se produisent partout dans I le plasmide, bien que diminuant la fréquence comme la distance entre les augmentations Ori plasmide (figure 3). Cette constatation implique que la transition ou "switch" de Pol I Pol III pendant ColE1 réplication plasmidique est beaucoup plus progressif que celui rapporté antérieurement, au moins dans nos conditions expérimentales 2, et est d'accord avec les études antérieures suggérant une redondance fonctionnelle entre Pol I et Pol III 11.
Notre protocole original décrit mutagenèse dans des cultures liquides (4). Ce protocole donne 0,41 mutations / Ko pour d <1000, c'est à dire au sein du BP 1000 adjacent à l'ADN / ARN interrupteur (tableau 1). Hypersaturation en laissant le liquide de culture dans le shaker pendant 3 jours sans l'ajout de toute nouvelle presse soulève la fréquence de mutation à 0,70 mutations / ko mais les résultats de rendement plasmidique très pauvres (moins de 1% par rapport au jour 1; données non présentées) ( Tableau 1). Nous présentons ici un protocole simplifié basé sur la transformation directe plaquage de la cible plasmide sur gélose solide à 37 ° C (figure 1). Cette procédure produit la plus grande fréquence de mutation / cycle de mutagenèse (0,92 mutations / Ko pour d <1000) et facilite grandement l'itération. L'évolution des conditions de culture d'un milieu solide affecte également le spectre de mutation (tableau 2). Mutagenèse ensemencement direct réduit le asymétrie marquée entre les substitutions de paires de bases complémentaires vu en culture liquide (comparer C → T vs G → A et A → G → T vs C). D'autre part, l'ensemencement direct a produit plus indels (de moins de 0,5% à 4%) et diminué le nombre de mutations G → A par 3 fois, conduisant à une surreprésentation de G / C mutations (74%). Globalement, nous pensons que la plus grande simplicité et une efficacité accrue du protocole ensemencement direct présentés ici l'emporte sur les avantages de la gamme un peu plus équilibrée mutation produite par mutagenèse liquide.
Dans le plasmide cible, les mutations générées par notre méthode ne sont pas limités à la séquence cible souhaitée. Cependant, l'évolution du roman activités biochimiques devrait dépendre de mutations dans le gène cible, car elle représente une approche qualitative plutôt que d'un changement quantitatif. Ainsi, en combinant nos mutagenèse plasmide avec le dépistage de mutants sur des plaques de gradient capitalise sur les atouts de notre système (les grandes bibliothèques et de la disponibilité de la sélection) pour l'évolution des nouvelles propriétés biochimiques. D'autres applications de mutagenèse aléatoire telles que l'optimisation des activités enzymatiques existants ou randomisant des domaines spécifiques d'un gène ne nécessitent clonage suivantes mutagenèse aléatoire. Dans ce cas, disposer de la bibliothèque dans un plasmide, par opposition à un produit d'amplification PCR améliore l'efficacité du clonage, en facilitant l'amplification et la restriction.
En somme, nous démontrons un protocole simple de créer une bibliothèque aléatoire mutantes pour un gène cible donné qui se distingue par sa simplicité et de la diversité des bibliothèques générés. Nous montrons comment cette méthode peut être couplée avec des sélections fonctionnelles pour l'évolution efficace de nouvelles activités biochimiques. En outre, notre in vivo généré les bibliothèques peuvent être facilement clonées, permettant la mutagenèse dirigée ou optimisation des activités existantes.
Déclarations de divulgation
Remerciements
Ce travail a été soutenu par K08 Prix CA116429-04 de MC et par une subvention du cancer Conquer maintenant (CONCERN) Fondation # 8501
matériels
| Name | Company | Catalog Number | Comments |
| carbenicillin | Cellgro | 46100R6 | 0.1mg/ml final |
| tetracycline | Fisher Scientific | BP7640 | 0.05mg/ml final |
| chloramphenicol | Genlantis | M120100 | 0.03mg/ml final |
| LB Agar Miller | Fisher Scientific | BP1425 | |
| LB Broth Miller | Difco Laboratories | 244620 | |
| Soft Agar | Difco Laboratories | 214580 | Made in house |
| Acridine Orange | Sigma-Aldrich | A38401-1 | |
| Antifoam B emulsion | Sigma-Aldrich | A5757 | |
| Glycerol | Acros Organics | 332030025 | |
| 100x100x15mm sq dish | Fisher Scientific | 0875711A | |
| 100mm rnd dish | Fisher Scientific | 0875712A | |
| Microscope slide 25x75x1mm | Gold Seal | 3048 | |
| Electroporator 2510 | Eppendorf | ||
| Electroporator 2510 | Eppendorf | ||
| 2mm gap tubes | Molecular BioProducts | 5520 | |
| Zippy mini prep kit | Zymo Research Corp. | D4020 |
Références
- Camps, M. Modulation of ColE1-like plasmid replication for recombinant gene expression. Recent Pat DNA Gene Seq. 4, 58-73 (2010).
- Itoh, T., Tomizawa, J. Initiation of replication of plasmid ColE1 DNA by RNA polymerase, ribonuclease H, and DNA polymerase I. Cold Spring Harb Symp Quant Biol. 43 Pt 1, 409-417 (1979).
- Polisky, B. ColE1 replication control circuitry: sense from antisense. Cell. 55, 929-932 (1988).
- Camps, M., Naukkarinen, J., Johnson, B. P., Loeb, L. A. Targeted gene evolution in Escherichia coli using a highly error-prone DNA polymerase I. Proc Natl Acad Sci U S A. 100, 9727-9732 (2003).
- Shinkai, A., Loeb, L. A. In vivo mutagenesis by Escherichia coli DNA polymerase I. Ile(709) in motif A functions in base selection. J Biol Chem. 276, 46759-46764 (2001).
- Uyemura, D., Lehman, I. R. Biochemical characterization of mutant forms of DNA polymerase I from Escherichia coli. I. The polA12 mutation. J Biol Chem. 251, 4078-4084 (1976).
- Sedgwick, B., Robins, P., Lindahl, T. Direct removal of alkylation damage from DNA by AlkB and related DNA dioxygenases. Methods Enzymol. 408, 108-120 (2006).
- Aharoni, A., Gaidukov, L., Khersonsky, O., Mc, Q. G. S., Roodveldt, C., Tawfik, D. S. The 'evolvability' of promiscuous protein functions. Nat Genet. 37, 73-76 (2005).
- Elena, S. F., Lenski, R. E. Evolution experiments with microorganisms: the dynamics and genetic bases of adaptation. Nat Rev Genet. 4, 457-469 (2003).
- Itoh, T., Tomizawa, J. FoFormation of an RNA primer for initiation of replication of ColE1 DNA by ribonuclease H. Proc Natl Acad Sci U S A. 77, 2450-2454 (1980).
- Bryan, S. K., Moses, R. E. Sufficiency of the Klenow fragment for survival of polC(Ts) pcbA1 Escherichia coli at 43 degrees. C. J Bacteriol. 170, 456-458 (1988).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon