Method Article
Mutagenese und Functional Selection Protocols for Directed Evolution von Proteinen in E. coli
In diesem Artikel
Zusammenfassung
Hier zeigen wir ein einfaches Protokoll, um eine zufällige Mutantenbibliothek für eine bestimmte Zielsequenz zu schaffen. Wir zeigen, wie diese Methode, die in vivo in Escherichia coli durchgeführt wird, mit funktionellen Selektionen gekoppelt werden kann, um neue enzymatische Aktivitäten entwickeln.
Zusammenfassung
Die effiziente Erzeugung der genetischen Vielfalt stellt einen unschätzbaren molekulare Werkzeug, um die DNA-Synthese-Label, um einzigartige molekulare Signaturen zu erstellen oder Proteine im Labor entwickelt werden können. Hier präsentieren wir ein Protokoll, das die Erzeugung von großen (> 10 11) Mutantenbibliotheken für eine bestimmte Zielsequenz ermöglicht. Diese Methode basiert auf der Replikation eines ColE1 Plasmid-Kodierung der gewünschten Reihenfolge von einer low-fidelity-Variante der DNA-Polymerase I (LF-Pol I). Das Ziel Plasmid wird in eine Mutatorstamm von E. verwandelt coli und verchromt auf festen Medien, wodurch zwischen 0,2 und 1-Mutationen / kb, je nach Lage des Zielgens. Höhere Mutation Frequenzen werden durch Iteration dieses Prozesses der Mutagenese erreicht. Im Vergleich zu alternativen Methoden der Mutagenese, steht unser Protokoll durch seine Einfachheit, da keine Klonen oder PCR beteiligt sind. So ist unsere Methode ideal für Mutationsanalysen Kennzeichnung von Plasmiden oder anderen Pol I-Vorlagen oder um große Teile der Sequenz Raum für die Entwicklung der Tätigkeiten, die nicht in das ursprüngliche Ziel zu erkunden. Die enge räumliche Steuerung, die PCR oder randomisierte Oligonukleotid-basierte Methoden bieten auch durch nachfolgende Klonierung von bestimmten Abschnitten der Bibliothek erreicht werden. Hier bieten wir Protokolle zeigen, wie man einen zufälligen Mutanten-Bibliothek zu erstellen und wie Drogen-Selektionen in E. etablieren coli Mutanten ausstellenden neuen biochemischen Aktivitäten zu identifizieren.
Protokoll
I. Erzeugung einer Zufallszahl Mutant Bibliothek
Pol I vermittelt Einleitung ColE1 Plasmidreplikation (besprochen in (1-3). Unsere Methode der Mutagenese bei Einlegen einer Zielsequenz in einer ColE1 Plasmid neben der Herkunft oder Replikation und Vermehrung in Zellen, die Low-Fidelity DNA-Polymerase I basiert (LF-Pol I). LF-Pol I ist ein mutierter DNA-Polymerase I-Kodierung drei Mutationen, die die Treue der Replikation, nämlich I709N (in Motiv A), A759R (in Motiv B) und D424A (Inaktivierung Korrekturlesen) 4,5 Rückgang . LF-Pol I wird in einer E. coli-Stamm, JS200, dass ein Temperatur-sensitives Allel des Pol I (polA12) (6) hat zum Ausdruck gebracht, so dass LF-Pol I wird die vorherrschende Aktivität bei 37 ° C. Die Replikation der Zielsequenz in polA12 Zellen unter restriktiven Bedingungen führt zur Erzeugung eines zufälligen Mutanten-Bibliothek. Mutagenesis effizienter ist in gesättigten Kulturen 4. Aus Gründen, die noch unklar sind, Mutagenese nicht kontinuierlich, dh die Mutation Frequenz nicht linear mit der Anzahl Generationen einmal die Kultur erreicht Sättigung, auch wenn Zellen dürfen weiter ausbauen in frischen Medien. Daher eine weitere Erhöhung der Mutation Belastung der Bibliothek erfordert iterative Runden von Mutagenese und Plasmid-Rückgewinnung. Hier haben wir Protokolle für LF-Pol I Mutagenese liefern. Beachten Sie, dass die Protokolle hier vorgestellten erheblich worden im Vergleich zu unseren ursprünglichen Beschreibung 4 haben, um Iteration des Prozesses zur Erreichung der gewünschten Mutation Belastung (Abb. 1) zu erleichtern vereinfacht.
Materialien
- Cells: JS200 recA718 polA12 (ts) uvrA155 trpE65 lon-11 sula
- JS200 WT-Pol I: JS200 Zellen, die Wildtyp (WT) Pol I
- JS200 LF-Pol I: JS200 Ausdruck low fidelity (LF) Pol I
- Readout Stamm: JS200 WT-Pol I oder (für die Komplementierung) eine Belastung fehlt eine spezifische Aktivität
- Zielplasmid
- Plasmide mit einem ColE1 Replikation mit dem Ziel kloniert in
1. Vor Mutagenese: Vorbereitung von elektro-kompetenten JS200 LF-Pol I-Zellen
- Wählen Sie ein einzelnes JS200 LF-Pol I Kolonie von einer LB-Platte bei 30 ° C (permissive Bedingungen) über Nacht gezüchtet mit den entsprechenden Antibiotika-Selektion für das Plasmid tragende LF-Pol I (0.03mg / ml Chloramphenicol) und Ort der Kolonie in einem Test Röhrchen mit 8 ml LB mit Chloramphenicol. Die Kultur bei 30 ° C und Schütteln bei 250rpm über Nacht.
- Am Morgen, erweitern Sie die Kultur, indem die 8 ml JS200 LF-Pol I in einen Kolben mit 400 mL LB mit Chloramphenicol. Lassen Sie die Kultur bei 30 ° C wachsen unter Schütteln bei 250rpm, bis es eine A 600 von 0,4-0,7 (in der Regel 3-4h) erreicht.
- Sobald bei einer A 600 von 0,4-0,7, chill die Kultur auf nassem Eis für 15 Minuten.
- Übertragen Sie die gekühlte Kultur einen geeigneten Behälter für die Zentrifugation. Pellet-Zellen durch Zentrifugation bei 4 ° C. Abgießen des Überstandes, und fügen Sie 10 ml steril und gekühlt (auf nassem Eis) 10% Glycerin in den Behälter und erneut zu suspendieren die Zellen mit einer serologischen Pipette.
- Übertragen Sie die resuspendierten Zellen in eine 50 ml konischen Rohr. Füllen Sie den konischen Rohr bis zur 45 ml Marke mit 10% Glycerin und dann Zentrifuge für 15 Minuten bei 4 ° C und 4000rpm.
- Abgießen des Überstandes, fügen Sie noch einmal 10 ml 10% Glycerin zu dem konischen Rohr, und wieder aussetzen Zellen unter Verwendung eines serologische oder normale Pipette. Wieder füllen Sie das konische Röhre, die 45 ml Marke mit 10% Glycerin und Zentrifuge für 15 Minuten bei 4 ° C und 4000rpm. Wiederholen Sie diesen Vorgang noch zweimal, um alle Spuren von Salzen zu entfernen.
- Nach dem letzten Schleudern, resuspendieren des Pellets von Zellen in gleichen Teilen 10% Glycerin (dh re-suspend 2 mL der Zellen mit 2 ml 10% Glycerin).
- Aliquot zwischen 100 und 500 ul ul der suspendierten Zellen in mehrere Storage-Röhren. Quick-Freeze die Zellen auf Trockeneis und dann speichern Sie sie bei -80 ° C.
- Vor der Verwendung der Zellen für die elektro-kompetenten Transformationen, tauen die Zellen langsam auf Eis.
2. Mutagenese: Transforming the Zielplasmid
- Pipette 40 ul der elektro-kompetenten JS200 LF-Pol I-Zellen und zwischen 30-250ng des Ziels Plasmid-DNA in einem 2mm Lücke Elektroporationsküvette.
Hinweis # 1: A ColE1 Plasmid GFP kann durch Mutagenese in parallel mit dem Zielgen als Kontrolle durchgeführt werden. Nach Abschluss der Anzeige Schritt können GFP auf LB Agarplatten und visualisiert werden für die Mutagenese. Kolonien, die dunkel oder dunkel erscheinen enthalten inaktivierende Mutationen. - Pulse die Mischung in der Elektroporator bei 1800V. Überprüfen Sie die Zeitkonstante (T C), um eine einheitliche Elektroporation Bedingungen für jede Probe zu gewährleisten, idealerweise T C = 5-6 sek.
- Recover der Zelle / DNA-Mischung in 1 ml LB-Bouillon für 40 Minuten bei 37 ° C (restrictive Bedingungen) Schütteln bei 250 Umdrehungen pro Minute.
- Tafel 50 ul der Erholung Kultur auf einem 100mm LB-Agar-Petrischale bis 37 vorgewärmten ° C, die sowohl Chloramphenicol und eine entsprechende Konzentration eines Antibiotikums Auswahl für die Ziel-Plasmid.
Hinweis: # 2: Richten Sie die Platte die Zellen bei einer "near Rasen" Konzentration. A "in der Nähe Rasen" Konzentration wird als eine deutliche, aber unzählige Kolonien (> 1000 Kolonien / 100mm Schale) definiert. Die Verdünnung vernickelt, wenn überhaupt, wie elektro-kompetenten Zellen abhängen. Wenn die Zellen sind nicht sehr elektro-kompetenten und eine "near Rasen" ist nicht durch Ausplattieren der Erholung Kultur "ordentlich", dann Zentrifuge die Erholung Kultur für 2 Minuten bei 4000rpm, giesst die überstehende, resuspendieren die Zellen in 50 erreichbar ul LB-Medium und die Platte der Kultur. - Inkubieren Sie die Petri dishe (s) über Nacht bei 37 ° C.
3. Mutagenese: Plasmid Erholung
- Am nächsten Tag, waschen Sie die Petrischalen mit einer Pipette 2 ml LB-Medium über die Zellen. Übertragen Sie die Bakterienkolonien von der LB-Agar-Petrischalen auf die LB Brühe durch "schrubben" sie von der Platte mit einem sterilisierten dreieckige Glasstab. 1 mL LB-Medium zuerst, sammeln die Wasch-und wiederholen Sie den Vorgang mit dem zweiten ml LB-Bouillon.
- Isolieren Sie die Plasmid-DNA von der Platte zu waschen. (Diese Plasmid-DNA bildet die Bibliothek).
Hinweis # 3: Die Wäsche aus dem LB-Platte gesammelt werden können, zu dicht, um Mini-Prep in seiner Gesamtheit. Wenn dies der Fall ist, mini-prep die maximale zugeteilten Betrag von Ihrem Mini-Prep-Kit (in der Regel daran beteiligt Verdünnen Sie Ihre Wäsche auf OD = 1 und prepping ~ 3 mL der verdünnten Kultur) oder skalieren bis zu einer maxi-prep
4. Iteration
- Beschränken 1 g der isolierten Plasmid-DNA mit einem Restriktionsenzym, dass die LF-Pol I Plasmid linearisiert, aber nicht schneiden Sie Ihre Ziel-Plasmid (Supplementary Abb. 1).
- Clean up the Restriktionsverdau mit einem DNA Purification Kit.
Hinweis # 4: Dieser Schritt entfernt alle Spuren von dem Restriktionsenzym und deren Puffer. Dieser Schritt ist wichtig, damit die Salzkonzentration niedrig für nachfolgende elektro-kompetenten Transformationen. - Re-Transformation 30-250ng der eingeschränkten Ziel-Plasmid-Bibliothek wieder in frischen JS200 LF-Pol I-Zellen in die Bibliothek durch den folgenden Runden von Mutagenese setzen.
Hinweis # 5: Linearisierung der Pol I-Plasmid mit einem Restriktionsenzym sichergestellt, dass nur das Ziel-Plasmid transformiert wird. - Wiederholen Sie die Abschnitte 2 und 3.
5. Anzeige
- Beschränken Sie die isolierte Plasmid-DNA mit einem Restriktionsenzym (en), die beide das Ziel und die Plasmid-Pol I Plasmid linearisiert. Führen Sie das zu verdauen auf einem 1% Agarosegel auf Quantität und Qualität der Plasmide zu gewährleisten. Beschränken ~ 400ng der isolierten Plasmid-DNA ist in der Regel ausreichend für die Analyse.
- Beschränken 1 g der isolierten Plasmid-DNA mit einem Restriktionsenzym, dass die LF-Pol I Plasmid linearisiert, aber nicht schneiden Sie Ihre Ziel-Plasmid.
- Clean up the Restriktionsverdau mit einem DNA Purification Kit.
- Transform der eingeschränkten Zielplasmid Bibliothek in ein Auslesen anstrengen, um die Mutationen zu charakterisieren.
II. Mutant Screen und Trait Analysis Using Gradient Growth Plates.
Um zu veranschaulichen, wie die große genetische Vielfalt in unseren Bibliotheken, um eine funktionale Auswahl gekoppelt werden kann, hier bieten wir ein Protokoll für die medikamentöse funktionale Auswahl in vivo in E. coli. Diese Methode ist auf Wachstum entlang einer Droge Gradienten auf festen Agar-Basis. Dies ermöglicht die gleichzeitige Charakterisierung von mehreren (bis zu 12) Proben über einen Bereich von Konzentrationen, einen breiteren Dynamikbereich als eine einzige medikamentöse Behandlung. Ein weiterer Vorteil ist, dass die nicht-linearen Auslesen von diesem Assay moderate Unterschiede in Lebensfähigkeit, in einem 2-fachen Bereich Puffer. Somit stellt diese Zytotoxizität-Resistenzassay eine robuste und schnelle Möglichkeit, um Mutantenbibliotheken auszuwählen und die phänotypische Profil der einzelnen Mutanten zu bestimmen. Abb.. 2 zeigt ein Beispiel für jede dieser Verwendungen: Panel A zeigt die Auswahl der einzelnen Mutanten aus einem menschlichen oxidative Demethylase ABH2 Bibliothek. Kolonien, die über dem WT Schwelle für erhöhten Schutz vor Zytotoxizität durch die Methylierungsmittel methyl Methansulfonat (MMS) 7 verursacht ausgewählt. Panel B zeigt ein Beispiel von Gradienten für einzelne Klon Charakterisierung verwendet. Die Höhe der Widerstand gegen die dritte Generation Cephalosporin-Antibiotikum Cefotaxim ist auf einem Agar-Gradienten für WT β-Lactamase und für zwei extended-spectrum-Mutanten, R164H und E104K R164S G267R 4 dargestellt. Beachten Sie, dass abhängig von der Stärke der beobachteten Effekte, mehr als eine einzelne Platte kann für eine angemessene Quantifizierung erforderlich: Während die 0.4mg/ml Gradienten ermöglicht den direkten Vergleich zur Kontrollgruppe Klone, die Beständigkeit der stärksten Mutanten können nur gegründet mit einer höheren Konzentration werdentration von Cefotaxim (4mg/mL).
Materialien
- Ausstattung
- 100x100x15mm quadratischen Petrischalen (Fisher Sci # 0875711A)
- 100mm rund Petrischale
- 25x75x1mm Glasobjektträger (Fischer Sci # 1255015)
- 50 ml Tube Studium
- Medien
- LB-Agar: geschmolzen und äquilibriert in einem Wasserbad bei 56 ° C
Hinweis # 6: Temperatur der Medien kann die Stabilität und damit Aktivität des Wirkstoffs oder der Verbindung geprüft beeinflussen. - Soft-Agar: geschmolzen und äquilibriert in einem Wasserbad auf 42 ° C
- LB-Agar: geschmolzen und äquilibriert in einem Wasserbad bei 56 ° C
1. Der Bau des Gradienten
- Mark zehn Spuren, gleichmäßig über eine Kante der Unterseite des Platzes Petrischale verteilt.
- Die Schale auf einer schiefen Ebene, so dass die untere Kante markiert erhöht ist 7mm;
ein dickes Gras oder anderen flachen Gegenstand kann als Unterstützung eingesetzt werden, um das Gericht zu erheben. Gießen Sie 25 ml warmem (~ 56 ° C) LB-Agar auch mit einer entsprechenden Konzentration der Auswahl Agenten in die geneigte Schale gemischt. Dies ist die unterste Schicht des Gradienten. Sicherstellen, dass die LB-Agar gleichmäßig überzieht die Petrischale, so dass die erhöhte, markierte Ende der Schale ~ 1mm von LB-Agar enthält und dem abgesenkten Teil enthält ~ 8mm von LB-Agar. Dann lassen Agar für 10-15 Minuten.
Hinweis # 7: Für hydrophobe Auswahl Agenten, 0,1% Tensid (Entschäumer B-Emulsion) kann auf die LB-Agar hinzugefügt werden, um Federung und eine gleichmäßige Verteilung des Medikaments zu erleichtern. Add Tensid zu den warmen sterile LB-Agar mit kräftigem Schütteln vor der Zugabe der Auswahl Agenten. Das Tensid sollte auszusetzen, wie ein feiner Nebel aus kleinen Tröpfchen, große Tropfen zeigen die Medien ist zu heiß und kann eine gleichmäßige Verteilung der Testsubstanz zu hemmen. - Nach den ersten 25 ml LB-Agar ausgehärtet ist, wird die Schale auf einer ebenen Fläche bewegt. Anschließend wird 25 ml LB-Agar ohne Selektionsmittel gegossen, um die erste LB-Agar-Oberfläche zu überlagern. Dies ist die oberste Schicht des Gradienten. Sicherstellen, dass die LB-Agar deckt die gesamte Oberfläche der unteren Schicht. Deckel mit Deckel schräg zur Belüftung und ermöglichen Agar für 10-15 Minuten.
Hinweis Nr. 8: Seien Sie sich bewusst von Aerosol-Gefahren und möglichen Verflüchtigung der Testverbindung; pour Gradienten in einem chemischen oder biologischen Schutzhaube, wenn durch chemische Sicherheitsanforderungen vorgeschrieben.
Hinweis # 9: Gradient Speisen sollten innerhalb von 4h verwendet werden, um die Steigung der Drogen-oder Testverbindung Konzentration zu bewahren.
2. Stamp Übertragung von Bakterien
- Weichagar sollte äquilibriert bis 42 ° C. Transfer 2 ml Flüssigkeit Weichagar in den Deckel oder Boden eines 100mm rund Petrischale. Pipette 40 ul der Bakterienkultur in die Soft-Agar und dann rockt die Platte zu mischen.
Hinweis # 10: Das Wachstum der Bühne einer Bakterienkultur kann seine Reaktion auf ein Medikament oder eine Testverbindung Auswirkungen. Kulturen in der log-Phase oder Übernacht-Kulturen in der stationären Phase sollten konsequent für einheitliche Ergebnisse verwendet werden. Die Zelldichte können auch windschief Ergebnisse, also alle Kulturen sollten verdünnt abgestimmt A 600 Dichtewerte haben. Übernacht-Kulturen sollten verdünnt, um eine A 600 Dichte von weniger als 1,0 haben, werden - Bestreichen Sie die lange Kante des Glas-Objektträger mit den Soft-Agar-Mischung. Dann Ausrichten der beschichteten Rand der Folie mit der unteren Marke (von niedrigen zu hohen Konzentration) auf den Gradienten Gericht, berühren Sie die Folie auf die Oberfläche des Agar. Ein Soft-Touch genügt, um ein Band der Soft-Agar, um den Verlauf Oberfläche zu übertragen. Die Folie wird dann beiseite zur Reinigung und Wiederverwendung gesetzt.
- Wiederholen Sie diesen Vorgang, für den Rest der bakteriellen Proben. Referenzproben sollten auf jeden Gradienten Gericht aufgenommen werden, wenn mehrere Gradienten ausgeführt wird.
- Inkubieren Sie die Steigung Teller von oben nach unten über Nacht bei 37 ° C. Die Zeiten und Temperaturen der Inkubation kann für verschiedene Auslesen Bakterienstämme, aber über Nacht bei 37 ° C unterscheiden ist in der Regel ausreichend für sichtbares Wachstum.
3. Imaging und Analyse von Wachstum
- Imaging: Nach Wachstum über Nacht, Verläufe können direkt abgebildet werden oder fixiert und mit einer Lösung von 0,2 mg / ml Acridinorange in 95% EtOH, um den Kontrast zu erhöhen. Die Platte wird bei Raumtemperatur für 5 min inkubiert, in der Färbelösung, dann mit 95% EtOH gewaschen und anschließend über einen UV-Licht-Box abgebildet.
Hinweis # 11: Achten Sie darauf, Kolonien von der Platte bündig, sondern Rock-Färbelösung und wäscht über die Platte und entfernen Sie die Lösungen von Ecken. - Für Phänotyp-Analyse der einzelnen Mutanten Plasmiden, ist der Abstand von Wachstum gegen den Konzentrationsgradienten zu einem Standard auf jedem Gefälle normalisiert. Diese relativen Werte können dann über Steigungen verglichen werden.
Hinweis # 12: Abhängig von der Art der zytotoxischen Wirkung, eine scharfe Kante oder eine diffuse Kante kann beobachtet werden (vgl. zB Panels A und B in Abb. 2). Im Falle der diffusen Kanten, ist es ratsam, an den Rand o messenf kontinuierliches Wachstum, da die einzelnen Kolonien zu einer erhöhten Variabilität zeigen, neigen. - Bei Bibliotheks-Auswahl werden die einzelnen Kolonien, die bei höheren Konzentrationen als die elterlichen Wildtyp Kontrolle wachsen isoliert und sequenziert, um Mutationen Beitrag zum erhöhten Schutz zu identifizieren.
III. Repräsentative Ergebnisse:
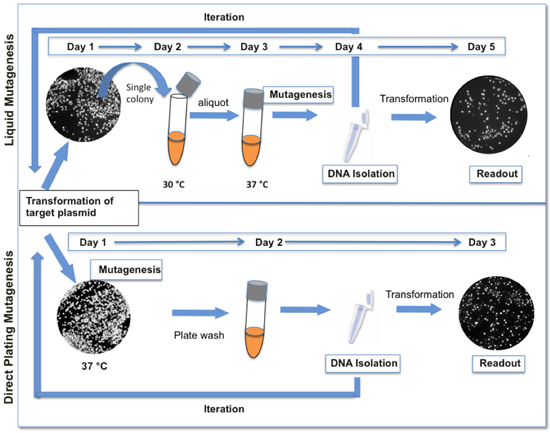
Abbildung 1. Vergleich zwischen Flüssigkeit und direkte Beschichtung Mutagenese Protokolle. Die direkte Beschichtung Mutagenese Protokoll hier (unten) dargestellt ist schneller und benötigt weniger Schritte als unsere ursprüngliche Flüssigkeit Mutagenese-Protokoll (oben). Wenn GFP als Reporter verwendet wird, sind Variationen in der Fluoreszenz-Indikator für die genetische Vielfalt in der Bibliothek vorhanden. In der Regel einen Zyklus von Mutagenese führt zu 12-18% Kolonien mit deutlich erniedrigter Fluoreszenz.
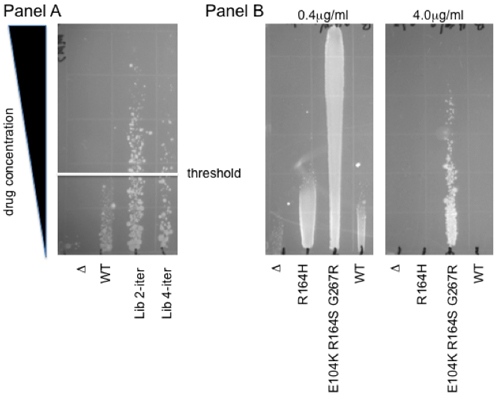
Abbildung 2. Gradient Widerstand Assays. Panel A-Auswahl für eine erhöhte Resistenz gegenüber Methyl-Methansulfonat (MMS). Plasmid-Bibliotheken des menschlichen oxidative Demethylase ABH2 wurden für eine erhöhte Resistenz gegenüber MMS ausgewählt. Zwei solcher Bibliotheken, die zwei und vier Iterationen der Mutagenese-Protokoll sind im Vergleich zu den elterlichen Wildtyp (WT) und dem leeren Vektor (Δ) dargestellt. Die weiße Linie zeigt die Grenze, ab der einzelnen Mutantenkolonien für weitere phänotypische Analyse isoliert wurden. Panel B Cefotaxim Schutz, bezeichnend für extended-spectrum β-Lactamase-Aktivität. R164H und E104K R164H G267R, zwei Mutanten von beta-Lactamase zuvor identifizierten folgende LF- Pol I Mutagenese gekoppelt Aztreonam Auswahl 4 sind auf einem 0.4μg/mL und 4μg/mL Cefotaxim Gradienten gezeigt. Beachten Sie, dass das Wildtyp-β-Lactamase-Enzym keinen Schutz gegenüber Zellen, die einen leeren Vektor Δ verleiht. Daher stellen diese Mutanten die Entwicklung eines neuen biochemischen Aktivität 8,9.
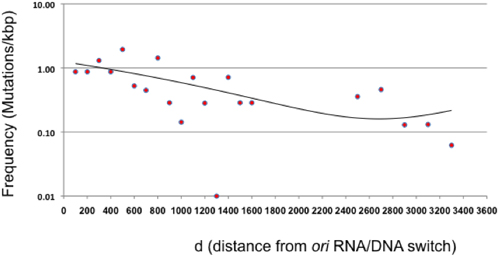
Abbildung 3. Mutation Frequenz als Funktion des Abstandes von ori (d). Die Mutation Frequenz nach einem einzigen Zyklus der direkten Beschichtung Mutagenese ist für 100 bp Abständen in Bezug auf die RNA / DNA-Schalter des ColE1 Replikation gezeigt. Der Bereich zwischen 1600 und 2400 nicht vertreten ist, weil β-Lactamase es sich nicht um neutrale Ziel. Die Punkte über β-Lactamase repräsentieren 200 bp Abständen angesichts der insgesamt niedrigen Frequenz von Mutationen in diesem Bereich. Der Trend (binomischen Gleichung, mit R 2 = 0,41) ist als Linie dargestellt.

Ergänzende Abbildung 1. LF Pol I-haltigen Pol I-Plasmid. Eine Sequence (FASTA-Format). Sequence Informationen zur Bestimmung der geeigneten Restriktionsenzym (s) zu verwenden, wenn die Linearisierung der Pol I Plasmid entweder für Iteration (Erzeugung einer zufälligen Mutation Bibliothek Schritt 4) oder Auslesen (Schritt 5). B Allgemeine Funktionen und Restriktionskarte des Plasmids. Die Lage des pSC101 Replikationsursprung, die Chloramphenicol-Resistenz-Marker (CAT) und LF-Pol I-Gen vorgestellt werden. Die Lage der einzelnen Restriktionsstellen ist gut ausgeschildert.
| Bibliothek | Direkte Plating | Flüssigkeit (1 Tag) | Flüssigkeit (3 Tage) | |
| Mutationen (#) | 95 | 40 | 142 | |
| Klone sequenziert | 288 | 96 | 190 | |
| Insgesamt Coverage (bp) | 182.000 | 102.000 | 213.000 | |
| Mutation freq (x10 3 bp) | 0,52 | 0,39 | 0,67 | |
| Freq d <1000 (x10 3 bp) | 0,92 | 0,41 | 0,70 |
Tabelle 1. Mutation Frequenzen Frequenzen (ausgedrückt als Anzahl der Mutationen / bp) für d <1000, also innerhalb der 1000 bp neben dem RNA / DNA-Schalter für drei Mutagenese Protokolle:. Direkten Beschichtung, Flüssigkeitssättigung (1 Tag), und flüssige hypersaturation ( 3 Tage).
| Direkte Plating | Flüssigkeit | ||
| Mutationen (#) | 95 | 182 | |
| Spectrum (%) | |||
| A bis G | A bis G | 6,3 | 19,2 |
| T zu C | 4,2 | 3,8 | |
| C zu T | G bis A | 27,4 | 13,7 |
| C zu T | 35,8 | 35,2 | |
| A bis T | A bis T | 5,3 | 8,2 |
| T zu A | 5,3 | 7,1 | |
| T zu G | T zu G | 1,1 | 1,1 |
| A bis C | 0,0 | 2,2 | |
| G zu T | G zu T | 1,1 | 2,7 |
| C nach A | 2,1 | 2,7 | |
| C bis G | C bis G | 2,1 | 1,1 |
| G zu C | 5,3 | 2,7 | |
| Indels | Ins | 3,2 | 0,0 |
| Del | 1,1 | 0,0 | |
| A bis N | 11,6 | 29,7 | |
| G bis N | 33,7 | 19,2 | |
| T zu N | 10,5 | 12,1 | |
| C zu N | 40,0 | 39,0 | |
| Ts | 73,7 | 72,0 | |
| Fernseher | 22,1 | 28,0 | |
| Indels | 4,2 | 0 |
Tabelle 2. Metrics der Mutation für die direkte Beschichtung und flüssige Mutagenese. Die Tabelle zeigt die Anzahl der beobachteten Mutationen (in Reihe) und die Mutation Spektrum (in%) nach einem einzigen Zyklus der Mutagenese. Das Spektrum ist nach unten durch komplementäre Paare gebrochen, von Nukleotid-Änderungen und durch die Art der Mutation.
Diskussion
Dieser Artikel stellt eine Mutagenese-Protokoll, das die Erzeugung von großen zufälligen Mutanten-Bibliotheken ermöglicht, ohne die Notwendigkeit zum Klonen oder PCR. Diese Methode basiert auf fehleranfällige Replikation eines Plasmids kodierend für eine Sequenz von Interesse basiert. In der Theorie Mutationen weitgehend sollten von 100-300 bp unmittelbar hinter dem RNA / DNA-Schalter befindet eingeschränkt werden, produziert von der Größe des Führers Strang Intermediat durch Pol I 2,10. Wir fanden, dass unter den Bedingungen in diesem Protokoll vorgestellt, Pol I-Mutationen in der ganzen Plasmid auftreten, obwohl abnehmende Häufigkeit der Entfernung aus dem Plasmid ori erhöht (Abbildung 3). Diese Erkenntnis impliziert, dass der Übergang oder "switch" von Pol I zu Pol III während ColE1 Plasmidreplikation viel langsamer als bisher berichtet wird, zumindest unter unseren experimentellen Bedingungen 2, und stimmt mit früheren Studien, in denen eine funktionelle Redundanz zwischen Pol I und Pol III 11.
Unsere ursprüngliche Protokoll beschriebene Mutagenese in flüssigen Kulturen (4). Dieses Protokoll liefert 0,41 Mutationen / kb für d <1000, also innerhalb der 1000 bp neben dem RNA / DNA-Schalter (Tabelle 1). Hypersaturation, indem man die Flüssigkeit Kultur in den Shaker für 3 Tage ohne den Zusatz von frischen Medien stellt sich die Mutationsrate auf 0,70 Mutationen / kb aber die Ergebnisse in einem sehr schlechten Plasmidausbeute (weniger als 1% im Vergleich zu Tag 1; Daten nicht gezeigt) ( Tabelle 1). Hier präsentieren wir ein vereinfachtes Protokoll direkt plating die Umwandlung der Zielplasmid auf Agar bei 37 ° C auf (Abbildung 1). Dieses Verfahren produziert die größte Häufigkeit der Mutation / Zyklus von Mutagenese (0,92 Mutationen / kb für d <1000) und erleichtert Iteration. Ändern Kulturbedingungen auf festen Medien wirkt sich auch auf die Mutation Spektrum (Tabelle 2). Direkte Beschichtung Mutagenese reduziert die deutliche Asymmetrie zwischen komplementären Basenpaar-Substitutionen in flüssiger Kultur gesehen (vgl. C → T vs G → A und A → G vs T → C). Auf der anderen Seite, produziert direkte Beschichtung mehr indels (von weniger als 0,5% bis 4%) und verringerte sich die Zahl der A → G Mutationen durch 3-fache, was zu einer Überrepräsentation von G / C-Mutationen (74%). Insgesamt glauben wir, dass der größere Einfachheit und erhöhte Effizienz der direkten Beschichtung Protokoll hier präsentierten die Vorteile der etwas ausgeglichener Mutationsspektrum mit flüssigem Mutagenese erzeugt überwiegt.
Im Rahmen des Ziels Plasmid, sind die Mutationen, die durch unsere Methode generiert nicht zu dem gewünschten Ziel-Sequenz beschränkt. Allerdings sollte die Entwicklung von neuartigen biochemischen Aktivitäten abhängig sein Mutationen innerhalb des Zielgens, da es eine eher qualitative als quantitative Veränderung darstellt. So kombinieren wir unsere Plasmid Mutagenese mit Screening von Mutanten auf Gradienten-Platten nutzt die Stärken unseres Systems (große Bibliotheken und Verfügbarkeit der Auswahl) für die Entwicklung von neuen biochemischen Eigenschaften. Weitere Anwendungen von Zufallsmutagenese wie die Optimierung der bestehenden enzymatischen Aktivitäten oder randomizing bestimmte Bereiche eines Gens erfordern Klonen folgenden Zufallsmutagenese. In diesem Fall haben die Bibliothek in ein Plasmid zu einer PCR-Amplifikation Produkt Gegensatz verbessert die Effizienz des Klonens durch die Erleichterung Verstärkung und Beschränkung.
In Summe zeigen wir ein einfaches Protokoll, um eine zufällige Mutantenbibliothek für einen bestimmten Zielgens, das sich durch seine Einfachheit und für die Vielfalt der Bibliotheken generiert erstellen. Wir zeigen, wie diese Methode mit funktionellen Selektionen für die effiziente Entwicklung von neuen biochemischen Aktivitäten gekoppelt werden kann. Darüber hinaus können unsere in vivo generierten Bibliotheken werden einfach geklont, so dass ortsspezifische Mutagenese oder Optimierung der bestehenden Aktivitäten.
Offenlegungen
Danksagungen
Diese Arbeit wurde von K08 Auszeichnung CA116429-04 bis MC und durch einen Zuschuss aus dem Conquer Cancer Now (Sorge) Stiftung # 8501 unterstützt
Materialien
| Name | Company | Catalog Number | Comments |
| carbenicillin | Cellgro | 46100R6 | 0.1mg/ml final |
| tetracycline | Fisher Scientific | BP7640 | 0.05mg/ml final |
| chloramphenicol | Genlantis | M120100 | 0.03mg/ml final |
| LB Agar Miller | Fisher Scientific | BP1425 | |
| LB Broth Miller | Difco Laboratories | 244620 | |
| Soft Agar | Difco Laboratories | 214580 | Made in house |
| Acridine Orange | Sigma-Aldrich | A38401-1 | |
| Antifoam B emulsion | Sigma-Aldrich | A5757 | |
| Glycerol | Acros Organics | 332030025 | |
| 100x100x15mm sq dish | Fisher Scientific | 0875711A | |
| 100mm rnd dish | Fisher Scientific | 0875712A | |
| Microscope slide 25x75x1mm | Gold Seal | 3048 | |
| Electroporator 2510 | Eppendorf | ||
| Electroporator 2510 | Eppendorf | ||
| 2mm gap tubes | Molecular BioProducts | 5520 | |
| Zippy mini prep kit | Zymo Research Corp. | D4020 |
Referenzen
- Camps, M. Modulation of ColE1-like plasmid replication for recombinant gene expression. Recent Pat DNA Gene Seq. 4, 58-73 (2010).
- Itoh, T., Tomizawa, J. Initiation of replication of plasmid ColE1 DNA by RNA polymerase, ribonuclease H, and DNA polymerase I. Cold Spring Harb Symp Quant Biol. 43 Pt 1, 409-417 (1979).
- Polisky, B. ColE1 replication control circuitry: sense from antisense. Cell. 55, 929-932 (1988).
- Camps, M., Naukkarinen, J., Johnson, B. P., Loeb, L. A. Targeted gene evolution in Escherichia coli using a highly error-prone DNA polymerase I. Proc Natl Acad Sci U S A. 100, 9727-9732 (2003).
- Shinkai, A., Loeb, L. A. In vivo mutagenesis by Escherichia coli DNA polymerase I. Ile(709) in motif A functions in base selection. J Biol Chem. 276, 46759-46764 (2001).
- Uyemura, D., Lehman, I. R. Biochemical characterization of mutant forms of DNA polymerase I from Escherichia coli. I. The polA12 mutation. J Biol Chem. 251, 4078-4084 (1976).
- Sedgwick, B., Robins, P., Lindahl, T. Direct removal of alkylation damage from DNA by AlkB and related DNA dioxygenases. Methods Enzymol. 408, 108-120 (2006).
- Aharoni, A., Gaidukov, L., Khersonsky, O., Mc, Q. G. S., Roodveldt, C., Tawfik, D. S. The 'evolvability' of promiscuous protein functions. Nat Genet. 37, 73-76 (2005).
- Elena, S. F., Lenski, R. E. Evolution experiments with microorganisms: the dynamics and genetic bases of adaptation. Nat Rev Genet. 4, 457-469 (2003).
- Itoh, T., Tomizawa, J. FoFormation of an RNA primer for initiation of replication of ColE1 DNA by ribonuclease H. Proc Natl Acad Sci U S A. 77, 2450-2454 (1980).
- Bryan, S. K., Moses, R. E. Sufficiency of the Klenow fragment for survival of polC(Ts) pcbA1 Escherichia coli at 43 degrees. C. J Bacteriol. 170, 456-458 (1988).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten