Method Article
Protocolli di selezione mutagenesi e funzionale per l'evoluzione Regia di proteine in E. coli
In questo articolo
Riepilogo
Qui mostriamo un semplice protocollo per creare una libreria casuale mutante per una sequenza bersaglio determinato. Mostriamo come questo metodo, che viene eseguita in vivo in Escherichia coli, possono essere accoppiati con selezioni funzionali ad evolvere nuove attività enzimatiche.
Abstract
La generazione efficiente della diversità genetica rappresenta un prezioso strumento molecolare che può essere utilizzato per etichettare la sintesi del DNA, per creare uniche firme molecolari, o per evolvere le proteine in laboratorio. Qui, vi presentiamo un protocollo che consente la generazione di grandi dimensioni (> 10 11) biblioteche mutante per una sequenza bersaglio determinato. Questo metodo si basa sulla replica di un plasmide codifica ColE1 sequenza desiderata da una bassa fedeltà variante della DNA polimerasi I (LF-Pol I). L'obiettivo plasmide si trasforma in un ceppo di E. mutatore coli e placcato su un supporto solido, producendo tra 0,2 e 1 mutazioni / kb, a seconda della posizione del gene bersaglio. Frequenze di mutazione superiori si ottengono iterando questo processo di mutagenesi. Rispetto a metodi alternativi di mutagenesi, il nostro protocollo si distingue per la sua semplicità, in quanto non la clonazione o PCR sono coinvolti. Così, il nostro metodo è ideale per l'etichettatura mutazionali di plasmidi o altri modelli io Pol o per esplorare ampie sezioni di sequenza spazio per l'evoluzione delle attività non presenti nel target originale. Lo stretto controllo dello spazio che la PCR o randomizzati oligonucleotide a base di offrire metodi possono anche essere ottenuto attraverso la clonazione di successive specifiche sezioni della biblioteca. Qui forniamo protocolli che mostra come creare una libreria casuale mutante e le modalità per stabilire farmaco a base di selezioni in E. coli per identificare mutanti espositrici nuove attività biochimiche.
Protocollo
Generazione I. di una biblioteca casuale Mutant
Pol I media di iniziazione ColE1 replica plasmide (recensione a (1-3). Il nostro metodo di mutagenesi si basa sul posizionamento di una sequenza target in un plasmide ColE1 adiacente l'origine o la replicazione e la propagazione in cellule che esprimono a bassa fedeltà DNA polimerasi I (LF-Pol I). LF-Pol I è una DNA polimerasi mutante ho codifica tre mutazioni che diminuiscono la fedeltà della replicazione, cioè I709N (motivo in A), A759R (motivo in B) e D424A (inattivando correzione di bozze) 4,5 . LF-Pol I è espresso in un ceppo di E. coli, JS200, che ha una temperatura-sensibili allele di Pol I (polA12) (6) in modo che LF-Pol I diventa l'attività predominante a 37 ° C. La replica del sequenza bersaglio in polA12 cellule in condizioni restrittive risultati nella generazione di una libreria casuale mutante. Mutagenesi è più efficiente nelle culture saturi 4. Per motivi che sono ancora poco chiare, mutagenesi non è continua, cioè la frequenza di mutazione non aumenta linearmente con il numero di generazioni, una volta che la coltura raggiunga la saturazione, anche se le cellule sono autorizzati a espandersi ulteriormente in mezzi freschi. conseguenza, aumentando ulteriormente il carico di mutazione della biblioteca richiede iterativo giri di mutagenesi e di recupero plasmide. Qui forniamo protocolli per la LF-Pol mutagenesi io. Si noti che i protocolli qui presentati sono stati notevolmente semplificati rispetto ai nostri 4 descrizione originale al fine di facilitare iterazione del processo per il raggiungimento del carico mutazione desiderata (Fig. 1).
Materiale
- Celle: JS200 recA718 polA12 (ts) uvrA155 trpE65 lon-11 sula
- JS200 WT-Pol I: JS200 cellule che esprimono wild-type (WT) Pol I
- JS200 LF-Pol I: JS200 esprimendo bassa fedeltà (LF) Pol I
- Lettura ceppo: JS200 WT-Pol I o (per complementazione), un ceppo privo di una specifica attività
- Obiettivo plasmidi
- Plasmide contenente una origine ColE1 di replica con il gene bersaglio clonato nel
1. Prima di Mutagenesi: Preparazione di elettro-competenti JS200 LF-Pol cellule I
- Scegliere un singolo JS200 LF-Pol colonia mi da una piastra di LB cresciuto a 30 ° C (condizioni permissive) durante la notte la selezione appropriata contenente antibiotico per il cuscinetto plasmide LF-Pol I (0.03mg / mL cloramfenicolo) e posizionare la colonia in un test provetta contenente 8 ml di LB con cloramfenicolo. Far crescere la coltura a 30 ° C e agitazione a 250rpm notte.
- Al mattino, espandere la cultura versando l'8 mL di JS200 LF-Pol io in un pallone contenente 400 ml di LB con cloramfenicolo. Lasciate che la cultura crescere a 30 ° C sotto agitazione a 250rpm fino a raggiungere una A 600 del ,4-0,7 (di solito 3-4 ore).
- Una volta a una A 600 del 0,4-0,7, chill la cultura in ghiaccio umido per 15 minuti.
- Trasferire la cultura refrigerate a un contenitore adeguato per centrifugazione. Cellule pellet per centrifugazione a 4 ° C. Eliminare il sopranatante e aggiungere 10 ml di sterile e fredda (in ghiaccio umido), glicerolo 10% al contenitore e risospendere le cellule usando una pipetta sierologica.
- Trasferire la risospensione delle cellule in una provetta da 50 ml. Riempire il tubo conico fino alla mL segno 45 con 10% glicerolo, quindi si centrifuga per 15 minuti a 4 ° C e 4000rpm.
- Eliminare il sopranatante, aggiungere 10 ml una volta di più del 10% glicerolo al tubo conico, e risospendere le cellule usando una pipetta sierologica o normale. Ancora una volta, riempire il tubo conico a ml segno 45 con 10% glicerolo e centrifugare per 15 minuti a 4 ° C e 4000rpm. Ripetere questa operazione altre due volte per rimuovere ogni traccia di sali.
- Dopo la centrifuga finale, risospendere il pellet di cellule in parte pari al 10% glicerolo (cioè risospendere 2 ml di cellule con 2 mL di glicerolo al 10%).
- Aliquota tra 100 ul e 500 ml di cellule sospese in tubi di memorizzazione diversi. Quick-congelare le cellule in ghiaccio secco e poi conservarli a -80 ° C.
- Prima di utilizzare le cellule per elettro-competente trasformazioni, scongelare le cellule lentamente sul ghiaccio.
2. Mutagenesi: Trasformare l'obiettivo plasmide
- Pipettare 40 ml di elettro-competente JS200 LF-Pol cellule I e tra i 30-250ng del DNA bersaglio plasmide nella cuvetta elettroporazione divario 2mm.
Nota # 1: Un ColE1 plasmide contenente GFP può essere effettuata attraverso mutagenesi in parallelo con il gene bersaglio come controllo. Dopo il completamento della fase di lettura, GFP può essere placcato su piastre di agar LB e visualizzati per la mutagenesi. Le colonie che appaiono scuri o poco illuminati contengono mutazioni inattivanti. - Pulse il composto nella elettroporatore a 1800V. Controllare la costante di tempo (T C) per assicurare condizioni uniformi di elettroporazione per ogni campione; idealmente T C = 5-6 sec.
- Recuperare il cellulare / DNA miscela in 1 ml di brodo LB per 40 minuti a 37 ° C (rescondizioni trictive) agitando a 250 giri al minuto.
- Tavola 50 microlitri della cultura di recupero su un 100mm piatto agar LB Petri pre-riscaldato a 37 ° C che contiene sia cloramfenicolo e di un'adeguata concentrazione di un antibiotico per selezionare il target plasmide.
Nota: # 2: Puntare alla piastra le cellule ad un "vicino prato" concentrazione. Un "prato vicino" la concentrazione è definito come una serie distinte, ma di innumerevoli colonie (> 1000 colonie / piatto 100 mm). La diluizione placcato, se del caso, dipenderà da come elettro-competente le cellule sono. Se le cellule non sono molto elettro-competente e un "prato vicino" non è raggiungibile dalla placcatura cultura recupero "pulito", quindi centrifugare la cultura di recupero per 2 minuti a 4000rpm, versare il sopranatante, risospendere le cellule in 50 l di brodo LB e la piastra della cultura. - Incubare la dishe Petri (s) durante la notte a 37 ° C.
3. Mutagenesi: recupero plasmidi
- Il giorno dopo, lavare i piatti Petri pipettando 2 ml di brodo LB sulle celle. Trasferire le colonie batteriche dai piatti LB agar Petri al brodo LB da "scrubbing" loro fuori il piatto con un sterilizzato triangolo a forma di bacchetta di vetro. Aggiungere 1 ml di brodo LB primo, raccogliere il lavaggio e ripetere la procedura con la seconda ml di brodo LB.
- Isolare il DNA plasmidico dal lavare piatto. (Questo DNA plasmidico costituisce la biblioteca).
Nota # 3: Il lavaggio raccolti dalla piastra di LB può essere troppo denso per mini-prep nella sua interezza. Se questo è il caso, mini-prep assegnato il massimo importo per il tuo mini-kit di preparazione (in genere, questo ha comportato diluendo il lavaggio a OD = 1 e durante la preparazione ~ 3 mL della coltura diluita) o la scala fino ad un maxi-prep
4. Iterazione
- Limitare 1μg del DNA isolata plasmide con un enzima di restrizione che linearizza I LF-Pol plasmide, ma non tagliare il tuo obiettivo plasmide (supplementare Fig. 1).
- Pulire il digest restrizione utilizzando un kit di purificazione del DNA.
Nota # 4: Questo passaggio rimuove tutte le tracce del enzima di restrizione e il suo buffer. Questo passo è essenziale per mantenere bassa la concentrazione di sale per le successive trasformazioni elettro-competente. - Ri-trasformare 30 250ng del ristretto target plasmide indietro libreria in fresco JS200 LF-Pol ho le cellule di mettere le librerie, attraverso cicli successivi di mutagenesi.
Nota # 5: la linearizzazione Pol ho plasmide usando un enzima di restrizione assicura solo l'obiettivo plasmide si trasforma. - Ripetere le sezioni 2 e 3.
5. Lettura
- Limitare il DNA isolato plasmide con un enzima di restrizione (s) che linearizza sia l'obiettivo plasmide e la Pol. I plasmide. Esegui il digest di un 1% gel per garantire la quantità e la qualità dei plasmidi. Limitare ~ 400ng del DNA isolata plasmide è solitamente sufficiente per l'analisi.
- Limitare 1μg del DNA isolata plasmide con un enzima di restrizione che linearizza I LF-Pol plasmide, ma non tagliare il tuo obiettivo plasmide.
- Pulire il digest restrizione utilizzando un kit di purificazione del DNA.
- Trasformare la biblioteca limitato obiettivo plasmide in un ceppo di lettura per caratterizzare le mutazioni.
II. Schermo mutanti e analisi Trait utilizzando piastre di crescita sfumatura.
Al fine di illustrare come la grande diversità genetica presente nelle nostre biblioteche può essere accoppiato ad una selezione funzionale, qui mettiamo a disposizione un protocollo per la selezione funzionale a base di droga in vivo in E. coli. Questo metodo si basa sulla crescita lungo un gradiente di droga su agar solido. Questo permette la caratterizzazione simultanea di più campioni (fino a 12) su una gamma di concentrazioni, fornendo una gamma dinamica più ampia rispetto a un trattamento singolo farmaco. Un altro vantaggio è che la non-lineare di lettura di questo saggio buffer differenze moderata della redditività, all'interno di una gamma di 2 volte. Così, questo citotossicità resistenza test fornisce un mezzo robusto e rapido per selezionare le biblioteche mutante e di determinare il profilo del fenotipo dei mutanti singoli. Fig. 2 mostra un esempio di ciascuno di questi usi: pannello mostra una selezione di mutanti singoli da un essere umano demetilasi ossidativo ABH2 biblioteca. Colonie in crescita al di sopra della soglia di WT sono selezionati per una maggiore protezione da citotossicità causata dalla metilazione solfonato metano metilico agente (MMS) 7. Pannello B mostra un esempio di sfumature utilizzate per la caratterizzazione clone individuali. Il livello di resistenza alla terza generazione di cefalosporine cefotaxime antibiotico è indicato su una pendenza agar per WT β-lattamasi e per due a spettro esteso mutanti, R164H e E104K R164S G267R 4. Si noti che a seconda della forza degli effetti osservati, più di un piatto unico può essere necessario per la quantificazione adeguata: mentre il gradiente 0.4mg/ml permette di confronto diretto con i cloni di controllo, il livello di resistenza dei mutanti più potenti possono solo essere stabilita utilizzando una concentrazione più altastrazione di cefotaxime (4mg/ml).
Materiale
- Attrezzatura
- 100x100x15mm piazza Petri (Fisher Sci # 0875711A)
- 100 millimetri Petri rotonda
- 25x75x1mm microscopio vetrino (Fischer Sci # 1255015)
- Tubo da 50 ml è laureato
- Media
- LB agar: sciolto ed equilibrato in un bagno d'acqua a 56 ° C
Nota # 6: temperatura dei media possono influenzare la stabilità e quindi l'attività del farmaco o composti in esame. - Morbido Agar: sciolto ed equilibrato in un bagno d'acqua a 42 ° C
- LB agar: sciolto ed equilibrato in un bagno d'acqua a 56 ° C
1. Costruzione del gradiente
- Segna dieci corsie, distanziati in modo uniforme su uno spigolo del fondo del piatto piazza Petri.
- Porre la capsula su un tale che il bordo inferiore segnata è elevata pendenza 7 millimetri;
un'erba fitta o un altro oggetto piatto può essere usato come supporto per elevare il piatto. Versare 25 mL di caldo (~ 56 ° C) LB agar mescolato bene con un'adeguata concentrazione dell'agente selezionando nel piatto inclinato. Questo è lo strato inferiore della sfumatura. Assicurarsi che l'agar LB in modo uniforme riveste la capsula di Petri in modo che la sopraelevata, fine marcato del piatto contiene ~ 1 mm di agar LB e la parte abbassato contiene ~ 8mm di agar LB. Poi permettono di impostare agar per 10-15 minuti.
Nota # 7: per idrofobo agenti selezione, 0,1% tensioattivo (emulsione antischiuma B) possono essere aggiunti alla agar LB per facilitare la sospensione e la distribuzione uniforme del farmaco. Aggiungi tensioattivo a caldo l'agar LB sterile con vigorosa agitazione prima dell'aggiunta dell'agente selezione. Il tensioattivo sospenda come una nebbia sottile di piccole gocce, grandi gocce di indicare i mezzi di comunicazione è troppo caldo e possono inibire la distribuzione uniforme del farmaco di prova. - Dopo i primi 25 mL di agar LB ha indurito, il piatto viene spostato in una superficie piana. Prossimo, 25 mL di agar LB senza l'agente di selezione viene versato per sovrapporre la prima superficie agar LB. Questo è lo strato superiore del gradiente. Assicurarsi che l'agar LB copre l'intera superficie dello strato di fondo. Coprire con coperchio di traverso per la ventilazione, e consentono di impostare agar per 10-15 minuti.
Nota # 8: Essere consapevoli dei rischi di aerosol e volatilizzazione potenziale di composto in esame; versare gradienti in una cappa chimica o biologica, se prescritti da requisiti di sicurezza chimica.
Nota # 9: piatti gradiente deve essere utilizzato entro 4 ore per preservare il gradiente del farmaco o composto concentrazione di prova.
2. Timbro trasferimento di batteri
- Agar molle devono essere equilibrati a 42 ° C. Trasferimento 2 ml di liquido agar molle nel coperchio o sul fondo di un 100mm piatto tondo Petri. Pipettare 40 microlitri della coltura batterica in agar molle e poi mescolare a dondolo della piastra.
Nota # 10: La fase di crescita di una coltura batterica possono influenzare la risposta ad un farmaco o di composto in esame. Culture in fase di log o culture pernottamento in fase stazionaria deve essere usato con coerenza per risultati uniformi. Densità cellulare può anche inclinare risultati relativi, così tutte le culture devono essere diluite a indovinati A 600 valori di densità. Culture durante la notte deve essere diluito per avere un A 600 densità inferiore a 1,0 - Rivestire il lato lungo del vetrino da microscopio con la miscela agar soffice. Poi, allineando il bordo rivestito della diapositiva con il segno di fondo (dal basso verso l'alta concentrazione) sul piatto pendenza, toccare lo scivolo sulla superficie del agar. Un tocco morbido è sufficiente per trasferire un nastro di agar molle in superficie gradiente. Il vetrino viene poi accantonato per la pulizia e il riutilizzo.
- Ripetere questo processo, per il resto dei campioni batterici. Campioni di riferimento dovrebbe essere incluso in ogni piatto gradiente se più sfumature sono in esecuzione.
- Incubare la top piatto pendenza verso il basso durante la notte a 37 ° C. Tempi e temperature di incubazione può variare per la lettura diversi ceppi batterici, ma durante la notte a 37 ° C è solitamente sufficiente per la crescita visibile.
3. Imaging e analisi della crescita
- Imaging: dopo la crescita durante la notte, le sfumature possono essere direttamente ripreso o fissate e colorate con una soluzione di 0,2 mg / ml acridina arancio nel 95% EtOH, per aumentare il contrasto. La piastra è incubata a temperatura ambiente per 5 minuti, nella soluzione colorante, poi lavato con EtOH 95% e poi ripreso nel corso di un light box UV.
Nota # 11: Fare attenzione a non lavare le colonie dalla piastra di roccia, ma piuttosto la soluzione colorante e lava sopra la piastra e togliere le soluzioni da angoli. - Per l'analisi dei singoli plasmidi fenotipo mutante, la distanza di crescita contro il gradiente di concentrazione è normalizzata a uno standard su ogni pendenza. Questi valori relativi possono quindi essere confrontati attraverso sfumature.
Nota # 12: A seconda della natura degli effetti citotossici, uno spigolo vivo o un bordo più diffuso può essere osservato (cfr. per esempio pannelli A e B in Fig. 2). Nel caso di bordi diffuso, si consiglia di misurare il bordo of continua crescita, come singole colonie tendono a mostrare una maggiore variabilità. - Per la selezione biblioteca, singole colonie che crescono a concentrazioni superiori al controllo parentale wild-type sono isolati e sequenziati per identificare le mutazioni che contribuiscono a una maggiore protezione.
III. Rappresentante dei risultati:
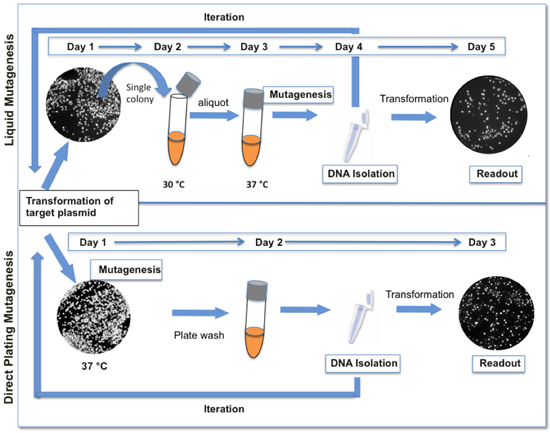
Figura 1. Confronto tra protocolli di placcatura liquido e diretto mutagenesi. Il protocollo di mutagenesi diretta placcatura qui presentati (in basso) è più veloce e richiede meno passaggi rispetto al nostro protocollo originale mutagenesi liquido (in alto). Quando GFP è usato come un reporter, variazioni di fluorescenza sono indicativi della diversità genetica presente in biblioteca. Tipicamente, un ciclo di risultati mutagenesi nel 12-18% colonie con livelli sensibilmente diminuito di fluorescenza.
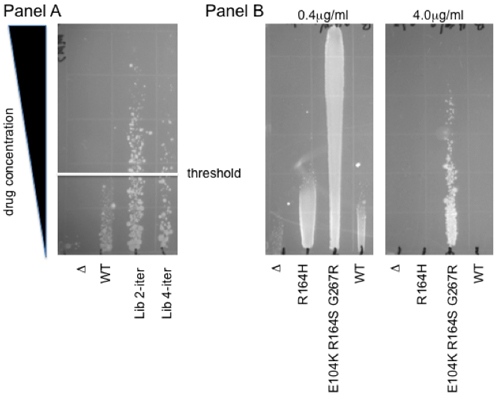
Figura 2. Gradiente di resistenza saggi. Un pannello di selezione per una maggiore resistenza al metil-metanosolfonato (MMS). Librerie plasmidi della demetilasi ABH2 umano ossidativo sono stati selezionati per una maggiore resistenza a MMS. Due biblioteche quali rappresentano due e quattro iterazioni del protocollo di mutagenesi sono indicati rispetto al tipo parentale selvatica (WT) e il vettore vuoto (Δ). La linea bianca indica la soglia oltre la quale sono state isolate singole colonie mutanti per ulteriori analisi fenotipica. Pannello B protezione Cefotaxime, indicativo di spettro esteso β-lattamasi attività. R164H e E104K R164H G267R, due mutanti di beta-lattamasi precedentemente individuati dopo LF- Pol mutagenesi ho accoppiato alla selezione aztreonam 4, vengono visualizzati su un 0.4μg/mL e un gradiente 4μg/mL cefotaxime. Si noti che il wild-type β-lattamasi enzima conferisce alcuna protezione rispetto a cellule che esprimono un vettore vuoto, Δ. Pertanto, questi mutanti rappresentano l'evoluzione di una nuova attività biochimica 8,9.
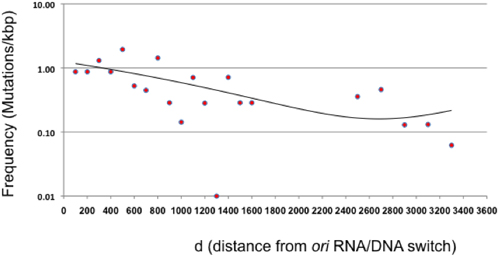
Figura 3. Frequenza di mutazione in funzione della distanza dalla ori (d). La frequenza di mutazione a seguito di un singolo ciclo di mutagenesi placcatura diretta viene mostrato per intervalli di 100 bp rispetto al DNA / RNA interruttore della provenienza ColE1 di replica. L'area tra 1600 e 2400 non è rappresentato perché β-lattamasi non rappresenta un obiettivo neutrale. I punti al di là di β-lattamasi rappresentano intervalli di 200 bp, data la frequenza complessivo della mutazione in questo settore. La tendenza (equazione binomiale, con un R 2 = 0.41) è mostrata come una linea.

Supplementare Figura 1. Pol LF I-Pol contenenti I plasmide. Una sequenza (in formato FASTA). Informazioni di sequenza per determinare l'enzima di restrizione appropriato (s) da utilizzare durante la linearizzazione I Pol plasmide sia per iterazione (generazione di un passaggio casuale biblioteca mutazione 4) o lettura (punto 5). B Caratteristiche generali e mappa di restrizione del plasmide. Il posizione dell'origine pSC101 di replica, il marcatore di resistenza cloramfenicolo (CAT) e LF-Pol gene io sono presentati. La posizione dei singoli siti di restrizione è indicato pure.
| Biblioteca | Placcatura diretta | Liquido (1 giorno) | Liquido (3 giorni) | |
| Mutazioni (#) | 95 | 40 | 142 | |
| Cloni sequenziati | 288 | 96 | 190 | |
| Copertura totale (bp) | 182.000 | 102.000 | 213.000 | |
| Freq mutazione (x10 3 bp) | 0,52 | 0,39 | 0,67 | |
| Freq d <1000 (x10 3 bp) | 0,92 | 0,41 | 0,70 |
Tabella 1. Frequenze di mutazione Frequenze (espresso come # di mutazioni / bp) per d <1000, cioè entro il 1000 bp adiacente al DNA / RNA interruttore, per tre protocolli di mutagenesi:. Placcatura diretta, la saturazione del liquido (1 giorno), e hypersaturation liquido ( 3 giorni).
| Placcatura diretta | Liquido | ||
| Mutazioni (#) | 95 | 182 | |
| Spectrum (%) | |||
| A a G | A a G | 6,3 | 19,2 |
| T a C | 4,2 | 3,8 | |
| C a T | G ad A | 27,4 | 13,7 |
| C a T | 35,8 | 35,2 | |
| Dalla A alla T | Dalla A alla T | 5,3 | 8,2 |
| T ad A | 5,3 | 7,1 | |
| T a G | T a G | 1,1 | 1,1 |
| Dalla A alla C | 0,0 | 2,2 | |
| G a T | G a T | 1,1 | 2,7 |
| C alla A | 2,1 | 2,7 | |
| C a G | C a G | 2,1 | 1,1 |
| G a C | 5,3 | 2,7 | |
| Indel | Ins | 3,2 | 0,0 |
| Del | 1,1 | 0,0 | |
| Dalla A alla N | 11,6 | 29,7 | |
| G a N | 33,7 | 19,2 | |
| T per N | 10,5 | 12,1 | |
| C a N | 40,0 | 39,0 | |
| Ts | 73,7 | 72,0 | |
| TV | 22,1 | 28,0 | |
| Indel | 4,2 | 0 |
Tabella 2. Metriche di mutazione per la placcatura diretta e mutagenesi liquido. La tabella presenta il numero di mutazioni osservate (in numero) e lo spettro di mutazione (in%) a seguito di un singolo ciclo di mutagenesi. Lo spettro è suddiviso per coppie complementari, dai cambiamenti nucleotide, e dal tipo di mutazione.
Discussione
Questo articolo presenta un protocollo di mutagenesi che consente la generazione di grandi librerie casuali mutante senza la necessità di clonazione o PCR. Questo metodo è basato sul soggetto a errori di replica di un plasmide codifica di una sequenza di interesse. In teoria, le mutazioni devono essere in gran parte limitato ai 100-300 pb situato immediatamente a valle del DNA / RNA interruttore, la dimensione del leader filo intermedia prodotta da Pol I 2,10. Abbiamo trovato che, nelle condizioni presentate in questo protocollo, Pol mutazioni ho verificarsi in tutto il plasmide, anche se diminuisce la frequenza come la distanza dagli aumenti plasmide ori (Figura 3). Questa scoperta implica che la transizione o "switch" da Pol I, Pol III ColE1 durante la replicazione del plasmide è molto più graduale di quanto riportato in precedenza, almeno nelle nostre condizioni sperimentali 2, e concorda con studi precedenti suggeriscono una ridondanza funzionale tra Pol I e Pol III 11.
Il nostro protocollo originale descritta mutagenesi in colture liquide (4). Questo protocollo produce mutazioni 0,41 / kb per d <1000, cioè entro il 1000 bp adiacente al DNA / RNA interruttore (Tabella 1). Hypersaturation lasciando la coltura liquida nello shaker per 3 giorni senza l'aggiunta di eventuali nuovi mezzi di comunicazione aumenta la frequenza di mutazione mutazioni a 0,70 / kb, ma si traduce in pessime resa plasmide (meno dell'1% rispetto al giorno 1; dati non riportati) ( Tabella 1). Qui vi presentiamo un protocollo semplificato basato sulla placcatura direttamente la trasformazione del target plasmide su agar solido a 37 ° C (Figura 1). Questo procedimento produce la maggior frequenza di mutazione / ciclo di mutagenesi (0,92 mutazioni / kb per d <1000) e facilita notevolmente l'iterazione. Modifica condizioni di coltura di terreni solidi colpisce anche lo spettro di mutazione (Tabella 2). Mutagenesi placcatura diretta riduce la marcata asimmetria tra sostituzioni di coppie di basi complementari visto in coltura liquida (confrontare C → T vs G → A e A → G vs T → C). D'altra parte, la placcatura diretto prodotto più indel (da meno 0,5% al 4%) e diminuito il numero di mutazioni G → A per 3 volte, portando ad una sovrarappresentazione di G / C mutazioni (74%). Nel complesso, crediamo che la maggiore semplicità e maggiore efficienza del protocollo placcatura diretta qui presentati supera i benefici dello spettro di mutazione leggermente più equilibrata prodotto da mutagenesi liquido.
All'interno dell'obiettivo plasmide, le mutazioni generate dal nostro metodo non si limitano alla sequenza target desiderato. Tuttavia, l'evoluzione del romanzo di attività biochimiche dovrebbe dipendere da mutazioni nel gene bersaglio, in quanto rappresenta una qualitativa piuttosto che un cambiamento quantitativo. Così, unendo le nostre mutagenesi plasmide con proiezione di mutanti su piastre gradiente sfrutta i punti di forza del nostro sistema (librerie di grandi dimensioni e disponibilità di selezione) per l'evoluzione di nuove proprietà biochimiche. Altre applicazioni di mutagenesi casuale come ottimizzare gli attuali attività enzimatiche o randomizing aree specifiche di un gene hanno bisogno di clonazione seguenti mutagenesi casuale. In questo caso, essendo la biblioteca in un plasmide in contrapposizione ad un prodotto di amplificazione di PCR migliora l'efficienza della clonazione, facilitando l'amplificazione e restrizione.
In sintesi, si dimostra un semplice protocollo per creare una libreria casuale mutante di un gene bersaglio, dato che si distingue per la sua semplicità e per la diversità delle librerie generate. Mostriamo come questo metodo può essere accoppiato con le selezioni per l'evoluzione funzionale efficiente di nuove attività biochimiche. Inoltre, il nostro in vivo generata librerie possono essere facilmente clonati, permettendo mutagenesi sito-specifica o di ottimizzazione delle attività già esistenti.
Divulgazioni
Riconoscimenti
Questo lavoro è stato sostenuto da CA116429 premio K08-04 a MC e da una sovvenzione da parte del Conquer Cancer Now (preoccupazione) fondazione # 8501
Materiali
| Name | Company | Catalog Number | Comments |
| carbenicillin | Cellgro | 46100R6 | 0.1mg/ml final |
| tetracycline | Fisher Scientific | BP7640 | 0.05mg/ml final |
| chloramphenicol | Genlantis | M120100 | 0.03mg/ml final |
| LB Agar Miller | Fisher Scientific | BP1425 | |
| LB Broth Miller | Difco Laboratories | 244620 | |
| Soft Agar | Difco Laboratories | 214580 | Made in house |
| Acridine Orange | Sigma-Aldrich | A38401-1 | |
| Antifoam B emulsion | Sigma-Aldrich | A5757 | |
| Glycerol | Acros Organics | 332030025 | |
| 100x100x15mm sq dish | Fisher Scientific | 0875711A | |
| 100mm rnd dish | Fisher Scientific | 0875712A | |
| Microscope slide 25x75x1mm | Gold Seal | 3048 | |
| Electroporator 2510 | Eppendorf | ||
| Electroporator 2510 | Eppendorf | ||
| 2mm gap tubes | Molecular BioProducts | 5520 | |
| Zippy mini prep kit | Zymo Research Corp. | D4020 |
Riferimenti
- Camps, M. Modulation of ColE1-like plasmid replication for recombinant gene expression. Recent Pat DNA Gene Seq. 4, 58-73 (2010).
- Itoh, T., Tomizawa, J. Initiation of replication of plasmid ColE1 DNA by RNA polymerase, ribonuclease H, and DNA polymerase I. Cold Spring Harb Symp Quant Biol. 43 Pt 1, 409-417 (1979).
- Polisky, B. ColE1 replication control circuitry: sense from antisense. Cell. 55, 929-932 (1988).
- Camps, M., Naukkarinen, J., Johnson, B. P., Loeb, L. A. Targeted gene evolution in Escherichia coli using a highly error-prone DNA polymerase I. Proc Natl Acad Sci U S A. 100, 9727-9732 (2003).
- Shinkai, A., Loeb, L. A. In vivo mutagenesis by Escherichia coli DNA polymerase I. Ile(709) in motif A functions in base selection. J Biol Chem. 276, 46759-46764 (2001).
- Uyemura, D., Lehman, I. R. Biochemical characterization of mutant forms of DNA polymerase I from Escherichia coli. I. The polA12 mutation. J Biol Chem. 251, 4078-4084 (1976).
- Sedgwick, B., Robins, P., Lindahl, T. Direct removal of alkylation damage from DNA by AlkB and related DNA dioxygenases. Methods Enzymol. 408, 108-120 (2006).
- Aharoni, A., Gaidukov, L., Khersonsky, O., Mc, Q. G. S., Roodveldt, C., Tawfik, D. S. The 'evolvability' of promiscuous protein functions. Nat Genet. 37, 73-76 (2005).
- Elena, S. F., Lenski, R. E. Evolution experiments with microorganisms: the dynamics and genetic bases of adaptation. Nat Rev Genet. 4, 457-469 (2003).
- Itoh, T., Tomizawa, J. FoFormation of an RNA primer for initiation of replication of ColE1 DNA by ribonuclease H. Proc Natl Acad Sci U S A. 77, 2450-2454 (1980).
- Bryan, S. K., Moses, R. E. Sufficiency of the Klenow fragment for survival of polC(Ts) pcbA1 Escherichia coli at 43 degrees. C. J Bacteriol. 170, 456-458 (1988).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon