Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Un método paso a paso para la reconstitución de un transportador ABC en partículas lipídicas Nanodisc
En este artículo
Resumen
Nanodiscos son pequeñas partículas discoidales que incorporan proteínas de membrana en un pequeño parche de doble capa de fosfolípidos. Proporcionamos un protocolo visual que muestra la incorporación paso a paso del transportador de MalFGK2 en un disco.
Resumen
El nanodisc es una partícula discoidal (~ 10-12 nm grande) que las proteínas de membrana trampa en un pequeño parche de doble capa de fosfolípidos. El nanodisc es una opción particularmente atractiva para el estudio de proteínas de membrana, especialmente en el contexto de las interacciones ligando-receptor. El método por primera vez por Sligar y colegas se basa en las propiedades anfipáticas de una ingeniería altamente a-helicoidal andamio de proteínas derivadas de la apolipoproteína A1. Las caras hidrófobas de la proteína andamio interactúan con los de acilo graso cadenas laterales de la bicapa de lípido, mientras que las regiones polares frente al entorno acuoso. Los análisis de proteínas de membrana en nanodiscos tienen ventajas significativas sobre liposoma porque las partículas son pequeñas, homogéneas y solubles en agua. Además, los métodos bioquímicos y biofísicos normalmente reservados para las proteínas solubles se pueden aplicar, y desde ambos lados de la membrana. En este protocolo visual, se presenta una reconstitución paso a paso de un carácter bienracterizado ABC transportador bacteriano, el macho-2 MalFGK complejo. La formación del disco es un proceso de auto-ensamblaje que depende de las interacciones hidrofóbicas que tienen lugar durante la eliminación progresiva del detergente. Se describen los pasos esenciales y se destaca la importancia de elegir una correcta proteína-lípido con el fin de limitar la formación de agregados y partículas más grandes de liposomas como polidispersos. Calidad simples controles, tal como cromatografía de filtración en gel, electroforesis en gel nativo y la espectroscopia de dispersión de luz dinámica asegurar que los discos han sido correctamente reconstituido.
Protocolo
Proceso de reconstitución global
El proceso de reconstitución se inicia mediante la mezcla de la proteína de membrana andamio (MSP) con el complejo purificado MalFGK 2 en presencia de solubilizadas en detergente fosfolípidos. El paso es seguido por la eliminación lenta del detergente por un material de poliestireno adsorbente llamado Bio-Beads o Amberlite (Figura 1). El proceso de auto-ensamblaje ocurre muy probablemente debido a las interacciones apolares entre los fosfolípidos hidrófobos, el complejo MalFGK 2 y la superficie de la proteína anfipática MSP. El producto final es una partícula discoide formado por dos moléculas de envolver alrededor de la MSP 2 MalFGK complejo. Las partículas se separan de los aductos y los agregados por ultra-centrifugación y cromatografía analítica de exclusión por tamaño. Las partículas se caracterizan por electroforesis en gel nativo y espectroscopia de dispersión de luz dinámica.
título "> 1. preparación de la membrana andamio de proteínas, MSP- El Su-etiquetados MSP (versión MSP1D1 1) se produce a partir pMSP1D1 plásmido en E. coli BL21 (DE3) indujo a una DO de 600 ~ 0,5 con 0,5 mM de isopropil β-D-1-tiogalactopiranósido durante 3 horas a 37 ° C.
- Las células se recogieron por centrifugación a 5.000 xg durante 10 min a 4 ° C y se resuspendieron en tampón que contenía TSG10 100 mM fluoruro de fenilmetanosulfonilo.
- Las células se lisaron con una prensa francesa (3x) a 8.000 psi y el material insoluble se eliminó por centrifugación a 5.000 xg durante 10 min a 4 ° C. La fracción soluble que contiene el MSP se aísla por ultra-centrifugación a 125.000 xg durante 45 min.
- El MSP se purifica mediante cromatografía de níquel quelante de uso de ~ 1,5 ml Ni Sepharose HP en TSG10 buffer. Los contaminantes se eliminan por lavado con TSG10 tampón que contiene 50 mM de imidazol. El MSP se eluye con tampón que contiene TSG10 600 mMimidazol. La proteína purificada se dializó en tampón TSG10 y almacenado en -70 º C a una concentración de proteína de ~ 10-15 mg / ml.
2. Preparación de la MalFGK 2 Complejo
- El MalFGK 2 compleja, su etiquetado en el extremo C-terminal de Malk, se expresa a partir del plásmido pBAD22-FGK en E. coli BL21 (DE3) y se indujo a una DO de 600 ~ 0,5 con 0,2% de L-arabinosa durante 3 horas a 37 ° C.
- Después de la lisis celular y centrifugación como en el paso 1,3, el sedimento de membrana se resuspende en tampón TSG20 a una concentración final de 5 mg / ml. El material se solubiliza con w / v 1% de n-dodecil-β-maltopiranósido (DDM) durante 3 horas a 4 ° C con agitación suave.
- El material insoluble se elimina por ultra-centrifugación como en el paso 1,3 y el sobrenadante que contiene el solubilizado 2 MalFGK complejo se recogió y se purificó como en el paso 1,4, pero sin diálisis.
- Further purificación se realizó mediante cromatografía de filtración en gel en tampón TSGD en un Superdex 200 HR 10/300 columna a un caudal de 0,5 ml / min.
3. Preparación de fosfolípidos
- Una E. lípidos totales coli extracto disuelto en cloroformo se separó en alícuotas de 1.000 nmol en tubos de microcentrífuga tornillos de cabeza. El disolvente se evaporó bajo una corriente suave de nitrógeno y se secó adicionalmente durante una noche en un desecador a vacío.
- La película de lípido se disuelve en tampón TS a una concentración de 5 nM final y vortex vigorosamente, y se sonicó. Los lípidos disueltos aparecerá ligeramente opaco debido a su insolubilidad en general en tampón TS acuoso, pero debe permanecer en suspensión. DDM se añade a una concentración final de 0,5% (~ 10 mM), en la que la solución se vuelva clara.
- La mezcla de lípidos se coloca en un baño de agua y el pulso se sonicó durante 5 veces para ~ 5 seg. La mezcla de lípidos se almacenan a 4 ° C durante un máximo de 1 week.
4. Preparación de Bio-Beads
- Aproximadamente el 10-15 ml (volumen seco) Bio-Beads se coloca en un tubo de 50 ml.
- Las perlas se lavaron sucesivamente con 50 ml de metanol al 100%, 95% de etanol, milliQ H 2 O, y finalmente tampón TS (dos veces cada uno).
- Las perlas lavadas se almacenan a 4 ° C en tampón de ~ 10 ml de TS.
5. Nanodisc Reconstitución
- El MSP se diluye en tampón TSGD a la concentración final de ~ 7 mg / ml (~ 0,3 mM).
- ~ 2 nmol de 2 purificado MalFGK complejo se mezclan a una proteína: MSP: lípido de 1:3:60 o 1:3:400 en tampón TSGD. La concentración final es de 6 micras MalFGK 2, 18 mM y lípidos MSP 360 micras (1:3:60) o 2,4 mM (lípidos 1:3:400). El volumen final es de 300 l. La concentración final de DDM es 0,08% (~ 1,6 mM). La concentración final de glicerol depende de la cantidad de lípido añadido, pero sigue siendo alrededor de 5-10% v / v
- ~ 50 l de Bio-Bead suspensión se añadió al tubo y se incuba la mezcla durante toda la noche en un agitador a 4 º C.
- Las perlas se sedimentan por gravedad y la solución se pipeteó a través de una punta estrecha para evitar en la medida Bio-Beads como sea posible.
- Grandes precipitados se separan por ultra-centrifugación a 100.000 xg durante 20 min. Los discos se purificó por cromatografía de filtración en gel como en el paso 2,4 en TSG10 tampón. Las fracciones que contenían los discos se reúnen y una alícuota se vuelve a inyectar en la columna de filtración en gel mismo para probar la estabilidad de la preparación.
- Los discos purificadas pueden almacenarse a -70 ° C durante un período prolongado de tiempo. Después de la descongelación en hielo, los discos debe ser sometido a ultra-centrifugación como en el paso 5,5 para eliminar el potencial de precipitados. Una alícuota se analizó por cromatografía de filtración en gel para garantizar que la agregación o precipitación significativa no se produjo durante la descongelaciónproceso.
6. Electroforesis en gel nativo
- 1 M de nanodiscos (~ 0,2 mg / ml) se analizan por PAGE nativa 6, 7 (Tris-HCl pH 8,8, 4-12%) para evaluar la calidad de la reconstitución (Figura 3A).
- La unión de macho (1 M) a la 2-complejo MalFGK reconstituyó en nanodiscos también es evaluado por nativo-PAGE (Figura 3A).
- Después de la electroforesis, el gel se tiñe con azul de Coomassie durante 10 min, y se decoloró durante ~ 1 hr.
7. Dispersión de luz dinámica (DLS)
- Nanodiscos se purificó por cromatografía de filtración en gel como en el paso 2,4 en TSG10 tampón utilizando un Superdex 200 HR 10/300 columna a un caudal de 0,1 ml / min. Las fracciones que contenían nanodiscos se reúnen y se concentran a ~ 10 mg / ml con un filtro Amicon centrífuga.
- La muestra se filtra dos veces (0,22 micras filtro) antes del análisis por DLS utilizandoun instrumento DynaPro NANOSTAR (Wyatt Technology) en un volumen de 1 l cubeta de cuarzo interno. Los datos se ajustaron usando el software de dinámica (Wyatt Technology) para estimar el diámetro y el peso molecular de las partículas.
8. ATPasa Medidas
- La actividad de ATPasa de MalFGK 2-reconstituidas nanodiscos se determina mediante un ensayo colorimétrico 8.
- 1 M de discos purificadas y 1 mM de ATP se mezclan con cantidades crecientes de MalE y se incubaron a 37 ° C durante 20 min. La liberación de fosfato inorgánico se midió a 660 nm.
- La cantidad de fosfato liberado se comparó con una curva estándar generada con una solución estándar de fósforo.
9. Los resultados representativos
Los nanodiscos se purificó por cromatografía de filtración en gel (Figura 2A, izquierda). El cromatograma muestra que la mayoría de la reconstituida dISCS (negro traza) se eluyen como un solo pico, mientras que los discos hechos con exceso de lípidos (curva roja) eluyen en el volumen vacío y como una serie de picos anchos. La calidad de los nanodiscos se analiza adicionalmente por electroforesis en gel nativo y espectroscopia de dispersión de luz dinámica (DLS). Reconstituidos correctamente los discos migran como una banda fuerte en el gel mientras que los reconstituida en presencia de un exceso de lípidos migrar como frotis (Figura 2A, derecha). El análisis por DLS muestra que la población disco es homogéneo con un diámetro medio de 11,4 nm (Figura 2B). Los discos reconstituidas tienen un peso molecular aparente de 215 kDa basado en la aproximación DLS. Las muestras reconstituidas en la presencia de exceso de lípidos de visualización ampliamente distribuido radios alrededor de 100 nm, que es típico de las muestras no homogéneas.
La calidad de la MalFGK 2 complejo se evaluó por electroforesis en gel nativo y su actividad por ATPasalas mediciones (Figura 3). El macho proteína de unión a maltosa se une con una afinidad alta a la MalFGK 2 transportador 9, 10. Utilizando electroforesis no desnaturalizante en gel, es posible detectar un complejo entre el macho y MalFGK 2 (Figura 3A). La estimulación de la actividad de la ATPasa MÄLK 2 por MalE se muestra en la Figura 3B.
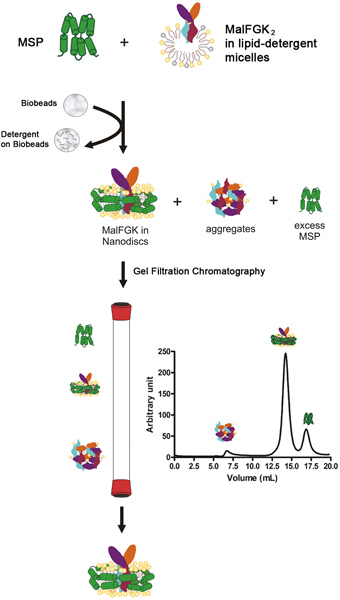
Figura 1. Diagrama de flujo típico para el protocolo de reconstitución.
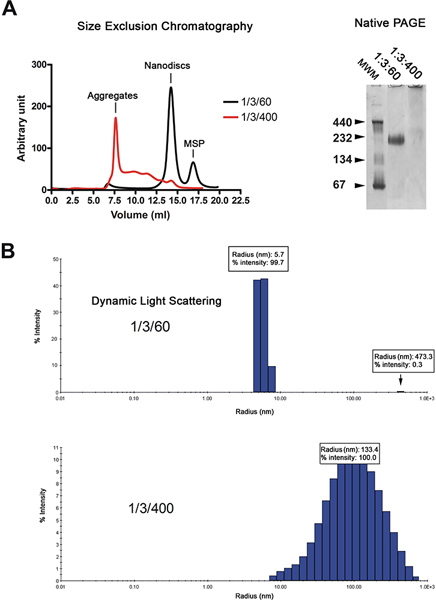
Figura 2. Control de calidad de la preparación nanodisc. A. Análisis de filtración en gel (Superdex 200 HR 10 / columna de 300) de los nanodiscos reconstituido en relación baja en lípidos (01/03/60; trazo negro) o la relación de lípido alta (1/3/400; trazo rojo). Electroforesis en gel nativo de la preparación del disco mismo. Molecular marcadores de peso en kDa se indican. B. La luz dinámica de análisis de dispersión de la preparación de un mismo disco. Haga clic aquí para ampliar la cifra .

Figura 3. Análisis de la MalFGK 2-nanodisc partículas. A. Análisis Gel cambio de MalE incubaron con cantidades crecientes de MalFGK 2 nanodisc partículas. B. actividad ATPasa de la MalFGK 2-nanodisc partículas como una función de la concentración masculina.
Discusión
Se describe un procedimiento sencillo para la reconstitución del transportador de maltosa en nanodiscos. El transportador es ATPasa activa y la interacción con el macho pareja de unión soluble puede ser recreada (Figura 3). La reconstitución exitosa del transportador en nanodiscos abrir el camino para un análisis adicional biofísica y bioquímica. De particular interés será el análisis sistemático de la ATPasa y la actividad Malk maltosa transporte en el detergente, liposomas y nanodiscos. Tra...
Divulgaciones
No hay conflictos de interés declarado.
Agradecimientos
Este trabajo fue apoyado por el Instituto Canadiense de Investigación en Salud. CSC fue financiado por una beca postdoctoral de las Ciencias Naturales e Ingeniería de Investigación de Canadá. FD es un Tier II Cátedra de Investigación de Canadá.
Materiales
| Name | Company | Catalog Number | Comments | ||||||||||||||||||||||||||||||
| Nombre del reactivo | Empresa | Número de catálogo | Comentarios | ||||||||||||||||||||||||||||||
| Amicon Ultra-4 50K filtro centrífugo | Millipore | UFC805008 | Seguir el protocolo del fabricante para el uso apropiado | ||||||||||||||||||||||||||||||
| Bio-Beads SM-2 Adsorbente | Bio-Rad | 152-3920 | |||||||||||||||||||||||||||||||
| E. coli lípidos totales | Avanti Polar Lipids | 100500C | Disuelto en cloroformo, según sea apropiado para manejar un disolvente orgánico | ||||||||||||||||||||||||||||||
| Ni Sepharose HP resina | GE Healthcare | 17-5268-01 | |||||||||||||||||||||||||||||||
| Fósforo solución estándar | Sigma-Aldrich | P3869 | |||||||||||||||||||||||||||||||
| pMSP1D1 | Addgene | 20061 | |||||||||||||||||||||||||||||||
| Superdex 200 HR 10/300 | GE Healthcare | 17-5172-01 | |||||||||||||||||||||||||||||||
| Tabla I. Los reactivos específicos. | |||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||
Referencias
- Denisov, I. G., Ginkova, Y. V., Lazarides, A. A., Sligar, S. G. Directed self-assembly of monodisperse phospholipid bilayer Nanodiscs with controlled size. J. Am. Chem. Soc. 126, 3477-3487 (2004).
- Boldog, T., Grimme, S., Li, M., Sligar, S. G., Hazelbauer, G. L. Nanodiscs separate chemoreceptor oligomeric states and reveal their signaling properties. Proc. Natl. Acad. Sci. USA. 103, 11509-11514 (2006).
- Bass, B. J., Denisov, I. G., Sligar, S. G. Homotropic cooperativity of monomeric cytochrome P450 3A4 in a nanoscale native bilayer environment. J. Biol. Chem. 282, 7066-7076 (2007).
- Alami, M., Dalal, K., Lelj-Garolla, B., Sligar, S. G., Duong, F. Nanodiscs unravel the interaction between the SecYEG channel and its cytosolic partner SecA. EMBO J. 26, 1995-2004 (2007).
- Mi, L. -. Z., Grey, M. J., Nishida, N., Walz, T., Lu, C., Springer, T. A. Functional and structural stability of the epidermal growth factor receptor in detergent micelles and phospholipid nanodiscs. Biochemistry. 47, 10314-10323 (2008).
- Schägger, H., Cramer, W. A., von Jagow, G. Analysis of molecular masses and oligomeric states of protein complexes by blue native electrophoresis and isolation of membrane protein complexes by two-dimensional native electrophoresis. Anal. Biochem. 217, 220-230 (1994).
- Dalal, K., Duong, F. Reconstitution of the SecY translocon in Nanodiscs. Methods Mol. Biol. 619, 145-156 (2010).
- Lanzetta, P. A., Alvarez, L. J., Reinach, P. S., Candia, O. A. An improved assay for nanomole amounts of inorganic phosphate. Anal. Biochem. 100, 95-97 (1979).
- Davidson, A. L., Dassa, E., Orelle, C., Chen, J. Structure, function and evolution of bacterial ATP-binding cassette systems. Microbiol. Mol. Biol. Rev. 72, 317-364 (2008).
- Bordignon, E., Grote, M., Schneider, E. The maltose ATP-binding cassette transporter in the 21st century-towards a structural dynamic perspective on its mode of action. Mol. Microbiol. 77, 1354-1366 (2010).
- Alvarez, F. J., Orelle, C., Davidson, A. L. Functional reconstitution of an ABC transporter for use in electron paramagnetic resonance spectroscopy. J. Am. Chem. Soc. 132, 9513-9515 (2010).
- Ritchie, T. K., Grinkova, Y. V., Bayburt, T. H., Denisov, I. G., Zolnerciks, J. K., Atkins, W. M., Sligar, S. G. Reconstitution of membrane proteins in phospholipid bilayer Nanodiscs. Methods Enzymol. 464, 211-231 (2009).
- Glück, J. M., Koenig, B. W., Willbold, D. Nanodiscs allow the use of integral membrane proteins as analytes in surface plasmon resonance studies. Anal. Biochem. 408, 46-52 (2011).
- Wan, C. -. P. L., Chiu, M. H., Wu, X., Lee, S. K., Prenner, E. J., Weers, P. M. M. Apolipoprotein-induced conversion of phosphatidylcholine bilayer vesicles into nanodisks. Biochim. Biophys. Acta (BBA). 1808, 606-613 (2011).
- Nath, A., Trexler, A. J., Koo, P. K., Miranker, A. D., Atkins, W. M., Rhoades, E. Single-molecule fluorescence spectroscopy using phospholipid bilayer Nanodiscs. Methods Enzymol. 472, 89-117 (2010).
- Denisov, I. G., Sligar, S. G. Cytochromes P450 in Nanodiscs. Biochim. Biophys. Acta. 1814, 223-229 (2011).
- Zhang, X. X., Chan, C. S., Bao, H., Fang, Y., Foster, L. J., Duong, F. Nanodiscs and SILAC-based mass spectrometry to identify a membrane protein interactome. J. Proteome Res. , (2011).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados