Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Une méthode étape par étape pour la reconstitution d'un transporteur ABC dans des particules lipidiques Nanodisc
Dans cet article
Résumé
NANODISQUES sont de petites particules discoïdes qui incorporent des protéines membranaires dans un petit lopin de bicouche phospholipidique. Nous fournir un protocole visuelle montre que l'incorporation étape par étape du transporteur MalFGK2 dans un disque.
Résumé
Le nanodisc est une particule en forme de disque (~ 10-12 nm grande) que les protéines membranaires piège dans une petite parcelle de bicouche phospholipidique. Le nanodisc est une option particulièrement intéressante pour l'étude des protéines membranaires, en particulier dans le contexte de interactions ligand-récepteur. Le procédé mis au point par Sligar et ses collaborateurs est basée sur les propriétés amphipathiques d'une ingénierie très une hélice protéine extraite de l'échafaudage apolipoprotéines A1. Les faces hydrophobes de la protéine d'échafaudage interagir avec les acyles gras des chaînes latérales de la bicouche lipidique, alors que les régions polaires face à l'environnement aqueux. L'analyse des protéines membranaires ont NANODISQUES avantages significatifs par rapport liposome, car les particules sont petites, homogène et soluble dans l'eau. En outre, les méthodes biochimiques et biophysiques normalement réservés à des protéines solubles peuvent être appliqués, et de part et d'autre de la membrane. Dans ce protocole visuelle, nous présentons une reconstitution étape par étape, d'un caractère bientérisée transporteur ABC bactérien, le mâle-MalFGK complexe 2. La formation du disque est un processus d'auto-assemblage qui dépend des interactions hydrophobes qui ont lieu au cours de l'élimination progressive du détergent. Nous décrivons les étapes essentielles et nous soulignons l'importance de choisir un bon protéine-lipide rapport afin de limiter la formation d'agrégats et les grands polydisperses liposomes comme des particules. Contrôle qualité simples telles que la chromatographie de filtration sur gel, l'électrophorèse sur gel natif et dynamique spectroscopie de diffusion de lumière en sorte que les disques ont été correctement reconstitué.
Protocole
Processus de reconstitution globale
Le processus de reconstitution commence par mélanger la protéine d'échafaudage membranaire (MSP) avec le complexe purifié MalFGK 2 en présence d'un détergent-solubilisées phospholipides. L'étape est suivie de l'élimination lente de la lessive par un matériau adsorbant en polystyrène appelé Bio-Beads ou Amberlite (figure 1). Le processus d'auto-assemblage se produit probablement en raison des interactions entre les phospholipides apolaires hydrophobes, le 2 MalFGK complexes et la surface de la protéine MSP amphipathique. Le produit final est une particule discoïde composé de deux molécules d'emballage MSP à travers le MalFGK complexe 2. Les particules sont séparées des produits d'addition et d'agrégats par ultra-centrifugation analytique et chromatographie d'exclusion stérique. Les particules sont caractérisées par une électrophorèse sur gel natif et dynamique spectroscopie de diffusion de lumière.
titre "> 1. Préparation de la membrane des protéines d'échafaudage, MSP- Le son MSP-marqué (version MSP1D1 1) est produit à partir du plasmide dans E. pMSP1D1 coli BL21 (DE3) induite à DO 600 ~ 0,5 avec 0,5 mM d'isopropyl β-D-1-thiogalactopyranoside pendant 3 heures à 37 ° C.
- Les cellules sont récoltées par centrifugation à 5000 xg pendant 10 min à 4 ° C et remises en suspension dans TSG10 tampon contenant 100 uM fluorure de phénylméthanesulfonyle.
- Les cellules sont lysées avec une presse française (3x) à 8.000 livres par pouce carré et l'insoluble est éliminé par centrifugation à 5000 xg pendant 10 min à 4 ° C. La fraction soluble contenant la MSP est isolé par ultracentrifugation à 125.000 g pendant 45 min.
- Le MSP est purifié par chromatographie de chélation nickel en utilisant ~ 1,5 ml Ni Sepharose HP TSG10 tampon. Contaminants sont éliminés par lavage avec TSG10 tampon contenant 50 mM d'imidazole. Le MSP est éluée avec un tampon contenant TSG10 600 mMimidazole. La protéine purifiée est dialysée dans un tampon TSG10 et stocké dans -70 ° C à une concentration en protéine de ~ 10-15 mg / ml.
2. Préparation de la MalFGK 2 Complexe
- Le 2 MalFGK complexe, sa-marquées à l'extrémité C-terminale de MalK, est exprimée à partir du plasmide pBAD22-FGK dans E. coli BL21 (DE3) et induit à DO 600 ~ 0,5 avec 0,2% de L-arabinose pendant 3 heures à 37 ° C.
- Suite à la lyse des cellules et centrifugation comme dans l'étape 1,3, le culot est remis en suspension dans la membrane TSG20 tampon à une concentration finale de 5 mg / ml. Le matériau est solubilisé à 1% p / v de n-dodécyl-β-maltopyranoside (DDM) pendant 3 heures à 4 ° C sous agitation douce.
- L'insoluble est éliminé par ultra-centrifugation comme à l'étape 1.3 et le surnageant contenant le solubilisée 2 MalFGK complexe est recueilli et purifié comme à l'étape 1,4, mais sans dialyse.
- Further purification est réalisée par chromatographie par filtration sur gel dans un tampon TSGD sur une colonne Superdex 200 HR 10/300 de colonne à un débit de 0,5 ml / min.
3. Préparation des phospholipides
- Un E. coli lipidique totale extraire dissous dans du chloroforme est séparée en 1.000 nmol aliquotes dans des tubes à centrifuger de vis d'assemblage. Le solvant est évaporé sous un léger courant d'azote et séché encore pendant une nuit dans un dessiccateur sous vide.
- Le film lipidique est dissoute dans un tampon TS à une concentration de 5 nM final et vortexé vigoureusement et ultrasons. Les lipides dissous apparaîtra légèrement opaque en raison de leur insolubilité générale dans le tampon TS aqueuse, mais ils doivent rester en suspension. DDM est ajouté à une concentration finale de 0,5% (~ 10 mM), à laquelle la solution devient claire.
- Le mélange de lipides est placé dans un bain d'eau et d'impulsions ultrasons pendant 5 heures à ~ 5 sec. Le mélange de lipides est stocké à 4 ° C pendant un maximum de 1 week.
4. Préparation de Bio-Beads
- Environ 10-15 ml (volume sec) Bio-Beads est placé dans un tube de 50 ml.
- Les perles sont lavées successivement avec 50 ml de méthanol à 100%, éthanol à 95%, milliQ H 2 O, et enfin tampon TS (deux fois).
- Les billes lavées sont conservés à 4 ° C dans 10 ml de tampon ~ TS.
5. Reconstitution Nanodisc
- Le MSP est dilué dans du tampon TSGD à la concentration finale d'environ 7 mg / ml (~ 0,3 mM).
- ~ 2 nmol de 2 purifié MalFGK complexe sont mélangés à une protéine: PVC: lipide rapport 1:3:60 ou dans un tampon TSGD 1:3:400. La concentration finale est de 6 uM MalFGK 2, 18 uM MSP et les lipides pM 360 (1:3:60) ou 2,4 mM de lipides (1:3:400). Le volume final est de 300 ul. La concentration finale est DDM 0,08% (~ 1,6 mM). La concentration finale en glycérol dépend de la quantité de lipide ajouté mais reste autour de 5-10% v / v
- ~ 50 pi de Bio-perle suspension est ajouté dans le tube et le mélange est incubé pendant une nuit sur une table à bascule à 4 ° C.
- Les billes sont sédimentées par gravité et la solution est introduit à la pipette à travers une pointe étroite à éviter autant Bio-Beads que possible.
- Gros précipités sont éliminés par ultra-centrifugation à 100.000 xg pendant 20 min. Les disques sont purifiées par chromatographie par filtration sur gel comme à l'étape de 2,4 TSG10 tampon. Les fractions contenant les disques sont mis en commun et une partie aliquote doit être réinjecté dans la colonne de filtration sur gel même de vérifier la stabilité de la préparation.
- Les disques purifiés peuvent stocké à -70 ° C pendant une période de temps prolongée. Après le dégel de la glace, les disques doivent être soumis à l'ultra-centrifugation comme à l'étape 5.5 à retirer potentiel précipités. Une aliquote doit être analysé par chromatographie de filtration sur gel afin de s'assurer que l'agrégation ou une précipitation importante n'a pas eu lieu lors de la décongélationprocessus.
6. Électrophorèse sur gel natif
- 1 uM de NANODISQUES (~ 0,2 mg / ml) sont analysés par PAGE native 6, 7 (Tris-HCl pH 8,8, 4-12%) pour évaluer la qualité de la reconstitution (figure 3A).
- La liaison de l'Homme (1 uM) à la 2-complexe MalFGK reconstitué dans NANODISQUES est également évalué en natif-PAGE (figure 3A).
- Après électrophorèse, le gel est coloré au bleu de Coomassie pendant 10 min, et décolorés pendant ~ 1 heure.
7. Diffusion de la lumière dynamique (DLS)
- NANODISQUES sont purifiées par chromatographie par filtration sur gel comme dans l'étape 2,4 à l'aide d'un tampon TSG10 Superdex 200 HR 10/300 de colonne à un débit de 0,1 ml / min. Les fractions contenant NANODISQUES sont réunies et concentrées à environ 10 mg / ml avec un filtre Amicon centrifuge.
- L'échantillon est filtré deux fois (0,22 um filtre) avant l'analyse par DLS utilisantun DynaPro nanostar instrument (Wyatt Technology) dans un volume intérieur 1 pl cuvette en quartz. Les données s'effectue à l'aide du logiciel DYNAMIQUE (Wyatt Technology) pour estimer le diamètre et le poids moléculaire des particules.
8. Mesures ATPase
- L'activité ATPase de MalFGK 2-reconstituées NANODISQUES est déterminée en utilisant un dosage colorimétrique 8.
- 1 uM de disques purifiés et 1 mM d'ATP sont mélangés avec des quantités croissantes de MalE et incubées à 37 ° C pendant 20 minutes. La libération de phosphate inorganique est mesurée à 660 nm.
- La quantité de phosphate libéré est comparée à une courbe d'étalonnage générée avec une solution standard de phosphore.
9. Les résultats représentatifs
Les NANODISQUES sont purifiés par chromatographie de filtration sur gel (Figure 2A, à gauche). Le chromatogramme montre que la majorité de la reconstitution dCSI (trace noire) éluer forme d'un pic unique, tandis que les disques à base de lipides en excès (rouge trace) éluent dans le volume de vide et comme une série de pics larges. La qualité des NANODISQUES est ensuite analysée par électrophorèse sur gel natif et dynamique spectroscopie diffusion de la lumière (DLS). Disques correctement reconstitué migrer comme une bande nette sur le gel alors que ceux reconstitué en présence d'un excès de lipides comme un test de migration (figure 2A, à droite). Analyse par DLS montre que la population est homogène disque avec un diamètre moyen de 11,4 nm (Figure 2B). Les disques reconstituées ont un poids moléculaire apparent de 215 kDa sur la base de l'approximation DLS. Les échantillons reconstitués en présence d'excès de lipides affichage largement distribué rayons autour de 100 nm, ce qui est typique pour les échantillons non homogènes.
La qualité de la MalFGK complexe 2 est évaluée par électrophorèse sur gel natif et son activité ATPase parmesures (figure 3). Le mâle protéine liant le maltose se lie avec une haute affinité pour le transporteur 2 MalFGK 9, 10. Utilisation non électrophorèse sur gel dénaturant, il est possible de détecter un complexe entre l'homme et MalFGK 2 (figure 3A). La stimulation de l'activité ATPase MalK 2 par mâle est montré sur la figure 3B.
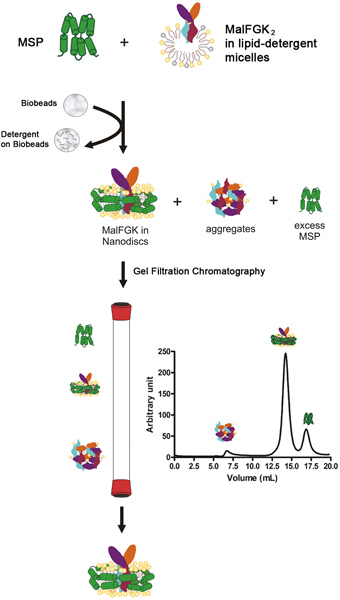
Figure 1. Organigramme type pour le protocole de reconstitution.
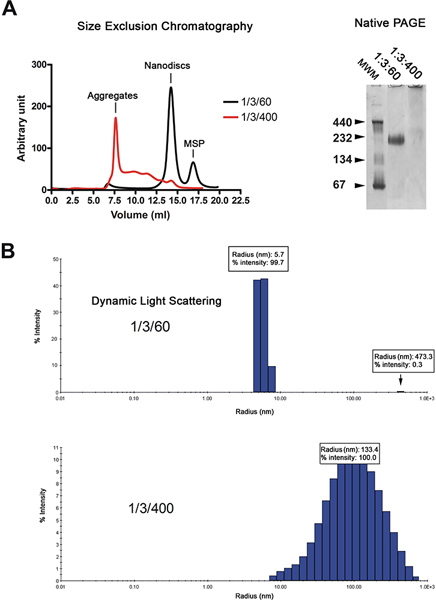
Figure 2. Contrôle de la qualité de la préparation nanodisc. A. Analyse par filtration sur gel (Superdex 200 HR 10/300 colonnes) des NANODISQUES reconstitué à lipide faible (03/01/60; trace noire) ou le taux de lipides élevé (1/3/400; trace rouge). Électrophorèse sur gel natif de la préparation même disque. Molmarqueurs de poids ecular à kDa sont indiquées. B. Analyse dynamique diffusion de la lumière de la préparation même disque. Cliquez ici pour agrandir la figure .

Figure 3. L'analyse de la MalFGK 2-nanodisc particules. A. L'analyse de retard sur gel de MalE incubés avec des quantités croissantes de MalFGK 2-nanodisc particules. B. ATPase de la MalFGK 2-nanodisc particules en fonction de la concentration de sexe masculin.
Discussion
Nous décrivons une procédure simple pour la reconstitution du transporteur maltose en NANODISQUES. Le transporteur est ATPase active et l'interaction avec le mâle partenaire de liaison soluble peut être recréé (figure 3). La reconstitution réussie du transporteur en NANODISQUES ouvrir la voie à l'analyse biophysique et biochimique supplémentaire. D'un intérêt particulier sera l'analyse systématique de l'ATPase MalK et de l'activité de transport du maltose dans du dé...
Déclarations de divulgation
Aucun conflit d'intérêt déclaré.
Remerciements
Ce travail a été soutenu par l'Institut canadien de recherche en santé. SCC a été financé par une bourse de recherche postdoctorale en sciences naturelles et en génie du Canada. FD est une chaire de recherche du Canada.
matériels
| Name | Company | Catalog Number | Comments | ||||||||||||||||||||||||||||||
| Nom du réactif | Entreprise | Numéro de catalogue | Commentaires | ||||||||||||||||||||||||||||||
| Amicon Ultra-4 50K filtre centrifuge | Millipore | UFC805008 | Suivre le protocole du fabricant pour une utilisation correcte | ||||||||||||||||||||||||||||||
| Bio-Beads SM-2 Adsorbant | Bio-Rad | 152-3920 | |||||||||||||||||||||||||||||||
| E. coli lipides totaux | Avanti Polar Lipids | 100500C | Dissous dans du chloroforme, gérer comme il convient pour un solvant organique | ||||||||||||||||||||||||||||||
| Ni Sepharose HP résine | GE Healthcare | 17-5268-01 | |||||||||||||||||||||||||||||||
| Solution étalon de phosphore | Sigma-Aldrich | P3869 | |||||||||||||||||||||||||||||||
| pMSP1D1 | Addgene | 20061 | |||||||||||||||||||||||||||||||
| Superdex 200 HR 10/300 | GE Healthcare | 17-5172-01 | |||||||||||||||||||||||||||||||
| Tableau I. réactifs spécifiques. | |||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||
Références
- Denisov, I. G., Ginkova, Y. V., Lazarides, A. A., Sligar, S. G. Directed self-assembly of monodisperse phospholipid bilayer Nanodiscs with controlled size. J. Am. Chem. Soc. 126, 3477-3487 (2004).
- Boldog, T., Grimme, S., Li, M., Sligar, S. G., Hazelbauer, G. L. Nanodiscs separate chemoreceptor oligomeric states and reveal their signaling properties. Proc. Natl. Acad. Sci. USA. 103, 11509-11514 (2006).
- Bass, B. J., Denisov, I. G., Sligar, S. G. Homotropic cooperativity of monomeric cytochrome P450 3A4 in a nanoscale native bilayer environment. J. Biol. Chem. 282, 7066-7076 (2007).
- Alami, M., Dalal, K., Lelj-Garolla, B., Sligar, S. G., Duong, F. Nanodiscs unravel the interaction between the SecYEG channel and its cytosolic partner SecA. EMBO J. 26, 1995-2004 (2007).
- Mi, L. -. Z., Grey, M. J., Nishida, N., Walz, T., Lu, C., Springer, T. A. Functional and structural stability of the epidermal growth factor receptor in detergent micelles and phospholipid nanodiscs. Biochemistry. 47, 10314-10323 (2008).
- Schägger, H., Cramer, W. A., von Jagow, G. Analysis of molecular masses and oligomeric states of protein complexes by blue native electrophoresis and isolation of membrane protein complexes by two-dimensional native electrophoresis. Anal. Biochem. 217, 220-230 (1994).
- Dalal, K., Duong, F. Reconstitution of the SecY translocon in Nanodiscs. Methods Mol. Biol. 619, 145-156 (2010).
- Lanzetta, P. A., Alvarez, L. J., Reinach, P. S., Candia, O. A. An improved assay for nanomole amounts of inorganic phosphate. Anal. Biochem. 100, 95-97 (1979).
- Davidson, A. L., Dassa, E., Orelle, C., Chen, J. Structure, function and evolution of bacterial ATP-binding cassette systems. Microbiol. Mol. Biol. Rev. 72, 317-364 (2008).
- Bordignon, E., Grote, M., Schneider, E. The maltose ATP-binding cassette transporter in the 21st century-towards a structural dynamic perspective on its mode of action. Mol. Microbiol. 77, 1354-1366 (2010).
- Alvarez, F. J., Orelle, C., Davidson, A. L. Functional reconstitution of an ABC transporter for use in electron paramagnetic resonance spectroscopy. J. Am. Chem. Soc. 132, 9513-9515 (2010).
- Ritchie, T. K., Grinkova, Y. V., Bayburt, T. H., Denisov, I. G., Zolnerciks, J. K., Atkins, W. M., Sligar, S. G. Reconstitution of membrane proteins in phospholipid bilayer Nanodiscs. Methods Enzymol. 464, 211-231 (2009).
- Glück, J. M., Koenig, B. W., Willbold, D. Nanodiscs allow the use of integral membrane proteins as analytes in surface plasmon resonance studies. Anal. Biochem. 408, 46-52 (2011).
- Wan, C. -. P. L., Chiu, M. H., Wu, X., Lee, S. K., Prenner, E. J., Weers, P. M. M. Apolipoprotein-induced conversion of phosphatidylcholine bilayer vesicles into nanodisks. Biochim. Biophys. Acta (BBA). 1808, 606-613 (2011).
- Nath, A., Trexler, A. J., Koo, P. K., Miranker, A. D., Atkins, W. M., Rhoades, E. Single-molecule fluorescence spectroscopy using phospholipid bilayer Nanodiscs. Methods Enzymol. 472, 89-117 (2010).
- Denisov, I. G., Sligar, S. G. Cytochromes P450 in Nanodiscs. Biochim. Biophys. Acta. 1814, 223-229 (2011).
- Zhang, X. X., Chan, C. S., Bao, H., Fang, Y., Foster, L. J., Duong, F. Nanodiscs and SILAC-based mass spectrometry to identify a membrane protein interactome. J. Proteome Res. , (2011).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon