È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
A Step-by-step Metodo per la ricostituzione di un Transporter ABC in particelle lipidiche Nanodisc
In questo articolo
Riepilogo
Nanodiscs sono piccole particelle discoidali che incorporano proteine di membrana in una piccola zona di doppio strato di fosfolipidi. Forniamo un protocollo visivo che mostra il passo-passo incorporazione del trasportatore MalFGK2 in un disco.
Abstract
Il nanodisc è una particella discoidale (~ 10-12 nm di grandi dimensioni) che le proteine di membrana trappola in una piccola zona di doppio strato di fosfolipidi. Il nanodisc è un'opzione particolarmente interessante per studiare proteine di membrana, in particolare nel contesto delle interazioni ligando-recettore. Il metodo introdotto da Sligar e colleghi si basa sulle proprietà di un anfipatiche altamente ingegnerizzato ad a-elica proteina scaffold derivato dal apolipoproteina A1. Le facce idrofobe della proteina scaffold interagire con i grassi acil catene laterali del doppio strato lipidico che le regioni polari affrontare l'ambiente acquoso. Analisi di proteine di membrana in nanodiscs hanno vantaggi significativi rispetto liposoma perché le particelle sono piccole, omogenea e idrosolubile. Inoltre, metodi biochimici e biofisici normalmente riservati alle proteine solubili possono essere applicati, e da entrambi i lati della membrana. In questo protocollo visiva, presentiamo un passo-passo ricostituzione di un carattere benterizzato trasportatore batterica ABC, il maschio-MalFGK 2 complessi. La formazione del disco è un processo di auto-assemblaggio che dipende da interazioni idrofobiche che avvengono durante la rimozione progressiva del detersivo. Si descrivono le fasi essenziali e sottolineano l'importanza di una corretta scelta proteina-lipide rapporto al fine di limitare la formazione di aggregati e grandi polidispersi liposoma-come particelle. Qualità semplici controlli, come la cromatografia di gel filtrazione, elettroforesi su gel nativo e dinamico spettroscopia di dispersione della luce in modo che i dischi sono stati adeguatamente ricostituito.
Protocollo
Ricostituzione del processo globale
Il processo inizia ricostituzione miscelando la membrana proteina scaffold (MSP) con il complesso purificato MalFGK 2 in presenza di detergente-solubilizzate fosfolipidi. La fase è seguita dalla rimozione lenta del detergente da un materiale adsorbente polistirene chiamato Bio-Beads o Amberlite (Figura 1). L'auto-assemblaggio processo si verifica molto probabilmente a causa delle interazioni tra i fosfolipidi apolari idrofobici, la MalFGK 2 complessi e la superficie della proteina MSP anfipatica. Il prodotto finale è una particella discoide fatta di due molecole di MSP incarto attorno al complesso 2 MalFGK. Le particelle vengono separate dalle addotti e aggregati di ultra-centrifugazione analitica e cromatografia ad esclusione dimensionale. Le particelle sono caratterizzate da native-gel elettroforesi e spettroscopia dispersione della luce dinamica.
titolo "> 1. Preparazione della membrana impalcatura proteica, MSP- Il suo tag-MSP (versione MSP1D1 1) è prodotto da pMSP1D1 plasmidi in E. coli BL21 (DE3) indotta a OD 600 ~ 0,5 con 0,5 mM isopropil β-D-1-tiogalattopiranoside per 3 ore a 37 ° C.
- Le cellule vengono raccolte mediante centrifugazione a 5000 xg per 10 min a 4 ° C e risospese in tampone contenente 100 TSG10 fluoruro phenylmethanesulfonyl pM.
- Le cellule vengono lisate con una pressa French (3x) a 8000 psi e il materiale insolubile è rimosso per centrifugazione a 5000 xg per 10 min a 4 ° C. La frazione solubile contenente l'MSP è isolato da ultra-centrifugazione a 125.000 xg per 45 min.
- La MSP è purificato dal nichel-chelante cromatografia usando ~ 1,5 ml Ni Sepharose HP TSG10 buffer. Contaminanti vengono lavati via con TSG10 tampone contenente 50 mM imidazolo. La MSP è eluita con TSG10 tampone contenente 600 mMimidazolo. La proteina purificata è dializzato in TSG10 tampone e conservati in -70 ° C ad una concentrazione proteica di ~ 10-15 mg / ml.
2. Preparazione del Complex 2 MalFGK
- Il complesso MalFGK 2, la-etichettato al C-terminale della MalK, viene espressa dal plasmide pBAD22-FGK in E. coli BL21 (DE3) e indotto a OD 600 ~ 0,5 con 0,2% di L-arabinosio per 3 ore a 37 ° C.
- Seguendo la lisi cellulare e centrifugazione come nel passo 1,3, il pellet viene risospeso in membrana TSG20 tampone ad una concentrazione finale di 5 mg / ml. Il materiale viene solubilizzato con 1% w / v n-dodecil-β-maltopyranoside (DDM) per 3 ore a 4 ° C con agitazione delicata.
- Il materiale insolubile è rimosso per centrifugazione ultra-come nel passo 1.3 e il surnatante contenente il solubilizzata MalFGK 2 complesso viene raccolto e purificato come nel passo 1.4, ma senza dialisi.
- Inopurificazione r è ottenuta mediante cromatografia di filtrazione su gel in tampone TSGD su un Superdex 200 HR 10/300 colonna ad una velocità di flusso di 0,5 ml / min.
3. Preparazione di fosfolipidi
- Un E. lipidi coli totale estratto sciolto in cloroformio è separato in 1000 nmol aliquote in provette con tappo a vite microcentrifuga. Il solvente viene evaporato sotto leggera corrente di azoto ed essiccato ulteriormente durante la notte in un essiccatore sotto vuoto.
- Il film lipidico viene disciolto in tampone TS a 5 nM concentrazione finale e vortexati vigorosamente e sonicata. I lipidi disciolti appariranno leggermente opaco a causa della loro insolubilità generale in tampone acquoso TS, ma devono rimanere in sospensione. DDM viene aggiunto ad una concentrazione finale di 0,5% (~ 10 mM), in cui la soluzione diventa limpida.
- La miscela lipidica è posto in un bagno di acqua e impulsi sonicata per 5 volte per ~ 5 sec. La miscela lipidica è conservato a 4 ° C per un massimo di 1 week.
4. Preparazione di Bio-Beads
- Circa 10-15 ml (volume secco) Bio-Beads viene posto in una provetta da 50 ml.
- Le perle vengono successivamente lavato con 50 ml di metanolo al 100%, 95% etanolo, MilliQ H 2 O, e infine tampone TS (due volte).
- Le perle vengono lavate conservato a 4 ° C in 10 ml di tampone ~ TS.
5. Nanodisc Ricostituzione
- La MSP è diluito in tampone TSGD alla concentrazione finale di ~ 7 mg / ml (~ 0,3 mM).
- ~ 2 nmol purificato MalFGK due complessi sono miscelati in una proteina: MSP: rapporto di 1:3:60 lipidi o 1:3:400 in tampone TSGD. La concentrazione finale è di 6 mM MalFGK 2, 18 MSP pM e lipidi pM 360 (1:3:60) e 2.4 mM di lipidi (1:3:400). Il volume finale è 300 ml. La concentrazione DDM finale è 0,08% (~ 1,6 mm). La concentrazione finale di glicerolo dipende dalla quantità di lipidi aggiunto ma rimane circa 5-10% v / v
- ~ 50 pl di Bio-Bead sospensione viene aggiunto alla provetta e la miscela è incubata per una notte su una tavola oscillante a 4 ° C.
- Le perle vengono sedimentate per gravità e la soluzione viene pipettato attraverso una punta stretta per evitare il più Bio-Beads possibile.
- Grande precipitati vengono rimossi dalla ultra-centrifugazione a 100.000 xg per 20 min. I dischi sono purificati mediante cromatografia di gel filtrazione, come nel passo 2.4 in TSG10 buffer. Frazioni contenenti i dischi vengono raggruppati ed una aliquota deve essere reimmesso sulla stessa colonna di filtrazione su gel per verificare la stabilità del preparato.
- I dischi purificate possono conservati a -70 ° C per un periodo prolungato di tempo. Dopo lo scongelamento in ghiaccio, i dischi devono essere sottoposti ad ultra-centrifugazione come nel passaggio 5,5 per rimuovere potenziale precipitati. Una aliquota deve essere analizzata mediante cromatografia di gel filtrazione per garantire che l'aggregazione significativa o precipitazione non verificarsi durante lo scongelamentoprocesso.
6. Elettroforesi su gel nativo
- 1 pM di nanodiscs (~ 0,2 mg / ml) sono analizzati mediante PAGE nativa 6, 7 (Tris-HCl pH 8,8, 4-12%) per valutare la qualità della ricostituzione (Figura 3A).
- Il legame di maschio (1 mM) al 2-complesso MalFGK ricostituito in nanodiscs è anche valutata nativo-PAGE (Figura 3A).
- Dopo l'elettroforesi, il gel viene colorato con blu Coomassie per 10 min, e decolorato per ~ 1 ora.
7. Light Scattering Dinamico (DLS)
- Nanodiscs vengono purificati mediante cromatografia di gel filtrazione, come al punto 2.4 in TSG10 tampone usando un Superdex 200 HR 10/300 colonna ad una velocità di flusso di 0,1 ml / min. Frazioni contenenti nanodiscs sono riunite e concentrate a ~ 10 mg / ml con un filtro Amicon centrifugo.
- Il campione viene filtrato due volte (0,22 micron filtro) prima dell'analisi da DLS utilizzanoun DYNAPRO nanostar strumento (Wyatt Technology) in un volume di 1 cuvetta microlitri interna quarzo. Dati è montato mediante il software DYNAMICS (Wyatt Technology) per stimare il diametro e peso molecolare delle particelle.
8. ATPasi Misure
- L'attività di ATPasi MalFGK 2-ricostituite nanodiscs viene determinato utilizzando un saggio colorimetrico 8.
- 1 pM di dischi purificati e 1 mM ATP si mescolano con quantità crescenti di sesso maschile e incubate a 37 ° C per 20 min. Il rilascio di fosfato inorganico è misurata a 660 nm.
- La quantità di fosfato liberato viene confrontata con una curva standard generata con una soluzione standard Fosforo.
9. Risultati rappresentativi
Le nanodiscs vengono purificati mediante cromatografia di filtrazione su gel (Figura 2A, a sinistra). Il cromatogramma mostra che la maggior parte del ricostituito dCSI (traccia nera) eluire come picco singolo, mentre i dischi realizzati con lipidi in eccesso (traccia rossa) eluiti nel volume di vuoto e da una serie di picchi di massima. La qualità dei nanodiscs è ulteriormente analizzati mediante elettroforesi su gel nativo e spettroscopia light scattering dinamico (DLS). Dischi correttamente ricostituite migrare come banda tagliente sul gel mentre quelli ricostituito in presenza di un eccesso di lipidi migrare come striscio (Figura 2A, destra). Analisi per DLS mostra che la popolazione disco è omogenea con un diametro medio di 11,4 nm (Figura 2B). I dischi ricostituiti hanno un peso molecolare apparente di 215 kDa basato sul ravvicinamento DLS. Campioni ricostituiti in presenza di eccesso di lipidi visualizzano ampiamente distribuiti raggi circa 100 nm, che è tipico per campioni non omogenei.
La qualità del complesso 2 MalFGK viene valutata native-gel elettroforesi e la sua attività da ATPasimisure (Figura 3). Il maschio maltosio legame con le proteine si lega con alta affinità per il trasportatore 2 MalFGK 9, 10. Utilizzo non denaturante elettroforesi su gel, è possibile rilevare un complesso tra maschio e MalFGK 2 (Figura 3A). La stimolazione della attività 2 MalK ATPasi da Male è illustrato nella figura 3B.
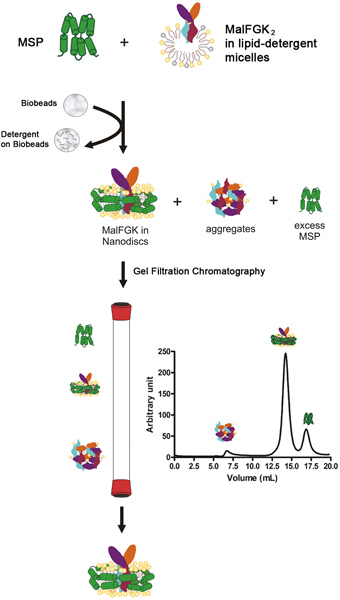
Figura 1. Tipico diagramma di flusso per il protocollo ricostituzione.
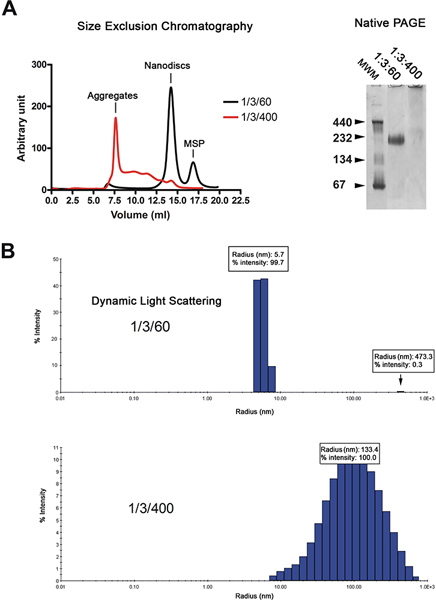
Figura 2. Il controllo di qualità della preparazione nanodisc. A. Filtrazione analisi Gel (Superdex 200 HR 10/300 colonne) delle nanodiscs ricostituito con rapporto di lipidi bassa (1/3/60; traccia nera) o il rapporto lipidi alta (1/3/400; traccia rossa). Elettroforesi su gel nativo del preparato stesso disco. Molmarcatori di peso ecular in kDa sono indicati. B. Analisi luce dinamica dispersione del preparato stesso disco. Clicca qui per ingrandire la figura .

Figura 3. Analisi del MalFGK 2-nanodisc particelle. A. Spostamento analisi Gel di Malè incubate con quantità crescenti di MalFGK 2 nanodisc particelle. B. ATPasi del MalFGK 2 nanodisc particelle in funzione della concentrazione di Malè.
Discussione
Descriviamo una semplice procedura per la ricostituzione del trasportatore maltosio in nanodiscs. Il trasportatore è ATPasi attiva e l'interazione con il maschio solubile partner di legame può essere ricreato (Figura 3). La ricostituzione di successo del trasportatore in nanodiscs aprire la strada a ulteriori analisi biofisica e biochimica. Di particolare interesse sarà l'analisi sistematica della ATPasi MalK e attività di trasporto in maltosio detersivo, liposomi e nanodiscs. Trasportatori ...
Divulgazioni
Nessun conflitto di interessi dichiarati.
Riconoscimenti
Questo lavoro è stato sostenuto dal Canadian Institute of Health Research. CSC è stato finanziato da una borsa di studio post-dottorato della scienze naturali e ingegneria Research Council del Canada. FD è un Tier II Canada Research Chair.
Materiali
| Name | Company | Catalog Number | Comments | ||||||||||||||||||||||||||||||
| Nome del reagente | Azienda | Numero di catalogo | Commenti | ||||||||||||||||||||||||||||||
| Amicon Ultra-4 50K filtro centrifugo | Millipore | UFC805008 | Segui protocollo del produttore per un uso corretto | ||||||||||||||||||||||||||||||
| Bio-Beads SM-2 Adsorbente | Bio-Rad | 152-3920 | |||||||||||||||||||||||||||||||
| E. coli lipidi totali | Avanti Polar Lipids | 100500C | Disciolto in cloroformio, come appropriato per gestire un solvente organico | ||||||||||||||||||||||||||||||
| Ni sefarosio HP resina | GE Healthcare | 17-5268-01 | |||||||||||||||||||||||||||||||
| Fosforo soluzione standard | Sigma-Aldrich | P3869 | |||||||||||||||||||||||||||||||
| pMSP1D1 | Addgene | 20061 | |||||||||||||||||||||||||||||||
| Superdex 200 HR 10/300 | GE Healthcare | 17-5172-01 | |||||||||||||||||||||||||||||||
| Tabella I. reagenti specifici. | |||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||
Riferimenti
- Denisov, I. G., Ginkova, Y. V., Lazarides, A. A., Sligar, S. G. Directed self-assembly of monodisperse phospholipid bilayer Nanodiscs with controlled size. J. Am. Chem. Soc. 126, 3477-3487 (2004).
- Boldog, T., Grimme, S., Li, M., Sligar, S. G., Hazelbauer, G. L. Nanodiscs separate chemoreceptor oligomeric states and reveal their signaling properties. Proc. Natl. Acad. Sci. USA. 103, 11509-11514 (2006).
- Bass, B. J., Denisov, I. G., Sligar, S. G. Homotropic cooperativity of monomeric cytochrome P450 3A4 in a nanoscale native bilayer environment. J. Biol. Chem. 282, 7066-7076 (2007).
- Alami, M., Dalal, K., Lelj-Garolla, B., Sligar, S. G., Duong, F. Nanodiscs unravel the interaction between the SecYEG channel and its cytosolic partner SecA. EMBO J. 26, 1995-2004 (2007).
- Mi, L. -. Z., Grey, M. J., Nishida, N., Walz, T., Lu, C., Springer, T. A. Functional and structural stability of the epidermal growth factor receptor in detergent micelles and phospholipid nanodiscs. Biochemistry. 47, 10314-10323 (2008).
- Schägger, H., Cramer, W. A., von Jagow, G. Analysis of molecular masses and oligomeric states of protein complexes by blue native electrophoresis and isolation of membrane protein complexes by two-dimensional native electrophoresis. Anal. Biochem. 217, 220-230 (1994).
- Dalal, K., Duong, F. Reconstitution of the SecY translocon in Nanodiscs. Methods Mol. Biol. 619, 145-156 (2010).
- Lanzetta, P. A., Alvarez, L. J., Reinach, P. S., Candia, O. A. An improved assay for nanomole amounts of inorganic phosphate. Anal. Biochem. 100, 95-97 (1979).
- Davidson, A. L., Dassa, E., Orelle, C., Chen, J. Structure, function and evolution of bacterial ATP-binding cassette systems. Microbiol. Mol. Biol. Rev. 72, 317-364 (2008).
- Bordignon, E., Grote, M., Schneider, E. The maltose ATP-binding cassette transporter in the 21st century-towards a structural dynamic perspective on its mode of action. Mol. Microbiol. 77, 1354-1366 (2010).
- Alvarez, F. J., Orelle, C., Davidson, A. L. Functional reconstitution of an ABC transporter for use in electron paramagnetic resonance spectroscopy. J. Am. Chem. Soc. 132, 9513-9515 (2010).
- Ritchie, T. K., Grinkova, Y. V., Bayburt, T. H., Denisov, I. G., Zolnerciks, J. K., Atkins, W. M., Sligar, S. G. Reconstitution of membrane proteins in phospholipid bilayer Nanodiscs. Methods Enzymol. 464, 211-231 (2009).
- Glück, J. M., Koenig, B. W., Willbold, D. Nanodiscs allow the use of integral membrane proteins as analytes in surface plasmon resonance studies. Anal. Biochem. 408, 46-52 (2011).
- Wan, C. -. P. L., Chiu, M. H., Wu, X., Lee, S. K., Prenner, E. J., Weers, P. M. M. Apolipoprotein-induced conversion of phosphatidylcholine bilayer vesicles into nanodisks. Biochim. Biophys. Acta (BBA). 1808, 606-613 (2011).
- Nath, A., Trexler, A. J., Koo, P. K., Miranker, A. D., Atkins, W. M., Rhoades, E. Single-molecule fluorescence spectroscopy using phospholipid bilayer Nanodiscs. Methods Enzymol. 472, 89-117 (2010).
- Denisov, I. G., Sligar, S. G. Cytochromes P450 in Nanodiscs. Biochim. Biophys. Acta. 1814, 223-229 (2011).
- Zhang, X. X., Chan, C. S., Bao, H., Fang, Y., Foster, L. J., Duong, F. Nanodiscs and SILAC-based mass spectrometry to identify a membrane protein interactome. J. Proteome Res. , (2011).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon