Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Eine Schritt-für-Schritt-Verfahren zur Rekonstitution eines ABC-Transporter in Nanodisc Lipid Particles
In diesem Artikel
Zusammenfassung
Nanoscheiben sind kleine scheibenförmige Partikel, die Membranproteine in einem kleinen Fleckchen Phospholipiddoppelschicht integrieren. Wir stellen eine visuelle Protokoll, das die Schritt-für-Schritt Einarbeitung des MalFGK2 Transporters zu einer Scheibe zeigt.
Zusammenfassung
Die nanodisc ist eine scheibenförmige Teilchen (~ 10-12 nm groß), dass trap Membranproteine in einem kleinen Fleckchen Phospholipiddoppelschicht. Die nanodisc ist eine besonders attraktive Option zur Untersuchung Membranproteine, insbesondere im Kontext von Ligand-Rezeptor-Wechselwirkungen. Die Methode erstmals von Sligar und Kollegen auf den amphipathischen Eigenschaften eines technisch hoch ein-helikalen Gerüstprotein aus dem Apolipoprotein A1 abgeleitet. Die hydrophoben Flächen des Gerüstprotein interagieren mit den Fettacyl Seitenketten der Lipiddoppelschicht während die polaren Regionen der wässrigen Umgebung zugewandt. Analysen von Membranproteinen in Nanoscheiben deutliche Vorteile gegenüber Liposom weil die Teilchen kleine, homogene und wasserlösliche sind. Zusätzlich können biochemische und biophysikalische Methoden normalerweise löslichen Proteinen vorbehalten angewandt werden, und von beiden Seiten der Membran. In dieser visuellen Protokoll, präsentieren wir eine Schritt-für-Schritt Rekonstitution eines gut charakterisiertgekenn zeichnet bakteriellen ABC-Transporter, die männlich-MalFGK 2-Komplex. Die Bildung der Platte eine Selbstorganisation, die auf hydrophoben Wechselwirkungen stattfinden während der progressiven Entfernung des Detergens abhängig ist. Wir beschreiben die wesentlichen Schritte und wir die Bedeutung der Wahl eines korrekten Protein-zu-Lipid-Verhältnis, um die Bildung von Aggregaten und größer polydispersen Liposomen-ähnliche Partikel zu begrenzen. Einfache Qualitätskontrollen wie Gelfiltrationschromatographie, native Gelelektrophorese und dynamische Lichtstreuung Spektroskopie sicherzustellen, dass die Scheiben wurden ordnungsgemäß wiederhergestellt.
Protokoll
Insgesamt Rekonstitutionsverfahren
Die Rekonstitution Prozess beginnt durch Mischen der Membran Gerüstprotein (MSP) mit dem gereinigten MalFGK 2-Komplex in Gegenwart von Detergens-solubilisierten Phospholipide. Der Schritt wird durch die langsame Entfernen des Detergens durch ein Adsorptionsmittel Polystyrolmaterial genannte Bio-Beads oder Amberlite (Abbildung 1) verfolgt. Selbststartendes Montageprozess tritt am wahrscheinlichsten aufgrund der Wechselwirkungen zwischen den apolaren hydrophoben Phospholipide, die 2 MalFGK Komplexes und der Oberfläche der MSP amphipathisches Protein. Das Endprodukt ist eine scheibenförmige Teilchen von zwei Molekülen MSP Umschlingung der MalFGK 2-Komplex hergestellt. Die Partikel werden von den Addukten und Aggregate durch Ultrazentrifugation und analytische Größenausschluss-Chromatographie getrennt. Die Partikel werden durch native-Gelelektrophorese und dynamische Lichtstreuung Spektroskopie charakterisiert.
Titel "> 1. Vorbereitung der Membrane Scaffold Protein, MSP- Die his-markierte MSP (Version MSP1D1 1) wird von dem Plasmid in E. hergestellt pMSP1D1 coli BL21 (DE3) bei OD 600 ~ 0,5 mit 0,5 mM Isopropyl-β-D-1-thiogalactopyranosid für 3 h induziert bei 37 ° C.
- Die Zellen werden durch Zentrifugation bei 5.000 xg für 10 min bei 4 ° C geerntet und in TSG10 Puffer mit 100 uM Phenylmethansulfonylfluorid.
- Die Zellen werden mit einem Französisch Presse (3x) bei 8000 psi lysiert und das unlösliche Material wird durch Zentrifugation bei 5000 × g für 10 min entfernt wird bei 4 ° C. Die lösliche Fraktion, die das MSP durch Ultrazentrifugation bei 125.000 × g für 45 min isoliert.
- Der MSP wird durch Nickel-Chelat-Chromatographie mit ~ 1,5 ml Ni Sepharose HP in TSG10 Puffer gereinigt. Verunreinigungen werden mit auswärts TSG10 Puffer, der 50 mM Imidazol gewaschen. Die MSP ist mit TSG10 Puffer mit 600 mM eluiertImidazol. Das gereinigte Protein wird in TSG10 Puffer dialysiert und in -70 ° C bei einer Proteinkonzentration von ~ 10-15 mg / ml.
2. Vorbereitung der MalFGK 2 Complex
- Die MalFGK 2-Komplex, an dem C-Terminus von MalK his-markierte, wird aus dem Plasmid-pBAD22 FGK exprimiert in E. coli BL21 (DE3) und induziert bei OD 600 ~ 0,5 mit 0,2% L-Arabinose für 3 Stunden bei 37 ° C.
- Nach Zellaufschluß und Zentrifugation wie in Schritt 1.3 wird die Membran Pellet in TSG20 Puffer in einer Endkonzentration von 5 mg / ml resuspendiert. Das Material wird mit 1% w / v n-Dodecyl-β-maltopyranosid (DDM) während 3 Stunden bei 4 ° C unter vorsichtigem Schütteln gelöst.
- Das unlösliche Material wird durch Ultrazentrifugation nach Stufe 1.3 entfernt und der Überstand, der das solubilisierte MalFGK 2-Komplex wird gesammelt und gereinigt, wie in Schritt 1.4, aber ohne Dialyse.
- Further Reinigung erfolgt durch Gelfiltrationschromatographie in TSGD Puffer auf einer Superdex 200 HR 10/300 Säule erreicht bei einer Fließgeschwindigkeit von 0,5 ml / min.
3. Vorbereitung der Phospholipide
- Ein E. coli insgesamt Lipidextrakt in Chloroform gelöst in 1.000 nmol Aliquots in Schraubverschluss Mikrozentrifugenröhrchen getrennt. Das Lösungsmittel wird unter einem leichten Stickstoffstrom eingedampft und getrocknet weitere Nacht in einem Vakuumexsikkator.
- Der Lipidfilm wird in TS-Puffer bei 5 nM Endkonzentration gelöst und gevortext kräftig und beschallt. Die gelösten Lipide erscheint leicht opak aufgrund ihrer allgemeinen Unlöslichkeit in wässrigen TS-Puffer, aber sie sollten in der Schwebe bleiben. DDM wird bis zu einer Endkonzentration von 0,5% (~ 10 mM), bei dem die Lösung klar wird zugegeben.
- Das Lipidgemisch wird in ein Wasserbad gestellt und Puls beschallt 5 mal für ~ 5 sek. Das Lipidgemisch wird bei 4 ° C für maximal 1 wee gespeichertenk.
4. Vorbereitung der Bio-Beads
- Etwa 10-15 ml (Volumen im trockenen Zustand) Bio-Beads wird in einem 50 ml Röhrchen gegeben.
- Die Perlen werden nacheinander mit 50 ml 100% Methanol, 95% Ethanol, milliQ H 2 O und schließlich TS-Puffer (zweimal) gewaschen.
- Die gewaschenen Kügelchen werden bei 4 ° C in 10 ml ~ TS-Puffer.
5. Nanodisc Rekonstitution
- Der MSP in TSGD Puffer bei einer Endkonzentration von ~ 7 mg / ml (~ 0,3 mM) verdünnt.
- MSP: Lipid-Verhältnis von 1:3:60 oder 1:3:400 in TSGD Puffer ~ 2 nmol von gereinigtem MalFGK 2-Komplex an ein Protein gemischt. Die Endkonzentration beträgt 6 uM MalFGK 2, 18 und 360 pM MSP uM Lipide (1:3:60) oder 2,4 mM Lipide (1:3:400). Das Endvolumen beträgt 300 ul. Die endgültige Konzentration DDM beträgt 0,08% (~ 1,6 mm). Die Endkonzentration von Glycerin ist abhängig von der Menge an Lipid zugesetzt bleibt aber etwa 5-10% v / v;
- ~ 50 ul Bio-Bead Suspension wird in das Röhrchen gegeben und die Mischung wird über Nacht auf einem Schüttler Tabelle bei 4 ° C inkubiert
- Die Perlen werden durch Schwerkraft sedimentiert und die Lösung wird durch eine schmale Spitze pipettiert, so viel Bio-Beads wie möglich zu vermeiden.
- Große Präzipitate werden durch Ultrazentrifugation bei 100.000 × g für 20 min entfernt. Die Scheiben werden durch Gelfiltrationschromatographie, wie in Schritt 2.4 in TSG10 Puffer gereinigt. Fraktionen, welche die Scheiben werden gepoolt und ein Aliquot sollte auf demselben Gelfiltrationssäule, um die Stabilität der Zubereitung testen erneut injiziert werden.
- Die gereinigten Scheiben können bei -70 ° C über einen längeren Zeitraum gelagert. Nach dem Auftauen auf Eis, sollten die Scheiben Ultrazentrifugation wie in Schritt 5,5 unterzogen werden, um zu entfernen potenziellen ausfällt. Ein Aliquot sollte durch Gelfiltrationschromatographie analysiert werden, um sicherzustellen, dass eine nennenswerte Aggregation oder Ausfällung nicht während des Auftauens auftretenProzess.
6. Nativer Gelelektrophorese
- 1 uM Nanoscheiben (~ 0,2 mg / ml) werden durch native PAGE 6, 7 (Tris-HCl pH 8,8, 4-12%), um die Qualität der Rekonstitution (3A) zu beurteilen analysiert.
- Die Bindung der männlichen (1 uM) zur MalFGK 2-Komplex rekonstituiert in Nanoscheiben wird auch durch native-PAGE (3A) beurteilt.
- Nach der Elektrophorese wird das Gel mit Coomassieblau für 10 min gefärbt und entfärbt für ~ 1 Stunde.
7. Dynamic Light Scattering (DLS)
- Nanoscheiben werden durch Gelfiltrationschromatographie, wie in Schritt 2.4 in TSG10 Puffer unter Verwendung einer Superdex 200 HR 10/300 Säule bei einer Fließgeschwindigkeit von 0,1 ml / min gereinigt. Enthaltenden Fraktionen werden vereinigt und Nanoscheiben konzentriert, um ~ 10 mg / ml mit einer Amicon Filterzentrifuge.
- Die Probe wird zweimal (0,22 um Filter) gefiltert vor der Analyse unter Verwendung von DLSein DynaPro NANOSTAR Instrument (Wyatt Technology) in einem 1 ul Innenvolumen Quarzküvette. Daten werden unter Verwendung des eingepasst DYNAMICS Software (Wyatt Technology), um den Durchmesser und das Molekulargewicht der Teilchen zu schätzen.
8. ATPase Messungen
- Die ATPase-Aktivität von 2-MalFGK rekonstituierten Nanoscheiben wird unter Verwendung eines kolorimetrischen Assay 8.
- 1 uM des gereinigten Scheiben und 1 mM ATP zusammen mit steigenden Mengen von männlichen und bei 37 ° C für 20 min gemischt. Die Freisetzung von anorganischem Phosphat wird bei 660 nm gemessen.
- Die Menge an Phosphat-Freigabe mit einer Standardkurve, die mit einer Standardlösung erzeugte Phosphor verglichen.
9. Repräsentative Ergebnisse
Die Nanoscheiben werden durch Gelfiltrationschromatographie (2A, links) gereinigt. Das Chromatogramm zeigt, dass die Mehrheit der rekonstituierten dISCs (schwarze Kurve) eluieren als einzelner Peak, während Discs mit überschüssigem Lipide (rote Linie) eluieren im Leervolumen und als eine Reihe von breiten Peaks hergestellt. Die Qualität der Nanoscheiben wird weiter durch native Gelelektrophorese und dynamische Lichtstreuung Spektroskopie (DLS) analysiert. Richtig rekonstituierten Discs als scharfe Bande auf dem Gel wandern während diejenigen rekonstituierten in Gegenwart eines Überschusses von Lipiden als eine Schmierschicht (2A, rechts) zu migrieren. Analyse durch DLS zeigt, dass die Scheibe Population homogen mit einem durchschnittlichen Durchmesser von 11,4 nm (2B) ist. Die rekonstituierten Scheiben haben ein scheinbares Molekulargewicht von 215 kDa auf dem DLS-Näherung berechnet. Proben rekonstituiert in Gegenwart eines Überschusses an Lipiden Display weithin Radien um 100 nm verteilt sind, was typisch ist für inhomogene Proben.
Die Qualität der MalFGK 2-Komplex wird durch native-Gelelektrophorese und seine Aktivität durch ATPase beurteiltMessungen (Abbildung 3). Die Maltosebindeprotein MalE bindet mit hoher Affinität an den Transporter MalFGK 2 9, 10. Verwendung nicht-denaturierender Gelelektrophorese, ist es möglich, einen Komplex zwischen männlichen und MalFGK 2 (Abbildung 3A) zu detektieren. Die Stimulation des MalK 2 ATPase-Aktivität von männlichen in 3B gezeigt.
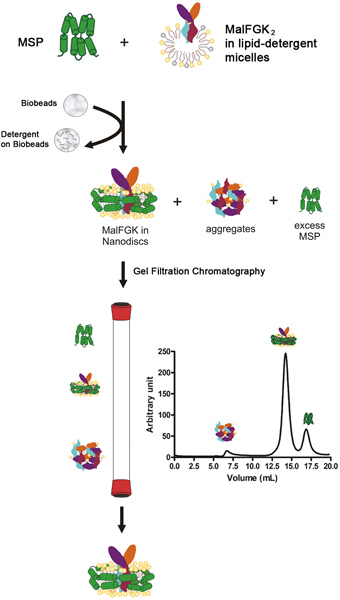
Abbildung 1. Typische Ablaufplan für die Wiederherstellung Protokoll.
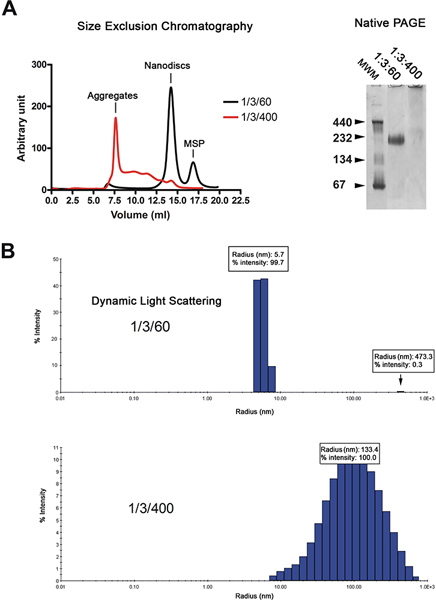
Abbildung 2. Qualitätskontrolle der nanodisc Vorbereitung. A. Gelfiltrationsanalyse (Superdex 200 HR 10/300 Spalte) der Nanoscheiben rekonstituiert bei niedrigen Lipid-Verhältnis (1/3/60; schwarze Kurve) oder hoher Lipid-Verhältnis (1/3/400, rote Kurve). Nativer Gelelektrophorese derselben Disk Zubereitung. Molecular Gewicht Marker in kDa angegeben. B. dynamische Lichtstreuung Analyse der gleichen Disc Vorbereitung. Klicken Sie hier für eine größere Abbildung zu sehen .

Abbildung 3. Analyse des MalFGK 2-nanodisc Teilchen. A. Gel-Shift-Analyse MalE mit steigenden Mengen MalFGK 2-nanodisc Partikeln inkubiert. B. ATPase Aktivität des MalFGK 2-nanodisc Teilchen als Funktion der männlichen Konzentration.
Diskussion
Wir beschreiben ein einfaches Verfahren für die Wiederherstellung der Maltose Transporter in Nanoscheiben. Der Transporter ist ATPase aktiv und die Interaktion mit dem löslichen Bindungspartner MalE können wiederhergestellt (Abbildung 3). Die erfolgreiche Rekonstitution des Transporters in Nanoscheiben öffnen den Weg für weitere biophysikalische und biochemische Analyse. Von besonderem Interesse ist dabei die systematische Analyse werden die MalK ATPase und Maltose Transportaktivität in Wasch-, Li...
Offenlegungen
Keine Interessenskonflikte erklärt.
Danksagungen
Diese Arbeit wurde von der Canadian Institute of Health Research unterstützt. CSC wurde von einem Postdoc-Stipendium von der Natural Sciences and Engineering Research Council of Canada finanziert. FD ist ein Tier-II Canada Research Chair.
Materialien
| Name | Company | Catalog Number | Comments | ||||||||||||||||||||||||||||||
| Name des Reagenzes | Firma | Katalog-Nummer | Kommentare | ||||||||||||||||||||||||||||||
| Amicon Ultra-4 50K Zentrifugalfilter | Millipore | UFC805008 | Folgen Sie dem Protokoll des Herstellers für die ordnungsgemäße Verwendung | ||||||||||||||||||||||||||||||
| Bio-Beads SM-2 Adsorbent | Bio-Rad | 152-3920 | |||||||||||||||||||||||||||||||
| E. coli Gesamtlipide | Avanti Polar Lipids | 100500C | Gelöst in Chloroform, zu behandeln, wie es sich für ein organisches Lösungsmittel | ||||||||||||||||||||||||||||||
| Ni-Sepharose HP Harz | GE Healthcare | 17-5268-01 | |||||||||||||||||||||||||||||||
| Phosphor Standardlösung | Sigma-Aldrich | P3869 | |||||||||||||||||||||||||||||||
| pMSP1D1 | Addgene | 20061 | |||||||||||||||||||||||||||||||
| Superdex 200 HR 10/300 | GE Healthcare | 17-5172-01 | |||||||||||||||||||||||||||||||
| Tabelle I. Spezifische Reagenzien. | |||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||
Referenzen
- Denisov, I. G., Ginkova, Y. V., Lazarides, A. A., Sligar, S. G. Directed self-assembly of monodisperse phospholipid bilayer Nanodiscs with controlled size. J. Am. Chem. Soc. 126, 3477-3487 (2004).
- Boldog, T., Grimme, S., Li, M., Sligar, S. G., Hazelbauer, G. L. Nanodiscs separate chemoreceptor oligomeric states and reveal their signaling properties. Proc. Natl. Acad. Sci. USA. 103, 11509-11514 (2006).
- Bass, B. J., Denisov, I. G., Sligar, S. G. Homotropic cooperativity of monomeric cytochrome P450 3A4 in a nanoscale native bilayer environment. J. Biol. Chem. 282, 7066-7076 (2007).
- Alami, M., Dalal, K., Lelj-Garolla, B., Sligar, S. G., Duong, F. Nanodiscs unravel the interaction between the SecYEG channel and its cytosolic partner SecA. EMBO J. 26, 1995-2004 (2007).
- Mi, L. -. Z., Grey, M. J., Nishida, N., Walz, T., Lu, C., Springer, T. A. Functional and structural stability of the epidermal growth factor receptor in detergent micelles and phospholipid nanodiscs. Biochemistry. 47, 10314-10323 (2008).
- Schägger, H., Cramer, W. A., von Jagow, G. Analysis of molecular masses and oligomeric states of protein complexes by blue native electrophoresis and isolation of membrane protein complexes by two-dimensional native electrophoresis. Anal. Biochem. 217, 220-230 (1994).
- Dalal, K., Duong, F. Reconstitution of the SecY translocon in Nanodiscs. Methods Mol. Biol. 619, 145-156 (2010).
- Lanzetta, P. A., Alvarez, L. J., Reinach, P. S., Candia, O. A. An improved assay for nanomole amounts of inorganic phosphate. Anal. Biochem. 100, 95-97 (1979).
- Davidson, A. L., Dassa, E., Orelle, C., Chen, J. Structure, function and evolution of bacterial ATP-binding cassette systems. Microbiol. Mol. Biol. Rev. 72, 317-364 (2008).
- Bordignon, E., Grote, M., Schneider, E. The maltose ATP-binding cassette transporter in the 21st century-towards a structural dynamic perspective on its mode of action. Mol. Microbiol. 77, 1354-1366 (2010).
- Alvarez, F. J., Orelle, C., Davidson, A. L. Functional reconstitution of an ABC transporter for use in electron paramagnetic resonance spectroscopy. J. Am. Chem. Soc. 132, 9513-9515 (2010).
- Ritchie, T. K., Grinkova, Y. V., Bayburt, T. H., Denisov, I. G., Zolnerciks, J. K., Atkins, W. M., Sligar, S. G. Reconstitution of membrane proteins in phospholipid bilayer Nanodiscs. Methods Enzymol. 464, 211-231 (2009).
- Glück, J. M., Koenig, B. W., Willbold, D. Nanodiscs allow the use of integral membrane proteins as analytes in surface plasmon resonance studies. Anal. Biochem. 408, 46-52 (2011).
- Wan, C. -. P. L., Chiu, M. H., Wu, X., Lee, S. K., Prenner, E. J., Weers, P. M. M. Apolipoprotein-induced conversion of phosphatidylcholine bilayer vesicles into nanodisks. Biochim. Biophys. Acta (BBA). 1808, 606-613 (2011).
- Nath, A., Trexler, A. J., Koo, P. K., Miranker, A. D., Atkins, W. M., Rhoades, E. Single-molecule fluorescence spectroscopy using phospholipid bilayer Nanodiscs. Methods Enzymol. 472, 89-117 (2010).
- Denisov, I. G., Sligar, S. G. Cytochromes P450 in Nanodiscs. Biochim. Biophys. Acta. 1814, 223-229 (2011).
- Zhang, X. X., Chan, C. S., Bao, H., Fang, Y., Foster, L. J., Duong, F. Nanodiscs and SILAC-based mass spectrometry to identify a membrane protein interactome. J. Proteome Res. , (2011).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten