Method Article
Los protocolos para la implementación de un
* Estos autores han contribuido por igual
En este artículo
Erratum Notice
Resumen
Este protocolo de cinco días se esbozan todos los pasos, el equipo y el software complementario necesario para la creación y ejecución de un eficiente Escherichia coli endógeno basado sistema de expresión libre de células TX-TL a partir de cero. Con los reactivos, el protocolo de toma 8 horas o menos a la configuración de una reacción, recopilar y procesar datos.
Resumen
Sistemas de expresión de ideales sin células pueden emular teóricamente un entorno celular in vivo en un control en la plataforma vitro. 1 Esto es útil para expresar las proteínas y los circuitos genéticos de una manera controlada, así como para proporcionar un entorno de creación de prototipos para la biología sintética. 2,3 Para lograr este último objetivo, los sistemas de expresión de células libres que preservan mecanismos de transcripción-traducción Escherichia coli endógenas son capaces de reflejar con mayor precisión la dinámica celular in vivo que los basados en T7 RNA polimerasa de transcripción. Se describe la preparación y ejecución de un desarrollo endógeno E. eficiente la transcripción-traducción basada coli (TX-TL) sistema de expresión libre de células que pueden producir cantidades equivalentes de proteína como los sistemas basados en T7 en una reducción de costes del 98% a los sistemas comerciales similares. 4,5 La preparación de tampones y extracto celular en bruto se descrito, así como la ejecución de untres tubos de reacción TX-TL. El protocolo completo dura cinco días para preparar y produce material suficiente para un máximo de 3.000 reacciones individuales en una preparación. Una vez preparado, cada reacción tiene menos de 8 horas desde la configuración hasta la recolección y análisis de datos. Mecanismos de regulación de la transcripción y exógena a la E. coli, tales como represores lac / tet y ARN polimerasa de T7, se puede complementar. 6 propiedades endógenos, tales como ARNm y las tasas de degradación de ADN, también se puede ajustar. 7 El sistema de expresión libre de células TX-TL se ha demostrado para gran ensamblaje escala del circuito, la exploración de los fenómenos biológicos, y la expresión de proteínas bajo las dos promotores T7 y endógenos. 6,8 modelos matemáticos de acompañamiento están disponibles. 9,10 El sistema resultante tiene aplicaciones únicas en la biología sintética como un entorno de creación de prototipos, o "TX- protoboard biomolecular TL ".
Introducción
Tecnología de expresión libre de células se inició en la década de 1950 como puramente traslacional, avanzando año más tarde para incluir mecanismos de transcripción-traducción acopladas utilizando el ADN del bacteriófago T7. 11,12 Desde entonces, se han realizado numerosos esfuerzos para optimizar la creación de extracto celular crudo (o E . extracto coli S30). 13,14 Estas optimizaciones incluyen la prolongación de la síntesis de proteínas libre de células a través de la regeneración de ATP o modificaciones de tensión y reduciendo el tiempo de protocolo y el costo. existen 15-17 sistemas de expresión de células libres alternativos que uso reconstituido componentes en lugar de crudo extracto de células para la expresión. 5 Tanto extracto celular crudo y métodos de reconstitución se han desarrollado para uso comercial.
Con el advenimiento de la biología sintética, existe una mayor necesidad de una plataforma bien caracterizado para probar y expresar módulos y circuitos biológicos de ingeniería. 18,19 Esta plataforma debe serversátil, bien caracterizado, fácil de manipular, y se centró en los componentes suministrados por el usuario. A pesar de haber desarrollado la mitad de un siglo antes, los sistemas libres de células basadas en E. coli intrínsecamente compartir estos requisitos, ya que son una representación simplificada en vitro de procesos celulares sin la complejidad de crecimiento y el metabolismo. Además, todos los conocimientos básicos de trabajo en vivo en E. coli se aplica fácilmente a E. sistemas libres de células de E. coli.
Aunque los sistemas de expresión de células libres pueden tener aplicaciones en la biología sintética, hasta la fecha el objetivo de la mayoría de los sistemas de expresión de células libres ha sido la maximización de la proteína y la producción de metabolitos. Esto se logra mediante el uso de bacteriófago T7 transcripción de secuencias conducidos por promotores T7. 20 Aunque la expresión es eficiente y robusta, estos sistemas sirven a un propósito altamente especializado. Métodos de regulación de la célula son limitados, las plantillas de ADN de destino deben serrediseñados para incluir promotores T7, y ciertas secuencias tales como complejos ribosómicos no se pueden transcribir y ensamblados. 21,22 sistemas de expresión libres de células existentes son incapaces de mantener altos rendimientos preservando al mismo tiempo los mecanismos de regulación endógenos, una versatilidad necesaria para la biología sintética.
Hemos desarrollado una E. endógena sistema de expresión libre de células coli que conserva la eficiencia de la expresión de la proteína demostrado por los sistemas anteriores pero añade versatilidad adicional al permitir la expresión y la regulación sobre la base de ambos mecanismos (T7 u otros) endógeno y exógeno. El protocolo descrito aquí está basada originalmente en Kigawa et al. (2004) y Liu et al. (2005), pero tiene importantes modificaciones. Se utiliza Mg y K-glutamato sobre Mg-y K-acetato de etilo para aumentar la eficiencia, quita 2-mercaptoetanol, y la lisis de las células usando un cordón-batidor. 17,23,24 bolas-latidos se elige sobre la homogeneización,métodos basados en la presión, o sonicación debido a su menor coste y rendimientos comparables a los sistemas de la competencia. 23 ácido 3-fosfoglicérico (3-PGA) se utiliza como la fuente de energía ya que se encontró para dar rendimientos de proteínas superiores en comparación con el fosfato de creatina y fosfoenolpiruvato. 4,25 Nuestro sistema puede producir hasta 0,75 mg / ml de proteína indicadora utilizando un promotor con sede en sigma70 con los operadores-fago lambda o un promotor T7 impulsado, de forma similar a los rendimientos procedentes de otros sistemas comerciales. 4,6 Cinco días son necesarias para producir todos los reactivos necesarios (Figura 1). Además, proporciona una reducción de costes del 98% en comparación con los sistemas comparables de células libres de comerciales - costes de material son de $ 0.11 por 10 l de reacción, que se eleva a 0,26 dólares, con mano de obra incluida (Figura 2).
Protocolo
1. Preparación extracto crudo de la célula
Preparación de extracto celular crudo durante tres días se necesitan dos personas para llevar a cabo de manera eficiente. El protocolo consiste funcionalmente de tres partes: crecimiento del cultivo (etapa 1.1 al paso 1,11), lisis celular (paso 1,12 al paso 1,37), y extraer la clarificación (paso 1,38 al paso 1,52). Se presenta dividida en día para mayor comodidad. Extracto ideal puede producir 0,75 mg / ml de deGFP de plásmido pbest-OR2-OR1-Pr-UTR1-deGFP-T500 (Addgene # 40019), y tiene una concentración de extracto celular en bruto entre 27-30 mg / ml de proteína. 4 Sin embargo , extracto de características varían de lote a lote. La siguiente receta suministra suficiente para aproximadamente 3000 reacciones individuales (6 ml de extracto celular en bruto). Si la escala hacia abajo, se recomienda utilizar no menos de 1/6 de los valores dados aquí. Por falta de tiempo, no se recomienda la ampliación.
Día 1
- Preparar los medios de cultivo bacteriano, cultura de la placa, y los medios de comunicación los suplementos como se describe en la Tabla 1. Ver Material Complementario 1 para las recetas.
- . Cepa BL21-racha de Rosetta2 de -80 ° C sobre una placa de agar 2xYT + P + cm y se incuba durante al menos 15 horas a 37 ° C o hasta que las colonias son fácilmente visibles Nota: Cloranfenicol (Cm) se utiliza para seleccionar para un plásmido codifica ARNt raros en la cepa BL21-Rosetta2.
Día 2
- Preparar tampones y suplementos como se describe en la Tabla 2. Ver Material Complementario 1 para las recetas.
- Preparar y esterilizar el material necesario para el día 3, que incluye: 6 x 4 matraces Erlenmeyer de L con una cubierta de papel de aluminio (autoclave), botellas de centrífuga estériles 4 x 1 L, embudo (autoclave), 100 g de 0,1 mm perlas de vidrio (en autoclave), 2 revolver-bares (autoclave), 1 L y 500 ml aforado (autoclave), 2 x 1 vasos L (en autoclave), 3 ml jeringa con 18 agujas G (estériles), 2-3 flBoyas de avena, 2-3 10k casetes de diálisis MWCO (estéril), cubetas.
- Preparar mini-cultura 1. Añadir 4 ml de medio 2xYT + P y 4 l de Cm a un tubo de cultivo estéril 12 ml y pre-caliente a 37 ° C durante 30 min.
- Inocular mini-cultura 1 con una colonia de la P + + placa de agar 2xYT Cm. Incubar a 220 rpm, 37 º C durante 8 h.
- 7 horas y 30 minutos más tarde, preparar mini-cultura 2. Añadir 50 ml de medio 2xYT + P y 50 l de Cm a un estéril matraz Erlenmeyer de 250 ml y pre-calentamiento hasta 37 º C durante 30 min.
- Inocular Mini-cultura 2 con 100 l de mini-cultura 1 y se incuba a 220 rpm, 37 º C durante 8 h.
Día 3
- Pesar cuatro 50 ml tubos Falcon estériles vacíos y masa registro en la tabla 3. Chill tubos Falcon en el hielo, los cuales se utilizarán posteriormente en el paso 1.18.
- 7 horas y 30 minutos después de la etapa 1.8, se preparan los medios de comunicación definitiva cultivo bacteriano. El uso de un estéril 1 L cilindro graduado, la transferencia de 660 ml de 2xYT + P i mediosnto cada uno de seis 4 matraces Erlenmeyer L y pre-caliente a 37 ° C durante 30 min Nota:. 4 L o mayor se recomienda matraces Erlenmeyer para la aireación adecuada.
- Añadir 6,6 ml de mini-cultivo de 2 en cada matraz Erlenmeyer de 4 l. Incubar a 220 rpm, 37 ° C hasta que el cultivo alcanza una DO de 1,5-2,0 a 600 nm (correspondiente a la fase de crecimiento semilogarítmica). Compruebe periódicamente OD con una dilución 1:10 de cultivo para la precisión Este paso debe tomar no más de 3 horas - 3 hr 45 min;. Rápido crecimiento y la colección durante la fase semilogarítmica es fundamental para la calidad del extracto.
- Inmediatamente después de crecimiento, transferir todas las culturas de manera uniforme en cuatro botellas de centrífuga de 1 L y centrifugar a 5000 xg durante 12 min a 4 ° C para sedimentar las células bacterianas.
- Aunque la centrifugación, la preparación completa de búfer S30A añadiendo 4 ml de DTT 1 M a 2 L de S30A previamente preparado. Mezclar y mantener tampón en hielo.
- Una vez finalizada la centrifugación, eliminar completamente sobrenadante del paso 1,12 por decanting y borrando las botellas de centrífuga en una toalla de papel estéril.
- Añadir 200 ml de tampón de S30A a 4 º C a cada una de las cuatro botellas de centrífuga, y agitar vigorosamente hasta que las botellas sedimento se solubilizó completamente sin grumos restantes. Centrifugar las cuatro botellas en 5000 g durante 12 min a 4 ° C.
- Retire completamente sobrenadante del paso anterior por decantación y secar las botellas de centrífuga en una toalla de papel estéril.
- Repita los pasos 1.15 y 1.16.
- Añadir 40 ml de tampón S30A a 4 º C a cada botella de centrifugación. . Transfiera cada pellet y la combinación S30A en un tubo Falcon refrigerada de 1,9) Nota: Este paso es transferir los pellets en un recipiente pequeño.
- Centrifugar los tubos Falcon a 2000 g durante 8 min a 4 ° C. Eliminar el sobrenadante por decantación.
- Re-centrifugue los tubos Falcon a 2.000 g durante 2 min a 4 ° C. Eliminar completamente el sobrenadante residual mediante una pipeta. Mantener en hielo.
- Pesar la fnuestros tubos Falcon con pellet y masa registro en la tabla 3. Calcula la masa de pellets, volumen de tampón S30A es necesario, y la masa de los granos necesarios sobre la base de las fórmulas específicas de la Tabla 3.
- Correcta carga y de cuentas latido del pellet es fundamental para hacer el extracto de la calidad, y es el paso más difícil. Se recomienda revisar el video antes de intentar. Si no se evitan las burbujas de aire y distribuir granos uniformemente resultará en el extracto de ineficiente.
- Añadir la cantidad de tampón S30A calculado en la Tabla 3 a cada tubo Falcon, vórtice hasta homogeneidad, y volver a hielo.
- Mientras se mantiene los otros tubos Falcon en hielo, añadir perlas de forma intermitente a un solo tubo Falcon en tres alícuotas, cada uno con 1/3 de las perlas totales. Después de la adición de cada alícuota de perlas, de vórtice durante 30 segundos. Coloque el tubo de Falcon en el hielo entre los pasos de vórtice y después vórtice final. Después se añade la última alícuota, garantizarperlas se distribuyen de manera uniforme. Se debe formar una pasta espesa.
- Prepare una punta de pipeta de 5 ml (volumen) cortando el extremo utilizando una hoja de afeitar estéril para crear una abertura de 3-4 mm. Dial pipeta 2 ml Nota: Los diferentes conjuntos de pipetas y puntas proporcionan diferentes cantidades de succión que puede no ser suficiente para tirar y soltar la solución de células cordón grueso, una punta de pipeta de 1 ml con fines eliminado puede ser usado en lugar..
- Coloque 20 tubos de cuentas que superaron en hielo.
- Verifique alta viscosidad de la solución de célula-perla utilizando pipeta modificado. Debe ser viscoso hasta el punto de apenas sale de la punta de la pipeta durante la eyección. Si es demasiado viscoso, vuelva a ajustar la punta de la pipeta según la etapa 1.25. Si no es lo suficientemente viscoso, perlas se pueden añadir en incrementos de (masa de pellets * 0,05), a una masa máxima (masa de pellets * 5.1). Después de cada adición de perlas, vortex durante 30 segundos y volver al hielo. Vea la Figura 3a para una demostración de la viscosidad.
- Retire la solución de células de cuentas de tubo Falcon utilizando pipeta modificado, y transferir a un tubo de talón superando estéril, llenándolo de tres cuartos de su capacidad con una solución de células de perlas. Gira muy brevemente (1s) en un contador mini-centrifugadora para eliminar las burbujas de aire y sin redistribuir cuentas. Vea las Figuras 3b-d para imágenes fijas de tubo perlas paliza carga.
- Terminar añadiendo solución de células del grano para formar un menisco cóncavo.
- Añadir una pequeña gota de solución de células de cuentas en el interior de una tapa de tubo de bolas-de latir, teniendo cuidado de no obstaculizar el borde exterior de la tapa, de lo contrario, el tubo de talón superando no lo hará sufficientl cerca y. Toque en la tapa en una superficie plana y compruebe que no haya burbujas de aire en la parte inferior de la tapa.
- Tape el tubo de cuentas-golpes con el casquillo del grano, superando en el paso anterior. Mano a asistente de golpes de talón. Si se hace correctamente, la tapa debe estar bien sellado, no hay burbujas de aire Should ser visible, y poco solución de células talón (si los hay) se debe volcar fuera. Vuelva a realizar el proceso de carga si hay burbujas de aire visible o la tapa no hace cerrar completamente.
- Tubo Falcon Vortex desde el paso 1,24 con la solución de células del grano restante para asegurar una distribución uniforme de los granos. Repita los pasos 1.28 a 1.31 hasta que el tubo Falcon está vacía y, a continuación, repita los pasos 1.24 a 1.31 por cada tubo Falcon adicional.
- Conducta pasos 1,33-1,38 simultáneamente. Haga que toma auxiliar lleno de talones golpeando tubos de 1.31 y el lugar en el hielo. Una vez que dos tubos de cuentas-superando llenas se han recogido y han sido en hielo durante al menos un minuto, comience a latir de perlas.
- Batir un tubo durante 30 segundos a 46 rpm. Coloque boca abajo sobre hielo durante 30 segundos sin dejar de batir el otro tubo.
- Repita el paso anterior de tal manera que cada tubo de talón superando llenado se ha de superar para 1 min total.
- Repita los pasos 1,33-1,35 hasta 8 tubos de perlas llenas de batido (o el importe máximo de la centrífuga puede sostener) hAVE ha procesado. Entonces, la construcción de un aparato de filtro de 15 ml Falcon (Figura 3e). Añadir un nuevo casquillo del grano-de latir,-parte plana hacia arriba, al fondo de un Falcon de 15 ml. A continuación, retire el tapón del tubo de talón superando procesado y la columna micro-cromatografía presione firmemente en el extremo del tubo de talón superando procesada hasta que esté completamente sellada. Encajar off final de elución de la columna micro-cromatografía, y coloque la columna micro-cromatografía, elución termina abajo, en el tubo de perlas abalorios vacía. Coloque este complejo en 15 ml Falcon. Repita el procedimiento para todos los 8 tubos de cuentas-superando llenas; mantener en hielo cuando se complete.
- Centrífuga 8 aparatos de filtro, tubo Falcon destapado, en 6000 g durante 5 min a 4 ° C para separar el extracto de sedimento y de perlas.
- Verifique cada tubo perlas paliza ha producido extracto viable. Extracto correctamente batir no será turbia, y el sedimento tendrá dos capas distintas. Deseche todos los tubos turbios, y transferir el sobrenadante de los tubos no turbias i1,75 ml tubos de microcentrífuga nto individuales, teniendo tan poco pellet posible. Mantener en hielo hasta que se hayan procesado todos los tubos de cuentas-de latir. Vea la Figura 3f comparar un cordón tubo-paliza correctamente vs incorrectamente procesada.
- Tubos de centrífuga de microcentrífuga de paso anterior a 12.000 g durante 10 min a 4 ° C.
- Transfer-pellet libre sobrenadante en tubos de cuentas que superaron vacíos utilizando una pipeta, consolidando 500 l en un nuevo tubo de talones de latir.
- Incubar paso anterior, con tapas de cuentas-superando retirados, a 220 rpm, 37 º C durante 80 min. Este paso digiere restante ácidos nucleicos utilizando exonucleasas endógenos liberados durante el proceso de cuentas-el gotear, y se puede hacer de pie el tubo de talones golpeando en un tubo de cultivo de tejidos.
- Preparar materiales de diálisis. Preparación completa búfer S30B añadiendo 2 ml de DTT 1 M a 2 L de S30B previamente preparado. Mezclar y añadir 900 ml en cada uno de dos ererile 1 L vasos. Añadir un agitador magnético estéril en cada vaso, mantener a 4 ° C.
- Después del paso 1.41, extracto debe ser turbia. Consolidar extracto en alícuotas de 1,5 ml en 1,75 ml de tubos de microcentrífuga, y se centrifuga a 12.000 g durante 10 min a 4 ° C.
- Con una pipeta, consolidar sobrenadante libre de pellet en 15 ml tubos Falcon en el hielo, y mezclar bien tapando el tubo y de inversión. Ahorra 10 l de sobrenadante en hielo para el paso de 1.47.
- Determine la cantidad total de extracto producido, e hidratar el número necesario de 10k MWCO casetes de diálisis sumergiendo en S30B durante 2 minutos, suponiendo 2,5 ml de extracto por casete.
- Cargue cassettes con 2,5 ml de extracto. Cada vaso puede tardar hasta 2 cassettes; dializar, revolviendo, a 4 º C durante 3 horas Nota: la carga parcial de casetes es aceptable.. Diálisis aumenta el rendimiento de producción de proteínas.
- Durante el paso anterior, caracterizar la proteína extractoconcentración con un ensayo de Bradford, utilizando extracto de salvado en el paso 1.44. Ver Material Suplementario 2 para más detalles.
- Después de la diálisis es completa, extracto de alícuota por 1,5 ml en 1,75 ml de tubos de microcentrífuga. Centrifugar a 12000 g durante 10 min a 4 ° C. Un sedimento se forma en la parte inferior del tubo.
- Consolidar sobrenadante claro desde el paso anterior con la pipeta en un tubo Falcon de 15 ml en hielo. Homogeneizar por inversión de 5-10x.
- Sobre la base de concentración determinada por Bradford en el paso 1,47, determinar la cantidad de extracto de alícuota en 1,75 ml de tubos individuales. Cada tubo individual debe tener un volumen con 810-900 mg de proteína total. Extracto debe tener una concentración total de proteína mayor que 27 mg / ml. Este paso requiere asistencia para llevar a cabo convenientemente Nota:. Extracto Alícuota por debajo de 30 mg / ml en 30 ml de alícuotas, y la escala si la concentración es mayor, por ejemplo, el extracto de alícuota a 28 mg / ml por 30 l, y el extracto de alícuota a 32mg / ml de 28,1 l.
- Alícuota de extracto siguiente paso 1.50, teniendo cuidado de evitar burbujas. Extracto de flash-congelación en nitrógeno líquido Nota:. Alícuotas con las burbujas se pueden eliminar por centrifugación a 10.000 xg durante 30 segundos a 4 ° C.
- Retire los tubos de nitrógeno líquido utilizando un colador y guardar inmediatamente a -80 ° C. Seguridad: Usar gafas de protección; las tapas de los tubos de extracto podrían desprenderse debido a la diferencia de temperatura entre el nitrógeno líquido y la temperatura ambiente.
2. Aminoácidos Preparación de la Solución
Solución de aminoácidos debe ser preparado de forma masiva. La siguiente receta utiliza un kit completo de estrategia en tiempo real de Aminoácidos Sampler, el suministro suficiente para aproximadamente 11.000 reacciones individuales. Si Reducción de escala, se recomienda utilizar no menos de la mitad de un kit. Cada aminoácido en el stock se suministra a 1,5 ml, 168 mM, a excepción de la leucina en 140 mM. La composición final de la solución de aminoácidos es:leucina, 5 mM, todos los demás aminoácidos, 6 mM. Esta es la concentración de trabajo 4x.
- Quite todos los 20 aminoácidos entre -20 ° C y descongelar a temperatura ambiente. Una vez descongelado, agitar hasta que se disuelven los aminoácidos, incubando a 37 ° C si es necesario. Después se disuelven aminoácidos, poner todos los aminoácidos en hielo excepto por Asn, Phe, y Cys, que se mantuvo a temperatura ambiente. Cys puede no disolverse completamente.
- En hielo, añadir 12 ml de agua estéril a un tubo Falcon de 50 ml estéril.
- Añadir 1,5 ml de cada aminoácido en el siguiente orden, teniendo cuidado de vórtice el tubo Falcon después de cada adición y mantener la solución en hielo: Ala, Arg, Asn, Asp, Gln, Glu, Gly, His, Ile, Lys, Met, Phe, Pro, Ser, Thr, Val, Trp, Tyr, Leu, Cys. Cys se pueden añadir como una suspensión. Después de la adición, agitar hasta que la solución es relativamente clara, incubando a 37 ° C si es necesario. Cys puede no disolverse completamente.
- Alícuota amino solución de ácido en 50 tubos en26 l cada uno en el hielo. Alícuota el resto a 500 l por tubo en hielo. Los 26 ml de alícuotas se utilizarán para la calibración de extracto, mientras que los 500 ml de alícuotas se utilizarán para la preparación de tampón. Mientras alícuotas, el vórtice de stock principal con frecuencia para evitar una distribución desigual de la suspensión.
- Alícuotas congeladas de Flash en nitrógeno líquido y se almacenan a -80 ° C. Seguridad: Usar gafas de protección; las tapas de los tubos de extracto podrían desprenderse debido a la diferencia de temperatura entre el nitrógeno líquido y la temperatura ambiente.
- Opcional: Realice un ensayo de actividad de la solución de aminoácidos recién hecho en contra de soluciones de aminoácidos previamente realizadas.
3. Energía Preparación de la Solución
Solución La energía se utiliza tanto para la calibración de extracto celular crudo y para la creación de tampón, y se debe preparar en grandes cantidades. La siguiente receta suministra suficiente para aproximadamente 10.000 reacciones individuales. Si la escala hacia abajo, es recommended utilizar no menos de 1/24 de los valores dados aquí. A medida que la solución de la energía es un costo económico significativo, los usuarios de primera vez pueden quieren preparar en escala 1/24. La composición final de la energía solución es: HEPES, pH 8 700 mM, ATP 21 mM, GTP 21 mM, CTP 12,6 mM, UTP 12,6 mM, ARNt 2,8 mg / ml, CoA 3,64 mM, NAD 4,62 mM, AMPc 10,5 mM, ácido folínico 0,95 mM, espermidina 14 mM, 3-PGA 420 mM. Este es 14x la concentración de trabajo. Si se desea, cada elemento individual en la Tabla 4 se puede almacenar a -80 ° C para su uso posterior.
- Eliminar todos los productos químicos en la Tabla 4 de -80 ° C, -20 ° C, o 4 º C a temperatura ambiente durante 30 min.
- Preparar soluciones madre como se describe en la Tabla 4. Ver Material Complementario 1 para las recetas. Coloque todas las soluciones en el hielo después de la preparación.
- En un tubo Falcon de 15 ml, añadir en el siguiente orden, teniendo cuidado de vórtice del tubo Falcon después de cada adición y mantener las soluciones on hielo: 3,6 ml de 2 M de HEPES, 144 l de agua, 1,39 ml de mezcla de nucleótidos, 576 l 50 mg / ml de ARNt, 576 l 65 mM de CoA, 276 l mM de NAD 175, 170 l mM de cAMP 650, 288 l 33,9 mM de ácido folínico , 144 l 1 M espermidina, y 3,09 ml de 1,4 M 3-PGA.
- Energy Solution alícuota en 50 tubos en 7 l cada uno en el hielo. Alícuota el resto a 150 l por tubo en hielo. Los 7 ml de alícuotas se utilizarán para la calibración de extracto, mientras que los 150 ml de alícuotas se utilizarán para la preparación de tampón. Mientras alícuotas, el vórtice de la frecuencia principal.
- Alícuotas congeladas de Flash en nitrógeno líquido y se almacenan a -80 ° C. Seguridad: Usar gafas de protección; las tapas de los tubos de extracto podrían desprenderse debido a la diferencia de temperatura entre el nitrógeno líquido y la temperatura ambiente.
- Opcional: Realice un ensayo de actividad del recién hecha Energy Solution contra Energy Solutions previamente realizadas.
4. Preparación Buffer
BufPreparación fer requiere la realización de crudo celular Extraer Preparación de Aminoácido Preparación de soluciones, y Energía Preparación de soluciones. Cada buffer es exclusivo de un lote de extracto celular crudo. Mg-glutamato, K-glutamato, y la TDT (en ese orden) son optimizados en esta sección para producir reacciones con los niveles máximos de expresión. El siguiente protocolo utiliza una plantilla pre-escrita, TXTL_e (plantilla) _calibration_JoVE.xlsx (material suplementario 3), para calibrar extracto celular crudo pre-preparados y preparar buffer. Sin embargo, también se puede calibrar extracto celular crudo y preparar tampón sin la plantilla mediante la optimización de Mg-glutamato, K-glutamato, y DTT manualmente y la creación de tampón tal que, junto con el extracto, es 75% de un volumen de reacción total. Si la calibración manual, las condiciones finales de la reacción se pueden encontrar en el paso 5.
- Complete el formulario de "Datos Generales".
- Descongelar en hielo 100 mM de Mg-glutamato (4 ° C), 3 M K-glutamato (4 ° C), 6mM Solución de Aminoácidos (26 l, -80 ° C), la solución de Energía (7 l, -80 ° C), 100 mM de DTT (-20 ° C), ADN de control positivo (-20 ° C), 40% de PEG- . 8000 (4 ° C), el extracto celular en bruto (-80 ° C), y agua (4 ° C) Nota: Utilice 1 nM concentración de trabajo pbest-OR2-OR1-Pr-UTR1-deGFP-T500 (plásmido Addgene 40019) para el control positivo (excitación 485 nm, emisión 525 nm), u otra referencia que produce alta intensidad de señal. 4
- Preparar siete 10,5 reacciones mu l, probando una gama de 4-10 mM adicional de Mg-glutamato, por alícuotas de cantidades Conjunto De Mg-glutamato en tubos de microcentrífuga individuales. Nota: Aunque 10,5 reacciones mu l se preparan inicialmente, la reacción final es 10 l.
- Prepare la mezcla maestra, como se indica en la plantilla en "la calibración Mg-glutamato", añadiendo un extra de 80 mM de K-glutamato. Mantenga en hielo y agitar después de la adición de cada artículo Nota:. Los valores dados aquí y en la plantilla sonAdemás de las cantidades de Mg-glutamato, K-glutamato, y DTT presente en el tampón S30B utilizado para hacer extracto celular crudo.
- Añadir mezcla maestra a las muestras que contienen Mg-glutamato y preparar reacciones. Consulte los pasos 5.10 a 5.13 para obtener instrucciones detalladas.
- Ejecutar de reacción a 29 ° C, ya sea en una incubadora o un lector de placas.
- . Determinar la concentración de Mg-glutamato óptimo nivel de la expresión final y la máxima tasa de expresión de la proteína (Figura 4a) Nota: Los tiempos de ejecución varían según el experimento, pero suelen durar menos de 8 horas.
- Repita los pasos 4.2 a 4.7 para K-glutamato en "calibración K glutamato," establecer los niveles de Mg-glutamato a los encontrados en el paso 4.7.
- Repita los pasos 4.2 a 4.7 para la TDT en "Calibración de la TDT", se fija el contenido de Mg-glutamato a los encontrados en el paso 4.7 y los niveles de K-glutamato a los encontrados en el paso 4.8 Nota:. Hemos encontrado que añade la TDT no afecta significativamente al final los niveles de expresión.
- Usovalores encontrados en la calibración en "composición Buffer" para determinar la composición de tampón que estar preparado. Sobre la base de la cantidad de extracto celular crudo producido, una receta mezcla maestra se produce por un importe conjunto de buffers.
- Descongelar las alícuotas que se enumeran en el maestro receta mezcla en hielo. Una vez descongelado, preparar mezcla maestra, manteniendo en hielo y agitación en vórtex después de la adición de cada elemento.
- Alícuota por cantidad indicada en "composición Buffer". Tubos de protección de Flash-congelación en nitrógeno líquido. Aunque alícuotas, vórtice del afuste principal con frecuencia.
- Retire los tubos de nitrógeno líquido utilizando un colador y guardar inmediatamente a -80 ° C. Seguridad: Usar gafas de protección; las tapas de los tubos de extracto podrían desprenderse debido a la diferencia de temperatura entre el nitrógeno líquido y la temperatura ambiente.
5. Ejecución experimental de una reacción TX-TL
Condiciones de reacción finales son: 8.9 a 9.9 mg / ml proteen (a partir de extracto crudo), 4,5 mM-10.5 mM de Mg-glutamato, 40-160 mM de K-glutamato, 0,33-3,33 mM de DTT, 1,5 mM de cada aminoácido excepto leucina, leucina 1,25 mM, HEPES 50 mM, 1,5 mM de ATP y GTP, CTP 0,9 mM y UTP, 0,2 mg / ml de ARNt, 0,26 mM de CoA, 0,33 mM de NAD, 0,75 mM AMPc, 0,068 mM de ácido folínico, espermidina 1 mM, 30 mM de 3-PGA, 2% de PEG-8000. Un TX básica -TL reacción consta de 3 partes (tubos): extracto celular en bruto, de amortiguamiento y de ADN. La relación es: tampón 75% y el extracto, DNA 25% Las reacciones pueden variar en volumen, y usamos 10 l por convención para minimizar el volumen de reacción y permitir que se ejecuta en una placa de 384 pocillos.. Volúmenes más grandes requieren agitación para la oxigenación adecuada. El siguiente protocolo utiliza una plantilla pre-escrita, TXTL_JoVE.xlsx (material suplementario 4), para llevar a cabo una reacción de 10 l. Los productos en púrpura indican los valores de entrada del usuario, y artículos en azul indican reactivos adicionales para añadir a la reacción. Sin embargo, también se puede llevar a cabo una reacción de ingeniohout la plantilla siguiendo las condiciones de reacción descritas anteriormente.
- Complete el formulario de "Datos Generales".
- En "Maestro Preparación Mezclar", inserte el valor del porcentaje de extracto del paso 4.1 en el cuadro de color púrpura.
- Diseñe el experimento in silico usando el "Maestro Preparación Mix" (filas 10 a 17) y "Preparación de ADN" (filas 19 a 50) secciones. En general, las constantes se pueden poner en la sección "Preparación Maestro Mix", mientras que las variables se pueden poner en la sección "Preparación de ADN". Minimizar muestras por experimento para evitar la evaporación de la muestra y experimental sesgo hora de inicio. Vea la Figura 6 para una configuración de ejemplo.
- En "Maestro Preparación Mezclar," añadir reactivos, tales como inductores o proteínas, que irán en todas las muestras a una concentración constante. Comenzando por la fila 14, llene las áreas sombreadas de color azul, manteniendo un reactivo a cada línea. Las unidades son proporciones relativas.
- En la sección "Preparación de ADN", añade ADN que será específi muestraIC. ID de la muestra # 1 y # 2 se corresponden con los controles positivos y negativos, respectivamente. ID de la muestra # 3 y arriba son modificables por el usuario para el ADN, la concentración de valores en ng / l, la longitud en pares de bases, la concentración final deseada en nM, y repeticiones (de 10 reacciones mu l). La cantidad de ADN de valores para alcanzar la concentración final deseada se calcula automáticamente. . El total a través de las sumas de las filas a 10,5 * n, donde n es el número de repeticiones Nota: Aunque el volumen de reacción final es 10 l, los cálculos suponen un volumen total de 10,5 l por reacción, para tener en cuenta el volumen perdido durante el pipeteado.
- En la sección "Preparación de ADN", añadir los reactivos o de ADN adicional que será específico de la muestra a las columnas azules. Concentraciones de ADN Stock en nM se pueden calcular en "Preparación de ADN", mientras que los reactivos específicos de ejemplo requieren cálculo manual basado en un volumen de reacción total de 10.5 * n. Los volúmenes introducidos se restan de la cantidad de agua de la misma fila.
- Retire Numbe necesariaR de tubos de tampón, extracto celular en bruto, y el control positivo bajo "Tubos de descongelar," de -20 ° C o -80 ° C y descongelar en hielo.
- Preparar las muestras de ADN. Para cada muestra Identificación, alícuota a cabo el ADN indicado, el agua, y elementos suministrados por el usuario por la sección "Preparación de ADN" en un tubo de microcentrífuga, a temperatura ambiente Nota:. Para evitar la pérdida de muestra, calibrados recientemente pipetas y de bajo palillo Se recomiendan puntas de pipeta y tubos de microcentrífuga.
- Cuando se descongelan tubos de paso 5.7, preparar la mezcla maestra que consta de tampón, extracto, y ningún artículo globales suministrados por el usuario sobre la base de las cajas de color naranja-sombreada, mantener en hielo y agitación en vórtex después de la adición de cada elemento Nota:. Extracto es extremadamente viscoso. Alícuotas con burbujas se pueden eliminar por centrifugación a 10.000 xg durante 30 segundos a 4 ° C.
- Añadir la cantidad de mezcla maestra se indica en las celdas naranjas en "Preparación de ADN" (columna O) para cada muestra de ADN, y mantenga a temperatura ambientetura. Tratar esto como la hora de inicio de la reacción.
- Vortex cada muestra, y se centrifuga a 10.000 xg durante 30 segundos a temperatura ambiente para reducir cualquier muestra residual y para reducir las burbujas.
- Si la realización de la reacción en tubos de microcentrífuga, incubar directamente a 29 ° C. . De lo contrario, una pipeta 10 l de muestra en una placa de 384 pocillos Nota: Reacciones en volúmenes superiores a los 10 l pueden requerir agitación para la oxigenación.
- Placa Centrifugar a 4000 xg durante 30 segundos a temperatura ambiente para reducir cualquier muestra residual y para reducir las burbujas. Sellar la placa después para evitar la evaporación.
- Ejecute reacción a 29 ° C. Nota: Los tiempos de ejecución varían dependiendo de experimento, pero por lo general una duración de menos de 8 horas.
Resultados
Hemos presentado un protocolo de cinco días para la preparación de una Escherichia coli endógena sistema de expresión libre de células TX-TL basado. Una línea de tiempo de la muestra para la creación de los reactivos - extracto celular en bruto y de amortiguamiento - se puede encontrar en la Figura 1. Una vez creado, éstos se pueden almacenar a -80 ° C durante hasta un año. Una vez creados los reactivos, la configuración experimental y ejecución se puede hacer en menos de 8 horas.
Además, hemos optimizado las condiciones de expresión del sistema de expresión libre de células TX-TL. Otras adiciones proporcionadas por el usuario, tales como tampones o soluciones de ADN, deben ser calibrados para la toxicidad de antemano. Por ejemplo, diferentes métodos de procesamiento de los resultados en plásmidos de expresión diferente debido al contenido de sal. También hemos probado el efecto de tampón de elución Tris-Cl en la eficiencia de reacción (Figura 5).
Un ejemplo de calibración extracto celular crudo, refiriéndose paso 04.01 a 04.09, se muestraen la Figura 4a. En general, nuestros experimentos muestran que el extracto celular en bruto es más sensible a los niveles de Mg-glutamato, seguido de niveles K-glutamato. Para demostrar el sistema de expresión libre de células, construimos y probamos un circuito de retroalimentación negativa basada en tet represión. 26 (Figura 6). En el sistema de expresión libre de células, el mismo recorrido del circuito con y sin ATC muestra un punto final cambia la expresión de 7 veces de reportero deGFP después de ocho horas de expresión. Aunque este experimento no requiere inductores o represores globales, si es necesario se pueden añadir en "Maestro Preparación de la mezcla."
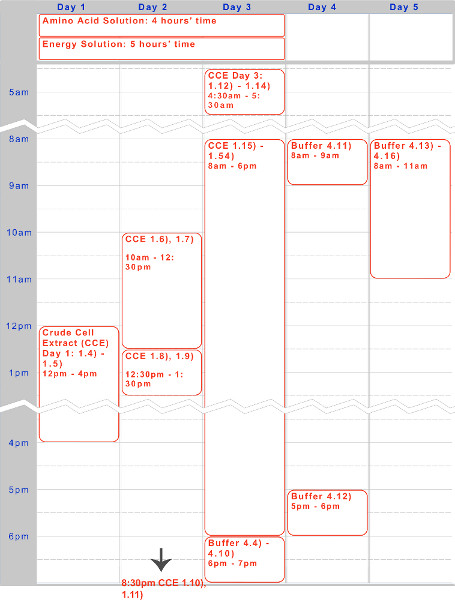
Figura 1. Línea de tiempo para el extracto celular en bruto, solución de aminoácidos, y preparación de la solución de energía. Un Timelin de cinco días E para una ejecución típica del protocolo se ha dado anteriormente, optimizado para incubaciones durante la noche y pasos de trabajo durante el día.
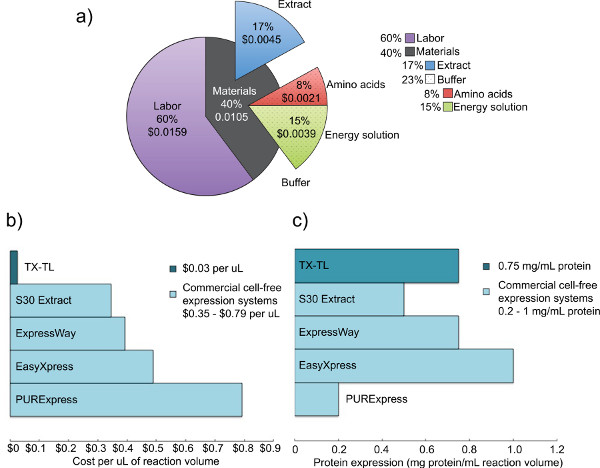
Figura 2. Costos y análisis de la expresión de competir extractos celulares crudos. a) Desglose de los costos de mano de obra y materiales del sistema de expresión libre de células TX-TL. Sobre la base de los costos de reactivos a partir de diciembre de 2012, y los costos de mano de obra de $ 14 por hora. B) Comparación de la TX-TL costos del sistema de expresión libre de células frente a otros sistemas comerciales. Los costos se dividen por l, aunque los volúmenes de reacción pueden variar según el kit. C) Comparación de la TX-TL libre de células rendimiento sistema de expresión frente a otros sistemas comerciales. Rendimiento de expresión de proteínas determinada por las normas del fabricante./ 50762fig2large.jpg "target =" _blank "> Haga clic aquí para ver más grande la figura.

Figura 3. La carga y el procesamiento de un tubo de perlas de latir. a) Demostración de la viscosidad correcta de solución célula-perla. Solución Cell-grano tendrá una viscosidad depende de muchos factores, incluyendo la cantidad de tampón S30A añadido, la cantidad de cuentas de agregados, y el tiempo pasado en el hielo. B) Carga de cordón tubo-batir antes de la centrifugación de mesa rápido. La centrifugación elimina las burbujas acumuladas durante la carga. C) de burbujas a la superficie después de la centrifugación tablero de la mesa. El tamaño de las burbujas puede variar, ya que pueden hacer estallar o eliminar con una punta de pipeta d) de tubo de bolas-superando completamente lleno antes de la limitación.. Se forma un menisco en el cordón de-batiendo tube, y la tapa tiene suficiente para cubrir y causar pequeñas cantidades para llenar en exceso. e) Aparato de filtro cargado correctamente. Estos pueden ser reutilizados. F) Comparación de la correcta vs cordón tubo-superando incorrectamente procesada. El tubo de la izquierda es un tubo bien-beat - que cuenta con un pequeño y bien delineada capa superior, y un sobrenadante claro. El tubo de la derecha es subóptima, en base a la, segunda capa nebuloso más grande y el sobrenadante turbio. Tubos que son subóptimas no deben someterse a un procesamiento adicional.
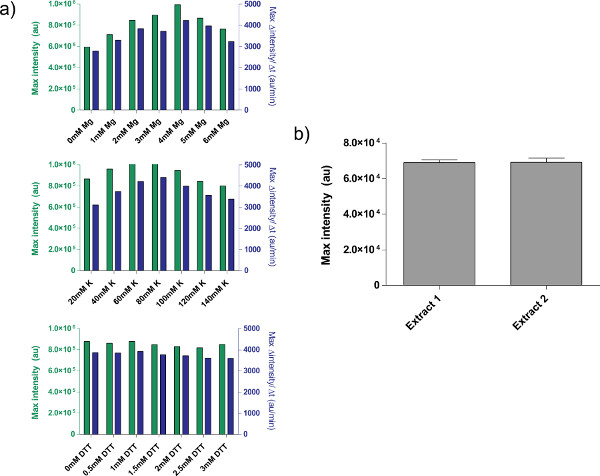
Figura 4. Propiedades de preparaciones de extracto crudo. a) parcelas de calibración típicas para extracto celular en bruto. Extracto crudo se calibra para el Mg-glutamato adicional, K-glutamato, y los niveles de TDT, en ese orden. Se muestra fluorescenc punto finale después de 8 horas, así como la tasa máxima de producción de proteína en base a un promedio móvil de 12 minutos. Sobre la base de estas parcelas, un rango aceptable de Mg-glutamato adicional es 4 mM, K-glutamato es 60-80 mM, y DTT es 0-3 mM. Tenga en cuenta que cada extracto en bruto necesita ser calibrado de forma independiente para estas tres variables. B) Variación de preparaciones de extractos. Se muestra de punto final de fluorescencia de dos extractos crudos preparados en fechas diferentes; barras de error son de 1 desviación estándar de tres carreras independientes en diferentes días. Haga clic aquí para ver más grande la figura .
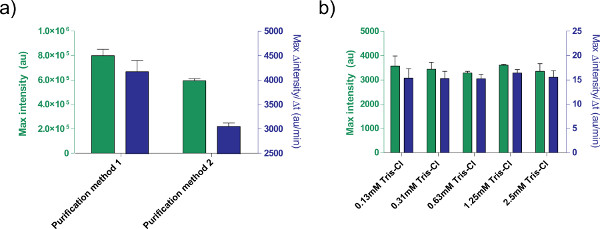
Figura 5. Efectos de la solución de ADN en la eficacia de la expresión. a) Comparación de dos purificación diferentemétodos para el procesamiento de plásmidos. 1 nM de pbest-OR2-OR1-Pr-UTR1-deGFP-T500 se prepara usando solamente un kit de Spin Miniprep Qiaprep (método de purificación 1) o post-procesado con un kit de purificación de PCR QIAquick (método de purificación 2). Se muestra la fluorescencia de punto final después de 8 horas, así como la tasa máxima de producción de proteína en base a un promedio móvil de 12 minutos. Las barras de error son de 1 desviación estándar de cuatro carreras independientes en diferentes días. B) Efecto del tampón de elución (Tris-Cl). Diferentes concentraciones de Tris-Cl se comparan en una reacción de expresión libre de células basado en la expresión de 1 nM de pbest-OR2-OR1-Pr-UTR1-deGFP-T500. Las concentraciones dadas son concentraciones finales de Tris-Cl en la reacción; tampón de elución utilizado es 10 mM de Tris-Cl. Las barras de error son de 1 desviación estándar de tres carreras independientes en diferentes días. Haga clic aquí para ver más grande la figura .
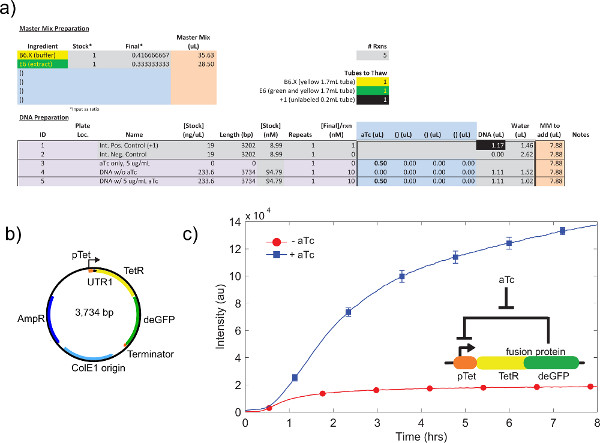
Figura 6. Muestra de ejecución TX-TL de un bucle de retroalimentación negativa. a) Ejemplo de configuración de una reacción de ejecución libre de células. Tests "en" contra "de" estado de la curva de retroalimentación negativa, con controles positivos y negativos. B) El plásmido mapa de bucle de retroalimentación negativa. C) Los resultados representativos. Los datos reflejan experimento en a) y b), con el control negativo se resta de la señal. Circuito genético muestra en la inserción. Las barras de error son de 1 desviación estándar de tres carreras independientes en diferentes días. Haga clic aquí para ver más grande la figura .
| Nombre | Concentración | Cantidad | Esterilización | Notas |
| El cloranfenicol (Cm) | 34 mg / ml en etanol | 1 ml | Se esteriliza con filtro (0.22 M) | Puede ser hecho en volúmenes más grandes almacenados a -20 ° C para su uso posterior. |
| 2xYT + P + placa de agar Cm | 31 g / L 2xYT, 40 mM de fosfato de potasio dibásico, 22 mM de fosfato de potasio monobásico, 34 mg / ml de cloranfenicol | 1 placa | Autoclave | |
| 2xYT + P multimedia | 31 g / L 2xYT, 40 mM de fosfato de potasio dibásico, 22 mM de fosfato de potasio monobásico | 4 L | Autoclave |
Tabla 1. Reactivos para el día 1 del protocolo de extracto crudo de la célula.
| Nombre | Concentración | Cantidad | Esterilización | Notas |
| Tris base | 2 M | 250 ml | Se esteriliza con filtro (0.22 M) o en autoclave | Se puede almacenar a temperatura ambiente. |
| TDT | 1 M | 6 ml | Se esteriliza con filtro (0.22 M) | Puede ser hecho en volúmenes más grandes y se almacenó a -20 ° C para su uso posterior. |
| Búfer S30A | 14 mM de Mg-glutamato, 60 mM de K-glutamato, mM de Tris, pH 7,7 50 | 2 L | Autoclave | Para llegar a pH 7,7, valorar con ácido acético. Añadir TDT a 2 mM de concentración final, justo antes de su uso. Almacenar a 4 ° C. |
| Búfer S30B | 14 mM de Mg-glutamato, 60 mM de K-glutamato, ~ 5 mM de Tris, pH 8,2 | 2 L | Autoclave | Para llegar a pH 8,2, se valora con 2 MTris. Añadir TDT a 1 mM concentración final justo antes de su uso. Almacenar a 4 ° C. |
Tabla 2. Reactivos para el día 2 del protocolo de extracto crudo de la célula.
| Halcón | ||||
| 1 | 2 | 3 | 4 | |
| Vacío 50 ml Falcon (g) | ||||
| 50 ml Falcon con pellet (g) | ||||
| Masa de pellets (50 ml Falcon con pellet - vacío 50 ml Falcon) (g) | ||||
| Volumen de tampón S30A añadir (masa de pellets * 0.9) (ml) | ||||
| Masa total de cuentas para agregar (masa de pellets * 5.0) (g) | ||||
Tabla 3. Búfer S30A y calculadora de masa del grano, para el día 3 de protocolo de extracto crudo de la célula.
| Nombre | Concentración | Cantidad | Esterilización | Notas |
| HEPES | 2 M, pH 8 | 4 ml | Ninguno | Para llegar a un pH de 8, titule con KOH. |
| Nucleotide Mix | 156 mM de ATP y GTP, CTP y UTP mM, de pH 7,5 94 | 1,5 ml | Ninguno | Para llegar a un pH de 7,5, titule con KOH. |
| tRNA | 50 mg / ml | 600 l | Ninguno | |
| CoA | 65 mM | 600 l | Ninguno | |
| NAD | 175 mM, pH 7.5-8 | 300 l | Ninguno | Para llegar a pH 7,5-8, titule con Tris 2 M. |
| cAMP | 650 mM, pH 8 | 200 l | Ninguno | Para llegar a un pH de 8, titule con Tris 2 M. |
| El ácido folínico | 33,9 mM | 300 l | Ninguno | Aunque se necesitan sólo 300 l, receta en suplementario es de 1,15 ml. |
| La espermidina | 1 M | 150 l | Ninguno | Almacenar a 4 ° C, el calor a 37 ° C para fundir. |
| 3-PGA | 1,4 M, pH 7,5 | 3,2 ml | Ninguno | Para llegar a un pH de 7,5, se valora con Tris 2 M. |
Tabla 4. Reactivos para prepararse para el protocolo Energy Solution.
Material complementario 1. Recetas para los artículos.
El cloranfenicol, 34 mg / ml: Preparar 0,51 g de cloranfenicol y añadir etanol a 15 ml. Se esteriliza con filtro (0.22 M), alícuota de 1 ml tubos, almacenar a -20 ° C para su uso posterior.
2xYT + P + placa de agar Cm: Preparar 1,24 g 2xYT, 1,6 ml de fosfato de potasio dibásico @ solución 1 M, 0,88 ml de solución de fosfato monobásico de potasio @ 1 M, 0,6 g de agar, y el agua a 40 ml. Autoclave. Dejar enfriar a 50 ° C y añadir 40 l Cm. Alícuota de 25 ml en una placa de 100 x 15 mm Petri, y deje que se enfríe durante una hora.
2xYT + P los medios de comunicación: Preparar 124 g 2xYT, 160 ml de fosfato de potasio dibásico @ solución 1 M, solución de 88 ml de fosfato de potasio monobásico @ 1 M, y agua a 4 L. Aliquot hacia 2 x 1,88 L y 0,24 L. Autoclave.
Tris base, 2 M: Prepare 60,57 g Tris base y agua hasta 250 ml. Esterilizar, almacenar a temperatura ambiente para su uso posterior.
TDT, 1 M: Preparar 2,31 g TDT y el agua a 15 ml. Se esteriliza con filtro (0.22 M), alícuota de 1 ml tubos, almacenar a -20 ° C para su uso posterior.
Búfer S30A: Prepare 10,88 g Mg-glutamato y 24,39 g K-glutamato, 50 ml de Tris a 2 M, ácido acético (pH 7,7), y el agua a 2 L. Autoclave, se almacenan a 4 ° C, añadir 4 ml 1 M DTT antes de su uso.
Búfer S30B: Prepare 10,88 g Mg-glutamato y 24,39 g K-glutamato, Tris 2 M (pH 8,2), y el agua a 2 L. Autoclave, se almacenan a 4 ° C, añadir 2 ml de 1 M DTT antes de su uso.
HEPES: Preparar 1,91 g de HEPES (MW 238,21), KOH (a pH 8), y agua a 4 ml.
ARNt: Prepare 30 mg de ARNt y agua a 600 l.
CoA: Preparar 30 mg de CoA (MW 767,53) y agua a 600 l.
NAD: Añadir 34,83 mg de NAD (MW 663,43), Tris a 2 M (a pH 7,5-8), y agua a 300 l. (Añadir 27 l de Tris a 2 M para llevar la solución a pH 7,5-8).
cAMP: Agregar 42,80 mg de cAMP (PM 329,22), Tris 2 M (pH 8), y el agua a 200 l. (Añadir 73 l de Tris a 2 M para llevar la solución a pH 8).
Ácido folínico (33,9 mM): Para 20 mg de sal de calcio de ácido folínico sólido (MW 511.5), añadir 1,15 ml de agua.
La espermidina: Preparar 23,55 l de espermidina (MW 145,25) y agua a 150 l. Preparar a temperatura ambiente después de la fusión brevemente a 37 ° C.
3-PGA: Añadir 1,03 g de 3-PGA (MW 230,02), Tris a 2 M (a pH 7,5), y agua a 3,2 ml. (Añadir 1,73 ml de Tris a 2 M para llevar la solución a pH 7,5).
Nucleotide Mix: Añadir 145 mg de ATP dipotásico dihidrato de sal (MW 619.4), 133 mg de sal disódica GTP (MW 567,14), 79,4 mg de sal disódica dihidrato CTP (MW 563,16), 82,6 mg de dihidrato de la sal trisódica UTP (MW 586,12) , KOH a 15% de dilución (a pH 7,5), y agua a 1,5 ml. (Añadir 353 l de KOH a 15% de dilución para llevar la solución a pH 7,5).
Material suplementario 2. Ensayo de Bradford.
- Eliminar los agentes Bradford de 4 ° C y se pondrá a la temperatura ambiente.
- Preparar 50 l de BSA estándar a 1 mg / ml y a 0,1 mg / ml.
- Preparar 40 l de 20x de dilución del extracto de la etapa 1.47.
- Añadir 800 l de agua a 7 cubetas.
- Preparar cubetas estándar para 0 mg / ml, 1 mg / ml (10 l 0,1 mg / ml de BSA), 2 mg / ml (20 l 0,1 mg / ml de BSA), 4 mg / ml (4 l 1 mg / ml de BSA) , 6 mg / ml (6 l 1 mg / ml de BSA).
- Preparar cubetas experimentales para 2 l de muestra y 4 l de muestra.
- Añadir 200 l de agente de Bradford a cada cubeta y mezclar bien bY pipeteado. Incubar a temperatura ambiente durante al menos 10 min.
- Producir curva estándar a una DO de 595 nm usando cubetas de paso 6.5. Rechazar curva estándar si r 2 <0,95.
- Determinar la concentración del extracto a una DO de 595 nm utilizando cubetas desde el paso 6.6.
Material complementario 3. Tampón de hoja de cálculo de calibración.
Ver TXTL_e (plantilla) _calibration_JoVE.xlsx .
Material suplementario 4. Expresión libre de células ejecutar hojas de cálculo.
Discusión
Sistema de expresión libre de células TX-TL basado La Escherichia coli endógeno descrito aquí es una reacción simple-to-run de tres tubos que pueden tener menos de ocho horas de creado para la recopilación de datos. El proceso de creación de todos los reactivos requiere un total de cinco días de tiempo (con requisitos laborales importantes en un solo día), pero produce extracto crudo de 3.000 reacciones y el tampón de decisiones reactivos para 10.000 reacciones (Figura 1). Por otra parte, el extracto crudo y amortiguan de decisiones reactivos son estables durante al menos 1 año a -80 ° C, lo que permite múltiples usos de una preparación de 4. A $ 0,11 por 10 l de reacción (0,26 dólares incluyendo mano de obra), los costos son 98% más bajo que comparables sistemas comerciales (Figura 2).
Hay algunas limitaciones sin resolver, sin embargo, para el sistema. La eficiencia final de cada preparación de extracto celular crudo puede variar en función de la aptitud del usuario y de las condiciones ambientales, aunque tla variación del rendimiento RUEBAS es entre 5-10% (Figura 4b). Como resultado, la variabilidad de lote a lote en tanto la expresión de punto final y en la dinámica de la expresión son de esperar. Estas variaciones probablemente se mantendrá hasta que el extracto está completamente caracterizado, o hasta que la creación de extracto está totalmente automatizado. Si el sistema de expresión libre de células se utiliza para llevar a cabo experimentos cuantitativos sensibles, es aconsejable ejecutar todos los experimentos con el mismo lote de extracto celular crudo. El rendimiento de un solo lote extracto celular crudo, unos 3.000 reacciones, debe ser suficiente para los cursos típicas experimentales. Aunque sospechamos variación puede ser removido por la ampliación y la automatización del procedimiento, tales intentos implicarían una inversión sustancial de recursos.
Además, si bien los niveles de punto final de expresión son razonablemente fáciles de determinar, aún queda trabajo por hacer en la dinámica de la comprensión intrínsecas al sistema libre de células. Se sabe que tanto COMPETENCIA recursosn de recursos y limitación puede afectar la dinámica de expresión. Por ejemplo, limitada Sigma endógeno 70 puede dar lugar a un régimen de saturar con el aumento de la plantilla de ADN que produce un perfil de expresión análoga a la de nucleótidos o aminoácidos agotamiento. 9,27 Sin embargo, la dinámica no tienen que ser entendidos completamente para utilizar el sistema. Para los aumentos puros de rendimiento, optimización se puede hacer mediante métodos de aprendizaje automático. 28 Cuestiones de competencia por los recursos y la limitación pueden ser abordadas por los modelos matemáticos verificó utilizando datos experimentales.
El protocolo que se presenta aquí está optimizado para una cepa BL21-Rosetta2, pero es generalizable a otras E. cepas de E. coli. Modificaciones en BL21-Rosetta2, tales como la eliminación del gen que codifica la proteasa lon y la adición de genes que codifican los ARNt raros, permiten para la producción máxima de proteínas. Hemos tratado el protocolo con otras dos cepas de extracto - sólo BL21 y un golpe de gracia-una BL21 trxAd encontraron 50% menos de producción de proteína. Nuestra hipótesis es que los rendimientos disminuyen de manera similar cuando se utilizan otras cepas. Otros cambios en los parámetros, tales como la conmutación de medio de crecimiento 2xYT para LB y otros caldos ricos, han dado lugar a la producción de proteína disminuido.
Sistemas de expresión sin células utilizando tanto la maquinaria de transcripción-traducción endógeno y exógeno y los mecanismos de regulación tienen amplias aplicaciones en proteínas y expresión metabolito y de la biología sintética. 3,29 En vez de estar limitado a los circuitos T7-regulada, se puede imaginar la producción de biomoléculas complejas en un entorno controlable por el usuario usando una mezcla de E. nativa promotores coli y mecanismos de transcripción y regulación suministrados exógenamente. Sin limitaciones de la división celular y el metabolismo, la variabilidad en los circuitos sintéticos tales como el Represilador o en las vías metabólicas de ingeniería tales como los que producen artemisinina se puede reducir o mejor entendida. 30,31 Have utilizado estas ventajas para implementar interruptores genéticos, así como para entender sigma factor de secuestro de 9,32 Dicha tecnología también puede formar la columna vertebral de la "mínima" o células "artificiales" -. pequeño, bien caracterizado y unidades encarnados autosuficientes de extracto de 33,34.
En última instancia, esperamos usos inmediatos de este sistema de expresión libre de células endógenas como un entorno de creación de prototipos para la biología sintética. Apodado el "TX-TL breadboard biomolecular", el sistema de expresión libre de células proporciona un entorno controlable donde los circuitos sintéticos cuyo destino final es la expresión in vivo pueden someterse a rondas de prototipos - ciclos de pruebas en el plásmido básico, lineal, o ADN sintetizado químicamente, seguido por el análisis y la modificación rápida. Rondas prototipos pueden ser ayudados por los modelos matemáticos predictivos que se están desarrollando actualmente. Mediante la eliminación de la clonación y la manipulación en vivo para circuitos no finales, anticipamos ingenieríatiempos de ciclo niería para ser reducidos a 1-3 días en lugar de los estándar actuales semana.
Divulgaciones
Los autores declaran que no tienen intereses financieros en competencia.
Agradecimientos
Agradecemos Jongmin Kim, Dan Siegal-Gaskins, Anu Thubagere, y Enoc Yeung asistencia racionalización del protocolo, y Clare Chen y Lee Barclay para la ayuda en las primeras etapas del proyecto. Este material está basado en trabajo apoyado en parte por la Agencia de Proyectos de Investigación Avanzada de Defensa (DARPA / MTO) Programa Living Fundiciones, número de contrato HR0011-12-C-0065 (DARPA / CMO.ZZS está también apoyado por un Científico Médico UCLA / Caltech Programa de becas de formación y de un Departamento de Defensa, la Fuerza Aérea Oficina de Investigación Científica, Defensa Nacional de Ciencia e Ingeniería de Posgrado (NDSEG) Fellowship, 32 CFR 168. Las opiniones y conclusiones contenidas en este documento son las de los autores y no debe interpretarse como la representación de oficialmente las políticas, ya sea expresa o implícita, de la Agencia de Proyectos de Investigación Avanzada de Defensa o el Gobierno de los EE.UU..
Materiales
| Name | Company | Catalog Number | Comments |
| 2xYT | MP biomedicals | 3012-032 | |
| 3-PGA | Sigma-Aldrich | P8877 | |
| ATP | Sigma-Aldrich | A8937 | |
| Bacto-agar | BD Diagnostics | 214010 | |
| Bead-beating tubes (polypropylene microvials) | BioSpec | 522S | |
| Beads, 0.1mm dia. | BioSpec | 11079101 | |
| BL21 Rosetta 2 E. coli strain | Novagen | 71402 | |
| Bradford BSA Protein Assay Kit | Bio-rad | 500-0201 | |
| cAMP | Sigma-Aldrich | A9501 | |
| Chloramphenicol | Sigma-Aldrich | C1919 | |
| CoA | Sigma-Aldrich | C4282 | |
| CTP | USB | 14121 | |
| Cuvettes, 1.5ml | Fisher | 14-955-127 | |
| DTT | Sigma-Aldrich | D0632 | |
| Folinic acid | Sigma-Aldrich | F7878 | |
| GTP | USB | 16800 | |
| HEPES | Sigma-Aldrich | H6147 | |
| K-glutamate | Sigma-Aldrich | G1149 | |
| Mg-glutamate | Sigma-Aldrich | 49605 | |
| Micro Bio-Spin Chromatography Columns | Bio-Rad | 732-6204 | |
| NAD | Sigma-Aldrich | N6522 | |
| Nunc 384-well optical bottom plates | Thermo-Scientific | 142761 | |
| Nunc sealing tape | Thermo-Scientific | 232701 | |
| PEG-8000 | Promega | V3011 | |
| Potassium phosphate dibasic solution | Sigma-Aldrich | P8584 | |
| Potassium phosphate monobasic solution | Sigma-Aldrich | P8709 | |
| RTS Amino Acid Sampler | 5 Prime | 2401530 | |
| Slide-A-Lyzer Dialysis Cassettes, 10k MWCO (Kit) | Thermo-Scientific | 66382 | |
| Spermidine | Sigma-Aldrich | 85558 | |
| Tris base | Fischer | BP1521 | |
| tRNA (from E. coli) | Roche Applied Science | MRE600 | |
| UTP | USB | 23160 | |
| 1L Centrifuge Bottle | Beckman-Coulter | A98813 | This is specific for Avanti J-series; obtain equivalent size for centrifuge in use. |
| 4L Erlenmeyer Flask | Kimble Chase | 26500-4000 | |
| Avanti J-26XP Centrifuge | Beckman-Coulter | 393127 | Or 1L-capable centrifuge equivalent. |
| Forma 480 Orbital Shaker | Thermo Scientific | 480 | Or chest-size 6x4L shaker equivalent. |
| JLA-8.1000 Rotor | Beckman-Coulter | 363688 | Or 1L-capable, 5000 x g rotor equivalent for centrifuge. |
| Mini-Beadbeater-1 | BioSpec | 3110BX | |
| Supplemental Material 1. Recipes for Items. Chloramphenicol, 34 mg/ml: Prepare 0.51 g chloramphenicol and add ethanol to 15 ml. Filter sterilize (0.22 μM), aliquot to 1 ml tubes, store at -20 °C for later use. 2xYT+P+Cm agar plate: Prepare 1.24 g 2xYT, 1.6 ml potassium phosphate dibasic solution @ 1 M, 0.88 ml potassium phosphate monobasic solution @ 1 M, 0.6 g agar, and water to 40 ml. Autoclave. Let cool to 50 °C and add 40 μl Cm. Aliquot 25 ml into a 100x15 mm petri dish, and let cool for an hour. 2xYT+P media: Prepare 124 g 2xYT, 160 ml potassium phosphate dibasic solution @1 M, 88 ml potassium phosphate monobasic solution @ 1 M, and water to 4 L. Aliquot out into 2x1.88 L and 0.24 L. Autoclave. Tris base, 2 M: Prepare 60.57 g Tris base and water to 250 ml. Sterilize, store at RT for later use. DTT, 1 M: Prepare 2.31 g DTT and water to 15 ml. Filter sterilize (0.22 μM), aliquot to 1 ml tubes, store at -20 °C for later use. S30A buffer: Prepare 10.88 g Mg-glutamate and 24.39 g K-glutamate, 50 ml Tris at 2M, acetic acid (to pH 7.7), and water to 2 L. Autoclave, store at 4 °C, add 4 ml 1 M DTT before use. S30B buffer: Prepare 10.88 g Mg-glutamate and 24.39 g K-glutamate, Tris at 2 M (to pH 8.2), and water to 2 L. Autoclave, store at 4 °C, add 2 ml 1 M DTT before use. HEPES: Prepare 1.91 g HEPES (MW 238.21), KOH (to pH 8), and water to 4 ml. tRNA: Prepare 30 mg of tRNA and water to 600 μl. CoA: Prepare 30 mg of CoA (MW 767.53) and water to 600 μl. NAD: Add 34.83 mg of NAD (MW 663.43), Tris at 2 M (to pH 7.5-8), and water to 300 μl. (Add 27 μl of Tris at 2 M to bring the solution to pH 7.5-8). cAMP: Add 42.80 mg of cAMP (MW 329.22), Tris at 2 M (to pH 8), and water to 200 μl. (Add 73 μl of Tris at 2 M to bring the solution to pH 8). Folinic Acid (33.9 mM): To 20 mg of solid folinic acid calcium salt (MW 511.5), add 1.15 ml water. Spermidine: Prepare 23.55 μl of spermidine (MW 145.25) and water to 150 μl. Prepare at room temperature after melting briefly at 37 °C. 3-PGA: Add 1.03 g of 3-PGA (MW 230.02), Tris at 2 M (to pH 7.5), and water to 3.2 ml. (Add 1.73 ml of Tris at 2 M to bring the solution to pH 7.5). Nucleotide Mix: Add 145 mg of ATP dipotassium salt dihydrate (MW 619.4), 133 mg of GTP disodium salt (MW 567.14), 79.4 mg of CTP disodium salt dihydrate (MW 563.16), 82.6 mg of UTP trisodium salt dihydrate (MW 586.12), KOH at 15% dilution (to pH 7.5), and water to 1.5 ml. (Add 353 μl of KOH at 15% dilution to bring the solution to pH 7.5). Supplemental Material 2. Bradford Assay.
See TXTL_e(template)_calibration_JoVE.xlsx. Supplemental Material 4. Cell-free expression run spreadsheet. See TXTL _JoVE.xlsx. | |||
Referencias
- Noireaux, V., Bar-Ziv, R., Libchaber, A. Principles of cell-free genetic circuit assembly. Proceedings of the National Academy of Sciences of the United States of America. 100, 12672-12677 (2003).
- He, M. Y., He, Y. Z., Luo, Q., Wang, M. R. From DNA to protein: No living cells required. Process Biochem. 46, 615-620 (2011).
- Forster, A. C., Church, G. M. Synthetic biology projects in vitro. Genome Res. 17, 1-6 (1101).
- Shin, J., Noireaux, V. Efficient cell-free expression with the endogenous E. Coli RNA polymerase and sigma factor 70. Journal of biological engineering. 4, 8 (2010).
- Shimizu, Y., et al. Cell-free translation reconstituted with purified components. Nat Biotechnol. 19, 751-755 (2001).
- Shin, J., Noireaux, V. An E. coli Cell-Free Expression Toolbox: Application to Synthetic Gene Circuits and Artificial Cells. Acs Synth Biol. 1, 29-41 (2012).
- Shin, J., Noireaux, V. Study of messenger RNA inactivation and protein degradation in an Escherichia coli cell-free expression system. Journal of biological engineering. 4, 9 (2010).
- Shin, J., Jardine, P., Noireaux, V. Genome Replication, Synthesis, and Assembly of the Bacteriophage T7 in a Single Cell-Free Reaction. Acs Synth Biol. 1, 408-413 (2012).
- Siegal-Gaskins, D., Noireaux, V., Murray, R. M., Pao, L., Abramovitch, D. Biomolecular resource utilization in elementary cell-free gene circuits. , 1531-1536 (2013).
- Karzbrun, E., Shin, J., Bar-Ziv, R. H., Noireaux, V. Coarse-grained dynamics of protein synthesis in a cell-free system. Physical review letters. 106, 048104 (2011).
- Hoagland, M. B., Stephenson, M. L., Scott, J. F., Hecht, L. I., Zamecnik, P. C. Soluble Ribonucleic Acid Intermediate in Protein Synthesis. J Biol Chem. 231, 241-257 (1958).
- Wood, W. B., Berg, P. Effect of Enzymatically Synthesized Ribonucleic Acid on Amino Acid Incorporation by a Soluble Protein-Ribosome System from Escherichia Coli. Proceedings of the National Academy of Sciences of the United States of America. 48, 94 (1962).
- Zubay, G. In-Vitro Synthesis of Protein in Microbial Systems. Annu Rev Genet. 7, 267-287 (1973).
- Pratt, J. M., Hames, B. D., Higgins, S. J. . Transcription and Translation: A Practical Approach. , 179-209 (1984).
- Kim, H. C., Kim, D. M. Methods for energizing cell-free protein synthesis. Journal of bioscience and bioengineering. 108, 1-4 (2009).
- Michel-Reydellet, N., Calhoun, K., Swartz, J. Amino acid stabilization for cell-free protein synthesis by modification of the Escherichia coli genome. Metabolic engineering. 6, 197-203 (2004).
- Liu, D. V., Zawada, J. F., Swartz, J. R. Streamlining Escherichia coli S30 extract preparation for economical cell-free protein synthesis. Biotechnology progress. 21, 460-465 (2005).
- Andrianantoandro, E., Basu, S., Karig, D. K., Weiss, R. Synthetic biology: new engineering rules for an emerging discipline. Molecular systems biology. 2, 2006.0028 (2006).
- Kwok, R. Five hard truths for synthetic biology. Nature. 463, 288-290 (2010).
- Tabor, S., Richardson, C. C. A bacteriophage T7 RNA polymerase/promoter system for controlled exclusive expression of specific genes. Proceedings of the National Academy of Sciences of the United States of America. 82, 1074-1078 (1985).
- Lewicki, B. T., Margus, T., Remme, J., Nierhaus, K. H. Coupling of rRNA transcription and ribosomal assembly in vivo. Formation of active ribosomal subunits in Escherichia coli requires transcription of rRNA genes by host RNA polymerase which cannot be replaced by bacteriophage T7 RNA polymerase. Journal of molecular biology. 231, 581-593 (1993).
- Iskakova, M. B., Szaflarski, W., Dreyfus, M., Remme, J., Nierhaus, K. H. Troubleshooting coupled in vitro transcription-translation system derived from Escherichia coli cells: synthesis of high-yield fully active proteins. Nucleic acids research. 34, e135 (2006).
- Kigawa, T., et al. Preparation of Escherichia coli cell extract for highly productive cell-free protein expression. Journal of structural and. 5, 63-68 (2004).
- Matsuda, T., et al. Improving cell-free protein synthesis for stable-isotope labeling. Journal of biomolecular. NMR. 37, 225-229 (2007).
- Sitaraman, K., et al. A novel cell-free protein synthesis system. Journal of biotechnology. 110, 257-263 (2004).
- Becskei, A., Serrano, L. Engineering stability in gene networks by autoregulation. Nature. 405, 590-593 (2000).
- Maeda, H., Fujita, N., Ishihama, A. Competition among seven Escherichia coli sigma subunits: relative binding affinities to the core RNA polymerase. Nucleic acids research. 28, 3497-3503 (2000).
- Caschera, F., et al. Coping with complexity: machine learning optimization of cell-free protein synthesis. Biotechnology and bioengineering. 108, 2218-2228 (2011).
- Hodgman, C. E., Jewett, M. C. Cell-free synthetic biology: thinking outside the cell. Metabolic engineering. 14, 261-269 (2012).
- Elowitz, M. B., Leibler, S. A synthetic oscillatory network of transcriptional regulators. Nature. 403, 335-338 (2000).
- Tsuruta, H., et al. High-level production of amorpha-4,11-diene, a precursor of the antimalarial agent artemisinin, in Escherichia coli. Plos One. 4, e4489 (2009).
- Gardner, T. S., Cantor, C. R., Collins, J. J. Construction of a genetic toggle switch in Escherichia coli. Nature. 403, 339-342 (2000).
- Jewett, M. C., Forster, A. C. Update on designing and building minimal cells. Current opinion in biotechnology. 21, 697-703 (2010).
- Noireaux, V., Libchaber, A. A vesicle bioreactor as a step toward an artificial cell assembly. Proceedings of the National Academy of Sciences of the United States of America. 101, 17669-17674 (2004).
Erratum
Formal Correction: Erratum: Protocols for Implementing an Escherichia coli Based TX-TL Cell-Free Expression System for Synthetic Biology
Posted by JoVE Editors on 1/01/1970. Citeable Link.
A correction to Figure 5's legend has been made for the article Protocols for Implementing an Escherichia coli Based TX-TL Cell-Free Expression System for Synthetic Biology. Method 1 and method 2 have been switched.Z
The figure legend was update from:
Figure 5. Effects of DNA solution on expression efficiency. a) Comparison of two different purification methods for processing plasmids. 1 nM of pBEST-OR2-OR1-Pr-UTR1-deGFP-T500 is prepared using only a QiaPrep Spin Miniprep Kit (Purification method 1) or post-processed with a QiaQuick PCR purification kit (Purification method 2).
to
Figure 5. Effects of DNA solution on expression efficiency. a) Comparison of two different purification methods for processing plasmids. 1 nM of pBEST-OR2-OR1-Pr-UTR1-deGFP-T500 is prepared using only a QiaPrep Spin Miniprep Kit (Purification method 2) or post-processed with a QiaQuick PCR purification kit (Purification method 1).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados