Method Article
Protocolos para implementação de uma
* Estes autores contribuíram igualmente
Neste Artigo
Erratum Notice
Resumo
Este protocolo de cinco dias descreve todos os passos, equipamentos e software suplementar necessário para a criação e execução de um Escherichia coli endógena eficiente baseado TX-TL livre de células sistema de expressão a partir do zero. Com reagentes, o protocolo leva 8 horas ou menos para configurar uma reação, coletar e processar dados.
Resumo
Sistemas ideais de expressão isentos de células, pode, teoricamente, emular um ambiente celular in vivo de uma forma controlada em plataforma vitro. Uma Isto é útil para a expressão de proteínas e circuitos genéticos de uma forma controlada, bem como para proporcionar um ambiente de prototipagem por biologia sintética. 2,3 Para alcançar o segundo objetivo, sistemas de expressão livre de células que preservam endógeno Escherichia coli mecanismos de transcrição-tradução é capaz de refletir com mais precisão in vivo dinâmica celular do que aqueles baseados em T7 RNA polimerase de transcrição. Nós descrevemos a preparação e execução de um E. endógena eficiente 4,5 base coli transcrição-tradução (TX-TL) livre de células sistema de expressão que pode produzir quantidades equivalentes de proteína como sistemas baseados em T7 em uma redução de custos de 98% para os sistemas comerciais similares. A preparação de tampões e extrato celular bruto são descrito, bem como a realização de umtrês tubo de reacção TX-TL. O protocolo inteiro leva cinco dias para preparar e rende material suficiente para até 3000 reações individuais em uma preparação. Depois de preparado, cada reação leva menos de 8 horas de configuração para a coleta de dados e análise. Mecanismos de regulação e transcrição exógeno ao E. coli, tais como repressores lac / tet e T7 ARN polimerase, pode ser completada. 6 propriedades endógenas, tais como ARNm e as taxas de degradação de ADN, pode também ser ajustado. 7 O sistema de expressão isento de células TX-TL tem sido demonstrado para os grandes montagem escala circuito, explorando fenômenos biológicos, e expressão de proteínas em ambos os promotores T7 e endógenos. 6,8 modelos matemáticos de acompanhamento estão disponíveis. 9,10 O sistema resultante tem aplicações exclusivas em biologia sintética como um ambiente de prototipagem, ou "TX- placa de ensaio TL biomolecular ".
Introdução
Expressão tecnologia sem celular começou na década de 1950 como puramente translacional, avançando anos mais tarde para incluir mecanismos de transcrição-tradução juntamente com DNA bacteriófago T7. 11,12 Desde então, foram feitos inúmeros esforços para otimizar a criação de extrato celular bruto (ou E extrato. coli S30). 13,14 Essas otimizações incluem prolongar a síntese de proteína livre de células através da regeneração de ATP ou modificações de tensão e reduzindo o tempo de protocolo e custo. existem 15-17 sistemas alternativos de expressão livre de celulares que usam reconstituído componentes em vez de bruto extracto de células para a expressão. 5 Ambos extracto celular bruto e métodos de reconstituição foram desenvolvidos para uso comercial.
Com o advento da biologia sintética, existe um aumento da necessidade para uma plataforma bem caracterizado para testar e expressar módulos e circuitos biológicas modificadas. 18,19 Esta plataforma tem de serversátil, bem caracterizada, simples de manipular, e focada em componentes fornecidos pelo usuário. Apesar de ter sido desenvolvido meio século antes, os sistemas livres de células baseado em E. coli intrinsecamente compartilhar esses requisitos, pois eles são uma representação simplificada em vitro de processos celulares, sem a complexidade de crescimento e metabolismo. Além disso, todo o conhecimento fundamental de trabalho em vivo em E. coli aplica prontamente para E. sistemas livres de células de E. coli.
Embora os sistemas de expressão livre de células pode ter aplicações em biologia sintética, até à data o objetivo da maioria dos sistemas de expressão livre de células tem sido a maximização de proteína e rendimento de metabólito. Isto é conseguido através da utilização do bacteriófago T7 de transcrição de sequências conduzidos pelos promotores de T7. 20 Embora a expressão eficiente e robusta, estes sistemas servem um propósito altamente especializado. Métodos de regulação celular são limitadas, modelos de DNA-alvo deve serredesenhado para incluir promotores T7, e certas sequências, como complexos ribossomais não pode ser transcrito e montado. 21,22 existentes sistemas de expressão livre de células são incapazes de manter altos rendimentos preservando mecanismos reguladores endógenos, a versatilidade necessária para a biologia sintética.
Nós desenvolvemos uma E. endógeno sistema de expressão de E. coli isento de células que mantém a eficiência da expressão proteica demonstrado pelos sistemas anteriores, mas adiciona versatilidade adicional, permitindo a expressão e regulação baseada em ambos (T7 ou outros mecanismos) endógeno e exógeno. O protocolo aqui descrito é baseado no Kigawa et al. (2004) e Liu et al. (2005), mas não tem modificações significativas. Ele utiliza-Mg e K-glutamato sobre Mg e K-acetato para o aumento da eficiência, remove 2-mercaptoetanol, e lisa células usando um talão-batedor. 17,23,24 Bead-surra é escolhido em detrimento de homogeneização,métodos baseados em pressão, ou de ultra-sons, devido ao seu menor custo e rendimentos comparáveis aos sistemas concorrentes. 23 ácido 3-phosphoglyceric (3-PGA) é utilizado como fonte de energia, uma vez que foi encontrada para dar rendimentos de proteína de qualidade superior quando comparada com o fosfato de creatina e fosfoenolpiruvato. 4,25 nosso sistema pode produzir-se de 0,75 mg / ml de proteína repórter usando um promotor à base de sigma70 com operadores lambda-fago ou um promotor de T7-driven, semelhante aos rendimentos de outros sistemas comerciais. 4,6 Cinco dias são necessárias para produzir todos os reagentes necessários (Figura 1). Além disso, proporciona uma redução de custos de 98% em comparação com os sistemas livres de células comerciais comparáveis - os custos de material são de US $ 0,11 por 10 mL de reação, que aumenta para $ 0,26 com mão de obra incluída (Figura 2).
Protocolo
1. Extracto de células bruto Preparação
Preparando extrato celular bruto durante três dias necessárias duas pessoas para conduzir de forma eficiente. O protocolo funcionalmente consiste em três partes: crescimento da cultura (passo 1.1 para o passo 1.11), lise celular (passo 1.12 para o passo 1.37), e extrair esclarecimento (passo 1,38 para o passo 1.52). Apresenta-se dividido em dias por conveniência. Extracto ideal pode produzir 0,75 mg / ml de deGFP de plasmídeo pBEST-OR2 OR1-Pr-UTR1-deGFP-T500 (Addgene # 40019), e tem uma concentração de extracto celular bruto entre 27-30 mg / ml de proteína. Quatro entanto , as características de extracto pode variar de lote para lote. A receita a seguir fornece o suficiente para cerca de 3.000 reações individuais (6 ml de extrato celular bruto). Se escalar para baixo, é recomendado o uso de nada menos do que 1/6 de valores aqui. Devido a limitações de tempo, ampliação não é recomendado.
Dia 1
- Preparar meios de cultura bacteriana, culplaca estrutura, mídia e suplementos, como descrito na Tabela 1. Veja material suplementar 1 para receitas.
- . Streak BL21-Rosetta2 estirpe de -80 ° C sobre uma placa de agar 2xYT + P + Cm e incubar durante pelo menos 15 horas a 37 ° C, ou até que as colónias são facilmente visíveis Nota: cloranfenicol (Cm) é utilizado para seleccionar um plasmídeo codificação tRNAs raros na cepa BL21-Rosetta2.
Dia 2
- Prepare tampões e suplementos, como descrito na Tabela 2. Veja material suplementar 1 para receitas.
- Preparar e esterilizar materiais necessários para o dia 3, incluindo: 6 x 4 frascos L de Erlenmeyer com tampa de alumínio (autoclavado), 4 x 1 L frascos de centrífuga estéreis, funil (autoclave), 100 g de 0,1 mm esferas de vidro (autoclavado), 2 agitar-bares (autoclavado), 1 L e 500 ml cilindro graduado (autoclavado), 2 x 1 copos L (autoclavados), 3 ml seringa com 18 agulhas G (estéreis), 2-3 flbóias de aveia, 2-3 10k MWCO cassetes de diálise (estéril), tinas.
- Prepare mini-cultura 1. Adicionar 4 ml de meio 2 x YT + P e 4 ul de Cm para um tubo de cultura de 12 ml estéril e pré-aquecer a 37 ° C durante 30 min.
- Inocular mini-cultura 1, com uma colônia do 2xYT + P + placa Cm agar. Incubar a 220 rpm, 37 ° C durante 8 horas.
- 7 horas e 30 minutos mais tarde, preparar mini-cultura 2. Adicionar 50 ml de meio 2 x YT + P e 50 ul de Cm para um estéril Erlenmeyer de 250 ml e pré-aquecer a 37 ° C durante 30 min.
- Inocular mini-cultura 2 com 100 ul de mini-cultura 1 e incuba-se a 220 rpm, 37 ° C durante 8 horas.
Dia 3
- Pesar quatro estéreis de 50 ml tubos Falcon vazios e massa registro na Tabela 3. Descontraia tubos Falcon no gelo, que serão posteriormente utilizados na etapa 1.18.
- 7 horas e 30 min após o passo 1.8, preparar meios de cultura bacteriana final. Usando um estéril 1 L cilindro graduado, transferência de 660 ml de meio 2xYT + P into cada um dos seis frascos de 4 L de Erlenmeyer e pré-aquecer a 37 ° C durante 30 minutos Nota:. 4 L ou maior matrás é recomendado para arejamento adequado.
- Adicionar 6,6 ml de mini-cultura 2 em cada Erlenmeyer 4 L. Incubar a 220 rpm, 37 ° C até a cultura atingir uma densidade óptica de 1,5-2,0 a 600 nm (o que corresponde a fase de crescimento semi-log). Verifique periodicamente OD com uma diluição de 1:10 da cultura para a exatidão Este passo não deve demorar mais do que 3 horas - 3 horas 45 min,. Rápido crescimento e coleta durante a fase mid-log é fundamental para a qualidade do extrato.
- Imediatamente após o crescimento, todas as culturas transferir uniformemente em quatro frascos de centrífuga de 1 L e centrifugar a 5000 g durante 12 min a 4 ° C para sedimentar as células bacterianas.
- Enquanto centrifugação, preparação completa tampão S30A, adicionando 4 ml de 1 M DTT a 2 L de previamente preparada S30A. Misture e manter tampão no gelo.
- Quando centrifugação estiver concluído, remova completamente o sobrenadante do passo 1,12 por decaNTING e secar as garrafas de centrífuga em uma toalha de papel estéril.
- Adicionar 200 mL de tampão de S30A a 4 ° C a cada um dos quatro frascos de centrífuga, e agitar os frascos vigorosamente até sedimento é solubilizado completamente sem grumos restantes. Centrifugar as quatro garrafas a 5.000 g durante 12 min a 4 ° C.
- Completamente remover o sobrenadante do passo anterior por decantação e secar as garrafas de centrífuga em uma toalha de papel estéril.
- Repita os passos de 1.15 e 1.16.
- Adicionar 40 ml de tampão S30A a 4 ° C a cada garrafa centrífuga. . Transfira cada bolinha e combinação S30A em um tubo Falcon refrigerados de 1,9) Nota: Este passo é transferir as bolinhas em um recipiente menor.
- Centrifugar os tubos Falcon a 2000 g durante 8 min a 4 ° C. Remover o sobrenadante por decantação.
- Re-centrifuga os tubos Falcon a 2000 g durante 2 min a 4 ° C. Completamente remover o sobrenadante residual com uma pipeta. Mantenha no gelo.
- Pesar a fnossos tubos Falcon com pellet e massa registro na Tabela 3. Calcular massa sedimento, o volume de tampão necessário S30A, e massa de grânulos necessários com base nas fórmulas específicas na Tabela 3.
- Correto carga e talão batida do pellet é fundamental para fazer extrato de qualidade, e é o passo mais desafiador. Recomenda-se a rever o vídeo antes de tentar. A falha para evitar bolhas de ar e distribuir grânulos uniformemente irá resultar em extracto ineficiente.
- Adicionar a quantidade de tampão S30A calculados na Tabela 3 para cada tubo de Falcon, de vórtice até estar homogénea, e retornar para o gelo.
- Embora mantendo os outros tubos Falcon de gelo, adicionar grânulos de forma intermitente a um único tubo de Falcon em três alíquotas, cada uma usando 1/3 do total dos grânulos. Após a adição de cada aliquota de grânulos, em vórtice durante 30 seg. Coloque o tubo Falcon no gelo entre as etapas vórtice e depois vórtice final. Depois da última alíquota é adicionado, garantiresferas são distribuídas uniformemente. A pasta grossa deve ser formado.
- Prepare um 5 ml (volume) ponteira cortando o final usando uma lâmina de barbear estéril para criar uma abertura de 3-4 mm. Dial pipeta de 2 ml Nota: diferentes conjuntos de pipetas e pontas fornecer diferentes quantidades de sucção que pode não ser suficiente para puxar e libertar solução de células do grânulo de espessura, com uma ponta de pipeta de 1 ml com extremidade removido pode ser utilizado em substituição..
- Colocar 20 tubos talão batendo no gelo.
- Verifique alta viscosidade da solução de células-talão usando pipeta modificado. Ele deve ser viscoso para o ponto de saída apenas a ponta da pipeta durante a ejecção. Se for muito viscoso, reajuste ponteira acordo com o passo 1,25. Se não for suficientemente viscosa, os grânulos podem ser adicionados em passos de (massa sedimento * 0,05), para um peso máximo (massa sedimento * 5.1). Após cada adição de grânulos, de vórtice durante 30 segundos e regressar ao gelo. Veja a Figura 3-A para uma demonstração de viscosidade.
- Remova a solução de células do grânulo de tubo Falcon utilizando pipeta modificado, e transferir para um tubo de batendo talão estéril, enchendo-o três quartos cheio com solução de células do grânulo. Gire extremamente brevemente (1s) em um balcão de mini-centrífuga para remover as bolhas de ar, sem redistribuição de contas. Ver Figuras 3b-d para imagens estáticas de carregamento tubo talão-de bater.
- Termine adicionando solução de células grânulo para formar um menisco côncavo.
- Adicione uma pequena gota de solução de células grânulo para o interior de uma tampa do tubo batendo talão, tomando cuidado para não impedir o lábio fora da tampa, caso contrário, o tubo batendo talão não vai fechar sufficientl y. Toque a tampa sobre uma superfície plana e verifique se não há bolhas de ar na parte inferior da tampa.
- Tapar o tubo batendo grânulo com a tampa batendo talão do passo anterior. Mão para assistente de talão de bater. Se feito corretamente, a tampa deve ser hermeticamente fechado, sem bolhas de ar shoulestaria visível, e pouco (se houver) solução de células talão deve transbordar. Refazer o processo de carregamento, se as bolhas de ar são visíveis ou a tampa não fechar totalmente.
- Vortex tubo Falcon de passo 1,24 com a solução de células talão restante para assegurar uma distribuição uniforme de contas. Repita os passos de 1,28-1,31 até tubo Falcon está vazia e, depois, repetir os passos 1,24-1,31 para cada tubo Falcon adicional.
- Conduta passos 1,33-1,38 simultaneamente. Já assistente take cheio de tubos de 1,31 batendo talão e colocar no gelo. Uma vez que dois tubos batendo talão preenchidos foram recolhidos e foram no gelo por pelo menos um minuto, comece talão de bater.
- Bata um tubo de 30 segundos a 46 rpm. Coloque de cabeça para baixo no gelo por 30 segundos, enquanto batendo o outro tubo.
- Repita o passo anterior de tal forma que cada tubo batendo talão preenchido foi vencer por 1 min total.
- Repita os passos de 1,33-1,35 até 8 tubos cheios talão batendo (ou a quantidade máxima da centrífuga pode segurar) have foi processado. Em seguida, constroem aparelho de filtro a partir de 15 ml Falcon (Figura 3e). Adicionar um novo cap-batendo grânulo,-parte plana virada para cima, para o fundo de um 15 ml Falcon. Em seguida, retire a tampa do tubo batendo talão processados e imprensa coluna micro-cromatografia firmemente extremidade do tubo batendo talão processada até que esteja completamente selado. Pressão em final de eluição de coluna micro-cromatografia, e coloque coluna micro-cromatografia de eluição acabar para baixo, em vazio tubo talão beading. Coloque este complexo em 15 ml Falcon. Repita o procedimento para todos os 8 tubos batendo talão cheias; manter em gelo quando completa.
- Centrífuga 8 aparelhos de filtro, o tubo Falcon destapado, a 6.000 g durante 5 min a 4 ° C para separar o extracto de sedimento e de grânulos.
- Verifique cada tubo talão espancamento produziu extrato viável. Extracto propriamente bater não será turva, e o sedimento terá duas camadas distintas. Descarte todos os tubos turvas, e transferir o sobrenadante de tubos não turvas i1,75 ml tubos de micro-centrífuga nto individuais, tendo tão pouco pellet possível. Mantenha no gelo até que todos os tubos de bater talão foram processadas. Veja a Figura 3-F comparar um tubo talão espancamento corretamente vs incorretamente processados.
- Tubos de centrífuga de micro-centrífuga a partir do passo anterior, em 12,000 g durante 10 min a 4 ° C.
- Transfira o sobrenadante em tubos talão batendo vazio usando uma pipeta, consolidando 500 mL em um novo tubo batendo talão livre de pelota.
- Incubar passo anterior, equipados com tampas de bater talão removidos, a 220 rpm, 37 ° C durante 80 min. Este passo digere restantes ácidos nucleicos utilizando exonucleases endógenos libertados durante o processo de perolização-grânulo, e pode ser feito através do tubo de pé-de bater grânulo em um tubo de cultura de tecidos.
- Preparar materiais de diálise. Preparação tampão completa S30B por adição de 2 ml de 1 M DTT a 2 L de previamente preparada S30B. Misture e adicione 900 ml em cada um dos dois ªerile 1 L copos. Adicionar agitador magnético estéril em cada copo, manter a 4 ° C.
- Depois do passo 1,41, extrato deve olhar turvo. Consolidar extracto em 1,5 ml de alíquotas de 1,75 ml em tubos de micro-centrífuga, e centrifugar a 12000 g durante 10 min a 4 ° C.
- Usando uma pipeta, consolidar sobrenadante livre de pellet em 15 ml tubos Falcon no gelo, e misture bem por tampar o tubo e inversora. Economize 10 ml de sobrenadante no gelo para a etapa 1.47.
- Determinar a quantidade total de extracto produzido, e hidratar o número necessário de 10k cassetes de diálise de MWCO submergindo em S30B durante 2 minutos, assumindo que 2,5 ml de extracto por cassete.
- Coloque cassetes com 2,5 ml do extrato. Cada proveta pode levar até 2 cassetes; diálise, agitação, a 4 ° C durante 3 horas Nota: carga parcial de cassetes é aceitável.. Dialisadora aumenta o rendimento da produção da proteína.
- Durante o passo anterior, caracterizar a proteína extractoconcentração com um ensaio de Bradford, utilizando extrato salvou na etapa 1.44. Veja material suplementar 2 para mais detalhes.
- Após a diálise é completa, extrato de alíquota de 1,5 ml em 1,75 ml tubos de micro-centrífuga. Centrifugar a 12.000 g durante 10 min a 4 ° C. Um sedimento vai formar-se no fundo do tubo.
- Consolidar claro sobrenadante do passo anterior por pipetagem em um tubo Falcon de 15 ml no gelo. Homogeneizar por inversão de 5-10x.
- Com base na concentração determinada por Bradford no passo 1.47, determinar a quantidade de extracto individuais Alíquota em tubos de 1,75 ml. Cada tubo indivíduo deve ter um volume de 810-900 mg de proteína total. Extrair deve ter uma concentração de proteína total maior do que 27 mg / ml. Esta etapa requer assistência para realizar expediente Nota:. Extrato alíquota abaixo de 30 mg / ml em 30 mL alíquotas e escala se a concentração é maior, por exemplo, o extrato de alíquota de 28 mg / ml por 30 mL, e extrato de alíquota em 32mg / ml por 28,1 ul.
- Extrato alíquota seguinte passo 1,50, tomando cuidado para evitar bolhas. Extracto de inflamação por congelação em azoto líquido Nota:. Alíquotas com bolhas pode ser removido por centrifugação a 10.000 xg durante 30 seg a 4 ° C.
- Remova os tubos de nitrogênio líquido usando uma peneira e armazenar imediatamente a -80 ° C. Segurança: Usar óculos de proteção; as tampas de tubos de extrato pode sair devido à diferença de temperatura entre nitrogênio líquido e temperatura ambiente.
2. Amino Acid Solution Preparação
Solução de aminoácidos devem ser preparados em massa. A receita a seguir utiliza um kit completo de RTS Aminoácidos Sampler, fornecendo o suficiente para aproximadamente 11.000 reações individuais. Se dimensionamento para baixo, é recomendável usar não menos do que metade de um kit. Cada aminoácido no estoque é fornecido a 1,5 ml, 168 mM, excepto para a leucina na 140 mM. A composição final de solução de aminoácidos é:leucina, 5 mM, todos os outros aminoácidos, 6 mM. Esta é a concentração de trabalho de 4x.
- Remove todos os 20 aminoácidos a partir de -20 ° C e descongelamento à temperatura ambiente. Uma vez descongelado, vortex até aminoácidos dissolver, incubando a 37 ° C, se necessário. Depois de aminoácidos são dissolvidos, colocar todos os aminoácidos excepto em gelo para Asn, Phe, e Cys, que são mantidas à temperatura ambiente. Cys pode não se dissolvem totalmente.
- Em gelo, adicionar 12 ml de água estéril para um tubo Falcon de 50 ml estéril.
- Adicionar 1,5 ml de cada um dos aminoácidos, na seguinte ordem, tendo o cuidado de vortex do tubo Falcon após cada adição e para manter a solução em gelo: Ala, Arg, Asn, Asp, Gln, Glu, Gli, His, lie, Lys, Met, Fen, Pro, Ser, Thr, Vai, Trp, Tyr, Leu, Cys. Cys pode ser adicionado como uma suspensão. Depois disso, vortex até que a solução é relativamente claro, incubando a 37 ° C, se necessário. Cys pode não se dissolvem totalmente.
- Solução de aminoácidos Alíquota em 50 tubos de26 mL cada no gelo. Alíquota o resto em 500 mL por tubo no gelo. Os 26 mL alíquotas serão utilizadas para a calibração de extracto, enquanto as alíquotas de 500 uL irá ser utilizado para a preparação de tampão. Enquanto alíquotas, vórtice do estoque principal frequentemente para evitar a distribuição desigual de suspensão.
- Alíquotas do Flash congelamento em nitrogênio líquido e armazenar a -80 ° C. Segurança: Usar óculos de proteção; as tampas de tubos de extrato pode sair devido à diferença de temperatura entre nitrogênio líquido e temperatura ambiente.
- Opcional: Realizar um ensaio da atividade de solução de aminoácidos recém-feita contra soluções de aminoácidos feitas anteriormente.
3. Preparação de Solução de energia
Solução de energia é utilizada tanto para a calibragem de extracto bruto de células e para a criação de tampão, e devem ser preparados em grandes quantidades. A receita a seguir fornece o suficiente para cerca de 10 mil reações individuais. Se escalar para baixo, é recomeNDED usar nada menos do que 1/24 dos valores aqui apresentados. Como a solução de energia é um custo monetário significativo, os usuários primeira vez pode querer preparar em escala 1/24. A composição final de solução de energia é: HEPES 700 mM pH 8, 21 mM de ATP, GTP 21 mM, CTP 12,6 mM, 12,6 mM de UTP, ARNt 2,8 mg / ml, 3,64 mM de CoA, NAD 4,62 mM, AMPc 10,5 mM, ácido folínico 0,95 mM, espermidina 14 mM, 3-PGA 420 mM. Esta é a concentração de trabalho de 14x. Se desejado, cada artigo individual na Tabela 4 pode ser armazenada a -80 ° C para uso posterior.
- Remove todos os produtos químicos na Tabela 4 a partir de -80 ° C, -20 ° C, ou 4 ° C até à temperatura ambiente durante 30 min.
- Preparar soluções de reserva, tal como descrito na Tabela 4. Veja material suplementar 1 para receitas. Coloque todas as soluções no gelo após a preparação.
- Num tubo Falcon de 15 ml, adicionar, na seguinte ordem, tendo o cuidado de vortex do tubo Falcon após cada adição e para manter as soluções on gelo: 3,6 ml de 2 M de HEPES, 144 mL de água, 1,39 ml de mistura de nucleótidos, 576 ul de 50 mg / ml de ARNt, 576 ul mM de CoA-65, 276 ul de 175 mM de NAD, 170 ul de 650 mM de cAMP, 288 ul 33,9 ácido folínico mM , 144 ul de 1 M espermidina, e 3,09 ml de 1,4 M de 3-PGA.
- Solução Energia Alíquota em 50 tubos de 7 mL cada no gelo. Alíquota o resto em 150 mL por tubo no gelo. Os 7 mL alíquotas serão utilizadas para a calibração de extracto, enquanto os 150 mL alíquotas será utilizado para a preparação de tampão. Enquanto alíquotas, vórtice do estoque principal frequentemente.
- Alíquotas do Flash congelamento em nitrogênio líquido e armazenar a -80 ° C. Segurança: Usar óculos de proteção; as tampas de tubos de extrato pode sair devido à diferença de temperatura entre nitrogênio líquido e temperatura ambiente.
- Opcional: Realizar um ensaio de recém-feitos Solution Energia contra Energy Solutions feitas anteriormente atividade.
4. Tampão de Preparação
Buffer Preparação exige a realização de celular extrato bruto de Preparação de Aminoácidos Solução Preparação e Energia Solução Preparação. Cada buffer é exclusivo para um lote de extrato celular bruto. Mg-glutamato, K-glutamato e DTT (nessa ordem) são otimizados nesta seção para produzir reações com níveis máximos de expressão. O protocolo a seguir utiliza um modelo de pré-escrita, TXTL_e (modelo) _calibration_JoVE.xlsx (Material Suplementar 3), para calibrar extrato celular bruto pré-preparados e preparar buffer. No entanto, também se pode calibrar extrato celular bruto e preparar tampão sem o modelo por meio da otimização Mg-glutamato, K-glutamato e DTT manualmente e configurar o buffer de tal forma que, juntamente com o extrato, é 75% de um volume total de reacção. Se a calibragem manualmente, condições de final de reacção pode ser encontrada na etapa 5.
- Preencha o formulário "Dados Gerais".
- Descongelar em gelo 100 mM de Mg-glutamato (4 ° C), 3 H K-glutamato (4 ° C), 6Solução mM Aminoácidos (26 mL, -80 ° C), Solução de energia (7 mL, -80 ° C), DTT 100 mM (-20 ° C), o DNA de controlo positivo (-20 ° C), 40% de PEG- . 8,000 (4 ° C), extracto celular bruto (-80 ° C), e água (4 ° C) Nota: O uso de 1 nM de concentração de trabalho pBEST-OR2 OR1-Pr-UTR1-deGFP-T500 (Addgene plasmídeo 40019) para o controlo positivo (excitação 485 nm, emissão 525 nm), ou outra referência que produz o sinal de alta intensidade 4.
- Prepare sete 10,5 reações ul, testando uma série de 4-10 mM Mg-glutamato adicional, por alíquotas definidas quantidades de estoque Mg-glutamato em tubos de micro-centrífuga individuais. Nota: Embora 10,5 ul reacções são inicialmente preparado, o final da reacção é de 10 uL.
- Prepare a mistura principal conforme indicado no molde sob a "calibração Mg-glutamato," a adição de um adicional de 80 mM de K-glutamato. Manter em gelo e vórtex após a adição de cada item Nota:. Os valores aqui apresentados e no modelo sãopara além das quantidades de Mg-glutamato, K-glutamato, e DTT presente no tampão usado para fazer S30B extracto celular bruto.
- Adicione a mistura de mestre para amostras contendo Mg-glutamato e preparar reações. Veja os passos 5,10-5,13 para instruções detalhadas.
- Executar reacção a 29 ° C, quer numa incubadora ou num leitor de placas.
- . Determinar concentração de Mg-glutamato ideal por nível no final de expressão e taxa máxima de expressão da proteína (Figura 4-A) Nota: Tempos de Execução variar de acordo com experiência, mas geralmente duram menos de 8 horas.
- Repita os passos 4.2 a 4.7 para o K-glutamato em "K-glutamato de calibração", estabelecendo os níveis de Mg-glutamato às encontradas no passo 4.7.
- Repita os passos 4.2 a 4.7 para a TDT em "calibração TDT", que fixa os teores Mg-glutamato aos encontrados no passo 4.7 e os níveis de K-glutamato aos encontrados no passo 4.8 Nota:. Descobrimos que acrescentou que a TDT não afecta significativamente o final de os níveis de expressão.
- UsoOs valores encontrados na calibração sob "a composição do tampão" para determinar a composição de tampão a ser preparado. Com base na quantidade de extrato celular bruto produzido, uma receita mistura principal é produzido por um valor conjunto de buffers.
- Descongele alíquotas conforme listado na receita mestre mix no gelo. Uma vez descongelada, a preparação da mistura principal, mantendo em gelo e centrifugação, depois da adição de cada item.
- Alíquota por montante declarado em "composição de Buffer". Tubos de tampão Flash-congelamento em nitrogênio líquido. Enquanto alíquotas, vórtice o estoque principal freqüência.
- Remova os tubos de nitrogênio líquido usando uma peneira e armazenar imediatamente a -80 ° C. Segurança: Usar óculos de proteção; as tampas de tubos de extrato pode sair devido à diferença de temperatura entre nitrogênio líquido e temperatura ambiente.
5. Execução Experimental de uma Reação TX-TL
Condições de reação finais são: 8,9-9,9 mg / ml proteem (a partir de extracto em bruto), 4,5 mM 10,5 mM-Mg-glutamato, 40-160 mM de K-glutamato, DTT 0,33-3,33 mM, 1,5 mM de cada aminoácido, excepto leucina, leucina 1,25 mM, HEPES 50 mM, ATP 1,5 mM e GTP, CTP e UTP 0,9 mM, 0,2 mg / ml de ARNt, CoA 0,26 mM, 0,33 mM de NAD, cAMP 0,75 mM, 0,068 mM de ácido folínico, 1 mM de espermidina, 30 mM de 3-PGA, 2% de PEG-8000. uma base TX -TL reacção tem três partes (tubos): extracto celular bruto, tampão, e ADN. A proporção é: tampão de 75% e extrato, 25% do DNA As reações podem variar de volume, e nós usamos 10 ml por convenção para minimizar o volume de reação e permitir que funciona em uma placa de 384 poços.. Volumes maiores requerem agitação para a oxigenação adequada. O protocolo a seguir utiliza um modelo de pré-escrita, TXTL_JoVE.xlsx (Material Suplementar 4), para realizar uma reação de 10 mL. Itens em roxo indicam valores-de entrada do usuário, e os itens em azul indicam reagentes adicionais para adicionar à reação. No entanto, também se pode realizar uma sagacidade reaçãohout o modelo, seguindo condições de reação descritas acima.
- Preencha o formulário "Dados Gerais".
- Em "Master Mix Preparação", inserir o valor percentual de extrato a partir do passo 4.1 na caixa roxa.
- Projete seu experimento in silico usando o "Master Mix Preparação" (linhas 10-17) e "DNA Preparação" (linhas 19-50) seções. Geralmente, as constantes podem ser colocados na seção "Master Mix Preparação", enquanto que as variáveis podem ser colocados na seção "Preparação de DNA". Minimizar amostras por experimento para evitar a evaporação da amostra e viés experimental hora de início. Veja a Figura 6 para uma configuração de amostra.
- Em "Master Mix Preparação", adicionar os reagentes, tais como indutores ou proteínas, que vão em todas as amostras com uma concentração constante. Começando com a linha 14, preencha as áreas sombreadas azuis, mantendo um reagente para cada linha. Unidades são proporções relativas.
- Em "Preparação de DNA", adicionar DNA que será específi amostraic. IDs amostra # 1 e # 2 correspondem aos controles positivos e negativos, respectivamente. IDs de amostra # 3 e acima são repetições (de 10 reacções ul) pelo usuário modificável para DNA, a concentração de ações em ng / mL, comprimento em pares de bases, concentração final desejada em nM, e. A quantidade de DNA de ações para atingir a concentração final desejada é calculado automaticamente. . Total entre os montantes de linha a 10,5 * n, onde n é o número de repetições Nota: Embora o volume final da reacção é de 10 uL, os cálculos assumem um volume total de 10,5 mL por reacção, para ter em conta o volume perdido durante a pipetagem.
- Em "Preparação de DNA", adicionar reagentes ou DNA adicional que será específico para amostra colunas azuis. As concentrações da ADN em nm pode ser calculado de acordo com "Preparação de ADN", enquanto que os reagentes específicos de exemplo exigem o cálculo manual com base num volume de reacção total de 10,5 * n. Os volumes introduzidos são subtraídos para fora do volume de água da mesma linha.
- Remover numbe necessáriar de tubos de tampão, o extracto celular em bruto, e o controlo positivo em "tubos para descongelar," a partir de -20 ° C ou -80 ° C e descongelar em gelo.
- Preparar as amostras de DNA. Para cada amostra ID, alíquota o DNA indicada, água e itens fornecidos pelo usuário por seção "Preparação de DNA" em um tubo de micro-centrífuga, à temperatura ambiente Nota:. Para evitar a perda de amostra, recentemente pipetas e baixo-stick calibrado pontas para pipetas e tubos de micro-centrífuga são recomendados.
- Quando tubos da etapa 5.7 são descongelados, prepare a mistura de mestre-tampão, extrato, e todos os itens fornecidos pelo usuário globais com base nas caixas-shaded laranja, mantendo no gelo e centrifugação, após a adição de cada item Nota:. Extract é extremamente viscoso. Alíquotas com bolhas pode ser removido por centrifugação a 10.000 xg durante 30 seg a 4 ° C.
- Adicione a quantidade de mistura principal indicado nas células laranja em "DNA Preparação" (coluna O) para cada amostra de DNA, e mantê-la em temperatura ambienteture. Trate isso como o tempo de início de reação.
- Vortex de cada amostra, e centrifugar a 10000 xg durante 30 segundos à temperatura ambiente para fazer descer qualquer amostra residual e para reduzir as bolhas.
- Se a condução da reacção em tubos de micro-centrífuga, incubar directamente a 29 ° C. . Caso contrário, pipeta 10 ul de amostra numa placa de 384 poços Nota: As reacções em volumes maiores do que 10 mL pode requerer agitação para oxigenação.
- Centrifugar a placa de 4.000 xg durante 30 segundos a temperatura ambiente para reduzir qualquer amostra residual e para reduzir as bolhas. Selar placa depois para evitar a evaporação.
- Execute reação a 29 ° C. Nota: Runtimes variam de acordo com experiência, mas geralmente duram menos de 8 horas.
Resultados
Nós apresentamos um protocolo de cinco dias para a preparação de uma Escherichia coli endógeno com base TX-TL sistema de expressão isento de células. A linha do tempo da amostra para a criação de reagentes - extracto celular bruto e tampão - pode ser encontrada na Figura 1. Uma vez criado, estes podem ser armazenados a -80 ° C durante até um ano. Depois de reagentes são criados, configuração e execução experimental pode ser feito em menos de 8 horas.
Além disso, optimizado as condições do sistema de expressão isento de células TX-TL de expressão. Outras adições fornecidos pelo usuário, tais como tampões ou soluções de DNA, deve ser calibrado para a toxicidade de antemão. Por exemplo, os diferentes métodos de transformação plasmídeos resultar na expressão diferente devido ao teor de sal. Também testámos o efeito de tampão de eluição de Tris-Cl a eficiência da reacção (Figura 5).
Um exemplo de calibração extracto celular em bruto, referindo passo 4,1-4,9, é mostradona figura 4a. Em geral, as nossas experiências demonstram que o extracto celular bruto é mais sensível aos níveis de Mg-glutamato, seguida por níveis K-glutamato. Para demonstrar o sistema de expressão isento de células, construiu-se e testou um circuito de retroalimentação negativa com base na repressão tet. 26 (Figura 6). No sistema de expressão livre de células, o mesmo circuito prazo com e sem aTc mostra uma de 7 vezes de ponto final a mudança de expressão deGFP repórter após oito horas de expressão. Embora nesta experiência não requer indutores globais ou repressores, se necessário, eles podem ser adicionados em "Master Mix Preparação."
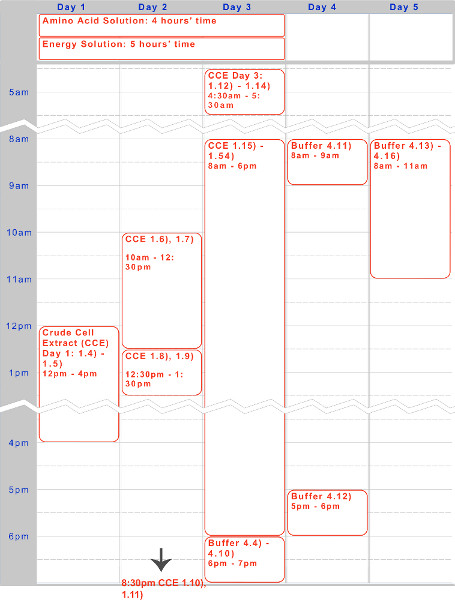
Figura 1. Cronograma para extrato celular bruto, solução de aminoácidos, e preparação da solução de energia. Uma timelin cinco dias e para uma execução típica do protocolo é dado acima, otimizado para incubações durante a noite e as etapas de trabalho durante o dia.
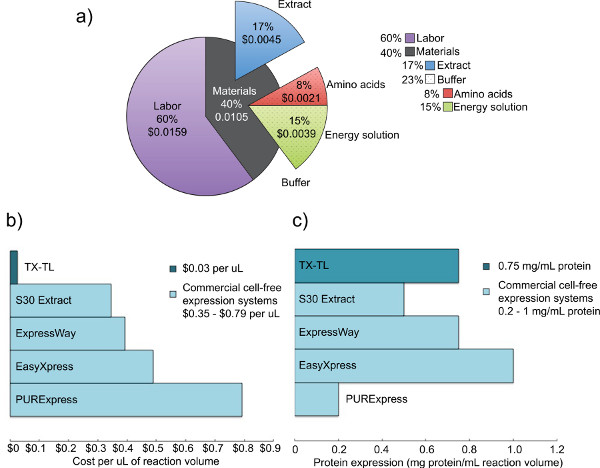
Figura 2. Custo e análise de extratos de células bruto competindo expressão. a) Composição dos custos de mão de obra e materiais do sistema de expressão livre de células TX-TL. Com base nos custos de reagentes a partir de dezembro de 2012, e os custos trabalhistas de US $ 14 por hora. B) Comparação de TX-TL livres de células custos do sistema de expressão versus outros sistemas comerciais. Os custos são divididos por mL, apesar de volumes de reação pode variar por kit. C) Comparação de TX-TL livre de células rendimento sistema de expressão versus outros sistemas comerciais. Expressão da proteína rendimento determinado por normas fabricante./ 50762fig2large.jpg "target =" _blank "> Clique aqui para ver maior figura.

Figura 3. Carregando e processamento de um tubo de talão-de bater. a) Demonstração da correta viscosidade da solução de células-pérola. Solução Cell-talão terá uma viscosidade depende de muitos fatores, incluindo a quantidade de tampão S30A adicionado, a quantidade de contas adicionadas, e passou um tempo no gelo. B) Carregamento de tubo batendo talão antes rápida centrifugação mesa. A centrifugação remove bolhas acumuladas durante o carregamento. C) Bolhas tona após tabletop centrifugação. O tamanho das bolhas irá variar, pois eles podem ser estalado ou removida utilizando uma ponta de pipeta d) do tubo-batendo grânulo completamente preenchido antes de nivelamento.. Um menisco é formado na batendo talão tube, ea tampa tem o suficiente para cobrir e causar pequenas quantidades para transbordar. e) aparelho de filtro corretamente carregado. Estes podem ser reutilizados. F) Comparação das correctamente vs tubo batendo cordão processado de forma incorrecta. O tubo da esquerda é um tubo bem-beat - apresenta um pequeno e bem delineado camada superior, e sobrenadante muito claro. O tubo da direita é sub-óptima, com base na maior, a segunda camada turva e nebulosa o sobrenadante. Tubos que são de qualidade inferior não devem ser submetidos a processamento adicional.
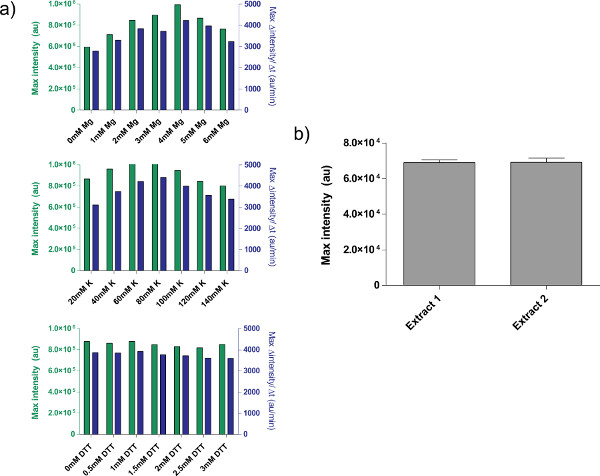
Figura 4. Propriedades de preparações de extrato bruto. a) parcelas de calibração típicas para extrato celular bruto. O extrato bruto é calibrado para o Mg-glutamato adicional, K-glutamato, e os níveis de DTT, nessa ordem. É mostrado endpoint fluorescence depois de 8 horas, bem como a taxa máxima de produção de proteínas com base em uma média móvel de 12 minutos. Com base nestes lotes, um intervalo aceitável de Mg-glutamato adicional é de 4 mM, K-glutamato é de 60-80 mM, e DTT é 0-3 mM. Note-se que cada extrato bruto precisa ser calibrado de forma independente para estas três variáveis. B) Variação de preparações de extrato. Endpoint fluorescência de dois extratos preparados em datas diferentes é mostrada; barras de erro são um desvio padrão de três corridas independentes em dias diferentes. Clique aqui para ver maior figura .
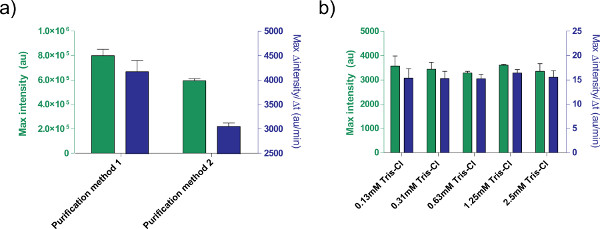
Figura 5. Efeitos da solução de DNA sobre a eficiência de expressão. a) Comparação de dois purificação diferentemétodos para a transformação de plasmídeos. 1 nM de pBEST-OR2-OR1-Pr-UTR1-deGFP-T500 é preparado utilizando apenas um Kit Qiaprep rotação Miniprep (método de purificação 1) ou pós-processado com um kit de purificação Qiaquick PCR (método de purificação 2). É mostrado o ponto final de fluorescência após 8 horas, bem como a taxa máxima de produção de proteínas com base em uma média móvel de 12 minutos. As barras de erro são um desvio padrão de quatro ensaios independentes em dias diferentes. B) Efeito de tampão de eluição (Tris-Cl). Diferentes concentrações de Tris-Cl são comparados numa reacção de expressão isento de células com base na expressão de 1 nM de pBEST-OR2 OR1-Pr-UTR1-deGFP-T500. As concentrações dadas são as concentrações finais de Tris-Cl na reacção; tampão de eluição utilizado é 10 mM Tris-Cl. As barras de erro são um desvio padrão de três corridas independentes em dias diferentes. Clique aqui para ver maior figura .
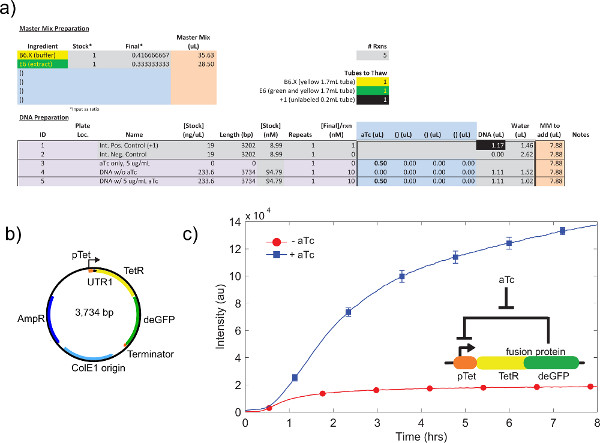
Figura 6. Amostra TX-TL de execução de um ciclo de feedback negativo. a) configuração Amostra de uma reação execução livre de células. Testes "na" versus "off" estado de loop de feedback negativo, com controles positivos e negativos. B) plasmídeo mapa de ciclo de feedback negativo. C) Os resultados representativos. Dados reflecte experimento em a) e b), com controlo negativo subtraído do sinal. Circuito genético mostrado na inserção. As barras de erro são um desvio padrão de três corridas independentes em dias diferentes. Clique aqui para ver maior figura .
| Nome | Concentração | Quantidade | Esterilização | Notas |
| Cloranfenicol (Cm) | 34 mg / ml em etanol | 1 ml | Filtro de esterilizar (0,22 mM) | Pode ser feito em grandes volumes armazenados a -20 ° C para uso posterior. |
| 2xYT + P + placa Cm agar | 31 g / L de 2 x YT, 40 mM de fosfato dibásico de potássio, 22 mM de fosfato de potássio, 34 ug / ml de cloranfenicol | 1 placa | Autoclave | |
| 2xYT + P mídia | 31 g / L de 2 x YT, 40 mM de fosfato dibásico de potássio, 22 mM de fosfato de potássio monobásico | 4 G | Autoclave |
Tabela 1. Reagentes para dia 1 de protocolo extrato celular bruto.
| Nome | Concentração | Quantidade | Esterilização | Notas |
| Tris base | 2 M | 250 ml | Filtro de esterilizar (0,22 mM) ou autoclave | Pode ser armazenada à temperatura ambiente. |
| TDT | 1 M | 6 ml | Filtro de esterilizar (0,22 mM) | Pode ser feito em grandes volumes e armazenado a -20 ° C para uso posterior. |
| Tampão S30A | 14 mM de Mg-glutamato, 60 mM de K-glutamato, 50 mM Tris, pH 7,7 | 2 L | Autoclave | Para atingir pH 7,7, titula-se com ácido acético. Adicionar DTT 2 mM concentração final imediatamente antes da utilização. Armazenar a 4 ° C. |
| Tampão S30B | 14 mM de Mg-glutamato, 60 mM de K-glutamato, ~ 5 mM de Tris, pH 8,2 | 2 L | Autoclave | Para atingir pH 8,2, titular com 2MTris. Adicionar DTT a 1 mM concentração final imediatamente antes da utilização. Armazenar a 4 ° C. |
Tabela 2. Reagentes para o dia 2 de protocolo extrato celular bruto.
| Falcão | ||||
| 1 | 2 | 3 | 4 | |
| Vazio de 50 ml Falcon (g) | ||||
| 50 ml Falcon com pellet (g) | ||||
| Massa Pellet (50 ml Falcon com pellet - vazio 50 ml Falcon) (g) | ||||
| Volume de tampão S30A adicionar (massa sedimento * 0.9) (ml) | ||||
| Massa total de contas para adicionar (massa pellet * 5.0) (g) | ||||
Tabela 3. Tampão S30A e calculadora massa talão, para o dia 3 de protocolo extrato celular bruto.
| Nome | Concentração | Quantidade | Esterilização | Notas |
| HEPES | 2 M, pH 8 | 4 ml | Nenhum | Para chegar a pH 8, titular com KOH. |
| Nucleotide Mix | ATP e GTP 156 mM, CTP e UTP, 7,5 mM de pH 94 | 1,5 ml | Nenhum | Para atingir pH 7,5, titular com KOH. |
| tRNA | 50 mg / ml | 600 mL | Nenhum | |
| CoA | 65 mM | 600 mL | Nenhum | |
| NAD | 175 mM, pH 7,5-8 | 300 ul | Nenhum | Para atingir pH 7,5-8, titular com Tris a 2 M. |
| cAMP | 650 mM, pH 8 | 200 ul | Nenhum | Para chegar a pH 8, titular com Tris a 2 M. |
| O ácido folínico | 33,9 mM | 300 ul | Nenhum | Apesar de serem necessárias apenas 300 mL, em receita suplementar é de 1,15 ml. |
| Espermidina | 1 M | 150 ul | Nenhum | Armazenar a 4 ° C, aquecer a 37 ° C para derreter. |
| 3-PGA | 1,4 M, pH 7,5 | 3,2 ml | Nenhum | Para atingir pH 7,5, titular com Tris a 2 M. |
Tabela 4. Reagentes para se preparar para o protocolo Energy Solution.
Material suplementar 1. Receitas para itens.
O cloranfenicol, 34 mg / ml: Preparar 0,51 g cloranfenicol e adicionar etanol a 15 ml. Filtro de esterilizar (0,22 mM), alíquota de 1 ml tubos, armazenar a -20 ° C para uso posterior.
2xYT + P + Cm placa de ágar: Preparar 1,24 g 2xYT, solução dibásico 1,6 ml de fosfato de potássio @ 1 M, 0,88 ml de solução de fosfato de potássio @ 1 M, 0,6 g de agar e água a 40 ml. Autoclave. Deixar arrefecer para 50 ° C e adicionar 40 ul Cm. Alíquota de 25 ml em uma placa de Petri de 100 x 15 mm, e deixe esfriar por uma hora.
2xYT + P media: Preparar 124 g de 2 x YT, solução dibásica de fosfato de 160 ml de potássio @ 1 M, solução de 88 ml de fosfato de potássio monobásico @ 1 M e de água a 4 L. Aliquot fora em 2 x 1,88 L e 0,24 L. Autoclave.
Tris base, 2 M: Prepare 60,57 g de base Tris e água para 250 ml. Esterilizar, armazenar à temperatura ambiente para uso posterior.
DTT, 1 M: Preparar 2,31 g DTT e água para 15 ml. Filtro de esterilizar (0,22 mM), alíquota de 1 ml tubos, armazenar a -20 ° C para uso posterior.
Tampão S30A: Preparar 10,88 g de Mg-glutamato e 24,39 g de K-glutamato, 50 ml de Tris a 2 M, ácido acético (para pH 7,7), e água a 2 L. Autoclave, armazenar a 4 ° C, adicionar 4 ml de 1 M DTT antes do uso.
Tampão S30B: Preparar 10,88 g de Mg-glutamato e 24,39 g de K-glutamato, Tris a 2 M (a pH 8,2), e água a 2 L. Autoclave, armazenar a 4 ° C, adicionar 2 ml de DTT 1 M antes da utilização.
HEPES: Preparar 1,91 g de HEPES (MW 238,21), KOH (a pH 8), e água até 4 ml.
ARNt: Preparar 30 mg de tRNA e água para 600 ul.
CoA: Preparar 30 mg de CoA (MW 767,53) e água para 600 ul.
NAD: Adicionar 34,83 mg de NAD (MW 663,43), Tris a 2 M (a pH 7,5-8), e água para 300 ml. (Adicionar 27 ul de Tris a 2 M para trazer a solução para pH 7,5-8).
AMPc: Adicionar 42,80 mg de AMPc (MW 329,22), Tris a 2 M (a pH 8), e água para 200 ul. (Adicionar 73 ul de Tris a 2 M para trazer a solução para pH 8).
Ácido folinico (33,9 mM): 20 mg de sal de cálcio de ácido folínico sólido (PM 511,5), adicionar 1,15 ml de água.
Espermidina: Prepare 23,55 mL de espermidina (MW 145,25) e água para 150 mL. Prepare à temperatura ambiente após a fusão brevemente a 37 ° C.
3-PGA: Adicionar 1,03 g de 3-PGA (MW 230.02), Tris a 2 M (a pH 7,5), e água a 3,2 ml. (Adicionar 1,73 ml de Tris a 2 M para levar a solução a pH 7,5).
Nucleotide Mix: Adicionar 145 mg de ATP dipotássico dihidratado sal (MW 619,4), 133 mg de GTP sal dissódico (MW 567,14), 79,4 mg de sal dissódico di-hidratado CTP (MW 563,16), 82,6 mg de sal trissódico dihidratado UTP (MW 586,12) , KOH a uma diluição de 15% (até pH 7,5), e água a 1,5 ml. (Adicionar 353 mL de KOH a 15% de diluição para levar a solução a pH 7,5).
Material suplementar 2. Ensaio de Bradford.
- Retirar agente Bradford de 4 ° C e ajustado para a temperatura ambiente.
- Preparar 50 ul de BSA padrão a 1 mg / ml e de 0,1 mg / ml.
- Prepare 40 mL de 20x de diluição do extrato do passo 1.47.
- Adicionar 800 mL de água para 7 tinas.
- Prepare cuvetes padrão para 0 mg / mL, 1 mg / ml (10 ul de 0,1 mg / ml BSA), 2 mg / ml (20 ul de 0,1 mg / ml de BSA), 4 mg / ml (4 ul de 1 mg / ml BSA) , 6 mg / ml (6 ul de 1 mg / ml BSA).
- Prepare cuvetes experimentais para 2 mL de amostra e 4 ul de amostra.
- Adicionar 200 ul de agente de Bradford a cada cuvete e misturar bem by pipetagem. Incubar à temperatura ambiente durante pelo menos 10 min.
- Produção da curva padrão em OD 595 nm, utilizando cuvetes do passo 6.5. Rejeitar curva padrão, se r 2 <0,95.
- Determinar a concentração de extrato de OD 595 nm usando tinas do passo 6.6.
Material suplementar 3. Tampão planilha de calibração.
Veja TXTL_e (modelo) _calibration_JoVE.xlsx .
Material suplementar 4. Expressão livre-Cell executar planilha.
Discussão
TX-TL sistema de expressão livre de células baseado A Escherichia coli endógeno descrito aqui é uma reação fácil de executar três tubo que pode levar menos de oito horas de configurar a coleta de dados. O processo de criação de todos os reagentes exige cinco dias de tempo total (com exigências de trabalho significativas em apenas um dia), mas produz extrato bruto de 3.000 reações e buffer de tomada de reagentes para 10.000 reações (Figura 1). Além disso, o extrato bruto e buffer de tomada de reagentes são estáveis por pelo menos 1 ano a -80 ° C, o que permite usos múltiplos de uma preparação. 4 Por US $ 0,11 por 10 reação mL (0,26 dólar incluindo trabalho), os custos são 98% menores do que comparáveis sistemas comerciais (Figura 2).
Há algumas limitações não resolvidos, no entanto, para o sistema. A eficiência final de cada preparação de extracto celular bruto pode variar de acordo com a proficiência utilizador e das condições ambientais, apesar de tTípica variação de rendimento situa-se entre 5-10% (Figura 4b). Como um resultado disso, a variabilidade de lote para lote, em ambos expressão de ponto final e na dinâmica de expressão devem ser esperados. Essas variações provavelmente permanecerá até que o extrato é totalmente caracterizado ou até a criação de extrato é totalmente automatizado. Se o sistema de expressão isento de células é utilizado para conduzir as experiências quantitativas sensíveis, é aconselhável para executar todas as experiências com o mesmo lote de extracto celular bruto. O rendimento a partir de um único lote extracto celular em bruto, de cerca de 3000 reacções, deve ser suficiente para cursos experimentais típicos. Embora nós suspeitamos variação pode ser removido por ampliação e automatizar o procedimento, tais tentativas envolveria um investimento substancial de recursos.
Além disso, embora os níveis de expressão de ponto final são razoavelmente fácil de determinar, mais trabalho precisa ser feito de compreensão dinâmica intrínseca ao sistema livre de células. Sabe-se que tanto competitio recurson e limitação de recursos pode afetar a dinâmica de expressão. Por exemplo, Sigma endógena limitada 70 pode resultar em um regime de saturação com o aumento da produção de um molde de ADN de perfil de expressão análogo ao de depleção de nucleótidos ou de aminoácidos. 9,27 Contudo, dinâmica não tem que ser totalmente entendido a utilizar o sistema. Para aumentos puros de produtividade, otimização pode ser feito por métodos de aprendizado de máquina. 28 questões de concorrência de recursos e limitação pode ser dirigida por modelos matemáticos verificado utilizando dados experimentais.
O protocolo aqui apresentado é otimizado para uma cepa BL21-Rosetta2, mas é generalizável a outros E. coli. Modificações em BL21-Rosetta2, tais como a remoção do gene que codifica a protease lon e a adição de genes que codificam para tRNAs raros, para permitir que a produção de proteína máxima. Tentamos o protocolo com duas outras cepas de extrato - BL21 só e uma BL21 trxA nocaute-and encontrou 50% menos produção de proteína. Nossa hipótese é que os rendimentos da mesma forma diminuir quando se utiliza outras cepas. Outras mudanças nos parâmetros, como alternar meio de crescimento 2xYT para LB e outros caldos ricos, resultaram em produção de proteína diminuiu.
Sistemas de expressão livre de celulares que utilizam tanto endógenos e exógenos maquinaria de transcrição-tradução e mecanismos de regulação têm larga aplicação em proteínas e expressão de metabólitos e da biologia sintética. 3,29 Em vez de se limitar aos circuitos regulados-T7, pode-se imaginar a produzir biomoléculas complexas em um ambiente controlável pelo usuário usando uma mistura de E. nativa promotores de E. coli e os mecanismos de transcrição e regulação exogenamente fornecidos. Sem limitações de divisão celular e o metabolismo, a variabilidade em circuitos sintéticos, tais como o repressilator ou em vias metabólicas de engenharia, tais como aqueles que produzem artemisinina pode ser reduzida ou melhor compreendida. 30,31 Nós have usado essas vantagens para implementar interruptores genéticos, bem como para entender sigma fator seqüestro 9,32 Essa tecnologia também pode formar a espinha dorsal do "mínimo" ou células "artificiais" -. pequeno, bem caracterizados e unidades encarnados auto-suficientes de extrato. 33,34
Em última análise, nós antecipamos usos imediatos deste sistema de expressão livre de células endógenas como um ambiente de prototipagem para a biologia sintética. Apelidado de "TX-TL biomolecular placa de ensaio", o sistema de expressão isento de células fornece um ambiente controlado, onde circuitos sintéticos destinasse para a expressão in vivo pode ser submetido a ciclos de prototipagem - ciclos de ensaio com o plasmídeo básico, linear, ou ADN sintetizado quimicamente, seguido por análise e modificação rápida. Rounds prototipagem pode ser auxiliada por modelos matemáticos preditivos sendo desenvolvidos atualmente. Ao remover a clonagem e manipulação vivo para circuitos não-finais, prevemos engenhariatempos de ciclo nharia de ser reduzida para 1-3 dias em vez de padrão das correntes semanas.
Divulgações
Os autores declaram que não têm interesses financeiros concorrentes.
Agradecimentos
Agradecemos Jongmin Kim, Dan Siegal-Gaskins, Anu Thubagere, e Enoque Yeung de assistência racionalização do protocolo, e Clare Chen e Barclay Lee para a assistência nos estágios iniciais do projeto. Este material é baseado no trabalho apoiado em parte pela Agência de Projetos de Pesquisa Avançada de Defesa (DARPA / MTO) programa Viver Fundições, número do contrato HR0011-12-C-0065 (DARPA / CMO.ZZS é também apoiado por um cientista médico UCLA / Caltech Programa de Formação de comunhão e por um DoD, Força Aérea Escritório de Pesquisa Científica, Ciência de Defesa Nacional e de Pós-Graduação de Engenharia (NDSEG) Fellowship, 32 CFR 168. As opiniões e conclusões contidas neste documento são de responsabilidade dos autores e não deve ser interpretada como representando oficialmente as políticas, de forma expressa ou implícita, da Agência de Projetos de Pesquisa Avançada de Defesa ou do Governo dos EUA.
Materiais
| Name | Company | Catalog Number | Comments |
| 2xYT | MP biomedicals | 3012-032 | |
| 3-PGA | Sigma-Aldrich | P8877 | |
| ATP | Sigma-Aldrich | A8937 | |
| Bacto-agar | BD Diagnostics | 214010 | |
| Bead-beating tubes (polypropylene microvials) | BioSpec | 522S | |
| Beads, 0.1mm dia. | BioSpec | 11079101 | |
| BL21 Rosetta 2 E. coli strain | Novagen | 71402 | |
| Bradford BSA Protein Assay Kit | Bio-rad | 500-0201 | |
| cAMP | Sigma-Aldrich | A9501 | |
| Chloramphenicol | Sigma-Aldrich | C1919 | |
| CoA | Sigma-Aldrich | C4282 | |
| CTP | USB | 14121 | |
| Cuvettes, 1.5ml | Fisher | 14-955-127 | |
| DTT | Sigma-Aldrich | D0632 | |
| Folinic acid | Sigma-Aldrich | F7878 | |
| GTP | USB | 16800 | |
| HEPES | Sigma-Aldrich | H6147 | |
| K-glutamate | Sigma-Aldrich | G1149 | |
| Mg-glutamate | Sigma-Aldrich | 49605 | |
| Micro Bio-Spin Chromatography Columns | Bio-Rad | 732-6204 | |
| NAD | Sigma-Aldrich | N6522 | |
| Nunc 384-well optical bottom plates | Thermo-Scientific | 142761 | |
| Nunc sealing tape | Thermo-Scientific | 232701 | |
| PEG-8000 | Promega | V3011 | |
| Potassium phosphate dibasic solution | Sigma-Aldrich | P8584 | |
| Potassium phosphate monobasic solution | Sigma-Aldrich | P8709 | |
| RTS Amino Acid Sampler | 5 Prime | 2401530 | |
| Slide-A-Lyzer Dialysis Cassettes, 10k MWCO (Kit) | Thermo-Scientific | 66382 | |
| Spermidine | Sigma-Aldrich | 85558 | |
| Tris base | Fischer | BP1521 | |
| tRNA (from E. coli) | Roche Applied Science | MRE600 | |
| UTP | USB | 23160 | |
| 1L Centrifuge Bottle | Beckman-Coulter | A98813 | This is specific for Avanti J-series; obtain equivalent size for centrifuge in use. |
| 4L Erlenmeyer Flask | Kimble Chase | 26500-4000 | |
| Avanti J-26XP Centrifuge | Beckman-Coulter | 393127 | Or 1L-capable centrifuge equivalent. |
| Forma 480 Orbital Shaker | Thermo Scientific | 480 | Or chest-size 6x4L shaker equivalent. |
| JLA-8.1000 Rotor | Beckman-Coulter | 363688 | Or 1L-capable, 5000 x g rotor equivalent for centrifuge. |
| Mini-Beadbeater-1 | BioSpec | 3110BX | |
| Supplemental Material 1. Recipes for Items. Chloramphenicol, 34 mg/ml: Prepare 0.51 g chloramphenicol and add ethanol to 15 ml. Filter sterilize (0.22 μM), aliquot to 1 ml tubes, store at -20 °C for later use. 2xYT+P+Cm agar plate: Prepare 1.24 g 2xYT, 1.6 ml potassium phosphate dibasic solution @ 1 M, 0.88 ml potassium phosphate monobasic solution @ 1 M, 0.6 g agar, and water to 40 ml. Autoclave. Let cool to 50 °C and add 40 μl Cm. Aliquot 25 ml into a 100x15 mm petri dish, and let cool for an hour. 2xYT+P media: Prepare 124 g 2xYT, 160 ml potassium phosphate dibasic solution @1 M, 88 ml potassium phosphate monobasic solution @ 1 M, and water to 4 L. Aliquot out into 2x1.88 L and 0.24 L. Autoclave. Tris base, 2 M: Prepare 60.57 g Tris base and water to 250 ml. Sterilize, store at RT for later use. DTT, 1 M: Prepare 2.31 g DTT and water to 15 ml. Filter sterilize (0.22 μM), aliquot to 1 ml tubes, store at -20 °C for later use. S30A buffer: Prepare 10.88 g Mg-glutamate and 24.39 g K-glutamate, 50 ml Tris at 2M, acetic acid (to pH 7.7), and water to 2 L. Autoclave, store at 4 °C, add 4 ml 1 M DTT before use. S30B buffer: Prepare 10.88 g Mg-glutamate and 24.39 g K-glutamate, Tris at 2 M (to pH 8.2), and water to 2 L. Autoclave, store at 4 °C, add 2 ml 1 M DTT before use. HEPES: Prepare 1.91 g HEPES (MW 238.21), KOH (to pH 8), and water to 4 ml. tRNA: Prepare 30 mg of tRNA and water to 600 μl. CoA: Prepare 30 mg of CoA (MW 767.53) and water to 600 μl. NAD: Add 34.83 mg of NAD (MW 663.43), Tris at 2 M (to pH 7.5-8), and water to 300 μl. (Add 27 μl of Tris at 2 M to bring the solution to pH 7.5-8). cAMP: Add 42.80 mg of cAMP (MW 329.22), Tris at 2 M (to pH 8), and water to 200 μl. (Add 73 μl of Tris at 2 M to bring the solution to pH 8). Folinic Acid (33.9 mM): To 20 mg of solid folinic acid calcium salt (MW 511.5), add 1.15 ml water. Spermidine: Prepare 23.55 μl of spermidine (MW 145.25) and water to 150 μl. Prepare at room temperature after melting briefly at 37 °C. 3-PGA: Add 1.03 g of 3-PGA (MW 230.02), Tris at 2 M (to pH 7.5), and water to 3.2 ml. (Add 1.73 ml of Tris at 2 M to bring the solution to pH 7.5). Nucleotide Mix: Add 145 mg of ATP dipotassium salt dihydrate (MW 619.4), 133 mg of GTP disodium salt (MW 567.14), 79.4 mg of CTP disodium salt dihydrate (MW 563.16), 82.6 mg of UTP trisodium salt dihydrate (MW 586.12), KOH at 15% dilution (to pH 7.5), and water to 1.5 ml. (Add 353 μl of KOH at 15% dilution to bring the solution to pH 7.5). Supplemental Material 2. Bradford Assay.
See TXTL_e(template)_calibration_JoVE.xlsx. Supplemental Material 4. Cell-free expression run spreadsheet. See TXTL _JoVE.xlsx. | |||
Referências
- Noireaux, V., Bar-Ziv, R., Libchaber, A. Principles of cell-free genetic circuit assembly. Proceedings of the National Academy of Sciences of the United States of America. 100, 12672-12677 (2003).
- He, M. Y., He, Y. Z., Luo, Q., Wang, M. R. From DNA to protein: No living cells required. Process Biochem. 46, 615-620 (2011).
- Forster, A. C., Church, G. M. Synthetic biology projects in vitro. Genome Res. 17, 1-6 (1101).
- Shin, J., Noireaux, V. Efficient cell-free expression with the endogenous E. Coli RNA polymerase and sigma factor 70. Journal of biological engineering. 4, 8 (2010).
- Shimizu, Y., et al. Cell-free translation reconstituted with purified components. Nat Biotechnol. 19, 751-755 (2001).
- Shin, J., Noireaux, V. An E. coli Cell-Free Expression Toolbox: Application to Synthetic Gene Circuits and Artificial Cells. Acs Synth Biol. 1, 29-41 (2012).
- Shin, J., Noireaux, V. Study of messenger RNA inactivation and protein degradation in an Escherichia coli cell-free expression system. Journal of biological engineering. 4, 9 (2010).
- Shin, J., Jardine, P., Noireaux, V. Genome Replication, Synthesis, and Assembly of the Bacteriophage T7 in a Single Cell-Free Reaction. Acs Synth Biol. 1, 408-413 (2012).
- Siegal-Gaskins, D., Noireaux, V., Murray, R. M., Pao, L., Abramovitch, D. Biomolecular resource utilization in elementary cell-free gene circuits. , 1531-1536 (2013).
- Karzbrun, E., Shin, J., Bar-Ziv, R. H., Noireaux, V. Coarse-grained dynamics of protein synthesis in a cell-free system. Physical review letters. 106, 048104 (2011).
- Hoagland, M. B., Stephenson, M. L., Scott, J. F., Hecht, L. I., Zamecnik, P. C. Soluble Ribonucleic Acid Intermediate in Protein Synthesis. J Biol Chem. 231, 241-257 (1958).
- Wood, W. B., Berg, P. Effect of Enzymatically Synthesized Ribonucleic Acid on Amino Acid Incorporation by a Soluble Protein-Ribosome System from Escherichia Coli. Proceedings of the National Academy of Sciences of the United States of America. 48, 94 (1962).
- Zubay, G. In-Vitro Synthesis of Protein in Microbial Systems. Annu Rev Genet. 7, 267-287 (1973).
- Pratt, J. M., Hames, B. D., Higgins, S. J. . Transcription and Translation: A Practical Approach. , 179-209 (1984).
- Kim, H. C., Kim, D. M. Methods for energizing cell-free protein synthesis. Journal of bioscience and bioengineering. 108, 1-4 (2009).
- Michel-Reydellet, N., Calhoun, K., Swartz, J. Amino acid stabilization for cell-free protein synthesis by modification of the Escherichia coli genome. Metabolic engineering. 6, 197-203 (2004).
- Liu, D. V., Zawada, J. F., Swartz, J. R. Streamlining Escherichia coli S30 extract preparation for economical cell-free protein synthesis. Biotechnology progress. 21, 460-465 (2005).
- Andrianantoandro, E., Basu, S., Karig, D. K., Weiss, R. Synthetic biology: new engineering rules for an emerging discipline. Molecular systems biology. 2, 2006.0028 (2006).
- Kwok, R. Five hard truths for synthetic biology. Nature. 463, 288-290 (2010).
- Tabor, S., Richardson, C. C. A bacteriophage T7 RNA polymerase/promoter system for controlled exclusive expression of specific genes. Proceedings of the National Academy of Sciences of the United States of America. 82, 1074-1078 (1985).
- Lewicki, B. T., Margus, T., Remme, J., Nierhaus, K. H. Coupling of rRNA transcription and ribosomal assembly in vivo. Formation of active ribosomal subunits in Escherichia coli requires transcription of rRNA genes by host RNA polymerase which cannot be replaced by bacteriophage T7 RNA polymerase. Journal of molecular biology. 231, 581-593 (1993).
- Iskakova, M. B., Szaflarski, W., Dreyfus, M., Remme, J., Nierhaus, K. H. Troubleshooting coupled in vitro transcription-translation system derived from Escherichia coli cells: synthesis of high-yield fully active proteins. Nucleic acids research. 34, e135 (2006).
- Kigawa, T., et al. Preparation of Escherichia coli cell extract for highly productive cell-free protein expression. Journal of structural and. 5, 63-68 (2004).
- Matsuda, T., et al. Improving cell-free protein synthesis for stable-isotope labeling. Journal of biomolecular. NMR. 37, 225-229 (2007).
- Sitaraman, K., et al. A novel cell-free protein synthesis system. Journal of biotechnology. 110, 257-263 (2004).
- Becskei, A., Serrano, L. Engineering stability in gene networks by autoregulation. Nature. 405, 590-593 (2000).
- Maeda, H., Fujita, N., Ishihama, A. Competition among seven Escherichia coli sigma subunits: relative binding affinities to the core RNA polymerase. Nucleic acids research. 28, 3497-3503 (2000).
- Caschera, F., et al. Coping with complexity: machine learning optimization of cell-free protein synthesis. Biotechnology and bioengineering. 108, 2218-2228 (2011).
- Hodgman, C. E., Jewett, M. C. Cell-free synthetic biology: thinking outside the cell. Metabolic engineering. 14, 261-269 (2012).
- Elowitz, M. B., Leibler, S. A synthetic oscillatory network of transcriptional regulators. Nature. 403, 335-338 (2000).
- Tsuruta, H., et al. High-level production of amorpha-4,11-diene, a precursor of the antimalarial agent artemisinin, in Escherichia coli. Plos One. 4, e4489 (2009).
- Gardner, T. S., Cantor, C. R., Collins, J. J. Construction of a genetic toggle switch in Escherichia coli. Nature. 403, 339-342 (2000).
- Jewett, M. C., Forster, A. C. Update on designing and building minimal cells. Current opinion in biotechnology. 21, 697-703 (2010).
- Noireaux, V., Libchaber, A. A vesicle bioreactor as a step toward an artificial cell assembly. Proceedings of the National Academy of Sciences of the United States of America. 101, 17669-17674 (2004).
Erratum
Formal Correction: Erratum: Protocols for Implementing an Escherichia coli Based TX-TL Cell-Free Expression System for Synthetic Biology
Posted by JoVE Editors on 1/01/1970. Citeable Link.
A correction to Figure 5's legend has been made for the article Protocols for Implementing an Escherichia coli Based TX-TL Cell-Free Expression System for Synthetic Biology. Method 1 and method 2 have been switched.Z
The figure legend was update from:
Figure 5. Effects of DNA solution on expression efficiency. a) Comparison of two different purification methods for processing plasmids. 1 nM of pBEST-OR2-OR1-Pr-UTR1-deGFP-T500 is prepared using only a QiaPrep Spin Miniprep Kit (Purification method 1) or post-processed with a QiaQuick PCR purification kit (Purification method 2).
to
Figure 5. Effects of DNA solution on expression efficiency. a) Comparison of two different purification methods for processing plasmids. 1 nM of pBEST-OR2-OR1-Pr-UTR1-deGFP-T500 is prepared using only a QiaPrep Spin Miniprep Kit (Purification method 2) or post-processed with a QiaQuick PCR purification kit (Purification method 1).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados