Method Article
Protokolle für die Implementierung einer
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Erratum Notice
Zusammenfassung
Das Fünf-Tage-Protokoll beschreibt alle Schritte, Geräte und Zusatzsoftware notwendig für die Schaffung und den Betrieb einer effizienten endogenen Escherichia coli basierten TX-TL zellfreien Expressionssystem von Grund auf neu. Mit Reagenzien, nimmt das Protokoll 8 Stunden oder weniger, um das Setup eine Reaktion, sammeln und Prozessdaten.
Zusammenfassung
Ideal zellfreie Expressionssysteme können theoretisch emulieren einen zellulären in vivo-Umgebung in einem in vitro-Plattform gesteuert. 1 Dies ist nützlich für die Expression von Proteinen und genetischen Schaltungen kontrolliert, sowie zum Bereitstellen einer Prototypumgebung für synthetische Biologie. 2,3 Um letzteres Ziel zu erreichen, zellfreie Expressionssysteme, die erhalten endogenen Escherichia coli Transkriptions-Translations-Mechanismen sind in der Lage, in vivo zellulären Dynamik als solche auf Basis von T7-RNA-Polymerase-Transkription genauer widerspiegeln. Wir beschreiben die Vorbereitung und Durchführung eines effizienten endogenen E. coli basiert Transkriptions-Translations-(TX-TL) zellfreien Expressionssystem, das äquivalente Mengen an Protein, wie T7-basierten Systemen zu einer Kostenreduktion bis zu 98% ähnlich wie kommerzielle Systeme produzieren kann. 4,5 Die Herstellung von Puffern und rohem Zellextrakt beschrieben, sowie die Ausführung einesdrei U-TX-TL-Reaktion. Das gesamte Protokoll dauert fünf Tage, um vorzubereiten und liefert genug Material für bis zu 3000 einzelne Reaktionen in einer Zubereitung. Einmal hergestellt, nimmt jeder Reaktion unter 8 Stunden von der Einrichtung der Datensammlung und-analyse. Mechanismen der Regulation und Transkription exogener E. coli, wie lac / Tet-Repressoren und T7-RNA-Polymerase, ergänzt werden. 6 Endogene Eigenschaften, wie mRNA und DNA-Abbauraten können ebenfalls eingestellt werden. 7 Der TX-TL zellfreien Expressionssystem für nachgewiesen groß Maßstab Schaltungsanordnung, die Erkundung der biologischen Phänomene und Expression von Proteinen unter beiden T7-und endogene Promotoren. 6,8 Begleitende mathematische Modelle zur Verfügung stehen. 9,10 Das resultierende System hat einzigartige Anwendungen in der Synthetischen Biologie als Prototyping-Umgebung, oder "TX- TL biomolekularen Steckbrett. "
Einleitung
Zellfreie Expression Technologie begann in den 1950er Jahren als rein translatorische, voran Jahre später gekoppelten Transkriptions-Translationsmechanismen umfassen unter Verwendung von T7 Bakteriophagen-DNA. 11,12 Seitdem wurden zahlreiche Anstrengungen unternommen worden, um die Erstellung von rohen Zellextrakt (oder E optimieren . coli S30-Extrakt). 13,14 Diese Optimierungen umfassen Verlängerung zellfreien Proteinsynthese durch ATP-Regeneration oder Belastung Modifikationen und Protokoll reduziert Zeit und Kosten. 15-17 Alternative zellfreie Expressionssysteme existieren, die Verwendung wiederhergestellten Komponenten anstelle von Rohöl Zellextrakt zur Expression. 5 Sowohl Zellrohextrakt und Rekonstitution Methoden für den kommerziellen Einsatz entwickelt worden.
Mit dem Aufkommen der synthetischen Biologie, gibt es einen erhöhten Bedarf für eine gut charakterisierte Plattform zu testen und Express entwickelt biologische Module und Schaltungen. 18,19 Diese Plattform mussvielseitig, gut charakterisiert, einfach zu manipulieren und auf Benutzer gelieferten Komponenten konzentriert. Obwohl er ein halbes Jahrhundert früher entwickelt, die zellfreie Systeme auf E. coli eigen teilen diese Anforderungen, da sie eine vereinfachte Darstellung der in-vitro-zelluläre Prozesse, ohne die Komplexität des Wachstums und des Stoffwechsels sind. Zusätzlich werden alle der grundlegenden Kenntnisse aus in vivo-Arbeit an E. coli gilt ohne weiteres auf E. coli zellfreien Systemen.
Obwohl zellfreien Expressionssystemen können Anwendungen in der synthetischen Biologie haben, um das Ziel der meisten zellfreie Expressionssysteme datieren ist die Maximierung der Protein-und Metabolit-Ausbeute. Dies wird durch Verwendung von T7-Bakteriophagen Transkription von Sequenzen durch T7-Promotoren gesteuert durchgeführt. 20. Obwohl die Expression ist effizient und robust, diese Systeme dienen einem hoch spezialisierten Zweck. Zellregulation Methoden sind begrenzt, muss Ziel-DNA-Vorlagen seinüberarbeitet, um T7-Promotoren sind, und bestimmte Sequenzen, wie ribosomale Komplexe können nicht transkribiert und zusammengebaut werden. 21,22 Bestehende zellfreie Expressionssysteme sind nicht in der Lage, hohe Ausbeuten zu erhalten, während die Erhaltung endogenen Regulationsmechanismen, eine Vielseitigkeit für synthetische Biologie erforderlich.
Wir haben eine endogene E. entwickelt coli zellfreien Expressionssystem, das die Effizienz der Proteinexpression von früheren Systemen nachgewiesen bewahrt aber fügt zusätzliche Vielseitigkeit, indem es die Expression und Regulation der Grundlage von sowohl endogenen als auch exogenen (T7 oder andere) Mechanismen. Das hier beschriebene Protokoll wird ursprünglich auf Kigawa et al. (2004) und Liu et al. (2005), hat aber wesentliche Änderungen. Es nutzt Mg-und K-Glutamat über Mg-und K-Acetat für mehr Effizienz, entfernt 2-Mercaptoethanol und lysiert Zellen mit einer Wulst-Beater. 17,23,24 Bead-Schläge über Homogenisierung gewählt,druckbasierten Verfahren oder Ultraschallbehandlung aufgrund ihrer geringeren Kosten und vergleichbaren Ausbeuten zu Konkurrenzsystemen. wird 23 3-Phosphoglycerinsäure (3-PGA) als Energiequelle verwendet, da festgestellt wurde, verglichen mit Kreatinphosphat überlegenen Ausbeuten und Protein Phosphoenolpyruvat. 4,25 Unser System kann bis zu 0,75 mg / ml Reporterprotein zu produzieren entweder mit einem sigma70 basierte Promotor mit Lambda-Phagen-Betreiber oder einen T7-Promotor angetrieben, ähnlich wie die Erträge aus anderen kommerziellen Systemen. 4,6 Fünf Tage sind erforderlich, um alle notwendigen Reagenzien (Abbildung 1) zu produzieren. Darüber hinaus bietet es eine 98% Kostenersparnis gegenüber vergleichbaren kommerziellen zellfreien Systemen - Materialkosten sind $ 0,11 pro 10 ul-Reaktion, die auf $ 0,26 mit der Arbeit enthalten (Abbildung 2) steigt.
Protokoll
1. Zellrohextrakt Vorbereitung
Vorbereiten Zellrohextrakt über drei Tage zwei Personen benötigt, um effizient durchzuführen. Das Protokoll funktionell besteht aus drei Teilen: Kultur-Wachstum (1,1 Schritt zu Schritt 1.11), Zell-Lyse (Schritt 1,12 bis 1,37 Schritt), und extrahieren Klärung (Schritt 1,38 bis 1,52 Schritt). Es wird präsentiert in Tage unterteilt für die Bequemlichkeit. Ideal Extrakt 0,75 mg / ml deGFP aus Plasmid pBEST-OR2-OR1-Pr-UTR1-deGFP-T500 (Addgene # 40019) und hat einen rohen Zellextrakt Konzentration zwischen 27-30 mg / ml Protein. 4 erzeugen jedoch , Extrakt Eigenschaften variieren von Charge zu Charge. Das folgende Rezept liefert für rund 3.000 Einzelreaktionen (6 ml Zellrohextrakt) genug. Wenn Verkleinerung, empfiehlt es sich, nicht weniger als 1/6 der angegebenen Werte zu verwenden. Aus Zeitgründen, die Aufstockung wird nicht empfohlen.
Tag 1
- Bereiten Bakterienkultur Medien, Kulturtur Platte und Medien ergänzt, wie in Tabelle 1 beschrieben. Siehe ergänzende Material ein für Rezepte.
- . Streifen BL21-Stamm Rosetta2 von -80 ° C auf eine 2 × YT + P + Cm Agarplatte und Inkubation für mindestens 15 h bei 37 ° C oder bis Kolonien sichtbar sind ohne Hinweis: Chloramphenicol (Cm) wird verwendet, um ein Plasmid auszuwählen seltene tRNAs kodieren, in der BL21-Rosetta2 Belastung.
Tag 2
- Bereiten Puffer und Nahrungsergänzungsmittel, wie in Tabelle 2 beschrieben. Siehe ergänzende Material ein für Rezepte.
- Vorbereiten und sterilisieren Materialien für Tag 3 erforderlich, einschließlich: 6 x 4 L Erlenmeyerkolben mit Aluminiumfolie Deckel (autoklaviert), 4 x 1 L steriles Zentrifugenflaschen, Trichter (autoklaviert), 100 g 0,1 mm Glasperlen (autoklaviert), 2 Stir-Bars (autoklaviert), 1 L und 500 ml Messzylinder (autoklaviert), 2 x 1 L Bechergläser (autoklaviert), 3 ml Spritze mit 18 G Nadeln (steril), 2-3 flHafer Bojen, 2-3 10k MWCO Dialysekassetten (steril), Küvetten.
- Bereiten Mini-Kultur ein. 4 ml 2 × YT-Medium + P und 4 ul cm bis 12 ml steriles Kulturröhrchen und Vorwärmen auf 37 ° C für 30 min.
- Impfen Mini-Kultur-1 mit einer Kolonie von der 2xYT + P + Cm-Agar-Platte. Inkubation bei 220 rpm, 37 ° C für 8 Stunden.
- 7 Stunden und 30 Minuten später, bereiten Mini-Kultur 2. 50 ml 2 × YT-Medium + P und 50 &mgr; l Cm in eine sterile 250 ml Erlenmeyerkolben und Vorwärmen auf 37 ° C für 30 min.
- Minikultur inokulieren 2 mit 100 ul Minikultur 1 und Inkubation bei 220 rpm, 37 ° C für 8 Stunden.
Tag 3
- Wiegen vier leere sterile 50 ml Falcon-Röhrchen und Plattenmasse in Tabelle 3. Chill-Falcon-Röhrchen auf Eis, diese werden anschließend in Schritt 1.18 verwendet werden.
- 7 Stunden und 30 Minuten nach Schritt 1.8, Vorbereitung Abschlussbakterienkultur-Medien. Mit einer sterilen 1 L Messzylinder, Transfer 660 ml 2 × YT + P Media into jeder von sechs 4 L Erlenmeyerkolben und Pre-warm bis 37 ° C für 30 min. Hinweis: 4 L oder größer Erlenmeyerkolben werden für die richtige Belüftung empfohlen.
- In 6,6 ml Mini-Kultur 2 in je 4 L Erlenmeyerkolben. Inkubation bei 220 rpm, 37 ° C, bis die Kultur eine OD von 1,5-2,0 bei 600 nm (entsprechend dem mittleren log-Wachstumsphase) erreicht. Überprüfen Sie regelmäßig mit einem OD 1.10 Kultur Verdünnung für Richtigkeit Dieser Schritt sollte nicht mehr als 3 Stunden dauern - 3 h 45 min;. Schnelle Wachstum und die Sammlung während der mid-log-Phase ist entscheidend für die Qualität Extrakt.
- Unmittelbar nach dem Wachstum, übertragen alle Kulturen gleichmäßig in vier 1 l-Zentrifugenflaschen und Zentrifugieren bei 5000 × g für 12 min bei 4 ° C, um bakterielle Zellen zu pelletieren.
- Während der Zentrifugation abgeschlossen S30A Pufferherstellung durch Zugabe von 4 ml 1 M DTT zu 2 l vorher hergestellt S30A. Mischen Sie und pflegen Puffer auf Eis.
- Bei der Zentrifugation beendet ist, Überstand vollständig aus Schritt 1.12 von Decanting und Blotting die Zentrifugenflaschen auf einer sterilen Papiertuch.
- In 200 ml S30A Puffer bei 4 ° C bis jeder der vier Zentrifugenflaschen, und schütteln Sie kräftig, bis die Flaschen Pellet wird vollständig ohne verbleibenden Klumpen gelöst. Zentrifugieren vier Flaschen bei 5000 g für 12 min bei 4 ° C.
- Überstand vollständig entfernen aus dem vorherigen Schritt durch Dekantieren und Lösch die Zentrifugenflaschen auf einer sterilen Papiertuch.
- Wiederholen Sie die Schritte 1.15 und 1.16.
- 40 ml S30A-Puffer bei 4 ° C zu jedem Zentrifugenflasche. . Übertragen Sie jede Tablette und S30A Kombination in ein gekühltes Falcon-Röhrchen von 1,9) Hinweis: Dieser Schritt ist, um die Pellets in einen kleineren Behälter übertragen.
- Zentrifugieren Sie die Falcon-Röhrchen bei 2000 g für 8 min bei 4 ° C Überstand entfernen durch Dekantieren.
- Re-Zentrifuge die Falcon-Röhrchen bei 2000 g für 2 min bei 4 ° C Vollständig zu entfernen Restüberstand mit einer Pipette. Halten Sie auf dem Eis.
- Wiegen Sie die funsere Falcon-Röhrchen mit Pellet-und Rekord Masse in Tabelle 3. Berechnen Pelletmasse, S30A Puffervolumen benötigt wird, und die Masse der Perlen auf die spezifischen Formeln in Tabelle 3 benötigt.
- Richtig beladen und Perlen Schlagen des Pellet ist wichtig, um die Qualität zu extrahieren, und ist die größte Herausforderung Schritt. Es wird empfohlen, um das Video, bevor Sie bewerten. Failure, um Luftblasen zu vermeiden und zu verteilen Perlen gleichmäßig in ineffizienten Extrakt führen.
- In der Höhe der S30A Puffer in Tabelle 3 bis jedes Falcon-Röhrchen, Vortex berechnet, bis sie homogen und werden wieder zu Eis.
- Während die anderen Falcon-Röhrchen auf Eis, fügen Perlen intermittierend zu einem einzigen Falcon-Röhrchen in drei Teilmengen, die jeweils mit 1/3 der Gesamt Perlen. Nach Zugabe von jeder Teilmenge von Perlen, Wirbel für 30 Sekunden. Zeigen Falcon-Röhrchen auf Eis zwischen Wirbel Schritte und nach der endgültigen Wirbel. Nach letzten aliquoten hinzugefügt werden, mussKügelchen gleichmäßig verteilt sind. Eine dicke Paste sollte gebildet werden.
- Eine 5 ml (Volumen) Pipettenspitze durch das Abschneiden der Ende mit einer sterilen Rasierklinge, ein 3-4 mm Öffnung zu schaffen. Dial Pipette 2 ml Hinweis: Verschiedene Sets und Pipettenspitzen bieten unterschiedliche Mengen von Sog, der nicht ausreicht, um zu ziehen und loslassen dicken Wulst-Zell-Lösung sein kann, eine 1 ml Pipettenspitze mit Ende entfernt werden kann, anstelle verwendet werden..
- Zeigen 20 Wulst-Prügel Röhrchen auf Eis.
- Stellen Sie sicher, hohe Viskosität von Zell-Bead-Lösung unter Verwendung von modifizierten Pipette. Es sollte viskos Punkt kaum Verlassen der Pipettenspitze während des Auswurfs ist. Wenn zu viskos, neu einstellen Pipettenspitze gemäß Schritt 1.25. Wenn nicht viskos genug ist, kann in Schritten von Perlen (Pelletmasse * 0,05) bis zu einer zulässigen Gesamtmasse von (Pelletmasse * 5.1) zugesetzt werden. Nach jeder Zugabe von Perlen, Wirbel für 30 Sekunden und zum Eis. Siehe Fig. 3a für eine Demonstration der Viskosität.
- Entfernen Wulst-Zell-Lösung von Falcon-Röhrchen mit modifizierten Pipette aufnehmen und in einem sterilen Perlenrohr schlagen, füllte es dreiviertel voll mit Korn-Zell-Lösung. Spin extrem kurz (1s) auf einen Zähler Mini-Zentrifuge, um Luftblasen zu entfernen, ohne Umverteilung von Perlen. Siehe Abb. 3b-d für Standbilder von Wulst-Schläge Rohrlade.
- Das Hinzufügen Wulst-Zell-Lösung, um einen konkaven Meniskus bilden.
- Fügen Sie einen sehr kleinen Tropfen Wulst-Zell-Lösung auf die Innenseite einer Kugel-Spiel gegen Rohrkappe, wobei Sie darauf achten, die Außenlippe der Kappe nicht behindern, sonst wird nicht der Wulst-schlagen Rohr nahe sufficientl y. Tippen Sie auf die Kappe auf einer ebenen Fläche und überprüfen, dass sich keine Luftblasen an der Unterseite der Kappe.
- Verschließe das Wulst-schlagen Rohr mit dem Wulst schlag Kappe aus dem vorherigen Schritt. Hand auf Assistent für Perle zu schlagen. Wenn es richtig gemacht, sollte der Deckel fest verschlossen werden, keine Luftblasen should sichtbar und wenig (wenn überhaupt) Wulst-Zell-Lösung sollte läuft. Wiederholen Sie den Ladevorgang, wenn die Luftblasen sichtbar oder die Kappe schließt nicht vollständig sind.
- Wirbel Falcon-Röhrchen aus Schritt 1.24 die verbleibenden Kügelchen Zell Lösung mäßige Verteilung der Kügelchen zu gewährleisten. Wiederholen Sie die Schritte, bis 1,28-1,31 Falcon Tube leer ist, wiederholen Sie die Schritte von 1,24 bis 1,31 für jede weitere Falcon-Röhrchen.
- Verhaltens Schritte 1,33-1,38 gleichzeitig. Haben Assistent Take gefülltes Kügelchen schlagRohre von 1,31 und auf Eis. Nach zwei gefüllte Kügelchen gegen Rohre wurden gesammelt und auf Eis für mindestens eine Minute gewesen, Perle beginnen zu schlagen.
- Schlagen Sie ein Rohr für 30 Sekunden bei 46 Umdrehungen pro Minute. Legen Sie den Kopf auf Eis für 30 sec, während gegen die andere Röhre.
- Wiederholen Sie den vorhergehenden Schritt, so dass jede gefüllte Kügelchen gegen Rohr wurde für 1 min Gesamt schlagen worden.
- Wiederholen Sie die Schritte 1,33-1,35 bis 8 gefüllt Wulst-Prügel Rohre (oder die maximale Höhe der Zentrifugen halten kann) hverarbeitet worden Ave. Dann konstruieren Filtergerät aus 15 ml Falcon (Abbildung 3e). Fügen Sie eine neue Perle-schlagen, flache Teil-Gesicht, auf den Boden einer 15 ml-Falcon. Dann entfernen Kappe aus verarbeiteten Wulst-Rohr schlagen und drücken Sie Mikro-Chromatographie-Säule fest auf Ende verarbeitet Perlenrohr schlagen, bis sie vollständig abgedichtet. Snap off Elution Ende der Mikro-Chromatographie-Säule, und legen Mikro-Chromatographie-Säule, Elution Ende nach unten, in leere Kügelchen-Sickenrohr. Legen Sie diese in komplexen 15 ml Falcon. Wiederholen Sie für alle 8 gefülltes Kügelchen-Rohre schlagen; auf Eis zu halten, wenn abgeschlossen.
- Zentrifuge 8 Filtervorrichtungen, Falcon Röhrchen geöffnet, bei 6.000 g für 5 min bei 4 ° C bis Extrakt und Pellet aus Perlen zu trennen.
- Stellen Sie sicher, jede Perle-Prügel Rohr lebensfähigen Extrakt hergestellt. Richtig schlagen Extrakt wird nicht trüb sein, und das Pellet wird zwei verschiedene Schichten haben. Entsorgen Sie alle trüben Röhren und den Überstand aus nicht trüben Rohre into Einzel 1,75 ml Mikrozentrifugenröhrchen, wobei so wenig wie möglich Pellet. Halten auf Eis, bis alle Wulst-Rohre schlagen verarbeitet wurden. Siehe Abbildung 3f Vergleich einer richtig gegen falsch verarbeitet Wulst-Prügel Röhre.
- Zentrifugenmikrozentrifugenröhrchen aus dem vorherigen Schritt bei 12.000 g für 10 min bei 4 ° C.
- Über Pellet freie Überstand in leere Kügelchen-Prügel Rohre mit einer Pipette, die Konsolidierung 500 ul in eine neue Perlenrohr schlagen.
- Inkubieren vorherigen Schritt, mit sicken schlagen Kappen entfernt, bei 220 rpm, 37 ° C für 80 min. Dieser Schritt verdaut verbleibenden Nukleinsäuren unter Verwendung endogenen Exonukleasen während der Wulst-Sicke freigesetzt und kann durch den Wulst-stand gegen Rohr bis in eine Gewebekulturröhrchen durchgeführt werden.
- Bereiten Dialyse Materialien. Komplette S30B Pufferherstellung durch Zugabe von 2 ml 1 M DTT zu 2 l vorher hergestellt S30B. Mix und fügen Sie 900 ml in jeweils zwei sterile 1 L Bechergläser. In sterilen Magnetrührer in jedem Becher, halten bei 4 ° C
- Nach Schritt 1.41 sollte Extrakt trübe aussehen. Konsolidieren Extrakt in 1,5 ml Aliquots in 1,75 ml Mikrozentrifugenröhrchen und Zentrifugieren bei 12000 g für 10 min bei 4 ° C.
- Mit einer Pipette, zu konsolidieren Pellet freie Überstand in 15 ml Falcon-Röhrchen auf Eis, und gut mischen durch Abdecken der Rohr-und Umkehr. Sparen Sie 10 ul der Überstand auf Eis für Schritt 1.47.
- Ermittlung der Gesamtmenge an Extrakt hergestellt und hydratisieren die notwendige Anzahl von 10K MWCO Dialysekassetten durch Eintauchen in S30B für 2 min, unter der Annahme, 2,5 ml Extrakt pro Kassette.
- Laden Kassetten mit 2,5 ml Extrakt. Jeder Becher kann bis zu 2 Kassetten nehmen, Dialyse, Rühren, bei 4 ° C für 3 Stunden Hinweis: Teilbelastung von Kassetten ist akzeptabel.. Dialyse erhöht die Proteinproduktionsausbeute.
- Im vorangegangenen Schritt charakterisieren ExtraktproteinKonzentration mit einem Bradford-Assay, wobei in Schritt 1.44 gespeichert Extrakt. Siehe Supplemental Material 2 für Details.
- Nach der Dialyse abgeschlossen ist, aliquoten Extrakt 1,5 ml in 1,75 ml Mikrozentrifugenröhrchen. Zentrifuge bei 12.000 g für 10 min bei 4 ° C Ein Pellet am Boden des Röhrchens zu bilden.
- Konsolidieren Sie klare Überstand aus dem vorherigen Schritt durch Pipettieren in ein 15 ml-Falcon-Röhrchen auf Eis. Homogenisieren durch Umdrehen 5-10x.
- Bezogen auf Konzentration von Bradford in Schritt 1.47 bestimmt, bestimmen Extraktmenge in einzelne 1,75-ml-Röhrchen aliquotiert. Jedes einzelne Rohr sollte mit einem Volumen von 810 bis 900 mg Gesamtprotein aufweisen. Auszug sollte ein Gesamtproteinkonzentration von mehr als 27 mg / ml haben. Dieser Schritt erfordert Unterstützung für zweckmäßig durchzuführen. Hinweis: Aliquot Extrakt unter 30 mg / ml in 30 ul Aliquots und Skalierung, wenn die Konzentration höher, beispielsweise aliquote Extrakt bei 28 mg / ml mit 30 ul und aliquoten Extrakt bei 32mg / ml um 28,1 ul.
- Aliquot Extrakt folgenden Schritt 1,50, wobei darauf zu Blasen zu vermeiden. Flash-Extrakt gefrier in flüssigem Stickstoff. Hinweis: Aliquots mit Blasen können durch Zentrifugieren bei 10.000 × g für 30 sec bei 4 ° C entfernt werden,
- Rohre von flüssigem Stickstoff mit einem Sieb entfernen und sofort bei -80 ° C Sicherheit: Schutzbrille tragen, die Kappen der Röhren-Extrakt kann sich lösen durch die Temperaturdifferenz zwischen flüssigem Stickstoff und Raumtemperatur.
2. Amino Acid Herstellung der Lösung
Aminosäurelösung sollte in Groß vorbereitet werden. Das folgende Rezept nutzt ein Komplettpaket der RTS-Aminosäure-Sampler, liefert genug für rund 11.000 Einzelreaktionen. Wenn Verkleinerung, wird empfohlen, auf nicht weniger als eine halbe Kit. Jede Aminosäure in die Aktie auf 1,5 ml, 168 mm geliefert, mit Ausnahme von Leucin bei 140 mM. Die endgültige Zusammensetzung der Aminosäurelösung ist:Leucin, 5 mm, alle anderen Aminosäuren, 6 mM. Dies ist 4x Arbeitskonzentration.
- Entfernen aller 20 Aminosäuren von -20 ° C und Auftauen bei Raumtemperatur. Einmal aufgetaut, bis Wirbel Aminosäuren lösen, Inkubation bei 37 ° C, falls erforderlich. Nach Aminosäuren gelöst sind, setzen alle Aminosäuren auf Eis außer Asn, Phe und Cys, die bei Raumtemperatur aufbewahrt werden. Cys möglicherweise nicht vollständig auflösen.
- Auf Eis, fügen Sie 12 ml steriles Wasser in ein steriles 50 ml Falcon-Röhrchen.
- 1,5 ml jeder Aminosäure in der folgenden Reihenfolge, kümmert sich um die Falcon-Röhrchen nach jeder Zugabe verwirbeln und die Lösung auf Eis zu halten: Ala, Arg, Asn, Asp, Gln, Glu, Gly, His, Ile, Lys, Met, Phe, Pro, Ser, Thr, Val, Trp, Tyr, Leu, Cys. Cys kann als Suspension zugegeben werden. Nach Zugabe, bis die Lösung Wirbel relativ klar, Inkubation bei 37 ° C, wenn nötig. Cys möglicherweise nicht vollständig auflösen.
- Aliquot Aminosäurelösung in 50 Röhrchen beiJeweils 26 ul auf Eis. Aliquot der Rest bei 500 ul pro Röhrchen auf Eis. Die 26 ul Aliquots wird zum Kalibrieren Extrakt verwendet werden, während die 500 ul Aliquots für die Vorbereitung Puffer verwendet werden. Während Aliquotierung, Vortex die wichtigsten Aktien häufig ungleiche Verteilung der Suspension zu vermeiden.
- Flash-Freeze-Aliquots in flüssigem Stickstoff und bei -80 ° C Sicherheit: Schutzbrille tragen, die Kappen der Röhren-Extrakt kann sich lösen durch die Temperaturdifferenz zwischen flüssigem Stickstoff und Raumtemperatur.
- Optional: Führen eines Aktivitätstests von neu gemacht Aminosäurelösung gegen bisherigen Amino Acid Solutions.
3. Energy Solution Vorbereitung
Energie Lösung sowohl zum Kalibrieren rohen Zellextrakt und zum Erzeugen Puffer verwendet wird, und sollte in Groß hergestellt werden. Das folgende Rezept liefert etwa 10000 Einzelreaktionen genug. Wenn Verkleinerung ist es empfnded um nicht weniger als 1/24 der angegebenen Werte zu verwenden. Wie der Energy Solution ist eine bedeutende monetären Kosten, zum ersten Mal Benutzer wollen auf 1/24 Maßstab herzustellen. Die endgültige Zusammensetzung des Energie-Lösung ist: HEPES pH 8, 700 mM, 21 mM ATP, GTP 21 mM, 12,6 mM CTP, UTP 12,6 mM, tRNA 2,8 mg / ml, 3,64 mM CoA, 4,62 mM NAD, 10,5 mM cAMP, Folinsäure 0,95 mM Spermidin 14 mM 3-PGA 420 mM. Das ist 14x Arbeitskonzentration. Falls gewünscht, kann jedes einzelne Element in der Tabelle 4 bei -80 ° C für die spätere Verwendung gespeichert werden.
- Entfernen aller Chemikalien in Tabelle 4 von -80 ° C, -20 ° C oder 4 ° C bis Raumtemperatur für 30 min.
- Stocklösungen, wie in Tabelle 4 beschrieben. Siehe ergänzende Material ein für Rezepte. Legen Sie alle Lösungen auf dem Eis nach der Zubereitung.
- In einem 15 ml-Falcon-Röhrchen, fügen Sie in der folgenden Reihenfolge, kümmert sich um die Falcon-Röhrchen nach jeder Zugabe und vortexen, um die Lösungen zu halten on Eis: 3,6 ml 2 M HEPES, 144 ul Wasser, 1,39 ml Nukleotid-Mix, 576 ul 50 mg / ml tRNA, 576 ul 65 mM CoA, 276 ul 175 mM NAD, 170 ul 650 mM cAMP, 288 ul 33,9 mM Folinsäure , 144 &mgr; l 1 M Spermidin und 3,09 ml 1,4 M 3-PGA.
- Aliquot Energy Solution in 50 Röhrchen mit 7 ul jeweils auf Eis. Aliquot der Rest bei 150 ul pro Röhrchen auf Eis. Die 7 ul Aliquots wird zum Kalibrieren Extrakt Während Aliquotierung, Vortex die wichtigsten Aktien häufig verwendet werden, während die 150 ul Aliquots für die Vorbereitung Puffer verwendet werden..
- Flash-Freeze-Aliquots in flüssigem Stickstoff und bei -80 ° C Sicherheit: Schutzbrille tragen, die Kappen der Röhren-Extrakt kann sich lösen durch die Temperaturdifferenz zwischen flüssigem Stickstoff und Raumtemperatur.
- Optional: Führen eines Aktivitätstests von neu gemacht Energy Solution gegen bisherigen Energy Solutions.
4. Buffer Vorbereitung
Buffer Vorbereitung erfordert den Abschluss der Zellrohextrakt Vorbereitung, Aminosäurelösung Vorbereitung, und Energy Solution Vorbereitung. Jeder Puffer ist einzigartig in einer Charge von rohen Zellextrakt. Mg-Glutamat, K-Glutamat und DTT (in dieser Reihenfolge) werden in diesem Abschnitt optimiert, um Reaktionen mit maximaler Ausdrucksebenen zu produzieren. Das folgende Protokoll nutzt eine bereits geschriebene Vorlage, TXTL_e (Vorlage) _calibration_JoVE.xlsx (Supplemental-Material 3), zu kalibrieren vorbereiteten rohen Zellextrakt und bereiten Puffer. Allerdings kann man auch kalibrieren rohen Zellextrakt herzustellen, und Puffer ohne Vorlage von Mg-Glutamat, K-Glutamat und DTT Optimierung manuell und Einrichten Puffer, so daß zusammen mit dem Extrakt, ist es 75% einer Gesamtreaktionsvolumen. Bei der manuellen Kalibrierung kann endgültigen Reaktionsbedingungen in Schritt 5 gefunden werden.
- Füllen Sie das "Allgemeine Daten" zu bilden.
- Auftauen auf Eis 100 mM Mg-glutamat (4 ° C), 3 M K-Glutamat (4 ° C), 6mM Aminosäurelösung (26 &mgr; l, 80 ° C), Energie-Lösung (7 &mgr; l, 80 ° C), 100 mM DTT (-20 ° C), positive Kontroll-DNA (-20 ° C), 40% PEG- . 8000 (4 ° C), rohen Zellextrakt (-80 ° C) und Wasser (4 ° C) Hinweis: Verwenden Sie 1 nM Arbeitskonzentration pBEST-OR 2-OR1-Pr-UTR1-deGFP-T500 (Addgene Plasmid-40019) für die positive Kontrolle (Anregung 485 nm, Emission 525 nm) oder eine andere Referenz, die hohe Signalintensität erzeugt. 4
- Bereiten sieben 10.5 ul Reaktionen Testen eines Bereich von 4-10 mM zusätzliche Mg-Glutamat durch Aliquotierung Satz Mengen an Lager Mg-Glutamat in einzelne Mikrozentrifugenröhrchen. Anmerkung: Obwohl 10,5 ul Reaktionen werden anfänglich hergestellt wird, ist die letzte Reaktion 10 ul.
- Bereiten Master-Mix, wie in der Vorlage unter angegeben "Mg-Glutamat-Kalibrierung", was einen zusätzlichen 80 mM K-Glutamat. Halten Sie sich auf Eis und Wirbel nach der Zugabe der einzelnen Elemente. Hinweis: Die hier und in der Vorlage angegebenen Werte sindzusätzlich zu den Mengen an Mg-Glutamat, K-Glutamat und DTT in der Gegenwart verwendet wird, um rohen Zellextrakt zu machen S30B Puffer.
- In Master-Mix, um Proben mit Mg-Glutamat und bereiten Reaktionen. Siehe Schritte 5,10-5,13 detaillierte Anleitung.
- Führen Reaktion bei 29 ° C, entweder in einem Brutschrank oder einem Plattenlesegerät.
- . Bestimmen Sie optimale Mg-Glutamat-Konzentration durch End-Expressionsniveau und die maximale Rate der Protein-Expression (Abbildung 4a) Hinweis: Laufzeiten variieren je nach Experiment aber in der Regel zuletzt unter 8 Stunden.
- Wiederholen Sie die Schritte 4.2 bis 4.7 für K-Glutamat unter "K-Glutamat-Kalibrierung"-Einstellung Mg-Glutamat Niveau wie in Schritt 4.7 gefunden.
- Wiederholen Sie die Schritte 4.2 bis 4.7 für DVB-T unter "DVB-T-Kalibrierung"-Einstellung Mg-Glutamat Niveau wie in Schritt 4.7 und K-Glutamat-Niveau wie in Schritt 4.8 gefunden. Hinweis: Wir haben festgestellt, dass DTT zugegeben nicht wesentlich beeinflusst End- Expressionsniveaus.
- VerwendenWerte der Kalibrierung unter "Pufferzusammensetzung" gefunden, um die Zusammensetzung des Puffers zu bestimmen, hergestellt werden. Bezogen auf die Menge an rohem Zellextrakt hergestellt wird, wird ein Master-Mix Rezept für eine bestimmte Menge von Puffern erzeugt.
- Auftauen Aliquots wie im Master-Mix auf Eis Rezept aufgelistet. Einmal aufgetaut, bereiten Master-Mix, halten auf Eis und Verwirbelung nach der Zugabe der einzelnen Elemente.
- Aliquot von Betrag ange unter "Puffer Zusammensetzung." Flash-Freeze-Puffer Röhrchen in flüssigem Stickstoff. Während Aliquotierung Wirbel der Hauptvorrat häufig aus.
- Rohre von flüssigem Stickstoff mit einem Sieb entfernen und sofort bei -80 ° C Sicherheit: Schutzbrille tragen, die Kappen der Röhren-Extrakt kann sich lösen durch die Temperaturdifferenz zwischen flüssigem Stickstoff und Raumtemperatur.
5. Experimentelle Durchführung einer TX-TL-Reaktion
Schlussreaktionsbedingungen sind: 8,9 bis 9,9 mg / ml protein (aus Roh-Extrakt), 4,5 mM-10.5 mM Mg-Glutamat, 40-160 mM K-Glutamat, 0,33-3,33 mM DTT, 1,5 mM jede Aminosäure außer Leucin, 1,25 mM Leucin, 50 mM HEPES, 1,5 mM ATP und GTP, 0,9 mM CTP und UTP, 0,2 mg / ml tRNA, 0,26 mM CoA, 0,33 mM NAD, 0,75 mM cAMP, 0,068 mM Folinsäure, 1 mM Spermidin, 30 mM 3-PGA, 2% PEG-8000. Ein Grund TX TL-Reaktion besteht aus 3 Teilen (Rohre): Zellrohextrakt, Puffer und DNA. Das Verhältnis ist: 75% Puffer und Extrakt, 25% DNA-Reaktionen können im Volumen variieren, und wir verwenden 10 ul durch Konvention zum Reaktionsvolumen zu minimieren und ermöglichen in einer 384-Well-Platte läuft.. Größere Volumina erfordern Agitation für die richtige Sauerstoffzufuhr. Das folgende Protokoll nutzt eine bereits geschriebene Vorlage, TXTL_JoVE.xlsx (Supplemental-Material 4), um eine 10 ul Reaktion durchzuführen. Angebote im lila zeigen Benutzer-Eingabewerte und Objekte in blau zeigen zusätzliche Reagenzien, um die Reaktion hinzuzufügen. Allerdings kann man auch eine Reaktion Witz führenhout die Vorlage, indem Sie oben skizzierten Reaktionsbedingungen.
- Füllen Sie das "Allgemeine Daten" zu bilden.
- Unter "Master Mix Vorbereitung," legen Sie die Extrakt Prozentwert aus Schritt 4.1 in die lila-Box.
- Entwerfen Sie Ihren Test in silico mit dem "Master Mix Vorbereitung" (Zeilen 10-17) und "DNA-Vorbereitung" (Zeilen 19-50) Abschnitte. Im Allgemeinen können Konstanten in den Abschnitt "Master Mix Vorbereitung" gestellt werden, während Variablen können in den Abschnitt "DNA-Vorbereitung" gestellt werden. Minimieren Proben pro Experiment zur Probenverdampfung und experimentelle Startzeit Verzerrungen zu vermeiden. Siehe Abbildung 6 für eine Probe-Setup.
- Unter "Master Mix Vorbereitung," add Reagenzien wie Induktoren oder Proteine, die in allen Proben bei einer konstanten Konzentration gehen. Ab Zeile 14, füllen Sie bitte die blau schraffierten Flächen, halten ein Reagenz, um jede Zeile. Die Einheiten sind relativen Verhältnisse.
- Unter "DNA-Vorbereitung," Add-DNA, die Probe spezifi sein wirdic. Probennummern # 1 und # 2 entsprechen den positiven und negativen Kontrollen. Proben-IDs # 3 und oben sind vom Benutzer veränderbar für die DNA-, Lager-Konzentration in ng / ul, Länge in Basenpaaren, gewünschte endgültige Konzentration in nM und Wiederholungen (von 10 ul Reaktionen). Die Anzahl der Aktien, um die gewünschten DNA-Endkonzentration erreichen wird automatisch berechnet. . Die Gesamtsummen für die Zeile auf 10,5 * n, wobei n die Anzahl der Wiederholungen Hinweis: Obwohl die endgültige Reaktionsvolumen 10 &mgr; l, die Berechnungen gehen von einem Gesamtvolumen von 10,5 &mgr; l pro Reaktion für Volumen beim Pipettieren verloren erklären.
- Unter "DNA-Vorbereitung," add Reagenzien oder zusätzliche DNA-Probe, die spezifisch für blaue Säulen sein werden. Auf DNA-Konzentrationen in nM unter "DNA-Vorbereitung," berechnet werden, während Probe spezifische Reagenzien erfordern manuelle Berechnung basierend auf einem Gesamtreaktionsvolumen von 10,5 * n. Die eingegebenen Mengen werden aus dem Wasservolumen des gleichen Zeile subtrahiert.
- Entfernen benötigten number von Röhren-Puffer, rohen Zellextrakt und positive Kontrolle unter "Tubes zu tauen", von -20 ° C oder -80 ° C und Auftauen auf Eis.
- Bereiten DNA-Proben. Für jede Probe ID aliquote aus der angegebenen DNA-, Wasser-, und Benutzer gelieferten Artikel pro Abschnitt "DNA-Vorbereitung" in einen Mikrozentrifugenröhrchen bei Raumtemperatur. Hinweis: zu Probenverlust zu vermeiden, vor kurzem kalibrierten Pipetten-und Low-Stick Pipettenspitzen und Mikrozentrifugenröhrchen werden empfohlen.
- Wenn Rohre aus Schritt 5.7 werden aufgetaut, bereiten Sie den Master-Mix, bestehend aus Puffer, extrahieren, und alle globalen Benutzer gelieferten Produkte auf der Basis der orange hinterlegten Feldern, halten auf Eis und Verwirbelung nach der Zugabe der einzelnen Elemente. Hinweis: Extract ist extrem viskos. Aliquots mit Blasen können durch Zentrifugieren bei 10.000 × g für 30 sec bei 4 ° C entfernt werden,
- In der Höhe der Master-Mix in den orangefarbenen Zellen unter "DNA-Vorbereitung" (Spalte O) in jede DNA-Probe zeigte, und halten bei Raumtemperaturtur. Behandeln Sie dies als Reaktion Startzeit.
- Wirbel jede Probe und Zentrifuge bei 10.000 × g für 30 Sekunden bei Raumtemperatur, um restliche Probe bringen und um Blasen zu reduzieren.
- Wenn die Durchführung Reaktion in Mikrozentrifugenröhrchen, Inkubation direkt bei 29 ° C . Andernfalls Pipette 10 ul Probe in ein 384-Well-Platte Hinweis: Reaktionen in Mengen von mehr als 10 ul kann Agitation für die Sauerstoffversorgung benötigen.
- Zentrifugenplatte bei 4.000 × g für 30 sec bei Raumtemperatur, um restliche Probe bringen und um Blasen zu reduzieren. Dichtungsplatte danach um Verdunstung zu verhindern.
- Führen Reaktion bei 29 ° C Hinweis: Laufzeiten variieren je nach Experiment je, aber in der Regel unter 8 Stunden dauern.
Ergebnisse
Wir haben einen Fünftage Protokoll zur Herstellung eines endogenen Escherichia coli basieren TX-TL zellfreien Expressionssystem dargestellt. Eine Probe Timeline für die Erstellung der Reagenzien - Zellrohextrakt und Puffer - ist in Abbildung 1 zu finden. Einmal erzeugt, so können diese bei -80 ° C für bis zu einem Jahr gelagert werden. Nach Reagenzien erstellt sind, können Versuchsaufbau und Durchführung in weniger als 8 Stunden durchgeführt werden.
Darüber hinaus optimiert die Expression Bedingungen des TX-TL zellfreien Expressionssystem. Andere Benutzer angegebenen Zusätze wie Puffer oder DNA-Lösungen sollten für Toxizität vorher kalibriert werden. Zum Beispiel ergeben sich unterschiedliche Verfahren der Verarbeitung Plasmide in anderen Ausdruck durch Salzgehalt. Wir testeten auch die Wirkung von Tris-Cl Elutionspuffer auf die Reaktionseffizienz (Abbildung 5).
Ein Beispiel eines rohen Zellextrakt Kalibrierung Bezugnahme Schritt 4,1 bis 4,9, dargestelltin Fig. 4a. Im allgemeinen zeigen unsere Versuche, dass der rohe Zellextrakt ist am empfindlichsten für Mg-Glutamat auf, gefolgt von K-Glutamat-Niveaus. Um die zellfreie Expressionssystem zu demonstrieren, konstruierten wir getestet und eine negative Rückkopplungsschleife auf der Basis tet Repression. 26 (Abbildung 6). In zellfreien Expressionssystem, die gleiche Schaltung Lauf mit und ohne ATC zeigt eine 7-fach-Endpunkt Ausdruck Wechsel deGFP Reporter nach acht Stunden des Ausdrucks. Obwohl dieses Experiment nicht erfordern globale Induktoren oder Repressoren, wenn nötig können sie unter "Master Mix Vorbereitung." Hinzugefügt werden
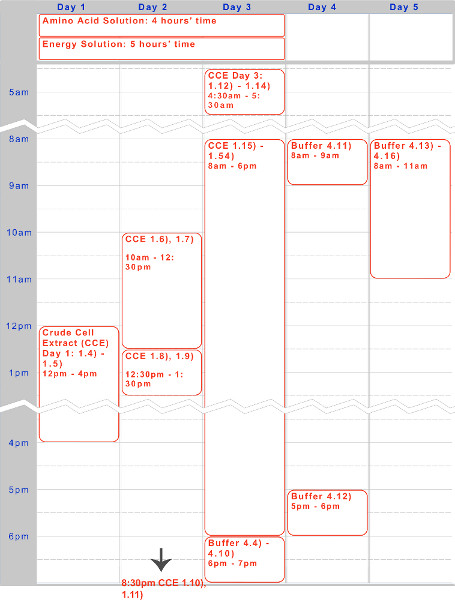
Fig. 1 ist. Timeline für rohen Zellextrakt, Aminosäure-Lösung und Energielösung Vorbereitung. Ein Fünf-Tages-timelin e für eine typische Ausführung des Protokolls ist oberhalb, da über Nacht Inkubationen und Tagesarbeitsschritte optimiert.
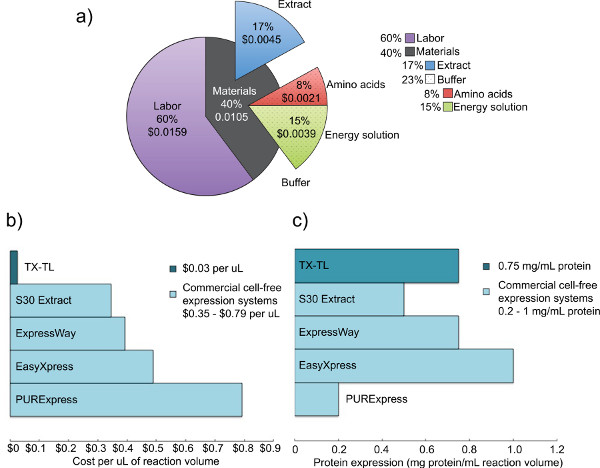
2. Kosten-und Expressionsanalyse von konkurrierenden rohen Zellextrakten. a) Aufschlüsselung der Kosten der Arbeit und Materialien der TX-TL zellfreien Expressionssystem. Basierend auf Kosten der Reagenzien im Dezember 2012 und der Arbeitskosten von $ 14 pro Stunde. B) Vergleich der TX-TL zellfreie Expressionssystemkosten im Vergleich zu anderen kommerziellen Systemen. Kosten werden pro ul gebrochen, obwohl Reaktionsvolumina pro Set. C) Vergleich der TX-TL zellfreien Expressionssystem Ausbeute im Vergleich zu anderen kommerziellen Systemen variieren. Proteinexpressionsausbeute von Herstellernormen festgelegt./ 50762fig2large.jpg "target =" _blank "> Klicken Sie hier für eine größere Abbildung zu sehen.

3. Be-und Verarbeitung von einer Wulst-schlagRohr. a) Demonstration der richtigen Viskosität von Zell-Bead-Lösung. Zell-Bead-Lösung wird eine Viskosität von vielen Faktoren abhängig, einschließlich Höhe der S30A-Puffer, Menge von Perlen aufgenommen, und die Zeit auf dem Eis verbracht. B) Laden der Wulst-Rohr schlagen vor schnellen Tisch Zentrifugieren. Die Zentrifugation entfernt Blasen während des Ladens angesammelt. C) Blasen nach Tisch Zentrifugation Beschichten. Die Größe der Blasen wird variieren, sie können aufgetaucht oder entfernt werden, mit einer Pipettenspitze d) vollständig gefülltes Kügelchen-Spiel gegen Rohr vor der Deckelung.. Ein Meniskus im Wulst-schlagen t gebildetube, und die Kappe hat genug zu decken und zu kleinen Mengen. füllen E) korrekt geladen Filtergerät. Diese können. F) Vergleich der richtig gegen falsch verarbeitet Perlen schlagen Rohr wiederverwendet werden. Das Rohr auf der linken Seite ist ein gut-Beat Rohr - es verfügt über eine kleine, gut abgegrenzte Oberschicht, und sehr klare Überstand. Das Rohr auf der rechten Seite nicht optimal sein, basierend auf dem größeren, trübe zweiten Schicht und der trübe Überstand. Rohre, die nicht optimal sind, sollten nicht weitere Verarbeitung erfolgt.
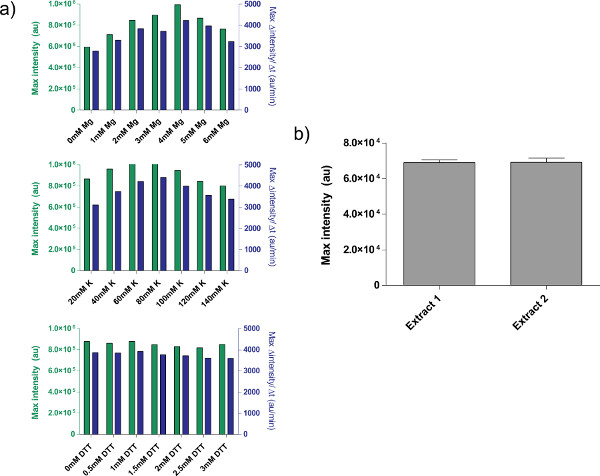
4. Eigenschaften der Rohextrakt Zubereitungen. a) Typische Eichkurven für Zellrohextrakt. Rohextrakt wird für weitere Mg-Glutamat, K-Glutamat und DTT Niveaus kalibriert ist, in dieser Reihenfolge. Dargestellt ist Endpunkt fluorescence nach 8 h, sowie maximale Rate der Proteinproduktion auf der Basis einer 12-minütigen Bewegungsdurchschnitt. Basierend auf diesen Grundstücken, ein akzeptables Angebot an Zusatz Mg-Glutamat ist 4 mM, K-Glutamat ist 60-80 mM, DTT und 0-3 mM. Beachten Sie, dass jedes Rohextrakt muss unabhängig für diese drei Variablen kalibriert werden. B) Änderung von Extraktpräparaten. Endpoint Fluoreszenz von zwei Rohextrakte an verschiedenen Tagen vorbereitet wird gezeigt, Fehlerbalken sind 1 Standardabweichung aus drei unabhängigen Läufen an verschiedenen Tagen. Klicken Sie hier, um eine größere Abbildung anzuzeigen .
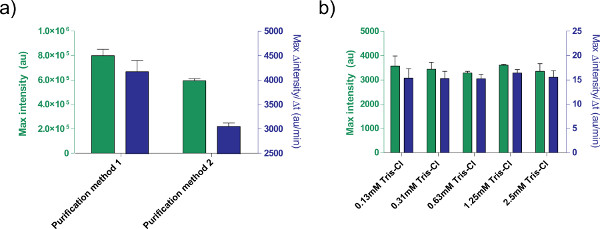
5. Auswirkungen der DNA-Lösung auf die Expression Effizienz. a) Vergleich von zwei unterschiedlichen ReinigungsVerfahren zur Verarbeitung von Plasmiden. 1 nM pBEST-OR 2-OR1-Pr-UTR1-deGFP-T500 ist mit nur einem QIAprep Spin Miniprep Kit (Reinigung Methode 1) oder mit einem QIAquick PCR Purification Kit nachbearbeitet vorbereitet (Reinigung Methode 2). Dargestellt ist Endpunkt Fluoreszenz nach 8 Stunden, sowie maximale Rate der Proteinproduktion auf der Basis einer 12-minütigen gleitenden Durchschnitt. Fehlerbalken sind 1 Standardabweichung aus vier unabhängigen Läufen an verschiedenen Tagen. B) Wirkung der Elutionspuffer (Tris-Cl). Verschiedene Konzentrationen von Tris-Cl in einer zellfreien Expressions Reaktion basierend auf der Expression von 1 nM pBEST-OR2-OR1-Pr-UTR1-deGFP-T500 verglichen. Angegebenen Konzentrationen sind Endkonzentrationen von Tris-Cl in der Reaktion; Elutionspuffer ist 10 mM Tris-Cl. Fehlerbalken sind 1 Standardabweichung aus drei unabhängigen Läufen an verschiedenen Tagen. Klicken Sie hier, um eine größere Abbildung anzuzeigen .
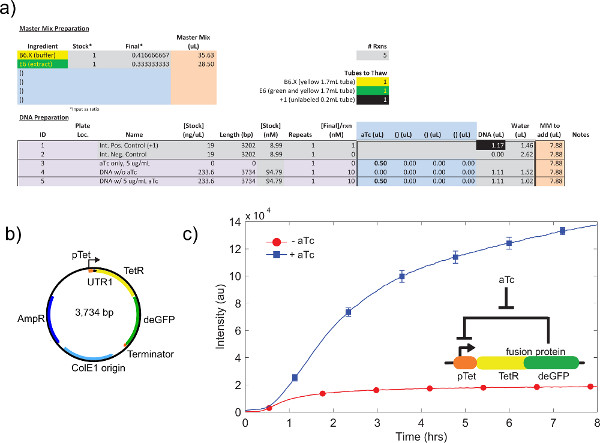
6. Beispiel TX-TL Lauf einer negativen Rückkopplungsschleife. a) Proben Einrichtung eines zellfreien Ausführung Reaktion. Tests "auf" gegen "Aus"-Zustand der negativen Rückkopplungsschleife, mit positiven und negativen Kontrollen. B) Plasmid Karte von negativen Rückkopplungsschleife. C) Repräsentative Ergebnisse. Daten spiegelt Experiment in a) und b) mit Negativkontrolle aus Signal subtrahiert. Genetische Schaltung in Einsatz gezeigt. Fehlerbalken sind 1 Standardabweichung aus drei unabhängigen Läufen an verschiedenen Tagen. Klicken Sie hier, um eine größere Abbildung anzuzeigen .
| Name | Konzentration | Menge | Sterilisation | Aufzeichnungen |
| Chloramphenicol (Cm) | 34 mg / ml in Ethanol | 1 ml | Filter zu sterilisieren (0,22 uM) | In größeren Mengen bei -20 º C zur späteren Verwendung gelagert werden. |
| 2xYT + P + Cm-Agar-Platte | 31 g / l 2 × YT, 40 mM Kaliumhydrogenphosphat, 22 mM monobasisches Kaliumphosphat, 34 ug / ml Chloramphenicol | 1 Platte | Autoklav | |
| 2xYT + P Media | 31 g / l 2 × YT, 40 mM Kaliumhydrogenphosphat, 22 mM Kaliumphosphat, monobasisches | 4 L | Autoklav |
Tabelle 1. Reagenzien für Tag 1 der Zellrohextrakt Protokoll.
| Name | Konzentration | Menge | Sterilisation | Aufzeichnungen |
| Tris-Base | 2 M | 250 ml | Filter zu sterilisieren (0,22 uM) oder Autoklaven | Kann bei Raumtemperatur gelagert werden. |
| DTT | 1 M | 6 ml | Filter zu sterilisieren (0,22 uM) | In größeren Mengen hergestellt und bei -20 ° C für die spätere Verwendung gespeichert werden. |
| S30A-Puffer | 14 mM Mg-Glutamat, 60 mM K-Glutamat, 50 mM Tris, pH 7,7 | 2 L | Autoklav | Um pH 7,7 zu erreichen, titriert mit Essigsäure. In DTT bis 2 mM Endkonzentration kurz vor der Verwendung. Lagern bei 4 ° C |
| S30B Puffer | 14 mM Mg-Glutamat, 60 mM K-Glutamat, ~ 5 mM Tris, pH 8,2 | 2 L | Autoklav | Auf pH 8,2 zu erreichen, titriert mit 2MTris. Hinzufügen DTT 1 mM Endkonzentration kurz vor der Verwendung. Lagern bei 4 ° C |
Tabelle 2. Reagenzien für Tag 2 von Zellrohextrakt Protokoll.
| Falke | ||||
| 1 | 2 | 3 | 4 | |
| Leere 50 ml Falcon (g) | ||||
| 50 ml Falcon mit Pellet (g) | ||||
| Pelletmasse (50 ml Falcon mit Pellets - leer 50 ml Falcon) (g) | ||||
| S30A Puffervolumen zu addieren (Pelletmasse * 0,9) (ml) | ||||
| Gesamtmasse von Perlen hinzufügen (Pelletmasse * 5.0) (g) | ||||
Tabelle 3. S30A Puffer-und Perlenmassen Rechner, für Tag 3 der Zellrohextrakt Protokoll.
| Name | Konzentration | Menge | Sterilisation | Aufzeichnungen |
| HEPES | 2 M, pH 8, | 4 ml | Keiner | Auf pH 8 zu erreichen, titriert mit KOH. |
| Nucleotide Mix | 156 mM ATP und GTP, 94 mM CTP und UTP, pH-Wert 7,5 | 1,5 ml | Keiner | Auf pH 7,5 zu erreichen, titriert mit KOH. |
| tRNA | 50 mg / ml | 600 ul | Keiner | |
| CoA | 65 mM | 600 ul | Keiner | |
| NAD | 175 mM, pH 7.5-8 | 300 ul | Keiner | Um pH-Wert von 7,5 bis 8 erreichen, titriert mit Tris bei 2 M. |
| cAMP | 650 mM, pH 8, | 200 ul | Keiner | Auf pH 8 zu erreichen, titriert mit Tris bei 2 M. |
| Folinsäure | 33,9 mM | 300 ul | Keiner | Obwohl nur 300 ul benötigt wird, ist Rezept für zusätzliche 1,15 ml. |
| Spermidin | 1 M | 150 ul | Keiner | Lagerung bei 4 ° C Hitze auf 37 ° C zu schmelzen. |
| 3-PGA | 1,4 M, pH 7,5 | 3,2 ml | Keiner | Auf pH 7,5 zu erreichen, titriert mit Tris bei 2 M. |
Tabelle 4. Reagenzien für Energy Solution-Protokoll vorzubereiten.
STADT Werkstoff 1. Rezepte für Gegenstände.
Chloramphenicol, 34 mg / ml: Bereiten 0,51 g Chloramphenicol und fügen Ethanol auf 15 ml. Filter zu sterilisieren (0,22 uM), aliquote 1-ml-Röhrchen bei -20 ° C für die spätere Verwendung.
2xYT + P + Cm-Agar-Platte: Bereiten 1,24 g 2xYT, 1,6 ml Kaliumhydrogenphosphat-Lösung @ 1 M, 0,88 ml Kaliumphosphat einbasischen Lösung @ 1 M, 0,6 g Agar und Wasser auf 40 ml. Autoklav. Abkühlen lassen bis 50 ° C und 40 &mgr; l Cm. Aliquot 25 ml in einen 100 x 15 mm Petrischale, und abkühlen lassen für eine Stunde.
2xYT + P Media: Vorbereiten 124 g 2xYT, 160 ml Kaliumhydrogenphosphat-Lösung @ 1 M, 88 ml Kaliumphosphat einbasischen Lösung @ 1 M, und Wasser auf 4 L. Aliquot heraus in 2 x 1,88 L und 0,24 L Autoklav.
Tris-Base, 2 M: Bereiten 60,57 g Tris-Base und Wasser auf 250 ml. Sterilisieren, Lagerung bei RT für die spätere Verwendung.
DVB-T, 1 M: Bereiten 2,31 g DTT und Wasser auf 15 ml. Filter zu sterilisieren (0,22 uM), aliquote 1-ml-Röhrchen bei -20 ° C für die spätere Verwendung.
S30A Puffer: Bereiten 10,88 g Mg-Glutamat und 24.39 g K-Glutamat, 50 ml Tris bei 2M, Essigsäure (pH-Wert 7,7) und Wasser auf 2 L Autoklav, Lagerung bei 4 ° C, 4 ml 1 M DTT vor der Verwendung.
S30B Puffer: Bereiten 10,88 g Mg-Glutamat und 24.39 g K-Glutamat, Tris bei 2 M (pH 8,2) und Wasser auf 2 L Autoklav, Lagerung bei 4 ° C, 2 ml 1 M DTT vor dem Gebrauch.
HEPES: Vorbereiten 1,91 g HEPES (MW 238,21), KOH (pH 8), und 4 ml Wasser zu.
tRNA: Vorbereitung 30 mg tRNA und Wasser zu 600 ul.
CoA: Bereiten 30 mg CoA (MW 767,53) und Wasser zu 600 ul.
NAD: Fügen 34.83 mg NAD (MW 663,43), Tris bei 2 M (pH 7,5-8) und Wasser auf 300 ul. (Add 27 ul Tris bei 2 M, um die Lösung auf pH 7.5-8 mitbringen).
cAMP hinzu: 42.80 mg cAMP (MW 329,22), Tris bei 2 M (pH 8) und Wasser auf 200 ul. (In 73 ul 2 M Tris bei, um die Lösung auf pH 8 zu bringen).
Folinsäure (33,9 mm): Zu 20 mg Fest Folinsäure Calciumsalz (MW 511,5), fügen 1,15 ml Wasser.
Spermidin: Bereiten 23,55 ul Spermidin (MW 145,25) und Wasser zu 150 ul. Vorbereitung bei Raumtemperatur nach kurzem Schmelzen bei 37 ° C
3-PGA: In 1,03 g 3-PGA (MW 230.02), bei 2 M Tris (pH 7,5) und 3,2 ml Wasser zu. (In 1,73 ml an 2 M Tris, die Lösung auf pH 7,5 zu bringen).
Nucleotide Mix: In 145 mg ATP Dikaliumsalzes Dihydrat (MW 619,4), 133 mg GTP Dinatriumsalz (MW 567,14), 79,4 mg CTP Dinatriumsalzdihydrat (MW 563,16), 82,6 mg UTP Trinatriumsalzes Dihydrat (MW 586,12) KOH in 15% iger Verdünnung (pH 7,5) und 1,5 ml Wasser zu. (In 353 &mgr; l KOH bei 15% Verdünnung, um den pH auf 7,5 zu bringen).
STADT Material 2. Bradford-Assay.
- Bradford entfernen Mittel von 4 ° C und bei Raumtemperatur einstellen.
- Vorbereitung 50 ul BSA-Standard in 1 mg / ml und 0,1 mg / ml.
- Bereiten Sie 40 ul 20x Verdünnung von Extrakt aus Schritt 1.47.
- In 800 ul Wasser bis 7 Küvetten.
- Vorbereitung Standardküvetten für 0 mg / ml, 1 mg / ml (10 &mgr; l 0,1 mg / ml BSA), 2 mg / ml (20 &mgr; l 0,1 mg / ml BSA), 4 mg / ml (4 &mgr; l 1 mg / ml BSA) , 6 mg / ml (6 ul 1 mg / ml BSA).
- Vorbereitung experimentellen Küvetten 2 ul Probe und 4 ul Probe.
- In 200 ul von Bradford Mittel zu jeder Küvette und gut mischen by Pipettieren. Inkubieren bei Raumtemperatur für mindestens 10 min.
- Produzieren Standardkurve bei OD 595 nm mit Küvetten aus Schritt 6.5. Ablehnen Standardkurve, wenn r 2 <0,95.
- Bestimmen Extrakt-Konzentration bei OD 595 nm mit Küvetten aus Schritt 6.6.
STADT Werkstoff 3. Puffer Kalibrierung Tabelle.
Siehe TXTL_e (Vorlage) _calibration_JoVE.xlsx .
Zusatzmaterial 4. Zellfreie Ausdruck laufen Tabellenkalkulation.
Diskussion
Die endogene Escherichia coli basierten hier beschriebenen TX-TL zellfreien Expressionssystem ist ein einfach-to-run drei U-Reaktion, die weniger als acht Stunden von bis zu Sammeln von Daten erfolgen kann. Der Prozess der Erstellung alle Reagenzien erfordert fünf Tage Zeit insgesamt (mit erheblichen Arbeitsaufwand nur an einem Tag), produziert aber Rohextrakt für 3000 Reaktionen und Pufferherstellung Reagenzien für 10.000 Reaktionen (Abbildung 1). Außerdem Rohextrakt und Pufferherstellung Reagenzien sind bei -80 ° C für mindestens 1 Jahr stabil, so dass für Mehrfachnutzung einer Zubereitung 4 Bei $ 0,11 pro 10 ul-Reaktion ($ 0,26 einschließlich Arbeits). Sind die Kosten 98% niedriger als bei vergleichbaren Kommerzielle Systeme (Abbildung 2).
Es gibt einige ungelöste Einschränkungen, jedoch zu dem System. Das Ende Effizienz jedes Zellrohextrakt Vorbereitung kann auf der Basis der Anwenderkenntnisse und den Umgebungsbedingungen variieren, obwohl typische Ertragsabweichung zwischen 5-10% (Abbildung 4b). Als Ergebnis sind Batch-zu-Batch-Variabilität im Endpunkt-Expression und der Expression Dynamik zu erwarten. Diese Schwankungen werden wahrscheinlich bleiben, bis Extrakt wird vollständig charakterisiert oder bis Extrakt Schöpfung ist voll automatisiert. Wenn der zellfreien Expressionssystem wird verwendet, um empfindliche quantitative Experimente durchzuführen, ist es ratsam, alle Experimente mit der gleichen Charge von Zellrohextrakt laufen. Die Ausbeute aus einer Zellrohextrakt Charge, etwa 3000 Reaktionen, sollte für typische experimentelle Kurse sein. Obwohl wir vermuten Variation kann durch die Skalierung und die Automatisierung der Verfahren entfernt werden, würden solche Versuche eine wesentliche Ressource Investitionen.
Zusätzlich, obwohl Endpunkt Expressionsspiegel sind relativ einfach zu bestimmen, muss mehr Arbeit zu verstehen Dynamik intrinsische zum zellfreien System durchgeführt werden. Es ist bekannt, dass sowohl Ressourcen-WETTBEWERBn-und Ressourcenbegrenzung kann Ausdruck Dynamik beeinflussen. Zum Beispiel können begrenzte endogenen Sigma-70 in einem Sättigungsregime mit erhöhter DNA-Matrize Herstellung eines Expressionsprofil analog zu der Nukleotid-oder Aminosäureabbau führen. 9,27 jedoch Dynamik müssen nicht vollständig verstanden, das System zu nutzen. Für reine Ertragssteigerungen kann die Optimierung von Maschine-Learning-Ansätze durchgeführt werden. 28. Fragen der Ressourcen Wettbewerb und Einschränkung kann durch mathematische Modelle angesprochen werden mit experimentellen Daten verifiziert.
Die hier vorgestellten Protokoll für ein BL21-Stamm Rosetta2 optimiert, aber ist verallgemeinerbar zu anderen E. coli-Stämme. Änderungen in BL21-Rosetta2, wie die Entfernung des Gens lon-Protease und die Zugabe von Genen seltenen tRNAs kodieren, ermöglichen maximale Proteinproduktion. BL21 und BL21 nur trxA Knockout-an - Wir haben das Protokoll mit zwei anderen Stämmen versucht Extraktd gefunden 50% weniger Proteinausbeute. Wir vermuten, dass die Renditen ähnlich, wenn andere Stämme zu verringern. Weitere Änderungen in den Parametern, wie z. B. Schalt 2xYT Wachstumsmedium für LB und andere reiche Brühen, haben zu einer verminderten Proteinausbeute führte.
Zellfreie Expression Systeme, die sowohl endogene als auch exogene Transkriptions-Translations-Maschinen und Regulationsmechanismen haben breite Anwendungen in der Protein-und Metabolit-Expression und in der synthetischen Biologie. 3,29 Anstatt auf T7-regulierten Stromkreise beschränkt, kann man sich vorstellen Herstellung von komplexen Biomolekülen in einer vom Benutzer steuerbare Einstellung mit einer Mischung aus nativen E. coli-Promotoren und exogen zugeführtem Transkriptions-und Regulationsmechanismen. Ohne Einschränkungen der Zellteilung und den Stoffwechsel, können Schwankungen in der synthetischen Schaltungen wie der repressilator oder in Stoffwechselwege entwickelt, wie solche Herstellung von Artemisinin reduziert oder besser verstanden. 30,31 Wir have verwendet, diese Vorteile zu genetischen Schalter zu implementieren, sowie Sigma-Faktor-Sequestrierung versteht 9,32 Diese Technologie kann auch das Rückgrat der "minimal" oder "künstliche" Zellen bilden -. kleinen, gut gekennzeichnet und autark verkörperten Einheiten Extrakt. 33,34
Letztendlich erwarten wir sofort Nutzungen dieser endogenen zellfreie Expressionssystem als Prototyping-Umgebung für synthetische Biologie. Den Spitznamen "TX-TL biomolekularen Steckbrett", das zellfreie Expressionssystem bietet eine Umgebung, in der steuerbaren Kunstkreise letztlich für die in vivo-Expression kann bestimmt Runden Prototyping unterziehen - Testzyklen auf Basisplasmid, linear, oder chemisch synthetisierte DNA, gefolgt durch Analyse und schnelle Änderung. Prototyping Runden kann durch mathematische Vorhersagemodelle entwickelt derzeit unterstützt werden. Durch das Entfernen von Klonen und in vivo-Manipulation für nicht-finale Kreise erwarten wir Ingenieuretechnik Zykluszeiten auf 1-3 Tage statt der aktuellen Wochen-Standard reduziert werden.
Offenlegungen
Die Autoren erklären, dass sie keine finanziellen Interessen konkurrieren.
Danksagungen
Wir danken Jongmin Kim, Dan Siegal-Gaskins, Anu Thubagere und Enoch Yeung für die Unterstützung Straffung des Protokolls und Clare Chen und Lee Barclay für die Unterstützung in den frühen Phasen des Projekts. Dieses Material wird auf der Arbeit zum Teil von der Defense Advanced Research Projects Agency (DARPA / MTO) unterstützt Gießereien Wohnprogramm, Vertragsnummer HR0011-12-C-0065 (DARPA / CMO.ZZS wird auch durch ein UCLA / Caltech Medical Scientist unterstützt auf Basis Training Program Kameradschaft und durch eine DoD, Air Force Office of Scientific Research, National Defense Science and Engineering Graduate (NDSEG) Fellowship, 32 CFR 168a. Die in diesem Dokument enthaltenen Ansichten und Rückschlüsse sind die der Autoren und sollte nicht als Darstellung interpretiert werden offiziell Politik, weder ausdrücklich noch stillschweigend, von der Defense Advanced Research Projects Agency oder der US-Regierung.
Materialien
| Name | Company | Catalog Number | Comments |
| 2xYT | MP biomedicals | 3012-032 | |
| 3-PGA | Sigma-Aldrich | P8877 | |
| ATP | Sigma-Aldrich | A8937 | |
| Bacto-agar | BD Diagnostics | 214010 | |
| Bead-beating tubes (polypropylene microvials) | BioSpec | 522S | |
| Beads, 0.1mm dia. | BioSpec | 11079101 | |
| BL21 Rosetta 2 E. coli strain | Novagen | 71402 | |
| Bradford BSA Protein Assay Kit | Bio-rad | 500-0201 | |
| cAMP | Sigma-Aldrich | A9501 | |
| Chloramphenicol | Sigma-Aldrich | C1919 | |
| CoA | Sigma-Aldrich | C4282 | |
| CTP | USB | 14121 | |
| Cuvettes, 1.5ml | Fisher | 14-955-127 | |
| DTT | Sigma-Aldrich | D0632 | |
| Folinic acid | Sigma-Aldrich | F7878 | |
| GTP | USB | 16800 | |
| HEPES | Sigma-Aldrich | H6147 | |
| K-glutamate | Sigma-Aldrich | G1149 | |
| Mg-glutamate | Sigma-Aldrich | 49605 | |
| Micro Bio-Spin Chromatography Columns | Bio-Rad | 732-6204 | |
| NAD | Sigma-Aldrich | N6522 | |
| Nunc 384-well optical bottom plates | Thermo-Scientific | 142761 | |
| Nunc sealing tape | Thermo-Scientific | 232701 | |
| PEG-8000 | Promega | V3011 | |
| Potassium phosphate dibasic solution | Sigma-Aldrich | P8584 | |
| Potassium phosphate monobasic solution | Sigma-Aldrich | P8709 | |
| RTS Amino Acid Sampler | 5 Prime | 2401530 | |
| Slide-A-Lyzer Dialysis Cassettes, 10k MWCO (Kit) | Thermo-Scientific | 66382 | |
| Spermidine | Sigma-Aldrich | 85558 | |
| Tris base | Fischer | BP1521 | |
| tRNA (from E. coli) | Roche Applied Science | MRE600 | |
| UTP | USB | 23160 | |
| 1L Centrifuge Bottle | Beckman-Coulter | A98813 | This is specific for Avanti J-series; obtain equivalent size for centrifuge in use. |
| 4L Erlenmeyer Flask | Kimble Chase | 26500-4000 | |
| Avanti J-26XP Centrifuge | Beckman-Coulter | 393127 | Or 1L-capable centrifuge equivalent. |
| Forma 480 Orbital Shaker | Thermo Scientific | 480 | Or chest-size 6x4L shaker equivalent. |
| JLA-8.1000 Rotor | Beckman-Coulter | 363688 | Or 1L-capable, 5000 x g rotor equivalent for centrifuge. |
| Mini-Beadbeater-1 | BioSpec | 3110BX | |
| Supplemental Material 1. Recipes for Items. Chloramphenicol, 34 mg/ml: Prepare 0.51 g chloramphenicol and add ethanol to 15 ml. Filter sterilize (0.22 μM), aliquot to 1 ml tubes, store at -20 °C for later use. 2xYT+P+Cm agar plate: Prepare 1.24 g 2xYT, 1.6 ml potassium phosphate dibasic solution @ 1 M, 0.88 ml potassium phosphate monobasic solution @ 1 M, 0.6 g agar, and water to 40 ml. Autoclave. Let cool to 50 °C and add 40 μl Cm. Aliquot 25 ml into a 100x15 mm petri dish, and let cool for an hour. 2xYT+P media: Prepare 124 g 2xYT, 160 ml potassium phosphate dibasic solution @1 M, 88 ml potassium phosphate monobasic solution @ 1 M, and water to 4 L. Aliquot out into 2x1.88 L and 0.24 L. Autoclave. Tris base, 2 M: Prepare 60.57 g Tris base and water to 250 ml. Sterilize, store at RT for later use. DTT, 1 M: Prepare 2.31 g DTT and water to 15 ml. Filter sterilize (0.22 μM), aliquot to 1 ml tubes, store at -20 °C for later use. S30A buffer: Prepare 10.88 g Mg-glutamate and 24.39 g K-glutamate, 50 ml Tris at 2M, acetic acid (to pH 7.7), and water to 2 L. Autoclave, store at 4 °C, add 4 ml 1 M DTT before use. S30B buffer: Prepare 10.88 g Mg-glutamate and 24.39 g K-glutamate, Tris at 2 M (to pH 8.2), and water to 2 L. Autoclave, store at 4 °C, add 2 ml 1 M DTT before use. HEPES: Prepare 1.91 g HEPES (MW 238.21), KOH (to pH 8), and water to 4 ml. tRNA: Prepare 30 mg of tRNA and water to 600 μl. CoA: Prepare 30 mg of CoA (MW 767.53) and water to 600 μl. NAD: Add 34.83 mg of NAD (MW 663.43), Tris at 2 M (to pH 7.5-8), and water to 300 μl. (Add 27 μl of Tris at 2 M to bring the solution to pH 7.5-8). cAMP: Add 42.80 mg of cAMP (MW 329.22), Tris at 2 M (to pH 8), and water to 200 μl. (Add 73 μl of Tris at 2 M to bring the solution to pH 8). Folinic Acid (33.9 mM): To 20 mg of solid folinic acid calcium salt (MW 511.5), add 1.15 ml water. Spermidine: Prepare 23.55 μl of spermidine (MW 145.25) and water to 150 μl. Prepare at room temperature after melting briefly at 37 °C. 3-PGA: Add 1.03 g of 3-PGA (MW 230.02), Tris at 2 M (to pH 7.5), and water to 3.2 ml. (Add 1.73 ml of Tris at 2 M to bring the solution to pH 7.5). Nucleotide Mix: Add 145 mg of ATP dipotassium salt dihydrate (MW 619.4), 133 mg of GTP disodium salt (MW 567.14), 79.4 mg of CTP disodium salt dihydrate (MW 563.16), 82.6 mg of UTP trisodium salt dihydrate (MW 586.12), KOH at 15% dilution (to pH 7.5), and water to 1.5 ml. (Add 353 μl of KOH at 15% dilution to bring the solution to pH 7.5). Supplemental Material 2. Bradford Assay.
See TXTL_e(template)_calibration_JoVE.xlsx. Supplemental Material 4. Cell-free expression run spreadsheet. See TXTL _JoVE.xlsx. | |||
Referenzen
- Noireaux, V., Bar-Ziv, R., Libchaber, A. Principles of cell-free genetic circuit assembly. Proceedings of the National Academy of Sciences of the United States of America. 100, 12672-12677 (2003).
- He, M. Y., He, Y. Z., Luo, Q., Wang, M. R. From DNA to protein: No living cells required. Process Biochem. 46, 615-620 (2011).
- Forster, A. C., Church, G. M. Synthetic biology projects in vitro. Genome Res. 17, 1-6 (1101).
- Shin, J., Noireaux, V. Efficient cell-free expression with the endogenous E. Coli RNA polymerase and sigma factor 70. Journal of biological engineering. 4, 8 (2010).
- Shimizu, Y., et al. Cell-free translation reconstituted with purified components. Nat Biotechnol. 19, 751-755 (2001).
- Shin, J., Noireaux, V. An E. coli Cell-Free Expression Toolbox: Application to Synthetic Gene Circuits and Artificial Cells. Acs Synth Biol. 1, 29-41 (2012).
- Shin, J., Noireaux, V. Study of messenger RNA inactivation and protein degradation in an Escherichia coli cell-free expression system. Journal of biological engineering. 4, 9 (2010).
- Shin, J., Jardine, P., Noireaux, V. Genome Replication, Synthesis, and Assembly of the Bacteriophage T7 in a Single Cell-Free Reaction. Acs Synth Biol. 1, 408-413 (2012).
- Siegal-Gaskins, D., Noireaux, V., Murray, R. M., Pao, L., Abramovitch, D. Biomolecular resource utilization in elementary cell-free gene circuits. , 1531-1536 (2013).
- Karzbrun, E., Shin, J., Bar-Ziv, R. H., Noireaux, V. Coarse-grained dynamics of protein synthesis in a cell-free system. Physical review letters. 106, 048104 (2011).
- Hoagland, M. B., Stephenson, M. L., Scott, J. F., Hecht, L. I., Zamecnik, P. C. Soluble Ribonucleic Acid Intermediate in Protein Synthesis. J Biol Chem. 231, 241-257 (1958).
- Wood, W. B., Berg, P. Effect of Enzymatically Synthesized Ribonucleic Acid on Amino Acid Incorporation by a Soluble Protein-Ribosome System from Escherichia Coli. Proceedings of the National Academy of Sciences of the United States of America. 48, 94 (1962).
- Zubay, G. In-Vitro Synthesis of Protein in Microbial Systems. Annu Rev Genet. 7, 267-287 (1973).
- Pratt, J. M., Hames, B. D., Higgins, S. J. . Transcription and Translation: A Practical Approach. , 179-209 (1984).
- Kim, H. C., Kim, D. M. Methods for energizing cell-free protein synthesis. Journal of bioscience and bioengineering. 108, 1-4 (2009).
- Michel-Reydellet, N., Calhoun, K., Swartz, J. Amino acid stabilization for cell-free protein synthesis by modification of the Escherichia coli genome. Metabolic engineering. 6, 197-203 (2004).
- Liu, D. V., Zawada, J. F., Swartz, J. R. Streamlining Escherichia coli S30 extract preparation for economical cell-free protein synthesis. Biotechnology progress. 21, 460-465 (2005).
- Andrianantoandro, E., Basu, S., Karig, D. K., Weiss, R. Synthetic biology: new engineering rules for an emerging discipline. Molecular systems biology. 2, 2006.0028 (2006).
- Kwok, R. Five hard truths for synthetic biology. Nature. 463, 288-290 (2010).
- Tabor, S., Richardson, C. C. A bacteriophage T7 RNA polymerase/promoter system for controlled exclusive expression of specific genes. Proceedings of the National Academy of Sciences of the United States of America. 82, 1074-1078 (1985).
- Lewicki, B. T., Margus, T., Remme, J., Nierhaus, K. H. Coupling of rRNA transcription and ribosomal assembly in vivo. Formation of active ribosomal subunits in Escherichia coli requires transcription of rRNA genes by host RNA polymerase which cannot be replaced by bacteriophage T7 RNA polymerase. Journal of molecular biology. 231, 581-593 (1993).
- Iskakova, M. B., Szaflarski, W., Dreyfus, M., Remme, J., Nierhaus, K. H. Troubleshooting coupled in vitro transcription-translation system derived from Escherichia coli cells: synthesis of high-yield fully active proteins. Nucleic acids research. 34, e135 (2006).
- Kigawa, T., et al. Preparation of Escherichia coli cell extract for highly productive cell-free protein expression. Journal of structural and. 5, 63-68 (2004).
- Matsuda, T., et al. Improving cell-free protein synthesis for stable-isotope labeling. Journal of biomolecular. NMR. 37, 225-229 (2007).
- Sitaraman, K., et al. A novel cell-free protein synthesis system. Journal of biotechnology. 110, 257-263 (2004).
- Becskei, A., Serrano, L. Engineering stability in gene networks by autoregulation. Nature. 405, 590-593 (2000).
- Maeda, H., Fujita, N., Ishihama, A. Competition among seven Escherichia coli sigma subunits: relative binding affinities to the core RNA polymerase. Nucleic acids research. 28, 3497-3503 (2000).
- Caschera, F., et al. Coping with complexity: machine learning optimization of cell-free protein synthesis. Biotechnology and bioengineering. 108, 2218-2228 (2011).
- Hodgman, C. E., Jewett, M. C. Cell-free synthetic biology: thinking outside the cell. Metabolic engineering. 14, 261-269 (2012).
- Elowitz, M. B., Leibler, S. A synthetic oscillatory network of transcriptional regulators. Nature. 403, 335-338 (2000).
- Tsuruta, H., et al. High-level production of amorpha-4,11-diene, a precursor of the antimalarial agent artemisinin, in Escherichia coli. Plos One. 4, e4489 (2009).
- Gardner, T. S., Cantor, C. R., Collins, J. J. Construction of a genetic toggle switch in Escherichia coli. Nature. 403, 339-342 (2000).
- Jewett, M. C., Forster, A. C. Update on designing and building minimal cells. Current opinion in biotechnology. 21, 697-703 (2010).
- Noireaux, V., Libchaber, A. A vesicle bioreactor as a step toward an artificial cell assembly. Proceedings of the National Academy of Sciences of the United States of America. 101, 17669-17674 (2004).
Erratum
Formal Correction: Erratum: Protocols for Implementing an Escherichia coli Based TX-TL Cell-Free Expression System for Synthetic Biology
Posted by JoVE Editors on 1/01/1970. Citeable Link.
A correction to Figure 5's legend has been made for the article Protocols for Implementing an Escherichia coli Based TX-TL Cell-Free Expression System for Synthetic Biology. Method 1 and method 2 have been switched.Z
The figure legend was update from:
Figure 5. Effects of DNA solution on expression efficiency. a) Comparison of two different purification methods for processing plasmids. 1 nM of pBEST-OR2-OR1-Pr-UTR1-deGFP-T500 is prepared using only a QiaPrep Spin Miniprep Kit (Purification method 1) or post-processed with a QiaQuick PCR purification kit (Purification method 2).
to
Figure 5. Effects of DNA solution on expression efficiency. a) Comparison of two different purification methods for processing plasmids. 1 nM of pBEST-OR2-OR1-Pr-UTR1-deGFP-T500 is prepared using only a QiaPrep Spin Miniprep Kit (Purification method 2) or post-processed with a QiaQuick PCR purification kit (Purification method 1).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten