Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Preparación y análisis de
En este artículo
Resumen
We demonstrate a method to generate 3D breast cancer surrogates, which can be cultured using a perfusion bioreactor system to deliver oxygen and nutrients. Following growth, surrogates are fixed and processed to paraffin for evaluation of parameters of interest. The evaluation of one such parameter, cell density, is explained.
Resumen
En tres dimensiones (3D) la cultura es un método más fisiológicamente relevantes para modelar el comportamiento celular in vitro de dos dimensiones la cultura. Los carcinomas, incluyendo los carcinomas de mama, son tejidos 3D complejas compuestas de células epiteliales de cáncer y los componentes del estroma, incluyendo fibroblastos y matriz extracelular (ECM). Sin embargo, la mayoría de los modelos in vitro de carcinoma de mama consisten solamente de las células epiteliales cancerosas, omitiendo el estroma y, por lo tanto, la arquitectura 3D de un tumor in vivo. modelado 3D apropiada del carcinoma es importante para la comprensión exacta de la biología del tumor, el comportamiento y la respuesta al tratamiento. Sin embargo, la duración de la cultura y el volumen de los modelos 3D está limitada por la disponibilidad de oxígeno y nutrientes en el cultivo. En este documento, se demuestra un método en el que las células de carcinoma de mama epiteliales y los fibroblastos del estroma se incorporan en ECM para generar un sustituto cáncer de mama en 3D que incluye estroma y se pueden cultivar como unaestructura 3D sólida o mediante el uso de un sistema de biorreactor de perfusión para entregar oxígeno y nutrientes. Después de la instalación y un período de crecimiento inicial, sustitutos pueden ser utilizados para las pruebas de drogas preclínica. Alternativamente, los componentes celulares y de la matriz de la madre sustituta pueden ser modificadas para abordar una variedad de cuestiones biológicas. Después del cultivo, sustitutos se fijaron y procesaron para parafina, de una manera similar a la manipulación de las muestras de carcinoma de mama clínica, para la evaluación de los parámetros de interés. La evaluación de uno de estos parámetros, la densidad de células presentes, se explica, en donde sistemas de software de análisis de imagen ImageJ y CellProfiler se aplican a fotomicrografías de secciones histológicas de sustitutos para cuantificar el número de células nucleadas por área. Esto se puede utilizar como un indicador del cambio en el número de células en el tiempo o el cambio en el número de células como resultado de diferentes condiciones de crecimiento y tratamientos.
Introducción
Tres modelos tridimensionales (3D) de cultivo que imitan con mayor precisión la arquitectura tumor y microentorno in vivo son importantes para los estudios dirigidos a diseccionar las complejas interacciones entre las células y su microambiente y para probar la eficacia de las terapias candidatos. Tumor impactos dimensionalidad de oxígeno y los gradientes de nutrientes, la uniformidad de la exposición al fármaco, el flujo / presión arterial intersticial, y la arquitectura 3D 1-4. La presencia de un microambiente estromal apropiado contribuye a la dimensionalidad tumor y las influencias señalización celular-ECM y la señalización paracrina entre las células del estroma y las células epiteliales malignas. Los efectos de la dimensionalidad del tumor y el microambiente de la función celular están bien establecidos, con los dos factores que alteran la respuesta a los fármacos 1,3,5-8. Además, la cinética de crecimiento celular, las tasas metabólicas y de señalización celular difieren entre las dos culturas dimensiones (2D) y la cultura en 3D, con estos factores affenexión respuesta celular 1,3,8-10.
In vitro, el microambiente sustituto tumor puede ser modulada mediante la inclusión de componentes de ECM representativos y las poblaciones de células del estroma. Células epiteliales malignos son influenciados por el ECM y las células del estroma asociadas con el cáncer, ya sea de una manera sinérgica / protección para promover la progresión tumoral o de una manera supresora para inhibir aún más la propagación 5,6,10 tumor. En cualquier contexto, el estroma puede afectar a la respuesta terapéutica y la administración de fármacos a través de la señalización paracrina y / o mediante el aumento de la presión intersticial en el tumor resulta en una disminución de suministro de fármaco de 1,6. Por lo tanto, la adición de ECM y las células del estroma en modelos preclínicos ayudará recapitular aspectos del tumor que no puede ser modelado bien en cultivo 2D.
En este documento un método para establecer sustitutos de cáncer de mama que incorporan un microambiente recapitulativo, incluyendo constituyentes y s ECMtromal células, en un volumen 3D se describe. En el carcinoma de mama, la población de células del estroma se predominantemente compuesta de cáncer asociado fibroblastos (CAF) y el ECM del estroma se compone principalmente de colágeno de tipo I con una menor proporción de componentes de la matriz que se encuentran en la membrana basal, incluyendo laminina y colágeno tipo IV 1,4,11-13. Por lo tanto, estos componentes del microambiente carcinoma de mama (es decir, CAF, colágeno I, y la membrana basal) se han incorporado en los sustitutos. Este método puede ser utilizado para generar sólidos, sustitutos 3D-un perfundido (Figura 1A) o puede ser adaptado para incluir la perfusión de medio a través de la madre sustituta a través de un sistema de biorreactor (Figura 1B). Ambos enfoques se describen aquí. Este método también podría ser modificado para incluir otros elementos del estroma, tales como macrófagos asociados al tumor, o para modelar otros tumores sólidos mediante el ajuste de los componentes celulares y de ECM, como sea apropiado.
Para el sustituto carcinoma de mama describe aquí, hemos utilizado el MDA-MB-231 (231) línea celular de cáncer de mama, la CAF previamente aislado de carcinoma de mama humano 14, y un ECM compuesto por 90% de colágeno I ( factor de crecimiento de material de 6 mg / ml) y 10% de reducción de la membrana basal (BM). El sustituto se cultiva bien en un 8 pocillos diapositiva cámara (sustituto sólido) o un sistema de biorreactor se utiliza para proporcionar la perfusión de nutrientes continua (sustituto perfundido). Cualquier sistema de biorreactor de perfusión que puede alojar un volumen de células que contiene ECM se puede utilizar 15. Como ejemplo, se describe la preparación de los sustitutos de tejido en nuestro sistema de biorreactor. Este sistema fue desarrollado internamente y no está disponible comercialmente. Debido a que nuestra atención se centra en la preparación y el análisis de los sustitutos de tejido 3D, no hemos entrado en una descripción más detallada con respecto a los detalles de fabricación y montaje de nuestro sistema de biorreactor. Sin embargo, una descripción detallada deeste sistema y su desarrollo ha sido publicado el 16. En este sistema de biorreactor, un canal de flujo de polidimetilsiloxano (PDMS) se utiliza para alojar el sustituto, que está soportado por una espuma de PDMS (formado usando métodos similares a los descritos por Calcagnile et al. 17). Este volumen es penetrado por 4 microcanales (cada uno de 400 m de diámetro) que son perfundidos continuamente por medio de una bomba a través de microphysiologic para suministrar oxígeno y nutrientes a la madre sustituta.análisis apropiado de los sustitutos es crucial para obtener la información pertinente a la función celular en respuesta al tratamiento u otras manipulaciones. Los sustitutos pueden ser analizadas por diversos métodos, incluyendo formación de imágenes directa de los sustitutos intactas mediante microscopía confocal o de otros medios de formación de imágenes no invasiva, análisis celular indirecto mediante el ensayo de los medios acondicionados, o perfundido, para los productos secretados, o análisis en cortes histológicos después de la fijación y el procesamiento aparafina. Uno de estos parámetros que pueden ser evaluados en los cortes histológicos es la densidad celular. Presentamos un método para medir la densidad de células (es decir, el número de células nucleadas por área de la sección), utilizando técnicas de procesamiento de imagen semi-automatizados aplicados a fotomicrografías de secciones histológicas de sustitución teñidas con hematoxilina y eosina (H & E). La densidad celular se puede utilizar como un indicador del cambio relativo en el número de células en el tiempo o que resulta de diferentes condiciones de crecimiento y tratamientos.
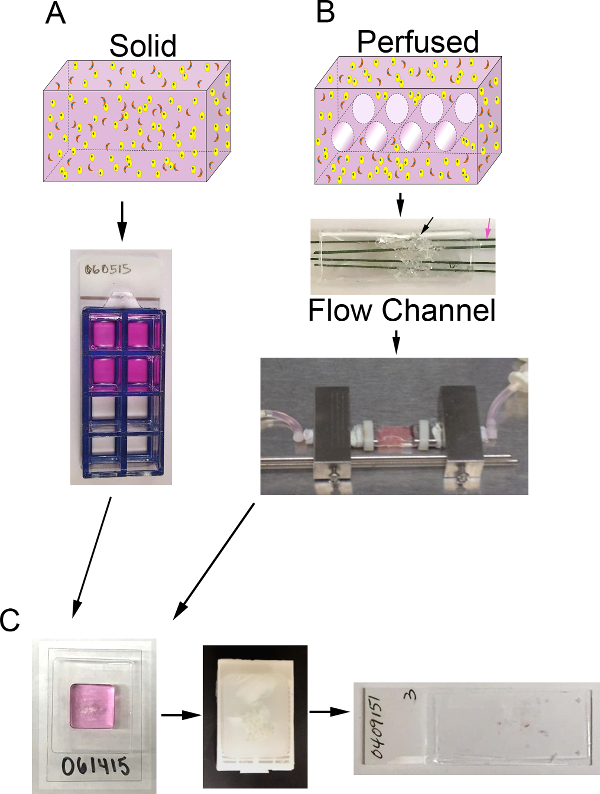
Figura 1. volumen 3D y el sistema de biorreactor. A) Representación esquemática del proceso para generar sustitutos 3D sólidos. Arriba: dibujos animados de volumen 3D sólido que contiene ECM (rosa), células de carcinoma epitelial (amarillo), y la CAF (naranja); En pocas palabras:. Vista desde arriba de sustitutos de 8 pocillos de diapositivas cámara que contiene B) Esquema del proceso para generar una perfusión sustitutos 3D. Arriba: cartoon de volumen 3D con canales para permitir la perfusión del medio y que contiene ECM (rosa), carcinoma de células epiteliales (amarillo), y la CAF (naranja); Medio: imagen del canal de flujo de PDMS que contiene PDMS espuma (flecha negro) para ser inyectado con células + ECM y penetrado por alambres de acero inoxidable recubiertos de polímero (flecha rosa) que miden 400 micras de diámetro; Inferior:. Imagen del canal de flujo PDMS que contiene un sustituto y conectado al sistema de biorreactor para permitir la perfusión del medio continuo (bomba peristáltica y depósito de medio que no se muestra) C) Imágenes de pasos de procesamiento para ambos sustitutos sólidos y perfundidos después del cultivo. A la izquierda: imagen de la criomolde que contiene la muestra de procesamiento de gel y sustituta; Medio: imagen de un bloque de parafina que contiene un sustituto fijo y procesados; Derecha:. Imagen de una placa de vidrio con un corte histológico teñido con HE y de un sustituto Haga clic aquí para ver unaversión más grande de esta figura.
Protocolo
1. Cultura de la célula
- Descongelar componente BM durante la noche a 4 ° C, en el hielo.
- medio caliente a 37 ° C. Para apoyar el crecimiento de ambas células 231 y CAF, utilice medio Eagle modificado de Dulbecco (DMEM) más suero bovino fetal al 10% (FBS).
Nota: Los medios utilizados dependerán del tipo de célula y los objetivos experimentales. - Retire medio de la placa de cultivo (10 cm) de células confluentes cerca de 231 y añadir 1,5 ml de tripsina / EDTA. Incubar durante 1 a 3 minutos a 37 ° C, el seguimiento para el desprendimiento de células. Una vez que las células comienzan a redondear y salir de la placa, detener la reacción mediante la adición de 3 ml del medio que contenía suero. Pipetear el medio y las células en un tubo cónico de 15 ml.
- Centrifugar el medio y las células en 150 xg durante 5 min. Eliminar el sobrenadante y volver a suspender el sedimento celular en 2 ml de medio.
- Contar el número de células por volumen utilizando azul tripán y un hemocitómetro. La viabilidad celular debe ser mayor que 90%.
- Repite elproceso con otros tipos de células que se incluirán en la madre sustituta. Para el sustituto se describe aquí, el proceso se repite con el CAF.
- Determinar el volumen apropiado de cada suspensión celular para obtener el número deseado de células.
Nota: Para obtener el sustituto se describe aquí, una densidad celular de 2,1 x 10 6 células por 100 l de ECM, con una proporción de 2: 1 de las células epiteliales a los fibroblastos (1,4 x 10 6 231 células y 7 x 10 5 CAF por 100 l ECM), se utiliza. Esta densidad celular es un buen punto de partida; sin embargo, la densidad celular óptima dependerá del tipo de célula, la duración de la cultura, y los objetivos del experimento. - Coloque el volumen apropiado de cada suspensión de células en un tubo cónico de 15 ml (un tubo para cada tipo de célula). Centrifugar el medio y las células en 150 xg durante 5 min.
- Después de la centrifugación, eliminar el sobrenadante, volver a suspender un tipo de célula en célulaagua de calidad de cultivo (178,8 l, véase la Tabla 1) y el otro tipo de célula en 10x DMEM (100 l, véase la Tabla 1, que contiene rojo de fenol para controlar el pH). Colocar los tubos que contienen las células en hielo y proceder rápidamente a la preparación de ECM a continuación. Limitar el tiempo que se mantienen las células en agua para preservar la viabilidad.
Nota: Los dos componentes se requieren 10x DMEM y agua de calidad para cultivo de células de la MEC; Por lo tanto, hemos decidido volver a suspender las células en ambos. Aquí se eligió arbitrariamente para volver a suspender las células en 231 agua de calidad de cultivo celular y CAF en 10x DMEM, aunque cualquiera de los tipos de células podría ser re-suspendió en cualquiera de estos dos componentes.
2. Preparación de las células en ECM (6 mg / ml de colágeno tipo I bovino + 10% BM)
Nota: Un ECM compuesta de colágeno 90% I + 10% BM fue elegido para modelo de carcinoma de mama invasivo debido a que el estroma tumoral en esta malignidad se compone principalmente de colágenoI con los componentes de la BM, tales como laminina, colágeno IV, y entactina, que comprende una porción más pequeña de la ECM 12,13,18,19.
- En el hielo, agregar componentes enumerados en la Tabla 1, en orden, en un tubo de microcentrífuga de 2 ml.
Nota: Esta cantidad es suficiente para 8 sustitutos sólidos o, usando el sistema de biorreactor descrito aquí, 4 sustitutos perfundidos.
| Preparación de células en ECM (6 mg / ml de colágeno bovino de tipo I + 10% BM) | |
| 178,8 μl | agua de calidad de cultivo celular que contiene el número deseado de 231 células (determinada anteriormente) |
| 606 l | El colágeno I (10 mg / ml bovina), añadir gota a gota |
| 100 l | membrana basal, descongelado |
| 100 l | 10x DMEM (que contiene rojo de fenol) con el número deseado de CAF (determinada anteriormente) |
| 15,2 l | 7,5% (v / v) Bicarbonato de sodio, añadir gota a gota |
Tabla 1. Preparación de células en ECM.
- Mezclar suavemente con la pipeta, evitando la formación de burbujas. Monitorear el nivel de pH usando el rojo de fenol en el 10x DMEM. Compruebe que la mezcla es una naranja / color rosado indica un pH de ~ 7. Si el pH es demasiado bajo (demasiado amarillo), añadir lentamente adicional 7.5% de bicarbonato de sodio una gota a la vez (~ 5 l) hasta que se alcanza el color apropiado.
- Mantener la mezcla en hielo y trabajar con rapidez para evitar la polimerización prematura ECM.
3. Preparación Sustituto
- Para los cultivos 3D sólidos (Figura 1A):
- Trabajando en una campana de cultivo de células utilizando una técnica estéril, la etiqueta de la tapa de un portaobjetos de cámara estéril 8 pocillos para indicar cualquier variación experimental en sustitutos.
- Mantener la diapositiva cámara en hielo para evitar la polimerización prematura ECM, lentamente pipeta 100 l de la mezcla de células + ECM en cada pocillo de la diapositiva cámara de 8 pocillos.
Nota: Medir con una pipeta la mezcla de células + ECM alrededor de los bordes de la primera así ayuda a distribuir mejor la mezcla de células + ECM en el pozo. - Incubar sustitutos a 37 ° C, 5% de CO2 durante 45 min para permitir la polimerización ECM.
- Después de la polimerización ECM, añadir 100 l de medios de cultivo a cada pocillo y se incuba a 37 ° C, 5% de CO 2 durante la duración del experimento,el cambio de medio de cultivo cada dos días.
- Para los cultivos 3D perfundidos en un sistema de biorreactor (Figura 1B):
- Esterilizar todos los componentes de biorreactores para la configuración de la cultura 3D (es decir, biorreactores, tubos, pinzas, y los accesorios necesarios para la configuración de biorreactor) utilizando el proceso específico para el biorreactor a utilizar.
- Para el sistema de biorreactor ejemplo utilizado aquí, utilizar una combinación de tratamiento en autoclave (por ejemplo, 12 min de exposición a 121,1 ° C con 15 min de secado) y la incubación en 70% de etanol durante 1 hr.
- Preparar y montar la parte del sistema de biorreactor que albergará la madre sustituta.
- Para el sistema de biorreactor ejemplo utilizado aquí, inserte una columna vertebral de espuma PDMS en el canal de flujo de PDMS tubo utilizando fórceps. Empuje cuatro (400 mM) alambres de acero inoxidable recubiertos de polímero en la espuma PDMS para generar microcanales paralelos.
- En una campana de cultivo de células, utilizando sterile técnica y una aguja de calibre 26 con la jeringuilla, inyectar la mezcla de células + ECM en el área de la biorreactor de perfusión diseñado para contener células. Proceder rápidamente con el paso siguiente.
- Para asegurar una distribución más uniforme de las células dentro de los sustitutos, colocar el componente biorreactor que aloja los sustitutos en un tubo cónico de 50 ml (bajo la campana de cultivo de células) y girar de forma continua en ~ 18 rpm mientras que la incubación a 37 ° C durante 45 minutos para permitir la ECM polimerización.
Nota: La rotación puede llevarse a cabo usando un rotador en la incubadora o un horno con un sistema incorporado en los rotadores, tal como un horno de hibridación a 37 ° C. - Conectar el conjunto biorreactor que contiene el sustituto de la bomba de perfusión utilizando las instrucciones del fabricante.
Nota: Los detalles de este proceso variará dependiendo del biorreactor y de la bomba que se utiliza.- Para el sistema de biorreactor ejemplo utilizado aquí, quitar los alambres de acero inoxidable antes de conectar el bioreamontaje ctor a la bomba.
- Iniciar la perfusión del medio (caudal mayor de 167,1 l / min; microcanal tensión de cizallamiento de 1 dina / cm 2) en una incubadora a 37 ° C, 5% de CO 2.
Nota: La tasa de perfusión del medio se puede ajustar, dependiendo de la configuración del biorreactor y de los objetivos y el diseño del experimento. - Continuar perfusión del medio durante la duración del experimento, el cambio de medio de cultivo cada siete días.
- Esterilizar todos los componentes de biorreactores para la configuración de la cultura 3D (es decir, biorreactores, tubos, pinzas, y los accesorios necesarios para la configuración de biorreactor) utilizando el proceso específico para el biorreactor a utilizar.
4. Sustituto de fijación y procesamiento (Figura 1C)
- criomoldes de etiquetas y cintas de tejido de plástico para la fijación y el procesamiento sustituta.
- A continuación, sustitutos EnCase en gel de procesamiento de muestras, que es un material acuoso que es líquido a temperaturas cálidas, pero solidifica a temperatura ambiente. Las asistencias de gel de procesamiento de muestras en mantener intactos los sustitutos durante el procesamiento y facilita el corte histológico 14,20-22.
- Derretir la muestra de procesamiento de gel en un baño de agua a 60 ° C para licuarlo, manteniendo a esta temperatura hasta que esté listo para su uso. Mueva el biorreactor con la madre sustituta a una cabina de bioseguridad.
- Pipeta aproximadamente 300 l espécimen gel de tratamiento en la parte inferior de la criomolde marcado (Figura 1C, panel izquierdo).
- El uso de una hoja de bisturí (Nº 10 preferido) y las pinzas retire con cuidado el sustituto del biorreactor o desde el pozo de un porta con cámara de 8 pocillos y lo coloca en el criomolde que contiene la muestra de procesamiento de gel.
Nota: tintes de marcación de tejido (véase Materiales / lista de los equipos para un ejemplo específico) de diferentes colores se puede utilizar para marcar sustitutos, lo que permite múltiples muestras que deben incluirse en un casete de tejido de una manera distinguible. - Pipeta aproximadamente 300 l muestra el procesamiento de gel para cubrir el sustituto en el criomolde y se incuba a 4 ° C durante 30 min a solidify.
- Una vez que el procesamiento de las muestras de gel se ha solidificado, retirar el gel de procesamiento de muestras que contiene el sustituto del criomolde, y colocarlo en un contenedor de tejido.
- Coloque el casete de tejido que contiene el sustituto en 10% de formalina tamponada neutra durante 10 a 12 horas a temperatura ambiente para permitir la fijación completa.
- Después de la fijación, mueva el casete de tejido que contiene el sustituto de etanol al 70% hasta que sea utilizado para la parafina.
Nota: La transferencia de la madre sustituta de formalina al etanol evita la sobre-fijación con formalina, que puede causar la pérdida de expresión de algunos epítopos 23. La longitud de tiempo en etanol no es crítica. Este cambio de fijador es importante si la madre sustituta será utilizado para inmunohistoquímica o inmunofluorescencia. El sustituto fijo está ahora listo para el procesamiento de parafina (Figura 1C, panel central). Este procesamiento se realiza típicamente en un procesador de tejido situado en un apropriately equipado laboratorio de histología. Un programa más corto, se recomienda debido al tamaño y naturaleza delicada de los sustitutos. 24
5. seccionamiento y tinción H & E (Figura 1C, panel derecho)
- Tras el procesamiento de los sustitutos a un bloque de parafina, la sección usando un microtomo estándar para el corte de, tejidos embebidos en parafina fijado en formol 24,25.
Nota: Esto puede llevarse a cabo en un laboratorio de histología cualificado, o en un laboratorio de investigación, si está debidamente equipado y con experiencia. El espesor de las secciones histológicas puede variar en función del uso previsto de las secciones; Sin embargo, por lo general utilizamos secciones que son 5 micras de espesor. La espuma PDMS utilizado aquí en los sustitutos perfundidos es fácilmente seccionada con un microtomo. - Coloque secciones sobre portaobjetos histológicos de vidrios planos.
- Después de seccionar, hornear las secciones histológicas a 58 ° C durante 10-12 horas para prepararse para la tincióncon hematoxilina y eosina (H & E). Hornear funde la parafina y también permite una mejor adherencia de las secciones de la lámina de vidrio.
- Tinción H & E:
- Configurar las estaciones descritas en la Tabla 2 en frascos de Coplin o platos de tinción de vidrio, dependiendo del número de diapositivas a manchar. Una vez que los reactivos se crean, mover las secciones a través de cada estación, con el fin, incubando durante el tiempo indicado por debajo de 24.
- Montar un cubreobjetos para cada diapositiva usando medio de montaje.
- Permita que el medio de montaje se sequen antes de exponer.
| H & E tinción | ||
| Estación | Solución | Hora |
| 1 | xileno | 5 minutos |
| 2 | xileno | 5 min |
| 3 | xileno | 5 minutos |
| 4 | 100% Etanol | 5 minutos |
| 5 | 100% Etanol | 5 minutos |
| 6 | 95% Etanol | 5 minutos |
| 7 | 95% Etanol | 5 minutos |
| 8 | Agua del grifo | 5 minutos |
| 9 | Agua desionizada | 5 minutos |
| 10 | hematoxilina 7211 | 5 minutos |
| 11 | Agua del grifo | 5 minutos |
| 12 | clarificador * | 10 inmersiones |
| * Richard Allan # 7401 o 70% de etanol + 0.5% de HCl | ||
| 13 | Agua del grifo | 5 minutos |
| 14 | Reactivo de azulado | 30 segundos |
| 15 | Agua del grifo | 5 minutos |
| dieciséis | 95% Etanol | 10 inmersiones |
| 17 | Eosina-Y | 1 minuto |
| 18 | 95% Etanol | 10 inmersiones |
| 19 | 95% Etanol | 10 inmersiones |
| 20 | 100% Etanol | 10 inmersiones |
| 21 | 100% Etanol | 10 inmersiones |
| 22 | 100% Etanol | 5 minutos |
| 23 | xileno | 10 inmersiones |
| 24 | Xileno | 5 minutos |
Tabla 2. tinción H & E.
6. Medición de la densidad celular
- Al menos una imagen de toda la sección histológica teñida con H & E de la madre sustituta usando microscopía de campo claro con un aumento de 400X, guardar las imágenes como archivos .tif.
Nota: El procesamiento de imágenes se describe sólo se ha completado utilizando imágenes en color. Aunque no probado, creemos que el mismo proceso también debe ser aplicable a las imágenes en escala de grises. - Descargar CellProfiler del Instituto Broad 26 (http://cellprofiler.org/download.shtml) y ImageJ de los Institutos Nacionales de Salud (http://rsb.info.nih.gov/ij/download.html), ambos los cuales son a disposición del público sin costo alguno.
- Para medir el área de la madre sustituta en cada imagen, ImageJ abierta, seleccione "Establecer mediciones" (en la pestaña "Analizar"), seleccione "Area" y luego seleccione "Ok & #34 ;.
- Abrir una imagen (archivo .tif) de la madre sustituta. Con la herramienta polígono en ImageJ (ver Figura 2), se rodeará el área de la madre sustituta en la imagen arrastrando el ratón y haciendo clic para que los puntos de anclaje. Usa los bordes de la ECM como una guía. Una vez esbozado, seleccione "medida" en la pestaña "Analizar".
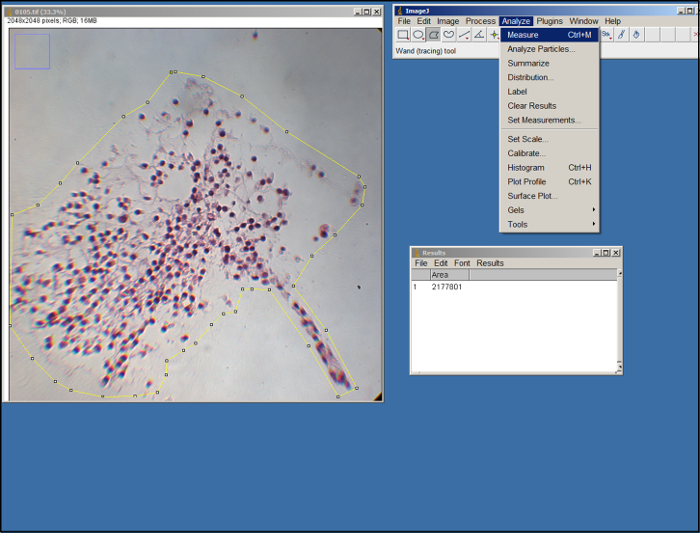
Figura 2. Análisis de ImageJ. Captura de pantalla de procesamiento de ImageJ. Haga clic aquí para ver una versión más grande de esta figura.
- Repita este procedimiento para cada imagen de la madre sustituta tejido. Guarde las mediciones y los identificadores de imagen correspondientes a un programa de hoja de cálculo.
- Cargar archivos de imagen utilizados para medir el área en ImageJ en CellProfiler arrastrando los archivos de imagen a la "Lista de archivos". Asignar un nombre a las imágenes importadas enel "NamesAndTypes" módulo de entrada y seleccione el tipo de imagen (es decir, "Color").
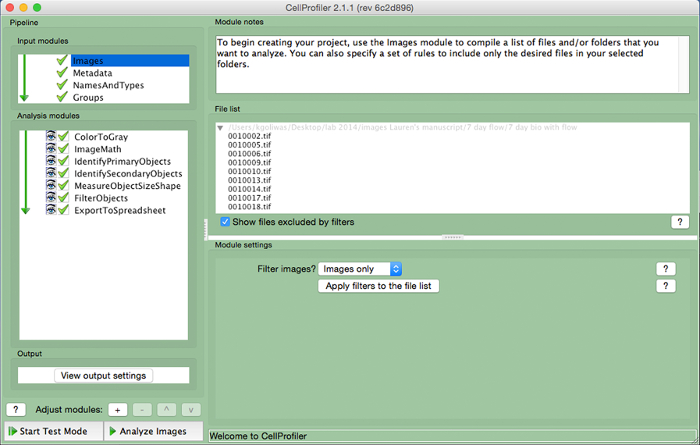
Figura 3. CellProfiler ejemplo tubería. Captura de pantalla de la tubería diseñada para medir el número de células nucleadas en CellProfiler. Haga clic aquí para ver una versión más grande de esta figura.
- Crear un análisis de tuberías que incluye los siguientes módulos para calcular el número de células por imagen, haciendo clic en el signo "+" al lado de "Ajuste del módulo" (Figura 3, en la parte inferior del panel de la izquierda). Añadir a cada uno de los módulos siguientes.
- Seleccione "ColorToGray" (Figura 4).
- Seleccione el nombre de la imagen de entrada desde el menú desplegable, el nombre de la imagen de salida, y seleccionar el tipo de imagen original ( es decir, si el color RGB de entrada de imagen).
Nota: no sería necesario Este módulo si el uso de imágenes en escala de grises
- Seleccione el nombre de la imagen de entrada desde el menú desplegable, el nombre de la imagen de salida, y seleccionar el tipo de imagen original ( es decir, si el color RGB de entrada de imagen).
- Seleccione "ImageMath" (Figura 5).
- Seleccione la operación "Invertir", el nombre de la imagen de salida, y selecciona la imagen en escala de grises en la pestaña de "seleccionar en primer lugar la imagen".
- Seleccione "IdentifyPrimaryObjects" (Figura 6).
- Seleccionar imagen de entrada (imagen después de la corrección de matemáticas), el nombre del objeto principal ser identificado (núcleos), y entrar en el rango de diámetro de los objetos que se miden en unidades de píxel (aproximadamente 25 a 65). Seleccione estrategia de "adaptación" umbral con el método de umbral "Otsu" con "tres clases". No cambie ningún otro parámetro de la configuración predeterminada.
Nota: intervalo óptimo de diámetros debe ser determinada por la apertura de una imagen en el módulo de imagen de entrada y midiendo el diámetro de los núcleos (es decir,el objeto primario) con la función de medida de longitud.
- Seleccionar imagen de entrada (imagen después de la corrección de matemáticas), el nombre del objeto principal ser identificado (núcleos), y entrar en el rango de diámetro de los objetos que se miden en unidades de píxel (aproximadamente 25 a 65). Seleccione estrategia de "adaptación" umbral con el método de umbral "Otsu" con "tres clases". No cambie ningún otro parámetro de la configuración predeterminada.
- Seleccione "IdentifySecondaryObjects" (Figura 7).
- Seleccione la imagen de entrada (imagen después de la corrección de matemáticas), seleccione los objetos de entrada (núcleos), y nombrar los objetos puedan ser identificados (células). Elegir el método de "propagación" para identificar los objetos secundarios, utilice el método "Otsu" estrategia "adaptativo" umbral y con "dos clases" minimizando "varianza ponderada". Seleccione "sin suavizado" y un factor de corrección del umbral 1, Límites superior e inferior de 0 y 1, y un factor de regularización de 0,05. No cambie ningún otro parámetro de la configuración predeterminada.
- Seleccione "MeasureObjectSizeShape" (Figura 8).
- Seleccione las celdas (objeto secundario) y los núcleos (objeto primario) como los objetos a medir.
- Seleccione "FilterObjects" (Figura 9 ).
- Nombrar los objetos de salida y seleccione los núcleos (objeto principal) como el objeto de filtrar.
- Mantenga los siguientes dos parámetros como los ajustes por defecto. Seleccione "AreaShape" como la medida para filtrar por categoría, y "FormFactor" como la medida.
- Seleccione "Sí" para filtrar utilizando un valor de medición mínimo y añadir un valor mínimo de 0,52.
- Seleccione "No" para filtrar usando una medida máxima.
- Seleccione "Sí" para retener los contornos de los objetos filtrados y el nombre de la imagen se indica.
- Seleccione "ExportToSpreadsheet" (Figura 10)
- Seleccionar el lugar donde guardar el archivo y el nombre de "archivos de salida".
- Una vez generado el análisis de tuberías (Pasos 6.7.1 a 6.7.7.1), start "modo de prueba" en CellProfiler y evaluar cada paso, incluyendo los núcleos que la imagen de prueba se filtran adecuadamente, para garantizar una óptima paparáme- fueron elegidos para identificar las células. La Figura 11 muestra una imagen de salida de CellProfiler siguiente de filtrado, donde los núcleos están correctamente identificados (en el círculo en verde), y el fondo es filtran.
- Una vez que se evalúe y determine que sea suficiente parámetros, guardar el proyecto y haga clic en "Analizar las imágenes". Este proyecto se puede utilizar en varias ocasiones para futuros análisis.
Nota: Una vez que se han establecido los parámetros, el programa está configurado para analizar todas las imágenes en la "Lista de archivos", de forma secuencial. Esto resultará en múltiples ventanas que se abren para cada imagen analizada, lo que provoca un tiempo de procesamiento más largo.- Para evitar un tiempo de proceso, haga clic en el icono del ojo en todos los módulos excepto "ExportToSpreadsheet" en la sección "módulos de análisis".
- Una vez que todas las imágenes de sustitución se han procesado por CellProfiler, abrir la hoja de cálculo que contiene los datos de la imagen generada por la célula Profiler y la hoja de cálculo que contiene el área medida con ImageJ. Copiar los datos filtrados núcleos (columna D de la hoja de cálculo CellProfiler) y los identificadores de imagen (columna I) y pegarlos en la hoja de cálculo que contiene los datos del área de medición.
- Calcular la suma de todas las mediciones obtenidas por el número de núcleos por imagen de la madre sustituta.
- Calcular la suma de las mediciones obtenidas para el área de sustituto de cada imagen de la madre sustituta. Se divide el área total medida por 1x10 6.
- Se divide el número total de núcleos por la medida del área total en el paso anterior para obtener un valor para el número de células por 1x10 6 pixeles 2.
- Seleccione "ColorToGray" (Figura 4).
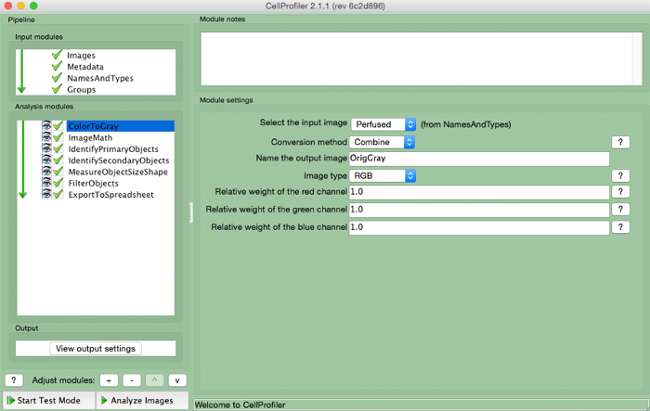
Figura 4. tubería CellProfiler: cambiar la imagen a escala de grises Captura de pantalla de módulo "ColortoGray"..s: //www.jove.com/files/ftp_upload/54004/54004fig4highres.jpg "target =" _ blank "> Haga clic aquí para ver una versión más grande de esta figura.
Figura 5. CellProfiler tubería:.. Invirtiendo la imagen de pantalla del módulo "ImageMath" Haga clic aquí para ver una versión más grande de esta figura.

Figura 6. CellProfiler tubería:.. Identificando núcleos Captura de pantalla del módulo "IdentifyPrimaryObjects" Haga clic aquí para ver una versión más grande de esta figura.
Figura 7. CellProfiler tubería:.. Que identifican las células Captura de pantalla del módulo "IdentifySecondaryObjects" Haga clic aquí para ver una versión más grande de esta figura.
Figura 8. CellProfiler tubería:.. Objetos de medición del módulo de pantalla "MeasureObjectSizeShape" Haga clic aquí para ver una versión más grande de esta figura.
Figura 9. CellProfiler tubería:. Filtrar objetos Captura de pantalla de "FilterObjects "módulo. Haga clic aquí para ver una versión más grande de esta figura.
Figura 10. CellProfiler tubería:.. La exportación de datos Captura de pantalla de módulo "ExportToSpreadsheet" Haga clic aquí para ver una versión más grande de esta figura.
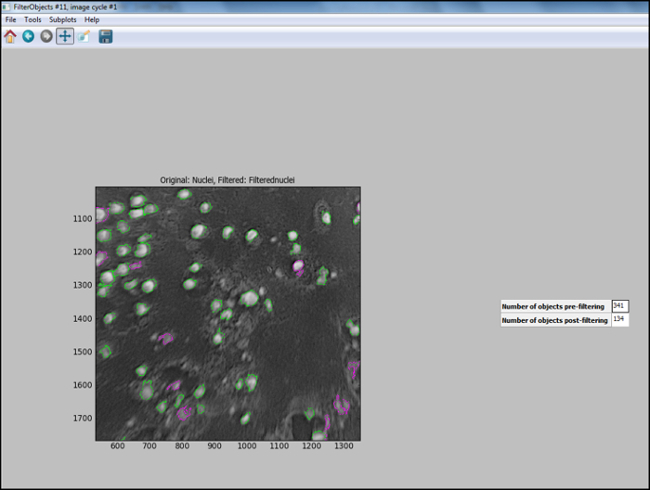
Figura 11. CellProfiler de salida después de la filtración. Captura de pantalla de salida en el generador de perfiles de células después de la filtración objeto. Haga clic aquí para conocer el Versi más grandesel de esta figura.
Resultados
Tanto sustitutos de cáncer de mama sólidos y perfundidos 3D se prepararon como se ha descrito anteriormente y se cultivaron durante 7 días. Posteriormente, sustitutos se fijaron, se procesaron para parafina, seccionadas y teñidas con hematoxilina y eosina, tal como se describe anteriormente. Se midió el número de células nucleadas por área (ambas células 231 y CAF) de cada sustituto. Como puede verse en la Figura 12, fotomicrografías representativas de las secc...
Discusión
En este documento, un método de cultivo 3D se ha descrito que incorpora componentes del microambiente del tejido, incluyendo la matriz extracelular (ECM) y fibroblastos estromales humanos, en un volumen que más de cerca los modelos de cáncer de mama humano para permitir el desarrollo de una morfología 3D recapitulativo . El método de cultivo 3D descrito es más representativo de la enfermedad humana de cultivo de células 2D tradicional en que múltiples tipos de células se incorporan en un volumen 3D de ECM. Se h...
Divulgaciones
The authors declare that they have no competing financial interests.
Agradecimientos
The University of Alabama at Birmingham Center for Metabolic Bone Disease performed the histologic processing and sectioning of surrogates. Southern Research (Birmingham, AL) provided support for the manufacture of the bioreactor system. Funding was provided by the United States Department of Defense Breast Cancer Research Program (BC121367).
Materiales
| Name | Company | Catalog Number | Comments |
| Dulbecco's Modified Eagel Medium 1x (DMEM) | Corning CellGro | 10-014-CV | |
| Fetal Bovine Serum (FBS) | Atlanta Biologicals | S11150 | |
| 0.25% Trypsin + 2.21 mM EDTA 1x | Corning | 25-053-CI | |
| Tissue Culture plates, 100 mm | CellTreat Scientific Products | 229620 | Sterile |
| Tissue Culture plates, 35 mm | CellTreat Scientific Products | 229638 | For PDMS foam formation |
| 9" Glass pipette | Fisher | 13-678-20D | Sterile |
| 10 ml pipette | CellTreat Scientific Products | 229210B | Sterile |
| 1,000 µl piptette tips | FisherBrand | 02-717-166 | Sterile Filtered |
| 200 µl pipette tips | FisherBrand | 02-717-141 | Sterile Filtered |
| 10 µl pipette tips | FisherBrand | 02-717-158 | Sterile Filtered |
| 15 ml conical tubes | CellTreat Scientific Products | 229410 | Sterile |
| 50 ml conical tubes | CellTreat Scientific Products | 229422 | Sterile |
| 1.5 ml microcentrifuge tubes | FisherBrand | 05-408-129 | Sterile |
| Trypan blue | Corning Cellgro | 25-900-CI | Sterile |
| Sylgard 184 | Electron Microscopy Sciences | 24236-10 | PDMS elastomer and curing agent. Used for our in-house bioreactor. |
| PDMS Foam | Made in-house for use in our in-house bioreactor. | ||
| High Concentration Bovine Collagen Type I | Advanced Biomatrix | 5133-A | FibriCol ~10 mg/ml |
| Growth Factor Reduced Matrigel (Basement Membrane) | Corning | 354230 | Basement membrane material |
| Sodium Bicarbonate | Sigma | S8761 | |
| Molecular Biology Grade Water | Fisher | BP2819-1 | |
| DMEM 10x | Sigma-Aldrich | D2429 | |
| Nunc Lab-Tek Chamber Slide System | Thermo Scientific | 177402 | 8-well |
| Bioreactor | Made in-house. | ||
| Spring-Back 304 Stainless Steel—Coated with PTFE polymer | McMaster-Carr | 1749T19 | Stainless steel wires to generate microchannels in our in-house bioreactor system. 0.016" Diameter |
| BioPharm Plus platinum-cured silicone pump tubing, L/S 14 | Masterflex | EW-96440-14 | For use in our in-house bioreactor system. Tubing ID: 1.6 mm, Hose barb size: 1/16 in. |
| 2-Stop Tubing Sets, non-flared PVC, 1.52 mm ID | Cole-Parmer | EW-74906-36 | For use in our in-house bioreactor system (with microperistalitic pump). |
| Six Channel precision micro peristaltic pump | Cole-Parmer | EW-74906-04 | For use with our in-house bioreactor system |
| Tuberculin Syringes | BD Medical | 309625 | 26 gauge 3/8 in. needle; Sterile |
| Dissecting Tissue Forceps | FisherBrand | 13-812-36 | 5.5 inch |
| Mini Tube Rotator | Boekel Scientific | 260750 | Equipment option for surrogate rotation. Used with carousel for 50 ml tubes (model number 260753) |
| 50 ml tube carousel | Boekel Scientific | 260753 | Used with mini tube rotator |
| Bambino Hybridization Oven | Boekel Scientific | 230301 | Equipment option for surrogate rotation |
| HistoGel Specimen Processing Gel | Thermo Scientific | HG-4000-012 | Specimen Processing Gel described in Step 5.2 |
| Cryomold | Andwin Scientific | 4566 | 15 mm x 15 mm x 5 mm |
| Tissue Marking Dye | Cancer Diagnostics, inc. | 03000P | Can be used to mark surrogates, allowing multiple samples to be included in one tissue cassette |
| Hinged tissue cassettes | FisherBrand | 22-272-416 | |
| Formalin | Fisher | 23-245-685 | |
| GoldSeal Plain Glass Slides | Thermo Scientific | 3048-002 | |
| Xylene | Fisher | X3P-1GAL | |
| Ethanol, 200 proof (100%), USP | Decon Laboratories, Inc. | 2805M | |
| Hematoxylin | Thermo Scientific Richard-Allan Scientific | 7211 | |
| Clarifier | Thermo Scientific Richard-Allan Scientific | 7401 | |
| Bluing Solution | Thermo Scientific Richard-Allan Scientific | 7301 | |
| Eosin Y | Thermo Scientific Richard-Allan Scientific | 7111 | |
| Cytoseal XYL mounting media | Thermo Scientific Richard-Allan Scientific | 83124 | |
| Coverslips | Fisher Scientific | 12-548-5G |
Referencias
- Hakanson, M., Textor, M., Charnley, M. Engineered 3D environments to elucidate the effect of environmental parameters on drug response in cancer. Integr Biol (Camb). 3 (1), 31-38 (2011).
- Horning, J. L., et al. 3-D tumor model for in vitro evaluation of anticancer drugs. Mol Pharm. 5 (5), 849-862 (2008).
- Dhiman, H. K., Ray, A. R., Panda, A. K. Three-dimensional chitosan scaffold-based MCF-7 cell culture for the determination of the cytotoxicity of tamoxifen. Biomaterials. 26 (9), 979-986 (2005).
- Place, A. E., Jin Huh, S., Polyak, K. The microenvironment in breast cancer progression: biology and implications for treatment. Breast Cancer Res. 13 (6), 227 (2011).
- Mao, Y., Keller, E. T., Garfield, D. H., Shen, K., Wang, J. Stromal cells in tumor microenvironment and breast cancer. Cancer Metastasis Rev. 32 (1-2), 303-315 (2013).
- Paulsson, J., Micke, P. Prognostic relevance of cancer-associated fibroblasts in human cancer. Semin Cancer Biol. 25, 61-68 (2014).
- Roskelley, C. D., Desprez, P. Y., Bissell, M. J. Extracellular matrix-dependent tissue-specific gene expression in mammary epithelial cells requires both physical and biochemical signal transduction. Proc. Natl. Acad. Sci. 91, 12378-12382 (1994).
- Pickl, M., Ries, C. H. Comparison of 3D and 2D tumor models reveals enhanced HER2 activation in 3D associated with an increased response to trastuzumab. Oncogene. 28 (3), 461-468 (2008).
- Ivascu, A., Kubbies, M. Rapid generation of single-tumor spheroids for high-throughput cell function and toxicity analysis. J Biomol Screen. 11 (8), 922-932 (2006).
- Lovitt, C. J., Shelper, T. B., Avery, V. M. Advanced cell culture techniques for cancer drug discovery. Biology (Basel). 3 (2), 345-367 (2014).
- Bergamaschi, A., et al. Extracellular matrix signature identifies breast cancer subgroups with different clinical outcome. J Pathol. 214 (3), 357-367 (2008).
- Oskarsson, T. Extracellular matrix components in breast cancer progression and metastasis. The Breast. 22, S66-S72 (2013).
- Kelley, L. C., Lohmer, L. L., Hagedorn, E. J., Sherwood, D. R. Traversing the basement membrane in vivo: A diversity of strategies. JBC. 204 (3), 291-301 (2014).
- Sadlonova, A., et al. Breast fibroblasts modulate epithelial cell proliferation in three-dimensional in vitro co-culture. Breast Cancer Res. 4, (2004).
- Wendt, D., Marsano, A., Jakob, M., Heberer, M., Martin, I. Oscillating perfusion of cell suspensions through three-dimensional scaffolds enhances cell seeding efficiency and uniformity. Biotechnol Bioeng. 84 (2), 205-214 (2003).
- Marshall, L. E., et al. Flow-perfusion bioreactor system for engineered breast cancer surrogates to be used in preclinical testing. J Tissue Eng Regen Med. , (2015).
- Calcagnile, P., Fragouli, D., Mele, E., Ruffilli, R., Athanassiou, A. Polymeric foams with functional nanocomposite cells. RSC Adv. 4, 19177-19182 (2014).
- Naba, A., Clauser, K. R., Lamar, J. M., Carr, S. A., Hynes, R. O. Extracellular matrix signatures of human mammary carcinoma identify novel metastasis promoters. eLife. 3, (2014).
- Lochter, A., Bissell, M. J. Involvement of extracellular matrix constituents in breast cancer. Semin Cancer Biol. 6 (3), 165-173 (1995).
- Joiner, K. S., Spangler, E. A. Evaluation of HistoGel-embedded specimens for use in veterinary diagnostic pathology. J Vet Diagn Invest. 24 (4), 710-715 (2012).
- Varsegi, G. M., Shidham, V. Cell Block Preparation from Cytology Specimen with Predominance of Individually Scattered Cells. J Vis Exp. (29), e1316 (2009).
- Sadlonova, A., et al. Human Breast Fibroblasts Inhibit Growth of the MCF10AT Xenograft Model of Proliferative Breast Disease. Am J Pathol. 170 (3), (2007).
- Otali, D., He, Q., Stockard, C. R., Grizzle, W. E. Preservation of immunorecognition by transferring cells from 10% neutral buffered formalin to 70% ethanol. Biotech Histochem. 88, 170-180 (2013).
- Webster, S. S., Jenkins, L., Burg, K. J. L. Histological Techniques for Porous, Absorbable, Polymeric Scaffolds, Used in Tissue Engineering. J Histotechnol. 26 (1), 57-65 (2003).
- Troy, T. -. C., Arabzadeh, A., Enikanolaiye, A., Lariviere, N., Turksen, K. Immunohistochemistry on Paraffin Sections of Mouse Epidermis Using Fluorescent Antibodies. J Vis Exp. (11), (2008).
- Carpenter, A. E., et al. CellProfiler: image analysis software for identifying and quantifying cell phenotypes. Genome Biol. 7 (10), R100 (2006).
- Kwon, Y. -. J., et al. Gli1 enhances migration and invasion via up-regulation of MMP-11 and promotes metastasis in ERa negative breast cancer cell lines. Clin Exp Metastasis. (28), (2011).
- Evilsizor, M. N., Ray-Jones, H. F., Lifshitz, J., Ziebell, J. Primer for Immunohistochemistry on Cryosectioned Rat Brain Tissue: Example Staining for Microglia and Neurons. J Vis Exp. (99), e52293 (2015).
- Pal, A., Kleer, C. G. Three dimensional cultures: a tool to study normal acinar architecture vs. malignant transformation of breast cells. J Vis Exp. (86), e51311 (2014).
- Hasselbach, L. A., et al. Optimization of High Grade Glioma Cell Culture from Surgical Specimens for Use in Clinically Relevant Animal Models and 3D Immunochemistry. J Vis Exp. (83), e51088 (2014).
- Foty, R. A Simple Hanging Drop Cell Culture Protocol for Generation of 3D Spheroids. J Vis Exp. (51), e2720 (2011).
- Materne, E. -. M., et al. The Multi-organ Chip - A Microfluidic Platform for Long-term Multi-tissue Coculture. J Vis Exp. (98), e52526 (2015).
- Sadlonova, A., et al. Identification of Molecular Distinctions Between Normal Breast-Associated Fibroblasts and Breast Cancer-Associated Fibroblasts. Cancer Microenviron. 2, 9-21 (2009).
- Wang, J. D., Douville, N. J., Takayama, S., ElSayed, M. Quantitative analysis of molecular absorption into PDMS microfluidic channels. Ann Biomed Eng. 40 (9), 1862-1873 (2012).
- Halldorsson, S., Lucumi, E., Gomez-Sjoberg, R., Fleming, R. M. Advantages and challenges of microfluidic cell culture in polydimethylsiloxane devices. Biosens Bioelectron. 63, 218-231 (2015).
- Regehr, K. J., et al. Biological implications of polydimethylsiloxane-based microfluidic cell culture. Lab Chip. 9 (15), 2132-2139 (2009).
- Burdett, E., Kasper, F. K., Mikos, A. G., Ludwig, J. A. Engineering tumors: a tissue engineering perspective in cancer biology. Tissue Eng Part B Rev. 16 (3), 351-359 (2010).
- Caruso, R. A., et al. Mechanisms of coagulative necrosis in malignant epithelial tumors (Review). Oncol Lett. 8 (4), 1397-1402 (2014).
- Elmore, S. Apoptosis: A Review of Programmed Cell Death. Toxicol Pathol. 35 (4), 495-516 (2007).
- Majno, G., Joris, I. Apoptosis, oncosis, and necrosis. An overview of cell death. Am J Pathol. 146 (1), 3-15 (1995).
- Ogino, S., et al. Molecular pathological epidemiology of epigenetics: emerging integrative science to analyze environment, host, and disease. Mod Pathol. 26 (4), 465-484 (2013).
- Otali, D., et al. Combined effects of formalin fixation and tissue processing on immunorecognition. Biotech Histochem. 84 (5), 223-247 (2009).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados