Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Préparation et analyse des
Dans cet article
Résumé
We demonstrate a method to generate 3D breast cancer surrogates, which can be cultured using a perfusion bioreactor system to deliver oxygen and nutrients. Following growth, surrogates are fixed and processed to paraffin for evaluation of parameters of interest. The evaluation of one such parameter, cell density, is explained.
Résumé
Trois dimensions de la culture (3D) est une méthode plus physiologiquement pertinents pour modéliser le comportement des cellules in vitro que deux cultures dimensionnel. Les cancers, y compris les cancers du sein sont des tissus 3D complexes composées de cellules cancéreuses épithéliales et stromales de composants, y compris les fibroblastes et la matrice extracellulaire (ECM). Mais la plupart des modèles in vitro de cancer du sein sont constitués uniquement de cellules epitheliales cancéreuses, en omettant le stroma et, par conséquent, l'architecture en 3D d'une tumeur in vivo. modélisation 3D appropriée du cancer est important pour la compréhension précise de la biologie de la tumeur, le comportement et la réponse au traitement. Toutefois, la durée de la culture et le volume des modèles 3D est limitée par la disponibilité de l'oxygène et des nutriments dans la culture. Ici, nous démontrons une méthode dans laquelle les cellules de carcinome du sein épithéliales et les fibroblastes du stroma sont incorporés dans l'ECM pour générer une porteuse de cancer du sein en 3D qui comprend stroma et peuvent être cultivées en tant questructure 3D solide ou en utilisant un système de bioréacteur à perfusion pour délivrer de l'oxygène et de nutriments. Après l'installation et une période de croissance initiale, les substituts peuvent être utilisés pour le dépistage des drogues préclinique. En variante, les composants cellulaires et de la matrice de la mère porteuse peuvent être modifiés pour répondre à une variété de questions biologiques. Après la culture, les mères porteuses sont fixées et traitées à la paraffine, d'une manière similaire à la manipulation d'échantillons de carcinome du sein clinique pour l'évaluation des paramètres d'intérêt. L'évaluation de l'un de ces paramètres, la densité de cellules présentes, il est expliqué, le cas des systèmes logiciels d'analyse d'image et ImageJ CellProfiler sont appliqués à des photomicrographies des coupes histologiques de mères porteuses pour quantifier le nombre de cellules nucléées par zone. Ceci peut être utilisé comme un indicateur de la variation du nombre de cellules au cours du temps ou la variation du nombre de cellules résultant de différentes conditions de croissance et les traitements.
Introduction
Trois modèles (3D) de culture de dimensions qui imitent plus précisément l'architecture de la tumeur et microenvironnement in vivo sont importantes pour des études visant à disséquer les interactions complexes entre les cellules et leur microenvironnement et pour tester l'efficacité des thérapies candidates. Tumeur impacts dimensionnalité de l' oxygène et des gradients de nutriments, l'uniformité de l' exposition au médicament, interstitielle débit / pression sanguine, et de l' architecture 3D 1-4. La présence d'un micro-environnement stromal approprié contribue à la dimensionnalité des tumeurs et des influences de signalisation cellule-ECM et la signalisation entre paracrine des cellules stromales et les cellules épithéliales malignes. Les effets de la dimensionnalité de la tumeur et le microenvironnement sur la fonction cellulaire sont bien établis, avec les deux facteurs altérant la réponse aux médicaments 1,3,5-8. En outre, la cinétique cellulaire de croissance, les taux métaboliques, et la signalisation cellulaire diffèrent entre les deux (2D) la culture et de la culture dimensionnelle en 3D, avec ces facteurs affection réponse cellulaire 1,3,8-10.
In vitro, la tumeur microenvironnement porteuse peut être modulée en incluant des composantes représentatives de la MEC et les populations de cellules stromales. Les cellules épithéliales malignes sont influencées par l'ECM et les cellules stromales associées au cancer soit d'une manière synergique / protecteur pour favoriser la progression tumorale ou d'une manière suppressive pour inhiber la propagation ultérieure de la tumeur 5,6,10. Dans les deux contexte, le stroma peut affecter la réponse thérapeutique et la délivrance de médicaments par l' intermédiaire d'une signalisation paracrine et / ou en augmentant la pression interstitielle dans la tumeur , entraînant une diminution de l' administration de médicaments à 1,6. Par conséquent, l'ajout de l'ECM et les cellules stromales dans des modèles précliniques aidera récapituler les aspects de la tumeur qui ne peut pas être bien modélisé dans la culture 2D.
Ici une méthode pour établir des substituts du cancer du sein qui incorporent un microenvironnement récapitulatifs, y compris les composants et s ECMtromal cellules dans un volume 3D est décrit. Dans le cancer du sein, la population de cellules stromales est principalement constitué de fibroblastes associés au cancer (FAC) et l'ECM stromales se compose essentiellement de collagène de type I avec une faible proportion de composants de la matrice que l'on trouve dans la membrane basale, y compris la laminine et du collagène de type IV 1,4,11-13. Par conséquent, ces composants du microenvironnement de carcinome du sein (c. -à- CAF, le collagène I, et la membrane basale) ont été incorporés dans les mères porteuses. Cette méthode peut être utilisée pour générer des solides, des substituts de l' ONU 3D perfusé (figure 1A) ou peut être adapté pour inclure la perfusion du milieu à travers le substitut par l' intermédiaire d' un système de bioréacteur (figure 1B). Les deux approches sont décrites ici. Cette méthode peut également être modifiée pour inclure d'autres éléments du stroma, telles que les macrophages associés aux tumeurs, ou pour modéliser d'autres tumeurs solides en ajustant les composants cellulaires et ECM, le cas échéant.
Pour la substitution de carcinome du sein décrit ici, nous avons utilisé la MDA-MB-231 (231) , la lignée cellulaire de cancer du sein, CAF précédemment isolé du carcinome du sein humain 14, et un ECM composé de 90% de collagène I ( le facteur de croissance matériau 6 mg / ml) et 10% de sous-sol réduite membrane (BM). Le substitut est soit cultivé dans un 8 puits lame de chambre (substitut solide) ou un système de bioréacteur est utilisé pour fournir une perfusion de nutriment continue (substitution perfusé). Tout système de bioréacteur à perfusion qui peut accueillir un volume de cellules ECM contenant peut être utilisé 15. A titre d'exemple, nous décrivons la préparation des succédanés de tissus dans le système de bioréacteur. Ce système a été développé en interne et ne sont pas disponibles dans le commerce. Parce que notre accent est mis sur la préparation et l'analyse des substituts de tissu 3D, nous ne sommes pas allés dans de nombreux détails sur les spécificités de la fabrication et l'assemblage de notre système de bioréacteur. Toutefois, une description détailléece système et son développement a été publié le 16. Dans ce système de bioréacteur, un canal d'écoulement (PDMS) est utilisé pour loger la porteuse, qui est supporté par une mousse de PDMS (formé en utilisant des procédés similaires à ceux décrits par Calcagnile et al. 17). Ce volume est traversé par des microcanaux 4 (chacun de 400 um de diamètre) qui sont continuellement perfusées par du milieu par l'intermédiaire d'une pompe à microphysiologic pour fournir de l'oxygène et des nutriments à la substitution.Une analyse appropriée des mères porteuses est crucial d'obtenir des informations pertinentes concernant la fonction cellulaire en réponse à un traitement ou d'autres manipulations. Les substituts peuvent être analysés par différents procédés, y compris l'imagerie directe des mères porteuses intactes par microscopie confocale ou d'autres moyens d'imagerie non invasive, l'analyse cellulaire indirecte par dosage du milieu conditionné ou perfusé, pour les produits sécrétés, ou des analyses sur des coupes histologiques après la fixation et le traitement àparaffine. Un tel paramètre qui peut être évalué sur des coupes histologiques est la densité cellulaire. Nous présentons une approche pour mesurer la densité cellulaire ( à savoir, le nombre de cellules nucléées par section zone) en utilisant des techniques de traitement d' image semi-automatisés appliqués aux photomicrographies des coupes histologiques de substitution colorées à l' hématoxyline et l' éosine (H & E). La densité cellulaire peut être utilisée comme un indicateur de la variation relative du nombre de cellules au cours du temps ou qui résulte de différentes conditions de croissance et les traitements.
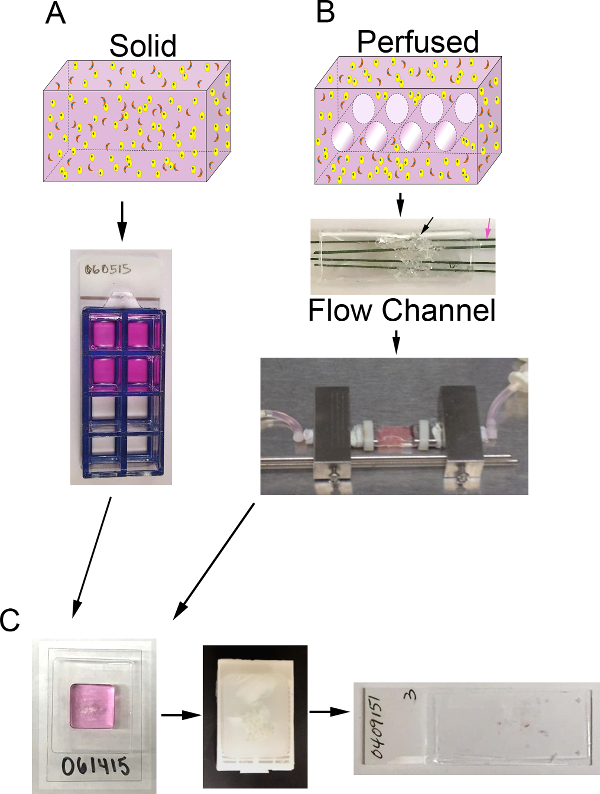
Figure 1. Volume 3D et système de bioréacteur. A) Schéma du processus pour produire des substituts 3D solides. Top: bande dessinée de volume 3D solide contenant ECM (rose), des cellules de carcinome épithéliales (jaune) et CAF (orange); Bottom:. Vue de dessus de 8 puits chambre diapositive contenant des substituts B) Schéma du processus pour générer perfusé mères porteuses 3D. Top: cartoon de volume 3D avec des canaux pour permettre perfusion de milieu et contenant ECM (rose), des cellules de carcinome épithéliales (jaune) et CAF (orange); Moyen: image de canal d'écoulement PDMS contenant de la mousse PDMS (flèche noire) à injecter avec la cellule + ECM et pénétrée par des fils d'acier inoxydable enduites de polymère (flèche rose) mesurant 400 m de diamètre; En bas:. L' image du canal d'écoulement de PDMS contenant une mère porteuse et connecté au système de bioréacteur pour permettre une perfusion de milieu continu (pompe péristaltique et le réservoir de support non représenté) , C) , les images des étapes de traitement pour les mères porteuses solides et perfusées après la culture. Gauche: image du cryomoule contenant l'échantillon de traitement du gel et de substitution; Moyen: l'image d'un bloc de paraffine contenant un substitut fixe et traité; A droite:. Image d'une lame de verre avec une coupe histologique H & E teinté d'un substitut S'il vous plaît cliquer ici pour voir uneune plus grande version de ce chiffre.
Protocole
1. Culture cellulaire
- Décongeler composante BM nuit à 4 ° C, sur la glace.
- milieu chaud à 37 ° C. Pour soutenir la croissance des deux 231 cellules et CAF, utiliser du milieu Eagle modifié Dulbecco (DMEM) plus 10% de sérum de veau fœtal (FBS).
Remarque: Les supports utilisés dépendent du type de cellule et les objectifs expérimentaux. - Retirer moyen de la boîte de culture (10 cm) de près de cellules confluentes 231 et ajouter 1,5 ml de trypsine / EDTA. Incuber pendant 1 à 3 minutes à 37 ° C, le suivi du détachement des cellules. Une fois que les cellules commencent à arrondir et se détacher de la plaque, arrêter la réaction en ajoutant 3 ml de sérum de milieu contenant. Pipeter le milieu et les cellules dans un tube conique de 15 ml.
- Centrifuger le milieu et les cellules à 150 g pendant 5 min. Eliminer le surnageant et remettre en suspension le culot cellulaire dans 2 ml de milieu.
- Comptez le nombre de cellules par volume à l'aide de bleu trypan et un hémocytomètre. La viabilité des cellules doit être supérieure à 90%.
- Répétez laprocédé avec d'autres types de cellules à inclure dans la substitution. Pour le substitut décrit ici, le processus est répété avec la CAF.
- Déterminer le volume approprié de chaque suspension cellulaire afin d'obtenir le nombre désiré de cellules.
Remarque: Pour la substitution décrit ici, une densité cellulaire de 2,1 x 10 6 cellules par 100 pi de l' ECM, avec un rapport de 2: 1 des cellules epitheliales à des fibroblastes (1,4 x 10 6 231 cellules et 7 x 10 5 CAF pour 100 ul ECM), est utilisé. Cette densité cellulaire est un bon point de départ; Cependant, la densité cellulaire optimale dépendra du type de cellule, la durée de la culture, et les objectifs de l'expérience. - Ajouter le volume approprié de chaque suspension cellulaire dans un tube conique de 15 ml (un tube pour chaque type de cellule). Centrifuger le milieu et les cellules à 150 g pendant 5 min.
- Après centrifugation, éliminer le surnageant, remettre en suspension un type de cellule dans la cellulel' eau de qualité culture (178,8 pi, voir le tableau 1) et l'autre type de cellule dans 10x DMEM (100 pi, voir le tableau 1, contenant du phénol rouge pour contrôler le pH). Placer les tubes contenant les cellules sur la glace et procéder rapidement à la préparation de l'ECM ci-dessous. Limitez le temps que les cellules restent dans l'eau pour préserver la viabilité.
Remarque: Les deux composants 10x DMEM et de l' eau cellulaire de qualité de culture sont nécessaires de l'ECM; par conséquent, nous avons choisi de remettre en suspension les cellules dans les deux. Ici , nous avons choisi arbitrairement remettre en suspension les cellules 231 en culture cellulaire eau de qualité et de la CAF en 10x DMEM, bien que les deux types de cellules pourrait être remis en suspension dans l' une de ces deux composantes.
2. Préparation de cellules dans l'ECM (6 mg / ml de collagène bovin de type I + 10% BM)
Remarque: Une ECM composée de 90% de collagène I + 10% BM a été choisi pour modéliser le carcinome du sein invasif car le stroma tumoral dans cette tumeur maligne est composée principalement de collagèneI avec des composants de la BM, telles que la laminine, le collagène IV, et l' entactine, comprenant une plus petite partie de la MEC 12,13,18,19.
- Sur la glace, ajouter des composants énumérés dans le tableau 1, dans l' ordre, dans un tube de microcentrifugeuse de 2 ml.
Remarque: Cette quantité est suffisante pour 8 substituts solides ou, en utilisant le système de bioréacteur décrit ici, 4 substituts perfusés.
| Préparation de cellules dans l' ECM (6 mg / ml de collagène bovin de type I + 10% BM) | |
| 178,8 μl | La culture cellulaire eau de qualité contenant le nombre souhaité de 231 cellules (déterminé ci-dessus) |
| 606 pi | Le collagène I (10 mg / ml de bovin), ajouter goutte à goutte |
| 100 ul | membrane basale, décongelé |
| 100 ul | 10x DMEM (contenant du phénol rouge) avec le nombre souhaité de la CAF (déterminé ci-dessus) |
| 15,2 pi | 7,5% (v / v) de bicarbonate de sodium, ajouter goutte à goutte |
Tableau 1. Préparation de cellules dans l' ECM.
- Mélanger doucement par pipetage, en évitant la formation de bulles. Surveiller le niveau de pH en utilisant le rouge de phénol dans le milieu DMEM 10x. Vérifiez que le mélange est une couleur orange / rose indiquant un pH de ~ 7. Si le pH est trop bas (trop jaune), ajouter lentement supplémentaire de 7,5% de bicarbonate de sodium, une goutte à la fois (~ 5 pi) jusqu'à ce que la couleur appropriée est atteinte.
- Gardez le mélange sur la glace et travailler rapidement pour empêcher la polymérisation de l'ECM prématurée.
3. Surrogate Préparation
- Pour les cultures 3D solides (Figure 1A):
- Travailler dans une hotte de culture cellulaire en utilisant une technique stérile, étiqueter le couvercle d'un 8 puits chambre diapo stérile pour indiquer toute variation expérimentale en substituts.
- Garder la lame de la chambre sur la glace pour empêcher la polymérisation de l'ECM prématurée, lentement Pipeter 100 pi du mélange de cellules + ECM dans chaque puits de la lame de la chambre 8 puits.
Remarque: pipetage du mélange de cellules + ECM autour des bords du premier puits permet de mieux répartir le mélange de cellules + ECM dans le puits. - Incuber mères porteuses à 37 ° C, 5% de CO 2 pendant 45 minutes pour permettre la polymérisation de l' ECM.
- Après la polymérisation de l' ECM, ajouter 100 milieux de culture ul à chaque puits et incuber à 37 ° C, 5% de CO 2 pendant toute la durée de l'expérience,la modification du milieu de culture tous les deux jours.
- Pour les cultures 3D perfusées dans un système de bioréacteur (figure 1B):
- Stériliser tous les composants de bioréacteurs pour la configuration de la culture 3D (ie, bioréacteurs, des tubes, des pinces et les raccords nécessaires pour la configuration du bioréacteur) en utilisant le procédé spécifique pour le bioréacteur à utiliser.
- Par exemple pour le système de bioréacteur utilisé ici, utiliser une combinaison d'autoclavage (par exemple, une exposition 12 min à 121,1 ° C , avec un séchage 15 min) et incubation dans 70% d' éthanol pendant 1 heure.
- Préparer et assembler la partie du système de bioréacteur qui abritera la mère porteuse.
- Pour le système de bioréacteur exemple utilisé ici, insérez un squelette de mousse de PDMS dans le canal d'écoulement PDMS tube à l'aide de forceps. Appuyez sur quatre (400 um) fils en acier inoxydable enduites de polymère dans la mousse de PDMS pour générer des microcanaux parallèles.
- Dans une hotte de culture cellulaire, en utilisant SterilTechnique e et une aiguille de calibre 26 avec une seringue, injecter le mélange de cellules + ECM dans la zone du bioréacteur de perfusion destinée à contenir des cellules. Procéder rapidement à l'étape suivante.
- Pour assurer une répartition plus uniforme des cellules à l'intérieur des mères porteuses, placer le composant de bioréacteur logeant les mères porteuses dans un tube conique de 50 ml (sous la hotte de culture cellulaire) et tourner de façon continue à environ 18 tours par minute pendant une incubation à 37 ° C pendant 45 min pour permettre l'ECM polymérisation.
Remarque: La rotation peut être complété en utilisant un rotateur dans l'incubateur ou un four avec un construit en rotateurs, comme une hybridation four réglé à 37 ° C. - Connecter l'ensemble de bioréacteur contenant le substitut à la pompe à perfusion en utilisant les instructions du fabricant.
Remarque: Les détails de ce processus varie en fonction du bioréacteur et de la pompe utilisée.- Pour le système de bioréacteur exemple utilisé ici, retirez les fils en acier inoxydable avant de connecter le bioreacteur ensemble à la pompe.
- Lancer la perfusion du milieu (de débit en vrac de 167,1 pi / min; microcanaux paroi contrainte de cisaillement de 1 dyne / cm 2) dans un incubateur à 37 ° C, 5% de CO 2.
Note: Le taux de perfusion du milieu peut être ajustée en fonction de la configuration du bioréacteur et les objectifs et la conception de l'expérience. - Continuer perfusion du milieu pendant toute la durée de l'expérience, en changeant le milieu de culture tous les sept jours.
- Stériliser tous les composants de bioréacteurs pour la configuration de la culture 3D (ie, bioréacteurs, des tubes, des pinces et les raccords nécessaires pour la configuration du bioréacteur) en utilisant le procédé spécifique pour le bioréacteur à utiliser.
4. Surrogate Fixation et traitement (figure 1C)
- cryomoules d'étiquetage et les cassettes de tissus en plastique pour la fixation et le traitement de substitution.
- Ensuite, les mères porteuses Encase dans un gel de traitement de l'échantillon, qui est une matière aqueuse qui est liquide à des températures élevées, mais se solidifie à température ambiante. Les passes de gel de traitement des échantillons en gardant les substituts intacts au cours du traitement et facilite histologiques sectionnant 14,20-22.
- Faire fondre l'échantillon de traitement du gel dans un bain d'eau C 60 ° à liquéfier, en gardant à cette température jusqu'à utilisation. Déplacez le bioréacteur avec la mère porteuse à une enceinte de sécurité biologique.
- Introduire à la pipette environ 300 pl du spécimen de gel de traitement dans le fond de la cryomoule marquée (figure 1C, le panneau de gauche).
- En utilisant une lame de scalpel (n ° 10 préféré) et une pince retirer délicatement le substitut du bioréacteur ou du puits d'une chambre diapositive 8 puits et placez-le dans le cryomoule contenant l'échantillon de traitement gel.
Nota: Les colorants de marquage de tissus (voir Matériaux / Liste d' équipement pour un exemple spécifique) de couleurs différentes peuvent être utilisées pour marquer les mères porteuses, permettant ainsi de multiples échantillons à inclure dans une cassette de tissus d'une manière reconnaissable. - Pipette environ 300 échantillons ul traitement gel pour couvrir la mère porteuse dans le cryomoule et incuber à 4 ° C pendant 30 min à solidify.
- Une fois que le gel de traitement des échantillons est solidifié, enlever le gel de traitement de l'échantillon contenant le substitut de la cryomoule, et le placer dans une cassette de tissu.
- Placer la cassette de tissus contenant la substitution dans 10% de formaline tamponnée neutre pendant 10 à 12 heures à la température ambiante pour permettre une fixation complète.
- Après la fixation, déplacer la cassette de tissus contenant le substitut éthanol à 70% jusqu'au traitement à la paraffine.
Note: Transfert de la mère porteuse de formaline à l' éthanol empêche over-fixation à la formaline qui peut entraîner une perte d'immunoréactivité de certains épitopes 23. La longueur du temps dans de l' éthanol ne soit pas critique. Ce changement de fixatif est important si la mère porteuse sera utilisée pour l' immunohistochimie ou immunofluorescence. Le substitut fixe est maintenant prêt pour le traitement à la paraffine (Figure 1C, panneau du milieu). Ce traitement est généralement effectué dans un processeur de tissu situé dans un appropriately équipé laboratoire d'histologie. Un programme plus court est recommandé en raison de la taille et de la nature délicate des substituts. 24
5. Sectionnement et H & E Coloration (figure 1C, panneau de droite)
- Après le traitement des substituts à un bloc de paraffine, section de les utiliser un microtome standard pour sectionner des tissus, enrobés de paraffine fixés au formol 24,25.
Note: Ceci peut être réalisé dans un laboratoire d'histologie qualifié, ou dans un laboratoire de recherche, si elle est correctement équipé et expérimenté. L'épaisseur des coupes histologiques peut varier en fonction de l'utilisation prévue des sections; cependant, nous utilisons généralement des sections qui sont 5 um d'épaisseur. La mousse PDMS utilisé ici dans les substituts perfusés est facilement sectionnée avec un microtome. - Placez les sections sur des lames lisses histologiques de verre.
- Après la coupe, cuire les coupes histologiques à 58 ° C pendant 10-12 heures pour se préparer à la colorationavec de l'hématoxyline et de l'éosine (H & E). Faire cuire au four fait fondre la paraffine et permet également une meilleure adhérence des coupes à la lame de verre.
- Coloration H & E:
- Mettre en place les stations décrites dans le Tableau 2 dans des bocaux Coplin ou plats de coloration de verre, en fonction du nombre de diapositives à tacher. Une fois que les réactifs sont mis en place, déplacer les sections à travers chaque station, dans l' ordre, l' incubation pendant la durée indiquée ci - dessous 24.
- Monter une lamelle à chaque diapositive en utilisant les médias de montage.
- Laisser un support de montage sécher avant de l'imagerie.
| H & E Coloration | ||
| Gare | Solution | Temps |
| 1 | Xylène | 5 min |
| 2 | Xylène | 5 min |
| 3 | Xylène | 5 min |
| 4 | 100% d'éthanol | 5 min |
| 5 | 100% d'éthanol | 5 min |
| 6 | Éthanol à 95% | 5 min |
| 7 | Éthanol à 95% | 5 min |
| 8 | l'eau du robinet | 5 min |
| 9 | Eau déminéralisée | 5 min |
| dix | hématoxyline 7211 | 5 min |
| 11 | l'eau du robinet | 5 min |
| 12 | Clarificateur * | 10 trempettes |
| * Richard Allan # 7401 ou 70% d'éthanol + 0,5% HCl | ||
| 13 | l'eau du robinet | 5 min |
| 14 | bleuissant réactif | 30 sec |
| 15 | l'eau du robinet | 5 min |
| 16 | Éthanol à 95% | 10 trempettes |
| 17 | Éosine-Y | 1 minute |
| 18 | Éthanol à 95% | 10 trempettes |
| 19 | Éthanol à 95% | 10 trempettes |
| 20 | 100% d'éthanol | 10 trempettes |
| 21 | 100% d'éthanol | 10 trempettes |
| 22 | 100% d'éthanol | 5 min |
| 23 | Xylène | 10 trempettes |
| 24 | Xylène | 5 min |
Tableau 2. H & E Coloration.
6. Mesure de densité cellulaire
- Image au moins une section entière H & E tachée histologiques de la mère porteuse en utilisant la microscopie à fond clair 400X, l'enregistrement des images en fichiers .tif.
Remarque: Le traitement de l' image décrite a seulement été complété en utilisant des images en couleur. Bien que non testé, nous croyons que le même traitement devrait également être applicable à des images en niveaux de gris. - Télécharger CellProfiler du Broad Institute 26 (http://cellprofiler.org/download.shtml) et ImageJ des National Institutes of Health (http://rsb.info.nih.gov/ij/download.html), les deux qui sont publiquement disponibles sans frais.
- Pour mesurer la surface de la mère porteuse dans chaque image, ImageJ ouverte, sélectionnez "Définir mesures" (sous l'onglet "Analyser"), sélectionnez «Zone», puis sélectionnez «Ok & #34 ;.
- Ouvrez une image (fichier .tif) de la mère porteuse. Utilisation de l'outil de polygone dans ImageJ (voir la figure 2), contour de la zone de la mère porteuse dans l'image en faisant glisser la souris et en cliquant pour faire des points d'ancrage. Utiliser les bords de la MEC comme un guide. Une fois décrit, sélectionnez "Mesure" sous l'onglet "Analyser".
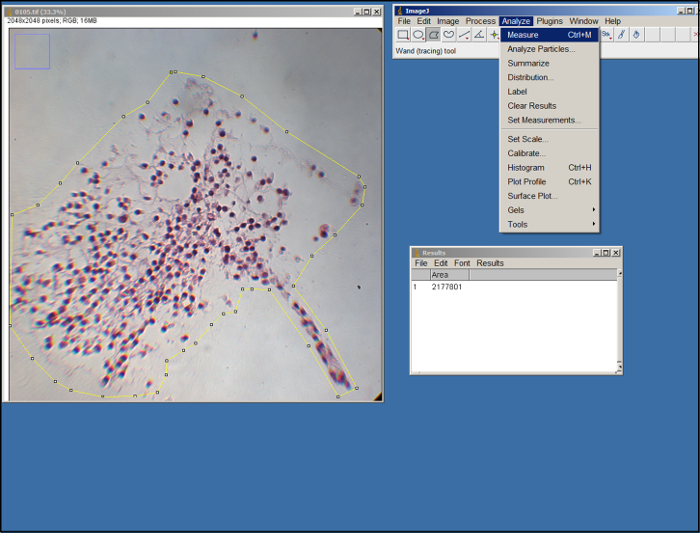
Figure 2. Analyse de ImageJ. Capture d'écran de traitement ImageJ. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
- Répétez l'opération pour chaque image de la mère porteuse des tissus. Enregistrer les mesures et les identificateurs d'image correspondant à un programme de feuille de calcul.
- Télécharger des fichiers d'image utilisés pour mesurer la zone en ImageJ dans CellProfiler en faisant glisser des fichiers image à la "Liste des fichiers". Attribuez un nom aux images importées dansle module et de saisie "NamesAndTypes" sélectionner le type d'image ( par exemple, «Image en couleur").
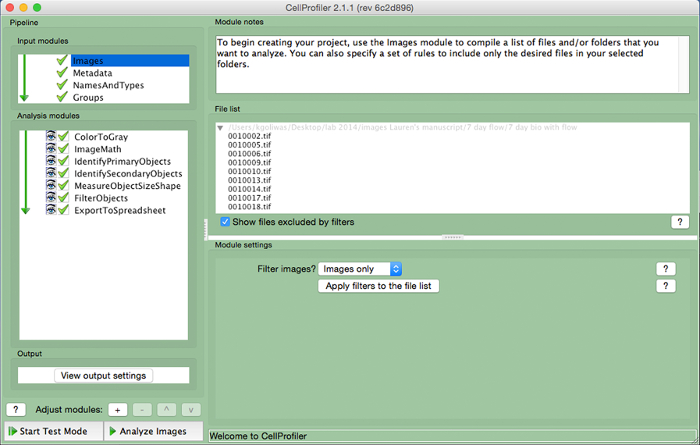
Figure 3. CellProfiler exemple pipeline. Capture d' écran du pipeline conçu pour mesurer le nombre de cellules nucléées dans CellProfiler. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
- Créer un pipeline d'analyse qui comprend les modules suivants pour calculer le nombre de cellules par image, en cliquant sur le signe "+" à côté de "Ajuster Module" (Figure 3, au bas du panneau de gauche). Ajouter chacun des modules ci-dessous.
- Sélectionnez "ColorToGray" (Figure 4).
- Sélectionnez le nom de l'image d'entrée à partir du menu déroulant, nommez l'image de sortie, et sélectionnez le type d'image d'origine ( ie, RVB si l' image couleur d' entrée).
Remarque: Ce module ne serait pas nécessaire si vous utilisez des images en niveaux de gris
- Sélectionnez le nom de l'image d'entrée à partir du menu déroulant, nommez l'image de sortie, et sélectionnez le type d'image d'origine ( ie, RVB si l' image couleur d' entrée).
- Sélectionnez "ImageMath" (Figure 5).
- Sélectionnez l'opération "Invert", nommez l'image de sortie, et sélectionnez l'image en niveaux de gris dans l'onglet "sélectionnez d'abord l'image".
- Sélectionnez "IdentifyPrimaryObjects" (figure 6).
- Sélectionner une image d'entrée (image après correction de maths), nommez l'objet principal d'être identifié (noyaux), et entrez la plage de diamètre pour les objets à mesurer en unités de pixels (environ 25 à 65). Sélectionnez "adaptative" stratégie de seuil avec la méthode de seuillage "Otsu" avec "trois classes". Ne pas modifier d'autres paramètres à partir des paramètres par défaut.
Remarque: La plage optimale des diamètres doit être déterminée par l' ouverture d' une image dans le module d'image d'entrée et en mesurant le diamètre des noyaux ( par exemple,l'objet principal) en utilisant l'outil de longueur de mesure.
- Sélectionner une image d'entrée (image après correction de maths), nommez l'objet principal d'être identifié (noyaux), et entrez la plage de diamètre pour les objets à mesurer en unités de pixels (environ 25 à 65). Sélectionnez "adaptative" stratégie de seuil avec la méthode de seuillage "Otsu" avec "trois classes". Ne pas modifier d'autres paramètres à partir des paramètres par défaut.
- Sélectionnez "IdentifySecondaryObjects" (figure 7).
- Sélectionnez l'image d'entrée (image après correction de mathématiques), sélectionnez les objets d'entrée (noyaux), et nommer les objets à identifier (cellules). Choisissez la méthode "de propagation" pour identifier les objets secondaires, utilisez la stratégie «adaptative» de seuil et la méthode "Otsu" avec "deux classes" minimisant "variance pondérée". Sélectionnez «sans lissage» et un facteur de correction de seuil de 1, bornes inférieures et supérieures de 0 et 1, et un facteur de régularisation de 0,05. Ne pas modifier d'autres paramètres à partir des paramètres par défaut.
- Sélectionnez "MeasureObjectSizeShape" (figure 8).
- Sélectionner les cellules (but secondaire) et les noyaux (objet primaire) comme des objets à mesurer.
- Sélectionnez "FilterObjects" (Figure 9 ).
- Nommez les objets de sortie et sélectionnez les noyaux (objet primaire) que l'objet à filtrer.
- Gardez les deux paramètres suivants selon les paramètres par défaut. Sélectionnez "AreaShape" comme la mesure de filtrer par catégorie, et "FormFactor" comme la mesure.
- Sélectionnez "Oui" pour filtrer en utilisant une valeur minimale de mesure et d'ajouter une valeur minimale de 0,52.
- Sélectionnez "Non" pour filtrer l'aide d'une mesure maximale.
- Sélectionnez "Oui" pour conserver les contours des objets filtrés et nommez l'image décrite.
- Sélectionnez "ExportToSpreadsheet" (Figure 10)
- Sélectionnez où enregistrer le fichier et le nom "Output Files".
- Après le pipeline d'analyse est générée (étapes 6.7.1 à 6.7.7.1), commencer "Mode Test" en CellProfiler et évaluer chaque étape, y compris les noyaux dans l'image de test sont filtrés de manière appropriée, pour assurer pa optimaleramètres ont été choisis pour identifier les cellules. La figure 11 montre une image de sortie de CellProfiler suivant filtrage, où les noyaux sont correctement identifiés (entourées en vert), et le fond est filtré.
- Une fois que les paramètres sont évalués et déterminés à être suffisante, enregistrez le projet, puis cliquez sur "l'analyse des images". Ce projet peut être utilisé à plusieurs reprises pour une analyse ultérieure.
Remarque: Une fois que les paramètres ont été établis, le programme est réglé pour analyser toutes les images dans la "Liste des fichiers", séquentiellement. Cela se traduira par plusieurs fenêtres d' ouverture de chaque image analysée, ce qui entraîne un temps de traitement plus long.- Pour éviter un temps de traitement plus, cliquez sur l'icône de l'œil sur tous les modules sauf "ExportToSpreadsheet" sous la section «modules d'analyse".
- Une fois que toutes les images de substitution ont été traitées par CellProfiler, ouvrez la feuille de calcul contenant les données d'image générées par Cell Profiler et la feuille contenant la zone mesurée avec ImageJ. Copiez les données filtrées noyaux (colonne D dans le tableur de CellProfiler) et les identificateurs d'image (colonne R) et les coller dans la feuille de calcul contenant les données de la zone de mesure.
- Calculer la somme de toutes les mesures obtenues pour le nombre de noyaux par image de la porteuse.
- Calculer la somme des mesures obtenues pour la zone de substitution de chaque image de la mère porteuse. Divisez la superficie totale mesurée par 1x10 6.
- Diviser le nombre total de noyaux par la mesure de la surface totale dans l'étape ci - dessus pour obtenir une valeur pour le nombre de cellules par 1x10 6 pixels 2.
- Sélectionnez "ColorToGray" (Figure 4).
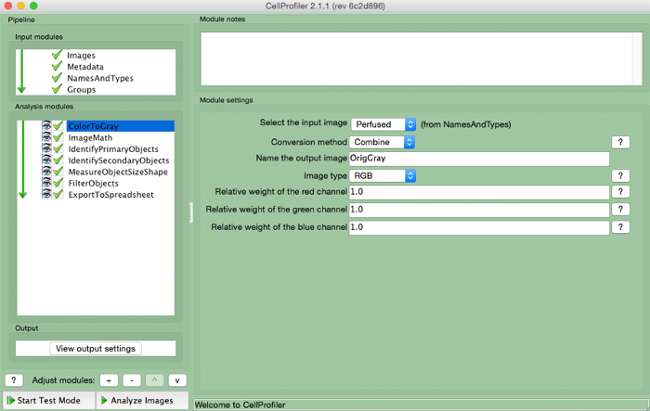
Figure pipeline 4. CellProfiler: changement d' image en niveaux de gris Capture de module "ColortoGray"..s: //www.jove.com/files/ftp_upload/54004/54004fig4highres.jpg "target =" _ blank "> S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
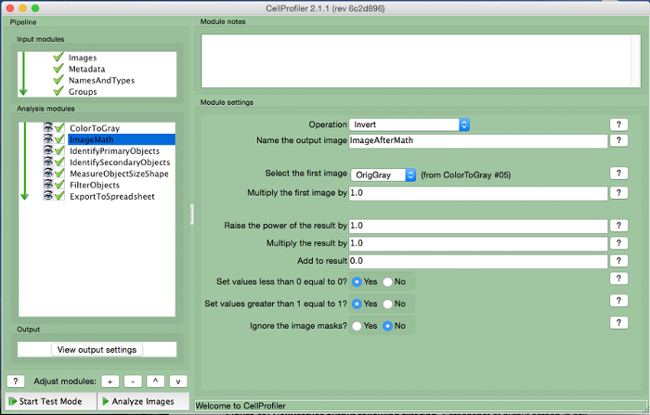
Figure 5. CellProfiler pipeline:.. L' image d' inversion Capture d'écran de module "ImageMath" S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.

Figure 6. CellProfiler pipeline:.. Noyaux d' identification Capture d' écran du module "IdentifyPrimaryObjects" S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
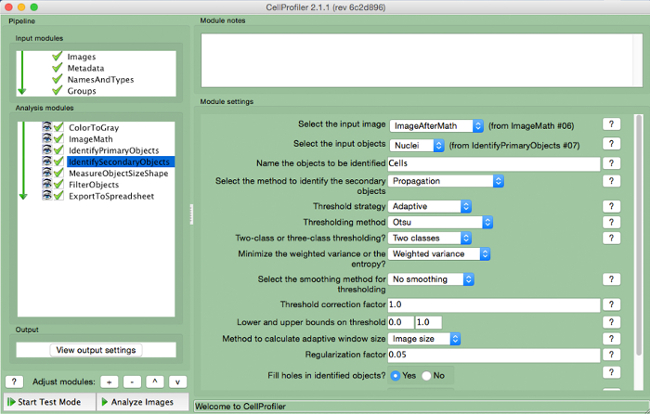
Figure 7. CellProfiler pipeline:.. Cellules d' identification Capture d'écran du module "IdentifySecondaryObjects" S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
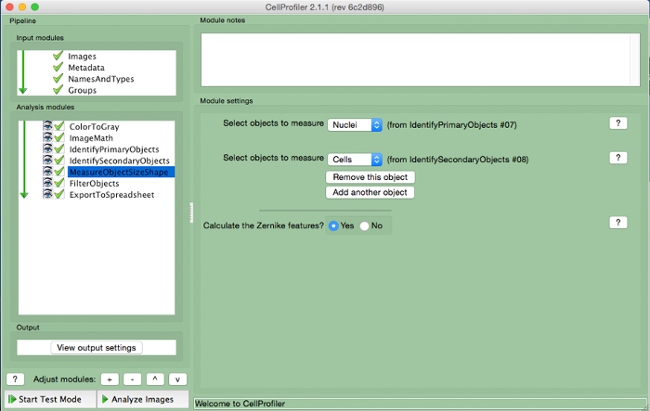
Figure 8. CellProfiler pipeline:.. Les objets de mesure Capture d' écran du module "MeasureObjectSizeShape" S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
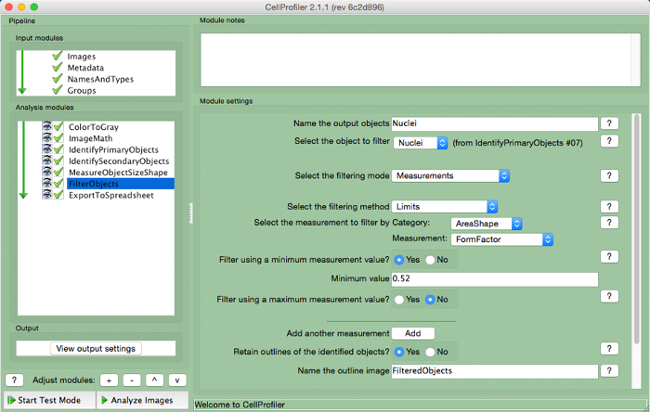
Figure 9. CellProfiler pipeline:. Filtrage des objets Capture d'écran de "FilterObjects "module. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
Figure 10. CellProfiler pipeline:.. L' exportation de données Capture d' écran du module "ExportToSpreadsheet" S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
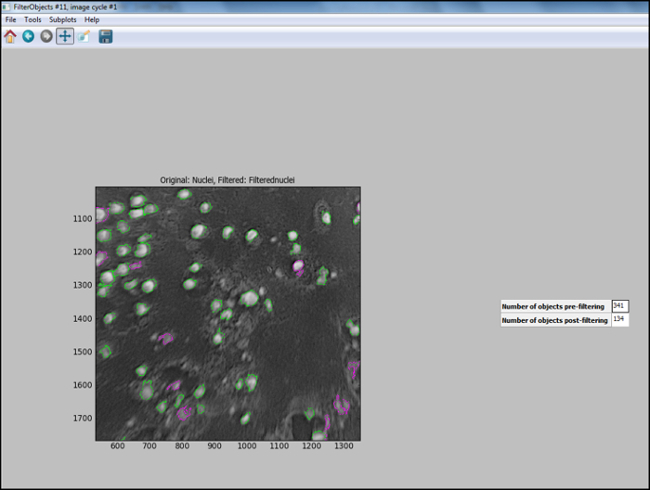
Figure 11. Sortie CellProfiler suivant filtrage. Capture d'écran de l' écran de sortie en profileur cellulaire après filtrage de l' objet. S'il vous plaît cliquer ici pour voir un plus grand versisur ce chiffre.
Résultats
Deux 3D mères porteuses de cancer du sein solides et perfusées ont été préparées comme décrit ci-dessus et cultivées pendant 7 jours. Par la suite, les mères porteuses sont fixées, traitées à la paraffine, sectionnées et colorées avec de l'hématoxyline et de l'éosine comme décrit ci-dessus. Le nombre de cellules nucléées par zone (les deux cellules 231 et CAF) de chaque porteuse a été mesurée. Comme on peut le voir sur la figure 12, photomic...
Discussion
Ici, un procédé de culture en 3D a été décrite qui incorpore les composants du micro-environnement tissulaire, y compris la matrice extracellulaire (ECM) et les fibroblastes du stroma de l'homme, dans un volume plus près des modèles de cancer du sein humain pour permettre le développement d'une morphologie en 3D récapitulatif . Le procédé de culture en 3D décrit est plus représentatif de la maladie chez l'homme que la culture cellulaire traditionnelle 2D en ce que plusieurs types de cellules son...
Déclarations de divulgation
The authors declare that they have no competing financial interests.
Remerciements
The University of Alabama at Birmingham Center for Metabolic Bone Disease performed the histologic processing and sectioning of surrogates. Southern Research (Birmingham, AL) provided support for the manufacture of the bioreactor system. Funding was provided by the United States Department of Defense Breast Cancer Research Program (BC121367).
matériels
| Name | Company | Catalog Number | Comments |
| Dulbecco's Modified Eagel Medium 1x (DMEM) | Corning CellGro | 10-014-CV | |
| Fetal Bovine Serum (FBS) | Atlanta Biologicals | S11150 | |
| 0.25% Trypsin + 2.21 mM EDTA 1x | Corning | 25-053-CI | |
| Tissue Culture plates, 100 mm | CellTreat Scientific Products | 229620 | Sterile |
| Tissue Culture plates, 35 mm | CellTreat Scientific Products | 229638 | For PDMS foam formation |
| 9" Glass pipette | Fisher | 13-678-20D | Sterile |
| 10 ml pipette | CellTreat Scientific Products | 229210B | Sterile |
| 1,000 µl piptette tips | FisherBrand | 02-717-166 | Sterile Filtered |
| 200 µl pipette tips | FisherBrand | 02-717-141 | Sterile Filtered |
| 10 µl pipette tips | FisherBrand | 02-717-158 | Sterile Filtered |
| 15 ml conical tubes | CellTreat Scientific Products | 229410 | Sterile |
| 50 ml conical tubes | CellTreat Scientific Products | 229422 | Sterile |
| 1.5 ml microcentrifuge tubes | FisherBrand | 05-408-129 | Sterile |
| Trypan blue | Corning Cellgro | 25-900-CI | Sterile |
| Sylgard 184 | Electron Microscopy Sciences | 24236-10 | PDMS elastomer and curing agent. Used for our in-house bioreactor. |
| PDMS Foam | Made in-house for use in our in-house bioreactor. | ||
| High Concentration Bovine Collagen Type I | Advanced Biomatrix | 5133-A | FibriCol ~10 mg/ml |
| Growth Factor Reduced Matrigel (Basement Membrane) | Corning | 354230 | Basement membrane material |
| Sodium Bicarbonate | Sigma | S8761 | |
| Molecular Biology Grade Water | Fisher | BP2819-1 | |
| DMEM 10x | Sigma-Aldrich | D2429 | |
| Nunc Lab-Tek Chamber Slide System | Thermo Scientific | 177402 | 8-well |
| Bioreactor | Made in-house. | ||
| Spring-Back 304 Stainless Steel—Coated with PTFE polymer | McMaster-Carr | 1749T19 | Stainless steel wires to generate microchannels in our in-house bioreactor system. 0.016" Diameter |
| BioPharm Plus platinum-cured silicone pump tubing, L/S 14 | Masterflex | EW-96440-14 | For use in our in-house bioreactor system. Tubing ID: 1.6 mm, Hose barb size: 1/16 in. |
| 2-Stop Tubing Sets, non-flared PVC, 1.52 mm ID | Cole-Parmer | EW-74906-36 | For use in our in-house bioreactor system (with microperistalitic pump). |
| Six Channel precision micro peristaltic pump | Cole-Parmer | EW-74906-04 | For use with our in-house bioreactor system |
| Tuberculin Syringes | BD Medical | 309625 | 26 gauge 3/8 in. needle; Sterile |
| Dissecting Tissue Forceps | FisherBrand | 13-812-36 | 5.5 inch |
| Mini Tube Rotator | Boekel Scientific | 260750 | Equipment option for surrogate rotation. Used with carousel for 50 ml tubes (model number 260753) |
| 50 ml tube carousel | Boekel Scientific | 260753 | Used with mini tube rotator |
| Bambino Hybridization Oven | Boekel Scientific | 230301 | Equipment option for surrogate rotation |
| HistoGel Specimen Processing Gel | Thermo Scientific | HG-4000-012 | Specimen Processing Gel described in Step 5.2 |
| Cryomold | Andwin Scientific | 4566 | 15 mm x 15 mm x 5 mm |
| Tissue Marking Dye | Cancer Diagnostics, inc. | 03000P | Can be used to mark surrogates, allowing multiple samples to be included in one tissue cassette |
| Hinged tissue cassettes | FisherBrand | 22-272-416 | |
| Formalin | Fisher | 23-245-685 | |
| GoldSeal Plain Glass Slides | Thermo Scientific | 3048-002 | |
| Xylene | Fisher | X3P-1GAL | |
| Ethanol, 200 proof (100%), USP | Decon Laboratories, Inc. | 2805M | |
| Hematoxylin | Thermo Scientific Richard-Allan Scientific | 7211 | |
| Clarifier | Thermo Scientific Richard-Allan Scientific | 7401 | |
| Bluing Solution | Thermo Scientific Richard-Allan Scientific | 7301 | |
| Eosin Y | Thermo Scientific Richard-Allan Scientific | 7111 | |
| Cytoseal XYL mounting media | Thermo Scientific Richard-Allan Scientific | 83124 | |
| Coverslips | Fisher Scientific | 12-548-5G |
Références
- Hakanson, M., Textor, M., Charnley, M. Engineered 3D environments to elucidate the effect of environmental parameters on drug response in cancer. Integr Biol (Camb). 3 (1), 31-38 (2011).
- Horning, J. L., et al. 3-D tumor model for in vitro evaluation of anticancer drugs. Mol Pharm. 5 (5), 849-862 (2008).
- Dhiman, H. K., Ray, A. R., Panda, A. K. Three-dimensional chitosan scaffold-based MCF-7 cell culture for the determination of the cytotoxicity of tamoxifen. Biomaterials. 26 (9), 979-986 (2005).
- Place, A. E., Jin Huh, S., Polyak, K. The microenvironment in breast cancer progression: biology and implications for treatment. Breast Cancer Res. 13 (6), 227 (2011).
- Mao, Y., Keller, E. T., Garfield, D. H., Shen, K., Wang, J. Stromal cells in tumor microenvironment and breast cancer. Cancer Metastasis Rev. 32 (1-2), 303-315 (2013).
- Paulsson, J., Micke, P. Prognostic relevance of cancer-associated fibroblasts in human cancer. Semin Cancer Biol. 25, 61-68 (2014).
- Roskelley, C. D., Desprez, P. Y., Bissell, M. J. Extracellular matrix-dependent tissue-specific gene expression in mammary epithelial cells requires both physical and biochemical signal transduction. Proc. Natl. Acad. Sci. 91, 12378-12382 (1994).
- Pickl, M., Ries, C. H. Comparison of 3D and 2D tumor models reveals enhanced HER2 activation in 3D associated with an increased response to trastuzumab. Oncogene. 28 (3), 461-468 (2008).
- Ivascu, A., Kubbies, M. Rapid generation of single-tumor spheroids for high-throughput cell function and toxicity analysis. J Biomol Screen. 11 (8), 922-932 (2006).
- Lovitt, C. J., Shelper, T. B., Avery, V. M. Advanced cell culture techniques for cancer drug discovery. Biology (Basel). 3 (2), 345-367 (2014).
- Bergamaschi, A., et al. Extracellular matrix signature identifies breast cancer subgroups with different clinical outcome. J Pathol. 214 (3), 357-367 (2008).
- Oskarsson, T. Extracellular matrix components in breast cancer progression and metastasis. The Breast. 22, S66-S72 (2013).
- Kelley, L. C., Lohmer, L. L., Hagedorn, E. J., Sherwood, D. R. Traversing the basement membrane in vivo: A diversity of strategies. JBC. 204 (3), 291-301 (2014).
- Sadlonova, A., et al. Breast fibroblasts modulate epithelial cell proliferation in three-dimensional in vitro co-culture. Breast Cancer Res. 4, (2004).
- Wendt, D., Marsano, A., Jakob, M., Heberer, M., Martin, I. Oscillating perfusion of cell suspensions through three-dimensional scaffolds enhances cell seeding efficiency and uniformity. Biotechnol Bioeng. 84 (2), 205-214 (2003).
- Marshall, L. E., et al. Flow-perfusion bioreactor system for engineered breast cancer surrogates to be used in preclinical testing. J Tissue Eng Regen Med. , (2015).
- Calcagnile, P., Fragouli, D., Mele, E., Ruffilli, R., Athanassiou, A. Polymeric foams with functional nanocomposite cells. RSC Adv. 4, 19177-19182 (2014).
- Naba, A., Clauser, K. R., Lamar, J. M., Carr, S. A., Hynes, R. O. Extracellular matrix signatures of human mammary carcinoma identify novel metastasis promoters. eLife. 3, (2014).
- Lochter, A., Bissell, M. J. Involvement of extracellular matrix constituents in breast cancer. Semin Cancer Biol. 6 (3), 165-173 (1995).
- Joiner, K. S., Spangler, E. A. Evaluation of HistoGel-embedded specimens for use in veterinary diagnostic pathology. J Vet Diagn Invest. 24 (4), 710-715 (2012).
- Varsegi, G. M., Shidham, V. Cell Block Preparation from Cytology Specimen with Predominance of Individually Scattered Cells. J Vis Exp. (29), e1316 (2009).
- Sadlonova, A., et al. Human Breast Fibroblasts Inhibit Growth of the MCF10AT Xenograft Model of Proliferative Breast Disease. Am J Pathol. 170 (3), (2007).
- Otali, D., He, Q., Stockard, C. R., Grizzle, W. E. Preservation of immunorecognition by transferring cells from 10% neutral buffered formalin to 70% ethanol. Biotech Histochem. 88, 170-180 (2013).
- Webster, S. S., Jenkins, L., Burg, K. J. L. Histological Techniques for Porous, Absorbable, Polymeric Scaffolds, Used in Tissue Engineering. J Histotechnol. 26 (1), 57-65 (2003).
- Troy, T. -. C., Arabzadeh, A., Enikanolaiye, A., Lariviere, N., Turksen, K. Immunohistochemistry on Paraffin Sections of Mouse Epidermis Using Fluorescent Antibodies. J Vis Exp. (11), (2008).
- Carpenter, A. E., et al. CellProfiler: image analysis software for identifying and quantifying cell phenotypes. Genome Biol. 7 (10), R100 (2006).
- Kwon, Y. -. J., et al. Gli1 enhances migration and invasion via up-regulation of MMP-11 and promotes metastasis in ERa negative breast cancer cell lines. Clin Exp Metastasis. (28), (2011).
- Evilsizor, M. N., Ray-Jones, H. F., Lifshitz, J., Ziebell, J. Primer for Immunohistochemistry on Cryosectioned Rat Brain Tissue: Example Staining for Microglia and Neurons. J Vis Exp. (99), e52293 (2015).
- Pal, A., Kleer, C. G. Three dimensional cultures: a tool to study normal acinar architecture vs. malignant transformation of breast cells. J Vis Exp. (86), e51311 (2014).
- Hasselbach, L. A., et al. Optimization of High Grade Glioma Cell Culture from Surgical Specimens for Use in Clinically Relevant Animal Models and 3D Immunochemistry. J Vis Exp. (83), e51088 (2014).
- Foty, R. A Simple Hanging Drop Cell Culture Protocol for Generation of 3D Spheroids. J Vis Exp. (51), e2720 (2011).
- Materne, E. -. M., et al. The Multi-organ Chip - A Microfluidic Platform for Long-term Multi-tissue Coculture. J Vis Exp. (98), e52526 (2015).
- Sadlonova, A., et al. Identification of Molecular Distinctions Between Normal Breast-Associated Fibroblasts and Breast Cancer-Associated Fibroblasts. Cancer Microenviron. 2, 9-21 (2009).
- Wang, J. D., Douville, N. J., Takayama, S., ElSayed, M. Quantitative analysis of molecular absorption into PDMS microfluidic channels. Ann Biomed Eng. 40 (9), 1862-1873 (2012).
- Halldorsson, S., Lucumi, E., Gomez-Sjoberg, R., Fleming, R. M. Advantages and challenges of microfluidic cell culture in polydimethylsiloxane devices. Biosens Bioelectron. 63, 218-231 (2015).
- Regehr, K. J., et al. Biological implications of polydimethylsiloxane-based microfluidic cell culture. Lab Chip. 9 (15), 2132-2139 (2009).
- Burdett, E., Kasper, F. K., Mikos, A. G., Ludwig, J. A. Engineering tumors: a tissue engineering perspective in cancer biology. Tissue Eng Part B Rev. 16 (3), 351-359 (2010).
- Caruso, R. A., et al. Mechanisms of coagulative necrosis in malignant epithelial tumors (Review). Oncol Lett. 8 (4), 1397-1402 (2014).
- Elmore, S. Apoptosis: A Review of Programmed Cell Death. Toxicol Pathol. 35 (4), 495-516 (2007).
- Majno, G., Joris, I. Apoptosis, oncosis, and necrosis. An overview of cell death. Am J Pathol. 146 (1), 3-15 (1995).
- Ogino, S., et al. Molecular pathological epidemiology of epigenetics: emerging integrative science to analyze environment, host, and disease. Mod Pathol. 26 (4), 465-484 (2013).
- Otali, D., et al. Combined effects of formalin fixation and tissue processing on immunorecognition. Biotech Histochem. 84 (5), 223-247 (2009).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon