このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
の調製と分析
要約
We demonstrate a method to generate 3D breast cancer surrogates, which can be cultured using a perfusion bioreactor system to deliver oxygen and nutrients. Following growth, surrogates are fixed and processed to paraffin for evaluation of parameters of interest. The evaluation of one such parameter, cell density, is explained.
要約
三次元(3D)培養は、二次元培養物よりもin vitroでの細胞の挙動をモデル化するために、より生理学的に関連する方法です。乳癌を含む癌は、線維芽細胞および細胞外マトリックス(ECM)を含む、がん上皮細胞および間質成分からなる複雑な3次元組織です。しかし、乳癌のほとんどのin vitroモデルは、したがって、間質を省略し、唯一の癌上皮細胞からなり、そして、in vivoでの腫瘍の3Dアーキテクチャ。癌の適切な3Dモデリングは、腫瘍生物学、行動、および治療に対する反応の正確な理解のために重要です。しかし、3Dモデルの文化とボリュームの持続時間は、培養中の酸素や栄養素の利用可能性によって制限されます。ここで、我々は、乳癌上皮細胞及び間質線維芽細胞は、間質を含むとして培養することができる3D乳癌サロゲートを生成するECMに組み込まれた方法を示します固体の3D構造または酸素および栄養素を送達するための灌流バイオリアクターシステムを使用することによって。セットアップおよび初期成長期に続いて、サロゲートは、前臨床薬物試験のために使用することができます。代替的に、代理の細胞およびマトリックスの構成要素は、生物学的問題の様々な対処するために変更することができます。培養後、サロゲートを固定し、目的のパラメータの評価のために、臨床的乳癌試料の処理と同様に、パラフィンに処理されます。 ImageJのとCellProfiler画像解析ソフトウェアシステムは、サロゲートの組織切片の顕微鏡写真に適用され、説明されているそのようなパラメータ、存在する細胞の密度の評価は、面積当たりの有核細胞の数を定量します。これは、時間の経過とともに細胞数の変化または変動する成長条件および治療から得られた細胞数の変化の指標として使用することができます。
概要
より正確にインビボでの腫瘍アーキテクチャおよび微小環境を模倣する三次元(3D)培養モデルは、細胞とその微小環境との間の複雑な相互作用を分析するために候補治療法の有効性を試験することを目的とした研究のために重要です。腫瘍次元への影響の酸素と栄養勾配、薬物暴露、間質圧/血液の流れ、および3Dアーキテクチャ1-4の均一性。適切な間質微小環境の存在は、腫瘍の次元と影響細胞のECMシグナリングおよびストローマ細胞と悪性上皮細胞間の傍分泌シグナル伝達に貢献しています。細胞機能に対する腫瘍の次元と微小環境の影響はよく両方の要因が、薬物応答1,3,5-8を変更することで、確立されています。さらに、細胞増殖動態、代謝率、および細胞シグナル伝達は、これらの要因がaffeで、3Dで2次元(2D)文化と文化の間で異なります細胞応答1,3,8-10を cting。
インビトロでは 、腫瘍微小環境代理代表ECM成分および間質細胞の集団を含むことによって調節することができます。悪性上皮細胞はさらに、腫瘍の伝播5,6,10を阻害するためにECMおよび癌関連間質細胞のいずれかによって腫瘍の進行を促進するための相乗/保護方法で、または抑制の方法で影響を受けています。いずれかの文脈では、基質は、薬物送達1,6を減少パラ分泌シグナル伝達を介して、および/ または、その結果、腫瘍内の間質圧力を増加させることによって、治療応答および薬物送達に影響を与えることができます。したがって、前臨床モデルへのECMとの間質細胞の添加は、2D培養で十分にモデル化することができない腫瘍の特徴を再現するのに役立ちます。
ECM成分とsを含む概括的微小環境を組み込む乳癌サロゲートを確立するために、本明細書の方法3D体積tromal細胞は、記載されています。乳癌では、間質細胞集団は主に癌関連線維芽細胞(CAF)および間質ECMから構成されているが、主としてラミニンおよびコラーゲンIV型を含む基底膜に見出されるマトリックス成分のより少ない割合でI型コラーゲンで構成されています1,4,11-13。したがって、乳癌の微小環境( すなわち、CAF、コラーゲンI、および基底膜)のこれらの構成要素は、サロゲートに組み込まれています。この方法は、固体の、非灌流3D代用物( 図1A)を生成するために使用することができ、またはバイオリアクターシステム( 図1B)を介してサロゲートを通して培地の灌流を含むように適合させることができます。両方のアプローチは、ここで説明されています。この方法はまた、腫瘍関連マクロファージのような他の間質要素を含むように改変され得る、または必要に応じて、細胞とECMの成分を調整することにより、他の固形腫瘍をモデル化します。
我々は、MDA-MB-231を利用している(231)乳癌細胞株、CAFは、以前にヒト乳癌14、及びIは、(90%のコラーゲンからなるECMから単離されました6 mg / mlで)および10%の増殖因子低減基底膜材料(BM)。サロゲートは、いずれか8ウェルチャンバースライド(固体サロゲート)に成長させるか、バイオリアクターシステムは、連続的栄養かん流(灌流サロゲート)を提供するために利用されます。 ECMを含有する細胞の量を収容することができる任意の灌流バイオリアクターシステム15を使用することができます。例として、私たちは私たちのバイオリアクターシステムにおける組織サロゲートの調製を記載します。このシステムは、社内で開発され、市販されていないました。ここで我々の焦点は、3D組織代替物の調製および分析上にあるので、私たちは私たちのバイオリアクターシステムの製造及び組み立ての仕様に関する広範な詳細に行っていません。しかしながら、詳細な説明このシステムとその開発は16を発表してきました。このバイオリアクターシステムにおいては、ポリジメチルシロキサン(PDMS)流路は、PDMSフォームでサポートされているサロゲートを収容するために使用される(Calcagnile ら 17によって記載されたものと同様の方法を用いて形成されます)。このボリュームは、連続的に代理に酸素と栄養を供給するためにmicrophysiologicポンプを介して媒体によって灌流されている4マイクロチャンネル(直径各400ミクロン)によって貫通されています。サロゲートの適切な分析は、治療または他の操作に応答した細胞機能に関する適切な情報を得るために重要です。サロゲートは、固定および処理後の分泌物、または組織切片上での分析のために、共焦点顕微鏡または非侵襲的イメージングの他の手段、馴化培地、または灌流液をアッセイすることによって間接的細胞解析を用いて、無傷のサロゲートの直接画像を含む様々な方法によって分析することができますにパラフィン。組織切片で評価することができるそのようなパラメータは、セル密度です。我々は、ヘマトキシリンおよびエオシン(H&E)で染色されたサロゲート組織切片の顕微鏡写真に適用される半自動画像処理技術を用いて細胞密度( すなわち、断面積当たりの有核細胞数)を測定するための一つのアプローチを提示します。細胞密度は、時間の経過または様々な増殖条件および処理の結果は、細胞数の相対的な変化の指標として用いることができます。
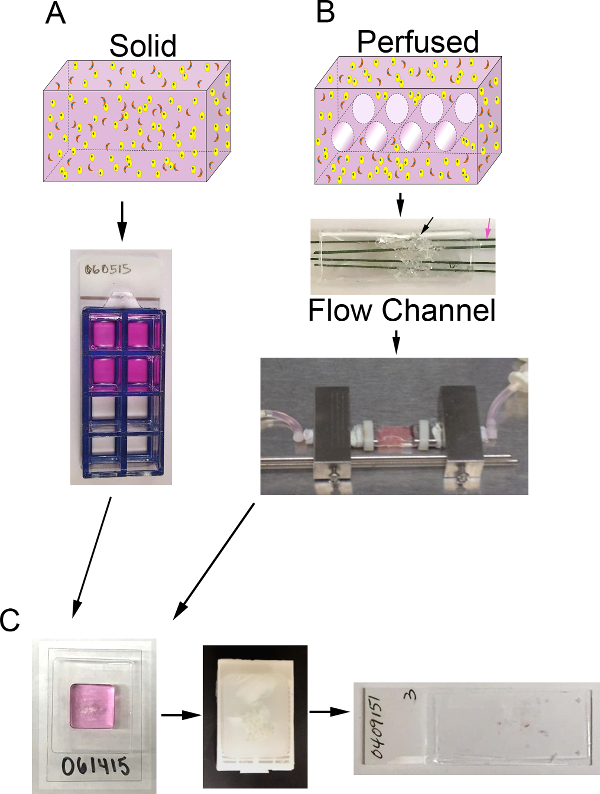
図1の3次元ボリュームとバイオリアクターシステム。 3Dソリッドサロゲートを生成するためのプロセスのA)概略図。トップ:ECMを含む固体3Dボリュームの漫画(ピンク)、上皮癌細胞(黄色)、およびCAF(オレンジ)。下:サロゲートを含む8ウェルチャンバースライドの上面図B)概略工程の灌流3Dのサロゲートを生成します。トップ:CA培地灌流およびECMを含む(ピンク)、上皮癌細胞(黄色)、およびCAF(オレンジ色)を可能にするためのチャネルを有する3Dボリュームのrtoon。中央:PDMSフォーム(黒矢印)を含むPDMS流路の画像は、細胞+ ECMを注入し、直径400μmの測定ポリマー被覆ステンレス鋼線(ピンク色矢印)によって貫通されます。底:サロゲートを含有する連続媒体灌流を可能にするバイオリアクターシステムに接続されたPDMS流路の画像(蠕動ポンプと媒体リザーバ図示せず)とC)培養後、固体及び灌流サロゲート両方の処理ステップの写真。左:ゲルとサロゲートを処理する検体を含むcryomoldの画像。ミドル:固定され、処理サロゲートを含むパラフィンブロックの画像。右:サロゲートのH&E染色した組織学的セクションとスライドガラスの画像表示するには、こちらをクリックしてください。この図の拡大版。
プロトコル
1.細胞培養
- 氷上、4℃で一晩BM成分を解凍。
- 37℃に温培地。両方の231細胞およびCAFの成長を支持するために、ダルベッコ改変イーグル培地(DMEM)+ 10%ウシ胎児血清(FBS)を使用します。
注: 使用される媒体は、細胞の種類および実験目的に依存するであろう。 - 近くコンフルエント231細胞の培養皿(10センチメートル)から培地を除去し、1.5ミリリットルのトリプシン/ EDTAを追加します。細胞分離のための監視、37℃で1~3分間インキュベートします。細胞がプレートを丸くし、オフに来て開始すると、血清を含む培地の3ミリリットルを加えて反応を停止します。 15ミリリットルコニカルチューブに培地および細胞をピペット。
- 5分間、150×gで培地および細胞を遠心分離します。上清を除去し、2 mlの培地中で細胞ペレットを再懸濁します。
- トリパンブルーおよび血球計数器を使用して、ボリューム当たりの細胞数をカウントします。細胞生存率は90%以上でなければなりません。
- 繰り返し他の細胞タイプのプロセスは、サロゲートに含まれます。ここで説明するサロゲートの場合、プロセスは、CAFを繰り返します。
- 所望の細胞数を得るために、各細胞懸濁液の適切な量を決定します。
注:1の線維芽細胞への上皮細胞の割合(1.4×10 6 231細胞を、100μlの あたり 7×10 5 CAF:2と ここで説明する代理、ECM100μl のあたり 2.1×10 6 細胞 の細胞密度について は、 ECM)が使用されます。この細胞密度は良い出発点です。しかしながら、最適な細胞密度は、細胞型、培養の時間、実験の目的に依存するであろう。 - 15ミリリットルコニカルチューブ(各細胞型のための1つのチューブ)に各細胞懸濁液の適切な量を置きます。 5分間、150×gで培地および細胞を遠心分離します。
- 遠心分離後、上清を除去し、再懸濁つの細胞型の細胞で培養グレードの水(178.8μL、 表1を参照)、10倍のDMEM中の他の細胞型(100μL、pH値を監視するために、フェノールレッドを含む、 表1を参照)。氷の上に細胞を含むチューブを置き、以下ECMの準備に迅速に進んでください。細胞は生存能力を維持するために水の中に残る時間を制限します。
注:ECMの両方の10×DMEM細胞培養グレードの水が必要とされるコンポーネント。したがって、我々は両方の中に再懸濁細胞を選択しました。いずれかの細胞型は、これらの2つの成分のいずれかに再懸濁することができるが、ここでは任意に、10倍DMEM中で細胞培養グレードの水とCAFで再懸濁231細胞に選びました。
ECM中の細胞の調製(6 mg / mlでウシI型コラーゲン+ 10%BM)
注意:90%のコラーゲンで構成ECMは、私はこの悪性腫瘍における腫瘍間質は、主にコラーゲンで構成されているので、10%BMは、浸潤性乳癌をモデル化するために選ばれました+ECM 12,13,18,19の小さい部分を含むようにラミニン、コラーゲンIV、およびエンタクチンなどBMの構成要素、と私。
- 氷上で、2mLの微小遠心管に、順番に、 表1に列挙された成分を加えます。
注: この量は8固体サロゲートまたは、ここで説明するバイオリアクターシステムを使用して、4灌流サロゲートのために十分です 。
| ECM内の細胞の調製 (6 mg / mlでウシI型コラーゲン+ 10%BM) | |
| 178.8μリットル | 231細胞(上記決定される)所望の数を含む細胞培養グレードの水 |
| 606μlの | コラーゲンは、I(10 mg / mlでウシ)、滴ずつを追加します |
| 100μlの | 解凍した基底膜、 |
| 100μlの | (上記で決定)CAFの所望の数の10倍DMEM(フェノールレッドを含みます) |
| 15.2μlの | 7.5%(v / v)の重炭酸ナトリウム、滴ずつ加えます |
ECM内のセルの表1.準備。
- 静かに気泡の形成を回避すること、ピペッティングにより混合します。 10倍DMEM中でフェノールレッドを使用してpHレベルを監視します。混合物は〜7のpHを示すオレンジ/ピンク色であることを確認してください。 pHが(余りに黄色)が低すぎる場合は、適切な色に到達するまで、ゆっくりと時間(〜5μL)でさらに7.5%重炭酸ナトリウム一滴を加えます。
- 氷の上で混合物を維持し、早期のECMの重合を防止するために、迅速に作業。
3.代理準備
- 固体3D培養( 図1A)の場合:
- 無菌技術を用いて細胞培養フードでの作業、サロゲート内の任意の実験変動を示すために、無菌の8ウェルチャンバースライドの蓋にラベルを付けます。
- ゆっくり8ウェルチャンバースライドの各ウェルに細胞+ ECM混合物の100μLをピペットで、早期のECMの重合を防止するために氷上でチャンバースライドを維持します。
注:よく最初のエッジの周りの細胞+ ECM混合物をピペッティングすることは、より良いウェル内の細胞+ ECM混合物を分配するのに役立ちます。 - ECMの重合を可能にするために、45分間、37℃、5%CO 2でサロゲートをインキュベートします。
- 、ECMの重合に続いて、各ウェルに100μlの培地を追加し、実験期間中、37℃、5%CO 2でインキュベート2日毎に培地を変えます。
- バイオリアクターシステム( 図1B)で灌流し、3D培養の場合:
- 3D培養のセットアップのためのすべてのバイオリアクターの成分を滅菌する( すなわち、バイオリアクターの設定に必要な生物反応器、チューブ、鉗子、および継手)バイオリアクターのための特定のプロセスを使用して、使用します。
- ここでの利用例のバイオリアクターシステムの場合は、( 例えば 、12分、15分乾燥し121.1°Cでの露光)とは、1時間、70%エタノール中でインキュベートオートクレーブ処理を組み合わせて使用します。
- サロゲートを収容するバイオリアクターシステムの一部を準備し、組み立てます。
- ここで使用される例のバイオリアクターシステムの場合は、ピンセットを用いてチューブのPDMS流路にPDMSフォームバックボーンを挿入します。並列マイクロチャネルを生成するために、PDMSフォームに4(400ミクロン)ポリマー被覆ステンレス鋼線を押してください。
- 細胞培養フード中で、sterilを使用電子技術とシリンジと26ゲージ針は、細胞を含むように設計された灌流バイオリアクターの領域への細胞+ ECM混合物を注入します。次のステップに迅速に進んでください。
- ECMを可能にするために45分間37℃でインキュベートしながら、代用内の細胞のより均一な分布を確実にするために、(細胞培養フード中)50mlコニカルチューブにサロゲートを収容するバイオリアクターのコンポーネントを配置し、連続〜18 rpmで回転させます重合。
注記: 回転は、オーブン37℃に設定したハイブリダイゼーションのように、回転子に組み込まれてインキュベータまたはオーブン内で回転体を用いて完成させることができます。 - 製造元の指示を使用して、灌流ポンプにサロゲートを含むバイオリアクターアセンブリを接続します。
注意: このプロセスの詳細は、使用されるバイオリアクターおよびポンプに依存して変化します。- ここで使用される例示的なバイオリアクターシステムの場合、bioreaを接続する前にステンレス鋼線を除去しますポンプへのCTORアセンブリ。
- 37℃、5%CO 2のインキュベーター中で、培地灌流(1ダイン/ cm 2のマイクロチャネル壁せん断応力1,671μL/分のバルク流速)を起動。
注: メディア灌流の速度を調整することができ、バイオリアクターのセットアップは目標および実験の設計に依存。 - 7日ごとに培地を変更し、実験期間中の媒体灌流を続けます。
- 3D培養のセットアップのためのすべてのバイオリアクターの成分を滅菌する( すなわち、バイオリアクターの設定に必要な生物反応器、チューブ、鉗子、および継手)バイオリアクターのための特定のプロセスを使用して、使用します。
4.代理固定および処理(図1C)
- ラベルcryomoldsと代理固定および処理のためのプラスチック製組織カセット。
- 次に、暖かい温度で液体であるが、室温で固化する水性材料である検体処理ゲル、で包むサロゲート。処理中に無傷のサロゲートを保ち、14,20-22を切片の組織学的を促進における検体処理ゲルを助けます。
- 使用する準備ができるまでこの温度で維持し、それを液化するために60℃の水浴中で検体処理ゲルを溶かします。バイオセーフティキャビネットに代理でバイオリアクターを移動します。
- ラベルされたcryomold( 図1C、左パネル)の底部にゲルを処理するピペットは約300μlの標本。
- メスの刃(第10号推奨)と鉗子を使用すると、慎重にバイオリアクターからまたは8ウェルチャンバースライドのウェルからサロゲートを削除し、ゲルを処理する検体を含むcryomoldにそれを置きます。
注: 異なる色の 色素を組織マーキングは、( 特定の例のための材料/機器リストを参照) サロゲートをマークするために使用することができ、それによって複数の試料を識別可能な方法で1つの組織カセットに含めることを可能にします。 - cryomoldにサロゲートをカバーし、solidifに4℃で30分間インキュベートするピペット約300μlの検体処理ゲルY。
- 検体処理ゲルが固化した後、cryomoldからサロゲートを含む検体処理ゲルを除去し、組織カセットにそれを置きます。
- 完全な固定を可能にするために、室温で10〜12時間、10%中性緩衝ホルマリンにサロゲートを含む組織カセットを置きます。
- 固定後、パラフィンに処理するまで70%エタノールを代用を含む組織カセットを移動させます。
注: エタノールにホルマリンからサ ロゲートを転送するには、いくつかのエピトープ23の免疫反応性の損失を引き起こす可能性がホルマリン過固定防ぎます。エタノール中の時間の長さは重要ではありません。サロゲートは、免疫組織化学または免疫蛍光のために使用される場合、固定剤のこの変化は重要です。固定代理は今( 図1C、中央のパネル)をパラフィンする処理の準備ができています。この処理は、典型的には、APPRに位置する組織のプロセッサで実行されますopriately組織学の研究室を装備。短いプログラムは、サイズとサロゲートのデリケートな性質のために推奨されます。24
5.セクショニングとH&E染色(図1C、右パネル)
- パラフィンブロックにサロゲートの処理に続いて、部分それらは、ホルマリン固定、パラフィン包埋組織切片24,25のための標準的なミクロトームを使用して。
注意: 適切に装備され、経験豊富な場合、これは、資格のある組織学の研究室で、または研究室で行うことができます。組織切片の厚さは、セクションの意図される用途に応じて変えることができます。しかし、我々は、典型的には厚さ5μmのあるセクションを使用します。灌流サロゲートにここで使用されるPDMSフォームは簡単にミクロトームで区分されています。 - プレーンなガラス組織学的スライド上にセクションを配置します。
- 切片化した後、染色の準備のために10から12時間58℃で組織切片を焼きますヘマトキシリンおよびエオシン(H&E)です。ベーキングは、パラフィンを溶融し、ガラススライドにセクションの優れた接着を可能にします。
- H&E染色:
- 染色するスライドの数に応じて、コプリンジャーまたはガラス染色皿の表2に記載のステーションをセットアップします。試薬が設定されると、24以下に示す時間インキュベートし、順番に、それぞれの局を介してのセクションを移動します。
- マウントメディアを使用して、各スライドにカバースリップをマウントします。
- 取り付けメディアが撮影前に乾燥することができます。
| H&E染色 | ||
| 駅 | 溶液 | 時間 |
| 1 | キシレン | 5分 |
| 2 | キシレン | 5分 |
| 3 | キシレン | 5分 |
| 4 | 100%エタノール | 5分 |
| 5 | 100%エタノール | 5分 |
| 6 | 95%エタノール | 5分 |
| 7 | 95%エタノール | 5分 |
| 8 | 水道水 | 5分 |
| 9 | 脱イオン水 | 5分 |
| 10 | ヘマトキシリン7211 | 5分 |
| 11 | 水道水 | 5分 |
| 12 | 清澄* | 10ディップ |
| *リチャード・アラン第74位01または70%エタノール+ 0.5%のHCl | ||
| 13 | 水道水 | 5分 |
| 14 | 青味試薬 | 30秒 |
| 15 | 水道水 | 5分 |
| 16 | 95%エタノール | 10ディップ |
| 17 | エオシンY | 1分 |
| 18 | 95%エタノール | 10ディップ |
| 19 | 95%エタノール | 10ディップ |
| 20 | 100%エタノール | 10ディップ |
| 21 | 100%エタノール | 10ディップ |
| 22 | 100%エタノール | 5分 |
| 23 | キシレン | 10ディップ |
| 24 | キシレン | 5分 |
表2のH&E染色。
6.細胞密度を測定します
- .tifファイルとして画像を保存し、400Xの倍率で明視野顕微鏡を使用して、サロゲートの画像の少なくとも1全体のH&E染色された組織学的セクション。
注: 説明した画像処理は、カラー画像を用いて完成されています。テストされていないが、我々は、同様の処理をもグレースケール画像に適用可能であるべきであると信じています。 - CellProfilerはブロード研究所26(http://cellprofiler.org/download.shtml)と国立衛生研究所からのImageJ(http://rsb.info.nih.gov/ij/download.html)、の両方からダウンロードこれは無償で公的に入手可能です。
- 各画像中のサロゲートの面積を測定するために、オープンImageJが、選択「エリア」(「分析」タブの下に)「測定値の設定」を選択し、「オーケー&#を選択します34 ;.
- サロゲートの画像(.tifファイル)を開きます。 ImageJのポリゴンツールを使用してマウスをドラッグすると、アンカーポイントを作るためにクリックすることにより、画像内のサロゲートの領域の輪郭を描く( 図2参照します)。ガイドとしてECMのエッジを使用してください。一度概説、「分析」タブで「測定」を選択します。
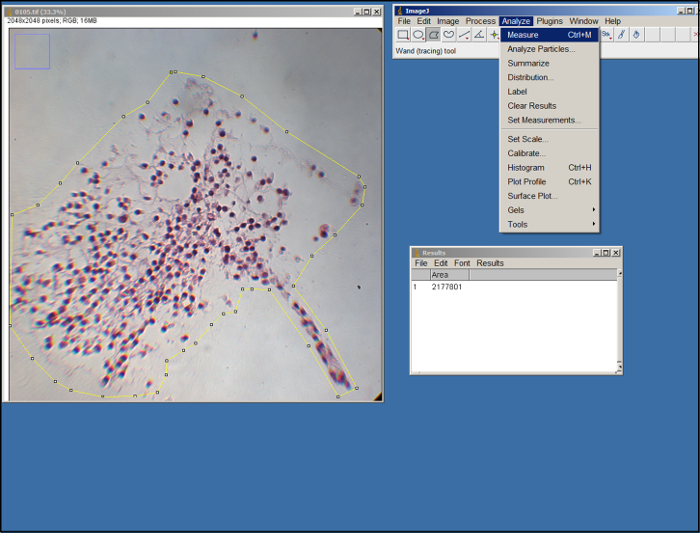
図2. ImageJの解析。 ImageJの処理のスクリーンショット。 この図の拡大版をご覧になるにはこちらをクリックしてください。
- 組織・サロゲートの画像ごとに繰り返します。スプレッドシートプログラムに測定し、対応する画像識別子を保存します。
- 「ファイルリスト」に画像ファイルをドラッグしてCellProfilerにImageJの中で面積を測定するために使用する画像ファイルをアップロードします。で取り込んだ画像に名前を割り当て「NamesAndTypes「入力モジュールと画像の種類( すなわち、「カラー」)を選択します。
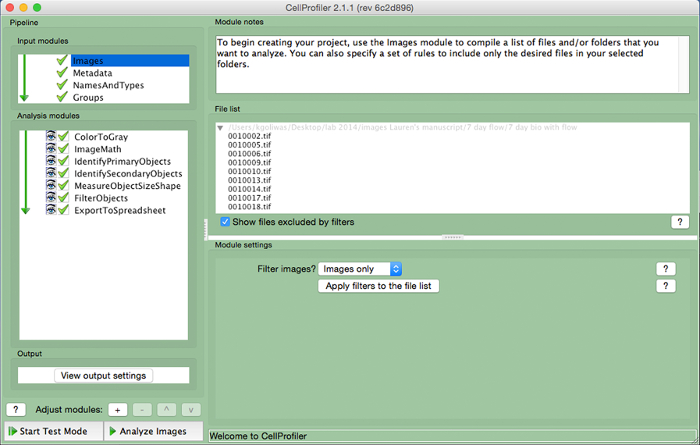
図3. CellProfiler例のパイプライン。CellProfiler中の有核細胞の数を測定するために設計されたパイプラインのスクリーンショット。 この図の拡大版をご覧になるにはこちらをクリックしてください。
- 次の(左パネルの下部にある図3、)「モジュールの調整」に「+」記号をクリックすることで、画像あたりの細胞数を計算するには、次のモジュールが含まれて解析パイプラインを作成します。以下に各モジュールを追加します。
- 「ColorToGray」( 図4)を選択します。
- <(ドロップダウンメニューから、入力画像名を選択し、出力画像に名前を付け、元の画像の種類を選択em>のすなわち、RGBカラー画像入力する場合)。
注: グレースケール画像を使用している場合、このモジュールが必要とされないであろう
- <(ドロップダウンメニューから、入力画像名を選択し、出力画像に名前を付け、元の画像の種類を選択em>のすなわち、RGBカラー画像入力する場合)。
- 「ImageMath」( 図5)を選択します。
- 「反転」操作を選択し、出力画像に名前を付けて、「最初の画像を選択」タブにグレースケール画像を選択します。
- 「IdentifyPrimaryObjects」( 図6)を選択します。
- 入力画像(数学補正後の画像)を選択し、識別されるべき第一の目的(核を)名前を付けて、ピクセル単位(約25〜65)で測定されるオブジェクトのための直径範囲を入力します。 「三つのクラス」と「大津」しきい値法と「適応」の閾値戦略を選択します。デフォルト設定から任意の他のパラメータを変更しないでください。
注: 直径の最適な範囲は、入力画像部の画像を開き、核の直径を測定することによって決定されるべきである(すなわち、小節の長さのツールを使用して、プライマリ・オブジェクト)。
- 入力画像(数学補正後の画像)を選択し、識別されるべき第一の目的(核を)名前を付けて、ピクセル単位(約25〜65)で測定されるオブジェクトのための直径範囲を入力します。 「三つのクラス」と「大津」しきい値法と「適応」の閾値戦略を選択します。デフォルト設定から任意の他のパラメータを変更しないでください。
- 「IdentifySecondaryObjects」( 図7)を選択します。
- 入力画像(数学補正後の画像)を選択し、入力オブジェクト(核)を選択し、識別されるオブジェクトに名前を付ける(セル)。二次オブジェクトを識別するために、「伝播」方法を選択し、「加重分散を「最小化」二つのクラス」と「適応」しきい値戦略と「大津」メソッドを使用します。 「スムージング」と閾値補正係数1、0と1の下限と上限、および0.05の正規化係数を選択します。デフォルト設定から任意の他のパラメータを変更しないでください。
- 「MeasureObjectSizeShape」( 図8)を選択します。
- オブジェクトを測定するように、細胞(二次オブジェクト)と核(プライマリ・オブジェクト)を選択します。
- 「FilterObjects」( 図9を選択)。
- 出力オブジェクトに名前を付け、フィルタにオブジェクトとして核(プライマリ・オブジェクト)を選択します。
- デフォルトの設定に従って、次の2つのパラメータを保管してください。測定などのカテゴリ別にフィルタリングするための測定として「areashapeが」、および「形状因子」を選択します。
- 最小測定値を使用してフィルタリングするには「はい」を選択し、0.52の最小値を追加します。
- 最大測定を使用してフィルタリングするには「いいえ」を選択します。
- フィルタリング対象物の輪郭を保持し、概説した画像に名前を付け、「はい」を選択します。
- 「ExportToSpreadsheet」( 図10)を選択します
- ここで、ファイルと名前「出力ファイル」を保存する]を選択します。
- 解析パイプラインは(ステップ6.7.7.1に6.7.1)生成された後、最適なPAを確保するために、テスト画像中の核が適切にフィルタリングされることを含めて、各ステップをCellProfilerで「テストモード」を起動し、評価ラメータは、細胞を同定するために選択した。 図11(緑の円内)核が正しく識別されているフィルタリング、以下CellProfilerから出力画像を示しており、背景が除外されています。
- パラメータが評価され、十分であると決定されると、プロジェクトを保存してから、「画像を分析」をクリックします。このプロジェクトは、将来の分析のために繰り返し使用することができます。
注パラメータが確立されたら、プログラムを順次、「ファイルリスト」内のすべての画像を分析するように設定されています。これは、より長い処理時間を引き起こし分析した各画像のために開いた複数のウィンドウになります。- 長い処理時間を回避するために、「解析モジュール」セクションの下に「ExportToSpreadsheet」を除くすべてのモジュールの目のアイコンをクリックします。
- すべての代理画像がCellProfilerによって処理された後、細胞により生成された画像データを含むスプレッドシートを開きプロファイラとImageJので測定エリアを含むスプレッドシート。フィルタリング核データ(CellProfilerスプレッドシートの列D)及び画像識別子(列R)をコピーし、測定面積データを含むスプレッドシートに貼り付けます。
- サロゲートの画像あたりの核の数に対して得られた測定値のすべての合計を計算します。
- サロゲートの各画像からサロゲート領域について得られた測定値の合計を計算します。 1×10 6によって測定された総面積を分割します。
- 1×10 6画素2あたりの細胞の数の値を取得するために上記のステップの総面積測定によって核の合計数を割ります。
- 「ColorToGray」( 図4)を選択します。
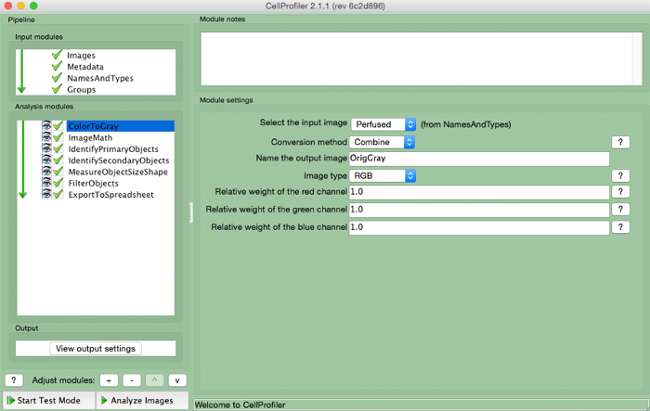
図4. CellProfilerパイプライン:グレースケールにイメージを変える 「ColortoGray」モジュールのスクリーンショット。S://www.jove.com/files/ftp_upload/54004/54004fig4highres.jpg "ターゲット=" _空白 ">この図の拡大版をご覧になるにはこちらをクリックしてください。
図5. CellProfilerパイプライン:反転画像 」ImageMath」モジュールのスクリーンショットこの図の拡大版をご覧になるにはこちらをクリックしてください。

図6. CellProfilerパイプライン:。。核を識別する 「IdentifyPrimaryObjects」モジュールのスクリーンショットこの図の拡大版をご覧になるにはこちらをクリックしてください。
図7. CellProfilerパイプライン:。。細胞を同定する 「IdentifySecondaryObjects」モジュールのスクリーンショットこの図の拡大版をご覧になるにはこちらをクリックしてください。
図8. CellProfilerパイプライン:オブジェクトを測定する 「MeasureObjectSizeShape」モジュールのスクリーンショットこの図の拡大版をご覧になるにはこちらをクリックしてください。
図9. CellProfilerパイプライン:オブジェクトのフィルタリング 」Fのスクリーンショット。ilterObjects「モジュール。 この図の拡大版をご覧になるにはこちらをクリックしてください。
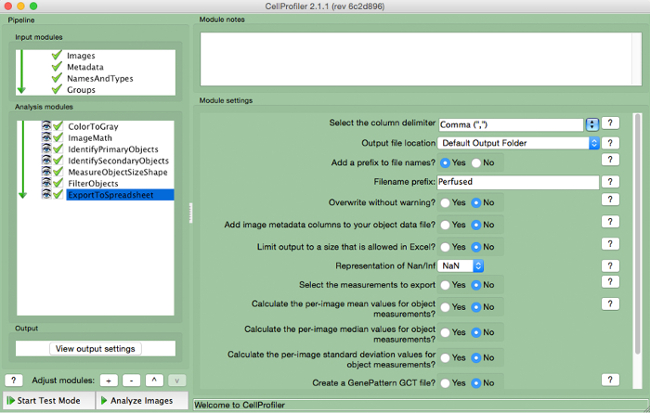
図10. CellProfilerパイプライン:。。データのエクスポート 」ExportToSpreadsheet」モジュールのスクリーンショットこの図の拡大版をご覧になるにはこちらをクリックしてください。
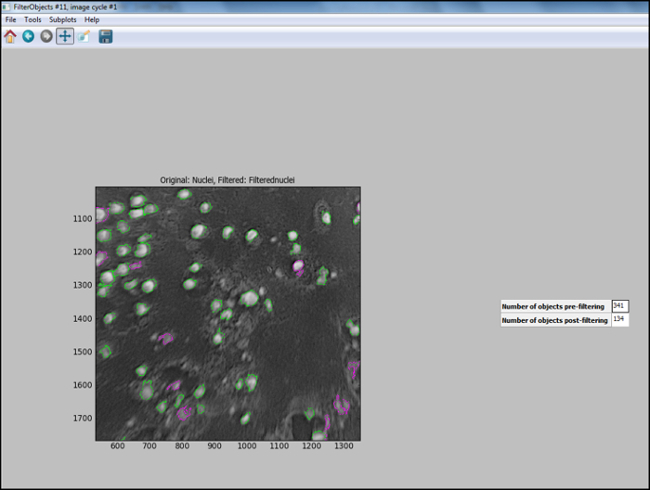
フィルタリング以下の図11. CellProfiler出力。オブジェクトのフィルタリング次電池プロファイラでの出力画面のスクリーンショット。 より大きなversiを表示するには、こちらをクリックしてください。この図の上。
結果
上述の7日間増殖させたような固体及び灌流の両方の3D乳癌代用物を調製しました。その後、サロゲートは、固定パラフィンように処理し、切片、および上記のように、ヘマトキシリンおよびエオシンで染色しました。各サロゲートの面積当たりの有核細胞の数(両方とも231細胞及びCAF)を測定しました。 図12に見られるように、H&E染色切片の代表的...
ディスカッション
本明細書において、3D培養の方法は、より密接にモデルヒト乳癌は概括的な3D形態の開発を可能にする量で、細胞外マトリックス(ECM)およびヒト間質線維芽細胞を含む組織の微小環境の成分を組み込むことが記載されています。記載の三次元培養法は、ECMの三次元ボリュームに組み込まれている、複数の細胞型における従来の2D細胞培養物よりも、ヒト疾患のより代表的です。組織構造、微?...
開示事項
The authors declare that they have no competing financial interests.
謝辞
The University of Alabama at Birmingham Center for Metabolic Bone Disease performed the histologic processing and sectioning of surrogates. Southern Research (Birmingham, AL) provided support for the manufacture of the bioreactor system. Funding was provided by the United States Department of Defense Breast Cancer Research Program (BC121367).
資料
| Name | Company | Catalog Number | Comments |
| Dulbecco's Modified Eagel Medium 1x (DMEM) | Corning CellGro | 10-014-CV | |
| Fetal Bovine Serum (FBS) | Atlanta Biologicals | S11150 | |
| 0.25% Trypsin + 2.21 mM EDTA 1x | Corning | 25-053-CI | |
| Tissue Culture plates, 100 mm | CellTreat Scientific Products | 229620 | Sterile |
| Tissue Culture plates, 35 mm | CellTreat Scientific Products | 229638 | For PDMS foam formation |
| 9" Glass pipette | Fisher | 13-678-20D | Sterile |
| 10 ml pipette | CellTreat Scientific Products | 229210B | Sterile |
| 1,000 µl piptette tips | FisherBrand | 02-717-166 | Sterile Filtered |
| 200 µl pipette tips | FisherBrand | 02-717-141 | Sterile Filtered |
| 10 µl pipette tips | FisherBrand | 02-717-158 | Sterile Filtered |
| 15 ml conical tubes | CellTreat Scientific Products | 229410 | Sterile |
| 50 ml conical tubes | CellTreat Scientific Products | 229422 | Sterile |
| 1.5 ml microcentrifuge tubes | FisherBrand | 05-408-129 | Sterile |
| Trypan blue | Corning Cellgro | 25-900-CI | Sterile |
| Sylgard 184 | Electron Microscopy Sciences | 24236-10 | PDMS elastomer and curing agent. Used for our in-house bioreactor. |
| PDMS Foam | Made in-house for use in our in-house bioreactor. | ||
| High Concentration Bovine Collagen Type I | Advanced Biomatrix | 5133-A | FibriCol ~10 mg/ml |
| Growth Factor Reduced Matrigel (Basement Membrane) | Corning | 354230 | Basement membrane material |
| Sodium Bicarbonate | Sigma | S8761 | |
| Molecular Biology Grade Water | Fisher | BP2819-1 | |
| DMEM 10x | Sigma-Aldrich | D2429 | |
| Nunc Lab-Tek Chamber Slide System | Thermo Scientific | 177402 | 8-well |
| Bioreactor | Made in-house. | ||
| Spring-Back 304 Stainless Steel—Coated with PTFE polymer | McMaster-Carr | 1749T19 | Stainless steel wires to generate microchannels in our in-house bioreactor system. 0.016" Diameter |
| BioPharm Plus platinum-cured silicone pump tubing, L/S 14 | Masterflex | EW-96440-14 | For use in our in-house bioreactor system. Tubing ID: 1.6 mm, Hose barb size: 1/16 in. |
| 2-Stop Tubing Sets, non-flared PVC, 1.52 mm ID | Cole-Parmer | EW-74906-36 | For use in our in-house bioreactor system (with microperistalitic pump). |
| Six Channel precision micro peristaltic pump | Cole-Parmer | EW-74906-04 | For use with our in-house bioreactor system |
| Tuberculin Syringes | BD Medical | 309625 | 26 gauge 3/8 in. needle; Sterile |
| Dissecting Tissue Forceps | FisherBrand | 13-812-36 | 5.5 inch |
| Mini Tube Rotator | Boekel Scientific | 260750 | Equipment option for surrogate rotation. Used with carousel for 50 ml tubes (model number 260753) |
| 50 ml tube carousel | Boekel Scientific | 260753 | Used with mini tube rotator |
| Bambino Hybridization Oven | Boekel Scientific | 230301 | Equipment option for surrogate rotation |
| HistoGel Specimen Processing Gel | Thermo Scientific | HG-4000-012 | Specimen Processing Gel described in Step 5.2 |
| Cryomold | Andwin Scientific | 4566 | 15 mm x 15 mm x 5 mm |
| Tissue Marking Dye | Cancer Diagnostics, inc. | 03000P | Can be used to mark surrogates, allowing multiple samples to be included in one tissue cassette |
| Hinged tissue cassettes | FisherBrand | 22-272-416 | |
| Formalin | Fisher | 23-245-685 | |
| GoldSeal Plain Glass Slides | Thermo Scientific | 3048-002 | |
| Xylene | Fisher | X3P-1GAL | |
| Ethanol, 200 proof (100%), USP | Decon Laboratories, Inc. | 2805M | |
| Hematoxylin | Thermo Scientific Richard-Allan Scientific | 7211 | |
| Clarifier | Thermo Scientific Richard-Allan Scientific | 7401 | |
| Bluing Solution | Thermo Scientific Richard-Allan Scientific | 7301 | |
| Eosin Y | Thermo Scientific Richard-Allan Scientific | 7111 | |
| Cytoseal XYL mounting media | Thermo Scientific Richard-Allan Scientific | 83124 | |
| Coverslips | Fisher Scientific | 12-548-5G |
参考文献
- Hakanson, M., Textor, M., Charnley, M. Engineered 3D environments to elucidate the effect of environmental parameters on drug response in cancer. Integr Biol (Camb). 3 (1), 31-38 (2011).
- Horning, J. L., et al. 3-D tumor model for in vitro evaluation of anticancer drugs. Mol Pharm. 5 (5), 849-862 (2008).
- Dhiman, H. K., Ray, A. R., Panda, A. K. Three-dimensional chitosan scaffold-based MCF-7 cell culture for the determination of the cytotoxicity of tamoxifen. Biomaterials. 26 (9), 979-986 (2005).
- Place, A. E., Jin Huh, S., Polyak, K. The microenvironment in breast cancer progression: biology and implications for treatment. Breast Cancer Res. 13 (6), 227 (2011).
- Mao, Y., Keller, E. T., Garfield, D. H., Shen, K., Wang, J. Stromal cells in tumor microenvironment and breast cancer. Cancer Metastasis Rev. 32 (1-2), 303-315 (2013).
- Paulsson, J., Micke, P. Prognostic relevance of cancer-associated fibroblasts in human cancer. Semin Cancer Biol. 25, 61-68 (2014).
- Roskelley, C. D., Desprez, P. Y., Bissell, M. J. Extracellular matrix-dependent tissue-specific gene expression in mammary epithelial cells requires both physical and biochemical signal transduction. Proc. Natl. Acad. Sci. 91, 12378-12382 (1994).
- Pickl, M., Ries, C. H. Comparison of 3D and 2D tumor models reveals enhanced HER2 activation in 3D associated with an increased response to trastuzumab. Oncogene. 28 (3), 461-468 (2008).
- Ivascu, A., Kubbies, M. Rapid generation of single-tumor spheroids for high-throughput cell function and toxicity analysis. J Biomol Screen. 11 (8), 922-932 (2006).
- Lovitt, C. J., Shelper, T. B., Avery, V. M. Advanced cell culture techniques for cancer drug discovery. Biology (Basel). 3 (2), 345-367 (2014).
- Bergamaschi, A., et al. Extracellular matrix signature identifies breast cancer subgroups with different clinical outcome. J Pathol. 214 (3), 357-367 (2008).
- Oskarsson, T. Extracellular matrix components in breast cancer progression and metastasis. The Breast. 22, S66-S72 (2013).
- Kelley, L. C., Lohmer, L. L., Hagedorn, E. J., Sherwood, D. R. Traversing the basement membrane in vivo: A diversity of strategies. JBC. 204 (3), 291-301 (2014).
- Sadlonova, A., et al. Breast fibroblasts modulate epithelial cell proliferation in three-dimensional in vitro co-culture. Breast Cancer Res. 4, (2004).
- Wendt, D., Marsano, A., Jakob, M., Heberer, M., Martin, I. Oscillating perfusion of cell suspensions through three-dimensional scaffolds enhances cell seeding efficiency and uniformity. Biotechnol Bioeng. 84 (2), 205-214 (2003).
- Marshall, L. E., et al. Flow-perfusion bioreactor system for engineered breast cancer surrogates to be used in preclinical testing. J Tissue Eng Regen Med. , (2015).
- Calcagnile, P., Fragouli, D., Mele, E., Ruffilli, R., Athanassiou, A. Polymeric foams with functional nanocomposite cells. RSC Adv. 4, 19177-19182 (2014).
- Naba, A., Clauser, K. R., Lamar, J. M., Carr, S. A., Hynes, R. O. Extracellular matrix signatures of human mammary carcinoma identify novel metastasis promoters. eLife. 3, (2014).
- Lochter, A., Bissell, M. J. Involvement of extracellular matrix constituents in breast cancer. Semin Cancer Biol. 6 (3), 165-173 (1995).
- Joiner, K. S., Spangler, E. A. Evaluation of HistoGel-embedded specimens for use in veterinary diagnostic pathology. J Vet Diagn Invest. 24 (4), 710-715 (2012).
- Varsegi, G. M., Shidham, V. Cell Block Preparation from Cytology Specimen with Predominance of Individually Scattered Cells. J Vis Exp. (29), e1316 (2009).
- Sadlonova, A., et al. Human Breast Fibroblasts Inhibit Growth of the MCF10AT Xenograft Model of Proliferative Breast Disease. Am J Pathol. 170 (3), (2007).
- Otali, D., He, Q., Stockard, C. R., Grizzle, W. E. Preservation of immunorecognition by transferring cells from 10% neutral buffered formalin to 70% ethanol. Biotech Histochem. 88, 170-180 (2013).
- Webster, S. S., Jenkins, L., Burg, K. J. L. Histological Techniques for Porous, Absorbable, Polymeric Scaffolds, Used in Tissue Engineering. J Histotechnol. 26 (1), 57-65 (2003).
- Troy, T. -. C., Arabzadeh, A., Enikanolaiye, A., Lariviere, N., Turksen, K. Immunohistochemistry on Paraffin Sections of Mouse Epidermis Using Fluorescent Antibodies. J Vis Exp. (11), (2008).
- Carpenter, A. E., et al. CellProfiler: image analysis software for identifying and quantifying cell phenotypes. Genome Biol. 7 (10), R100 (2006).
- Kwon, Y. -. J., et al. Gli1 enhances migration and invasion via up-regulation of MMP-11 and promotes metastasis in ERa negative breast cancer cell lines. Clin Exp Metastasis. (28), (2011).
- Evilsizor, M. N., Ray-Jones, H. F., Lifshitz, J., Ziebell, J. Primer for Immunohistochemistry on Cryosectioned Rat Brain Tissue: Example Staining for Microglia and Neurons. J Vis Exp. (99), e52293 (2015).
- Pal, A., Kleer, C. G. Three dimensional cultures: a tool to study normal acinar architecture vs. malignant transformation of breast cells. J Vis Exp. (86), e51311 (2014).
- Hasselbach, L. A., et al. Optimization of High Grade Glioma Cell Culture from Surgical Specimens for Use in Clinically Relevant Animal Models and 3D Immunochemistry. J Vis Exp. (83), e51088 (2014).
- Foty, R. A Simple Hanging Drop Cell Culture Protocol for Generation of 3D Spheroids. J Vis Exp. (51), e2720 (2011).
- Materne, E. -. M., et al. The Multi-organ Chip - A Microfluidic Platform for Long-term Multi-tissue Coculture. J Vis Exp. (98), e52526 (2015).
- Sadlonova, A., et al. Identification of Molecular Distinctions Between Normal Breast-Associated Fibroblasts and Breast Cancer-Associated Fibroblasts. Cancer Microenviron. 2, 9-21 (2009).
- Wang, J. D., Douville, N. J., Takayama, S., ElSayed, M. Quantitative analysis of molecular absorption into PDMS microfluidic channels. Ann Biomed Eng. 40 (9), 1862-1873 (2012).
- Halldorsson, S., Lucumi, E., Gomez-Sjoberg, R., Fleming, R. M. Advantages and challenges of microfluidic cell culture in polydimethylsiloxane devices. Biosens Bioelectron. 63, 218-231 (2015).
- Regehr, K. J., et al. Biological implications of polydimethylsiloxane-based microfluidic cell culture. Lab Chip. 9 (15), 2132-2139 (2009).
- Burdett, E., Kasper, F. K., Mikos, A. G., Ludwig, J. A. Engineering tumors: a tissue engineering perspective in cancer biology. Tissue Eng Part B Rev. 16 (3), 351-359 (2010).
- Caruso, R. A., et al. Mechanisms of coagulative necrosis in malignant epithelial tumors (Review). Oncol Lett. 8 (4), 1397-1402 (2014).
- Elmore, S. Apoptosis: A Review of Programmed Cell Death. Toxicol Pathol. 35 (4), 495-516 (2007).
- Majno, G., Joris, I. Apoptosis, oncosis, and necrosis. An overview of cell death. Am J Pathol. 146 (1), 3-15 (1995).
- Ogino, S., et al. Molecular pathological epidemiology of epigenetics: emerging integrative science to analyze environment, host, and disease. Mod Pathol. 26 (4), 465-484 (2013).
- Otali, D., et al. Combined effects of formalin fixation and tissue processing on immunorecognition. Biotech Histochem. 84 (5), 223-247 (2009).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved