Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Medición simultánea de HDAC1 y HDAC6 actividad en HeLa células usando UHPLC-MS
* Estos autores han contribuido por igual
En este artículo
Resumen
El presente método sirve para identificar inhibidores de la isoforma específica de histona deacetilasas (HDAC) en células HeLa por el análisis UHPLC-MS de múltiples sustratos. Este es un método libre de anticuerpos desarrollado para reflejar actividad HDAC1 y HDAC6 en la vida del ambiente celular, en contraste con solo isoform análisis sin células.
Resumen
La búsqueda de nuevos inhibidores de histona deacetilasa (HDAC) es de creciente interés en el descubrimiento de medicamentos. Isoforma selectividad ha sido el centro de atención desde la aprobación de la romidepsina, una clase que las HDACS inhibidor para el tratamiento del cáncer y la investigación clínica de los inhibidores de la HDAC6 específico para el mieloma múltiple. El presente método permite determinar la actividad inhibitoria de los compuestos de prueba en HDAC1 y HDAC6 en las células. La actividad de la isoforma se mide utilizando la cromatografía líquida de ultra alto rendimiento-análisis de espectrometría de masas (UHPLC-MS) de sustratos específicos que se incubaron con células HeLa tratadas y no tratadas. El método tiene la ventaja de reflejar la actividad HDAC endógena dentro del entorno de la célula, en contraste con ensayos bioquímicos sin células en isoformas aislado. Por otra parte, porque se basa en la cuantificación de sustratos sintéticos, el método no requiere el reconocimiento de anticuerpos de proteínas endógenas acetilados. Es fácilmente adaptable a varias líneas celulares y un proceso automatizado. El método ya ha demostrado ser útil en la búsqueda de compuestos HDAC6 selectivo en neuroblastos. Aquí se muestran resultados representativos con el estándar HDAC inhibidores tricostatina A (no específica), MS275 (HDAC1-específica), y tubastatin (HDAC6-específico) con células HeLa.
Introducción
Las HDACs pertenecen a una familia de enzimas capaces de deacetylate histonas dentro de la estructura de la cromatina. También tienen otros substratos de la proteína en el citosol y se encuentran en diferentes compartimentos celulares. Un total de 18 HDAC isoformas se han identificado hasta ahora y se han relacionado con varios mecanismos de la célula, incluyendo la regulación de factores de transcripción y expresión génica, así como la señalización celular y transporte1,2,3,4,5,6,7. Inhibidores de la HDAC catalíticos han surgido como potenciales medicamentos para terapia del cáncer. Más inhibidores de las HDACS actualmente aprobados por la FDA para el tratamiento del linfoma del T-cell y el mieloma múltiple y son inhibidores de las HDACS no específicos, tales como vorinostat (SAHA), belinostat y panobinostat8,9. Sin embargo, una serie de efectos secundarios se han asociado con inhibidores de la cacerola, y la búsqueda de moléculas pequeñas de la isoforma específica es un tema candente en descubrimiento de química y drogas medicinal. En consecuencia, la clase me romidepsina inhibidor selectivo (HDAC1-3 y HDAC8) es un medicamento ya aprobado10, mientras que los inhibidores específicos de la HDAC6 son actualmente bajo ensayos clínicos, con mayor potencial terapéutico en el mieloma múltiple11,12,13,14,15.
Selección de ensayos para caracterizar las HDACS inhibidores están basados en la incubación de un sustrato HDAC con una fuente enzimática (isoforma sola, extracto nuclear o lisado de célula). El sustrato es generalmente una secuencia de péptido pequeño que contiene un residuo de lisina de acetilo acoplado a un fluoróforo divisible (por ejemplo, cumarina), como N-(4-methyl-7-aminocoumarinyl)-Nα-(t-butoxycarbonyl)-Nω-acetyllysineamide (MAL)16. Para distinguir entre las actividades de la isoforma específica, análisis sin células separados que implica cada isoforma son necesarios y podrían no reflejar la actividad de la isoforma real en las células vivas. Específica de isoforma sustratos están comercialmente disponibles, como bencílico (S)-[1-(4-methyl-2-oxo-2H-chromen-7-ylcarbamoyl)-5-propionylaminopentyl]carbamate (MOCPAC, HDAC1 sustrato específico) y (éster terc-butílico ácido de S)-[5-acetylamino-1-(2-oxo-4-trifluoromethyl-2H-chromen-7-ylcarbamoyl)pentyl]carbamic (BATCP, sustrato específico HDAC6) (figura 1B). Sin embargo, una mezcla de varios sustrato que contiene MAL, MOCPAC y BATCP a las células vivas no permitirá la detección de los productos desacetilados de medición fluorométrica, dado que llevan el mismo fluoróforo escindibles.
El método aquí descrito permite la detección y cuantificación relativa de cada sustrato y su producto desacetilado en células HeLa, usando un análisis de múltiples sustrato seguido de UHPLC-ESI-MS/MS análisis17. Un análisis de las HDACS se lleva a cabo en las células HeLa que permitan la identificación directa de la actividad inhibitoria de las HDACS y de la especificidad de la prueba de compuestos sobre las HDACs endógenas. Hay un enfoque en HDAC1 y HDAC6, que son evaluados al mismo tiempo. Para lograr estas mediciones enzimáticas en un ensayo de incubación única, una mezcla de sustratos HDAC no específicos y específicos se agrega a las células HeLa tratadas y no tratadas sobre una placa de 96 pocillos. Tras un paso de incubación, las células son sometidas a lisis para liberar a los sustratos y sus productos de la reacción respectiva, que se separan y detectan utilizando un método de UHPLC-MS (figura 1). Los productos desacetilados de los sustratos MAL MOCPAC y BATCP son el MAL desacetilado (dMAL) MOCPAC desacetilado (dMOCPAC) y BATCP desacetilado (dBATCP), respectivamente. Curvas de dosis-respuesta se pueden construir con compuestos activos.
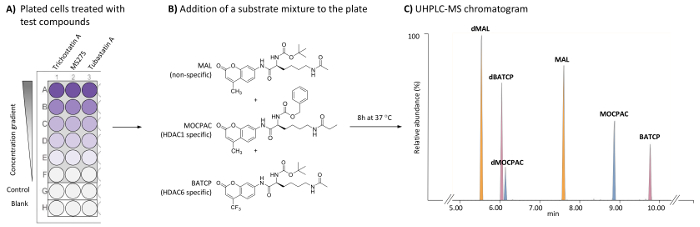
Figura 1: esquema General para el análisis de las HDACS basadas en células identificar inhibidores específicos de HDAC1 y HDAC6 por el análisis UHPLC-MS de múltiples sustratos. (A) esquema de una típica placa de 96 pocillos que contienen tratados (compuestos de prueba) y las células HeLa sin tratamiento (control), así como espacios libres de células. (B) estructura química de sustratos ha añadido como una mezcla (21 μm) a desacetilado de HDACs endógenas. (C) cromatograma típico UHPLC-MS que muestra los picos de los sustratos agregados (MAL MOCPAC y BATCP) y sus productos desacetilados (dMAL, dMOCPAC y dBACTP, respectivamente). Haga clic aquí para ver una versión más grande de esta figura.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. cultivo celular
Nota: Los siguientes pasos se realizan en una campana de cultivo de tejidos estándar. Se espera que la familiaridad con técnica estéril.
- Cultivo de células HeLa a confluencia del 80% en un matraz de cultivo celular T75 en MEM suplementado con 10% suero fetal bovino, penicilina G (100 U/mL) y estreptomicina (100 mg/mL).
Nota: Cultura, el tratamiento y pasos de lisis celular se llevan a cabo bajo flujo laminar, con pasos de incubación de células a 37 ° C en una atmósfera humidificada de 5% CO2 en condiciones estériles. - Lavan las células dos veces con 5 mL de DPBS, aspirar la DPBS y añadir 1 mL de reactivo de disociación celular. Incubar a 37 ° C durante 5 min separar las células.
- Añadir 9 mL de medio de cultivo MEM, bien suspender la suspensión de células y contar las células usando un hemocitómetro.
- Ajustar la densidad de la suspensión celular en 6 x 104 células/mL de MEM.
- Añadir 100 μl de la suspensión celular a control y prueba de los pozos de una placa de 96 pocillos estéril, fondo plano, tratada con cultivo de tejidos (figura 1A).
- Añadir 100 μl de medio de cultivo MEM a pozos en blanco de la placa de 96 pocillos (figura 1A).
- Incubar la placa de 96 pozos a 37 ° C durante 24 h.
Nota: Una incubación de 24 h es un momento adecuado para probar la actividad HDAC endógena en las células HeLa. Diferentes tipos de células con actividad HDAC desconocido pueden necesitar una evaluación timecourse de las células del control.
2. células: tratamiento con compuestos de prueba y sustratos HDAC
- Aspire el medio de los pozos de la placa de 96 pocillos.
- Añada 25 μl de 2 x test compuestos (p. ej., tricostatina A, MS275 y tubastatin A) en el MEM para prueba de pozos. Añada 25 μl de MEM a control y pozos en blanco.
Nota: La concentración de compuestos de la prueba debe ajustarse para proporcionar una gama de concentraciones de ensayo final por lo menos 5 (a una concentración por pozo). La concentración final de DMSO en la sustancia de ensayo no debe exceder 0.5% y debe ser el mismo en cada pocillo, incluyendo espacios en blanco y los controles. El volumen final de la incubación es de 50 μL por pocillo. A Trichostatin es probado en 5 concentraciones que van desde 500 hasta 1,95 nM (dilución 1:4). MS275 y tubastatin A son probados a 5 concentraciones que van desde 8.000 hasta 6.17 nM (dilución 1:6). La concentración de la prueba final deseado para cada sustancia de ensayo debe determinarse por cada usuario individual mediante la creación de una curva dosis-respuesta. - Añadir 25 μl de la mezcla de sustrato (42 μm de MAL, 42 μm de MOCPAC y 42 de la BATCP en MEM) a pozos de control y de prueba. Añadir 25 μl de MEM a pozos en blanco.
Nota: La concentración final de cada sustrato es 21 μm. MOCPAC y BATCP distinguir entre actividad de HDAC1 y HDAC6, respectivamente (figura 1B). Cuando no hay actividad se observa con los sustratos, el uso de MAL podría llevar potencialmente a la identificación de la actividad contra otra isoforma HDAC. - Incubar la placa de 96 pozos a 37 ° C durante 8 h en un 5% humidificado incubadora de CO2 .
Nota: Este tiempo de incubación es conveniente para las células HeLa y permite la HDAC endógeno interactuar con los sustratos sintéticos. Diferentes líneas celulares pueden requerir ajustes en la concentración de tiempo o sustrato de incubación.
3. la célula Lysis y preparación de muestras para UHPLC-ESI-MS/MS
PRECAUCIÓN: Los pasos de preparación de muestra usan productos químicos orgánicos, que son altamente inflamables y tóxicos por ingestión o inhalación. Utilice protección personal adecuada, como guantes y gafas de seguridad.
- Añadir 10 μl de tampón de x RIPA 6 (diluido de 10 x RIPA buffer) con inhibidor de la proteasa en cada pocillo de la placa de 96 pocillos.
- Detener la reacción añadiendo 160 μl de acetonitrilo frío a cada pozo y mezclar mediante pipeteo arriba y abajo.
Nota: Mantenga el acetonitrilo a-20 ° C antes de este paso. - Colocar la placa en un congelador de-80 ° C durante 10 minutos.
- Retire la placa del congelador y transferencia 220 μl del contenido de cada pocillo de una placa de 96 pocillos estériles, cónico-parte inferior (V abajo). Centrifugar la placa a 5.000 x g y 4 ° C durante 10 minutos.
Nota: Este paso tiene como objetivo eliminar las proteínas y sales. Si es necesario, las soluciones pueden ser transferidas a tubos de centrífuga cónicos individuales antes de centrifugar o filtradas a través de un conjunto de 96 pocillos, placas de filtro PVDF de 0,65 μm. El retiro acertado del precipitado es necesario antes del análisis UHPLC. - Transfiera 200 μL de sobrenadante (o filtrado) en una placa de 96 pocillos compatible con el sistema UHPLC y séllelo con papel pelable, soldadura usando un sellador de placas.
Nota: Evitar pipetear las pelotillas al retirar el sobrenadante. Si no puede realizarse inmediatamente el análisis UHPLC, la placa puede guardarse a 4 ° C por un máximo de 24 h hasta su análisis.
4. UHPLC/MS-MS. Análisis
- Preparar el sistema UHPLC rellenando con el sistema de fase móvil (95% A:5% B):
R: H2O, grado HPLC, que contiene 0.1% de ácido fórmico (preparar 1 L)
B: acetonitrilo, grado HPLC, que contiene 0.1% de ácido fórmico (preparar 1 L).
PRECAUCIÓN: La fase móvil contiene productos químicos orgánicos, que son altamente inflamables y tóxicos por ingestión o inhalación. Utilice protección personal adecuada, como guantes y gafas de seguridad. - Coloque la placa de 96 pocillos en el administrador muestra que contiene un sostenedor de la placa. Ejecutar las muestras según las condiciones UHPLC-ESI-MS/MS proporcionadas en la tabla 1.
Nota: Cada columna de la placa de 96 pocillos analítica debe tener así un control y un pozo en blanco (como se muestra en el esquema de la placa de la figura 1A). Los controles son representativos de 100% de actividad enzimática en la línea celular probado, durante los espacios en blanco comprobar si la fuente MS proporciona antecedentes consistentes. Picos de MS inusuales detectadas en un determinado espacio en blanco deben ser investigados para prevenir MS cambios de sensibilidad.
5. Análisis de datos
- Integrar el área de pico de cada sustrato y su producto desacetilado con un software de integración apropiado de UHPLC.
Nota: Utilizar parámetros de integración automática con un método de suavizado (media, tamaño de la ventana 3 y número de Lisa 2, por ejemplo). Usando las condiciones cromatográficas proporcionadas en la tabla 1, el orden de elución es dMAL (5,8 min), dBATCP (6,0 min), dMOCPAC (mínima 6,2), MAL (min 7,6), MOCPAC (8,9 min) y BATCP (9,8 min), como se muestra en la figura 1. - Para cada sustrato, calcular la proporción del área pico (desacetilado/acetilado) en cada cromatograma (muestras de control y de test).
- Calcular el porcentaje inhibición de la HDAC de cada muestra:
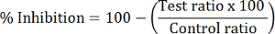
Nota: Inhibición de HDAC1 se calcula utilizando la proporción del área pico de dMOCPAC/MOCPAC; HDAC6 inhibición se calcula con dBATCP/BATCP. La relación dMAL/MAL puede utilizarse para expresar general inhibición de la HDAC.
Access restricted. Please log in or start a trial to view this content.
Resultados
Para demostrar la aplicación del método para identificar inhibidores selectivos y no selectivos para HDAC1 (clase I) y HDAC6 (clase IIb), las células HeLa fueron tratadas con compuestos estándar conocidos: tricostatina A (no selectivo entre HDAC1 y HDAC6)18y MS275 (inhibidor selectivo de HDAC1)19tubastatin (inhibidor selectivo de HDAC6)20. Mediante el método actual (figura 1), valore...
Access restricted. Please log in or start a trial to view this content.
Discusión
Inhibición de la HDAC es un tema candente en el descubrimiento de medicamentos, con un enfoque actual en HDAC6 selectivo inhibidores de cáncer terapia21. Selectividad HDAC es evaluado generalmente por una serie de ensayos de alto rendimiento, libres de células, que pretenden determinar la potencia inhibitoria hacia individuales HDAC isoformas21. Sin embargo, la selectividad de un inhibidor debe ser además confirmada en las células vivas por evaluar el estado de acetila...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Los autores reconocen costo acción CM1406 (epigenéticos Biología química). Investigación en esta publicación fue apoyada por la Fundación Pierre Mercier.
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| BATCP | Sigma-Aldrich | B4061 | |
| MAL | Sigma-Aldrich | SCP0168 | Synonym: BOC-Ac-Lys-AMC |
| MOCPAC | Sigma-Aldrich | M2195 | |
| MS275 | Sigma-Aldrich | EPS002 | |
| Trichostatin A | Sigma-Aldrich | T8552 | |
| Tubastatin A | Sigma-Aldrich | SML0044 | |
| Acetonitrile, HPLC grade | Fisher Scientific | 10660131 | |
| Formic acid, LC/MS grade | Fisher Scientific | 10596814 | |
| H2O, HPLC grade | distilled H2O filtered through a Milli-Q purification system | ||
| HeLa cells | ATCC | ATCC CRM-CCL-2 | |
| Cell dissociation reagent TrypLE Express | ThermoFisher Scientific | 12604013 | |
| DMSO, cell culture grade | Applichem | 146463 | |
| DPBS | ThermoFisher Scientific | 14190144 | |
| Fetal bovine serum | Biowest | S1810 | |
| MEM | ThermoFisher Scientific | 22561021 | |
| Penicillin-Streptomycin | Bioconcept | 4-01F00-H | |
| 10x RIPA buffer | Abcam | ab156034 | |
| SigmaFast protease inhibitor tablets | Sigma-Aldrich | S8820 | |
| 96-well plates, sterile, flat-bottom, tissue culture treated Corning | VWR | 29442-058 | |
| 96-well plates, non-sterile, V-bottom Corning | VWR | 29442-404 | used in the centrifugation step |
| 96-well plate, conical bottom, Nunc | ThermoFisher Scientific | 249944 | compatible with the Acquity UHPLC system |
| T75 cell culture flasks Corning | Sigma-Aldrich | CLS430641 | |
| Peelable heat sealing foil | Waters | 186002789 | |
| Acquity UPLC system | Waters | ||
| Eppendorf Centrifuge 5810 R | Fisher Scientific | 05-413-323 | |
| Integration software: MassLynx V4.1 | Waters | Catalog number not available | |
| Combi thermo-sealer SP-0669/240 | Waters | Catalog number not available | |
| Quattro micro API Tandem Quadrupole System | Waters | Catalog number not available |
Referencias
- Arrowsmith, C. H., Bountra, C., Fish, P. V., Lee, K., Schapira, M. Epigenetic protein families: a new frontier for drug discovery. Nat Rev Drug Discov. 11 (5), 384-400 (2012).
- Henikoff, S., Greally, J. M. Epigenetics, cellular memory and gene regulation. Curr Biol. 26 (14), R644-R648 (2016).
- Kim, C., et al. HDAC6 inhibitor blocks amyloid beta-induced impairment of mitochondrial transport in hippocampal neurons. PLoS One. 7 (8), (2012).
- Kawaguchi, Y., et al. The deacetylase HDAC6 regulates aggresome formation and cell viability in response to misfolded protein stress. Cell. 115 (6), 727-738 (2003).
- Zhao, Y., et al. Acetylation of p53 at lysine 373/382 by the histone deacetylase inhibitor depsipeptide induces expression of p21(Waf1/Cip1). Mol Cell Biol. 26 (7), 2782-2790 (2006).
- Berger, S. L. Histone modifications in transcriptional regulation. Curr Opin Genet Dev. 12 (2), 142-148 (2002).
- Simões-Pires, C., et al. HDAC6 as a target for neurodegenerative diseases: what makes it different from the other HDACs? Mol Neurodegener. 8 (7), 1-16 (2013).
- Mottamal, M., Zheng, S., Huang, T. L., Wang, G. Histone deacetylase inhibitors in clinical studies as templates for new anticancer agents. Molecules. 20 (3), 3898-3941 (2015).
- Manal, M., Chandrasekar, M. J., Gomathi Priya, J., Nanjan, M. J. Inhibitors of histone deacetylase as antitumor agents: A critical review. Bioorg Chem. 67, 18-42 (2016).
- Mack, G. S. To selectivity and beyond. Nat Biotech. 28 (12), 1259-1266 (2010).
- U.S. National Institutes of Health. Study of ACY-1215 alone and in combination with bortezomib and dexamethasone in multiple myeloma (ACY-1215). , Available from: https://clinicaltrials.gov/ct2/show/NCT01323751 (1211).
- U.S. National Institutes of Health. Study of ACY-1215 in combination with lenalidomide, and dexamethasone in multiple myeloma. , Available from: https://clinicaltrials.gov/ct2/show/NCT01583283 (2012).
- U.S. National Institutes of Health. 13ACY-1215 (ricolinostat) in combination with pomalidomide and low-dose dex in relapsed-and-refractory multiple myeloma. , Available from: https://clinicaltrials.gov/ct2/show/NCT01997840 (2013).
- U.S. National Institutes of Health. Study of ACY-241 alone and in combination with pomalidomide and dexamethasone in multiple myeloma. , Available from: https://clinicaltrials.gov/ct2/show/NCT02400242 (2015).
- Yee, A. J., et al. Ricolinostat plus lenalidomide, and dexamethasone in relapsed or refractory multiple myeloma: a multicentre phase 1b trial. Lancet Oncol. 17 (11), 1569-1578 (2016).
- Jung, M. Homogenous non-isotopic assays for histone deacetylase activity. Expert Opin Ther Pat. 13 (6), 935(2003).
- Zwick, V., Simões-Pires, C., Cuendet, M. Cell-based multi-substrate assay coupled to UHPLC-ESI-MS/MS for a quick identification of class-specific HDAC inhibitors. J Enzyme Inhibi Med Chem. 31 (1), 209-214 (2016).
- Khan, N., et al. Determination of the class and isoform selectivity of small-molecule histone deacetylase inhibitors. Biochem J. 409 (2), 581-589 (2008).
- Glaser, K. B., et al. Differential protein acetylation induced by novel histone deacetylase inhibitors. Biochem Biophys Res Commun. 325 (3), 683-690 (2004).
- Butler, K. V., et al. Rational design and simple chemistry yield a superior, neuroprotective HDAC6 inhibitor, tubastatin A. J Am Chem Soc. 132 (31), 10842-10846 (2010).
- Jung, M., Yong, K. -J., Velena, A., Lee, S. Cell-Based Assays for HDAC Inhibitor Hit Validation. Epigenetic Targets in Drug Discovery. Sippl, W., Jung, M. , Wiley-VCH. (2010).
- Milli, A., et al. Proteomic analysis of cellular response to novel proapoptotic agents related to atypical retinoids in human IGROV-1 ovarian carcinoma cells. J Proteome Res. 10 (3), 1191-1207 (2011).
- Zwick, V., et al. Synthesis of a selective HDAC6 inhibitor active in neuroblasts. Bioorg Med Chem Lett. 26 (20), 4955-4959 (2016).
- Ciossek, T., Julius, H., Wieland, H., Maier, T., Beckers, T. A homogeneous cellular histone deacetylase assay suitable for compound profiling and robotic screening. Anal Biochem. 372 (1), 72-81 (2008).
- Heltweg, B., Dequiedt, F., Marshall, B. L., Brauch, C., Yoshida, M., et al. Subtype selective substrates for histone deacetylases. J Med Chem. 47 (21), 5235-5243 (2004).
- Copeland, R. A. Evaluation of enzyme inhibitors in drug discovery. , Wiley. (2005).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados