Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Mesure simultanée de HDAC1 et de HDAC6 activité en HeLa cellules utilisant UHPLC-MS
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
La présente méthode sert à identifier les inhibiteurs de l’isoforme spécifique des histones désacétylases (HDAC) dans les cellules HeLa par l’analyse UHPLC-MS de plusieurs substrats. Il s’agit d’une méthode sans anticorps mis au point afin de tenir compte de l’activité HDAC1 et HDAC6 dans la vie cellule environnement, contrairement à l’isoforme unique acellulaire dosages.
Résumé
La recherche de nouveaux inhibiteurs d’histone désacétylase (HDAC) est d’un intérêt croissant pour la découverte de médicaments. Sélectivité de l’isoforme a été sous le feu des projecteurs depuis l’approbation du romidepsin, une classe j’ai HDAC inhibiteur pour le traitement du cancer et l’investigation clinique des inhibiteurs de HDAC6 spécifiques pour le myélome multiple. La présente méthode sert à déterminer l’activité inhibitrice des composés d’essai sur HDAC1 et HDAC6 dans les cellules. L’activité de l’isoforme est mesurée à l’aide de la chromatographie liquide à ultra performant – analyse de spectrométrie de masse (UHPLC-MS) des substrats spécifiques incubées en présence de cellules HeLa traitées et non traitées. La méthode a l’avantage de refléter l’activité HDAC endogène dans l’environnement de la cellule, à la différence des épreuves biochimiques acellulaires menées sur les isoformes isolés. En outre, parce qu’elle est fondée sur la quantification des substrats synthétiques, la méthode ne requiert pas la reconnaissance d’anticorps des protéines endogènes d’acétylés. Il est facilement adaptable à plusieurs lignées cellulaires et un processus automatisé. La méthode a déjà prouvé utile dans la recherche de composés HDAC6 sélectif en neuroblastes. Résultats représentatifs sont montrées ici avec le standard HDAC inhibiteurs trichostatine A (non spécifiques), MS275 (HDAC1-spécifique), et tubastatin un HDAC6 (spécifique) à l’aide de cellules HeLa.
Introduction
HDAC appartiennent à une famille d’enzymes capables de deacetylate des histones au sein de la structure de la chromatine. Aussi, ils ont d’autres substrats protéiques dans le cytosol et sont situés dans divers compartiments de la cellule. Un total de 18 HDAC isoformes ont été identifiés à ce jour et ont été associés à plusieurs mécanismes de la cellule, y compris la réglementation des facteurs de transcription et l’expression des gènes, ainsi que la signalisation de cellules et transport1,2,3,4,5,6,7. Inhibiteurs d’HDAC catalytiques sont apparues comme des médicaments thérapeutiques potentiels pour le traitement du cancer. La plupart inhibiteurs d’HDAC actuellement approuvés par la FDA sont pour le traitement du lymphome T et le myélome multiple et inhibiteurs d’HDAC non spécifiques, telles que le vorinostat (SAHA), belinostat et panobinostat8,9. Cependant, une série d’effets secondaires ont été associés à des inhibiteurs de pan, et la recherche pour les petites molécules isoforme spécifique est un sujet d’actualité en medicinal chemistry et découverte de médicaments. En conséquence, la classe j’ai romidepsin inhibiteur sélectif (HDAC1-3 et HDAC8) est un médicament déjà approuvé10, tandis que les inhibiteurs spécifiques HDAC6 sont en cours d’essais cliniques, avec l’accroissement du potentiel thérapeutique dans le myélome multiple11,12,13,14,15.
Dépistage des essais pour caractériser les HDAC inhibiteurs sont basées sur l’incubation d’un substrat HDAC avec une source enzymatique (isoforme unique, extrait nucléaire ou lysat cellulaire). Le substrat est généralement une séquence de petit peptide contenant un résidu de lysine acétyl couplé à un fluorophore CLIVABLES (p. ex., coumarine), par exemple N-(4-methyl-7-aminocoumarinyl)-Nα-(t-butoxycarbonyl)-Nω-acetyllysineamide (MAL)16. Pour distinguer entre les activités de l’isoforme spécifique, distincts acellulaire essais impliquant chaque isoforme sont nécessaires et peuvent ne pas refléter l’activité de l’isoforme réel dans les cellules vivantes. Substrats de l’isoforme spécifique sont disponibles dans le commerce, par exemple de benzyle (S)-[1-(4-methyl-2-oxo-2H-chromen-7-ylcarbamoyl)-5-propionylaminopentyl]carbamate (MOCPAC, HDAC1 substrat spécifique) et (ester d’acide tert-butyl S)-[5-acetylamino-1-(2-oxo-4-trifluoromethyl-2H-chromen-7-ylcarbamoyl)pentyl]carbamic (BATCP, HDAC6 substrat spécifique) (Figure 1 b). Cependant, un mélange de plusieurs substrat contenant du MAL, MOCPAC et BATCP donnée aux cellules vivantes ne permet la détection de chaque produit désacylés par mesurage fluorimétrique, étant donné qu’ils portent le même fluorophore CLIVABLES.
La méthode décrite ici permet la détection et la quantification relative de chacun des substrats et de son produit désacylé dans les cellules HeLa par dosage multi-substrat suivi UHPLC-ESI-MS/MS analyse17. Un dosage d’HDAC est mené sur les cellules HeLa pour permettre l’identification directe d’activité inhibitrice HDAC et de la spécificité des composés d’essai sur des HDACs endogènes. Il y a un accent sur HDAC1 et HDAC6, qui sont évaluées simultanément. Pour réaliser ces mesures enzymatiques dans un essai d’incubation unique, un mélange de substrats HDAC spécifiques et non spécifiques est ajouté aux cellules HeLa traitées et non traitées plaqués sur une plaque à 96 puits. Après une étape d’incubation, les cellules sont lysées pour libérer les substrats et leurs produits de réaction respectifs, qui sont séparés et détectés en utilisant une méthode UHPLC-MS (Figure 1). Les produits désacylés des substrats MAL, MOCPAC et BATCP sont les désacylés MAL (dMAL) et désacylé MOCPAC (dMOCPAC) désacylé BATCP (dBATCP), respectivement. Les courbes dose-réponse peuvent être construits avec des composés actifs.
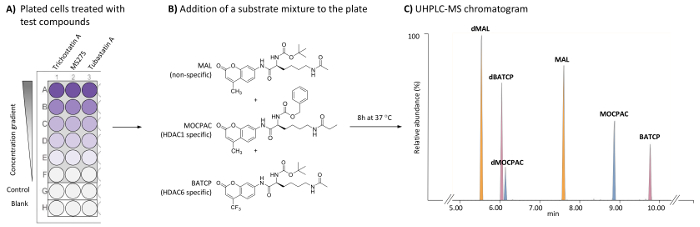
Figure 1 : schéma général pour le dosage des HDAC basés sur les cellules identifier les inhibiteurs spécifiques HDAC1 et HDAC6 par l’analyse UHPLC-SM de plusieurs substrats. (A) schéma d’une plaque 96 puits typique contenant traités (composés d’essai) et les cellules HeLa (témoin), ainsi que flans acellulaire. (B) la structure chimique des substrats ajouté sous forme d’un mélange (21 µM) à être désacétylé par HDACs endogènes. (C) UHPLC-MS typique chromatogramme montrant les pics des substrats ajoutés (MAL, MOCPAC et BATCP) et leurs produits désacylés (dMAL, dMOCPAC et dBACTP, respectivement). S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
Access restricted. Please log in or start a trial to view this content.
Protocole
1. Culture cellulaire
Remarque : Les étapes suivantes sont effectuées dans une hotte de vitroplants standard. Familiarité avec la technique de stérilisation est prévue.
- La culture de cellules HeLa à confluence de 80 % dans un flacon de culture cellulaire T75 dans MEM additionné de 10 % sérum de veau fœtal, la pénicilline G (100 U/mL) et à la streptomycine (100 mg/mL).
NOTE : Culture cellulaire, le traitement et mesures de lyse sont menées sous flux laminaire, avec les étapes d’incubation de cellules à 37 ° C dans une atmosphère humidifiée de 5 % de CO2 dans des conditions stériles. - Laver les cellules deux fois avec 5 mL de SPD, aspirer le SPD et ajouter 1 mL de réactif de dissociation cellulaire. Incuber à 37 ° C pendant 5 min détacher les cellules.
- Ajouter 9 mL de milieu de culture MEM, bien remettre en suspension la suspension cellulaire et compter les cellules à l’aide d’un hémocytomètre.
- Ajuster la densité de la suspension cellulaire à 6 x 104 cellules/mL dans MEM.
- Ajouter 100 µL de la suspension cellulaire au contrôle et essai des puits d’une plaque de 96 puits stérile, à fond plat, imprégnées de culture tissulaire (Figure 1 a).
- Ajouter 100 µL de milieu de culture MEM à vides puits de la plaque à 96 puits (Figure 1 a).
- Incuber à 37 ° C pendant 24 h la plaque à 96 puits.
Remarque : Une incubation de 24 h est un moment approprié pour tester l’activité HDAC endogène dans les cellules HeLa. Différents types de cellules ayant une activité HDAC inconnue peuvent besoin d’une évaluation timecourse des cellules contrôles.
2. cellule traitement avec des composés d’essai et substrats HDAC
- Aspirez le milieu provenant des puits de la plaque à 96 puits.
- Ajouter 25 µL de 2 x composés d’essai (p. ex., trichostatine A, MS275 et tubastatin A) en MEM pour tester des puits. Ajouter 25 µL de MEM pour contrôle et blancs.
Remarque : La concentration des composés d’essai doit être ajustée pour fournir une gamme de concentrations d’essai final au moins 5 (une concentration / puits). La concentration finale de DMSO dans la substance d’essai ne doit pas dépasser 0,5 % et devrait être le même dans chaque puits, y compris les blancs et les contrôles. Le volume final de l’incubation est 50 µL / puits. Trichostatine A est testé à 5 concentrations allant de 500 à 1,95 nM (dilution 1:4). MS275 et tubastatin A sont testés à 5 concentrations variant de 8 000 à 6.17 nM (dilution 1:6). La concentration finale souhaitée pour chaque substance d’essai doit être déterminée par chaque utilisateur individuel en créant une courbe dose-réponse. - Ajouter 25 µL du mélange substrat (42 µM de MAL, 42 µM de MOCPAC et 42 µM de BATCP en MEM) aux puits de contrôle et d’essai. Ajouter 25 µL de MEM aux puits vides.
Remarque : La concentration finale de chaque substrat est 21 µM. MOCPAC et BATCP faire la distinction entre activité HDAC1 et HDAC6, respectivement (Figure 1 b). Lorsque aucune activité n’est observée avec les substrats, l’utilisation du MAL pourrait entraîner l’identification de l’activité contre une autre isoforme HDAC. - Incuber la plaque 96 puits à 37 ° C pendant 8 h dans un humidifié 5 % CO2 incubateur.
NOTE : Ce temps d’incubation est adapté pour les cellules HeLa et permet l’HDAC endogène d’interagir avec les substrats synthétiques. Différentes lignées cellulaires peuvent nécessiter des ajustements dans la concentration de substrat et/ou de temps d’incubation.
3. cellule Lysis et préparation des échantillons pour UHPLC-ESI-MS/MS
ATTENTION : Les étapes de préparation d’échantillon utilisent des produits chimiques organiques, qui sont hautement inflammables et toxiques par ingestion ou inhalation. Porter une protection personnelle appropriée, comme des gants et des lunettes de sécurité.
- Ajouter 10 µL de 6 x RIPA tampon (dilué de 10 x RIPA tampon) additionné d’inhibiteur de la protéase dans chaque puits de la plaque à 96 puits.
- Arrêter la réaction en ajoutant 160 µL d’acétonitrile froide dans chaque puits et mélanger par pipetage de haut en bas.
Remarque : Maintenez l’acétonitrile à-20 ° C avant cette étape. - Placer la plaque dans un congélateur à-80 ° C pendant 10 min.
- Retirez la plaque du congélateur et transférer 220 µL du contenu de chaque puits d’une plaque à 96 puits non stérile, conique-fond (fond en V). Centrifuger la plaque à 5 000 x g et 4 ° C pendant 10 min.
Remarque : Cette étape vise à éliminer les protéines et les sels. Si nécessaire, les solutions peuvent être transférées à tubes à centrifuger coniques individuels avant la centrifugation ou filtrées à travers un ensemble de 96 puits, plaques de filtration 0,65 µm PVDF. La suppression réussie du précipité est nécessaire avant l’analyse UHPLC. - Transférer 200 µL de liquide surnageant (ou filtrat) dans une plaque à 96 puits compatible avec le système UHPLC et fermez-le hermétiquement avec du papier pelable, thermocollage, à l’aide d’un adhésif.
Remarque : Évitez de pipetage des granulés en enlevant les surnageants. Si l’analyse UHPLC ne peut pas être effectuée immédiatement, la plaque peut être stockée à 4 ° C pendant un maximum de 24h jusqu'à l’analyse.
4. UHPLC/MS-MS Analysis
- Préparer le système UHPLC en la remplissant avec le système de la phase mobile (95 % A:5 % B) :
R : H2O,-qualité HPLC, contenant 0,1 % d’acide formique (préparer 1 L)
B: acétonitrile, de qualité CLHP, contenant 0,1 % d’acide formique (préparer 1 L).
ATTENTION : La phase mobile contient des produits chimiques organiques, qui sont hautement inflammables et toxiques par ingestion ou inhalation. Porter une protection personnelle appropriée, comme des gants et des lunettes de sécurité. - Placer la plaque à 96 puits dans le gestionnaire de l’échantillon contenant un support de plaque. Exécuter les exemples dans les conditions de UHPLC-ESI-MS/MS prévues dans le tableau 1.
Remarque : Chaque colonne de la plaque à 96 puits analytique doit avoir bien un contrôle et un puits vide (tel que représenté dans le schéma de la plaque de la Figure 1 a). Les contrôles sont représentatifs de 100 % activité enzymatique dans la lignée de cellules testées, tandis que les blancs sont là pour vérifier si la source de MS fournit fond cohérent. Inhabituel MS pics détectés dans un espace donné devraient être étudiées pour éviter les changements de sensibilité MS.
5. analyse
- Intégrer la surface du pic de chacun des substrats et de son produit désacylé avec un logiciel d’intégration UHPLC approprié.
NOTE : Utiliser les paramètres d’intégration automatique avec une méthode de lissage (moyenne, taille de la fenêtre 3 et nombre de lisse 2, par exemple). En utilisant les conditions chromatographiques fournies dans le tableau 1, l’ordre d’élution est dMAL (5,8 min), dBATCP (min 6,0), dMOCPAC (min 6,2), MAL (7,6 min), MOCPAC (8,9 min) et BATCP (9,8 min), comme illustré à la Figure 1. - Pour chaque substrat, calculer le rapport d’aire de pic (désacétylé/acétylé) dans chaque chromatogramme (échantillons de test et de contrôle).
- Calculer le pourcentage d’inhibition HDAC de chaque échantillon :
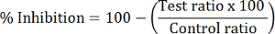
Remarque : HDAC1 l’inhibition est calculée selon le rapport d’aire de pic de dMOCPAC/MOCPAC ; Inhibition de HDAC6 est calculée avec dBATCP/BATCP. Le ratio dMAL/MAL peut être utilisé pour exprimer l’inhibition HDAC générale.
Access restricted. Please log in or start a trial to view this content.
Résultats
Pour illustrer l’application de la méthode pour identifier des inhibiteurs non sélectifs et sélectifs pour HDAC1 (classe I) et HDAC6 (classe IIb), les cellules HeLa ont été traités avec des composés standards connus : trichostatine A (non sélectif entre HDAC1 et HDAC6)18, MS275 (inhibiteur sélectif HDAC1)19et tubastatin (inhibiteur sélectif de HDAC6)20. À l’aide de la méthode actuelle (
Access restricted. Please log in or start a trial to view this content.
Discussion
HDAC l’inhibition est un sujet brûlant dans la découverte de médicaments, avec un accent mis sur les inhibiteurs sélectifs HDAC6 pour cancer thérapie21. Sélectivité des HDAC est habituellement évaluée par une série d’épreuves de haut-débit, acellulaire, qui ont l’intention de déterminer la puissance inhibitrice vers individuels HDAC isoformes21. Cependant, la sélectivité d’un inhibiteur doit être confirmée par ailleurs dans les cellules vivantes en ...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Les auteurs remercient coût Action CM1406 (biologie chimique épigénétique). Recherche rapporté dans cette publication a été financée par la Fondation Pierre Mercier.
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| BATCP | Sigma-Aldrich | B4061 | |
| MAL | Sigma-Aldrich | SCP0168 | Synonym: BOC-Ac-Lys-AMC |
| MOCPAC | Sigma-Aldrich | M2195 | |
| MS275 | Sigma-Aldrich | EPS002 | |
| Trichostatin A | Sigma-Aldrich | T8552 | |
| Tubastatin A | Sigma-Aldrich | SML0044 | |
| Acetonitrile, HPLC grade | Fisher Scientific | 10660131 | |
| Formic acid, LC/MS grade | Fisher Scientific | 10596814 | |
| H2O, HPLC grade | distilled H2O filtered through a Milli-Q purification system | ||
| HeLa cells | ATCC | ATCC CRM-CCL-2 | |
| Cell dissociation reagent TrypLE Express | ThermoFisher Scientific | 12604013 | |
| DMSO, cell culture grade | Applichem | 146463 | |
| DPBS | ThermoFisher Scientific | 14190144 | |
| Fetal bovine serum | Biowest | S1810 | |
| MEM | ThermoFisher Scientific | 22561021 | |
| Penicillin-Streptomycin | Bioconcept | 4-01F00-H | |
| 10x RIPA buffer | Abcam | ab156034 | |
| SigmaFast protease inhibitor tablets | Sigma-Aldrich | S8820 | |
| 96-well plates, sterile, flat-bottom, tissue culture treated Corning | VWR | 29442-058 | |
| 96-well plates, non-sterile, V-bottom Corning | VWR | 29442-404 | used in the centrifugation step |
| 96-well plate, conical bottom, Nunc | ThermoFisher Scientific | 249944 | compatible with the Acquity UHPLC system |
| T75 cell culture flasks Corning | Sigma-Aldrich | CLS430641 | |
| Peelable heat sealing foil | Waters | 186002789 | |
| Acquity UPLC system | Waters | ||
| Eppendorf Centrifuge 5810 R | Fisher Scientific | 05-413-323 | |
| Integration software: MassLynx V4.1 | Waters | Catalog number not available | |
| Combi thermo-sealer SP-0669/240 | Waters | Catalog number not available | |
| Quattro micro API Tandem Quadrupole System | Waters | Catalog number not available |
Références
- Arrowsmith, C. H., Bountra, C., Fish, P. V., Lee, K., Schapira, M. Epigenetic protein families: a new frontier for drug discovery. Nat Rev Drug Discov. 11 (5), 384-400 (2012).
- Henikoff, S., Greally, J. M. Epigenetics, cellular memory and gene regulation. Curr Biol. 26 (14), R644-R648 (2016).
- Kim, C., et al. HDAC6 inhibitor blocks amyloid beta-induced impairment of mitochondrial transport in hippocampal neurons. PLoS One. 7 (8), (2012).
- Kawaguchi, Y., et al. The deacetylase HDAC6 regulates aggresome formation and cell viability in response to misfolded protein stress. Cell. 115 (6), 727-738 (2003).
- Zhao, Y., et al. Acetylation of p53 at lysine 373/382 by the histone deacetylase inhibitor depsipeptide induces expression of p21(Waf1/Cip1). Mol Cell Biol. 26 (7), 2782-2790 (2006).
- Berger, S. L. Histone modifications in transcriptional regulation. Curr Opin Genet Dev. 12 (2), 142-148 (2002).
- Simões-Pires, C., et al. HDAC6 as a target for neurodegenerative diseases: what makes it different from the other HDACs? Mol Neurodegener. 8 (7), 1-16 (2013).
- Mottamal, M., Zheng, S., Huang, T. L., Wang, G. Histone deacetylase inhibitors in clinical studies as templates for new anticancer agents. Molecules. 20 (3), 3898-3941 (2015).
- Manal, M., Chandrasekar, M. J., Gomathi Priya, J., Nanjan, M. J. Inhibitors of histone deacetylase as antitumor agents: A critical review. Bioorg Chem. 67, 18-42 (2016).
- Mack, G. S. To selectivity and beyond. Nat Biotech. 28 (12), 1259-1266 (2010).
- U.S. National Institutes of Health. Study of ACY-1215 alone and in combination with bortezomib and dexamethasone in multiple myeloma (ACY-1215). , Available from: https://clinicaltrials.gov/ct2/show/NCT01323751 (1211).
- U.S. National Institutes of Health. Study of ACY-1215 in combination with lenalidomide, and dexamethasone in multiple myeloma. , Available from: https://clinicaltrials.gov/ct2/show/NCT01583283 (2012).
- U.S. National Institutes of Health. 13ACY-1215 (ricolinostat) in combination with pomalidomide and low-dose dex in relapsed-and-refractory multiple myeloma. , Available from: https://clinicaltrials.gov/ct2/show/NCT01997840 (2013).
- U.S. National Institutes of Health. Study of ACY-241 alone and in combination with pomalidomide and dexamethasone in multiple myeloma. , Available from: https://clinicaltrials.gov/ct2/show/NCT02400242 (2015).
- Yee, A. J., et al. Ricolinostat plus lenalidomide, and dexamethasone in relapsed or refractory multiple myeloma: a multicentre phase 1b trial. Lancet Oncol. 17 (11), 1569-1578 (2016).
- Jung, M. Homogenous non-isotopic assays for histone deacetylase activity. Expert Opin Ther Pat. 13 (6), 935(2003).
- Zwick, V., Simões-Pires, C., Cuendet, M. Cell-based multi-substrate assay coupled to UHPLC-ESI-MS/MS for a quick identification of class-specific HDAC inhibitors. J Enzyme Inhibi Med Chem. 31 (1), 209-214 (2016).
- Khan, N., et al. Determination of the class and isoform selectivity of small-molecule histone deacetylase inhibitors. Biochem J. 409 (2), 581-589 (2008).
- Glaser, K. B., et al. Differential protein acetylation induced by novel histone deacetylase inhibitors. Biochem Biophys Res Commun. 325 (3), 683-690 (2004).
- Butler, K. V., et al. Rational design and simple chemistry yield a superior, neuroprotective HDAC6 inhibitor, tubastatin A. J Am Chem Soc. 132 (31), 10842-10846 (2010).
- Jung, M., Yong, K. -J., Velena, A., Lee, S. Cell-Based Assays for HDAC Inhibitor Hit Validation. Epigenetic Targets in Drug Discovery. Sippl, W., Jung, M. , Wiley-VCH. (2010).
- Milli, A., et al. Proteomic analysis of cellular response to novel proapoptotic agents related to atypical retinoids in human IGROV-1 ovarian carcinoma cells. J Proteome Res. 10 (3), 1191-1207 (2011).
- Zwick, V., et al. Synthesis of a selective HDAC6 inhibitor active in neuroblasts. Bioorg Med Chem Lett. 26 (20), 4955-4959 (2016).
- Ciossek, T., Julius, H., Wieland, H., Maier, T., Beckers, T. A homogeneous cellular histone deacetylase assay suitable for compound profiling and robotic screening. Anal Biochem. 372 (1), 72-81 (2008).
- Heltweg, B., Dequiedt, F., Marshall, B. L., Brauch, C., Yoshida, M., et al. Subtype selective substrates for histone deacetylases. J Med Chem. 47 (21), 5235-5243 (2004).
- Copeland, R. A. Evaluation of enzyme inhibitors in drug discovery. , Wiley. (2005).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon