È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Misura simultanea di HDAC1 e HDAC6 attività in HeLa Cells utilizzando UHPLC-MS
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Il presente metodo serve a identificare isoforma-specifici inibitori dell'istone deacetilasi (HDAC) in cellule HeLa dall'analisi UHPLC-MS di substrati multipli. Questo è un metodo privo di anticorpi sviluppato per riflettere l'attività HDAC1 e HDAC6 del vita ambiente cellulare, in contrasto con le analisi senza cellula singola isoforma.
Abstract
La ricerca di nuovi inibitori delle istone deacetilasi (HDAC) è di crescente interesse nella scoperta della droga. Isoforma selettività è stato sotto i riflettori dopo l'approvazione del romidepsina, una classe ho HDAC inibitore per la terapia del cancro e l'indagine clinica degli inibitori HDAC6-specifico per il mieloma multiplo. Il presente metodo è utilizzato per determinare l'attività inibitoria di sostanze da testare su HDAC1 e HDAC6 nelle cellule. L'attività dell'isoforma è misurata usando la cromatografia liquida ultra-high-performance – analisi di spettrometria di massa (UHPLC-MS) di specifici substrati incubati con cellule HeLa trattate e non trattate. Il metodo ha il vantaggio di riflettere l'attività HDAC endogena all'interno dell'ambiente di cella, in contrasto con analisi senza cellula biochimiche condotte su isoforme isolato. Inoltre, perché si basa sulla quantificazione dei substrati sintetici, il metodo non richiede il riconoscimento anticorpale di proteine endogene acetilati. È facilmente adattabile a diverse linee cellulari e un processo automatizzato. Il metodo ha già dimostrato utile nella ricerca di composti HDAC6-selettiva in neuroblasti. Risultati rappresentativi sono indicati qui con standard HDAC inibitori tricostatina A (non-specifico), MS275 (HDAC1-specifica), e tubastatin un (HDAC6-specifico) utilizzando cellule HeLa.
Introduzione
HDAC appartengono ad una famiglia di enzimi in grado di deacetilano gli istoni all'interno della struttura della cromatina. Hanno anche altri substrati della proteina nel citosol e si trovano in vari compartimenti cellulari. Un totale di 18 HDAC isoforme sono state identificate finora e sono stati collegati con parecchi meccanismi delle cellule, compreso la regolazione di fattori di trascrizione e l'espressione genica, come pure la segnalazione delle cellule e trasporto1,2,3,4,5,6,7. Inibitori HDAC catalitici sono emersi come terapeutici potenziali farmaci per la terapia del cancro. La maggior parte delle HDAC inibitori attualmente approvati dalla FDA sono per il trattamento di linfoma a cellula T ed il mieloma multiplo e sono inibitori HDAC aspecifici come vorinostat (SAHA), belinostat e panobinostat8,9. Tuttavia, una serie di effetti collaterali sono stati associati con gli inibitori di pan, e la ricerca di piccole molecole isoforma-specifici è un tema caldo nella scoperta di droga e chimica medicinale. Di conseguenza, la classe ho romidepsina inibitore selettivo (HDAC1-3 e HDAC8) è un farmaco già approvato10, mentre gli inibitori HDAC6-specific sono attualmente oggetto di studi clinici, con maggiore potenziale terapeutico in mieloma multiplo11,12,13,14,15.
Lo screening di saggi per caratterizzare HDAC inibitori sono basati sull'incubazione di un substrato HDAC con un'origine enzimatica (singola isoforma, estratto nucleare o lysate delle cellule). Il substrato è solitamente una sequenza del peptide di piccole dimensioni contenente un residuo di lisina acetil accoppiato ad un fluoroforo spaccabili (ad es., cumarina), ad esempio N-(4-methyl-7-aminocoumarinyl)-Nα-(t-butoxycarbonyl)-Nω-acetyllysineamide (MAL)16. Per distinguere tra attività isoforma-specifici, analisi senza cellula separate che coinvolgono ciascuna isoforma sono necessari e potrebbero non riflettere l'attività dell'isoforma reale in cellule viventi. Isoforma-specifici substrati sono commercialmente disponibili, come benzilico (S)-[1-(4-methyl-2-oxo-2H-chromen-7-ylcarbamoyl)-5-propionylaminopentyl]carbamate (MOCPAC, HDAC1 specifico substrato) e (S)-[5-acetylamino-1-(2-oxo-4-trifluoromethyl-2H-chromen-7-ylcarbamoyl)pentyl]carbamic acid tert-butile (BATCP, HDAC6 substrato specifico) (Figura 1B). Tuttavia, una miscela di multi-substrata contenente MAL, MOCPAC e BATCP dato alle cellule viventi non permetterà la rilevazione dei singoli prodotti deacetylated da fluorometrica misurazione, dato che essi portano il fluoroforo spaccabili stesso.
Il metodo qui descritto consente la rilevazione e quantificazione relativa di ogni substrato ed il suo prodotto deacetylated in cellule HeLa usando un'analisi multi-substrata seguita da UHPLC-ESI-MS/MS analisi17. Un'analisi HDAC è condotto su cellule HeLa per consentire l'identificazione diretta di attivita ' inibitoria e della specificità della prova composti endogeni HDACs. C'è un focus su HDAC1 e HDAC6, che vengono valutati contemporaneamente. Per ottenere queste misurazioni enzimatiche in un'analisi di incubazione singola, una miscela di substrati HDAC aspecifici e specifici è aggiunto ai trattati e non trattate cellule HeLa placcate su una piastra a 96 pozzetti. A seguito di una fase di incubazione, le cellule vengono lisate per rilasciare i substrati ed i loro prodotti di reazione rispettiva, che sono separati e rilevato usando un metodo UHPLC-MS (Figura 1). I prodotti deacetylated dei substrati MAL, MOCPAC e BATCP sono il MAL deacetylated (dMAL), deacetylated MOCPAC (dMOCPAC) e deacetylated BATCP (dBATCP), rispettivamente. Curve dose-risposta possono essere costruite con composti attivi.
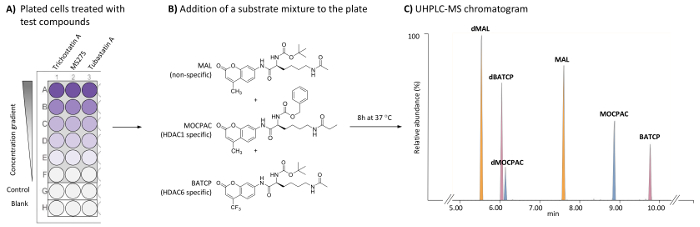
Figura 1: schema generale per il saggio HDAC basati su celle a identificare gli inibitori specifici HDAC1 e HDAC6 dall'analisi dei substrati multipli UHPLC-MS. (A) schema di una tipica piastra a 96 pozzetti contenenti trattati (test composti) e cellule HeLa non trattati (controllo), nonché spazi vuoti senza cellula. (B) struttura chimica dei substrati aggiunti come una miscela (21 µM ciascuna) per essere viene desacetilato agli acidi di HDACs endogeno. (C) UHPLC-MS tipico cromatogramma mostrando le cime dei substrati aggiunto (MAL, MOCPAC e BATCP) e loro prodotti deacetylated (dMAL, dMOCPAC e dBACTP, rispettivamente). Clicca qui per visualizzare una versione più grande di questa figura.
Access restricted. Please log in or start a trial to view this content.
Protocollo
1. coltura cellulare
Nota: I seguenti passaggi vengono eseguiti in una cappa di coltura di tessuti standard. Familiarità con tecnica sterile è previsto.
- Coltura di cellule HeLa al confluency di 80% in un matraccio di cultura cellulare T75 in MEM completati con 10% siero bovino fetale, penicillina G (100 U/mL) e la streptomicina (100 mg/mL).
Nota: Cultura, il trattamento e passaggi di lisi delle cellule sono condotti sotto flusso laminare, con fasi di incubazione delle cellule a 37 ° C, eseguite in atmosfera umidificata di 5% CO2 in condizioni sterili. - Lavare le cellule due volte con 5 mL di DPBS, aspirare il DPBS e aggiungere 1 mL di reagente di dissociazione delle cellule. Incubare a 37 ° C per 5 min staccare le cellule.
- Aggiungere 9 mL di terreno di coltura MEM, accuratamente Risospendere la sospensione cellulare e contare le celle utilizzando un emocitometro.
- Regolare la densità della sospensione delle cellule a 6 x 104 cellule/mL in MEM.
- Aggiungere 100 µ l di sospensione cellulare di controllo e test dei pozzetti di una piastra a 96 pozzetti sterile, fondo piatto, coltura del tessuto-trattati (Figura 1A).
- Aggiungere 100 µ l di terreno di coltura MEM a vuoti pozzetti della piastra 96 pozzetti (Figura 1A).
- Incubare la piastra a 96 pozzetti a 37 ° C per 24 h.
Nota: Un'incubazione di 24 ore è un tempo adatto per verificare l'attività endogena di HDAC in cellule HeLa. Diversi tipi di cellule con attività di HDAC sconosciuto potrebbero essere necessario una valutazione timecourse delle cellule di controllo.
2. cell trattamento con sostanze da testare e substrati HDAC
- Aspirare il mezzo dai pozzetti della piastra 96 pozzetti.
- Aggiungere 25 µ l di sostanze da testare: 2x (ad es., tricostatina A, MS275 e tubastatin A) in MEM di pozzi di prova. Aggiungere 25 µ l di MEM per controllo e pozzetti del bianco.
Nota: La concentrazione delle sostanze da testare è quello di essere regolato per fornire un intervallo di almeno 5 concentrazioni di prova finale (una concentrazione per pozzetto). La concentrazione finale di DMSO nel composto in esame non dovrebbe superare lo 0,5% e dovrebbe essere la stessa in ciascun pozzetto, compresi spazi vuoti e i controlli. Il volume di incubazione finale è di 50 µ l per pozzetto. Trichostatin A è stato testato in 5 concentrazioni che variano da 500 a 1,95 nM (diluizione 1:4). MS275 e tubastatin A sono testati a 5 concentrazioni che variano da 8.000 a 6,17 nM (diluizione 1:6). La concentrazione di prova finale desiderata per ogni sostanza in esame dovrebbe essere determinata da ogni singolo utente creando una curva dose-risposta. - Aggiungere 25 µ l della miscela substrato (42 µM di MAL, 42 µM di MOCPAC e 42 µM di BATCP in MEM) nei pozzetti di controllo e prova. Aggiungere 25 µ l di MEM per pozzetti del bianco.
Nota: La concentrazione finale di ogni substrato è 21 µM. MOCPAC e BATCP distinguere tra attività HDAC1 e HDAC6, rispettivamente (Figura 1B). Quando nessuna attività è osservata con quei substrati, l'uso di MAL potenzialmente potrebbe portare all'identificazione di attività contro un'altra isoforma HDAC. - Incubare la piastra a 96 pozzetti a 37 ° C per 8 h in un 5% umidificata incubatore a CO2 .
Nota: Questo tempo di incubazione è adatto per le cellule HeLa e permette le HDAC endogena di interagire con i substrati sintetici. Diverse linee cellulari possono richiedere adattamenti nella concentrazione di substrato e/o di tempo di incubazione.
3. cellula Lisi e preparazione del campione per UHPLC-ESI-MS/MS
Attenzione: La procedura di preparazione del campione utilizza prodotti chimici organici, che sono altamente infiammabile e tossico per ingestione o inalazione. Indossare adeguata protezione personale, come guanti e occhiali di sicurezza.
- Aggiungere 10 µ l di buffer di x RIPA 6 (diluito da 10 x RIPA buffer) completati con inibitore della proteasi in ciascun pozzetto della piastra 96 pozzetti.
- Bloccare la reazione aggiungendo 160 µ l di acetonitrile freddo in ciascun pozzetto e Miscelare pipettando su e giù.
Nota: Tenere l'acetonitrile a-20 ° C prima di questo passaggio. - Posizionare la piastra in un congelatore-80 ° C per 10 min.
- Rimuovere il piatto dal freezer e trasferire 220 µ l del contenuto di ciascun pozzetto di una piastra a 96 pozzetti non sterili, fondo conico (fondo V). Centrifugare la piastra a 5.000 x g e a 4 ° C per 10 min.
Nota: Questo passaggio mira a rimuovere le proteine e sali. Se necessario, le soluzioni possono essere trasferite ai tubi singoli conica per centrifuga prima della centrifugazione o filtrate attraverso una serie di 96 pozzetti, piastre filtranti di 0,65 µm PVDF. La rimozione di successo del precipitato è richiesta prima dell'analisi UHPLC. - Trasferire 200 µ l di supernatante (o filtrato) in una piastra a 96 pozzetti compatibile con il sistema UHPLC e sigillare con un foglio pelabile, termosaldatura, utilizzando un foglio sigillante.
Nota: Evitare di pipettaggio il pellet quando si rimuove i sovranatante. Se l'analisi UHPLC non può essere eseguita immediatamente, la piastra possa essere memorizzata a 4 ° C per un massimo di 24 h fino all'analisi.
4. UHPLC/MS-MS Analysis
- Preparare il sistema UHPLC riempiendolo con il sistema della fase mobile (95% A:5% B):
R: H2O, HPLC-grado, contenente acido formico 0.1% (preparare 1 L)
B: Acetonitrile, HPLC-grado, contenente acido formico 0.1% (preparare 1L).
Attenzione: La fase mobile contiene prodotti chimici organici, che sono altamente infiammabile e tossico per ingestione o inalazione. Indossare adeguata protezione personale, come guanti e occhiali di sicurezza. - La gestione di esempio contenente un portatarga, inserire la piastra a 96 pozzetti. Eseguire i campioni secondo le condizioni di UHPLC-ESI-MS/MS indicati nella tabella 1.
Nota: Ogni colonna della piastra 96 pozzetti analitica dovrebbe avere un controllo del bene ed un pozzetto vuoto (come raffigurato nello schema di Figura 1Apiastra). I controlli sono rappresentante del 100% di attività enzimatica nella linea cellulare testata, mentre gli spazii in bianco sono lì per verificare se l'origine di MS stia fornendo sfondo coerenza. Insolito MS picchi rilevati in un determinato campo vuoto dovrebbero essere studiati per impedire i cambiamenti di sensibilità di MS.
5. analisi dei dati
- Integrare l'area del picco di ogni substrato ed il suo prodotto deacetylated con un software di integrazione UHPLC appropriato.
Nota: Utilizzare i parametri di integrazione automatica con un metodo smoothing (media, dimensione della finestra 3 e numero di liscio 2, ad esempio). Utilizzando le condizioni cromatografiche indicate nella tabella 1, l'ordine di eluizione è dMAL (5,8 min), dBATCP (6,0 min), dMOCPAC (6,2 min), MAL (7,6 min), MOCPAC (8,9 min) e BATCP (9,8 min), come illustrato nella Figura 1. - Per ogni substrato, calcolare il rapporto tra area di picco (viene desacetilato agli acidi/acetilato) in ogni cromatogramma (campioni di controllo e test).
- Calcolare l'inibizione di HDAC percentuale di ciascun campione da testare:
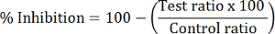
Nota: HDAC1 inibizione viene calcolata utilizzando il rapporto tra area di picco di dMOCPAC/MOCPAC; HDAC6 inibizione viene calcolata con dBATCP/BATCP. Il rapporto dMAL/MAL può essere utilizzato per esprimere generale inibizione di HDAC.
Access restricted. Please log in or start a trial to view this content.
Risultati
Per illustrare l'applicazione del metodo all'identificazione di inibitori selettivi e non selettivi per HDAC1 (classe I) e HDAC6 (classe IIb), le cellule HeLa sono state trattate con composti standard conosciuti: tricostatina A (non-selettivi tra HDAC1 e HDAC6)18, MS275 (HDAC1-selettivo inibitore)19e tubastatin (HDAC6-selettivo inibitore)20. Utilizzando il presente metodo (Figura 1), valo...
Access restricted. Please log in or start a trial to view this content.
Discussione
Inibizione di HDAC è un tema caldo nella scoperta della droga, con un focus corrente su HDAC6-selettivi per cancro terapia21. Selettività HDAC solitamente è valutata da una serie di saggi di alto-rendimento, senza cellula, che intende determinare la potenza inibitoria verso singoli HDAC isoforme21. Tuttavia, la selettività di un inibitore deve essere ulteriormente confermata in cellule viventi valutando lo stato di acetilazione di substrati di proteine endogene, quali g...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Gli autori riconoscono COST Action CM1406 (epigenetici Chemical Biology). Ricerca riportata in questa pubblicazione è stata sostenuta dalla Fondazione Pierre Mercier.
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| BATCP | Sigma-Aldrich | B4061 | |
| MAL | Sigma-Aldrich | SCP0168 | Synonym: BOC-Ac-Lys-AMC |
| MOCPAC | Sigma-Aldrich | M2195 | |
| MS275 | Sigma-Aldrich | EPS002 | |
| Trichostatin A | Sigma-Aldrich | T8552 | |
| Tubastatin A | Sigma-Aldrich | SML0044 | |
| Acetonitrile, HPLC grade | Fisher Scientific | 10660131 | |
| Formic acid, LC/MS grade | Fisher Scientific | 10596814 | |
| H2O, HPLC grade | distilled H2O filtered through a Milli-Q purification system | ||
| HeLa cells | ATCC | ATCC CRM-CCL-2 | |
| Cell dissociation reagent TrypLE Express | ThermoFisher Scientific | 12604013 | |
| DMSO, cell culture grade | Applichem | 146463 | |
| DPBS | ThermoFisher Scientific | 14190144 | |
| Fetal bovine serum | Biowest | S1810 | |
| MEM | ThermoFisher Scientific | 22561021 | |
| Penicillin-Streptomycin | Bioconcept | 4-01F00-H | |
| 10x RIPA buffer | Abcam | ab156034 | |
| SigmaFast protease inhibitor tablets | Sigma-Aldrich | S8820 | |
| 96-well plates, sterile, flat-bottom, tissue culture treated Corning | VWR | 29442-058 | |
| 96-well plates, non-sterile, V-bottom Corning | VWR | 29442-404 | used in the centrifugation step |
| 96-well plate, conical bottom, Nunc | ThermoFisher Scientific | 249944 | compatible with the Acquity UHPLC system |
| T75 cell culture flasks Corning | Sigma-Aldrich | CLS430641 | |
| Peelable heat sealing foil | Waters | 186002789 | |
| Acquity UPLC system | Waters | ||
| Eppendorf Centrifuge 5810 R | Fisher Scientific | 05-413-323 | |
| Integration software: MassLynx V4.1 | Waters | Catalog number not available | |
| Combi thermo-sealer SP-0669/240 | Waters | Catalog number not available | |
| Quattro micro API Tandem Quadrupole System | Waters | Catalog number not available |
Riferimenti
- Arrowsmith, C. H., Bountra, C., Fish, P. V., Lee, K., Schapira, M. Epigenetic protein families: a new frontier for drug discovery. Nat Rev Drug Discov. 11 (5), 384-400 (2012).
- Henikoff, S., Greally, J. M. Epigenetics, cellular memory and gene regulation. Curr Biol. 26 (14), R644-R648 (2016).
- Kim, C., et al. HDAC6 inhibitor blocks amyloid beta-induced impairment of mitochondrial transport in hippocampal neurons. PLoS One. 7 (8), (2012).
- Kawaguchi, Y., et al. The deacetylase HDAC6 regulates aggresome formation and cell viability in response to misfolded protein stress. Cell. 115 (6), 727-738 (2003).
- Zhao, Y., et al. Acetylation of p53 at lysine 373/382 by the histone deacetylase inhibitor depsipeptide induces expression of p21(Waf1/Cip1). Mol Cell Biol. 26 (7), 2782-2790 (2006).
- Berger, S. L. Histone modifications in transcriptional regulation. Curr Opin Genet Dev. 12 (2), 142-148 (2002).
- Simões-Pires, C., et al. HDAC6 as a target for neurodegenerative diseases: what makes it different from the other HDACs? Mol Neurodegener. 8 (7), 1-16 (2013).
- Mottamal, M., Zheng, S., Huang, T. L., Wang, G. Histone deacetylase inhibitors in clinical studies as templates for new anticancer agents. Molecules. 20 (3), 3898-3941 (2015).
- Manal, M., Chandrasekar, M. J., Gomathi Priya, J., Nanjan, M. J. Inhibitors of histone deacetylase as antitumor agents: A critical review. Bioorg Chem. 67, 18-42 (2016).
- Mack, G. S. To selectivity and beyond. Nat Biotech. 28 (12), 1259-1266 (2010).
- U.S. National Institutes of Health. Study of ACY-1215 alone and in combination with bortezomib and dexamethasone in multiple myeloma (ACY-1215). , Available from: https://clinicaltrials.gov/ct2/show/NCT01323751 (1211).
- U.S. National Institutes of Health. Study of ACY-1215 in combination with lenalidomide, and dexamethasone in multiple myeloma. , Available from: https://clinicaltrials.gov/ct2/show/NCT01583283 (2012).
- U.S. National Institutes of Health. 13ACY-1215 (ricolinostat) in combination with pomalidomide and low-dose dex in relapsed-and-refractory multiple myeloma. , Available from: https://clinicaltrials.gov/ct2/show/NCT01997840 (2013).
- U.S. National Institutes of Health. Study of ACY-241 alone and in combination with pomalidomide and dexamethasone in multiple myeloma. , Available from: https://clinicaltrials.gov/ct2/show/NCT02400242 (2015).
- Yee, A. J., et al. Ricolinostat plus lenalidomide, and dexamethasone in relapsed or refractory multiple myeloma: a multicentre phase 1b trial. Lancet Oncol. 17 (11), 1569-1578 (2016).
- Jung, M. Homogenous non-isotopic assays for histone deacetylase activity. Expert Opin Ther Pat. 13 (6), 935(2003).
- Zwick, V., Simões-Pires, C., Cuendet, M. Cell-based multi-substrate assay coupled to UHPLC-ESI-MS/MS for a quick identification of class-specific HDAC inhibitors. J Enzyme Inhibi Med Chem. 31 (1), 209-214 (2016).
- Khan, N., et al. Determination of the class and isoform selectivity of small-molecule histone deacetylase inhibitors. Biochem J. 409 (2), 581-589 (2008).
- Glaser, K. B., et al. Differential protein acetylation induced by novel histone deacetylase inhibitors. Biochem Biophys Res Commun. 325 (3), 683-690 (2004).
- Butler, K. V., et al. Rational design and simple chemistry yield a superior, neuroprotective HDAC6 inhibitor, tubastatin A. J Am Chem Soc. 132 (31), 10842-10846 (2010).
- Jung, M., Yong, K. -J., Velena, A., Lee, S. Cell-Based Assays for HDAC Inhibitor Hit Validation. Epigenetic Targets in Drug Discovery. Sippl, W., Jung, M. , Wiley-VCH. (2010).
- Milli, A., et al. Proteomic analysis of cellular response to novel proapoptotic agents related to atypical retinoids in human IGROV-1 ovarian carcinoma cells. J Proteome Res. 10 (3), 1191-1207 (2011).
- Zwick, V., et al. Synthesis of a selective HDAC6 inhibitor active in neuroblasts. Bioorg Med Chem Lett. 26 (20), 4955-4959 (2016).
- Ciossek, T., Julius, H., Wieland, H., Maier, T., Beckers, T. A homogeneous cellular histone deacetylase assay suitable for compound profiling and robotic screening. Anal Biochem. 372 (1), 72-81 (2008).
- Heltweg, B., Dequiedt, F., Marshall, B. L., Brauch, C., Yoshida, M., et al. Subtype selective substrates for histone deacetylases. J Med Chem. 47 (21), 5235-5243 (2004).
- Copeland, R. A. Evaluation of enzyme inhibitors in drug discovery. , Wiley. (2005).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon