Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
El ensayo de secuestro del lactato deshidrogenasa, Es un método Simple y confiable para determinar la actividad de autofagia secuestro a granel en células de mamíferos
* Estos autores han contribuido por igual
En este artículo
Resumen
Aquí se describe un protocolo simple y bien validado para medir la actividad de autofagia secuestro a granel en células de mamíferos. El método se basa en cuantificar la proporción de lactato deshidrogenasa (LDH) en fracciones celulares de sedimentables en comparación con los niveles de LDH celulares total.
Resumen
A granel la autofagia se caracteriza por el secuestro de grandes porciones de citoplasma en estructuras de doble/múltiple-membrane denominado autophagosomes. Aquí se describe un protocolo simple para controlar este proceso. Además, se proporcionan resultados típicos y validación experimental del método bajo condiciones de inducción autofagia en varios tipos de células mamíferos cultivadas. Durante autofagia a granel, autophagosomes secuestrar citosol y así también soluble proteínas citosólicas, junto a otras cargas de autofagia. LDH es estable y muy abundante, soluble enzima citosólica que selectivamente no es secuestrado en autophagosomes. La cantidad de secuestro de LDH por lo tanto refleja la cantidad de secuestro autofagia a granel. Determinar eficientemente y exactamente secuestro de LDH en las células, empleamos un protocolo basado en electrodisruption fraccionamiento que separa efectivamente sedimentables de LDH citosólica, seguido por la medida de la actividad enzimática en sedimentables fracciones versus muestras celulares. Secuestro de autofagia se determina restando la proporción de sedimentables LDH en las células no tratadas de eso en células tratadas. La ventaja de la prueba de captura de LDH es que da una medida cuantitativa de la autofagia secuestro de carga endógena, en comparación con otros métodos que o bien implican expresión ectópica de sondas de captura o proteasa semi-cuantitativo Análisis de marcadores de autofagia o receptores de protección.
Introducción
Autofagia (griego para "la comer") es un proceso evolutivo conservado vacuolar/lysosomal degradación de material intracelular. Al descubrimiento de los genes relacionados con la autofagia ("ATG"), que son importantes para la autofagia en levaduras y humanos, y la realización que la autofagia juega un papel importante en la salud y enfermedad (reconocido por el Premio Nobel en medicina o fisiología a 2016 Yoshinori Oshumi), autofagia ha convertido rápidamente en uno de los procesos más intensamente estudiados en biología de la célula1,2.
Macroautophagy (en lo sucesivo, "autofagia") es caracterizado por la expansión y plegamiento de cisternas de membrana intracelular ("phagophores") en estructuras selladas, membrane doble o múltiple ("autophagosomes") que secuestran eficazmente la enwrapped material del resto del citoplasma. Sobre fusión de autophagosomes con lisosomas, la membrana autophagosomal interna y la carga secuestrada es degradado y reciclado. Autophagosomes puede secuestrar material citoplásmico en aleatorizada (no-selectivas autofagia) y modales de selectivo (selectivas autofagia). A granel autofagia probablemente representa una mezcla de la autofagia no selectivo y selectiva.
En los años 60 y 70 ("la era morfológico" de la investigación de autofagia), secuestro de autofagia se evaluó principalmente a través de análisis ultraestructurales. En la década de 1980 y principios de la década de 1990 ("la era bioquímica") por Seglen y compañeros de trabajo — que estudió autofagia en hepatocitos primarios de rata — desarrollaron los primeros métodos para medir cuantitativamente la autofagia secuestro actividad3. Mediante estos ensayos, Seglen define y caracteriza los diferentes pasos de la vía autofagia lisosomal4,5, descubierto acuñó el amphisome6 (el producto de la fusión del endosome-autophagosome) y fue el primero en describir el papel de la fosforilación de la proteína en autofagia Reglamento7. Sin embargo, tras el descubrimiento de los ATGs en 1990 ("la era molecular") y la primera caracterización de una proteína de ATG8 mamífera, proteína asociada a microtúbulos 1A/1B-luz cadena 3 (LC3) en 20008, el uso de proteínas ATG como marcadores para la proceso de autofagia rápidamente ganó popularidad, y el más viejos y más laboriosos métodos bioquímicos se quedaron atrás. De hecho, durante los últimos 18 años, western blot y análisis de microscopía de fluorescencia de LC3 se han convertido en el más popular (y en muchos casos el único) medio de estudio de la autofagia en las células mamíferas. La ventaja es la facilidad relativa por la cual estos métodos pueden llevar a cabo. La desventaja es que uno está estudiando un componente del carro (LC3) en lugar de la carga real de autofagia. Esto es una desventaja bastante grave, porque la relación entre los Estados y el flujo de LC3 a través de la vía contra el secuestro y el flujo de carga es altamente confusa. De hecho, hemos demostrado que flujo de carga a granel puede ser mantenido en niveles elevados en condiciones donde no hay ningún flujo de LC3, a pesar de la presencia de conjugados LC3 en las celdas9. Por otra parte, hemos demostrado esa autofagia a granel se ve afectada por agotamiento de LC3 eficiente y es así probablemente LC3-independiente9. Este hallazgo más tarde ha sido confirmado por LC3 k.o. estudios10,11, que también indican que la mitofagia Parkin-dependiente (el selectiva autofagia de las mitocondrias) es independiente de LC310,11 .
En Resumen, existe una clara necesidad de ensayos de carga a controlar la actividad de autofagia. Forma óptima tales ensayos deben ser ampliamente aplicable, bien definidos y fáciles de realizar. En los últimos años hemos tomado un especial interés en el análisis del secuestro de LDH, que fue desarrollado por Seglen en la década de 198012y se basa en la transferencia de LDH citosólico a sedimentables, célula que contiene vacuolas autophagic de medición fracciones. LDH es una proteína citosólica estable y soluble que fácilmente Co es secuestrada cuando phagophores enwrap carga citoplásmico. Secuestro de LDH por lo tanto es una medida general de secuestro de autofagia. LDH se degrada exclusivamente por la vía autofagia lisosomal12. Así, en presencia de inhibidores de la degradación lisosomal, por ejemplo, bafilomycin A1 (Baf)13, directamente los efectos del tratamiento experimental reflejan alteraciones en la actividad de autofagia secuestro. En ausencia de inhibidores de la degradación, se puede medir el efecto neto de las alteraciones en el secuestro de la LDH y la degradación.
El ensayo de secuestro de LDH es ampliamente aplicable, puesto que la LDH se expresa altamente y ubicuo en todos los tipos de la célula y los niveles de LDH pueden cuantificarse con precisión por un ensayo enzimático14,15. Sin embargo, el original del protocolo12 , establecido en hepatocitos primarios de rata — era algo lento y requiere una gran cantidad de a partir de material así como un descarga eléctrica a la medida del condensador. De manera escalonada, poco a poco hemos transformado el ensayo en un método fácil y versátil. En primer lugar, el protocolo original fue adaptado para el uso en células de mamífero líneas16. En segundo lugar, el método era substancialmente reduce3,9. Tercero, se eliminaron varios pasos en el protocolo, incluyendo un cojín densidad laborioso paso17. Esto permitió al mismo tiempo una regionalización aún más el método, desde el punto de partida original del uso de una placa de 10 cm por ejemplo16 para usar un único pozo de una placa de 12 pozos por muestra (es decir, aproximadamente 15 dobleces menos a partir de material)17. En cuarto lugar, identificamos un aparato de electroporación comercial que podría sustituir el condensador descarga eléctrica por encargo del17.
Aquí se presenta nuestro protocolo más actualizada del ensayo LDH de secuestro, que incluye algunas otras simplificaciones del método con respecto a los previamente publicados17 . Además, se muestra un conjunto de resultados típicos obtenidos en un número de diferentes tipos de células, e importante, cuentan con varias líneas de validación experimental del método usando derribo farmacológico así como genético y enfoques de octavos de final. Para un esquema general de flujo del protocolo todo, vea la figura 1.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. célula siembra y tratamiento
-
Células adherentes en cultivo en frascos de cultivo de tejidos2 de 75 cm en una incubadora humidificada con 5% CO2 a 37 ° C, utilizando el medio de cultivo recomendado: para el tipo de célula en cuestión. Permitir a las células a crecer hasta llegar a una capa de la célula cerca del confluente.
Nota: Use Medio RPMI 1640 suplementado con 10% suero fetal bovino (FBS) de LNCaP, HEK293, fibroblastos embrionarios del ratón (MEFs), BJ, MCF-7 y las células RPE-1.- Lavar las células con 3 mL 37 ° C con tampón fosfato salino (PBS), pH 7,4. Sustituya el PBS por 3 mL 0.25% tripsina-ethylenediaminetetraacetate (EDTA) (w/v) e Incube el frasco en una incubadora humidificada con 5% CO2 a 37 ° C hasta que separe de las células (2-5 min).
- Resuspender las células separadas con 7 mL medio de cultivo que contiene 10% FBS. Mezclar una alícuota de 10 μl célula suspensión con 10 μl 0,4% azul de tripano en un tubo de microcentrífuga, utilizando una pipeta de 0.5-20 μl. Utilice la misma punta de pipeta para llenar inmediatamente una cuenta slide cámara y contar las células en un contador de células automatizado.
-
Preparar una adecuada dilución (ver nota abajo) de la suspensión de células de paso 1.1.2 con cultura media que contiene 10% FBS y las semillas 1 mL de la suspensión celular diluido en cada pocillo de una placa (superficie ~3.8 cm2) de 12-bien cultivo de tejidos utilizando aséptica técnica. Permitir el crecimiento en una incubadora humidificada con 5% CO2 a 37 ° C hasta alcanzar la densidad deseada de la célula, por ejemplo, 60 – 90% de confluencia en la cosecha.
Nota: La dilución de la suspensión de células que dará la confluencia celular deseada en la cosecha varía de célula tipo tipo de la célula, así como según la duración y tipo de tratamientos experimentales. Por lo tanto, esto debe empíricamente evaluarse en cada caso.- Para los experimentos que deben ser tratados y había cosechado 2 días después de siembra, semilla de 2.5 x 105 LNCaP, HEK293, o MCF-7 células, MEFs de4 5 x 10, 4 x 105 BJ o 1,5 x 105 1 RPE células en cada pocillo de la placa de la pozo 12.
- Para las células que se adhieren libremente, cubrir las placas con el tipo de recubrimiento recomendado para el tipo de la célula en cuestión. Para las células LNCaP (y HEK293) Utilice placas recubiertas con poly-D-lisina (PDL).
- Para ello, agregar 500 μl de PDL en 2,5 μg/mL en estéril de H2O para cada pozo e incubar las placas en un ambiente estéril durante 30 min a temperatura ambiente (20 – 25 ° C). Retire de la PDL con la succión y lave cada bien brevemente con 1 mL estéril H2O.
Nota: Generalmente, se realiza paso 1.2.1 sin cualquier tratamiento experimental. Sin embargo, si realizar ARNi, puede ser conveniente comenzar una transfección inversa con la siembra9.
-
Realice tratamientos experimentales en los pocillos duplicados o triplicados por condición.
- Por ejemplo, tratar las células con 50 nM de la Torin1 del inhibidor de mTOR, que generalmente es un eficiente inductor del secuestro de autofagia, o someter las células a la inanición aguda de suero y aminoácidos por lavar las células con 1 mL de Earle del aminoácido libre de equilibrado Medio de solución (EBSS) la sal y posteriormente incubar las células en 1 mL de EBSS en una incubadora humidificada con 5% CO2 a 37 ° C.
- Deja un conjunto de pozos sin tratar para definir niveles de sedimentables LDH de fondo.
- Agregue una cantidad de saturación de la bafilomycin del inhibidor de la captura posterior A1 (Baf)3,13,16,18 en la ausencia o presencia de los tratamientos experimentales, 3 – 4 h antes de la célula de la cosecha. Incubar las células en una incubadora humidificada con 5% CO2 a 37 ° C.
- Uso 100 nM Baf de LNCaP, HEK293, BJ, MCF-7 y las células de RPE-1 y 10 nM Baf para MEFs.
- Para tratamientos experimentales que tienen una duración de sólo 3-4 h (como ésos paso ejemplificada en 1.3.1 típicamente), agregue Baf simultáneamente con los tratamientos. Para más tratamientos experimentales, esperar 3-4 h antes de la cosecha y añadir 2 μl de 500 x concentrado Baf stock directamente en el medio.
- Mezclar por agitación de la placa inmediatamente después de la adición de Baf. En este punto también es recomendable añadir inhibidores de la captación de macroautophagic como controles, por ejemplo, 10 mM de la pan-Fosfoinositol 3-quinasa (PI3K) inhibidor de la 3-metil adenina (3MA)19o 10 μm de la selectivo PI3K clase III inhibidor de la SAR-40520.
2. cosecha y preparación para la Electrodisruption de la célula
- Al final del tratamiento, Aspire el medio con la succión y añadir solución de separación celular de μl 200 (precalentado a 37 ° C) a cada pocillo. Incubar a 37 ° C hasta que separe de las células (típicamente alrededor de 5 min).
Nota: Considerando que 0.25% tripsina-EDTA (w/v) pueden utilizarse en lugar de la solución de separación celular, este último contiene DNasa, que ayuda a reducir la viscosidad de las células separadas. Como el medio fondo se aspira, no es necesario lavar las células antes de la adición de solución de tripsina-EDTA o celda de separación. - Añadir 500 μl temperatura ambiente (20-25 ° C) PBS, pH 7,4, con 2% (p/v) de albúmina sérica bovina (BSA) a cada pocillo y resuspender con la pipeta hasta que no matas celulares visibles. Transferir inmediatamente la suspensión de células a tubos de microcentrífuga de 1,5 mL en hielo.
Nota: A menos que se indique lo contrario, realice todos los pasos subsiguientes en el hielo. - Sedimentos las células por centrifugación a 400 x g durante 5 min a 4 ° C.
- Cuidadosamente aspirar el sobrenadante (con succión) para salir de los gránulos de la célula tan secas como sea posible.
- Añadir 400 μl 10% (p/v) de sacarosa (en ultrapura H2O) a cada tubo.
3. membrana plasmática Electrodisruption y separación de fracciones celulares sedimentables y Total
- Resuspender el precipitado de células con una pipeta para obtener una suspensión unicelular cerca y transferir a una cubeta de electroporación de 4 mm.
Nota: Pipeteo altibajos ~ 10-15 veces, utilizando una pipeta de 100-1.000 μl, es generalmente suficiente. - Colocar la cubeta en un decaimiento exponencial onda electroporator y descarga un solo pulso eléctrico a 800 V, 25 μF y 400 Ω; Estos ajustes producen un pulso de 8 ms de duración.
-
Utilice una nueva punta de pipeta para transferir la disruptate de células a un tubo de microcentrífuga de 1,5 mL con 400 μL de solución de helada sacarosa tamponada con fosfato (monofosfato de sodio 100 mM, 2 mM Ditiotreitol (DTT), 2 mM EDTA y 1.75% de sacarosa, pH 7,5) y mezclar brevemente pipeteo.
- Opcional: Para verificar la eficiente membrana plasmática electrodisruption17, mezclar 10 μl de la disruptate celular diluida en el paso 3.3 con 10 μl 0,4% azul de tripano en un tubo de microcentrífuga de 1,5 mL. Traslado a una cámara de conteo y comprobar que el porcentaje de células positivas de azul de tripano es > 99%.
- Dejar la muestra en la cámara de conteo durante 30 min a temperatura ambiente (20 – 25 ° C) y verificar que se ha mantenido el porcentaje de células positivas de azul de tripano > 99%.
- Opcional: Para verificar que el electrodisruption no ha sido demasiado duro, es decir, no ha interrumpido los organelos intracelulares, realice pasos 1.1-3.3 como se describe anteriormente, pero utilice un material de partida más grande (un pozo de una placa de 6 pozos con una capa de células confluentes de ~ 80%), y 150 μl 10% de sacarosa paso 2.5 y 150 μL tampón fosfato solución sacarosa sin TDT paso 3.3.
- Utilice una pipeta cuidadosamente capa 200 μL de la solución de disruptate de celular diluida sobre un colchón de densidad 1,2 mL de medio de gradiente de densidad de 8% (w/v) con tampón fosfato (por ejemplo, 8% Nycodenz, fosfato de sodio 50 mM, 2.2% de sacarosa, 1 mM EDTA) en una centrífuga de 2 mL tubo. Centrifugue a 20.000 x g durante 45 minutos a 4 ° C en una microcentrífuga con función soft-mode (para suave aceleración y desaceleración) y con cuidado poner los tubos en hielo.
- Retire con cuidado 60 μL de la fracción superior de ~ 200 μL, asegurándose de que no cualquier solución medio gradiente de densidad y transferir a un tubo de microcentrífuga fresco.
Nota: Esto debe contener citosol de pureza excepcional, denominada "savia celular"21. - Prueba de la pureza de la fracción obtenida en el paso anterior, mediante la realización de análisis de western blot de organelo contiene proteínas, usando técnicas estándar y geles de gradiente de 4 – 20%16.
- Realizar por ejemplo immunoblotting para catepsina B21, citocromo c y isomerase del disulfuro de la proteína, para verificar que la descarga eléctrica en el paso 3.2 ha no interrumpido lisosomas, mitocondrias y retículo endoplásmico, respectivamente y el inmunoblot para LDH para verificar la presencia de una proteína citosólica en la savia celular.
- En paralelo, realizar immunoblotting en extractos de proteína procedente de la celda total disruptate solución16 para confirmar que los anticuerpos utilizados pueden detectar las proteínas contienen organelas que están siendo evaluadas.
- Opcional: Para verificar la eficiente membrana plasmática electrodisruption17, mezclar 10 μl de la disruptate celular diluida en el paso 3.3 con 10 μl 0,4% azul de tripano en un tubo de microcentrífuga de 1,5 mL. Traslado a una cámara de conteo y comprobar que el porcentaje de células positivas de azul de tripano es > 99%.
- Repita los pasos 3.1-3.3 para cada muestra.
- Quitar 550 μl de cada solución de disruptate de células diluidas (obtenido en el paso 3.3) para tubos de microcentrífuga de 2 mL que contiene 900 μl resuspensión helada tampón (monofosfato de sodio 50 mM, 1 mM TDT, 1 mM EDTA y 5.9% de sacarosa, pH 7.5) suplementado con 0,5% BSA y 0,01% Tween-20 y mezclar brevemente por pipeteo.
- Centrifugar a 18.000 x g por 45 min a 4 ° C para producir pellets que contiene "LDH sedimentada". Cuidadosamente aspirar el sobrenadante (con succión) para dejar las pelotillas tan secas como sea posible. Coloque las muestras en un congelador de-80 ° C.
- Transferir 150 μL de cada solución de disruptate de células diluidas (obtenido en el paso 3.3) a nuevos tubos y coloque las muestras en un congelador de-80 ° C. Usar estas muestras para determinar los niveles de "total LDH" en las células.
Nota: En este punto el experimento puede pausarse para como deseado.
4. LDH extracción y medición de la actividad enzimática de LDH
- Deshielo "LDH sedimentada" (del paso 3.6) y muestras de "total LDH" (de paso 3.7) en hielo.
- Añadir 300 μL de buffer de resuspensión helada que contiene 1,5% Triton X-405 a la "LDH total" las muestras (obtención de una concentración final de X-405 Triton de 1%). Rotar las muestras en un rodillo en una cámara fría (4 – 8 ° C) durante 30 minutos.
- Añadir 750 μl de buffer de resuspensión helada con 1% Triton X-405 a la LDH"sedimentada" muestras y resuspender el pellet con una pipeta hasta que se alcance una solución homogénea.
- Centrifugue las muestras en el paso 4.2 y 4.3 en 18.000 x g durante 5 min a 4 ° C a los desechos celulares sedimento disuelta.
- Mezcle 4 partes de imidazol frío 65 mM (piruvato de mM de 7.5)/0.75 de pH con una parte de imidazol frío 65 mM (pH 7.5) / 1,8 mM NADH para obtener una solución de trabajo que es estable durante al menos tres semanas a 4 ° C.
- Mezcle 3 – 30 μl de los sobrenadantes de paso 4.4 con 200 μL de la solución de trabajo de paso 4.5.
- Determinar la cantidad de LDH por medición de la actividad enzimática de LDH como la disminución de nicotinamida-adenin-dinucleótido (forma reducida) absorbancia (NADH) a 340 nm a 37 ° C en comparación con un estándar con una concentración conocida de la LDH. Mida la absorbancia hasta que la reacción ha acercado al terminar, es decir, hasta que la absorbancia a 340 nm ya no cambia con el tiempo.
Nota: Este es el clásico método bioquímico para medir la actividad LDH. Aunque el protocolo actual realiza la reacción a 37 ° C, también se puede realizar a temperatura ambiente (20 – 25 ° C), que es recomendable si haciendo espectrofotometría manual. El protocolo actual utiliza un instrumento multianalyzer robótico, que en forma automática mezcla las muestras con solución de trabajo en una placa de 96 pocillos y mide la absorbancia a 340 nm a 37 ° C cada 20 s durante 3 minutos. Después de eso, el software del instrumento calcula la concentración de LDH, expresada como unidades (U) L, comparando la cuesta de las mediciones de absorbancia con el tiempo en comparación con una curva estándar obtenido a través de la calibración con un estándar de LDH conocida concentración. El rango lineal de detección por este método es de 30 – 1.500 U/L. Como alternativa, existen una gran variedad de kits disponibles en el mercado para medir la LDH. Algunos de ellos se basan en la reacción enzimática a la generación de productos colorimétricas o fluorescentes, permitiendo la detección por otros medios que la espectrofotometría UV y con otras gamas de lineales de detección de acoplamiento.
5. cálculo del secuestro de LDH
- Calcular el porcentaje de LDH sedimentado a LDH total para cada muestra, tomar las diluciones y toma en cuenta:
LDH sedimentado (%) =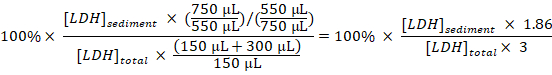
Nota: Durante los pasos 3.1-3.3 aproximadamente 50 μl se pierde debido a traslado dentro y fuera de la cubeta de electroporación. Calcular por lo tanto, de tener un total de 750 μl (en lugar de 800 μL) de disruptate de células diluidas en el paso 3.3. - Restar el porcentaje de sedimentación LDH obtenida en las muestras de las células no tratadas (paso 1.3.2) del porcentaje de sedimentación LDH obtenida en muestras de células tratadas experimentalmente y se dividen por el tiempo de tratamiento con Baf para obtener el porcentaje de LDH secuestrado por hora en el período de muestreo:
Secuestrado LDH (% / h) =
Access restricted. Please log in or start a trial to view this content.
Resultados
Usando el protocolo descrito aquí, la actividad de autofagia secuestro a granel en un número de líneas de diferentes células de mamífero, incluyendo LAPC4, DU145, Huh7, PNT2A, HeLa, VCaP, H3122, Hec1A, MCF-7, T47D, U2OS, PC3, G361, ratón (MEFs) los fibroblastos embrionarios, RPE-1, Se midió las células HEK293, BJ y LNCaP. Secuestro fue evaluado bajo condiciones basales (en medio completo, rico en nutrientes), o en células muy hambrientos de suero y aminoácidos (un bona fide<...
Access restricted. Please log in or start a trial to view this content.
Discusión
En Resumen, el protocolo descrito aquí representa un método fiable y ampliamente aplicable para monitorear la actividad de autofagia secuestro a granel en células de mamíferos. Comparado con el original método12,16, hemos eliminado una serie de pasos innecesarios, simplificado a varios de los pasos restantes e introdujo una sustancial reducción de resolución. Como resultado, el protocolo es mejorado en relación a costo y tiempo-eficiencia, y ahora se pued...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
Los autores no tienen conflicto de interés.
Agradecimientos
Este trabajo fue apoyado financieramente por el Consejo de investigación de Noruega, la Universidad de Oslo, la Anders Jahre Foundation, la Fundación de Nansen y el legado en la memoria de Henrik Homan. Agradecemos a Dr. Noboru Mizushima para el ATG5 + / + MEFs y ATG5/MEFs, Dr. Masaaki Komatsu para la ATG7 + / + MEFs y ATG7/MEFs y Dr. Shizuo Akira para el ATG9A + / + MEFs y ATG9A/MEFs. Agradecemos Frank Sætre asistencia técnica, y el Dr. por Seglen O. constructivas discusiones metodológicas.
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| 1.5 mL and 2 mL microcentrifuge tubes | Eppendorf | 211-2130 and 211-2120 | |
| 12-well plates | Falcon | 353043 | |
| Accumax cell detachment solution | Innovative Cell Technologies | A7089 | Keep aliquots at -20 °C for years, and in fridge for a few months |
| Bafilomycin A1 | Enzo | BML-CM110-0100 | Dissolve in DMSO |
| BJ cells | ATCC | CRL-2522 | use at passage <30 |
| Bovine serum albumin (BSA) | VWR | 422361V | |
| Burker counting chamber | Fisher Scientific | 139-658585 | |
| Countess Cell Counting Chamber Slides | ThermoFisher Scientfic | C10228 | |
| Countess II Automated Cell Counter | ThermoFisher Scientfic | AMQAX1000 | |
| Cover glass for the Burker counting chamber | Fisher Scientific | 139-658586 | |
| Criterion Tris-HCl Gel, 4–20%, 26-well, 15 µL, 13.3 cm x 8.7 cm (W x L) | Bio-Rad | 3450034 | |
| DTT | Sigma-Aldrich | D0632 | |
| Earle's balanced salt solution (EBSS) | Gibco | 24010-043 | conatains 0.1% glucose |
| EDTA | Sigma-Aldrich | E7889 | |
| Electroporation cuvette (4 mm) | Bio-Rad | 1652088 | |
| Exponential decay wave electroporator | BTX Harvard Apparatus | EMC 630 | |
| Fetal bovine serum (FBS) | Sigma | F7524 | 10% final concentration in RPMI 1640 medium |
| HEK293 cells | ATCC | CRL-1573 | |
| Imidazole | Sigma-Aldrich | 56750 | Autoclave a 65 mM solution and keep in fridge for months |
| Incubator; Autoflow IR Direct Heat CO2 incubator | NuAire | NU-5510E | |
| Lipofectamine RNAiMAX Transfection Reagent | ThermoFisher | 13778150 | |
| LNCaP cells | ATCC | CRL-1740 | use at passage <30 |
| 3-Methyl Adenine (3MA) | Sigma-Aldrich | M9281 | Stock 100 mM in RPMI in -20 °C. Heat stock to 65 °C for 10 min, and use at 10 mM final concentration |
| Refridgerated Microcentrifuge | Beckman Coulter Life Sciences | 368831 | |
| Refridgerated Microcentrifuge with soft-mode function | Eppendorf | Eppendorf 5417R | |
| MRT67307 hydrochloride (ULKi) | Sigma-Aldrich | SML0702 | Inhibits ULK kinase activity. Dissolve in DMSO. |
| MaxMat Multianalyzer instrument | Erba Diagnostics | PL-II | |
| MCF7 cells | ATCC | HTB-22 | |
| NADH | Merck-Millipore | 1.24644.001 | |
| Nycodenz | Axis-Shield | 1002424 | |
| Opti-MEM Reduced Serum Medium | ThermoFisher | 31985062 | |
| Phosphate-buffered saline (PBS) | Gibco | 20012-019 | |
| Pipette tips 3 (0.5-20 µL) | VWR | 732-2223 | Thermo Fischer ART Barrier tips |
| Pipette tips (1-200 µL) | VWR | 732-2207 | Thermo Fischer ART Barrier tips |
| Pipette tips (100-1,000 µL) | VWR | 732-2355 | Thermo Fischer ART Barrier tips |
| Pipettes | ThermoFisher | 4701070 | Finnpipette F2 GLP Kit |
| Poly-D-lysine | Sigma-Aldrich | P6407-10X5MG | Make a 1 mg/mL stock solution in sterile H2O. This solution is stable at -20 °C for at least 1 year. |
| Pyruvate | Merck-Millipore | 1066190050 | |
| RPE-1 cells (hTERT RPE-1) | ATCC | CRL-4000 | |
| RPMI 1640 | Gibco | 21875-037 | |
| SAR-405 | ApexBio | A8883 | Inhibits phosphoinositide 3-kinase class III (PIK3C3). Dissolve in DMSO. |
| Silencer Select Negative Control #1 (siCtrl) | ThermoFisher/Ambion | 4390843 | |
| Silencer Select ATG9-targeting siRNA (siATG9A) | ThermoFisher/Ambion | s35504 | |
| Silencer Select FIP200-targeting siRNA (siFIP200) | ThermoFisher/Ambion | s18995 | |
| Silencer Select ULK1-targeting siRNA (siULK1) | ThermoFisher/Ambion | s15964 | |
| Silencer Select ULK2-targeting siRNA (siULK2) | ThermoFisher/Ambion | s18705 | |
| Silencer Select GABARAP-targeting siRNA (siGABARAP) | ThermoFisher/Ambion | s22362 | |
| Silencer Select GABARAPL1-targeting siRNA (siGABARAPL1) | ThermoFisher/Ambion | s24333 | |
| Silencer Select GABARAPL2-targeting siRNA (siGABARAPL2) | ThermoFisher/Ambion | s22387 | |
| Sodium phosphate monobasic dihydrate (NaH2PO4 • 2H2O) | Merck-Millipore | 1.06580.1000 | |
| Sodium phosphate dibasic dihydrate (Na2HPO4 • 2H2O ) | Prolabo | 28014.291 | |
| Sucrose | VWR | 443816T | 10% final concentration in water; filter through 0.45 µm filter and keep in fridge for months |
| Thapsigargin | Sigma-Aldrich | T9033 | Inhibits the SERCA ER Ca2+ pump. Dissolve in DMSO. |
| Triton X-405 | Sigma-Aldrich | X405 | 1% final |
| Trypan Blue stain 0.4% | Molecular Probes | T10282 | |
| Trypsin-EDTA (0.25% w/v Trypsin) | Gibco | 25200-056 | |
| Tween-20 | Sigma-Aldrich | P2287 | 0.01% final |
Referencias
- Rubinsztein, D. C., Frake, R. A. Yoshinori Ohsumi's Nobel Prize for mechanisms of autophagy: from basic yeast biology to therapeutic potential. J R Coll Physicians Edinb. 46 (4), 228-233 (2016).
- Mizushima, N. The exponential growth of autophagy-related research: from the humble yeast to the Nobel Prize. FEBS Lett. 591 (5), 681-689 (2017).
- Seglen, P. O., et al. Macroautophagic cargo sequestration assays. Methods. 75, 25-36 (2015).
- Hoyvik, H., Gordon, P. B., Seglen, P. O. Use of a hydrolysable probe, [14C]lactose, to distinguish between pre-lysosomal and lysosomal steps in the autophagic pathway. Exp Cell Res. 166 (1), 1-14 (1986).
- Plomp, P. J., Gordon, P. B., Meijer, A. J., Hoyvik, H., Seglen, P. O. Energy dependence of different steps in the autophagic-lysosomal pathway. J Biol Chem. 264 (12), 6699-6704 (1989).
- Gordon, P. B., Seglen, P. O. Prelysosomal convergence of autophagic and endocytic pathways. Biochem Biophys Res Commun. 151 (1), 40-47 (1988).
- Holen, I., Gordon, P. B., Seglen, P. O. Protein kinase-dependent effects of okadaic acid on hepatocytic autophagy and cytoskeletal integrity. Biochem J. 284, Pt 3 633-636 (1992).
- Kabeya, Y., et al. LC3, a mammalian homologue of yeast Apg8p, is localized in autophagosome membranes after processing. Embo j. 19 (21), 5720-5728 (2000).
- Szalai, P., et al. Autophagic bulk sequestration of cytosolic cargo is independent of LC3, but requires GABARAPs. Exp Cell Res. 333 (1), 21-38 (2015).
- Nguyen, T. N., et al. Atg8 family LC3/GABARAP proteins are crucial for autophagosome-lysosome fusion but not autophagosome formation during PINK1/Parkin mitophagy and starvation. J Cell Biol. , (2016).
- Pontano Vaites, L., Paulo, J. A., Huttlin, E. L., Harper, J. W. Systematic analysis of human cells lacking ATG8 proteins uncovers roles for GABARAPs and the CCZ1/MON1 regulator C18orf8/RMC1 in macro and selective autophagic flux. Mol Cell Biol. , (2017).
- Kopitz, J., Kisen, G. O., Gordon, P. B., Bohley, P., Seglen, P. O. Nonselective autophagy of cytosolic enzymes by isolated rat hepatocytes. J Cell Biol. 111 (3), 941-953 (1990).
- Bowman, E. J., Siebers, A., Altendorf, K. Bafilomycins: a class of inhibitors of membrane ATPases from microorganisms, animal cells, and plant cells. Proc Natl Acad Sci U S A. 85 (21), 7972-7976 (1988).
- Hadjivassiliou, A. G., Rieder, S. V. The enzymatic assay of pyruvic and lactic acids. A definitive procedure. Clin Chim Acta. 19 (3), 357-361 (1968).
- Bergmeyer, H. U., Bernt, E. Methods of enzymatic analysis (2nd English ed). Bergmeyer, H. U. 2, Verlag Chemie. 574-579 (1974).
- Engedal, N., et al. Modulation of intracellular calcium homeostasis blocks autophagosome formation. Autophagy. 9 (10), 1475-1490 (2013).
- Luhr, M., et al. A Simple Cargo Sequestration Assay for Quantitative Measurement of Nonselective Autophagy in Cultured Cells. Methods Enzymol. 587, 351-364 (2017).
- Mousavi, S. A., et al. Effects of inhibitors of the vacuolar proton pump on hepatic heterophagy and autophagy. Biochim Biophys Acta. 1510 (1-2), 243-257 (2001).
- Seglen, P. O., Gordon, P. B. 3-Methyladenine: specific inhibitor of autophagic/lysosomal protein degradation in isolated rat hepatocytes. Proc Natl Acad Sci U S A. 79 (6), 1889-1892 (1982).
- Ronan, B., et al. A highly potent and selective Vps34 inhibitor alters vesicle trafficking and autophagy. Nat Chem Biol. 10 (12), 1013-1019 (2014).
- Saetre, F., Hagen, L. K., Engedal, N., Seglen, P. O. Novel steps in the autophagic-lysosomal pathway. Febs j. 282 (11), 2202-2214 (2015).
- Klionsky, D. J., et al. Guidelines for the use and interpretation of assays for monitoring autophagy (3rd edition). Autophagy. 12 (1), 1-222 (2016).
- Seglen, P. O., Overbye, A., Saetre, F. Sequestration assays for mammalian autophagy. Methods Enzymol. 452, 63-83 (2009).
- Hurley, J. H., Young, L. N. Mechanisms of Autophagy Initiation. Annu Rev Biochem. 86, 225-244 (2017).
- Thastrup, O., Cullen, P. J., Drobak, B. K., Hanley, M. R., Dawson, A. P. Thapsigargin, a tumor promoter, discharges intracellular Ca2+ stores by specific inhibition of the endoplasmic reticulum Ca2(+)-ATPase. Proc Natl Acad Sci U S A. 87 (7), 2466-2470 (1990).
- Kuma, A., et al. The role of autophagy during the early neonatal starvation period. Nature. 432 (7020), 1032-1036 (2004).
- Komatsu, M., et al. Impairment of starvation-induced and constitutive autophagy in Atg7-deficient mice. J Cell Biol. 169 (3), 425-434 (2005).
- Saitoh, T., et al. Atg9a controls dsDNA-driven dynamic translocation of STING and the innate immune response. Proc Natl Acad Sci U S A. 106 (49), 20842-20846 (2009).
- Gordon, P. B., Seglen, P. O. Autophagic sequestration of [14C]sucrose, introduced into rat hepatocytes by reversible electro-permeabilization. Exp Cell Res. 142 (1), 1-14 (1982).
- An, H., Harper, J. W. Systematic analysis of ribophagy in human cells reveals bystander flux during selective autophagy. Nat Cell Biol. 20 (2), 135-143 (2018).
- Fass, E., Shvets, E., Degani, I., Hirschberg, K., Elazar, Z. Microtubules support production of starvation-induced autophagosomes but not their targeting and fusion with lysosomes. J Biol Chem. 281 (47), 36303-36316 (2006).
- Velikkakath, A. K., Nishimura, T., Oita, E., Ishihara, N., Mizushima, N. Mammalian Atg2 proteins are essential for autophagosome formation and important for regulation of size and distribution of lipid droplets. Mol Biol Cell. 23 (5), 896-909 (2012).
- Øverbye, A., Sætre, F., Hagen, L. K., Johansen, H. T., Seglen, P. O. Autophagic activity measured in whole rat hepatocytes as the accumulation of a novel BHMT fragment (p10), generated in amphisomes by the asparaginyl proteinase, legumain. Autophagy. 7 (9), 1011-1027 (2011).
- Kominami, E., Hashida, S., Khairallah, E. A., Katunuma, N. Sequestration of cytoplasmic enzymes in an autophagic vacuole-lysosomal system induced by injection of leupeptin. J Biol Chem. 258 (10), 6093-6100 (1983).
- Rosado, C. J., Mijaljica, D., Hatzinisiriou, I., Prescott, M., Devenish, R. J. Rosella: a fluorescent pH-biosensor for reporting vacuolar turnover of cytosol and organelles in yeast. Autophagy. 4 (2), 205-213 (2008).
- Katayama, H., Kogure, T., Mizushima, N., Yoshimori, T., Miyawaki, A. A sensitive and quantitative technique for detecting autophagic events based on lysosomal delivery. Chem Biol. 18 (8), 1042-1052 (2011).
- Ogier-Denis, E., et al. A heterotrimeric Gi3-protein controls autophagic sequestration in the human colon cancer cell line HT-29. J Biol Chem. 270 (1), 13-16 (1995).
- Seglen, P. O., Gordon, P. B., Tolleshaug, H., Hoyvik, H. Use of [3H]raffinose as a specific probe of autophagic sequestration. Exp Cell Res. 162 (1), 273-277 (1986).
- Luhr, M., Sætre, F., Engedal, N. The Long-lived Protein Degradation Assay: an Efficient Method for Quantitative Determination of the Autophagic Flux of Endogenous Proteins in Adherent Cell Lines. Bio-protocol. 8 (9), e2836(2018).
- Ronning, O. W., Pettersen, E. O., Seglen, P. O. Protein synthesis and protein degradation through the cell cycle of human NHIK 3025 cells in vitro. Exp Cell Res. 123 (1), 63-72 (1979).
- Seglen, P. O., Grinde, B., Solheim, A. E. Inhibition of the lysosomal pathway of protein degradation in isolated rat hepatocytes by ammonia, methylamine, chloroquine and leupeptin. Eur J Biochem. 95 (2), 215-225 (1979).
- Seglen, P. O., Solheim, A. E. Valine uptake and incorporation into protein in isolated rat hepatocytes. Nature of the precursor pool for protein synthesis. Eur J Biochem. 85 (1), 15-25 (1978).
- Bauvy, C., Meijer, A. J., Codogno, P. Assaying of autophagic protein degradation. Methods Enzymol. 452, 47-61 (2009).
- Engedal, N., Seglen, P. O. Autophagy of cytoplasmic bulk cargo does not require LC3. Autophagy. 12 (2), 1-3 (2016).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados