Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Laktat-Dehydrogenase Sequestrierung Assay – Eine einfache und zuverlässige Methode zur Bestimmung Bulk Selbstverdauende Sequestration Aktivität in Säugerzellen
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Hier ist eine einfache und gut validierte Protokoll zur Messung der Masse selbstverdauende Sequestrierung Aktivität in Säugetierzellen beschrieben. Die Methode basiert auf der Quantifizierung des Anteils der Laktat-Dehydrogenase (LDH) im Sedimentable Zelle Fraktionen gegenüber dem gesamten zellulären LDH Niveau.
Zusammenfassung
Bulk Autophagie zeichnet sich durch die Sequestrierung große Teile der Zytoplasma in Doppel/multi-membrane Strukturen bezeichnet autophagosom. Hier ist ein einfaches Protokoll zur Überwachung dieses Prozesses beschrieben. Darüber hinaus sind typische Ergebnisse und experimentelle Validierung der Methode Autophagie-induzierenden Bedingungen in den verschiedenen Arten von kultivierten Säugerzellen vorgesehen. Während der Großteil Autophagie sequester autophagosom Zytosol, und damit auch lösliche cytosolischen Proteine, neben anderen selbstverdauende Ladung. LDH ist eine stabile und sehr reichlich, lösliche cytosolische Enzym, das nicht selektiv in autophagosom abgesondert wird. Die Höhe der LDH-Sequestrierung spiegelt daher die Menge an Masse selbstverdauende Sequestrierung. Um effizient und präzise LDH-Sequestrierung in Zellen bestimmen, setzen wir eine Electrodisruption-basierte Fraktionierung-Protokoll, die effektiv Sedimentable trennt sich von zytosolischen LDH, gefolgt durch eine Messung der Enzymaktivität in sedimentable Brüche im Vergleich zu ganzen Zellproben. Selbstverdauende-Sequestrierung wird durch Subtraktion des Anteils der Sedimentable LDH in den unbehandelten Zellen aus, dass im behandelten Zellen bestimmt. Der Vorteil des LDH-Sequestrierung Tests ist, dass es einem quantitativen Maß für die selbstverdauende Sequestrierung von endogenen Cargo, im Gegensatz zu anderen Methoden, dass entweder ektopische Expression von Sequestrierung Sonden oder semi-quantitativen Protease einbeziehen Schutz-Analysen der Autophagie Marker oder Rezeptoren.
Einleitung
Autophagie (griechisch für "Self-Essen") ist ein evolutionärer konservierten Prozess für vacuolar/lysosomalen Abbau der intrazellulären Material. Nach der Entdeckung der Autophagie-bezogene ("ATG") Gene, die für Autophagie in Hefe und Menschen wichtig sind, und die Erkenntnis, dass Autophagie spielt eine bedeutende Rolle in Gesundheit und Krankheit (anerkannt durch die 2016 den Nobelpreis für Medizin oder Physiologie Yoshinori Ohsumi), Autophagie ist eines der am intensivsten untersuchten Prozesse in der Zelle Biologie1,2schnell geworden.
Macroautophagy (nachfolgend "Autophagie") ist geprägt durch den Ausbau und Faltung der intrazellulären Membran Cisternen ("Phagophores") in versiegelten, Doppel oder membrane Strukturen ("autophagosom"), die effektiv absondern der eingehüllt Material vom Rest des Zytoplasmas. Bei der Verschmelzung von autophagosom mit Lysosomen ist die innere Autophagosomal Membran und die abgesonderten Ladung abgebaut und recycelt. Autophagosom kann zytoplasmatischen Material in zufälligen (nicht-selektiven Autophagie) und selektive (selektiven Autophagie) Manieren sequester. Bulk Autophagie am wahrscheinlichsten ist eine Mischung von nicht-selektiven und selektiven Autophagie.
In den 1960er und 70er Jahre ("die morphologische Ära" der Autophagie Forschung) wurde selbstverdauende Sequestrierung hauptsächlich durch Ultrastrukturforschung Analysen beurteilt. In den 1980er Jahren und Anfang der 90er Jahre ("der biochemischen Ära") pro Seglen und Mitarbeitern – der Autophagie in primären Ratte Hepatocytes studiert – entwickelt die ersten Methoden zur Messung quantitativ selbstverdauende Sequestrierung Aktivität3. Mit diesen Tests, Seglen definiert und charakterisiert die verschiedenen Schritte der selbstverdauende lysosomalen Pathway4,5, entdeckt und prägte die Amphisome6 (das Produkt der Fusion Endosom-Autophagosome) und war der erste beschreiben Sie die Rolle der Protein-Phosphorylierung Autophagie Verordnung7. Jedoch nach der Entdeckung der Stuka in den 1990 ("Molekulare Zeitalter") und die erste Charakterisierung eines Säugetiers ATG8 Proteins, Mikrotubuli-assoziierten Protein 1A/1 b-Light chain 3 (LC3) im Jahr 20008, die Verwendung von ATG Proteine als Marker für die selbstverdauende Prozess schnell an Popularität gewann, und der älteren und mühsamen biochemischen Methoden wurden zurückgelassen. In der Tat über die letzten 18 Jahre, western-Blot und Fluoreszenz-Mikroskopie-Analysen der LC3 geworden die mit Abstand beliebteste (und in vielen Fällen die einzige) Mittel der Autophagie in Säugetierzellen zu studieren. Der Vorteil ist die relative Leichtigkeit, mit der diese Methoden durchgeführt werden können. Der Nachteil ist, dass man eine Warenkorb-Komponente (LC3) anstatt tatsächliche selbstverdauende Fracht studiert. Dies ist ein ziemlich gravierender Nachteil, da die Beziehung zwischen den Staaten und/oder Fluss der LC3 durch den Weg gegen die Sequestrierung und Flussmittel Ladung sehr unklar ist. In der Tat haben wir gezeigt, dass Bulk Cargo Flussmittel auf hohem Niveau unter Bedingungen aufrechterhalten werden kann wo es keine LC3 Flussmittel, trotz der Anwesenheit von konjugierten LC3 in den Zellen9. Darüber hinaus haben wir bewiesen, dass Bulk Autophagie ist unbeeinflusst von effizienten LC3 Erschöpfung und somit wahrscheinlich LC3-unabhängige9. Dieser Befund wurde später bestätigt durch LC3 Knock-out-Studien10,11, in dem auch angeben, dass Parkin-abhängige Mitophagy (der selektiven Autophagie der Mitochondrien) unabhängig von LC310,11 .
Zusammenfassend lässt sich sagen, es ist ein klares muss für Fracht-based Assays, selbstverdauende Aktivitäten zu überwachen. Solche Tests sollte optimal breit anwendbar, gut definierten und leicht durchzuführen. In den letzten Jahren haben wir ein besonderes Interesse an der LDH-Sequestrierung Assay getroffen, die war in den 1980er Jahren12von pro Seglen entwickelt und basiert auf der Messung der Übertragung der cytosolischen LDH, Sedimentable, selbstverdauende Vakuole-haltigen Zelle Brüche. LDH ist ein stabiles, lösliche cytosolischen Protein, das bereitwillig Co abgesondert ist, wenn Phagophores zytoplasmatischen Fracht Einhüllen. Sequestrierung von LDH ist daher ein allgemeines Maß für selbstverdauende Sequestrierung. LDH wird ausschließlich durch die selbstverdauende lysosomalen Pathway12abgebaut. Also, im Beisein von lysosomalen Abbau-Inhibitoren, z. B. Bafilomycin A1 (Baf)13, experimentelle behandlungseffekte direkt Änderungen in selbstverdauende Sequestrierung Aktivität widerspiegeln. In Ermangelung von Abbau-Inhibitoren kann der Netto-Effekt der Änderungen in LDH-Sequestrierung und Abbau gemessen werden.
Die LDH-Sequestrierung Assay ist breit anwendbar, da LDH hoch und ubiquitär sich in alle Zelltypen drückt und LDH Niveaus durch eine enzymatische Assays14,15genau quantifiziert werden können. Aber das Original Protokoll12 — primäre Ratte Hepatocytes gegründet – war ziemlich zeitaufwändig und erforderte ein hohes Maß an Material sowie eine maßgeschneiderte elektrische Entladung Kondensator. In gewissem Sinne schrittweise haben wir eine einfache und vielseitige Methode allmählich Assays umgewandelt. Erstens war das ursprüngliche Protokoll für den Einsatz in Säugetierzellen Linien16angepasst. Zweitens war die Methode wesentlich herunterskalierten3,9. Dritte, mehrere Schritte im Protokoll wurden eliminiert, einschließlich ein mühsamer Dichte Polster Schritt17. Dies ermöglichte gleichzeitig eine noch weiter verkleinern der Methode, aus der ursprünglichen Ausgangspunkt mit einem 10 cm Teller pro Probe16 mit einem einzigen Brunnen aus einer 12-Well-Platte pro Probe (d.h.ca. 15-fold weniger ab Material)17. Viertens haben wir eine kommerzielle Elektroporation-Apparat, der die maßgeschneiderte elektrische Entladung Kondensator17ersetzen könnte.
Hier ist unsere aktuelle Protokoll des LDH-Sequestrierung Assays, enthält einige weiteren Vereinfachungen des Verfahrens im Vergleich zu den bisher veröffentlichten17 vorgestellt. Darüber hinaus eine Reihe von typischen Ergebnisse in einer Reihe von verschiedenen Zelltypen wird angezeigt, und wichtig ist, mehrere Textzeilen experimentelle Validierung der Methode mit pharmakologischen sowie genetische Zuschlag und Ko-Ansätze zur Verfügung gestellt. Ein Fluss Gesamtschema das ganze Protokoll Siehe Bild 1.
Protokoll
(1) Zelle Aussaat und Behandlung
-
Kultur adhärente Zellen in 75 cm2 Gewebe Kulturflaschen in einem befeuchteten Inkubator mit 5 % CO2 bei 37 ° C, mit dem bevorzugten Kulturmedium für den Zelltyp in Frage. Können Sie die Zellen wachsen, bis sie eine in der Nähe von Zusammenfluss Zellschicht erreichen.
Hinweis: Verwenden Sie RPMI 1640 Medium ergänzt mit 10 % fetalen bovine Serum (FBS) für LNCaP, HEK293, Maus embryonalen Fibroblasten (MEFs), BJ, MCF-7 und RPE-1-Zellen.- Waschen Sie die Zellen mit 3 mL 37 ° C Phosphat-gepufferte Kochsalzlösung (PBS), pH-Wert 7,4. Ersetzen der PBS mit 3 mL 0,25 % (w/V) Trypsin-Ethylenediaminetetraacetate (EDTA), und den Kolben in einem befeuchteten Inkubator mit 5 % CO2 bei 37 ° C inkubieren, bis die Zellen trennen (2 – 5 min).
- Aufschwemmen der freistehenden Zellen mit 7 mL Kulturmedium mit 10 % FBS. Mischen Sie eine 10 µL Zelle Aussetzung aliquoten mit 10 µL 0,4 % Trypan blau in einem Microcentrifuge Schlauch mit einer 0,5-20 µL PIPETTENSPITZE. Verwenden Sie die gleichen PIPETTENSPITZE sofort eine Zählung Kammer Folie füllen, und zählen Sie die Zellen in einem automatisierten Zelle Zähler.
-
Bereiten Sie einen geeigneten Verdünnung (siehe Hinweis unten) die Zellsuspension hindern, Schritt 1.1.2 Kultur mittlere mit 10 % FBS und Samen 1 mL verdünnter Zellsuspension in jede Vertiefung eines 12-Well-Zellkultur-Platte (Fläche ~3.8 cm2) mit aseptischen Technik. Lassen Sie Wachstum in einem befeuchteten Inkubator mit 5 % CO2 bei 37 ° C, bis die gewünschte Zelldichte erreicht wurde, z. B. 60 – 90 % Zusammenfluss bei der Ernte.
Hinweis: Die entsprechende Verdünnung der Zellsuspension, die die gewünschte Zelle Konfluenz bei der Ernte wird vom Zelltyp Zelltyp, sowie nach der Dauer und Art der experimentelle Behandlungen variiert. So muss dies empirisch in jedem Einzelfall beurteilt werden.- Für Experimente, die sollen beide behandelt und 2 Tage nach der Aussaat, Samen 2,5 x 105 LNCaP, HEK293, oder MCF-7 Zellen, 5 x 104 MEFs, 4 x 105 BJ oder 1,5 x 105 RPE-1-Zellen in jede Vertiefung des 12-Well-Platte geerntet.
- Für Zellen, die lose halten, Beschichten der Platten mit der Art der Beschichtung, die für den betreffenden Typ empfohlen. Verwenden Sie für LNCaP (und HEK293) Zellen Platten beschichtet mit Poly-D-Lysin (PDL).
- Zu diesem Zweck jedes gut 500 µL PDL bei 2,5 µg/mL in sterilen H2O hinzu, und inkubieren Sie die Platten in einer sterilen Umgebung für 30 min bei Raumtemperatur (20 – 25 ° C). Entfernen Sie die PDL mit Absaugung und waschen Sie auch kurz mit 1 mL sterile H2O.
Hinweis: In der Regel erfolgt Schritt 1.2.1 ohne jede experimentellen Behandlungen. Jedoch kann wenn RNAi durchführen, es eine umgekehrte Transfektion mit der Aussaat9zu starten sein.
-
Durchführen Sie experimentelle Behandlungen in doppelter oder dreifacher Brunnen pro Zustand.
- Zum Beispiel behandelt die Zellen mit 50 nM von der mTOR-Inhibitor Torin1, die in der Regel ist eine effiziente Induktor von selbstverdauende Sequestrierung oder vorbehaltlich der Zellen akute Serum und Aminosäure Hunger durch Waschen der Zellen mit 1 mL von Aminosäure-freie Earle ist ausgeglichen Salz-Lösung (EBSS) Medium, und anschließend die Zellen in 1 mL EBSS in einem befeuchteten Inkubator mit 5 % CO2 bei 37 ° c inkubieren
- Lassen Sie eine Reihe von Brunnen, die unbehandelt zur Hintergrund-Niveaus von LDH Sedimentable definieren.
- Fügen Sie eine Sättigung Menge von Post-Sequestrierung Inhibitor Bafilomycin A1 (Baf)3,13,16,18 in das Fehlen oder Vorhandensein der experimentelle Behandlungen, 3 – 4 h vor der Zelle Ernte. Inkubieren Sie die Zellen in einem befeuchteten Inkubator mit 5 % CO2 bei 37 ° c
- Einsatz 100 nM Baf für LNCaP, HEK293, BJ, MCF-7 und RPE-1-Zellen, und 10 nM Baf für MEFs.
- Für experimentelle Behandlungen, die haben eine Laufzeit von nur 3 – 4 h (wie diese beispielhaft in Schritt 1.3.1 haben in der Regel), fügen Sie Baf gleichzeitig mit den Behandlungen. Warten Sie für mehr experimentelle Behandlungen, bis 3-4 h vor der Ernte, und fügen Sie 2 µL eines 500 x Baf Lager direkt in das Medium konzentriert.
- Durch Rühren die Platte sofort nach der Zugabe der Baf mischen. An dieser Stelle ist es auch empfehlenswert, Macroautophagic Sequestrierung Inhibitoren als Steuerelemente, z. B. 10 mM von der Pan-Phosphoinositide 3-Kinase (PI3K) Hemmer 3-Methyl-Adenin (3MA)19oder 10 µM der selektiven PI3K Klasse III hinzufügen Inhibitor SAR-40520.
2. Handy-Ernte und Vorbereitung für Electrodisruption
- Am Ende des Behandlungszeitraums Aspirieren Sie das Medium mit Absaugung und jedes gut fügen Sie 200 µL Zelle Ablösung Lösung (vorgeheizt auf 37 ° C hinzu). Inkubieren Sie bei 37 ° C, bis die Zellen trennen (in der Regel ca. 5 min).
Hinweis: 0,25 % (w/V) Trypsin-EDTA statt der Zelle Ablösung Lösung verwendet werden kann, enthält diese DNase, wodurch die Viskosität der freistehenden Zellen zu verringern. Solange das Medium gründlich abgesaugt wird, ist es nicht notwendig, die Zellen vor Zugabe von Trypsin-EDTA oder Zelle Ablösung Lösung zu waschen. - Fügen Sie 500 µL Raumtemperatur (20-25 ° C) PBS, pH 7,4, mit 2 % (w/V) Rinderserumalbumin (BSA) in jede Vertiefung und erneut mit der Pipette bis keine Zelle Klumpen sichtbar sind. Übertragen Sie sofort die Zellsuspension auf 1,5 mL Mikrozentrifugenröhrchen auf Eis.
Hinweis: Sofern nicht anders angegeben, führen Sie alle weiteren Schritte auf dem Eis. - Sediment der Zellen durch Zentrifugation bei 400 X g für 5 min bei 4 ° C.
- Aspirieren Sie gründlich den überstand (mit Absaugung) um die Zelle Pellets so trocken wie möglich zu verlassen.
- Jedes Rohr 400 µL 10 % (w/V) Saccharose (in hochreinen H2O) hinzufügen.
(3) Plasmamembran Electrodisruption und Trennung von Sedimentable und Gesamt-Zelle Brüche
- Aufschwemmen der Zelle Pellet mit einer Pipette, in der Nähe von einzelligen Aussetzung zu erhalten, und übertragen Sie es auf einem 4 mm-Elektroporation-Küvette.
Hinweis: Pipettieren nach oben und unten ca. 10 – 15 Zeiten mit einer 100-1.000 µL PIPETTENSPITZE ist in der Regel ausreichend. - Legen Sie die Küvette in einem exponentiellen Zerfall Welle Electroporator ab und lassen Sie einen einzigen elektrischen Impuls bei 800 V, 25 µF und 400 Ω; Diese Einstellungen erzeugen einen Puls von ca. 8 ms Dauer.
-
Verwenden Sie eine neue PIPETTENSPITZE, um die Zelle Disruptate zu einem 1,5 mL Microcentrifuge Schlauch mit 400 µL eiskalte Phosphat-gepufferte Saccharoselösung (100 mM Natrium Monophosphate, 2 mM Dithiothreitol (DTT), 2 mM EDTA und 1,75 % Saccharose, pH 7,5) übertragen, und kurz mischen Sie, indem pipettieren.
- Optional: Überprüfen Sie effiziente Plasmamembran Electrodisruption17, 10 µL verdünnter Zelle Disruptate aus Schritt 3.3 mit 10 µL mischen 0,4 % Trypan blau in einem 1,5 mL Microcentrifuge Schlauch. Auf einer Zählkammer übertragen und stellen Sie sicher, dass der Anteil der positiven Zellen Trypan blau > 99 %.
- Lassen Sie die Probe in der Zählkammer für 30 min bei Raumtemperatur (20 – 25 ° C), und stellen Sie sicher, dass der Anteil der positiven Zellen Trypan blau geblieben > 99 %.
- Optional: Um sicherzustellen, dass der Electrodisruption nicht zu hart, das heißt, hat es nicht gestört, intrazelluläre Organellen, führen Sie Schritte 1.1 – 3,3, wie oben beschrieben, aber verwenden Sie eine größere Ausgangsmaterial (ein gut von einer 6-Well-Platte mit einem konfluierende Zellschicht von ~ 80 %), und 150 µL 10 % Saccharose in Schritt 2.5 und 150 µL Saccharose-Phosphat-gepufferte Lösung ohne DVB-t im Schritt 3.3 verwenden.
- Verwenden Sie eine Pipette, sorgfältig Schicht 200 µL der Zelle verdünnte Disruptate Lösung auf einem 1,2 mL Dichte Kissen von Phosphat-gepufferte 8 % (w/V) Dichte Gradienten Medium (z.B., 8 % Nycodenz, 50 mM Natriumphosphat, 2,2 % Saccharose, 1 mM EDTA) in einer 2 mL-Zentrifuge Rohr. Zentrifugieren bei 20.000 X g 45 min bei 4 ° C in einem Microcentrifuge mit Soft-Modus-Funktion (für sanfte Beschleunigung und Verzögerung), und die Rohre sorgfältig auf Eis gelegt.
- Entfernen Sie vorsichtig die ~ 200 µL Top Bruchteil, und achten Sie nicht auf jede Dichte Gradienten mittlere Lösung abholen, und übertragen auf einen frischen Microcentrifuge Schlauch 60 µL.
Hinweis: Dies sollte Zytosol von außergewöhnlicher Reinheit, genannt "Zellsaft"21enthalten. - Testen Sie die Reinheit des Bruches in den oben genannten Schritt durch western-Blot Analysen der Organellen enthalten Proteine, mit Standardtechniken und 4 – 20 % Steigung Gele16zu erhalten.
- Führen Sie z. B. Immunoblotting für Cathepsin B21, Cytochrom c und Protein umgeformt Isomerase, um sicherzustellen, dass der Stromschlag im Schritt 3.2 nicht Lysosomen, Mitochondrien oder endoplasmatische Retikulum bzw. gestört hat und Immunoblot für LDH, Vorhandensein eines zytosolischen Proteins in den Zellsaft zu überprüfen.
- Parallel führen Sie Immunoblotting auf Protein-Extrakte hergestellt aus der gesamten Zelle Disruptate Lösung16 zu bestätigen, dass die Antikörper verwendet die Organelle enthaltenen Proteine erkennen können, die geprüft werden.
- Optional: Überprüfen Sie effiziente Plasmamembran Electrodisruption17, 10 µL verdünnter Zelle Disruptate aus Schritt 3.3 mit 10 µL mischen 0,4 % Trypan blau in einem 1,5 mL Microcentrifuge Schlauch. Auf einer Zählkammer übertragen und stellen Sie sicher, dass der Anteil der positiven Zellen Trypan blau > 99 %.
- Wiederholen Sie die Schritte 3.1-3.3 für jede Probe.
- Entfernen Sie 550 µL aus jeder verdünnte Zelle Disruptate Lösung (erhältlich bei Schritt 3.3) auf 2 mL Mikrozentrifugenröhrchen mit 900 µL eiskalte Wiederfreisetzung Puffer (50 mM Natrium Monophosphate, 1 mM DVB-t, 1 mM EDTA und 5,9 % Saccharose, pH 7,5) ergänzt mit 0,5 % BSA und 0,01 % Tween-20 und Mix kurz durch pipettieren.
- Zentrifuge bei 18.000 X g 45 min bei 4 ° C zu produzieren pellets mit "sedimentierte LDH". Aspirieren Sie gründlich den überstand (mit Absaugung) um die Pellets so trocken wie möglich zu verlassen. Legen Sie die Proben in einem-80 ° C Gefrierschrank.
- 150 µL aus jeder verdünnte Zelle Disruptate Lösung (erhältlich bei Schritt 3.3), neue Röhren, und die Proben in einem-80 ° C Gefrierschrank. Verwenden Sie diese Beispiele, um die "totale LDH" Ebenen in den Zellen bestimmen.
Hinweis: An dieser Stelle kann das Experiment für angehalten werden so lange wie gewünscht.
(4) LDH Extraktion und Messung der LDH enzymatische Aktivität
- Die "sedimentierte LDH" (aus Schritt 3,6) und "total LDH" Proben (aus Schritt 3,7) auf Eis Auftauen.
- Fügen Sie 300 µL der eiskalte Wiederfreisetzung Puffer mit 1,5 % Triton X-405 auf der "Gesamt-LDH" Proben (Gewinnung einer Endkonzentration von Triton X-405 von 1 %). Drehen Sie die Proben auf einer Walze in einem kalten Zimmer (4 – 8 ° C) für 30 min.
- Fügen Sie 750 µL eiskalte Wiederfreisetzung Puffer mit 1 % Triton X-405 auf die "sedimentierte LDH" Proben hinzu und aufzuwirbeln Sie die Pellets mit einer Pipette, bis eine homogene Lösung erreicht ist.
- Zentrifugieren Sie Proben aus Schritt 4.2 und 4.3 bei 18.000 X g für 5 min bei 4 ° C, Sediment ungelösten zelltrümmer.
- Mix 4 Teile des kalten 65 mM Imidazol (pH 7.5)/0.75 mM Pyruvat mit einem Teil der kalten 65 mM Imidazol (pH 7,5) / 1,8 mM NADH eine funktionierende Lösung zu erhalten, die für mindestens drei Wochen bei 4 ° c stabil ist
- Mischen Sie 3 – 30 µL der Überstände ab Schritt 4.4 mit 200 µL der Schritt 4.5 funktionierende Lösung.
- Bestimmen Sie die Höhe des LDH durch LDH enzymatische Aktivität als der Rückgang der Nicotinamid-adenin-Dinucleotide (reduzierte Form) messen (NADH) Absorption bei 340 nm bei 37 ° C im Vergleich zu einem Standard mit einer bekannten LDH-Konzentration. Extinktion Messungen durchführen, bis die Reaktion abgeschlossen, d. h. bis die Absorption bei 340 genähert hat nm nicht mehr mit der Zeit ändert.
Hinweis: Dies ist die klassische biochemische Methode, LDH-Aktivität zu messen. Obwohl das aktuelle Protokoll die Reaktion bei 37 ° C durchführt, kann es auch bei Raumtemperatur (20 – 25 ° C), erfolgen die ist sinnvoll, wenn manuelle Spektrophotometrie zu tun. Das aktuelle Protokoll verwendet eine Roboter-multianalyzer Instrument, das in einer automatisierten Weise vermischt sich Proben mit Arbeitslösung in eine 96-Well-Platte, und misst die Absorption bei 340 nm bei 37 ° C alle 20 s 3 Minuten lang. Danach die Gerätesoftware berechnet die Konzentration von LDH, ausgedrückt als Einheiten (U) / L, durch den Vergleich der Neigung der Extinktion Messungen im Laufe der Zeit im Vergleich zu einer standard-Kurve zu erhalten, durch Kalibrierung mit einem Standard von bekannten LDH Konzentration. Des linearen Bereichs der Erkennung durch diesen Ansatz wird 30-1.500 U/L. Als Alternative gibt es eine Vielzahl von kommerziell erhältlichen Kits, LDH zu messen. Einige davon basieren auf Kupplung die enzymatische Reaktion auf die Generation der kolorimetrischen oder fluoreszierende Produkte, Erkennung durch andere Mittel als UV Spektralphotometrie und mit anderen linearen reicht der Erkennung aktivieren.
5. Berechnung der LDH-Sequestrierung
- Berechnung des Prozentsatzes des sedimentierten LDH, total LDH für jede Probe unter die Verdünnungen und Probenahme zu berücksichtigen:
Sedimentierte LDH (%) =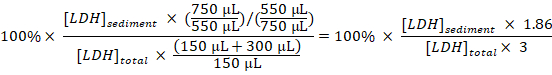
Hinweis: Während der Schritte ist 3.1-3.3 rund 50 µL verloren durch den Transfer in und aus der Elektroporation-Küvette. So aus mit insgesamt 750 µL (statt 800 µL) des verdünnten Zelle Disruptate im Schritt 3.3 berechnen. - Subtrahieren Sie den Prozentsatz der sedimentierte LDH erwarb die Proben aus unbehandelten Zellen (Schritt 1.3.2) aus dem Prozentsatz der sedimentierte LDH in Proben von experimentell behandelten Zellen und dividieren durch die Behandlungszeit mit Baf um den Prozentsatz der zu erhalten erhalten LDH pro Stunde in die Abfrageperiode abgesondert:
Sequestriert LDH (% / h) =
Ergebnisse
Mit dem Protokoll beschrieben hier, Masse selbstverdauende Sequestrierung Aktivität in einer Reihe von verschiedenen Säugetieren Zelllinien, einschließlich LAPC4, DU145, Huh7, PNT2A, HeLa, VCaP, H3122, Hec1A, T47D, U2OS, PC3, MCF-7, G361, Maus embryonale Fibroblasten (MEFs), RPE-1, HEK293, BJ und LNCaP Zellen gemessen wurde. Sequestrierung war unter basalen Bedingungen (im kompletten, nährstoffreichen Medium) bewertet, oder in Zellen verhungert akut für Serum und Aminosäuren (eine <...
Diskussion
Zusammenfassend lässt sich sagen ist das Protokoll hier beschrieben eine zuverlässigere und vielseitig einsetzbar-Methode, um lose selbstverdauende Sequestrierung in Säugetierzellen zu überwachen. Im Vergleich zu der ursprünglichen Methode12,16, haben wir etliche unnötige Schritte entfernt, einige der verbleibenden Schritte vereinfacht und eingeführt, eine erhebliche Herabsetzung. Infolgedessen das Protokoll in Bezug auf Kosten und Zeit-Effizienz erheblich...
Offenlegungen
Die Autoren haben keinen Interessenkonflikt.
Danksagungen
Diese Arbeit wurde von Research Council of Norway, der Universität von Oslo, Anders Jahre Foundation, der Nansen-Stiftung und das Erbe in der Erinnerung an Henrik Homan finanziell unterstützt. Wir danken Dr. Noboru Mizushima für die ATG5 + / + MEFs und ATG5/MEFs, Dr. Masaaki Komatsu für die ATG7 + / + MEFs und ATG7/MEFs und Dr. Shizuo Akira für die ATG9A + / + MEFs und ATG9A/MEFs. Wir danken Frank Sætre für technische Hilfe und Dr. pro O. Seglen für konstruktive methodische Diskussionen.
Materialien
| Name | Company | Catalog Number | Comments |
| 1.5 mL and 2 mL microcentrifuge tubes | Eppendorf | 211-2130 and 211-2120 | |
| 12-well plates | Falcon | 353043 | |
| Accumax cell detachment solution | Innovative Cell Technologies | A7089 | Keep aliquots at -20 °C for years, and in fridge for a few months |
| Bafilomycin A1 | Enzo | BML-CM110-0100 | Dissolve in DMSO |
| BJ cells | ATCC | CRL-2522 | use at passage <30 |
| Bovine serum albumin (BSA) | VWR | 422361V | |
| Burker counting chamber | Fisher Scientific | 139-658585 | |
| Countess Cell Counting Chamber Slides | ThermoFisher Scientfic | C10228 | |
| Countess II Automated Cell Counter | ThermoFisher Scientfic | AMQAX1000 | |
| Cover glass for the Burker counting chamber | Fisher Scientific | 139-658586 | |
| Criterion Tris-HCl Gel, 4–20%, 26-well, 15 µL, 13.3 cm x 8.7 cm (W x L) | Bio-Rad | 3450034 | |
| DTT | Sigma-Aldrich | D0632 | |
| Earle's balanced salt solution (EBSS) | Gibco | 24010-043 | conatains 0.1% glucose |
| EDTA | Sigma-Aldrich | E7889 | |
| Electroporation cuvette (4 mm) | Bio-Rad | 1652088 | |
| Exponential decay wave electroporator | BTX Harvard Apparatus | EMC 630 | |
| Fetal bovine serum (FBS) | Sigma | F7524 | 10% final concentration in RPMI 1640 medium |
| HEK293 cells | ATCC | CRL-1573 | |
| Imidazole | Sigma-Aldrich | 56750 | Autoclave a 65 mM solution and keep in fridge for months |
| Incubator; Autoflow IR Direct Heat CO2 incubator | NuAire | NU-5510E | |
| Lipofectamine RNAiMAX Transfection Reagent | ThermoFisher | 13778150 | |
| LNCaP cells | ATCC | CRL-1740 | use at passage <30 |
| 3-Methyl Adenine (3MA) | Sigma-Aldrich | M9281 | Stock 100 mM in RPMI in -20 °C. Heat stock to 65 °C for 10 min, and use at 10 mM final concentration |
| Refridgerated Microcentrifuge | Beckman Coulter Life Sciences | 368831 | |
| Refridgerated Microcentrifuge with soft-mode function | Eppendorf | Eppendorf 5417R | |
| MRT67307 hydrochloride (ULKi) | Sigma-Aldrich | SML0702 | Inhibits ULK kinase activity. Dissolve in DMSO. |
| MaxMat Multianalyzer instrument | Erba Diagnostics | PL-II | |
| MCF7 cells | ATCC | HTB-22 | |
| NADH | Merck-Millipore | 1.24644.001 | |
| Nycodenz | Axis-Shield | 1002424 | |
| Opti-MEM Reduced Serum Medium | ThermoFisher | 31985062 | |
| Phosphate-buffered saline (PBS) | Gibco | 20012-019 | |
| Pipette tips 3 (0.5-20 µL) | VWR | 732-2223 | Thermo Fischer ART Barrier tips |
| Pipette tips (1-200 µL) | VWR | 732-2207 | Thermo Fischer ART Barrier tips |
| Pipette tips (100-1,000 µL) | VWR | 732-2355 | Thermo Fischer ART Barrier tips |
| Pipettes | ThermoFisher | 4701070 | Finnpipette F2 GLP Kit |
| Poly-D-lysine | Sigma-Aldrich | P6407-10X5MG | Make a 1 mg/mL stock solution in sterile H2O. This solution is stable at -20 °C for at least 1 year. |
| Pyruvate | Merck-Millipore | 1066190050 | |
| RPE-1 cells (hTERT RPE-1) | ATCC | CRL-4000 | |
| RPMI 1640 | Gibco | 21875-037 | |
| SAR-405 | ApexBio | A8883 | Inhibits phosphoinositide 3-kinase class III (PIK3C3). Dissolve in DMSO. |
| Silencer Select Negative Control #1 (siCtrl) | ThermoFisher/Ambion | 4390843 | |
| Silencer Select ATG9-targeting siRNA (siATG9A) | ThermoFisher/Ambion | s35504 | |
| Silencer Select FIP200-targeting siRNA (siFIP200) | ThermoFisher/Ambion | s18995 | |
| Silencer Select ULK1-targeting siRNA (siULK1) | ThermoFisher/Ambion | s15964 | |
| Silencer Select ULK2-targeting siRNA (siULK2) | ThermoFisher/Ambion | s18705 | |
| Silencer Select GABARAP-targeting siRNA (siGABARAP) | ThermoFisher/Ambion | s22362 | |
| Silencer Select GABARAPL1-targeting siRNA (siGABARAPL1) | ThermoFisher/Ambion | s24333 | |
| Silencer Select GABARAPL2-targeting siRNA (siGABARAPL2) | ThermoFisher/Ambion | s22387 | |
| Sodium phosphate monobasic dihydrate (NaH2PO4 • 2H2O) | Merck-Millipore | 1.06580.1000 | |
| Sodium phosphate dibasic dihydrate (Na2HPO4 • 2H2O ) | Prolabo | 28014.291 | |
| Sucrose | VWR | 443816T | 10% final concentration in water; filter through 0.45 µm filter and keep in fridge for months |
| Thapsigargin | Sigma-Aldrich | T9033 | Inhibits the SERCA ER Ca2+ pump. Dissolve in DMSO. |
| Triton X-405 | Sigma-Aldrich | X405 | 1% final |
| Trypan Blue stain 0.4% | Molecular Probes | T10282 | |
| Trypsin-EDTA (0.25% w/v Trypsin) | Gibco | 25200-056 | |
| Tween-20 | Sigma-Aldrich | P2287 | 0.01% final |
Referenzen
- Rubinsztein, D. C., Frake, R. A. Yoshinori Ohsumi's Nobel Prize for mechanisms of autophagy: from basic yeast biology to therapeutic potential. J R Coll Physicians Edinb. 46 (4), 228-233 (2016).
- Mizushima, N. The exponential growth of autophagy-related research: from the humble yeast to the Nobel Prize. FEBS Lett. 591 (5), 681-689 (2017).
- Seglen, P. O., et al. Macroautophagic cargo sequestration assays. Methods. 75, 25-36 (2015).
- Hoyvik, H., Gordon, P. B., Seglen, P. O. Use of a hydrolysable probe, [14C]lactose, to distinguish between pre-lysosomal and lysosomal steps in the autophagic pathway. Exp Cell Res. 166 (1), 1-14 (1986).
- Plomp, P. J., Gordon, P. B., Meijer, A. J., Hoyvik, H., Seglen, P. O. Energy dependence of different steps in the autophagic-lysosomal pathway. J Biol Chem. 264 (12), 6699-6704 (1989).
- Gordon, P. B., Seglen, P. O. Prelysosomal convergence of autophagic and endocytic pathways. Biochem Biophys Res Commun. 151 (1), 40-47 (1988).
- Holen, I., Gordon, P. B., Seglen, P. O. Protein kinase-dependent effects of okadaic acid on hepatocytic autophagy and cytoskeletal integrity. Biochem J. 284, 633-636 (1992).
- Kabeya, Y., et al. LC3, a mammalian homologue of yeast Apg8p, is localized in autophagosome membranes after processing. Embo j. 19 (21), 5720-5728 (2000).
- Szalai, P., et al. Autophagic bulk sequestration of cytosolic cargo is independent of LC3, but requires GABARAPs. Exp Cell Res. 333 (1), 21-38 (2015).
- Nguyen, T. N., et al. Atg8 family LC3/GABARAP proteins are crucial for autophagosome-lysosome fusion but not autophagosome formation during PINK1/Parkin mitophagy and starvation. J Cell Biol. , (2016).
- Pontano Vaites, L., Paulo, J. A., Huttlin, E. L., Harper, J. W. Systematic analysis of human cells lacking ATG8 proteins uncovers roles for GABARAPs and the CCZ1/MON1 regulator C18orf8/RMC1 in macro and selective autophagic flux. Mol Cell Biol. , (2017).
- Kopitz, J., Kisen, G. O., Gordon, P. B., Bohley, P., Seglen, P. O. Nonselective autophagy of cytosolic enzymes by isolated rat hepatocytes. J Cell Biol. 111 (3), 941-953 (1990).
- Bowman, E. J., Siebers, A., Altendorf, K. Bafilomycins: a class of inhibitors of membrane ATPases from microorganisms, animal cells, and plant cells. Proc Natl Acad Sci U S A. 85 (21), 7972-7976 (1988).
- Hadjivassiliou, A. G., Rieder, S. V. The enzymatic assay of pyruvic and lactic acids. A definitive procedure. Clin Chim Acta. 19 (3), 357-361 (1968).
- Bergmeyer, H. U., Bernt, E., Bergmeyer, H. U. . Methods of enzymatic analysis (2nd English ed). 2, 574-579 (1974).
- Engedal, N., et al. Modulation of intracellular calcium homeostasis blocks autophagosome formation. Autophagy. 9 (10), 1475-1490 (2013).
- Luhr, M., et al. A Simple Cargo Sequestration Assay for Quantitative Measurement of Nonselective Autophagy in Cultured Cells. Methods Enzymol. 587, 351-364 (2017).
- Mousavi, S. A., et al. Effects of inhibitors of the vacuolar proton pump on hepatic heterophagy and autophagy. Biochim Biophys Acta. 1510 (1-2), 243-257 (2001).
- Seglen, P. O., Gordon, P. B. 3-Methyladenine: specific inhibitor of autophagic/lysosomal protein degradation in isolated rat hepatocytes. Proc Natl Acad Sci U S A. 79 (6), 1889-1892 (1982).
- Ronan, B., et al. A highly potent and selective Vps34 inhibitor alters vesicle trafficking and autophagy. Nat Chem Biol. 10 (12), 1013-1019 (2014).
- Saetre, F., Hagen, L. K., Engedal, N., Seglen, P. O. Novel steps in the autophagic-lysosomal pathway. Febs j. 282 (11), 2202-2214 (2015).
- Klionsky, D. J., et al. Guidelines for the use and interpretation of assays for monitoring autophagy (3rd edition). Autophagy. 12 (1), 1-222 (2016).
- Seglen, P. O., Overbye, A., Saetre, F. Sequestration assays for mammalian autophagy. Methods Enzymol. 452, 63-83 (2009).
- Hurley, J. H., Young, L. N. Mechanisms of Autophagy Initiation. Annu Rev Biochem. 86, 225-244 (2017).
- Thastrup, O., Cullen, P. J., Drobak, B. K., Hanley, M. R., Dawson, A. P. Thapsigargin, a tumor promoter, discharges intracellular Ca2+ stores by specific inhibition of the endoplasmic reticulum Ca2(+)-ATPase. Proc Natl Acad Sci U S A. 87 (7), 2466-2470 (1990).
- Kuma, A., et al. The role of autophagy during the early neonatal starvation period. Nature. 432 (7020), 1032-1036 (2004).
- Komatsu, M., et al. Impairment of starvation-induced and constitutive autophagy in Atg7-deficient mice. J Cell Biol. 169 (3), 425-434 (2005).
- Saitoh, T., et al. Atg9a controls dsDNA-driven dynamic translocation of STING and the innate immune response. Proc Natl Acad Sci U S A. 106 (49), 20842-20846 (2009).
- Gordon, P. B., Seglen, P. O. Autophagic sequestration of [14C]sucrose, introduced into rat hepatocytes by reversible electro-permeabilization. Exp Cell Res. 142 (1), 1-14 (1982).
- An, H., Harper, J. W. Systematic analysis of ribophagy in human cells reveals bystander flux during selective autophagy. Nat Cell Biol. 20 (2), 135-143 (2018).
- Fass, E., Shvets, E., Degani, I., Hirschberg, K., Elazar, Z. Microtubules support production of starvation-induced autophagosomes but not their targeting and fusion with lysosomes. J Biol Chem. 281 (47), 36303-36316 (2006).
- Velikkakath, A. K., Nishimura, T., Oita, E., Ishihara, N., Mizushima, N. Mammalian Atg2 proteins are essential for autophagosome formation and important for regulation of size and distribution of lipid droplets. Mol Biol Cell. 23 (5), 896-909 (2012).
- Øverbye, A., Sætre, F., Hagen, L. K., Johansen, H. T., Seglen, P. O. Autophagic activity measured in whole rat hepatocytes as the accumulation of a novel BHMT fragment (p10), generated in amphisomes by the asparaginyl proteinase, legumain. Autophagy. 7 (9), 1011-1027 (2011).
- Kominami, E., Hashida, S., Khairallah, E. A., Katunuma, N. Sequestration of cytoplasmic enzymes in an autophagic vacuole-lysosomal system induced by injection of leupeptin. J Biol Chem. 258 (10), 6093-6100 (1983).
- Rosado, C. J., Mijaljica, D., Hatzinisiriou, I., Prescott, M., Devenish, R. J. Rosella: a fluorescent pH-biosensor for reporting vacuolar turnover of cytosol and organelles in yeast. Autophagy. 4 (2), 205-213 (2008).
- Katayama, H., Kogure, T., Mizushima, N., Yoshimori, T., Miyawaki, A. A sensitive and quantitative technique for detecting autophagic events based on lysosomal delivery. Chem Biol. 18 (8), 1042-1052 (2011).
- Ogier-Denis, E., et al. A heterotrimeric Gi3-protein controls autophagic sequestration in the human colon cancer cell line HT-29. J Biol Chem. 270 (1), 13-16 (1995).
- Seglen, P. O., Gordon, P. B., Tolleshaug, H., Hoyvik, H. Use of [3H]raffinose as a specific probe of autophagic sequestration. Exp Cell Res. 162 (1), 273-277 (1986).
- Luhr, M., Sætre, F., Engedal, N. The Long-lived Protein Degradation Assay: an Efficient Method for Quantitative Determination of the Autophagic Flux of Endogenous Proteins in Adherent Cell Lines. Bio-protocol. 8 (9), e2836 (2018).
- Ronning, O. W., Pettersen, E. O., Seglen, P. O. Protein synthesis and protein degradation through the cell cycle of human NHIK 3025 cells in vitro. Exp Cell Res. 123 (1), 63-72 (1979).
- Seglen, P. O., Grinde, B., Solheim, A. E. Inhibition of the lysosomal pathway of protein degradation in isolated rat hepatocytes by ammonia, methylamine, chloroquine and leupeptin. Eur J Biochem. 95 (2), 215-225 (1979).
- Seglen, P. O., Solheim, A. E. Valine uptake and incorporation into protein in isolated rat hepatocytes. Nature of the precursor pool for protein synthesis. Eur J Biochem. 85 (1), 15-25 (1978).
- Bauvy, C., Meijer, A. J., Codogno, P. Assaying of autophagic protein degradation. Methods Enzymol. 452, 47-61 (2009).
- Engedal, N., Seglen, P. O. Autophagy of cytoplasmic bulk cargo does not require LC3. Autophagy. 12 (2), 1-3 (2016).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten