Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Le test de fixation du Lactate déshydrogénase — Une méthode Simple et fiable pour déterminer l’activité de séquestration autophagique en vrac dans des cellules de mammifères
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Un protocole simple et bien validé pour mesurer l’activité de séquestration autophagique en vrac dans des cellules de mammifères est décrit ici. La méthode est basée sur la quantification de la proportion de la lactate déshydrogénase (LDH) dans les fractions de cellules sédimentables par rapport aux niveaux LDH cellulaires totales.
Résumé
En vrac autophagie est caractérisée par la séquestration de larges portions du cytoplasme en double/multi-membrane structures appelées autophagosomes. Un protocole simple de contrôler ce processus est décrit ici. En outre, les résultats typiques et validation expérimentale de la méthode dans des conditions inductrices d’autophagie dans divers types de cellules mammifères cultivées sont fournis. Au cours de l’autophagie en vrac, autophagosomes séquestrer le cytosol et aussi des protéines cytosoliques solubles, aux côtés d’autres cargaisons autophagie. LDH est stable et très abondant, soluble enzyme cytosolique qui est non sélective séquestré dans autophagosomes. Le montant de la séquestration du LDH reflète donc la quantité de séquestration autophagique en vrac. Pour efficacement et précisément déterminer la séquestration de LDH dans les cellules, nous utilisons un protocole basé sur electrodisruption de fractionnement qui sépare efficacement sédimentables LDH cytosolique, suivie de la mesure de l’activité enzymatique dans sédimentables fractions par rapport à des échantillons de cellules entières. Séquestration de l’autophagie est déterminée en soustrayant la proportion de sédimentables LDH dans les cellules non traitées que dans les cellules traitées. L’avantage de l’essai de séquestration de LDH est qu’il donne une mesure quantitative de la séquestration autophagique du fret endogène, par opposition à d’autres méthodes que soit impliquent l’expression ectopique de sondes de séquestration ou semi-quantitatifs protéase analyses de la protection des récepteurs ou des marqueurs de l’autophagie.
Introduction
Autophagie (du grec « se manger ») est un processus évolutif et conservé pour dégradation lysosomale/vacuolaire du matériel intracellulaire. Lors de la découverte des gènes liés autophagie (« ATG »), qui sont importants pour l’autophagie chez les levures et les humains, et la réalisation que l’autophagie joue un rôle important dans la santé humaine et de la maladie (reconnu par le prix Nobel de médecine ou de physiologie à 2016 Yoshinori Ohsumi), autophagie est rapidement devenu un des procédés plus intensément étudiés en cellule biologie1,2.
Macroautophagy (ci-après dénommé « autophagie ») est caractérisée par l’expansion et pliage de saccules membranaires intracellulaire (« phagophores ») dans des structures scellées, double - ou multi - membrane (« autophagosomes ») séquestrent efficacement le enveloppé de matériel du reste du cytoplasme. Lors de la fusion des autophagosomes avec les lysosomes, la membrane intérieure autophagosomal et la cargaison séquestrée est dégradée et recyclés. Autophagosomes peut piéger le matériel cytoplasmique en aléatoire (autophagie non sélectif) et manières sélectives (autophagie sélective). En vrac autophagie probablement représente un mélange de l’autophagie non-sélective et sélective.
Dans les années 60 et 70 (« l’ère morphologique » de la recherche de l’autophagie), séquestration autophagique a été principalement évaluée au moyen d’analyses ultrastructurales. Dans les années 1980 et au début des années 1990 (« l’ère biochimique ») par Seglen et collègues de travail — qui a étudié l’autophagie dans les hépatocytes primaires de rat — mis au point les premières méthodes pour mesurer quantitativement la séquestration autophagique activité3. À l’aide de ces tests, Seglen défini et caractérise les différentes étapes de la voie autophagique lysosomal4,5, découvert et a inventé l’amphisome6 (le produit de la fusion de l’endosome-autophagosome) et fut le premier à décrire le rôle de la phosphorylation des protéines l’autophagie règlement7. Cependant, après la découverte des ATGs dans les années 1990 (« l’ère moléculaire ») et la première caractérisation d’une protéine ATG8 chez les mammifères, les protéines associées aux microtubules 1 a/1 b-light chain 3 (LC3) 20008, l’utilisation de protéines ATG comme marqueurs pour la processus autophagique rapidement gagné en popularité, et les plus âgés et plus laborieux des méthodes biochimiques ont été laissés. En fait, sur les 18 dernières années, les tache occidentale et les analyses de microscopie de fluorescence de LC3 sont devenues de loin la plus populaire (et dans bien des cas la seule) moyens d’étudier l’autophagie des cellules de mammifères. L’avantage est la facilité par laquelle ces méthodes peuvent être effectuées. L’inconvénient est que l'on étudie un composant du panier (LC3) plutôt que de réel autophagique fret. Il s’agit d’un inconvénient plutôt sérieux, parce que la relation entre les Etats et/ou les flux de LC3 par la voie contre la séquestration et de la cargaison n’est pas très claire. En fait, nous avons montré que le flux de cargaison en vrac peut être maintenue à des niveaux élevés dans des conditions où il n’y a aucun flux LC3, malgré la présence de LC3 conjugués dans les cellules9. En outre, nous avons démontré que l’autophagie en vrac n’est pas affectée par l’appauvrissement LC3 efficace et donc probablement est indépendant du LC39. Cette constatation est confirmée plus tard par LC3 études knock out10,11, qui indiquent également que mitophagy Parkin-dépendante (l’autophagie sélective des mitochondries) est indépendant de LC310,11 .
En résumé, il n’y a absolument besoin pour analyses fret surveiller l’activité de l’autophagie. Façon optimale ces tests devraient être largement applicable, bien définie et facile à exécuter. Au cours de ces dernières années, nous avons pris un intérêt particulier pour le test de fixation du LDH, qui a été développé par Seglen dans les années 198012et repose sur la mesure de la cession de LDH cytosolique à sédimentables, cellule de contenant des vacuoles autophagique fractions. LDH est une protéine cytosolique stable, soluble qui est séquestrée facilement conjointement au moment où phagophores enwrap fret cytoplasmique. Séquestration de LDH est donc une mesure générale de séquestration de l’autophagie. LDH est exclusivement dégradée par la voie autophagique lysosomal12. Ainsi, en présence d’inhibiteurs de dégradation lysosomale, par exemple, bafilomycine A1 (Baf)13, directement les effets du traitement expérimental reflètent altérations dans l’activité de séquestration autophagie. En l’absence d’inhibiteurs de la dégradation, l’effet net des altérations dans la séquestration de la LDH et de la dégradation peut être mesurée.
Le test de fixation du LDH est largement applicable, puisque la LDH s’exprime fortement et de façon ubiquitaire dans tous les types de cellules, et des niveaux LDH peuvent être quantifiées avec précision par un dosage enzymatique14,15. Toutefois, l’original du protocole12 — établi dans les hépatocytes primaires de rat — est plutôt fastidieux et requiert une grande quantité d’à partir de matériel comme un condensateur de décharge électrique sur mesure. De manière progressive, nous avons progressivement transformé l’essai dans une méthode simple et versatile. Tout d’abord, le protocole original a été adapté pour une utilisation dans des cellules de mammifères lignes16. Deuxièmement, la méthode a été substantiellement à échelle réduite de3,9. Troisièmement, plusieurs étapes dans le protocole ont été éliminés, y compris un coussin de densité laborieuse étape17. En même temps, cela a permis une réduction encore plus loin de la méthode, dans le point de départ original d’utiliser une plaque de 10 cm par exemple16 pour utiliser un seul puits d’une plaque de 12 puits par échantillon (soitenviron 15 fois moins à partir le matériel)17. Quatrièmement, nous avons identifié un appareil commercial électroporation qui pourrait remplacer le condensateur de décharge électrique sur mesure17.
Notre protocole à jour de l’essai de fixation du LDH, qui comprend quelques simplifications supplémentaires de la méthode par rapport à la publiées antérieurement17 est présenté ici. En outre, un ensemble de résultats typiques obtenus dans un certain nombre de différents types de cellules s’affiche, et important, plusieurs lignes de validations expérimentales de la méthode à l’aide de knockdown pharmacologique mais aussi génétique et approches knockout sont fournis. Pour un projet global de flux du protocole entier, voir la Figure 1.
Protocole
1. cellule ensemencement et traitement
-
Cellules adhérentes de la culture dans des flacons de culture de tissu de2 cm 75 dans un incubateur humidifié avec 5 % de CO2 à 37 ° C, en utilisant le milieu de culture privilégié pour le type de cellule en question. Laissez les cellules jusqu'à ce qu’ils atteignent une couche de cellules de proximité-anastomosé.
Remarque : Utiliser un milieu RPMI 1640 additionné de 10 % de sérum bovin fœtal (SVF) pour LNCaP, HEK293, fibroblastes embryonnaires de souris (MEFs), BJ, MCF-7 et cellules pre-1.- Laver les cellules avec 3 mL 37 ° C tampon phosphate salin (PBS), pH 7,4. Remplacez les PBS avec 3 mL de 0,25 % (p/v), la trypsine-éthylènediaminetétraacétate (EDTA) et incuber la fiole dans un incubateur humidifié avec 5 % de CO2 à 37 ° C jusqu'à ce que les cellules détachement (2 à 5 min).
- Remettre en suspension les cellules individuelles avec 7 mL de milieu de culture contenant 10 % FBS. Mélanger une quantité de suspension de cellules de 10 µL à 10 µL de 0,4 % le bleu Trypan dans un tube de microcentrifuge, à l’aide d’une pointe de pipette 0,5 – 20 µL. Utiliser la même pipette pour pourvoir immédiatement une diapositive comptage de la chambre et compter les cellules dans un compteur de cellules automatisées.
-
Préparer une dilution (voir note ci-dessous) de la suspension cellulaire en faisant étape 1.1.2 culture moyenne contenant 10 % FBS et semences 1 mL de la suspension cellulaire dilué dans chaque puits d’une plaque (superficie ~3.8 cm2) en culture de tissu de 12 puits utilisant aseptique technique. Permettre la croissance dans un incubateur humidifié avec 5 % de CO2 à 37 ° C jusqu'à ce que la densité de la cellule souhaitée est atteinte, par exemple, de 60 à 90 % confluence à la récolte.
Remarque : La dilution appropriée de la suspension cellulaire qui donnera à la confluence de la cellule souhaitée à la récolte varie de type cellulaire de type cellulaire, ainsi que selon la durée et le type de traitements expérimentaux. Par conséquent, cela doit être évaluée empiriquement dans chaque cas.- Pour des expériences qui doivent être tous deux traitement et récolté 2 jours après le semis, semences 2,5 x 105 LNCaP, HEK293, ou MCF-7 cellules, MEFs4 5 x 10, 4 x 105 BJ ou 1,5 x 105 pre-1 éléments dans chaque puits de la plaque de 12 puits.
- Pour les cellules qui adhèrent librement, enduire les plaques avec le type de revêtement recommandé pour le type de cellule en question. Pour les cellules LNCaP (et HEK293) utiliser plaques recouvertes de poly-D-lysine (PDL).
- À cette fin, ajouter 500 µL PDL à 2,5 µg/mL stérile H2O dans chaque puits et incuber les boîtes dans un environnement stérile pendant 30 min à température ambiante (20 à 25 ° C). Retirer le PDL avec aspiration et laver chaque bien brièvement avec 1 mL stérile H2O.
Remarque : En général, l’étape 1.2.1 est faite sans traitement expérimental. Toutefois, si vous effectuez l’ARNi, il peut être pratique démarrer une transfection inverse avec le semis9.
-
Effectuer des traitements expérimentaux dans les puits en doubles ou en triple par État.
- Par exemple, traiter les cellules avec 50 nM de la Torin1 inhibiteur de mTOR, qui généralement est un inducteur efficace de séquestration autophagique, ou sous réserve des cellules à la famine aiguë de sérum et d’acides aminés par laver les cellules avec 1 mL d’Earle exempte d’acides aminés est équilibré Support de la Solution (EBSS) de sel et ensuite Incuber les cellules dans 1 mL de EBSS dans un incubateur humidifié avec 5 % de CO2 à 37 ° C.
- Laisser un jeu de puits non traitée afin de définir des niveaux de fond des sédimentables LDH.
- Ajouter une quantité saturante de l’inhibiteur de la fixation du post bafilomycines A1 (Baf)3,13,16,18 en l’absence ou la présence des traitements expérimentaux, 3 – 4 h avant cellule la récolte. Incuber les cellules dans un incubateur humidifié avec 5 % de CO2 à 37 ° C.
- Utilisez les 100 nM Baf pour LNCaP, HEK293, BJ, MCF-7 et cellules pre-1 et 10 nM Baf pour MEFs.
- Pour les traitements expérimentaux qui ont une durée de seulement 3 à 4 h (comme ces étape illustré en 1.3.1 ont généralement), ajouter Baf simultanément avec les traitements. Pour plus de traitements expérimentaux, attendre 3-4 h avant la récolte et ajouter 2 µL d’une 500 x concentré Baf stock directement dans le milieu.
- Mélanger en agitant la plaque immédiatement après l’ajout de la Baf. À ce stade, il est également recommandé d’ajouter macroautophagic inhibiteurs de la séquestration des contrôles, par exemple, 10 mM de la pan-phosphoinositide 3-kinase (PI3K) inhibiteur de la 3-méthyl adénine (3MA)19ou 10 µM de la classe sélective du PI3K III inhibiteur de la SAR-40520.
2. récolte et préparation pour Electrodisruption des cellules
- À la fin de la période de traitement, aspirer d’aspiration au milieu et ajouter 200 µL de solution détachement de cellules (préchauffé à 37 ° C) dans chaque puits. Incuber à 37 ° C jusqu'à ce que les cellules détachement (généralement autour de 5 min).
NOTE : Considérant que 0,25 % (p/v), la trypsine-EDTA peuvent servir au lieu de la solution de détachement cellulaire, ce dernier contient DNase, qui permet de réduire la viscosité des cellules individuelles. Aussi longtemps que le milieu est complètement aspiré, il n’est pas nécessaire de laver les cellules avant l’addition de trypsine-EDTA ou cellule solution de détachement. - Ajouter 500 µL température ambiante (20-25 ° C) PBS, pH 7,4, contenant 2 % (p/v) d’albumine sérique bovine (BSA) dans chaque puits et remettre en suspension à l’aide de la pipette jusqu'à ce qu’aucun amas de cellules ne sont visibles. Transférer immédiatement la suspension cellulaire aux tubes de microcentrifuge de 1,5 mL sur la glace.
Remarque : Sauf indication contraire, effectuez toutes les étapes subséquentes sur la glace. - Sédiments les cellules par centrifugation à 400 x g pendant 5 min à 4 ° C.
- Bien aspirer le surnageant (par aspiration) pour laisser les granules cellulaires aussi sèches que possible.
- Ajouter 400 µL 10 % (p/v) de sucrose (dans ultrapure H2O) dans chaque tube.
3. Membrane plasmique Electrodisruption et séparation des Fractions de cellules sédimentables et Total
- Resuspendre le culot cellulaire avec une pipette pour obtenir une suspension de cellules individuelles proche et de le transférer à une cuvette d’électroporation de 4 mm.
NOTE : Pipetage va-et-vient ~ 10 à 15 fois, à l’aide d’une pointe de pipette 100 – 1 000 µL, est généralement suffisant. - Placez la cuve dans une électroporateur vague de décroissance exponentielle et acquitter une seule impulsion électrique à 800 V, 25 µF et 400 Ω ; ces paramètres de produisent une impulsion de durée ~ 8 ms.
-
Un nouvel embout de la pipette permet de transférer le disruptate de cellules dans un tube de microtubes de 1,5 mL contenant 400 µL de solution de saccharose glacée de tampon phosphate (monophosphate de sodium 100 mM, 2 mM le dithiothréitol (DTT), EDTA de 2 mM et 1,75 % de sucrose, pH 7,5) et mélanger brièvement en pipetage.
- Facultatif : Pour vérifier la membrane plasmique efficace electrodisruption17, mélanger 10 µL de la disruptate cellulaire dilués à l’étape 3.3 avec 10 µL de 0,4 % le bleu Trypan dans un tube de microtubes de 1,5 mL. Transférer vers une chambre de comptage et de vérifier que le pourcentage de cellules positives le bleu Trypan est > 99 %.
- Laisser l’échantillon dans la chambre de comptage pendant 30 min à température ambiante (20 à 25 ° C) et vérifiez que le pourcentage de cellules positives le bleu Trypan est resté > 99 %.
- Facultatif : Pour vérifier que l’electrodisruption n’a pas été trop sévère, autrement dit, il n’a pas perturbé organites intracellulaires, effectuez les étapes 1.1 – 3.3 comme décrit ci-dessus, mais utilisez un plus grand produit de départ (un puits d’une plaque de 6 puits avec une couche de cellules confluentes de ~ 80 %), et utiliser 150 µL 10 % de saccharose en étape 2.5 et 150 µL tampon phosphate une solution de saccharose sans TNT à l’étape 3.3.
- Utiliser une pipette à soigneusement couche 200 µL de la solution de disruptate cellule dilué sur un coussin de densité 1,2 mL de milieu dégradé densité de 8 % (p/v) tamponnée au phosphate (par exemple, 8 % Nycodenz, phosphate de sodium de 50 mM, 2,2 % de sucrose, EDTA 1 mM) dans une centrifugeuse de 2 mL tube. Centrifuger à 20 000 x g pendant 45 min à 4 ° C dans une micro-centrifugeuse avec fonction soft-mode (pour légère accélération et de décélération) et rangez soigneusement les tubes sur la glace.
- Retirez soigneusement les 60 µL de la fraction supérieure ~ 200 µL, en veillant pas à ramasser toute solution moyenne gradient de densité et transférer dans un tube de microcentrifuge fraîches.
Remarque : Cela devrait contenir cytosol d’une pureté exceptionnelle, appelée « suc cellulaire »21. - Test de la pureté de la fraction obtenue dans l’étape précédente, en effectuant des analyses par transfert western des protéines contenus organite, utilisant des techniques standards et de gels dégradé de 4 à 20 %16.
- Exécuter par exemple immunoblotting pour cathepsine B21, le cytochrome c et isomérase de bisulfure de protéine, pour vérifier que l’électrocution à l’étape 3.2 n'a pas perturbé les lysosomes, mitochondries ou réticulum endoplasmique, respectivement et immunoblot pour LDH pour vérifier la présence d’une protéine cytosolique dans le suc cellulaire.
- En parallèle, effectuer l’immunotransfert sur extraits protéiques issus de la cellule total disruptate solution16 pour confirmer que les anticorps utilisés peuvent détecter les protéines organelle autonomes qui sont évaluées.
- Facultatif : Pour vérifier la membrane plasmique efficace electrodisruption17, mélanger 10 µL de la disruptate cellulaire dilués à l’étape 3.3 avec 10 µL de 0,4 % le bleu Trypan dans un tube de microtubes de 1,5 mL. Transférer vers une chambre de comptage et de vérifier que le pourcentage de cellules positives le bleu Trypan est > 99 %.
- Répétez les étapes 3.1 – 3.3 pour chaque échantillon.
- Séparer la 550 µL de chaque solution de disruptate cellule dilué (obtenue à l’étape 3.3) tubes de microcentrifuge de 2 mL contenant 900 µL glacee tampon de resuspension (monophosphate de sodium de 50 mM, 1 mM DTT, EDTA 1 mM et 5,9 % de sucrose, pH 7,5) additionné de 0,5 % de BSA et 0,01 % Tween-20 et mélanger brièvement par pipetage.
- Centrifuger à 18 000 x g pendant 45 min à 4 ° C pour produire des granulés contenant « sédimentée LDH ». Bien aspirer le surnageant (par aspiration) pour laisser les granules aussi sèches que possible. Placer les échantillons dans un congélateur à-80 ° C.
- Transférer 150 µL de chaque solution de disruptate cellule dilué (obtenue à l’étape 3.3) dans de nouveaux tubes et placer les échantillons dans un congélateur à-80 ° C. Utiliser ces échantillons pour déterminer les niveaux de « total des LDH » dans les cellules.
Remarque : À ce stade, l’expérience peut être suspendue pour aussi longtemps que vous le souhaitez.
4. LDH Extraction et mesure de l’activité enzymatique de la LDH
- Décongeler la LDH « sédimentée » (de l’étape 3.6) et échantillons « total LDH » (de l’étape 3,7) sur la glace.
- Ajouter 300 µL de tampon de resuspension glacée contenant 1,5 % X-405 Triton à la LDH « totale » des échantillons (ce qui donnerait une concentration finale de Triton X-405 de 1 %). Faire pivoter les échantillons sur un rouleau dans une chambre froide (4 – 8 ° C) pendant 30 min.
- Ajouter 750 µL de tampon de resuspension glacée avec 1 % X-405 Triton à la LDH « sédimentée » échantillons et remettre en suspension les boulettes avec une pipette jusqu'à obtenir une solution homogène.
- Centrifuger les échantillons à l’étape 4.2 et 4.3 à 18 000 x g pendant 5 min à 4 ° C pour les sédiments non dissous les débris cellulaires.
- Mix 4 pièces de l’imidazole froid 65 mM (pH 7.5)/0.75 mM pyruvate avec une part de l’imidazole froid 65 mM (pH 7.5) / 1,8 mM NADH pour obtenir une solution de travail qui est stable pendant au moins trois semaines à 4 ° C.
- Mélanger 3 – 30 µL de surnageants de l’étape 4.4 avec 200 µL de la solution de travail étape 4.5.
- Déterminer le montant de la LDH en mesurant l’activité enzymatique LDH comme la baisse de la nicotinamide adénine dinucléotide (forme réduite) absorbance (NADH) à 340 nm à 37 ° C par rapport à un étalon avec une concentration connue de la LDH. Effectuer des mesures d’absorbance jusqu'à ce que la réaction a approché l’achèvement, c'est-à-dire jusqu'à l’absorbance à 340 nm ne change avec le temps.
NOTE : Ceci est la méthode biochimique classique pour mesurer l’activité de la LDH. Bien que le protocole actuel effectue la réaction à 37 ° C, elle peut également être réalisée à température ambiante (20 à 25 ° C), qui est conseillé si spectrophotométrie manuelle à faire. Le protocole actuel utilise un instrument robotique multianalyzer, qui, de manière automatisée, mélange d’échantillons avec la solution de travail dans une plaque à 96 puits et mesure l’absorbance à 340 nm à 37 ° C toutes les 20 s pendant 3 min. Par la suite, le logiciel de l’instrument calcule la concentration de la LDH, exprimée en unités (U) / L, en comparant la pente de la mesure d’absorbance dans le temps par rapport à une courbe d’étalonnage obtenue par étalonnage avec une norme de LDH connu concentration. La gamme linéaire de détection de cette approche est 30 – 1 500 U/L. Comme alternative, il existe une grande variété de kits disponibles dans le commerce pour mesurer la LDH. Certains d'entre eux sont basés sur la réaction enzymatique à la génération de produits colorimétriques ou fluorescentes, permettant la détection par d’autres moyens que la spectrophotométrie UV et avec les autres cuisinières linéaires de détection de couplage.
5. calcul de la séquestration du LDH
- Calculer le pourcentage de LDH sédimenté de LDH totale pour chaque échantillon, prenant les dilutions et d’échantillonnage en compte :
LDH sédimentée (%) =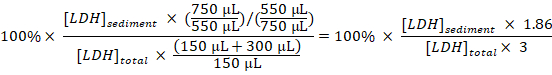
Remarque : Au cours des étapes 3.1 – 3.3 environ 50 µL est perdue en raison du transfert dans et hors de la cuvette d’électroporation. Ainsi, calculer d’avoir un total de 750 µL (au lieu de 800 µL) de disruptate cellule dilués à l’étape 3.3. - Soustraire le pourcentage de LDH sédimenté acquis dans les échantillons de cellules non traitées (étape 1.3.2) du pourcentage de LDH sédimenté acquis dans des échantillons de cellules traitées expérimentalement et diviser par la durée du traitement avec la Baf pour obtenir le pourcentage de LDH séquestré par heure pendant la période d’échantillonnage :
Séquestré LDH (% / h) =
Résultats
En utilisant le protocole décrit ici, activité autophagique séquestration en vrac dans un certain nombre de lignées cellulaires chez les mammifères, y compris LAPC4, DU145, Huh7, PNT2A, HeLa, VCaP, H3122, Hec1A, T47D, U2OS, PC3, MCF-7, G361, (MEFs) les fibroblastes embryonnaires de souris, pre-1, On a mesuré les cellules HEK293, BJ et LNCaP. Sequestration a été évaluée dans les conditions basales (dans un milieu complet, riche en nutriments), ou dans les cellules aiguë affamée...
Discussion
En résumé, le protocole décrit ici représente une méthode fiable et largement applicable pour surveiller l’activité de séquestration autophagique en vrac dans des cellules de mammifères. Par rapport à la méthode originale12,16, nous avons supprimé un certain nombre d’étapes inutiles, simplifié plusieurs des étapes restantes et introduit une substantielle réduction d’échelle. Par conséquent, le protocole est grandement amélioré par rapport...
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts.
Remerciements
Ce travail a été soutenu financièrement par le Conseil norvégien de la recherche, l’Université d’Oslo, le Anders Jahre Foundation, la Fondation de Nansen et l’héritage dans la mémoire de Henrik Homan. Nous remercions m. Noboru Mizushima pour le ATG5 + / + MEFs et ATG5-/-MEFs, Dr Masaaki Komatsu pour le ATG7 + / + MEFs et ATG7-/-MEFs et Dr Shizuo Akira pour le ATG9A + / + MEFs et ATG9A-/-MEFs. Nous remercions Frank Sætre pour assistance technique et Dr par O. Seglen pour des débats constructifs méthodologiques.
matériels
| Name | Company | Catalog Number | Comments |
| 1.5 mL and 2 mL microcentrifuge tubes | Eppendorf | 211-2130 and 211-2120 | |
| 12-well plates | Falcon | 353043 | |
| Accumax cell detachment solution | Innovative Cell Technologies | A7089 | Keep aliquots at -20 °C for years, and in fridge for a few months |
| Bafilomycin A1 | Enzo | BML-CM110-0100 | Dissolve in DMSO |
| BJ cells | ATCC | CRL-2522 | use at passage <30 |
| Bovine serum albumin (BSA) | VWR | 422361V | |
| Burker counting chamber | Fisher Scientific | 139-658585 | |
| Countess Cell Counting Chamber Slides | ThermoFisher Scientfic | C10228 | |
| Countess II Automated Cell Counter | ThermoFisher Scientfic | AMQAX1000 | |
| Cover glass for the Burker counting chamber | Fisher Scientific | 139-658586 | |
| Criterion Tris-HCl Gel, 4–20%, 26-well, 15 µL, 13.3 cm x 8.7 cm (W x L) | Bio-Rad | 3450034 | |
| DTT | Sigma-Aldrich | D0632 | |
| Earle's balanced salt solution (EBSS) | Gibco | 24010-043 | conatains 0.1% glucose |
| EDTA | Sigma-Aldrich | E7889 | |
| Electroporation cuvette (4 mm) | Bio-Rad | 1652088 | |
| Exponential decay wave electroporator | BTX Harvard Apparatus | EMC 630 | |
| Fetal bovine serum (FBS) | Sigma | F7524 | 10% final concentration in RPMI 1640 medium |
| HEK293 cells | ATCC | CRL-1573 | |
| Imidazole | Sigma-Aldrich | 56750 | Autoclave a 65 mM solution and keep in fridge for months |
| Incubator; Autoflow IR Direct Heat CO2 incubator | NuAire | NU-5510E | |
| Lipofectamine RNAiMAX Transfection Reagent | ThermoFisher | 13778150 | |
| LNCaP cells | ATCC | CRL-1740 | use at passage <30 |
| 3-Methyl Adenine (3MA) | Sigma-Aldrich | M9281 | Stock 100 mM in RPMI in -20 °C. Heat stock to 65 °C for 10 min, and use at 10 mM final concentration |
| Refridgerated Microcentrifuge | Beckman Coulter Life Sciences | 368831 | |
| Refridgerated Microcentrifuge with soft-mode function | Eppendorf | Eppendorf 5417R | |
| MRT67307 hydrochloride (ULKi) | Sigma-Aldrich | SML0702 | Inhibits ULK kinase activity. Dissolve in DMSO. |
| MaxMat Multianalyzer instrument | Erba Diagnostics | PL-II | |
| MCF7 cells | ATCC | HTB-22 | |
| NADH | Merck-Millipore | 1.24644.001 | |
| Nycodenz | Axis-Shield | 1002424 | |
| Opti-MEM Reduced Serum Medium | ThermoFisher | 31985062 | |
| Phosphate-buffered saline (PBS) | Gibco | 20012-019 | |
| Pipette tips 3 (0.5-20 µL) | VWR | 732-2223 | Thermo Fischer ART Barrier tips |
| Pipette tips (1-200 µL) | VWR | 732-2207 | Thermo Fischer ART Barrier tips |
| Pipette tips (100-1,000 µL) | VWR | 732-2355 | Thermo Fischer ART Barrier tips |
| Pipettes | ThermoFisher | 4701070 | Finnpipette F2 GLP Kit |
| Poly-D-lysine | Sigma-Aldrich | P6407-10X5MG | Make a 1 mg/mL stock solution in sterile H2O. This solution is stable at -20 °C for at least 1 year. |
| Pyruvate | Merck-Millipore | 1066190050 | |
| RPE-1 cells (hTERT RPE-1) | ATCC | CRL-4000 | |
| RPMI 1640 | Gibco | 21875-037 | |
| SAR-405 | ApexBio | A8883 | Inhibits phosphoinositide 3-kinase class III (PIK3C3). Dissolve in DMSO. |
| Silencer Select Negative Control #1 (siCtrl) | ThermoFisher/Ambion | 4390843 | |
| Silencer Select ATG9-targeting siRNA (siATG9A) | ThermoFisher/Ambion | s35504 | |
| Silencer Select FIP200-targeting siRNA (siFIP200) | ThermoFisher/Ambion | s18995 | |
| Silencer Select ULK1-targeting siRNA (siULK1) | ThermoFisher/Ambion | s15964 | |
| Silencer Select ULK2-targeting siRNA (siULK2) | ThermoFisher/Ambion | s18705 | |
| Silencer Select GABARAP-targeting siRNA (siGABARAP) | ThermoFisher/Ambion | s22362 | |
| Silencer Select GABARAPL1-targeting siRNA (siGABARAPL1) | ThermoFisher/Ambion | s24333 | |
| Silencer Select GABARAPL2-targeting siRNA (siGABARAPL2) | ThermoFisher/Ambion | s22387 | |
| Sodium phosphate monobasic dihydrate (NaH2PO4 • 2H2O) | Merck-Millipore | 1.06580.1000 | |
| Sodium phosphate dibasic dihydrate (Na2HPO4 • 2H2O ) | Prolabo | 28014.291 | |
| Sucrose | VWR | 443816T | 10% final concentration in water; filter through 0.45 µm filter and keep in fridge for months |
| Thapsigargin | Sigma-Aldrich | T9033 | Inhibits the SERCA ER Ca2+ pump. Dissolve in DMSO. |
| Triton X-405 | Sigma-Aldrich | X405 | 1% final |
| Trypan Blue stain 0.4% | Molecular Probes | T10282 | |
| Trypsin-EDTA (0.25% w/v Trypsin) | Gibco | 25200-056 | |
| Tween-20 | Sigma-Aldrich | P2287 | 0.01% final |
Références
- Rubinsztein, D. C., Frake, R. A. Yoshinori Ohsumi's Nobel Prize for mechanisms of autophagy: from basic yeast biology to therapeutic potential. J R Coll Physicians Edinb. 46 (4), 228-233 (2016).
- Mizushima, N. The exponential growth of autophagy-related research: from the humble yeast to the Nobel Prize. FEBS Lett. 591 (5), 681-689 (2017).
- Seglen, P. O., et al. Macroautophagic cargo sequestration assays. Methods. 75, 25-36 (2015).
- Hoyvik, H., Gordon, P. B., Seglen, P. O. Use of a hydrolysable probe, [14C]lactose, to distinguish between pre-lysosomal and lysosomal steps in the autophagic pathway. Exp Cell Res. 166 (1), 1-14 (1986).
- Plomp, P. J., Gordon, P. B., Meijer, A. J., Hoyvik, H., Seglen, P. O. Energy dependence of different steps in the autophagic-lysosomal pathway. J Biol Chem. 264 (12), 6699-6704 (1989).
- Gordon, P. B., Seglen, P. O. Prelysosomal convergence of autophagic and endocytic pathways. Biochem Biophys Res Commun. 151 (1), 40-47 (1988).
- Holen, I., Gordon, P. B., Seglen, P. O. Protein kinase-dependent effects of okadaic acid on hepatocytic autophagy and cytoskeletal integrity. Biochem J. 284, 633-636 (1992).
- Kabeya, Y., et al. LC3, a mammalian homologue of yeast Apg8p, is localized in autophagosome membranes after processing. Embo j. 19 (21), 5720-5728 (2000).
- Szalai, P., et al. Autophagic bulk sequestration of cytosolic cargo is independent of LC3, but requires GABARAPs. Exp Cell Res. 333 (1), 21-38 (2015).
- Nguyen, T. N., et al. Atg8 family LC3/GABARAP proteins are crucial for autophagosome-lysosome fusion but not autophagosome formation during PINK1/Parkin mitophagy and starvation. J Cell Biol. , (2016).
- Pontano Vaites, L., Paulo, J. A., Huttlin, E. L., Harper, J. W. Systematic analysis of human cells lacking ATG8 proteins uncovers roles for GABARAPs and the CCZ1/MON1 regulator C18orf8/RMC1 in macro and selective autophagic flux. Mol Cell Biol. , (2017).
- Kopitz, J., Kisen, G. O., Gordon, P. B., Bohley, P., Seglen, P. O. Nonselective autophagy of cytosolic enzymes by isolated rat hepatocytes. J Cell Biol. 111 (3), 941-953 (1990).
- Bowman, E. J., Siebers, A., Altendorf, K. Bafilomycins: a class of inhibitors of membrane ATPases from microorganisms, animal cells, and plant cells. Proc Natl Acad Sci U S A. 85 (21), 7972-7976 (1988).
- Hadjivassiliou, A. G., Rieder, S. V. The enzymatic assay of pyruvic and lactic acids. A definitive procedure. Clin Chim Acta. 19 (3), 357-361 (1968).
- Bergmeyer, H. U., Bernt, E., Bergmeyer, H. U. . Methods of enzymatic analysis (2nd English ed). 2, 574-579 (1974).
- Engedal, N., et al. Modulation of intracellular calcium homeostasis blocks autophagosome formation. Autophagy. 9 (10), 1475-1490 (2013).
- Luhr, M., et al. A Simple Cargo Sequestration Assay for Quantitative Measurement of Nonselective Autophagy in Cultured Cells. Methods Enzymol. 587, 351-364 (2017).
- Mousavi, S. A., et al. Effects of inhibitors of the vacuolar proton pump on hepatic heterophagy and autophagy. Biochim Biophys Acta. 1510 (1-2), 243-257 (2001).
- Seglen, P. O., Gordon, P. B. 3-Methyladenine: specific inhibitor of autophagic/lysosomal protein degradation in isolated rat hepatocytes. Proc Natl Acad Sci U S A. 79 (6), 1889-1892 (1982).
- Ronan, B., et al. A highly potent and selective Vps34 inhibitor alters vesicle trafficking and autophagy. Nat Chem Biol. 10 (12), 1013-1019 (2014).
- Saetre, F., Hagen, L. K., Engedal, N., Seglen, P. O. Novel steps in the autophagic-lysosomal pathway. Febs j. 282 (11), 2202-2214 (2015).
- Klionsky, D. J., et al. Guidelines for the use and interpretation of assays for monitoring autophagy (3rd edition). Autophagy. 12 (1), 1-222 (2016).
- Seglen, P. O., Overbye, A., Saetre, F. Sequestration assays for mammalian autophagy. Methods Enzymol. 452, 63-83 (2009).
- Hurley, J. H., Young, L. N. Mechanisms of Autophagy Initiation. Annu Rev Biochem. 86, 225-244 (2017).
- Thastrup, O., Cullen, P. J., Drobak, B. K., Hanley, M. R., Dawson, A. P. Thapsigargin, a tumor promoter, discharges intracellular Ca2+ stores by specific inhibition of the endoplasmic reticulum Ca2(+)-ATPase. Proc Natl Acad Sci U S A. 87 (7), 2466-2470 (1990).
- Kuma, A., et al. The role of autophagy during the early neonatal starvation period. Nature. 432 (7020), 1032-1036 (2004).
- Komatsu, M., et al. Impairment of starvation-induced and constitutive autophagy in Atg7-deficient mice. J Cell Biol. 169 (3), 425-434 (2005).
- Saitoh, T., et al. Atg9a controls dsDNA-driven dynamic translocation of STING and the innate immune response. Proc Natl Acad Sci U S A. 106 (49), 20842-20846 (2009).
- Gordon, P. B., Seglen, P. O. Autophagic sequestration of [14C]sucrose, introduced into rat hepatocytes by reversible electro-permeabilization. Exp Cell Res. 142 (1), 1-14 (1982).
- An, H., Harper, J. W. Systematic analysis of ribophagy in human cells reveals bystander flux during selective autophagy. Nat Cell Biol. 20 (2), 135-143 (2018).
- Fass, E., Shvets, E., Degani, I., Hirschberg, K., Elazar, Z. Microtubules support production of starvation-induced autophagosomes but not their targeting and fusion with lysosomes. J Biol Chem. 281 (47), 36303-36316 (2006).
- Velikkakath, A. K., Nishimura, T., Oita, E., Ishihara, N., Mizushima, N. Mammalian Atg2 proteins are essential for autophagosome formation and important for regulation of size and distribution of lipid droplets. Mol Biol Cell. 23 (5), 896-909 (2012).
- Øverbye, A., Sætre, F., Hagen, L. K., Johansen, H. T., Seglen, P. O. Autophagic activity measured in whole rat hepatocytes as the accumulation of a novel BHMT fragment (p10), generated in amphisomes by the asparaginyl proteinase, legumain. Autophagy. 7 (9), 1011-1027 (2011).
- Kominami, E., Hashida, S., Khairallah, E. A., Katunuma, N. Sequestration of cytoplasmic enzymes in an autophagic vacuole-lysosomal system induced by injection of leupeptin. J Biol Chem. 258 (10), 6093-6100 (1983).
- Rosado, C. J., Mijaljica, D., Hatzinisiriou, I., Prescott, M., Devenish, R. J. Rosella: a fluorescent pH-biosensor for reporting vacuolar turnover of cytosol and organelles in yeast. Autophagy. 4 (2), 205-213 (2008).
- Katayama, H., Kogure, T., Mizushima, N., Yoshimori, T., Miyawaki, A. A sensitive and quantitative technique for detecting autophagic events based on lysosomal delivery. Chem Biol. 18 (8), 1042-1052 (2011).
- Ogier-Denis, E., et al. A heterotrimeric Gi3-protein controls autophagic sequestration in the human colon cancer cell line HT-29. J Biol Chem. 270 (1), 13-16 (1995).
- Seglen, P. O., Gordon, P. B., Tolleshaug, H., Hoyvik, H. Use of [3H]raffinose as a specific probe of autophagic sequestration. Exp Cell Res. 162 (1), 273-277 (1986).
- Luhr, M., Sætre, F., Engedal, N. The Long-lived Protein Degradation Assay: an Efficient Method for Quantitative Determination of the Autophagic Flux of Endogenous Proteins in Adherent Cell Lines. Bio-protocol. 8 (9), e2836 (2018).
- Ronning, O. W., Pettersen, E. O., Seglen, P. O. Protein synthesis and protein degradation through the cell cycle of human NHIK 3025 cells in vitro. Exp Cell Res. 123 (1), 63-72 (1979).
- Seglen, P. O., Grinde, B., Solheim, A. E. Inhibition of the lysosomal pathway of protein degradation in isolated rat hepatocytes by ammonia, methylamine, chloroquine and leupeptin. Eur J Biochem. 95 (2), 215-225 (1979).
- Seglen, P. O., Solheim, A. E. Valine uptake and incorporation into protein in isolated rat hepatocytes. Nature of the precursor pool for protein synthesis. Eur J Biochem. 85 (1), 15-25 (1978).
- Bauvy, C., Meijer, A. J., Codogno, P. Assaying of autophagic protein degradation. Methods Enzymol. 452, 47-61 (2009).
- Engedal, N., Seglen, P. O. Autophagy of cytoplasmic bulk cargo does not require LC3. Autophagy. 12 (2), 1-3 (2016).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon