É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
O ensaio de sequestro de lactato desidrogenase — Um método simples e confiável para determinar a atividade de Autophagic de sequestro em massa em células de mamíferos
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Aqui, é descrito um protocolo simples e bem validado para medir a atividade de autophagic de sequestro em massa em células de mamíferos. O método baseia-se em quantificar a proporção de lactato desidrogenase (LDH) em frações de célula sedimentáveis em comparação com os níveis LDH totais do celulares.
Resumo
Autofagia em massa caracteriza-se por sequestro de grandes porções do citoplasma nas estruturas de duplo/multi-membrane denominado autophagosomes. Aqui um protocolo simples para monitorar este processo é descrito. Além disso, o típicos resultados e validação experimental do método sob condições indutoras autofagia em vários tipos de células de mamíferos cultivadas são fornecidos. Durante autofagia em massa, autophagosomes sequestrar citosol e desse modo também proteínas citosólica solúveis, juntamente com outras cargas autophagic. LDH é estável e altamente solúvel, abundante enzima citosólica que não seletivamente é sequestrada em autophagosomes. A quantidade de sequestro de LDH, portanto, reflete a quantidade de sequestro de autophagic em massa. Para eficiência e precisão determinar o sequestro de LDH em células, nós empregamos um protocolo baseado em electrodisruption de fracionamento que efetivamente separa sedimentáveis LDH citosólica, seguido de medida da atividade enzimática em sedimentáveis fracções contra amostras de células inteiras. Sequestro de autophagic é determinado subtraindo-se a proporção de sedimentáveis LDH em células não tratadas do que em células tratadas. A vantagem do ensaio do sequestro de LDH é que dá uma medida quantitativa da autophagic embargo da carga endógena, ao contrário de outros métodos que também envolvem expressão ectópica de sondas de sequestro ou semi-quantitativa de protease análises de proteção de marcadores de autofagia ou receptores.
Introdução
Autofagia (do grego para "Self comendo") é um processo evolutivo e conservado pela degradação vacuolar/lysosomal do material intracelular. Após a descoberta de genes relacionados com autofagia ("ATG"), que são importantes para a autofagia no fermento e seres humanos, e a realização que autofagia desempenha um papel significativo na saúde e na doença (reconhecido por 2016 Prêmio Nobel de medicina ou fisiologia de Yoshinori Ohsumi), autofagia rapidamente se tornou um dos processos mais intensamente estudados na célula biologia1,2.
Macroautophagy (doravante referida como "autofagia") é caracterizada pela expansão e dobramento de pequenos membrana intracelular ("phagophores") em estruturas seladas, double ou multi membrane ("autophagosomes") que efetivamente sequestram o enwrapped material do resto do citoplasma. Mediante a fusão de autophagosomes com lisossomos, membrana interna autophagosomal e a carga sequestrada é degradado e reciclados. Autophagosomes pode requisitar material citoplasmático em aleatória (não-seletivo autofagia) e modos seletiva (autofagia seletiva). Granel autofagia provavelmente representa uma mistura de autofagia não-seletivos e seletiva.
Nos anos 60 e 70 ("a morfológica era" da pesquisa de autofagia), sequestro de autophagic principalmente foi avaliado através de análises ultraestrutural. Na década de 1980 e início dos anos 90 ("a era bioquímico") por Seglen e colegas de trabalho — quem estudou autofagia em hepatócitos primários de rato — desenvolveu os primeiro métodos para medir quantitativamente o sequestro autophagic atividade3. Usando estes ensaios, Seglen definidos e caracterizados diferentes etapas da via lisossomal-autophagic4,5, descoberto e cunhou o amphisome6 (o produto da fusão endossomo-autophagosome) e foi o primeiro a Descreva o papel da fosforilação de proteínas em autofagia Regulamento7. No entanto, após a descoberta dos ATGs de 1990 ("a era molecular") e a primeira caracterização de uma proteína ATG8 mamíferos, proteína microtubule-associada 1A/1B-luz corrente 3 (LC3) em 20008, a utilização de proteínas ATG como marcadores para o autophagic processo rapidamente ganhou popularidade, e os mais velhos e mais trabalhosos métodos bioquímicos foram deixados para trás. Na verdade, durante os últimos 18 anos, borrão ocidental e análises de microscopia de fluorescência de LC3 tornaram-se de longe mais populares (e em muitos casos, o único) meio de estudar autofagia em células de mamíferos. A vantagem é a relativa facilidade pelo qual estes métodos podem ser realizados. A desvantagem é que um está estudando um componente de carrinho (LC3) ao invés de carga autophagic real. Esta é uma desvantagem bastante séria, porque a relação entre os Estados-Membros e/ou fluxo de LC3 através da via contra o sequestro e o fluxo de carga é altamente incerto. Na verdade, mostramos que o fluxo de carga em massa pode ser mantido em níveis elevados em condições onde não há nenhum fluxo LC3, apesar da presença de conjugados LC3 na9células. Além disso, temos demonstrado que autofagia em massa não é afetada pela depleção de LC3 eficiente e assim provavelmente é LC3-independente9. Este achado mais tarde foi confirmado por LC3 nocaute estudos10,11, que também indicam que mitophagy Parkin-dependente (a seletiva autofagia mitocondrial) é independente dos LC310,11 .
Em resumo, há uma clara necessidade ensaios baseados em carga monitorar a atividade do autophagic. Otimamente desses ensaios devem ser amplamente aplicável, bem definido e fácil de executar. Ao longo dos últimos anos, demos um interesse particular no ensaio de sequestro de LDH, que foi desenvolvido por Per Seglen na década de 198012e baseia-se na medição da transferência de LDH citosólico para sedimentáveis, célula vacúolo contendo autophagic frações. LDH é uma proteína citosólica estável, solúvel que prontamente co é sequestrada quando phagophores Wrap carga citoplasmática. Sequestro de LDH é, portanto, uma medida geral de sequestro autophagic. LDH é exclusivamente degradado pela via lisossomal-autophagic12. Assim, na presença de inibidores da degradação dos lisossomos, por exemplo, bafilomycin A1 (Baf)13, efeitos do tratamento experimental diretamente refletir alterações na atividade de sequestro autophagic. Na ausência de inibidores da degradação, o efeito líquido de alterações no sequestro de LDH e degradação pode ser medido.
O ensaio de sequestro de LDH é amplamente aplicável, desde que a LDH é altamente e ubiquitously expressa em todos os tipos de células, e os níveis LDH podem ser quantificados com precisão por um ensaio enzimático de14,15. No entanto, o original do protocolo12 — estabelecido em hepatócitos primários de rato — foi bastante demorado e exigiu uma alta quantidade de começar o material, bem como um capacitor feito por descarga elétrica. De uma forma faseada, nós transformaram gradualmente o ensaio em um método fácil e versátil. Em primeiro lugar, o protocolo original foi adaptado para uso em linhas de células de mamíferos16. Em segundo lugar, o método foi substancialmente downscaled3,9. Terceiro, várias etapas no protocolo foram eliminadas, incluindo uma almofada de densidade laborioso passo17. Isto permitiu simultaneamente uma regionalização ainda mais do método, do ponto de partida original do uso de uma placa de 10cm por exemplo16 usando um único poço de uma placa de 12 por amostra (ou seja, aproximadamente 15-fold menos começando material)17. Em quarto lugar, nós identificamos uma aparelho de eletroporação comercial que poderia substituir o capacitor de descarga elétrica sob medida17.
Aqui nosso protocolo mais atualizado do ensaio do sequestro de LDH, que inclui algumas simplificações adicionais do método em comparação com o anteriormente publicado17 é apresentado. Além disso, é mostrado um conjunto de resultados típicos obtidos em um número de diferentes tipos de células, e importante, várias linhas de validações experimentais do método usando o knockdown farmacológica, bem como genética e nocaute abordagens são fornecidas. Um regime global de fluxo do protocolo inteiro, veja a Figura 1.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. célula semeadura e tratamento
-
Células aderentes de cultura em frascos de cultura de tecidos de2 75 cm numa incubadora umidificado com 5% CO2 a 37 ° C, usando o meio de cultura preferido para o tipo de célula em questão. Permitir que as células a crescer até atingir uma camada de célula perto confluente.
Nota: Use RPMI 1640 suplementado com 10% soro bovino fetal (FBS) para LNCaP, HEK293, rato embrionários fibroblastos (MEFs), BJ, MCF-7 e as células de RPE-1.- Lave as células com 3ml 37 ° C fosfato salino (PBS), pH 7,4. Substitua a PBS 3 mL 0,25% (p/v) tripsina-etilenodiaminotetracético (EDTA) e incubar o frasco numa incubadora umidificado com 5% CO2 a 37 ° C, até que as células desanexar (2 – 5 min).
- Ressuspender as células isoladas com 7 mL de meio de cultura contendo 10% FBS. Misture uma alíquota de suspensão de células de 10 µ l com 10 µ l 0.4% Trypan azul em um tubo de microcentrifugadora, usando a ponta da pipeta 0,5 – 20 µ l. Use a ponta da pipeta mesmo para encher imediatamente um slide de câmara a contagem e contar as células em um contador automatizado de células.
-
Preparar um adequado (ver nota abaixo) diluição da suspensão de célula de etapa 1.1.2 usando cultura médio contendo 10% FBS, sementes e 1 mL da suspensão celular diluídos em cada poço de uma placa (área de superfície ~3.8 cm2) de cultura de tecidos de 12-poços de asséptico técnica. Permita o crescimento em uma incubadora umidificada com 5% CO2 a 37 ° C até a densidade da célula desejada tenha sido atingida, por exemplo, 60 – 90% confluência na colheita.
Nota: A diluição adequada da suspensão celular que dará a confluência da célula desejada na colheita pode variar do tipo de célula para o tipo de célula, bem como de acordo com a duração e o tipo de tratamentos experimentais. Assim, isto deve ser avaliado empiricamente em cada caso.- Para experiências que devem ser ambos trataram e colhidas 2 dias após a semeadura, semente 2,5 x 105 LNCaP, HEK293, ou MCF-7 células, 5 x 104 MEFs, 4 x 105 BJ ou 1,5 x 105 RPE-1 células em cada poço da placa 12-bem.
- Para as células que aderem frouxamente, revesti as placas com o tipo de revestimento recomendado para o tipo de célula em questão. Para células LNCaP (e HEK293) usar placas revestidas com poli-D-lisina (PDL).
- Para o efeito, adicionar 500 µ l PDL em 2,5 µ g/mL em estéril H2O para cada poço e incubar as placas em um ambiente estéril por 30 min à temperatura ambiente (20-25 ° C). Remover o PDL por sucção e lavar cada um bem brevemente com 1ml estéril H2O.
Nota: Geralmente, passo 1.2.1 é feito sem quaisquer tratamentos experimentais. No entanto, se executar RNAi, pode ser conveniente iniciar uma transfecção inversa com a semeadura9.
-
Realize tratamentos experimentais em duplicado ou triplicados poços por condição.
- Por exemplo, tratar as células com 50 nM do inibidor de mTOR Torin1, que geralmente é um eficiente indutor de sequestro de autophagic, ou submeter as células a inanição aguda do soro - e ácido aminado lavando as células com 1 mL de aminoácidos livres Earle é equilibrado Sal médio de solução (EBSS) e posteriormente, incubar as células em 1 mL de EBSS numa incubadora umidificado com 5% CO2 a 37 ° C.
- Deixe um conjunto de poços não for tratada, a fim de definir níveis de sedimentáveis LDH.
- Adicionar uma quantidade saturante do pós-sequestro inibidor bafilomycin A1 (Baf)3,13,16,18 na ausência ou presença dos tratamentos experimentais, 3 – 4 h antes de célula colheita. Incubar as células em uma incubadora umidificada com 5% CO2 a 37 ° C.
- Uso 100 nM Baf para LNCaP, HEK293, BJ, MCF-7 e células RPE-1 e 10 nM Baf para MEFs.
- Para tratamentos experimentais que têm uma duração de apenas 3-4 h (como aqueles passo exemplificado em 1.3.1 normalmente tem), adicione o Baf simultaneamente com os tratamentos. Para mais tratamentos experimentais, esperar até 3-4 h antes da colheita e adicionar 2 µ l de uma 500 x concentrado Baf estoque diretamente para o meio.
- Misture agitando a placa imediatamente após a adição do Baf. Neste momento também é aconselhável adicionar macroautophagic inibidores de sequestro como controles, por exemplo, 10 mM da pan-phosphoinositide 3-quinase (PI3K) inibidor 3-metil adenina (3MA)19ou 10 µM de PI3K selectivo classe III inibidor de SAR-40520.
2. celular colheita e preparação para Electrodisruption
- No final do período de tratamento, Aspire o meio com sucção e adicionar 200 solução de desprendimento de célula µ l (pre-aquecido a 37 ° C) para cada poço. Incube a 37 ° C até que as células desanexar (tipicamente em torno de 5 min).
Nota: Considerando que 0,25% (p/v) do Trypsin-EDTA pode ser usado em vez da solução de desprendimento de células, este último contém DNase, que ajuda a reduzir a viscosidade das células desanexadas. Enquanto o meio é completamente aspirado, não é necessário lavar as células antes da adição da solução de desprendimento do Trypsin-EDTA ou célula. - Adicionar 500 µ l da temperatura ambiente (20-25 ° C) PBS pH 7,4, contendo 2% (p/v) albumina de soro bovino (BSA) em cada poço e ressuspender com a pipeta até sem tufos de célula são visíveis. Transferi imediatamente a suspensão de eritrócitos para tubos de 1,5 mL microcentrifuga no gelo.
Nota: A menos que indicado o contrário, execute todas as etapas subsequentes no gelo. - Sedimento das células por centrifugação a 400 x g por 5 min a 4 ° C.
- Aspire cuidadosamente o sobrenadante (por sucção) para deixar as pelotas de célula seca como possível.
- Adicione 400 µ l 10% (p/v) de sacarose (em ultrapura H2O) para cada tubo.
3. membrana plasmática Electrodisruption e separação de frações Total e sedimentáveis --célula
- Ressuspender as células com uma pipeta para obter uma suspensão de célula única perto e transferi-lo para uma cubeta de eletroporação de 4 mm.
Nota: Pipetagem inconstante ~ 10 a 15 vezes, usando a ponta da pipeta 100 – 1.000 µ l, normalmente é suficiente. - Colocar a cubeta em um electroporator de onda de decaimento exponencial e descarga de um único pulso elétrico em 800 V, 25 µF e 400 Ω; essas configurações produzem um pulso de duração ~ 8 ms.
-
Use uma ponta de pipeta nova para transferir o disruptate de célula para um tubo de microcentrifugadora de 1,5 mL contendo 400 µ l solução gelada sacarose tampão fosfato (monofosfato de sódio de 100 mM, 2mm ditiotreitol (DTT), EDTA 2 mM e 1,75% de sacarose, pH 7,5) e misture brevemente por pipetagem.
- Opcional: Para verificar se a membrana de plasma eficiente electrodisruption17, misture 10 µ l do disruptate célula diluídos da etapa 3.3 com 10 µ l 0.4% Trypan azul em um tubo de microcentrifugadora de 1,5 mL. Transferir para uma câmara de contagem e verifique se a porcentagem de células positivas azul Trypan é > 99%.
- Deixe a amostra na câmara de contagem por 30 min à temperatura ambiente (20-25 ° C) e verifique se que a porcentagem de células positivas Trypan azul manteve-se > 99%.
- Opcional: Para verificar se o electrodisruption não tem sido muito dura, ou seja, não tem não interrompida organelas intracelulares, execute etapas 1.1 – 3.3 conforme descrito acima, mas use a maior matéria-prima (um poco de uma placa com uma camada de célula confluente de ~ 80% 6), e usar 150 µ l 10% de sacarose em solução de tampão fosfato sacarose µ l etapa 2.5 e 150 sem TDT na etapa 3.3.
- Usar uma pipeta para cuidadosamente camada 200 µ l da solução diluída de célula disruptate em cima de uma almofada de densidade de 1,2 mL de meio de gradiente de densidade de 8% (p/v) de tampão de fosfato (por exemplo, 8% Nycodenz, fosfato de sódio 50 mM, 2,2% de sacarose, 1 mM EDTA) em uma centrífuga de 2 mL tubo. Centrifugar a 20.000 x g durante 45 min a 4 ° C, numa microcentrifuga com função de modo suave (para suave aceleração e desaceleração) e com cuidado, coloque os tubos no gelo.
- Remova cuidadosamente a 60 µ l da fração superior ~ 200 µ l, certificando-se não pegar qualquer solução médio gradiente de densidade e transfira para um tubo de microcentrifugadora fresco.
Nota: Isto deve conter citosol de pureza excepcional, denominada "célula sap"21. - Teste a pureza da fração obtida na etapa acima, através da realização de análises de borrão ocidental de proteínas organela-contido, utilizando as técnicas convencionais e de géis gradiente de 4 – 20%16.
- Executar, por exemplo, immunoblotting para catepsina B21, citocromo c e isomerase de dissulfureto de proteína, para verificar que o choque elétrico em passo 3.2 não desestruturou lisossomos, mitocôndrias ou retículo endoplasmático, respectivamente e immunoblot para LDH para verificar a presença de uma proteína citosólica na seiva do celular.
- Em paralelo, execute immunoblotting em extratos de proteína feitos a partir de solução de disruptate a célula total16 para confirmar que os anticorpos usados podem detectar as proteínas contidas por organela que estão sendo avaliadas.
- Opcional: Para verificar se a membrana de plasma eficiente electrodisruption17, misture 10 µ l do disruptate célula diluídos da etapa 3.3 com 10 µ l 0.4% Trypan azul em um tubo de microcentrifugadora de 1,5 mL. Transferir para uma câmara de contagem e verifique se a porcentagem de células positivas azul Trypan é > 99%.
- Repita as etapas de 3.1-3.3 para cada amostra.
- Retire 550 µ l da solução de disruptate cada célula diluído (obtida na etapa 3.3) para tubos de microcentrifuga de 2 mL contendo 900 µ l gelada ressuspensão buffer (monofosfato de sódio 50 mM, 1 mM DTT, 1 mM EDTA e 5,9% de sacarose, pH 7,5) suplementado com 0,5% de BSA e 0,01% Tween-20 e misture brevemente por pipetagem.
- Centrifugar a 18.000 x g por 45 min a 4 ° C para produzir pelotas contendo "LDH sedimentada". Aspire cuidadosamente o sobrenadante (por sucção) para deixar as bolinhas tão seco quanto possível. Coloca as amostras em um freezer-80 ° C.
- Transferência de 150 µ l de cada solução de disruptate de célula diluído (obtida na etapa 3.3) para tubos novos e colocar as amostras em um freezer-80 ° C. Use estas amostras para determinar os níveis de "total de LDH" nas células.
Nota: Neste momento o experimento pode ser pausado para contanto que desejado.
4. LDH extração e avaliação da actividade enzimática de LDH
- Descongele o "LDH sedimentada" (da etapa 3.6) e "total de LDH" amostras (da etapa 3.7) no gelo.
- Adicione 300 µ l de tampão de ressuspensão gelada contendo 1,5% Triton X-405 para "LDH total" amostras (produzindo uma concentração final de Triton X-405 de 1%). Gire as amostras em um rolo em uma sala fria (4 – 8 ° C) por 30 min.
- Adicione 750 µ l de tampão de ressuspensão gelada com 1% Triton X-405 para "LDH sedimentada" amostras e ressuspender as pelotas com uma pipeta até que seja alcançada uma solução homogénea.
- Centrifugar as amostras da etapa 4.2 e 4.3 a 18.000 x g por 5 min a 4 ° C para restos celulares de sedimentos não dissolvido.
- Mix 4 peças do imidazol frio 65mm (piruvato de mM pH 7.5)/0.75 com uma parte do imidazole frio 65 mM (pH 7,5) / 1,8 mM NADH para obter uma solução de trabalho que é estável pelo menos três semanas a 4 ° C.
- Misture 3 – 30 µ l dos sobrenadantes da etapa 4.4 com 200 µ l da solução de trabalho passo 4.5.
- Determinar a quantidade de LDH, medindo a atividade enzimática de LDH como o declínio da nicotinamida adenina dinucleótido (forma reduzida) absorbância (NADH) 340 nm a 37 ° C, em comparação com um padrão com uma concentração conhecida de LDH. Executar medidas de absorvância até a reação se aproximou de conclusão, ou seja, até a absorbância em 340 nm já não muda com o tempo.
Nota: Este é o clássico método bioquímico para medir a atividade da LDH. Embora o atual protocolo realiza a reação a 37 ° C, ele também pode ser realizado à temperatura ambiente (20-25 ° C), que é aconselhável se fazer espectrofotometria manual. O protocolo atual usa um instrumento multianalyzer robótico, que, em uma forma automatizada, mistura de amostras com solução de trabalho em uma placa de 96 poços e mede a absorbância em 340 nm a 37 ° C cada 20 s por 3 min. Depois disso, o software de instrumento calcula a concentração de LDH, expressado como unidades (U) / L, comparando-se a inclinação das medições absorvância ao longo do tempo, em comparação com uma curva padrão obtida através de calibração com um padrão de LDH conhecido concentração. O intervalo linear de detecção por esta abordagem é 30 – 1.500 U/L. Como alternativa, existe uma grande variedade de kits comercialmente disponíveis para medir a LDH. Alguns deles são baseados na reação enzimática para a geração de produtos colorimétricos ou fluorescentes, permitindo a deteção por outros meios de espectrofotometria UV e com outras escalas lineares de deteção de acoplamento.
5. cálculo do sequestro de LDH
- Calcule a percentagem de LDH sedimentada a LDH total para cada amostra, tomando as diluições e amostragem em conta:
LDH sedimentada (%) =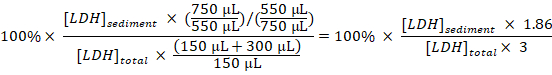
Nota: Durante as etapas 3.1 – 3.3 aproximadamente 50 µ l é perdida devido à transferência para dentro e fora da cubeta de eletroporação. Assim, calcule de ter um total de 750 µ l (em vez de 800 µ l) de disruptate de célula diluídos na etapa 3.3. - Subtrair a porcentagem de LDH sedimentada obtido nas amostras de células não tratadas (etapa 1.3.2) da porcentagem de LDH sedimentada, obtida em amostras de células tratadas experimentalmente e dividir pelo tempo de tratamento com Baf para obter a percentagem de LDH sequestrado por hora durante o período de amostragem:
Isolado de LDH (% / h) =
Access restricted. Please log in or start a trial to view this content.
Resultados
Usando o protocolo descrito aqui, a atividade de autophagic de sequestro em massa em um número de linhas de células de mamíferos diferentes, incluindo LAPC4, DU145, Huh7, PNT2A, HeLa, capnografia, H3122, Hec1A, MCF-7 T47D, U2OS, PC3, G361, mouse embrionários fibroblastos (MEFs), RPE-1, Células HEK293, BJ e LNCaP foi medido. Sequestro foi avaliado em condições basais (em médio completo, rico em nutrientes), ou em células agudamente faminta de soro e aminoácidos (uma bona fide...
Access restricted. Please log in or start a trial to view this content.
Discussão
Em resumo, o protocolo descrito aqui representa um método confiável e amplamente aplicável para monitorar a atividade de autophagic de sequestro em massa em células de mamíferos. Em comparação com o original método de12,16, ter removido um número de etapas desnecessárias, simplificamos várias das etapas restantes e introduziu uma substancial redução de escala. Como resultado, o protocolo é muito melhorado em relação ao custo e tempo-eficiência, e...
Access restricted. Please log in or start a trial to view this content.
Divulgações
Os autores não têm nenhum conflito de interesses.
Agradecimentos
Este trabalho foi apoiado financeiramente pelo Conselho de pesquisa da Noruega, da Universidade de Oslo, o Anders Jahre Foundation, Fundação Nansen e o legado na memória do Henrik Homan. Agradecemos o Dr. Noboru Mizushima para o ATG5 + / + MEFs e ATG5-/-MEFs, Dr. Masaaki Komatsu para o ATG7 + / + MEFs e ATG7-/-MEFs e Dr. Shizuo Akira para o ATG9A + / + MEFs e ATG9A-/-MEFs. Agradecemos Frank Sætre para assistência técnica e Dr. Per O. Seglen discussões construtivas metodológicas.
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| 1.5 mL and 2 mL microcentrifuge tubes | Eppendorf | 211-2130 and 211-2120 | |
| 12-well plates | Falcon | 353043 | |
| Accumax cell detachment solution | Innovative Cell Technologies | A7089 | Keep aliquots at -20 °C for years, and in fridge for a few months |
| Bafilomycin A1 | Enzo | BML-CM110-0100 | Dissolve in DMSO |
| BJ cells | ATCC | CRL-2522 | use at passage <30 |
| Bovine serum albumin (BSA) | VWR | 422361V | |
| Burker counting chamber | Fisher Scientific | 139-658585 | |
| Countess Cell Counting Chamber Slides | ThermoFisher Scientfic | C10228 | |
| Countess II Automated Cell Counter | ThermoFisher Scientfic | AMQAX1000 | |
| Cover glass for the Burker counting chamber | Fisher Scientific | 139-658586 | |
| Criterion Tris-HCl Gel, 4–20%, 26-well, 15 µL, 13.3 cm x 8.7 cm (W x L) | Bio-Rad | 3450034 | |
| DTT | Sigma-Aldrich | D0632 | |
| Earle's balanced salt solution (EBSS) | Gibco | 24010-043 | conatains 0.1% glucose |
| EDTA | Sigma-Aldrich | E7889 | |
| Electroporation cuvette (4 mm) | Bio-Rad | 1652088 | |
| Exponential decay wave electroporator | BTX Harvard Apparatus | EMC 630 | |
| Fetal bovine serum (FBS) | Sigma | F7524 | 10% final concentration in RPMI 1640 medium |
| HEK293 cells | ATCC | CRL-1573 | |
| Imidazole | Sigma-Aldrich | 56750 | Autoclave a 65 mM solution and keep in fridge for months |
| Incubator; Autoflow IR Direct Heat CO2 incubator | NuAire | NU-5510E | |
| Lipofectamine RNAiMAX Transfection Reagent | ThermoFisher | 13778150 | |
| LNCaP cells | ATCC | CRL-1740 | use at passage <30 |
| 3-Methyl Adenine (3MA) | Sigma-Aldrich | M9281 | Stock 100 mM in RPMI in -20 °C. Heat stock to 65 °C for 10 min, and use at 10 mM final concentration |
| Refridgerated Microcentrifuge | Beckman Coulter Life Sciences | 368831 | |
| Refridgerated Microcentrifuge with soft-mode function | Eppendorf | Eppendorf 5417R | |
| MRT67307 hydrochloride (ULKi) | Sigma-Aldrich | SML0702 | Inhibits ULK kinase activity. Dissolve in DMSO. |
| MaxMat Multianalyzer instrument | Erba Diagnostics | PL-II | |
| MCF7 cells | ATCC | HTB-22 | |
| NADH | Merck-Millipore | 1.24644.001 | |
| Nycodenz | Axis-Shield | 1002424 | |
| Opti-MEM Reduced Serum Medium | ThermoFisher | 31985062 | |
| Phosphate-buffered saline (PBS) | Gibco | 20012-019 | |
| Pipette tips 3 (0.5-20 µL) | VWR | 732-2223 | Thermo Fischer ART Barrier tips |
| Pipette tips (1-200 µL) | VWR | 732-2207 | Thermo Fischer ART Barrier tips |
| Pipette tips (100-1,000 µL) | VWR | 732-2355 | Thermo Fischer ART Barrier tips |
| Pipettes | ThermoFisher | 4701070 | Finnpipette F2 GLP Kit |
| Poly-D-lysine | Sigma-Aldrich | P6407-10X5MG | Make a 1 mg/mL stock solution in sterile H2O. This solution is stable at -20 °C for at least 1 year. |
| Pyruvate | Merck-Millipore | 1066190050 | |
| RPE-1 cells (hTERT RPE-1) | ATCC | CRL-4000 | |
| RPMI 1640 | Gibco | 21875-037 | |
| SAR-405 | ApexBio | A8883 | Inhibits phosphoinositide 3-kinase class III (PIK3C3). Dissolve in DMSO. |
| Silencer Select Negative Control #1 (siCtrl) | ThermoFisher/Ambion | 4390843 | |
| Silencer Select ATG9-targeting siRNA (siATG9A) | ThermoFisher/Ambion | s35504 | |
| Silencer Select FIP200-targeting siRNA (siFIP200) | ThermoFisher/Ambion | s18995 | |
| Silencer Select ULK1-targeting siRNA (siULK1) | ThermoFisher/Ambion | s15964 | |
| Silencer Select ULK2-targeting siRNA (siULK2) | ThermoFisher/Ambion | s18705 | |
| Silencer Select GABARAP-targeting siRNA (siGABARAP) | ThermoFisher/Ambion | s22362 | |
| Silencer Select GABARAPL1-targeting siRNA (siGABARAPL1) | ThermoFisher/Ambion | s24333 | |
| Silencer Select GABARAPL2-targeting siRNA (siGABARAPL2) | ThermoFisher/Ambion | s22387 | |
| Sodium phosphate monobasic dihydrate (NaH2PO4 • 2H2O) | Merck-Millipore | 1.06580.1000 | |
| Sodium phosphate dibasic dihydrate (Na2HPO4 • 2H2O ) | Prolabo | 28014.291 | |
| Sucrose | VWR | 443816T | 10% final concentration in water; filter through 0.45 µm filter and keep in fridge for months |
| Thapsigargin | Sigma-Aldrich | T9033 | Inhibits the SERCA ER Ca2+ pump. Dissolve in DMSO. |
| Triton X-405 | Sigma-Aldrich | X405 | 1% final |
| Trypan Blue stain 0.4% | Molecular Probes | T10282 | |
| Trypsin-EDTA (0.25% w/v Trypsin) | Gibco | 25200-056 | |
| Tween-20 | Sigma-Aldrich | P2287 | 0.01% final |
Referências
- Rubinsztein, D. C., Frake, R. A. Yoshinori Ohsumi's Nobel Prize for mechanisms of autophagy: from basic yeast biology to therapeutic potential. J R Coll Physicians Edinb. 46 (4), 228-233 (2016).
- Mizushima, N. The exponential growth of autophagy-related research: from the humble yeast to the Nobel Prize. FEBS Lett. 591 (5), 681-689 (2017).
- Seglen, P. O., et al. Macroautophagic cargo sequestration assays. Methods. 75, 25-36 (2015).
- Hoyvik, H., Gordon, P. B., Seglen, P. O. Use of a hydrolysable probe, [14C]lactose, to distinguish between pre-lysosomal and lysosomal steps in the autophagic pathway. Exp Cell Res. 166 (1), 1-14 (1986).
- Plomp, P. J., Gordon, P. B., Meijer, A. J., Hoyvik, H., Seglen, P. O. Energy dependence of different steps in the autophagic-lysosomal pathway. J Biol Chem. 264 (12), 6699-6704 (1989).
- Gordon, P. B., Seglen, P. O. Prelysosomal convergence of autophagic and endocytic pathways. Biochem Biophys Res Commun. 151 (1), 40-47 (1988).
- Holen, I., Gordon, P. B., Seglen, P. O. Protein kinase-dependent effects of okadaic acid on hepatocytic autophagy and cytoskeletal integrity. Biochem J. 284, Pt 3 633-636 (1992).
- Kabeya, Y., et al. LC3, a mammalian homologue of yeast Apg8p, is localized in autophagosome membranes after processing. Embo j. 19 (21), 5720-5728 (2000).
- Szalai, P., et al. Autophagic bulk sequestration of cytosolic cargo is independent of LC3, but requires GABARAPs. Exp Cell Res. 333 (1), 21-38 (2015).
- Nguyen, T. N., et al. Atg8 family LC3/GABARAP proteins are crucial for autophagosome-lysosome fusion but not autophagosome formation during PINK1/Parkin mitophagy and starvation. J Cell Biol. , (2016).
- Pontano Vaites, L., Paulo, J. A., Huttlin, E. L., Harper, J. W. Systematic analysis of human cells lacking ATG8 proteins uncovers roles for GABARAPs and the CCZ1/MON1 regulator C18orf8/RMC1 in macro and selective autophagic flux. Mol Cell Biol. , (2017).
- Kopitz, J., Kisen, G. O., Gordon, P. B., Bohley, P., Seglen, P. O. Nonselective autophagy of cytosolic enzymes by isolated rat hepatocytes. J Cell Biol. 111 (3), 941-953 (1990).
- Bowman, E. J., Siebers, A., Altendorf, K. Bafilomycins: a class of inhibitors of membrane ATPases from microorganisms, animal cells, and plant cells. Proc Natl Acad Sci U S A. 85 (21), 7972-7976 (1988).
- Hadjivassiliou, A. G., Rieder, S. V. The enzymatic assay of pyruvic and lactic acids. A definitive procedure. Clin Chim Acta. 19 (3), 357-361 (1968).
- Bergmeyer, H. U., Bernt, E. Methods of enzymatic analysis (2nd English ed). Bergmeyer, H. U. 2, Verlag Chemie. 574-579 (1974).
- Engedal, N., et al. Modulation of intracellular calcium homeostasis blocks autophagosome formation. Autophagy. 9 (10), 1475-1490 (2013).
- Luhr, M., et al. A Simple Cargo Sequestration Assay for Quantitative Measurement of Nonselective Autophagy in Cultured Cells. Methods Enzymol. 587, 351-364 (2017).
- Mousavi, S. A., et al. Effects of inhibitors of the vacuolar proton pump on hepatic heterophagy and autophagy. Biochim Biophys Acta. 1510 (1-2), 243-257 (2001).
- Seglen, P. O., Gordon, P. B. 3-Methyladenine: specific inhibitor of autophagic/lysosomal protein degradation in isolated rat hepatocytes. Proc Natl Acad Sci U S A. 79 (6), 1889-1892 (1982).
- Ronan, B., et al. A highly potent and selective Vps34 inhibitor alters vesicle trafficking and autophagy. Nat Chem Biol. 10 (12), 1013-1019 (2014).
- Saetre, F., Hagen, L. K., Engedal, N., Seglen, P. O. Novel steps in the autophagic-lysosomal pathway. Febs j. 282 (11), 2202-2214 (2015).
- Klionsky, D. J., et al. Guidelines for the use and interpretation of assays for monitoring autophagy (3rd edition). Autophagy. 12 (1), 1-222 (2016).
- Seglen, P. O., Overbye, A., Saetre, F. Sequestration assays for mammalian autophagy. Methods Enzymol. 452, 63-83 (2009).
- Hurley, J. H., Young, L. N. Mechanisms of Autophagy Initiation. Annu Rev Biochem. 86, 225-244 (2017).
- Thastrup, O., Cullen, P. J., Drobak, B. K., Hanley, M. R., Dawson, A. P. Thapsigargin, a tumor promoter, discharges intracellular Ca2+ stores by specific inhibition of the endoplasmic reticulum Ca2(+)-ATPase. Proc Natl Acad Sci U S A. 87 (7), 2466-2470 (1990).
- Kuma, A., et al. The role of autophagy during the early neonatal starvation period. Nature. 432 (7020), 1032-1036 (2004).
- Komatsu, M., et al. Impairment of starvation-induced and constitutive autophagy in Atg7-deficient mice. J Cell Biol. 169 (3), 425-434 (2005).
- Saitoh, T., et al. Atg9a controls dsDNA-driven dynamic translocation of STING and the innate immune response. Proc Natl Acad Sci U S A. 106 (49), 20842-20846 (2009).
- Gordon, P. B., Seglen, P. O. Autophagic sequestration of [14C]sucrose, introduced into rat hepatocytes by reversible electro-permeabilization. Exp Cell Res. 142 (1), 1-14 (1982).
- An, H., Harper, J. W. Systematic analysis of ribophagy in human cells reveals bystander flux during selective autophagy. Nat Cell Biol. 20 (2), 135-143 (2018).
- Fass, E., Shvets, E., Degani, I., Hirschberg, K., Elazar, Z. Microtubules support production of starvation-induced autophagosomes but not their targeting and fusion with lysosomes. J Biol Chem. 281 (47), 36303-36316 (2006).
- Velikkakath, A. K., Nishimura, T., Oita, E., Ishihara, N., Mizushima, N. Mammalian Atg2 proteins are essential for autophagosome formation and important for regulation of size and distribution of lipid droplets. Mol Biol Cell. 23 (5), 896-909 (2012).
- Øverbye, A., Sætre, F., Hagen, L. K., Johansen, H. T., Seglen, P. O. Autophagic activity measured in whole rat hepatocytes as the accumulation of a novel BHMT fragment (p10), generated in amphisomes by the asparaginyl proteinase, legumain. Autophagy. 7 (9), 1011-1027 (2011).
- Kominami, E., Hashida, S., Khairallah, E. A., Katunuma, N. Sequestration of cytoplasmic enzymes in an autophagic vacuole-lysosomal system induced by injection of leupeptin. J Biol Chem. 258 (10), 6093-6100 (1983).
- Rosado, C. J., Mijaljica, D., Hatzinisiriou, I., Prescott, M., Devenish, R. J. Rosella: a fluorescent pH-biosensor for reporting vacuolar turnover of cytosol and organelles in yeast. Autophagy. 4 (2), 205-213 (2008).
- Katayama, H., Kogure, T., Mizushima, N., Yoshimori, T., Miyawaki, A. A sensitive and quantitative technique for detecting autophagic events based on lysosomal delivery. Chem Biol. 18 (8), 1042-1052 (2011).
- Ogier-Denis, E., et al. A heterotrimeric Gi3-protein controls autophagic sequestration in the human colon cancer cell line HT-29. J Biol Chem. 270 (1), 13-16 (1995).
- Seglen, P. O., Gordon, P. B., Tolleshaug, H., Hoyvik, H. Use of [3H]raffinose as a specific probe of autophagic sequestration. Exp Cell Res. 162 (1), 273-277 (1986).
- Luhr, M., Sætre, F., Engedal, N. The Long-lived Protein Degradation Assay: an Efficient Method for Quantitative Determination of the Autophagic Flux of Endogenous Proteins in Adherent Cell Lines. Bio-protocol. 8 (9), e2836(2018).
- Ronning, O. W., Pettersen, E. O., Seglen, P. O. Protein synthesis and protein degradation through the cell cycle of human NHIK 3025 cells in vitro. Exp Cell Res. 123 (1), 63-72 (1979).
- Seglen, P. O., Grinde, B., Solheim, A. E. Inhibition of the lysosomal pathway of protein degradation in isolated rat hepatocytes by ammonia, methylamine, chloroquine and leupeptin. Eur J Biochem. 95 (2), 215-225 (1979).
- Seglen, P. O., Solheim, A. E. Valine uptake and incorporation into protein in isolated rat hepatocytes. Nature of the precursor pool for protein synthesis. Eur J Biochem. 85 (1), 15-25 (1978).
- Bauvy, C., Meijer, A. J., Codogno, P. Assaying of autophagic protein degradation. Methods Enzymol. 452, 47-61 (2009).
- Engedal, N., Seglen, P. O. Autophagy of cytoplasmic bulk cargo does not require LC3. Autophagy. 12 (2), 1-3 (2016).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados