Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Fluorescencia cuantitativa en hibridación situ (FISH) e inmunofluorescencia (IF) de productos genéticos específicos en células infectadas por KSHV
En este artículo
Resumen
Describimos un protocolo que utiliza la hibridación in situ por fluorescencia (FISH) para visualizar múltiples ARN herpesvirales dentro de células humanas infectadas lyéticamente, ya sea en suspensión o adherente. Este protocolo incluye la cuantificación de la fluorescencia produciendo una relación nucleocitotoplasmática y se puede extender para la visualización simultánea de proteínas huésped y virales con inmunofluorescencia (IF).
Resumen
La visión mecanicista proviene de un estudio cuidadoso y la cuantificación de ARN y proteínas específicas. Las ubicaciones relativas de estas biomoléculas en toda la célula en momentos específicos se pueden capturar con hibridación in situ por fluorescencia (FISH) e inmunofluorescencia (IF). Durante la infección por herpesvirus lítico, el virus secuestra la célula huésped para expresar preferentemente genes virales, causando cambios en la morfología celular y el comportamiento de las biomoléculas. Las actividades líticas se centran en fábricas nucleares, denominadas compartimentos de replicación viral, que sólo son discernibles con FISH y IF. Aquí describimos un protocolo adaptable de las técnicas de ARN FISH y IF para las células infectadas por el herpesvirus asociado al sarcoma de Kaposi (KSHV), tanto adherentes como en suspensión. El método incluye pasos para el desarrollo de oligonucleótidos específicos antisentido, DOBLE ARN FISH, RNA FISH con IF y cálculos cuantitativos de intensidades de fluorescencia. Este protocolo se ha aplicado con éxito a múltiples tipos de células, células no infectadas, células latentes, células líticas, cursos de tiempo y células tratadas con inhibidores para analizar las actividades espaciotemporales de ARN y proteínas específicos tanto del huésped humano como de Kshv.
Introducción
En su fase lítica (activa), los herpesvirus secuestran la célula huésped, causando cambios en la morfología celular y la localización de moléculas biológicas, para producir viriones. La base de las operaciones es el núcleo, donde el genoma viral del ADN de doble cadena se replica y empaqueta en una cáscara de proteína, llamada cápside1. Para empezar, el virus expresa sus propias proteínas, secuestrando maquinaria huésped y previniendo la expresión de genes de host no esenciales, un proceso llamado efecto de cierre del huésped. La mayor parte de esta actividad se localiza en regiones nucleares específicas libres de 4o,6-diamidino-2-fenilindolo (DAPI)llamadas compartimentos de replicación viral, compuestos por proteínas huésped y virales, ARN y ADN viral 2. La célula se revisa para proporcionar espacio y recursos para los compartimentos de replicación y, por lo tanto, el montaje de cápsides virales. Una vez que la cápside sale del núcleo, no está claro cómo la cápside está envuelta en el citoplasma para producir una partícula viral ligada a la membrana, también conocida como virión. La comprensión de la localización y los cambios espaciales de las biomoléculas víricas y del huésped durante la fase lítica proporciona una visión mecanicista más profunda de la disposición del compartimiento de replicación, el efecto de cierre del host, la vía de virión-salida y otros procesos relacionados con la infección y replicación del herpesviral.
Actualmente el mejor método para detectar y estudiar estos cambios es la visualización de proteínas y ARN en células infectadas con inmunofluorescencia (IF) e hibridación fluorescente in situ (FISH), respectivamente. El uso de un curso de tiempo con estas técnicas revela la localización de biomoléculas en puntos clave de la fase lítica o, simplemente, datos espaciotemporales. FISH y IF complementan otras técnicas bioquímicas, como la inhibición de un proceso celular (por ejemplo, la inhibición de la replicación del ADN viral), RT-qPCR (reacción en cadena de la polimerasa en tiempo real), la secuenciación de ARN, las manchas del norte, la espectrometría de masas, la hinchazón occidental y análisis de la producción de ADN viral, que puede proporcionar una imagen más global de las actividades celulares.
Desarrollamos estrategias de ARN FISH para examinar los productos de ARN a partir de genes específicos y un análisis computacional que calcula cuantitativamente la relación nucleocitotoplasmática de un producto genético específico. La preparación de la muestra, modificada a partirde publicaciones anteriores de Steitz y sus colegas 3,4, es relativamente fácil y se puede utilizar tanto para células adherentes como suspendidas. El protocolo también es adaptable para el uso simultáneo de múltiples estrategias de ARN FISH (doble ARN FISH) o RNA FISH con estrategias IF. El desarrollo de una estrategia específica de FISH es un reto, pero se describen sugerencias para mejorar el éxito. El análisis de datos descrito aquí es cuantitativo si se utilizan cuentas fluorescentes y marcadores fuertes de los límites del compartimiento y ofrece información adicional sobre las micrografías, información que elimina el sesgo de observación. El protocolo detallado está diseñado tanto para células latentes como líticas infectadas por el herpesvirus asociado al sarcoma de Kaposi (KSHV) y se puede utilizar con células no infectadas o células infectadas por otros herpesvirus5. Los métodos de cuantificación son aplicables a estudios sobre desplazamientos nucleocitotoplásmicos o relocalización entre compartimentos subcelulares en la mayoría de las células.
Protocolo
1. Diseño de oligonucnucleótidos antisentido de fluorescencia in situ (FISH) para detectar una transcripción específica de herpesviral
- Seleccione segmentos de 25 a 40 nt de la secuencia de ARN de interés y conviértalos en antisentido. Una estrategia FISH exitosa puede contener de uno hasta diez o más diferentes oligonucleótidos antisentido. Al seleccionar secuencias, tenga en cuenta lo siguiente:
- Si el ARN de interés contiene una región de repetición única, aproveche esta característica y diseñe un oligonucleótido antisentido para orientar la secuencia de repetición.
NOTA: Tycowski y sus colegas5 proporcionan un ejemplo de esta estrategia con ARN nuclear poliadenilado (PAN) de rhesus rhadinovirus (RRV). - Si el ARN de interés contiene un sitio conocido de unión a proteínas o estructura de bucle de tallo, diseñe oligonucleótidos que eviten estas regiones.
- Dependiendo de los objetivos de los experimentos, considere la secuencia intrónica y si diseñar o no un oligonucleótido antisentido para ella.
- Si el ARN de interés contiene una región de repetición única, aproveche esta característica y diseñe un oligonucleótido antisentido para orientar la secuencia de repetición.
- Realice análisis computacionales simples en las secuencias antisentido seleccionadas para garantizar la especificidad de unión y reducir la agregación del oligonucleótido antisentido.
- Las secuencias deben ser aproximadamente un 50% ricas en GC (alto contenido de guanina y citosina) y tener una temperatura de fusión en el rango de 60 a 70 oC.
- Utilice una herramienta de analizador de secuencias para seleccionar secuencias que no se autodimerizen ni formen horquillas con temperaturas de fusión superiores a 37 oC, la temperatura de hibridación.
- Realizar una búsqueda NCBI BLASTn (National Center for Biotechnology Information Basic Local Alignment Search Tool for nucleotide alignments) de las secuencias seleccionadas tanto contra el host como con los transcriptomes virales utilizando el ajuste "algo similar". Esta búsqueda identificará oligonucleótidos antisentido únicos que probablemente no se unirán a otras transcripciones de host o virales.
NOTA: Si los transcriptomas no están disponibles, realice la búsqueda BLAST con las secuencias genómicas. Es ideal si las búsquedas se realizan en las secuencias del virus aislado de las células infectadas utilizadas en el experimento porque las cepas silvestres tienden a diversificarse y contienen una combinación de secuencias de diferentes cepas de laboratorio.
- Ordene los oligonucleótidos de ADN purificados correspondientes a la secuencia anti-sentido que se ha verificado computacionalmente para que sea único y que sea probable que se una al ARN objetivo. No es necesario introducir modificaciones especiales en los oligonucleótidos.
- Pruebe los oligonucleótidos antisentido diseñados para la especificidad de unión por FISH y Northern blot.
- Utilizando una línea celular no infectada de la misma especie huésped (por ejemplo, 293T) e idealmente del mismo tipo de célula, llevar a cabo una transfección con un plásmido que exprese el ARN de interés de un promotor robusto (CMV, citomegalovirus) y uno con el vector vacío (por ejemplo, pcDNA3). Utilice un control positivo para la transfección, como la co-transfección con un plásmido GFP (proteína fluorescente verde) (por ejemplo, pmaxGFP) o el vector que contiene GFP.
NOTA: Es importante evitar las líneas celulares que han sido inmortalizadas utilizando el virus del herpesvirus Epstein-Barr (EBV) ya que hay similitudes de secuencia entre los herpesvirus. - Realice FISH como se describe en la sección 3 en ambos conjuntos de células con los oligonucleótidos antisentido como se describe en este protocolo. Deducir los candidatos exitosos comparando experimentos FISH con individuos, pares o conjuntos de los oligonucleótidos anti-sentido. Utilice un control positivo para el protocolo FISH, como el ARN SnRNA U2 (ARN nuclear pequeño), presente en 500.000 copias por núcleo celular humano6 (Tabla 1).
- La señal fluorescente debe ser específica y fuerte en la célula que contiene el ARN de interés. Diseñar oligonucleótidos antisentido adicionales para fortalecer la señal y eliminar los oligonucleótidos antisentido que se unen no específicamente de la consideración. La intensidad de la señal debe estar por encima del fondo y la autofluorescencia automática.
- Probar la especificidad de unión por northern blot.
- Utilizando una línea celular no infectada de la misma especie huésped (por ejemplo, 293T) e idealmente del mismo tipo de célula, llevar a cabo una transfección con un plásmido que exprese el ARN de interés de un promotor robusto (CMV, citomegalovirus) y uno con el vector vacío (por ejemplo, pcDNA3). Utilice un control positivo para la transfección, como la co-transfección con un plásmido GFP (proteína fluorescente verde) (por ejemplo, pmaxGFP) o el vector que contiene GFP.
2. Oligonucleótido y preparación celular
- Siguiendo las instrucciones del fabricante, utilice la transferencia terminal para etiquetar los oligonucleótidos antisentido con dioxigenina(DIG)-dUTP o, si se une fuertemente, directamente con un nucleótido fluorescente como Alexa Fluor 594-5-dUTP. Después del etiquetado, no es necesaria una purificación adicional. Almacene los oligonucleótidos etiquetados a -20 oC hasta varios años y en papel de aluminio si están etiquetados directamente para evitar el fotoblanqueo.
ADVERTENCIA: La solución de etiquetado contiene un material tóxico, cacodilato de potasio. Manipule las reacciones de etiquetado con guantes.
NOTA: Para conservar los recursos, se pueden etiquetar varios oligonucleótidos antisentido diferentes en una sola reacción. Este protocolo utiliza el etiquetado de 3'-end. El etiquetado interno es difícil porque el grupo químico (por ejemplo, DIG o el fluoróforo) queda atrapado o no puede entrar en el sitio activo de una polimerasa de ADN. Un experimento con dos ARN diferentes se puede realizar utilizando oligonucleótidos antisentido etiquetados directamente e inmunofluorescencia anti-DIG con un fluoróforo diferente (por ejemplo, FITC (fluoresceína) o Alexa Fluor 488 con Alexa Fluor 594). - Adherir las celdas a las diapositivas de ocho cámaras.
NOTA: Las diapositivas de ocho cámaras permiten varios experimentos simultáneos al tiempo que minimizan recursos preciosos como los anticuerpos. Una alternativa a los portaobjetos de ocho cámaras es una placa de cultivo de tejido de seis pocillos con tapas estándar (22 mm x 22 mm) que son estériles. Una disposición similar es posible con tapas circulares y una placa de cultivo de tejido de 24 pocillos. Para ambos, aumente los volúmenes mencionados en este protocolo en 10-15x (por ejemplo, solución de hibridación de 1,75 ml) y 4x (por ejemplo, solución de hibridación de 600 ml) respectivamente.- Para las células líticas adherentes, utilice 1x trippsina/PBS a 37oC y 5%CO2 durante 10 min para suspender las células y diluir al 60% de confluencia.
NOTA: Las líneas celulares adherentes utilizadas en los experimentos FISH incluían 293T, iSLK.2197e iSLK-BAC36 celdas8. - Aplicar 200 ml de suspensión celular en cada cámara de los portaobjetos estériles de ocho cámaras ypermitir el crecimiento de las semillas durante 12-24 h a 37 oC y 5% de CO 2. Ajuste según sea necesario para las células de crecimiento lento o rápido y para las células que son fácilmente dañadas por la trippsina.
NOTA: El objetivo es tener celdas espaciadas uniformemente firmemente fijadas a la diapositiva. Considere la posibilidad de inducir la fase lítica después de la adhesión si las células líticas son frágiles. Las conclusiones extraídas de experimentos con células iSLK son limitadas9. - Para las células de suspensión lítica, pre-tratar diapositivas de ocho cámaras con 1:10 poli L-lisina durante 5 minutos bajo la capucha de cultivo de tejido. A continuación, deje que los portaobjetos se sequen durante la noche a temperatura ambiente o 1 h a 65 oC. Incubar 800 l de células líticas a una concentración de 1 x 106 células/ml con las diapositivas con cámara durante 30 min a 1 h a 37oC y 5% co2 .
NOTA: Las células de suspensión se asentarán en una monocapa, adándose a la poli L-lisina y por lo tanto el exceso de células no son una preocupación en comparación con las células adherentes. Las células líticas que forman racimos de uva, si es posible, deben separarse por un suave vórtice o medios químicos. Es cierto que los autores no han tenido mucho éxito con tales recomendaciones en el caso de las células líticas BJAB-RRV-GFP. Si las células de suspensión no se adhieren bien, considere aumentar el tiempo o la concentración de la incubación de poli L-lisina.
- Para las células líticas adherentes, utilice 1x trippsina/PBS a 37oC y 5%CO2 durante 10 min para suspender las células y diluir al 60% de confluencia.
3. Fijación, Inmunofluorescencia (Opcional), Hibridación y Visualización de ARN virales
- Retire los medios y el exceso de células. A lo largo de este protocolo, utilice la succión al vacío para eliminar las soluciones y el micropipetesuave suave para agregar soluciones.
NOTA: La resistencia de un vacío se puede reducir colocando una punta de micropipeta de 200 ml sobre la pipeta Pasteur de vidrio. Sustituya la punta del micropipeta entre los pasos de lavado para evitar la contaminación. Cada paso de lavado debe realizarse rápidamente porque es imperativo que las células nunca se sequen. - Inmediatamente, fije las células con un 4% de formaldehído/PBS (salina tamponada en fosfato) preenfriado en hielo durante 30 minutos.
- Permeabilizar las células fijas con 200 ml de Triton-X/PBS preenfriado 0,5% (sal insa tamponada de fosfato) durante 10 minutos en hielo o 750 ml de etanol preenfriado 70% a 4 oC durante 1 h (min) a 7 d (máx.).
NOTA: Recoger muestras de proteína, ARN total y ADN genómico en el punto de fijación para garantizar la coherencia entre imágenes y ensayos bioquímicos. Todos los lavados a lo largo de este protocolo se realizan de la misma manera a menos que se especifique lo contrario. 70% etanol afloja el pegamento entre las cámaras y la corredera, lo que facilita la separación posterior, y también proporciona una pausa significativa en el protocolo. Sin embargo, utilice película de parafina alrededor de la corredera de la cámara para reducir la evaporación y comprobar el nivel del etanol en cada cámara aproximadamente cada 8 h. 70% etanol también aplana las células, haciendo una imagen más nítida, mientras que Triton-X no deshidrata las células y cambia la dimensiones de la celda.
- Permeabilizar las células fijas con 200 ml de Triton-X/PBS preenfriado 0,5% (sal insa tamponada de fosfato) durante 10 minutos en hielo o 750 ml de etanol preenfriado 70% a 4 oC durante 1 h (min) a 7 d (máx.).
- Retire las cámaras con cuidado para evitar que se agriete la corredera. Si el experimento incluye inmunofluorescencia (IF) de una proteína viral o huésped con un anticuerpo primario policlonal, realice el IF como se describe a continuación antes de proceder al ARN FISH. Si la inmunofluorescencia utiliza un anticuerpo primario monoclonal, realice la inmunofluorescencia como se describe en el paso 3.3.1 después del paso 3.11.
NOTA: Utilice un dispositivo de extracción fresco o uno con muy poco adhesivo sobrante proporcionado por el fabricante y alivie suavemente las cámaras para evitar que la diapositiva se agriete. El uso de 70% de etanol como reactivo permeabilizador durante 4 h reduce en gran medida la probabilidad de agrietamiento. En el caso de una grieta, continúe el protocolo en cámaras no afectadas por la grieta y tenga en cuenta la mayor tasa de oxidación de las diapositivas imperfectamente selladas (es decir, disminución de la vida útil de almacenamiento).- Enjuague las células con 1pbS preenfriado y bloquee con 4% de BSA (albúmina sérica bovina) /1x PBS pre-enfriado durante 30 min a 4 oC.
NOTA: El uso de BSA a lo largo de este protocolo limita el etiquetado inespecífico. - Retire la solución de bloqueo e incubar las células con 1:200 u otro anticuerpo primario policlonal en 0,1% de BSA/1x PBS durante 1 h a 4 oC. Luego lave tres veces con 1x PBS.
NOTA: Se utilizó un anticuerpo10 para la detección de SSB/ORF6 (proteína de unión de ADN de una sola cadena viral) a 1:200 dilución. - Incubar las células con un anticuerpo secundario con fluoróforo compatible con el anticuerpo de detección fish durante 1 h a 4 oC. Lávese tres veces con 1 pbS. A continuación, fijar con 4% de formaldehído/1x PBS para 10-15 min y permeabilizar con Triton-X o 70% etanol como se describió anteriormente antes de proceder a FISH. Cubra el portaobjetos con papel de aluminio para preservar la señal fluorescente y evitar el fotoblanqueo.
- Enjuague las células con 1pbS preenfriado y bloquee con 4% de BSA (albúmina sérica bovina) /1x PBS pre-enfriado durante 30 min a 4 oC.
- Lavar las células con 2 sSC (citrato de sodio salino) una vez y luego aplicar 45 l de solución de hibridación que consiste en 50% de formamida, 10% de sulfato de dextrano, 2x SSC, 0,1% BSA, 500 g/ml de ADN esperma de salmón, 125 g/ml de e. coli tRNA y 1 mM de vanya Complejos. Incubar durante 1 h a 37oC en una cámara de humedad que puede ser una placa Petri de 150 mm con toallitas estériles humedecidas.
NOTA: Prepare una solución de hibridación fresca al menos una hora antes de su uso. Disolver primero el sulfato de dextran en agua, vórtice con frecuencia e incubando en un baño de agua a 37 oC. - Calcule para tener una concentración sugerida de oligonucleótidos de 25 m en una solución de hibridación de 35-uL por cámara. Ajuste la concentración de oligonucleótido antisentido según sea necesario. Añadir agua destilada a los oligonucleótidos para llevar el volumen de desnaturalización a 10 l.
NOTA: Tras la reacción de etiquetado, los oligonucleótidos se almacenan en la solución alargada que contiene 0,18 M de cacodilato de potasio, Tris-HCl de 23 mM, 0,23 mg/mL BSA, 4,5 mM de CoCl2,18 mM EDTA, 2,7 mM de K-fosfato y 6,8 mM de KCl, 45 MM de 2 Mcaptoetanol, 45 mM 2-Mercaptoetanol, 45 MM 2-Mercaptoetanol, 45 MM 2-Mercaptoetanol, 45 MM 2-Mercapto, 0.02% Tritón X-100, y 2% glicerol. Las concentraciones son lo suficientemente altas como para que la dilución con agua lleve la solución de desnaturalización a concentraciones cercanas a 1x TE (10 mM Tris-HCl y 1 mM EDTA), un tampón de desnaturalización de oligonucleótidos estándar. - Desnaturalizar los oligonucleótidos con etiqueta DIG y/o Alexa Fluor 594 a 95 oC durante 5 min. A continuación, añada una solución de hibridación fresca de 35 l por cámara prevista a los oligonucleótidos desnaturalizados. Si se realiza el doble FISH, ambos conjuntos de oligonucleótidos antisentido pueden desnaturalizarse e hibridarse juntos.
- Quite la solución de pre-hibridación y, a continuación, agregue la solución de hibridación que contenga los oligonucleótidos etiquetados a las células. Incubar durante la noche en la cámara de humedad a 37 oC con papel de aluminio para proteger los oligonucleótidos etiquetados con fluoróforo.
NOTA: La incubación debe ser de al menos 10 h y no superior a 24 h. - Al día siguiente, lave las células dos veces con 2 sSC durante 10 minutos a 37 oC y luego dos veces con 1 SSC durante 10 min a 25 oC.
- Fijar las células con un 4% de formaldehído/1x PBS pre-enfriado durante 10-15 minutos sobre hielo. A continuación, lave las células con PBS tres veces y permeabilice durante 1 h con etanol pre-enfriado 70% o durante 10 minutos con 0.5% Triton-X/1x PBS pre-enfriado a 4oC.
- Incubar las células con 1:200 FITC anti-DIG en pre-enfriado 0.1% BSA/1x PBS durante 1 h a 4oC. Retire la solución de anticuerpos y lávela tres veces con 1 x PBS.
- Arreglar con un 4% de formaldehído/1x PBS pre-enfriado/1x PBS durante 10-15 min a 4oC y luego lavar tres veces con 1x PBS. Si realiza inmunofluorescencia para un huésped o proteína viral con un anticuerpo primario monoclonal, permeabilizar las células y luego realizar el protocolo IF descrito en el paso 3.3.1. De lo contrario, proceda a la tinción de DAPI.
- Incubar las células con 0,4 g/ml de DAPI en P.N. pre-enfriado 0.5% Triton-X/1x PBS durante 15 minutos sobre hielo y luego lavar tres veces con 1x PBS.
- Monte portaobjetos con perlas fluorescentes (opcional) y un medio de montaje. A continuación, sellar la cubierta a la diapositiva con esmalte de uñas transparente.
- Usando un microscopio confocal, recopile imágenes de las muestras dentro de una hora a una semana de realizar el protocolo a una ampliación de 630x. Aplique múltiples capas de esmalte de uñas para sellar el resbalón de la cubierta y prolongar la vida útil del fluoróforo reduciendo la tasa de oxidación.
NOTA: No utilice un medio de montaje que contenga DAPI. Al recopilar las imágenes, incluya la barra de escala en cada imagen para una cuantificación posterior. Las perlas fluorescentes sirven como controles de la intensidad de la fluorescencia entre los portaobjetos y las preparaciones de la muestra11. Adquiera imágenes en la sección media de la celda para la cuantificación bidimensional (2D) en el paso 4.
4. Cuantificación de imágenes FISH e IF para resaltar la localización subcelular y determinar la relación nucleocitotoplasmática de fluorescencia
- Realice análisis de imágenes en una pila montada de las diversas imágenes fusionadas y teñidas con fluorescentes para garantizar la consistencia. Establezca la escala del software de análisis de imágenes utilizando la barra de escala incluida cuando se recopilaron las imágenes.
- Para cuantificar la intensidad de la fluorescencia a través de varios canales y en referencia a la mancha DAPI nuclear, utilice una herramienta de línea y una función de perfil de trazado. A continuación, indique la línea de forma permanente en una copia de la imagen utilizando marcadores que no obstruyan o influyan en el juicio del espectador.
- Establecer criterios para guiar dónde se dibuja la línea, como un rastro que captura una diversidad de características topográficas, picos y valles, a lo largo de un eje central o una línea que no atraviesa áreas sobresaturadas.
NOTA: Estos rastros de línea representan la fluorescencia cruda en una célula y, por lo tanto, se limitan a las comparaciones de las ubicaciones de una mancha, no de la intensidad. Para comparar las intensidades de la misma mancha entre diapositivas, tratamientos o preparaciones, agregue un cordón fluorescente a la diapositiva como control interno durante el paso 3.13. El cordón fluorescente debe añadirse durante el proceso de montaje y detectarse con los mismos ajustes en el láser de excitación y el tubo fotomultiplicador (confocal).
- Establecer criterios para guiar dónde se dibuja la línea, como un rastro que captura una diversidad de características topográficas, picos y valles, a lo largo de un eje central o una línea que no atraviesa áreas sobresaturadas.
- Para cuantificar un cambio en la localización subcelular, calcule las proporciones nucleocitoplasmáticas de las células sometidas a diferentes tratamientos.
- Mida el área y la intensidad de fluorescencia cruda tanto del núcleo como del citoplasma utilizando la mancha DAPI nuclear para establecer el límite interno. Incluya controles nucleares y citoplasmasmáticos como un ARN nuclear (por ejemplo, ARN KSHV PAN) y ARN citoplasma (por ejemplo, ARNm GAPDH anfitrión). Por otra parte, calcule la intensidad de fondo para tres áreas similares a las celdas y promedialos los valores por píxel o2.
NOTA: Los valores de intensidad tienden a carecer de unidades, por lo que se utiliza el término 'unidades'. - Normalice los valores de intensidad bruta nuclear y celular determinando primero el fondo promedio para la misma área y luego restando ese valor individualizado de la intensidad bruta de la zona.
- Por ejemplo, un núcleo de una célula B lítica tiene un área de 133,4 m2 y una intensidad bruta de 75976 unidades, mientras que la intensidad de fondo de la misma señal fluorescente se determinó que era de 0,67 unidades por m2. La intensidad nuclear normalizada sería
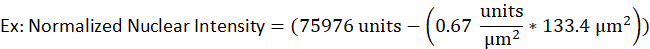
- Por ejemplo, un núcleo de una célula B lítica tiene un área de 133,4 m2 y una intensidad bruta de 75976 unidades, mientras que la intensidad de fondo de la misma señal fluorescente se determinó que era de 0,67 unidades por m2. La intensidad nuclear normalizada sería
- Introduzca los valores en la siguiente ecuación.

NOTA: Este cálculo controla los cambios en el área subcelular. La inducción lítica y los tratamientos farmacológicos pueden agrandar el núcleo o cambiar el tamaño de la célula, respectivamente. - Para interpretar los resultados, cree un trazado de bigote de caja. Una distribución equitativa de la señal fluorescente sería cercana a cero, mientras que una distribución nuclear favorecería un valor de relación positivo y una distribución citoplasmática sería tendencia hacia un valor de relación negativo.
- Mida el área y la intensidad de fluorescencia cruda tanto del núcleo como del citoplasma utilizando la mancha DAPI nuclear para establecer el límite interno. Incluya controles nucleares y citoplasmasmáticos como un ARN nuclear (por ejemplo, ARN KSHV PAN) y ARN citoplasma (por ejemplo, ARNm GAPDH anfitrión). Por otra parte, calcule la intensidad de fondo para tres áreas similares a las celdas y promedialos los valores por píxel o2.
Resultados
Los métodos FISH y IF detallados en este manuscrito se muestran en la Figura 1 junto con la cuantificación de los resultados por trazas de línea de intensidad fluorescente. Los resultados presentados aquí son semicuantitativos y ofrecen información sobre la localización, en lugar de en comparaciones entre las intensidades de diferentes manchas fluorescentes porque los experimentos no incluyeron un cordón fluorescente en la preparación de la diapositiv...
Discusión
El protocolo descrito en este informe se puede adaptar a diferentes tipos de células e incluye pasos para el doble ARN FISH y el ARN FISH con IF utilizando anticuerpos primarios monoclonales y policlonales. Aunque las diapositivas preparadas se suelen crear imágenes con un microscopio confocal, las imágenes se pueden realizar con un microscopio STED (agotamiento de emisiones estimulada) después de modificaciones del aumento de la concentración de anticuerpos y un medio de montaje diferente. Para un análisis mejorad...
Divulgaciones
Los autores no tienen conflictos de intereses que revelar.
Agradecimientos
Agradecemos a Jonathan Rodenfels, Kazimierz Tycowski y Johanna B. Withers por su asesoramiento sobre el análisis de datos. También agradecemos a G. Hayward por el anticuerpo anti-SSB. Este trabajo fue apoyado por las subvenciones T32GM007223 y T32AI055403 de los Institutos Nacionales de Salud (TKV) y la subvención NIH (CA16038) (a JAS). JAS es investigador del Instituto Médico Howard Hughes. Las figuras 1-3 y 1 se reprodujeron con permiso de la Sociedad Americana de Microbiología bajo una licencia Creative Commons Attribution de la siguiente publicación: Vallery, T. K., Withers, J. B., Andoh, J. A., Steitz, J. A. Kaposi's Sarcoma-Associated La acumulación de ARNm de herpesvirus en foci nucleares está influenciada por la replicación del ADN viral y el ARN nuclear no denilado no codificador viral. Revista de Virología. 92 (13), doi:10.1128/JVI.00220-18, (2018).
Materiales
| Name | Company | Catalog Number | Comments |
| AlexaFluor594-5-dUTP | Life Technologies | C1100 | |
| anti-DIG FITC | Jackson Lab Immunologicals | 200-092-156 | |
| Anti-Rabbit Secondary AlexaFluor594 Monoclonal Antibody | Invitrogen | A-11037 | Goat |
| Anti-SSB Antibody | N/A | N/A | Ref. Chiou et al. 2002 |
| BLASTn | NIH NCBI | N/A | Free Sequence Alignment Software |
| Dextran Sulfate | Sigma Aldrich | D8906 | Molecular Biology Grade |
| DIG-Oligonucleotide Tailing Kit | Sigma Roche | #03353583910 | 2nd Gen |
| Eight-Chamber Slides | Nunc Lab Tek II | #154453 | Blue seal promotes surface tension but separation by clear gel is also available. |
| Formamide | Sigma Aldrich | F9037 | Molecular Biology Grade |
| GAPDH Probes | Stellaris | SMF-2019-1 | Compatible with protocol, Quasar 670 |
| ImageJ | NIH, Bethesda, MD | N/A | Free Image Analysis Software, [http:rsb.info.nih.gov/ij/] |
| OligoAnalyzer | IDT | N/A | Free Oligonucleotide Analyzer |
| pcDNA3 | Invitrogen | A-150228 | |
| pmaxGFP | Amaxa | VDF-1012 | |
| Poly L-Lysine | Sigma Aldrich | P8920 | |
| Terminal Transferase | Sigma Roche | #003333574001 | |
| Vanadyl Ribonucleoside Complexes | NEB | S1402S | |
| Vectashield | Vector Laboratories, Inc. | H-1000 | DAPI within the mounting media scatters the light and reduces contrast. |
Referencias
- Amen, M. A., Griffiths, A. Packaging of Non-Coding RNAs into Herpesvirus Virions: Comparisons to Coding RNAs. Frontiers in Genetics. 2, 81 (2011).
- Schmid, M., Speiseder, T., Dobner, T., Gonzalez, R. A. DNA virus replication compartments. Journal of Virology. 88 (3), 1404-1420 (2014).
- Pawlicki, J. M., Steitz, J. A. Primary microRNA transcript retention at sites of transcription leads to enhanced microRNA production. Journal of Cell Biology. 182 (1), 61-76 (2008).
- Borah, S., Darricarrere, N., Darnell, A., Myoung, J., Steitz, J. A. A viral nuclear noncoding RNA binds re-localized poly(A) binding protein and is required for late KSHV gene expression. Public Library of Science Pathogens. 7 (10), e1002300 (2011).
- Tycowski, K. T., Shu, M. D., Borah, S., Shi, M., Steitz, J. A. Conservation of a triple-helix-forming RNA stability element in noncoding and genomic RNAs of diverse viruses. Cell Reports. 2 (1), 26-32 (2012).
- Weinberg, R. A., Penman, S. Small molecular weight monodisperse nuclear RNA. Journal of Molecular Biology. 38 (3), 289-304 (1968).
- Myoung, J., Ganem, D. Generation of a doxycycline-inducible KSHV producer cell line of endothelial origin: maintenance of tight latency with efficient reactivation upon induction. Journal of Virology Methods. 174 (1-2), 12-21 (2011).
- Brulois, K. F., et al. Construction and manipulation of a new Kaposi's sarcoma-associated herpesvirus bacterial artificial chromosome clone. Journal of Virology. 86 (18), 9708-9720 (2012).
- Sturzl, M., Gaus, D., Dirks, W. G., Ganem, D., Jochmann, R. Kaposi's sarcoma-derived cell line SLK is not of endothelial origin, but is a contaminant from a known renal carcinoma cell line. International Journal of Cancer. 132 (8), 1954-1958 (2013).
- Chiou, C. J., et al. Patterns of gene expression and a transactivation function exhibited by the vGCR (ORF74) chemokine receptor protein of Kaposi's sarcoma-associated herpesvirus. Journal of Virology. 76 (7), 3421-3439 (2002).
- Cole, R. W., Jinadasa, T., Brown, C. M. Measuring and interpreting point spread functions to determine confocal microscope resolution and ensure quality control. Nature Protocols. 6 (12), 1929-1941 (2011).
- Nakamura, H., et al. Global changes in Kaposi's sarcoma-associated virus gene expression patterns following expression of a tetracycline-inducible Rta transactivator. Journal of Virology. 77 (7), 4205-4220 (2003).
- Majerciak, V., Yamanegi, K., Zheng, Z. M. Gene structure and expression of Kaposi's sarcoma-associated herpesvirus ORF56, ORF57, ORF58, and ORF59. Journal of Virology. 80 (24), 11968-11981 (2006).
- Sun, R., Lin, S. F., Gradoville, L., Miller, G. Polyadenylylated nuclear RNA encoded by Kaposi sarcoma-associated herpesvirus. Proceedings of the National Academy Sciences U S A. 93 (21), 11883-11888 (1996).
- Vallery, T. K., Withers, J. B., Andoh, J. A., Steitz, J. A. Kaposi's Sarcoma-Associated Herpesvirus mRNA Accumulation in Nuclear Foci Is Influenced by Viral DNA Replication and Viral Noncoding Polyadenylated Nuclear RNA. Journal of Virology. 92 (13), (2018).
- Borah, S., Nichols, L. A., Hassman, L. M., Kedes, D. H., Steitz, J. A. Tracking expression and subcellular localization of RNA and protein species using high-throughput single cell imaging flow cytometry. RNA. 18 (8), 1573-1579 (2012).
- Bruce, A. G., et al. Quantitative Analysis of the KSHV Transcriptome Following Primary Infection of Blood and Lymphatic Endothelial Cells. Pathogens. 6 (1), (2017).
- Chen, C. P., et al. Kaposi's Sarcoma-Associated Herpesvirus Hijacks RNA Polymerase II To Create a Viral Transcriptional Factory. Journal of Virology. 91 (11), (2017).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados