JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
KSHV 감염 세포에서 특정 유전자 제품의 시투 혼성화 (FISH) 및 면역 형광 (IF)의 정량형 형광
요약
우리는 현탁액 또는 부착물에서 서정적으로 감염된 인간 세포 내의 다중 헤르페스 바이러스 RNA를 시각화하기 위해 situ 혼성화 (FISH)에서 형광을 활용하는 프로토콜을 설명합니다. 이 프로토콜은 뉴클레오세포질을 생성하는 형광의 정량화를 포함하고 면역형광(IF)을 가진 숙주 및 바이러스 성 단백질의 동시 시각화를 위해 확장될 수 있다.
초록
기계론적 통찰력은 특정 RNA 및 단백질의 신중한 연구 및 정량화에서 옵니다. 특정 시간에 세포 를 통해 이 생체 분자의 상대적인 위치는 위치 혼성화 (FISH) 및 면역 형광 (IF)에 있는 형광으로 붙잡을 수 있습니다. lytic 헤르페스 바이러스 감염 도중, 바이러스는 우대적으로 바이러스성 유전자를 표현하기 위하여 호스트 세포를 납치하고, 생체 분자의 세포 형태 그리고 행동에 있는 변경을 일으키는 원인이 됩니다. Lytic 활동은 원자력 공장을 중심으로, 물고기와 IF로만 식별 할 수있는 바이러스 복제 구획이라고합니다. 여기에서 우리는 카포시의 육종 관련 헤르페스 바이러스 (KSHV)에 대한 RNA FISH 및 IF 기술의 적응 프로토콜을 설명합니다, 모두 부착 및 현탁액. 이 방법은 특정 안티 센스 올리고뉴클레오티드, 이중 RNA FISH, IF를 이용한 RNA FISH, 및 형광 강도의 정량적 계산의 개발을 위한 단계를 포함한다. 이 프로토콜은 여러 세포 유형, 감염되지 않은 세포, 잠복 세포, 용리 세포, 시간 과정 및 억제물로 처리된 세포에 성공적으로 적용되어 인간 숙주 및 단백질 모두에서 특정 RNA 및 단백질의 시공간 활동을 분석했습니다. KSHV.
서문
그들의 용리 (활성) 단계에서, 헤르페스 바이러스는 숙주 세포를 납치하여 세포 형태와 생물학적 분자의 국소화의 변화를 일으켜 비리온을 생성합니다. 작전의 기초는 이중 가닥 DNA 바이러스 게놈이 capsid1에게불린 단백질 껍질로 복제되고 포장되는 핵입니다. 시작하기 위하여, 바이러스는 그것의 자신의 단백질을 표현하고, 호스트 기계를 납치하고 비필수 호스트 유전자의 발현을 방지합니다, 프로세스는 호스트 차단 효력을 불릴. 이 활동의 대다수는 특정 4′,6-diamidino-2-phenylindole (DAPI)가 없는 바이러스 성 복제 구획이라고 불리는, 호스트 및 바이러스 성 단백질,RNA 및 바이러스 DNA 2로 구성된 지역화된다. 셀은 복제 구획에 대한 공간과 자원을 제공하기 위해 정밀 검사되어 바이러스 성 캡시드의 조립을 합니다. 일단 capsid가 핵을 종료하면, 캡시드가 세포질에서 어떻게 비리온이라고도 하는 막 결합된 바이러스성 입자를 생성하기 위하여 포위되는지, 불분명합니다. 용해 단계 도중 호스트와 바이러스성 생체 분자 둘 다의 현지화 그리고 공간 교대의 이해는 복제 구획의 배열, 호스트 차단 효력, virion-egress 통로 및 그밖에 더 깊은 기계론적인 통찰력을 제공합니다 헤르페스 바이러스 감염 및 복제와 관련된 프로세스.
현재 이러한 변화를 감지하고 연구하는 가장 좋은 방법은 각각 동형형형화(FISH)에서 면역형광(IF)과 형광을 가진 감염된 세포에서 단백질과 RNA의 시각화입니다. 이러한 기술을 사용한 타임 코스를 사용하면 용해 단계의 핵심 지점에서 생체 분자의 국소화 또는 단순히 시공간 적 데이터를 알 수 있습니다. FISH 및 IF는 세포 과정의 억제(예를 들어, 바이러스 DNA 복제의 억제), RT-qPCR(실시간 중합효소 연쇄 반응), RNA 시퀀싱, 북부 블롯, 질량 분광법, 웨스턴 블로팅, 및 바이러스 DNA 생산의 분석, 그 세포 활동의 더 글로벌 그림을 제공할 수 있습니다.
우리는 특정 유전자에서 RNA 제품을 검사하는 RNA FISH 전략 및 특정 유전자 생성물의 뉴클레오세포 비율을 정량적으로 계산하는 계산 분석을 개발했습니다. Steitz 및 동료3,4에의해 이전 간행물에서 수정된 샘플 준비는 상대적으로 용이하며 부착 및 부유 셀 모두에 사용할 수 있습니다. 이 프로토콜은 또한 IF 전략과 다중 RNA FISH 전략 (이중 RNA FISH) 또는 RNA FISH의 동시 사용에 적응할 수 있습니다. 특정 FISH 전략의 개발은 어렵지만 성공을 개선하기 위한 제안이 설명되어 있습니다. 여기에 설명된 데이터 분석은 형광 비드와 구획 경계의 강력한 마커를 사용하고 현미경 조사에 대한 추가 통찰력을 제공하는 경우 정량적이며 관찰 편향을 제거하는 통찰력을 제공합니다. 상세 프로토콜은 Kaposi의 육종 관련 헤르페스 바이러스 (KSHV)에 의해 감염된 잠재 세포와 용염 세포를 위해 설계되었으며 다른 헤르페스 바이러스에 감염된 감염되지 않은 세포 또는 세포와 함께 사용할 수있습니다5. 정량의 방법은 대부분의 세포에 있는 subcellular 구획 사이 뉴클레오세포 세포 교대 또는 재국소화에 연구 결과에 적용할 수 있습니다.
프로토콜
1. 특정 헤르페스 바이러스 전사체를 검출하는 위치 (FISH) 안티 센스 올리고 뉴클레오티드에서 형광의 설계
- 관심 있는 RNA의 서열로부터 25~40개의 nt 세그먼트를 선택하고 안티센스로 변환한다. 성공적인 FISH 전략은 1개에서 10개 이상의 상이한 안티센스 올리고뉴클레오티드를 포함할 수 있다. 시퀀스를 선택할 때 다음을 고려하십시오.
- 관심 있는 RNA가 독특한 반복 영역을 포함하는 경우, 이 특징을 활용하고 반복 서열을 표적으로 하는 안티센스 올리고뉴클레오티드를 설계한다.
참고: Tycowski 및 동료 5는 rhesus rhadinovirus (RRV) 폴리아데닐화 핵 (PAN) RNA를 가진 이 전략의 보기를 제공합니다. - 관심 있는 RNA가 공지된 단백질 결합 부위 또는 줄기 루프 구조를 포함하는 경우, 이러한 부위를 피하는 올리고뉴클레오티드를 설계한다.
- 실험의 목표에 따라, 내향적 서열및 그것에 대한 안티센스 올리고뉴클레오티드를 설계할지 여부를 고려한다.
- 관심 있는 RNA가 독특한 반복 영역을 포함하는 경우, 이 특징을 활용하고 반복 서열을 표적으로 하는 안티센스 올리고뉴클레오티드를 설계한다.
- 선택된 안티센스 서열에 대한 간단한 계산 분석을 수행하여 결합 특이성을 보장하고 안티센스 올리고뉴클레오티드의 응집을 줄입니다.
- 서열은 약 50% GC-풍부(고구아닌 및 시토신 함량)이어야 하며 60~70°C 의 범위에서 용융 온도를 가져야 한다.
- 시퀀스 분석기 도구를 사용하여 혼성화 온도인 37°C 이상의 용융 온도로 헤어핀을 자체 이화하거나 형성하지 않는 서열을 선택합니다.
- NCBI BLASTn (뉴클레오티드 정렬을위한 국립 생명 공학 정보 기본 지역 정렬 검색 도구) '다소 유사한'설정을 사용하여 숙주 및 바이러스 성 전사체 모두에 대해 선택된 서열을 검색수행합니다. 이 검색은 다른 호스트 또는 바이러스 성 전사체에 결합하지 않을 독특한 안티 센스 올리고뉴클레오티드를 식별합니다.
참고: 전사체를 사용할 수 없는 경우 게놈 서열로 BLAST 검색을 수행합니다. 야생 균주가 다양화하고 다른 실험실 균주에서 서열의 조합을 포함하는 경향이 있기 때문에 실험에 사용되는 감염된 세포로부터 분리 된 바이러스로부터의 서열에 대한 검색이 수행되는 것이 이상적입니다.
- 정제된 DNA 올리고뉴클레오티드는 표적 RNA에 결합할 가능성이 유일하고 유일하도록 전산적으로 검증된 항센스 서열에 상응하는 것을 주문한다. 올리고뉴클레오티드에 특별한 수정을 도입할 필요가 없습니다.
- FISH 및 노던 블롯에 의한 결합 특이성을 위해 설계된 안티센스 올리고뉴클레오티드를 테스트합니다.
- 동일한 숙주 종(예를 들어, 293T)과 이상적으로 동일한 세포 유형으로부터의 감염되지 않은 세포주들을 사용하여, 강력한 프로모터(CMV, cytomegalovirus)로부터 관심 있는 RNA를 발현하는 플라스미드와 빈 벡터(예를 들어, pcDNA3)를 이용한 형질감염을 수행한다. GFP(녹색 형광 단백질) 플라스미드(예를 들어, pmaxGFP) 또는 GFP를 함유하는 벡터와의 공동 형질감염과 같은 형질감염에 대한 양성 대조군을 사용한다.
참고: 헤르페스바이러스 사이에 서열 유사성이 있기 때문에 헤르페스바이러스 엡스타인-바 바이러스(EBV)를 사용하여 불멸화된 세포주를 피하는 것이 중요합니다. - 본 프로토콜에 기재된 바와 같이 항센스 올리고뉴클레오티드를 가진 두 세포 세트상에서 섹션 3에 기재된 바와 같이 FISH를 수행한다. 물고기 실험을 안티센스 올리고뉴클레오티드의 개별, 쌍 또는 세트와 비교하여 성공적인 후보를 추론합니다. 인간 세포 핵 당 500,000 카피에서 존재하는 U2 snRNA(small nuclear RNA) FISH와 같은 FISH 프로토콜에 대한 양성 대조군을 사용한다 6(표 1).
- 형광 신호는 관심 있는 RNA를 포함하는 세포에서 특이적이고 강해야 한다. 신호를 강화하고 비특이적 고려에서 결합 안티 센스 올리고 뉴클레오티드를 제거하기 위해 추가 안티 센스 올리고 뉴클레오티드를 설계합니다. 신호 강도는 배경 및 자동 형광 이상이어야 합니다.
- 북부 블롯에 의한 테스트 바인딩 특이성.
- 동일한 숙주 종(예를 들어, 293T)과 이상적으로 동일한 세포 유형으로부터의 감염되지 않은 세포주들을 사용하여, 강력한 프로모터(CMV, cytomegalovirus)로부터 관심 있는 RNA를 발현하는 플라스미드와 빈 벡터(예를 들어, pcDNA3)를 이용한 형질감염을 수행한다. GFP(녹색 형광 단백질) 플라스미드(예를 들어, pmaxGFP) 또는 GFP를 함유하는 벡터와의 공동 형질감염과 같은 형질감염에 대한 양성 대조군을 사용한다.
2. 올리고뉴클레오티드 및 세포 제제
- 제조업체의 지침에 따라, 단말 트랜스퍼라제에 다이옥신(DIG)-dUTP와 함께 안티센스 올리고뉴클레오티드에 라벨을 붙이거나, 강하게 결합하는 경우 알렉사 플루오르 594-5-dUTP와 같은 형광 뉴클레오티드로 직접 라벨을 붙입니다. 라벨링 후 추가 정제가 필요하지 않습니다. 광표백을 방지하기 위해 직접 라벨을 붙인 경우 -20°C에서 최대 몇 년, 주석 호일에 표지된 올리고뉴클레오티드를 보관하십시오.
주의 : 라벨 링 솔루션에는 독성 물질 인 카코 디레이트 칼륨이 포함되어 있습니다. 장갑으로 라벨링 반응을 처리합니다.
참고: 자원을 보존하기 위해, 여러 가지 다른 안티 센스 올리고뉴클레오티드를 하나의 반응으로 표지할 수 있다. 이 프로토콜은 3'엔드 라벨링을 활용합니다. 화학군(예를 들어, DIG 또는 형광소)이 잡히거나 DNA 폴리머라제의 활성 부위에 들어갈 수 없기 때문에 내부 라벨링은 어렵다. 2개의 상이한 RNA를 이용한 실험은 상이한 플루오로포르(예를 들어, FITC(플루오레세인) 또는 알렉사 플루오르 594를 이용한 알렉사 플루오르 488)을 이용한 직접 표지된 안티센스 올리고뉴클레오티드 및 안티-DIG 면역형광을 사용하여 수행될 수 있다. - 8 챔버 슬라이드에 세포를 부착합니다.
참고 : 8 챔버 슬라이드는 항체와 같은 귀중한 자원을 최소화하면서 여러 가지 동시 실험을 허용합니다. 8 챔버 슬라이드에 대한 대안은 모두 멸균 표준 커버 슬립 (22mm x 22mm)와 여섯 잘 조직 배양 플레이트입니다. 원형 커버립과 24 웰 조직 배양 플레이트와 유사한 배열이 가능합니다. 둘 다, 이 프로토콜에 언급된 볼륨을 각각 10-15x(예를 들어, 1.75 mL 하이브리드화 솔루션) 및 4배(예를 들어, 600 μL 하이브리드화 용액)로 증가시다.- 부착성 용액 세포의 경우, 37°C에서 1x 트립신/PBS를 사용하고 10분 동안 5% CO2를 사용하여 세포를 일시 중단하고 60% 의 수축률로 희석하십시오.
참고 : FISH 실험에 사용되는 부착 세포주 에는 293T, iSLK.2197및 iSLK-BAC36 셀8이포함되었습니다. - 멸균 된 8 챔버 슬라이드의 각 챔버에 200 μL의 세포 현탁액을 적용하고 37 °C 및5 % CO2에서 12-24 시간 동안 종자 성장을 허용합니다. 느리거나 빠르게 성장하는 세포와 트립신에 의해 쉽게 손상된 세포에 대해 필요에 따라 조정하십시오.
참고: 목적은 슬라이드에 균등하게 간격을 두는 셀을 고정하는 것입니다. lytic 세포가 깨지기 쉬운 경우에 접착 후에 lytic 단계를 유도하는 것을 고려하십시오. iSLK 세포를 가진 실험에서 그려진 결론은9. - 용액 현탁액 세포의 경우, 조직 배양 후드 하에서 5 분 동안 1:10 폴리 L-리신으로 8 챔버 슬라이드를 미리 치료하십시오. 그런 다음 슬라이드를 실온에서 밤새 건조하거나 65°C에서 1시간 동안 건조시도록 둡니다. 37°C 및 5% CO2에서 30 분 내지 1 시간 동안 챔버 슬라이드와 함께 1 x 106 세포 /mL의 농도로 800 μL의 용리 세포를 배양합니다.
참고 : 서스펜션 세포는 폴리 L-리신에 집착하여 단층으로 정착하므로 과잉 세포는 부착 세포에 비해 문제가되지 않습니다. 포도 클러스터를 형성 하는 Lytic 세포, 가능 하면, 부드러운 소용돌이 또는 화학 수단에 의해 분리 되어야 한다. 명백하게, 저자는 lytic BJAB-RRV-GFP 세포의 경우에 그 같은 권고와 가진 많은 성공을 하지 않았습니다. 현탁액 세포가 잘 부착되지 않는 경우, 폴리 L-리신 배양시간 또는 농도를 증가시키면 고려하십시오.
- 부착성 용액 세포의 경우, 37°C에서 1x 트립신/PBS를 사용하고 10분 동안 5% CO2를 사용하여 세포를 일시 중단하고 60% 의 수축률로 희석하십시오.
3. 고정, 면역 형광 (선택 사항), 혼성화 및 바이러스 성 RNA의 시각화
- 미디어와 과잉 세포를 제거합니다. 이 프로토콜 전반에 걸쳐 진공 흡입을 사용하여 용액을 제거하고 부드러운 마이크로 파이펫팅을 사용하여 솔루션을 추가하십시오.
참고: 200 μL 마이크로파이펫 팁을 유리 파스퇴르 파이펫 위에 놓음으로써 진공의 강도를 줄일 수 있습니다. 오염을 방지하기 위해 세척 단계 사이에 마이크로 파이펫 팁을 교체하십시오. 각 세척 단계는 세포가 건조하지 않는 것이 필수적이기 때문에 신속하게 수행되어야합니다. - 즉시, 미리 냉각 된 4 % 포름 알데히드 / PBS (인산완충 식염수)로 세포를 얼음에 30 분 동안 고정하십시오. 200 μL 1x PBS로 세포를 3 회 씻어 4 °C로 냉각시키고 실온또는 얼음에서 5 분 동안 배양하십시오.
- 얼음에 10 분 동안 미리 냉각 된 0.5 % 트리톤 - X / PBS (인산완충 식염수)의 200 μL로 고정 된 세포를 퍼메아 빌화하거나 4 °C에서 70 % 에탄올의 750 μL을 1 시간 (분)에서 7 d (최대)까지.
참고: 고정 시점에서 단백질, 총 RNA 및 게놈 DNA 샘플을 수집하여 이미지와 생화학 적 검사 사이의 일관성을 보장합니다. 이 프로토콜 전체의 모든 워시스는 달리 명시되지 않는 한 동일한 방식으로 수행됩니다. 70% 에탄올은 챔버와 슬라이드 사이의 접착제를 느슨하게 하여 나중에 분리를 용이하게 하고, 또한 프로토콜에서 상당한 일시 정지를 제공한다. 그럼에도 불구하고, 챔버 슬라이드 주위에 파라핀 필름을 사용하여 증발을 줄이고 각 챔버에서 약 8 시간 70 % 에탄올의 수준을 확인하여 세포를 평평하게하고 선명한 이미지를 만드는 한편 Triton-X는 세포를 탈수시키고 변경하지 않습니다. 셀의 치수를 가합니다.
- 얼음에 10 분 동안 미리 냉각 된 0.5 % 트리톤 - X / PBS (인산완충 식염수)의 200 μL로 고정 된 세포를 퍼메아 빌화하거나 4 °C에서 70 % 에탄올의 750 μL을 1 시간 (분)에서 7 d (최대)까지.
- 슬라이드가 갈라지는 것을 방지하기 위해 챔버를 조심스럽게 제거하십시오. 실험이 폴리클론 원발성 항체를 가진 바이러스 또는 숙주 단백질의 면역형광(IF)을 포함하는 경우, RNA FISH로 진행하기 전에 아래에 설명된 바와 같이 IF를 수행한다. 면역형광이 단일클론 원발성 항체를 사용하는 경우, 3.11단계 후 3.3.1단계에서 설명한 대로 면역형광을 수행한다.
참고: 제조업체에서 제공하는 신선한 제거 장치 또는 남은 접착제가 거의 없는 장치를 사용하고 슬라이드가 균열되지 않도록 챔버를 부드럽게 완화하십시오. 70% 에탄올을 4시간 동안 투과 시약으로 사용하면 균열가능성이 크게 줄어듭니다. 균열의 경우, 균열의 영향을받지 않는 챔버에 프로토콜을 계속하고 불완전하게 밀봉 된 슬라이드의 높은 산화 속도 (즉, 저장 수명 감소)를 염두에 두어야합니다.- 미리 냉각된 1x PBS로 세포를 헹구고 미리 냉각된 4% BSA(소 혈청 알부민)/4°C에서 30분 동안 1x PBS로 차단합니다.
참고: 이 프로토콜 전체에 걸쳐 BSA를 사용하면 비특이적 라벨링이 제한됩니다. - 차단 용액을 제거하고 4 °C에서 1 시간 동안 0.1 % BSA / 1x PBS에서 1 :200 또는 다른 다각적 인 1 차 항체로 세포를 배양하십시오. 그런 다음 1x PBS로 세 번 씻으하십시오.
참고: SSB/ORF6(바이러스 성 단일 가닥 DNA 결합 단백질)의 검출을 위한 항체 10을 1:200 희석에서 사용하였다. - 4°C에서 1시간 동안 FISH 검출 항체와 호환되는 불소로 이차 항체로 세포를 배양한다. 1x PBS로 세 번 씻으소서. 그런 다음 4 % 포름 알데히드 / 1x PBS로 10-15 분 동안 수정하고 FISH로 진행하기 전에 앞에서 설명한 대로 트리톤-X 또는 70 % 에탄올로 투과합니다. 형광 신호를 보존하고 광 표백을 방지하기 위해 주석 호일로 커버 슬라이드.
- 미리 냉각된 1x PBS로 세포를 헹구고 미리 냉각된 4% BSA(소 혈청 알부민)/4°C에서 30분 동안 1x PBS로 차단합니다.
- 세포를 2x SSC(식염수 구연산염)로 한 번 씻은 다음 50% 포르마미드, 10% 덱스트란 황산염, 2x SSC, 0.1% BSA, 500 μg/mL 연어 정자 DNA, 125 μg/mL 대장균 tRNA, 1M의 대장균 tRNA, 1M의 성모네액으로 구성된 45 μL의 혼성화 용액을 적용합니다. 단지. 습한 멸균 물티슈로 150 mm 페트리 접시가 될 수 있는 습도 챔버에서 37°C에서 1시간 동안 배양한다.
참고: 사용하기 최소 한 시간 전에 신선한 혼성화 솔루션을 준비하십시오. 먼저 물에 덱스렌 황산염을 용해, 자주 소용돌이 와 37 °C 수조에서 배양. - 챔버당 35-uL 혼성화 용액에서 25 μM 올리고뉴클레오티드의 제안된 농도를 가지도록 계산한다. 필요에 따라 안티 센스 올리고 뉴클레오티드의 농도를 조정합니다. 올리고뉴클레오티드에 증류수를 첨가하여 변성 량을 10 μL로 가져옵니다.
참고 : 라벨 링 반응에 따라, 올리고뉴클레오티드는 0.18M 칼륨 카코디산염, 23mM 트리스-HCl, 0.23 mg/mL BSA, 4.5 mM CoCl2,18 mM EDTA, 2.7 mM K-인산염, 6.8 mM KCl, 45μg/mm을 함유하는 담금질 용액에 저장됩니다. 0.02% 트리톤 X-100, 2% 글리세롤. 농도는 물로 희석하면 표준 올리고뉴클레오티드 변성 완충제인 1x TE(10 mM Tris-HCl 및 1 mM EDTA)에 가까운 농도로 변성 용액을 가져올 만큼 충분히 높다. - DIG- 및/또는 알렉사 플루오르 594-표지 된 올리고뉴클레오티드를 95°C에서 5분 동안 변성시. 이어서 변성된 올리고뉴클레오티드에 의도된 챔버당 35 μL 의 신선한 혼성화 용액을 첨가한다. 이중 FISH를 수행하는 경우, 두 세트의 안티센스 올리고뉴클레오티드는 함께 변성되고 혼성화될 수 있다.
- 사전 혼성화 용액을 제거한 다음 표지된 올리고뉴클레오티드를 함유하는 혼성화 용액을 세포에 추가한다. 불소 표지 된 올리고뉴클레오티드를 보호하기 위해 주석 호일로 37 °C에서 습도 챔버에서 밤새 배양하십시오.
참고 : 배양은 적어도 10 시간 이상이어야하며 24 h 이상이어야합니다. - 다음날, 세포를 37°C에서 10분 동안 2x SSC로 두 번 세척한 다음 25°C에서 10분 동안 1x SSC로 두 번 세척합니다.
- 얼음에 10-15 분 동안 미리 차가운 4 % 포름 알데히드 / 1x PBS로 세포를 고정합니다. 그런 다음 세포를 PBS로 3회 세척하고 미리 냉각된 70% 에탄올로 1시간 동안 또는 4°C에서 미리 냉각된 0.5% 트리톤-X/1x PBS로 10분 동안 투과합니다.
- 4 °C에서 1 시간 동안 미리 냉각 된 0.1 % BSA / 1x PBS에서 1 :200 안티 DIG FITC로 세포를 배양하십시오. 항체 용액을 제거하고 1x PBS로 세 번 씻으하십시오.
- 4°C에서 10-15분 동안 미리 냉장 된 4 % 포름알데히드 / 1 x PBS로 고치고 1 x PBS로 세 번 씻으하십시오. 단일 클론 원발성 항체를 가진 숙주 또는 바이러스 성 단백질에 대한 면역 형광을 수행하는 경우, 세포를 투과한 다음 단계 3.3.1에 설명 된 IF 프로토콜을 수행한다. 그렇지 않으면 DAPI 염색을 진행합니다.
- 0.4 μg/mL DAPI로 세포를 미리 냉각된 0.5% 트리톤-X/1x PBS로 얼음에 15분 동안 배양한 다음 1x PBS로 3회 세척합니다.
- 형광 비드(선택 사항)와 마운팅 매체가 있는 슬라이드를 장착합니다. 그런 다음 뚜껑을 뚜렷한 매니큐어로 슬라이드에 밀봉합니다.
- 공초점 현미경을 사용하여 630배 배율로 프로토콜을 수행한 후 1시간에서 1주일 이내에 샘플의 이미지를 수집합니다. 커버 슬립을 밀봉하고 산화 속도를 감소시켜 플루오로포레 수명을 연장하기 위해 매니큐어의 여러 코트를 적용합니다.
참고: DAPI 함유 마운팅 매체를 사용하지 마십시오. 이미지를 수집할 때 나중에 정량화를 위해 각 이미지에 축척 막대를 포함합니다. 형광 비드는 슬라이드와 시료 제제11사이의 형광 강도의 대조군역할을 한다. 4단계에서 2차원(2D) 정량화를 위해 셀의 중간부에서 이미지를 수집합니다.
4. FISH 및 IF 이미지의 정량화하여 세포외 국소화를 강조하고 형광의 뉴클레오세포 비율을 결정합니다.
- 일관성을 보장하기 위해 다양한 형광 염색 및 병합 된 이미지의 조립 된 스택에서 이미지 분석을 수행합니다. 이미지를 수집할 때 포함된 축척 막대를 사용하여 이미지 분석 소프트웨어의 배율을 설정합니다.
- 여러 채널에서 핵 DAPI 얼룩을 참조하여 형광 강도를 정량화하려면 선 도구와 플롯 프로파일 함수를 사용합니다. 그런 다음 뷰어의 판단을 방해하거나 영향을 미치지 않는 마커를 사용하여 이미지 사본에 선을 영구적으로 표시합니다.
- 중앙 축을 따라 지형 피처, 봉우리 및 계곡의 다양성을 캡처하는 추적 또는 과포화 영역을 통과하지 않는 선과 같이 선이 그려지는 위치를 안내하는 기준을 설정합니다.
참고: 이 선 추적은 세포에 있는 원시 형광을 묘사하고 그러므로 얼룩의 위치의 비교로 제한됩니다, 강도가 아닙니다. 슬라이드, 트리트먼트 또는 제제 간에 동일한 얼룩의 강도를 비교하려면 3.13 단계에서 내부 제어로 슬라이드에 형광 비드를 추가합니다. 형광 비드는 장착 공정 중에 첨가되어야 하며 여기 레이저 및 광증선튜브(공초점)에서 동일한 설정으로 검출되어야 합니다.
- 중앙 축을 따라 지형 피처, 봉우리 및 계곡의 다양성을 캡처하는 추적 또는 과포화 영역을 통과하지 않는 선과 같이 선이 그려지는 위치를 안내하는 기준을 설정합니다.
- 세포 외 국소화의 변화를 정량화하려면 다른 치료를 받는 세포의 뉴클레오세포 비율을 계산합니다.
- 핵 DAPI 얼룩을 이용하여 핵 및 세포질 둘 다의 면적 및 원시 형광 강도를 측정하여 내부 경계를 설정한다. 핵 RNA(예를 들어, KSHV PAN RNA) 및 세포질 RNA(예를 들어, 호스트 GAPDH mRNA)와 같은 핵 및 세포질 조절을 포함한다. 또한 세 개의 셀과 같은 영역에 대한 배경 강도를 계산하고 픽셀 또는 μm2당값을 평균화합니다.
참고: 강도 값은 단위가 부족하여 '단위'라는 용어가 사용됩니다. - 먼저 동일한 영역에 대한 평균 배경을 결정한 다음 영역의 원시 강도에서 개별화된 값을 빼서 핵 및 셀룰러 원시 강도 값을 모두 정규화합니다.
- 예를 들어, 용리B 세포의 핵은 133.4 μm2의 면적과 75976 단위의 원시 강도를 가지며 동일한 형광 신호에 대한 배경 강도는 μm2당0.67 단위로 결정되었다. 정상화된 핵 강도는
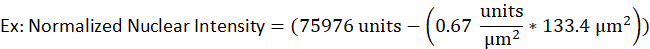
- 예를 들어, 용리B 세포의 핵은 133.4 μm2의 면적과 75976 단위의 원시 강도를 가지며 동일한 형광 신호에 대한 배경 강도는 μm2당0.67 단위로 결정되었다. 정상화된 핵 강도는
- 다음 방정식에 값을 입력합니다.

참고: 이 계산은 세포 외 영역의 변화에 대한 제어입니다. 용액 유도 및 약물 치료는 핵을 확대하거나 각각 세포의 크기를 변경할 수 있습니다. - 결과를 해석하려면 상자 수염 플롯을 만듭니다. 형광 신호의 균등 분포는 0에 가깝고 핵 분포는 양수 비율 값을 선호하고 세포질 분포는 음수 비율 값으로 추세입니다.
- 핵 DAPI 얼룩을 이용하여 핵 및 세포질 둘 다의 면적 및 원시 형광 강도를 측정하여 내부 경계를 설정한다. 핵 RNA(예를 들어, KSHV PAN RNA) 및 세포질 RNA(예를 들어, 호스트 GAPDH mRNA)와 같은 핵 및 세포질 조절을 포함한다. 또한 세 개의 셀과 같은 영역에 대한 배경 강도를 계산하고 픽셀 또는 μm2당값을 평균화합니다.
결과
이 원고에 자세히 설명된 FISH 및 IF 방법은 형광 강도의 선 자취에 의한 결과의 정량화와 함께 그림 1에 나와 있습니다. 여기에 제시된 결과는 반정적이며, 실험이 슬라이드 준비에 형광 비드를 포함하지 않았기 때문에 다른 형광 얼룩의 강도 사이의 비교보다는 지역화에 대한 통찰력을 제공합니다. 도 1은 또한 세포질 및 핵 영?...
토론
본 보고서에 기재된 프로토콜은 상이한 세포 유형에 적응될 수 있으며, 단일클론 및 폴리클로날 1차 항체를 모두 사용하는 경우와 함께 이중 RNA FISH 및 RNA FISH에 대한 단계를 포함한다. 준비된 슬라이드는 전형적으로 공초점 현미경으로 이미지화되지만, 증가된 항체 농도 및 상이한 장착 매체의 변형 후 STED(자극방출 고갈) 현미경으로 영상을 수행할 수 있다. 개별 세포의 향상된 분석을 위해, 이 프...
공개
저자는 공개할 이해상충이 없습니다.
감사의 말
조나단 로덴펠스, 카지미에츠 티코스키, 요한나 B. 위더스에게 데이터 분석에 대한 조언을 해주셔서 감사합니다. 우리는 또한 반대로 SSB 항체를 위한 G. Hayward 감사합니다. 이 작품은 국립 보건원 (TKV)과 NIH 교부금 (CA16038)에서 T32GM007223 및 T32AI05403 (JAS에)에 의해 지원되었습니다. JAS는 하워드 휴즈 의학 연구소의 조사자입니다. 그림 1-3 및 표 1은 다음 출판물에서 크리에이티브 커먼즈 저작자 표시 라이센스에 따라 미국 미생물학 협회의 허가를 받아 복제되었습니다: Vallery, T. K., 위더스, J. B., 안도, J. A., 스테이츠, J. A. 카포시의 육종 관련 핵 포시에 헤르페스 바이러스 mRNA 축적은 바이러스 DNA 복제 및 바이러스 비코딩 폴리아데닐화 핵 RNA에 의해 영향을 받습니다. 바이러스학의 전표. 92 (13), doi:10.1128/JVI.00220-18, (2018).
자료
| Name | Company | Catalog Number | Comments |
| AlexaFluor594-5-dUTP | Life Technologies | C1100 | |
| anti-DIG FITC | Jackson Lab Immunologicals | 200-092-156 | |
| Anti-Rabbit Secondary AlexaFluor594 Monoclonal Antibody | Invitrogen | A-11037 | Goat |
| Anti-SSB Antibody | N/A | N/A | Ref. Chiou et al. 2002 |
| BLASTn | NIH NCBI | N/A | Free Sequence Alignment Software |
| Dextran Sulfate | Sigma Aldrich | D8906 | Molecular Biology Grade |
| DIG-Oligonucleotide Tailing Kit | Sigma Roche | #03353583910 | 2nd Gen |
| Eight-Chamber Slides | Nunc Lab Tek II | #154453 | Blue seal promotes surface tension but separation by clear gel is also available. |
| Formamide | Sigma Aldrich | F9037 | Molecular Biology Grade |
| GAPDH Probes | Stellaris | SMF-2019-1 | Compatible with protocol, Quasar 670 |
| ImageJ | NIH, Bethesda, MD | N/A | Free Image Analysis Software, [http:rsb.info.nih.gov/ij/] |
| OligoAnalyzer | IDT | N/A | Free Oligonucleotide Analyzer |
| pcDNA3 | Invitrogen | A-150228 | |
| pmaxGFP | Amaxa | VDF-1012 | |
| Poly L-Lysine | Sigma Aldrich | P8920 | |
| Terminal Transferase | Sigma Roche | #003333574001 | |
| Vanadyl Ribonucleoside Complexes | NEB | S1402S | |
| Vectashield | Vector Laboratories, Inc. | H-1000 | DAPI within the mounting media scatters the light and reduces contrast. |
참고문헌
- Amen, M. A., Griffiths, A. Packaging of Non-Coding RNAs into Herpesvirus Virions: Comparisons to Coding RNAs. Frontiers in Genetics. 2, 81 (2011).
- Schmid, M., Speiseder, T., Dobner, T., Gonzalez, R. A. DNA virus replication compartments. Journal of Virology. 88 (3), 1404-1420 (2014).
- Pawlicki, J. M., Steitz, J. A. Primary microRNA transcript retention at sites of transcription leads to enhanced microRNA production. Journal of Cell Biology. 182 (1), 61-76 (2008).
- Borah, S., Darricarrere, N., Darnell, A., Myoung, J., Steitz, J. A. A viral nuclear noncoding RNA binds re-localized poly(A) binding protein and is required for late KSHV gene expression. Public Library of Science Pathogens. 7 (10), e1002300 (2011).
- Tycowski, K. T., Shu, M. D., Borah, S., Shi, M., Steitz, J. A. Conservation of a triple-helix-forming RNA stability element in noncoding and genomic RNAs of diverse viruses. Cell Reports. 2 (1), 26-32 (2012).
- Weinberg, R. A., Penman, S. Small molecular weight monodisperse nuclear RNA. Journal of Molecular Biology. 38 (3), 289-304 (1968).
- Myoung, J., Ganem, D. Generation of a doxycycline-inducible KSHV producer cell line of endothelial origin: maintenance of tight latency with efficient reactivation upon induction. Journal of Virology Methods. 174 (1-2), 12-21 (2011).
- Brulois, K. F., et al. Construction and manipulation of a new Kaposi's sarcoma-associated herpesvirus bacterial artificial chromosome clone. Journal of Virology. 86 (18), 9708-9720 (2012).
- Sturzl, M., Gaus, D., Dirks, W. G., Ganem, D., Jochmann, R. Kaposi's sarcoma-derived cell line SLK is not of endothelial origin, but is a contaminant from a known renal carcinoma cell line. International Journal of Cancer. 132 (8), 1954-1958 (2013).
- Chiou, C. J., et al. Patterns of gene expression and a transactivation function exhibited by the vGCR (ORF74) chemokine receptor protein of Kaposi's sarcoma-associated herpesvirus. Journal of Virology. 76 (7), 3421-3439 (2002).
- Cole, R. W., Jinadasa, T., Brown, C. M. Measuring and interpreting point spread functions to determine confocal microscope resolution and ensure quality control. Nature Protocols. 6 (12), 1929-1941 (2011).
- Nakamura, H., et al. Global changes in Kaposi's sarcoma-associated virus gene expression patterns following expression of a tetracycline-inducible Rta transactivator. Journal of Virology. 77 (7), 4205-4220 (2003).
- Majerciak, V., Yamanegi, K., Zheng, Z. M. Gene structure and expression of Kaposi's sarcoma-associated herpesvirus ORF56, ORF57, ORF58, and ORF59. Journal of Virology. 80 (24), 11968-11981 (2006).
- Sun, R., Lin, S. F., Gradoville, L., Miller, G. Polyadenylylated nuclear RNA encoded by Kaposi sarcoma-associated herpesvirus. Proceedings of the National Academy Sciences U S A. 93 (21), 11883-11888 (1996).
- Vallery, T. K., Withers, J. B., Andoh, J. A., Steitz, J. A. Kaposi's Sarcoma-Associated Herpesvirus mRNA Accumulation in Nuclear Foci Is Influenced by Viral DNA Replication and Viral Noncoding Polyadenylated Nuclear RNA. Journal of Virology. 92 (13), (2018).
- Borah, S., Nichols, L. A., Hassman, L. M., Kedes, D. H., Steitz, J. A. Tracking expression and subcellular localization of RNA and protein species using high-throughput single cell imaging flow cytometry. RNA. 18 (8), 1573-1579 (2012).
- Bruce, A. G., et al. Quantitative Analysis of the KSHV Transcriptome Following Primary Infection of Blood and Lymphatic Endothelial Cells. Pathogens. 6 (1), (2017).
- Chen, C. P., et al. Kaposi's Sarcoma-Associated Herpesvirus Hijacks RNA Polymerase II To Create a Viral Transcriptional Factory. Journal of Virology. 91 (11), (2017).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유