É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Hibridação in situ da fluorescência quantitativa (peixes) e immunofluorescence (se) de produtos específicos do gene em pilhas KSHV-infectadas
Neste Artigo
Resumo
Nós descrevemos um protocolo que utiliza a hibridação in situ da fluorescência (peixes) para visualizar RNAs herpética múltiplas dentro das pilhas humanas liticamente contaminadas, na suspensão ou no aderente. Este protocolo inclui a quantificação da fluorescência produzindo uma relação Mimivírus e pode ser estendido para o visualização simultâneo do anfitrião e das proteínas virais com imunofluorescência (se).
Resumo
A introspecção mechanistic chega do estudo cuidadoso e da quantificação de RNAs e de proteínas específicos. As posições relativas destas biomoléculas durante todo a pilha em épocas específicas podem ser capturadas com a hibridação in situ da fluorescência (peixes) e a imunofluorescência (se). Durante a infecção por herpesvírus lítico, o vírus seqüestra a célula hospedeira para expressar preferencialmente genes virais, causando alterações na morfologia celular e no comportamento das biomoléculas. As atividades líticas são centradas em fábricas nucleares, denominadas compartimentos de replicação viral, que são perceptível apenas com FISH e IF. Aqui nós descrevemos um protocolo adaptável de peixes do RNA e se as técnicas para Kaposi ' s sarcoma-associou o Herpesvirus (KSHV)-pilhas infectadas, aderente e na suspensão. O método inclui etapas para o desenvolvimento de oligonucleotides antisense específicos, peixes dobro do RNA, peixes do RNA com se, e cálculos quantitativos de intensidades da fluorescência. Este protocolo foi aplicado com sucesso aos tipos múltiplos da pilha, às pilhas não infectadas, às pilhas latentes, às pilhas Lytic, aos tempo-cursos, e às pilhas tratadas com os nervos inibidores para analisar as atividades armazenamento de RNAs e de proteínas específicas do anfitrião humano e KSHV.
Introdução
Em sua fase lítica (ativa), os herpesvírus sequestram a célula hospedeira, causando alterações na morfologia celular e localização de moléculas biológicas, para produzir virions. A base das operações é o núcleo, onde o genoma viral do DNA duplo-encalhado é replicado e empacotado em um escudo da proteína, chamado um capsídeo1. Para começar, o vírus expressa suas próprias proteínas, seqüestro máquinas hospedeiras e impedindo a expressão de genes de acolhimento não essenciais, um processo denominado o efeito de desligamento do hospedeiro. A maioria desta atividade é localizada a 4 ′, 6-diamidino-2-phenylindole (DAPI)-regiões nucleares livres chamadas compartimentos virais da replicação, compreendidos das proteínas do anfitrião e do viral, do RNAs, e do ADN viral2. A célula é revisado para fornecer espaço e recursos para os compartimentos de replicação e, portanto, montagem de capsids virais. Uma vez que o capsídeo sai do núcleo, como o capsídeo é envolvido no citoplasma para produzir uma partícula viral ligada à membrana, também conhecida como um virion, não é clara. A compreensão da localização e dos deslocamentos espaciais de ambos os hospedeiros e biomoléculas virais durante a fase lítica fornece uma introspecção mecanicista mais profunda no arranjo do compartimento da duplicação, do efeito do desligamento do anfitrião, da via do virion-saída, e do outro processos relacionados à infecção herpética e à replicação.
Atualmente, o melhor método para detectar e estudar essas alterações é a visualização de proteínas e RNAs em células infectadas com imunofluorescência (IF) e hibridação in situ fluorescente (FISH), respectivamente. O uso de um curso de tempo com essas técnicas revela a localização de biomoléculas em pontos-chave da fase lítica ou simplesmente, dados espaciotemporais. FISH e IF complementam outras técnicas bioquímicas, como a inibição de um processo celular (por exemplo, inibição da replicação viral do DNA), RT-qPCR (reação em cadeia da polimerase em tempo real), sequenciamento de RNA, manchas do Norte, espectrometria de massas, western blotting e análise da produção de DNA viral, que pode fornecer uma imagem mais global das atividades celulares.
Nós desenvolvemos estratégias do RNA Fish para examinar os produtos do RNA dos genes específicos e uma análise computacional que calcule quantitativamente a relação Mimivírus de um produto específico do gene. A preparação da amostra, modificada a partir de publicações anteriores por Steitz e colegas3,4, érelativamente fácil e pode ser usada tanto para as células aderentes como as suspensas. O protocolo também é adaptável para o uso simultâneo de múltiplas estratégias de RNA Fish (duplo RNA FISH) ou RNA FISH com estratégias IF. O desenvolvimento de uma estratégia específica de FISH é desafiador, mas as sugestões para melhorar o sucesso são delineadas. A análise de dados descrita aqui é quantitativa se os grânulos fluorescentes e os marcadores fortes de limites do compartimento são usados e oferecem a introspecção adicional nas micrografias, introspecção que remove o viés da observação. O protocolo detalhado é projetado para as pilhas latentes e Lytic contaminadas por Kaposi ' o Herpesvirus sarcoma-associado de s (KSHV) e pode ser usado com as pilhas ou as pilhas não contaminadas por outros Herpesvirus5. Os métodos de quantificação são aplicáveis a estudos sobre turnos nucleocitímicos ou relocalização entre compartimentos subcelulares na maioria das células.
Protocolo
1. projeto de oligonucleotídeos antisense in situ da fluorescência (peixes) para detectar um transcrito herpética específico
- Selecione 25 a 40 segmentos do NT da seqüência do RNA do interesse e converta-o para ser antisense. Uma estratégia bem sucedida dos peixes pode conter de um até dez ou mais oligonucleotides antisense diferentes. Ao selecionar sequências, considere o seguinte:
- Se o RNA do interesse contem uma região original da repetição, a seguir capitalizar nesta característica e projete um oligonucleotide antisense para alvejar a seqüência da repetição.
Nota: tycowski e os colegas5 fornecem um exemplo desta estratégia com Rhesus rhadinovirus (RRV) o RNA nuclear poliadenilado (Pan). - Se o RNA do interesse contiver um local proteína-obrigatório conhecido ou uma estrutura do haste-laço, projete oligonucleotídeos que evitam estas regiões.
- Dependendo dos objetivos das experiências, considere a seqüência intronic e se ou não projetar um oligonucleotide antisense para ele.
- Se o RNA do interesse contem uma região original da repetição, a seguir capitalizar nesta característica e projete um oligonucleotide antisense para alvejar a seqüência da repetição.
- Realize análises computacionais simples nas sequências antisense selecionadas para garantir a especificidade de ligação e reduzir a agregação do oligonucleotídeo antisense.
- As sequências devem ser aproximadamente 50% GC-Rich (alto teor de guanina e citosina) e têm uma temperatura de fusão na faixa de 60 a 70 ° c.
- Use uma ferramenta de analisador de sequência para selecionar sequências que não se autodimerizam ou formam grampos com temperaturas de fusão acima de 37 ° c, a temperatura de hibridação.
- Realize um NCBI BLASTn (National Center for Biotechnology Information Basic local alinhamento Search Tool para alinhamentos de nucleotídeo) busca das sequências selecionadas contra o hospedeiro e transcriptomos virais usando a configuração ' um pouco semelhante '. Esta pesquisa identificará os oligonucleotídeos antisense originais que não associarão provavelmente a outros hospedeiros ou transcritos virais.
Nota: se os transcriptomas não estiverem disponíveis, realize a pesquisa BLAST com as sequências genómicas. É ideal se as buscas são executadas nas seqüências do vírus isolado das pilhas contaminadas usadas no experimento porque as tensões selvagens tendem a diversificar e conter uma combinação de seqüências das tensões diferentes do laboratório.
- Ordem purificada DNA oligonucleotídeos correspondente à seqüência de anti-Sense que foi verificado computacionalmente para ser único e susceptível de se ligar ao RNA alvo. Nenhuma modificação especial precisa de ser introduzida nos oligonucleotides.
- Teste os oligonucleotídeos antisense projetados para a especificidade obrigatória pelo peixe e pelo Borrão do Norte.
- Usando uma célula-linha não infectada a partir da mesma espécie hospedeira (por exemplo, 293T) e idealmente o mesmo tipo de célula, realizar uma transfecção com um plasmídeo expressando o RNA de interesse de um promotor robusto (CMV, Citomegalovírus) e um com o vetor vazio (por exemplo, pcDNA3). Use um controle positivo para o transfection tal como o co-transfection com um plasmídeo de GFP (proteína fluorescente verde) (por exemplo, pmaxGFP) ou o vetor que contem GFP.
Nota: é importante evitar as células-linhas que foram imortalizadas usando o vírus de Epstein-Barr do Herpesvirus (EBV) desde que há umas semelhanças da seqüência entre Herpesvirus. - Realize os peixes como descrito na seção 3 em ambos os jogos das pilhas com os oligonucleotídeos antisense como descritos neste protocolo. Deduce os candidatos bem sucedidos, comparando experimentos com peixes individuais, pares, ou conjuntos de oligonucleotides anti-Sense. Use um controle positivo para o protocolo FISH como o snRNA U2 (RNA nuclear pequeno) FISH, presente em 500.000 exemplares por núcleo de células humanas6 (tabela 1).
- O sinal fluorescente deve ser específico e forte na célula que contém o RNA de interesse. Projete oligonucleotídeos antisense adicionais para fortalecer o sinal e remover oligonucleotídeos antisense que ligam nonespecificamente da consideração. A intensidade do sinal deve estar acima do fundo e autofluorescência.
- Teste de especificidade de ligação por Northern blot.
- Usando uma célula-linha não infectada a partir da mesma espécie hospedeira (por exemplo, 293T) e idealmente o mesmo tipo de célula, realizar uma transfecção com um plasmídeo expressando o RNA de interesse de um promotor robusto (CMV, Citomegalovírus) e um com o vetor vazio (por exemplo, pcDNA3). Use um controle positivo para o transfection tal como o co-transfection com um plasmídeo de GFP (proteína fluorescente verde) (por exemplo, pmaxGFP) ou o vetor que contem GFP.
2. oligonucleotídeo e preparação de células
- Seguindo as instruções do fabricante, use o terminal transferase para etiquetar os oligonucleotídeos antisense com dioxigenin (escavação)-dUTP ou, se fortemente encadernação, diretamente com um nucleotide fluorescente como o Alexa fluor 594-5-dUTP. Após a rotulagem, a purificação adicional não é necessária. Armazene oligonucleotídeos etiquetados em-20 ° c até diversos anos e na folha de estanho se etiquetado diretamente para impedir o photobranqueamento.
PRECAUÇÃO: a solução de rotulagem contém um material tóxico, cacodylate de potássio. Lidar com reações de rotulagem com luvas.
Nota: para conservar recursos, diversos oligonucleotídeos antisentidos diferentes podem ser etiquetados em uma reação. Este protocolo utiliza 3 '-End rotulagem. A rotulagem interna é desafiante porque o grupo químico (por exemplo, DIG ou o fluorophore) começ travado ou é incapaz de incorporar o local ativo de um polymerase do ADN. Um experimento com duas RNAs diferentes pode ser realizado usando oligonucleotídeos antisense diretamente rotulados e imunofluorescência anti-Dig com um fluoróforo diferente (por exemplo, FITC (Fluorescein) ou Alexa fluor 488 com Alexa fluor 594). - Adira as células aos slides de oito câmaras.
Nota: as corrediças da oito-Câmara permitem diversos experimentos simultâneos ao minimizar recursos preciosos como anticorpos. Uma alternativa às corrediças da oito-câmara é uma placa da cultura do tecido de seis poços com os COVERSLIP padrão (22 milímetros x 22 milímetros) que são ambos estéreis. Um arranjo similar é possível com COVERSLIP circulares e uma placa da cultura do tecido 24-well. Para ambos, aumente os volumes mencionados neste protocolo por 10-15x (por exemplo, solução da hibridação de 1,75 mL), e 4x (por exemplo, solução da hibridação de 600 μL) respectivamente.- Para células líticas aderentes, use 1x Trypsin/PBS a 37 ° c e 5% CO2 por 10 min para suspender as células e diluir para 60% de confluência.
Nota: as linhagens celulares aderentes utilizadas em experimentos com FISH incluíram 293T, iSLK. 2197e células islk-BAC368. - Aplique 200 μL de suspensão celular em cada câmara das lâminas estéreis de oito câmaras e permita o crescimento das sementes para 12-24 h a 37 ° c e 5% CO2. Ajuste conforme necessário para células de crescimento lento ou rápido e para células que são facilmente danificadas pela tripsina.
Observação: o objetivo é ter células uniformemente espaçadas firmemente conectadas ao slide. Considere induzir a fase lítica após a adesão se as células líticas são frágeis. As conclusões extraídas das experiências com pilhas de iSLK são limitadas9. - Para suspensão-células líticas, pré-tratar oito-câmara desliza com 1:10 poli L-lisina por 5 min a cultura do tecido Hood. Em seguida, deixe as lâminas para secar durante a noite à temperatura ambiente ou 1 h a 65 ° c. Incubar 800 μL de células líticas numa concentração de 1 x 106 células/ml com as lâminas de câmara durante 30 min a 1 h a 37 ° c e 5% co2.
Nota: as células de suspensão se instalarão em uma monocamada, aderindo à L-lisina poli e, portanto, as células em excesso não são uma preocupação em comparação com as células aderentes. Células líticas que formam aglomerados de uva, se possível, devem ser separadas por vortexing suave ou meios químicos. Reconhecidamente, os autores não tiveram muito sucesso com tais recomendações no caso das células Lytic BJAB-RRV-GFP. Se as células de suspensão não aderirem bem, considere aumentar o tempo ou a concentração da incubação de L-lisina poli.
- Para células líticas aderentes, use 1x Trypsin/PBS a 37 ° c e 5% CO2 por 10 min para suspender as células e diluir para 60% de confluência.
3. fixação, imunofluorescência (opcional), hibridação, e visualização de RNAs virais
- Remova a mídia e as células em excesso. Ao longo deste protocolo, use sucção a vácuo para remover soluções e micropipetagem suave para adicionar soluções.
Nota: a força de um vácuo pode ser reduzida colocando uma ponta de micropipeta de 200 μL sobre a pipeta Pasteur de vidro. Substitua a ponta da micropipeta entre as etapas de lavagem para evitar a contaminação. Cada etapa da lavagem deve ser executada rapidamente porque é imperativo que as pilhas nunca secam. - Imediatamente, fixar as células com pré-refrigerados 4% formaldeído/PBS (fosfato-tampão salina) no gelo por 30 min. Lave as células três vezes com 200 μL 1X PBS refrigerado a 4 ° c e incubar por 5 min à temperatura ambiente ou no gelo.
- Permeabilize as células fixas com 200 μL de pré-refrigerados 0,5% Triton-X/PBS (salina tamponada por fosfato) por 10 min no gelo ou 750 μL de etanol pré-70 resfriado a 4 ° c por 1 h (min) a 7 d (máx.).
Nota: colete proteínas, RNA total e amostras de DNA genômico no ponto de fixação para garantir a consistência entre as imagens e os ensaios bioquímicos. Todas as lavas ao longo deste protocolo são executadas da mesma maneira, a menos que especificado de outra forma. 70% etanol solta a cola entre as câmaras e o slide, o que facilita a separação posterior, e também fornece uma pausa significativa no protocolo. No entanto, use película de parafina em torno da corrediça da câmara para reduzir a evaporação e verificar o nível do etanol em cada câmara sobre cada 8 h. 70% etanol também aplaina as células, fazendo uma imagem mais nítida, enquanto Triton-X não desidrata as células e alterar o dimensões da célula.
- Permeabilize as células fixas com 200 μL de pré-refrigerados 0,5% Triton-X/PBS (salina tamponada por fosfato) por 10 min no gelo ou 750 μL de etanol pré-70 resfriado a 4 ° c por 1 h (min) a 7 d (máx.).
- Retire as câmaras cuidadosamente para evitar rachaduras no slide. Se o experimento incluir imunofluorescência (IF) de uma proteína viral ou hospedeira com um anticorpo primário policlononal, realize o IF como descrito abaixo antes de prosseguir para o RNA FISH. Se a imunofluorescência usa um anticorpo preliminar monoclonal, a seguir execute a imunofluorescência como descrito na etapa 3.3.1 após a etapa 3,11.
Nota: Use um dispositivo fresco da remoção ou um com o adesivo restante muito pequeno fornecido pelo fabricante e facilitem delicadamente as câmaras fora para impedir que a corrediça quebre. Usando 70% de etanol como o reagente de permeabilização para 4 h reduz consideravelmente a probabilidade de rachaduras. No caso de uma rachadura, continue o protocolo em câmaras não afetadas pela rachadura e seja consciente da taxa de oxidação mais elevada de corrediças imperfeitamente seladas (isto é a vida de armazenamento diminuída).- Lave as células com 1X PBS pré-refrigerado e bloqueie com 4% de BSA pré-refrigerada (albumina sérica bovina)/1X PBS por 30 min a 4 ° c.
Nota: o uso de BSA ao longo deste protocolo limita a rotulagem não específica. - Remover a solução de bloqueio e incubar as células com 1:200 ou outro anticorpo primário policlononal em 0,1% BSA/1X PBS por 1 h a 4 ° c. Em seguida, lave três vezes com 1X PBS.
Nota: um anticorpo10 para a deteção de SSB/ORF6 (proteína de ligação de ADN single-encalhado viral) foi usado em 1:200 diluição. - Incubar as células com um anticorpo secundário com fluoróforo compatível com o anticorpo detectando peixes por 1 h a 4 ° c. Lave três vezes com 1X PBS. Em seguida, fixar com 4% formaldeído/1X PBS para 10-15 min e Permeabilize com tanto Triton-X ou 70% etanol como descrito anteriormente antes de prosseguir para FISH. Deslize a tampa com folha de estanho para preservar o sinal fluorescente e evitar o fotobranqueamento.
- Lave as células com 1X PBS pré-refrigerado e bloqueie com 4% de BSA pré-refrigerada (albumina sérica bovina)/1X PBS por 30 min a 4 ° c.
- Lave as células com 2x SSC (citrato de sódio fisiológico) uma vez e aplique 45 μL de solução de hibridação consistindo de 50% de formamida, 10% de sulfato de dextrano, 2x SSC, 0,1% BSA, 500 μg/mL de DNA de esperma de salmão, 125 μg/mL e. coli tRNA e 1 mm de ribonucleosídeo Complexos. Incubar por 1 h a 37 ° c em uma câmara de humidade que pode ser um prato de Petri de 150 mm com Toalhetes esterilizados umedecidos.
Nota: Prepare uma solução de hibridação fresca pelo menos uma hora antes do uso. Dissolva o sulfato de dextrano na água primeiramente, vortexing freqüentemente e incubando em um banho de água de 37 ° c. - Calcule para ter uma concentração sugerida de 25 μm oligonucleotídeos na solução da hibridação 35-UL por a câmara. Ajuste a concentração do oligonucleotide antisense como necessário. Adicione a água destilada aos oligonucleotídeos para trazer o volume da desnaturação a 10 μL.
Nota: após a reação de rotulagem, os oligonucleotídeos são armazenados na solução apagada contendo 0,18 M de cacodylate de potássio, 23 mM Tris-HCl, 0,23 mg/mL BSA, 4,5 mM CoCl2, 18 mm EDTA, 2,7 mm K-fosfato, e 6,8 mm kcl, 45 μm 2-Mercaptoetanol, 0, 2% Triton X-100, e 2% glicerol. As concentrações são suficientemente elevadas para que a diluição com água traga a solução de desnaturação para concentrações próximas de 1x TE (10 mM de Tris-HCl e 1 mM de EDTA), um tampão de desnaturação de oligonucleotídeo padrão. - Desnaturar o Dig-e/ou Alexa fluor 594-rotulados oligonucleotídeos em 95 ° c por 5 min. Em seguida, adicione 35 μL de solução de hibridação fresca por câmara pretendida aos oligonucleotídeos desnaturados. Se a realização de duplo peixe, ambos os conjuntos de anti-Sense oligonucleotídeos pode ser desnaturado e hibridizados juntos.
- Remova a solução da pré-hibridação e adicione então a solução da hibridação que contem os oligonucleotídeos etiquetados às pilhas. Incubar durante a noite na câmara da umidade em 37 ° c com folha de estanho para proteger os oligonucleotides fluorophore-etiquetados.
Nota: incubação deve ser pelo menos 10 h e não mais do que 24 h. - No dia seguinte, lave as células duas vezes com 2x SSC por 10 min a 37 ° c e, em seguida, duas vezes com 1x SSC por 10 min a 25 ° c.
- Fixar as células com pre-refrigerado 4% formaldeído/1X PBS para 10-15 min no gelo. Em seguida, lave as células com PBS três vezes e permeabilize para 1 h com pré-refrigerados 70% etanol ou por 10 min com pré-refrigerados 0,5% Triton-X/1X PBS a 4 ° c.
- Incubar as células com 1:200 anti-DIG FITC na pré-refrigerada 0,1% BSA/1X PBS por 1 h a 4 ° c. Retire a solução de anticorpos e lave três vezes com 1X PBS.
- Fixar com pre-refrigerado 4% formaldeído/1X PBS para 10-15 min a 4 ° c e depois lavar três vezes com 1X PBS. Se a realização de imunofluorescência para um hospedeiro ou proteína viral com um anticorpo primário monoclonal, permeabilize as células e, em seguida, executar o protocolo IF descrito na etapa 3.3.1. Caso contrário, proceder à coloração DAPI.
- Incubar as células com 0,4 μg/mL de DAPI no pré-resfriado 0,5% Triton-X/1X PBS por 15 min no gelo e depois lave três vezes com 1X PBS.
- Monte corrediças com grânulos fluorescentes (opcionais) e um meio de montagem. Em seguida, selar a lamínula para o slide com verniz de unha claro.
- Usando um microscópio confocal, colete imagens das amostras dentro de uma hora a uma semana de executar o protocolo na ampliação 630x. Aplique várias camadas de unha polonês para selar o deslizamento da tampa e para prolongar a vida fluoróforo, reduzindo a taxa de oxidação.
Observação: não use um meio de montagem contendo DAPI. Ao coletar as imagens, inclua a barra de escala em cada imagem para posterior quantificação. Os grânulos fluorescentes servem como controles da intensidade da fluorescência entre corrediças e preparações da amostra11. Adquira imagens na seção central da célula para a quantificação bidimensional (2D) na etapa 4.
4. quantificação de imagens de Fish e If para destacar a localização subcelular e para determinar a razão Mimivírus da fluorescência
- Execute a análise de imagem em uma pilha montada das várias imagens fluorescentes-manchadas e fundidas para assegurar a consistência. Defina a escala do software de análise de imagem usando a barra de escala incluída quando as imagens foram coletadas.
- Para quantificar a intensidade de fluorescência em vários canais e em referência à mancha nuclear de DAPI, use uma ferramenta de linha e uma função de perfil de plotagem. Em seguida, indique a linha permanentemente em uma cópia da imagem usando marcadores que não obstruem ou influenciem o julgamento do espectador.
- Estabeleça critérios para orientar onde a linha é desenhada, como um traço que capta uma diversidade de características topográficas, picos e vales, ao longo de um eixo central ou uma linha que não atravessa áreas supersaturadas.
Nota: Estes traços da linha descrevem a fluorescência crua em uma pilha e são limitados assim às comparações das posições de uma mancha, não de intensidade. Para comparar intensidades da mesma mancha entre corrediças, tratamentos, ou preparações, adicione um grânulo fluorescente à corrediça como um controle interno durante a etapa 3,13. O cordão fluorescente deve ser adicionado durante o processo de montagem e detectado com as mesmas configurações no laser de excitação e tubo fotomultiplicador (confocal).
- Estabeleça critérios para orientar onde a linha é desenhada, como um traço que capta uma diversidade de características topográficas, picos e vales, ao longo de um eixo central ou uma linha que não atravessa áreas supersaturadas.
- Para quantificar uma mudança na localização subcelular, calcule os rácios Mimivírus das pilhas que submetem-se aos tratamentos diferentes.
- Meça a área e a intensidade crua da fluorescência do núcleo e do citoplasma usando a mancha nuclear de DAPI para ajustar o limite interno. Inclua controles nucleares e citoplasmáticos, como um RNA nuclear (por exemplo, RNA PAN KSHV) e RNA citoplasmático (por exemplo, hospedeiro GAPDH mRNA). Além disso, calcule a intensidade de fundo para três áreas semelhantes a células e a média dos valores por pixel ou μm2.
Observação: os valores de intensidade tendem a faltar unidades e, portanto, o termo "unidades" é usado. - Normalize os valores de intensidade bruta nuclear e celular determinando primeiro o fundo médio para a mesma área e, em seguida, subtraindo esse valor individualizado da intensidade bruta da área.
- Por exemplo, um núcleo de uma célula B lítica tem uma área de 133,4 μm2 e uma intensidade bruta de 75976 unidades, enquanto a intensidade de fundo para o mesmo sinal fluorescente foi determinada como sendo 0,67 unidades por μm2. A intensidade nuclear normalizada seria
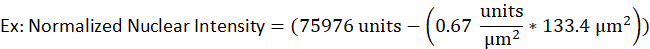
- Por exemplo, um núcleo de uma célula B lítica tem uma área de 133,4 μm2 e uma intensidade bruta de 75976 unidades, enquanto a intensidade de fundo para o mesmo sinal fluorescente foi determinada como sendo 0,67 unidades por μm2. A intensidade nuclear normalizada seria
- Insira os valores na equação a seguir.

Nota: este cálculo controla as alterações na área subcelular. A indução lítica e os tratamentos medicamentoso podem ampliar o núcleo ou alterar o tamanho da célula, respectivamente. - Para interpretar os resultados, crie um gráfico de Whisker de caixa. Uma distribuição igual do sinal fluorescente seria próxima de zero, enquanto que uma distribuição nuclear favoreceria um valor positivo da relação e uma distribuição cytoplasmic tenderia para um valor negativo da relação.
- Meça a área e a intensidade crua da fluorescência do núcleo e do citoplasma usando a mancha nuclear de DAPI para ajustar o limite interno. Inclua controles nucleares e citoplasmáticos, como um RNA nuclear (por exemplo, RNA PAN KSHV) e RNA citoplasmático (por exemplo, hospedeiro GAPDH mRNA). Além disso, calcule a intensidade de fundo para três áreas semelhantes a células e a média dos valores por pixel ou μm2.
Resultados
Os métodos FISH e IF detalhados neste manuscrito são mostrados na Figura 1 , juntamente com a quantificação dos resultados por traços de linha de intensidade fluorescente. Os resultados aqui apresentados são semiquantitativos e oferecem insight sobre a localização, em vez de comparações entre intensidades de diferentes manchas fluorescentes, pois experimentos não incluem um cordão fluorescente na preparação da lâmina. A Fig...
Discussão
O protocolo descrito neste relatório pode ser adaptado aos tipos diferentes da pilha e inclui etapas para peixes dobro do RNA e peixes do RNA com o se que usa anticorpos preliminares monoclonais e polyclonal. Embora as corrediças preparadas sejam imaged tipicamente com um microscópio confocal, a imagem latente pode ser executada com um microscópio de STED (depleção de emissão estimulada) após modificações da concentração aumentada do anticorpo e de um meio de montagem diferente. Para análise aprimorada de c?...
Divulgações
Os autores não têm conflitos de interesse para divulgar.
Agradecimentos
Agradecemos a Jonathan Rodenfels, Kazimierz Tycowski e Johanna B. Withers para aconselhamento sobre análise de dados. Agradecemos também ao G. Hayward pelo anticorpo anti-SSB. Este trabalho foi apoiado por subsídios T32GM007223 e T32AI055403 dos institutos nacionais de saúde (TKV) e concessão de NIH (CA16038) (para JAS). JAS é um investigador do Howard Hughes Medical Institute. As figuras 1-3 e a tabela 1 foram reproduzidas com a permissão da sociedade americana para microbiologia uma licença Creative Commons Attribution da seguinte publicação: Vallery, T. K., Withers, J. B., Andoh, J. A., Steitz, J. A. Kaposi ' s sarcoma-Associated A acumulação de mRNA do Herpesvirus em focos nucleares é influenciada pela replicação viral do ADN e pelo RNA nuclear Polyadenylated noncoding viral. Jornal de Virology. 92 (13), doi: 10.1128/jvi. 00220-18, (2018).
Materiais
| Name | Company | Catalog Number | Comments |
| AlexaFluor594-5-dUTP | Life Technologies | C1100 | |
| anti-DIG FITC | Jackson Lab Immunologicals | 200-092-156 | |
| Anti-Rabbit Secondary AlexaFluor594 Monoclonal Antibody | Invitrogen | A-11037 | Goat |
| Anti-SSB Antibody | N/A | N/A | Ref. Chiou et al. 2002 |
| BLASTn | NIH NCBI | N/A | Free Sequence Alignment Software |
| Dextran Sulfate | Sigma Aldrich | D8906 | Molecular Biology Grade |
| DIG-Oligonucleotide Tailing Kit | Sigma Roche | #03353583910 | 2nd Gen |
| Eight-Chamber Slides | Nunc Lab Tek II | #154453 | Blue seal promotes surface tension but separation by clear gel is also available. |
| Formamide | Sigma Aldrich | F9037 | Molecular Biology Grade |
| GAPDH Probes | Stellaris | SMF-2019-1 | Compatible with protocol, Quasar 670 |
| ImageJ | NIH, Bethesda, MD | N/A | Free Image Analysis Software, [http:rsb.info.nih.gov/ij/] |
| OligoAnalyzer | IDT | N/A | Free Oligonucleotide Analyzer |
| pcDNA3 | Invitrogen | A-150228 | |
| pmaxGFP | Amaxa | VDF-1012 | |
| Poly L-Lysine | Sigma Aldrich | P8920 | |
| Terminal Transferase | Sigma Roche | #003333574001 | |
| Vanadyl Ribonucleoside Complexes | NEB | S1402S | |
| Vectashield | Vector Laboratories, Inc. | H-1000 | DAPI within the mounting media scatters the light and reduces contrast. |
Referências
- Amen, M. A., Griffiths, A. Packaging of Non-Coding RNAs into Herpesvirus Virions: Comparisons to Coding RNAs. Frontiers in Genetics. 2, 81 (2011).
- Schmid, M., Speiseder, T., Dobner, T., Gonzalez, R. A. DNA virus replication compartments. Journal of Virology. 88 (3), 1404-1420 (2014).
- Pawlicki, J. M., Steitz, J. A. Primary microRNA transcript retention at sites of transcription leads to enhanced microRNA production. Journal of Cell Biology. 182 (1), 61-76 (2008).
- Borah, S., Darricarrere, N., Darnell, A., Myoung, J., Steitz, J. A. A viral nuclear noncoding RNA binds re-localized poly(A) binding protein and is required for late KSHV gene expression. Public Library of Science Pathogens. 7 (10), e1002300 (2011).
- Tycowski, K. T., Shu, M. D., Borah, S., Shi, M., Steitz, J. A. Conservation of a triple-helix-forming RNA stability element in noncoding and genomic RNAs of diverse viruses. Cell Reports. 2 (1), 26-32 (2012).
- Weinberg, R. A., Penman, S. Small molecular weight monodisperse nuclear RNA. Journal of Molecular Biology. 38 (3), 289-304 (1968).
- Myoung, J., Ganem, D. Generation of a doxycycline-inducible KSHV producer cell line of endothelial origin: maintenance of tight latency with efficient reactivation upon induction. Journal of Virology Methods. 174 (1-2), 12-21 (2011).
- Brulois, K. F., et al. Construction and manipulation of a new Kaposi's sarcoma-associated herpesvirus bacterial artificial chromosome clone. Journal of Virology. 86 (18), 9708-9720 (2012).
- Sturzl, M., Gaus, D., Dirks, W. G., Ganem, D., Jochmann, R. Kaposi's sarcoma-derived cell line SLK is not of endothelial origin, but is a contaminant from a known renal carcinoma cell line. International Journal of Cancer. 132 (8), 1954-1958 (2013).
- Chiou, C. J., et al. Patterns of gene expression and a transactivation function exhibited by the vGCR (ORF74) chemokine receptor protein of Kaposi's sarcoma-associated herpesvirus. Journal of Virology. 76 (7), 3421-3439 (2002).
- Cole, R. W., Jinadasa, T., Brown, C. M. Measuring and interpreting point spread functions to determine confocal microscope resolution and ensure quality control. Nature Protocols. 6 (12), 1929-1941 (2011).
- Nakamura, H., et al. Global changes in Kaposi's sarcoma-associated virus gene expression patterns following expression of a tetracycline-inducible Rta transactivator. Journal of Virology. 77 (7), 4205-4220 (2003).
- Majerciak, V., Yamanegi, K., Zheng, Z. M. Gene structure and expression of Kaposi's sarcoma-associated herpesvirus ORF56, ORF57, ORF58, and ORF59. Journal of Virology. 80 (24), 11968-11981 (2006).
- Sun, R., Lin, S. F., Gradoville, L., Miller, G. Polyadenylylated nuclear RNA encoded by Kaposi sarcoma-associated herpesvirus. Proceedings of the National Academy Sciences U S A. 93 (21), 11883-11888 (1996).
- Vallery, T. K., Withers, J. B., Andoh, J. A., Steitz, J. A. Kaposi's Sarcoma-Associated Herpesvirus mRNA Accumulation in Nuclear Foci Is Influenced by Viral DNA Replication and Viral Noncoding Polyadenylated Nuclear RNA. Journal of Virology. 92 (13), (2018).
- Borah, S., Nichols, L. A., Hassman, L. M., Kedes, D. H., Steitz, J. A. Tracking expression and subcellular localization of RNA and protein species using high-throughput single cell imaging flow cytometry. RNA. 18 (8), 1573-1579 (2012).
- Bruce, A. G., et al. Quantitative Analysis of the KSHV Transcriptome Following Primary Infection of Blood and Lymphatic Endothelial Cells. Pathogens. 6 (1), (2017).
- Chen, C. P., et al. Kaposi's Sarcoma-Associated Herpesvirus Hijacks RNA Polymerase II To Create a Viral Transcriptional Factory. Journal of Virology. 91 (11), (2017).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados