Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
KSHV Enfekte Hücrelerde Spesifik Gen Ürünlerinin Situ Hibridizasyonunda (FISH) ve İmmünfloresansında (IF) Kantitatif Floresan
Bu Makalede
Özet
Biz situ hibridizasyon floresan kullanan bir protokol tarif (FISH) lytically enfekte insan hücrelerinin içinde birden fazla herpesviral RNA görselleştirmek için, süspansiyon veya yapışık. Bu protokol, nükleositoplazmik oran üreten floresan ların niceliğini içerir ve konak ve viral proteinlerin immünoresans (IF) ile eşzamanlı olarak görüntülenmesi için uzatılabilir.
Özet
Mekanistik içgörü dikkatli çalışma ve belirli RNA ve proteinlerin niceliksel gelir. Bu biyomoleküllerin belirli zamanlarda hücre boyunca göreceli konumları, yerinde hibridizasyon (FISH) ve immünofloresan (IF) floresan ile yakalanabilir. Litik herpesvirüs enfeksiyonu sırasında, virüs tercihen viral genleri ifade etmek için konak hücre kaçırArak hücre morfolojisi ve biyomoleküllerin davranış değişikliklerine neden. Litik faaliyetler nükleer fabrikalarda, viral çoğaltma bölmeleri olarak adlandırılan ve sadece FISH ve IF ile ayırt edilebilen merkezler. Burada Kaposi sarkomu ile ilişkili herpesvirus (KSHV) enfekte hücreleri için RNA FISH ve IF teknikleri nin uyarlanabilir bir protokolünü açıklıyoruz, hem yapışık hem de süspansiyon. Yöntem, spesifik anti-duyu oligonükleotidlerin, çift RNA FISH, IF ile RNA FISH ve floresan yoğunluklarının nicel hesaplamalarının geliştirilmesi için gerekli adımları içerir. Bu protokol, birden fazla hücre tipine, enfekte olmayan hücrelere, gizli hücrelere, litik hücrelere, zaman-kurslara ve inhibitörlerle tedavi edilen hücrelere, hem insan konakından hem de insan konakçısından gelen spesifik RNA'ların ve proteinlerin spatiotemporal aktivitelerini analiz etmek için başarıyla uygulanmıştır. KSHV.
Giriş
Litik (aktif) fazlarında, herpesvirüsler konak hücreyi kaçırArak hücre morfolojisinde ve biyolojik moleküllerin lokalizasyonunda değişikliklere yol açarak virions üretirler. Operasyonların temeli çekirdektir, burada çift iplikli DNA viral genomu çoğaltılır ve bir protein kabuğuna paketlenir, capsid1olarak adlandırılır. Başlamak için, virüs kendi proteinlerini ifade eder, konak makine kaçırma ve gerekli olmayan konak genlerin ifade sini önleme, bir süreç konak kapatma etkisi olarak adlandırDı. Bu aktivitenin çoğunluğu belirli lokalize 4′,6-diamidino-2-fenilindole (DAPI)-ücretsiz nükleer bölgeler viral çoğaltma bölmeleri olarak adlandırılan, hem konak ve viral proteinler oluşan, RNA, ve viral DNA2. Hücre çoğaltma bölmeleri ve böylece viral capsids montajı için yer ve kaynak sağlamak için elden geçirilir. Kapsid çekirdekten çıktıktan sonra, kapsidin sitoplazmada nasıl sarsılmış olduğu, virion olarak da bilinen zara bağlı viral parçacık lar ürettiği belirsizdir. Litik fazda hem konak hem de viral biyomoleküllerin lokalizasyonu ve mekansal kaymalarının anlaşılması, çoğaltma bölmesinin düzenlenmesi, konak kapatma etkisi, virion-çıkış yolu ve diğer herpesviral enfeksiyon ve replikasyon ile ilgili süreçler.
Şu anda bu değişiklikleri tespit etmek ve incelemek için en iyi yöntem, sırasıyla immünfloresansan (IF) ve floresan in situ hibridizasyon (FISH) ile enfekte hücrelerde protein ve RNA görselleştirme olduğunu. Bu tekniklerle bir zaman-ders kullanımı litik faz ın önemli noktalarında biyomoleküllerin lokalizasyonunu ya da basitçe, spatiotemporal verileri ortaya çıkarır. FISH ve IF, hücresel sürecin inhibisyonu (örn. viral DNA replikasyonunun inhibisyonu), RT-qPCR (gerçek zamanlı polimeraz zincir reaksiyonu), RNA sıralama, Kuzey lekeleri, kütle spektrometresi, Batı lekeleme ve hücresel faaliyetlerin daha küresel bir resim sağlayabilir viral DNA üretiminin analizi.
RNA ürünlerini belirli genlerden incelemek için RNA FISH stratejileri ve belirli bir gen ürününün nükleositoplazmik oranını nicel olarak hesaplayan bir hesaplama analizi geliştirdik. Örnek hazırlama, Steitz ve meslektaşları tarafından önceki yayınlardan modifiye3,4, nispeten kolay ve hem yapışık ve askıda hücreler için kullanılabilir. Protokol aynı zamanda birden fazla RNA FISH stratejilerinin (çift RNA FISH) veya IF stratejileri ile RNA FISH'in eşzamanlı kullanımı için de uyarlanabilir. Belirli bir FISH stratejisinin geliştirilmesi zordur, ancak başarıyı artırmak için öneriler özetlenmiştir. Burada açıklanan veri analizi, floresan boncuklar ve bölme sınırlarının güçlü belirteçleri kullanılıyorsa niceldir ve mikrograflar, gözlem önyargısını ortadan kaldıran içgörü hakkında ek bilgiler sunar. Ayrıntılı protokol Kaposi sarkomu ile ilişkili herpesvirus (KSHV) tarafından enfekte hem gizli hem de litik hücreler için tasarlanmıştır ve diğer herpesviruses tarafından enfekte olmayan hücreler veya hücreler ile kullanılabilir5. Sayısallaştırma yöntemleri, çoğu hücredeki hücre altı bölmeler arasında nükleositoplazmik kaymalar veya yeniden lokalizasyon çalışmaları için geçerlidir.
Protokol
1. Belirli bir herpesviral transkript tespit etmek için yerinde floresan tasarımı (FISH) anti-sense oligonükleotidler
- İlgi çekici RNA dizisinden 25 ila 40 nt segmentleri seçin ve anti-sense'e dönüştürün. Başarılı bir FISH stratejisi bir kadar on veya daha fazla farklı anti-sense oligonükleotid içerebilir. Dizileri seçerken aşağıdakileri göz önünde bulundurun:
- İlgi alanı benzersiz bir yineleme bölgesi içeriyorsa, bu özellikten yararlanın ve yineleme dizisini hedeflemek için bir anti-sense oligonükleotid tasarlayın.
NOT: Tycowski ve meslektaşları5 rhesus rhadinovirus (RRV) poliainkaredilmiş nükleer (PAN) RNA ile bu stratejinin bir örnek sağlar. - RNA ilgi bilinen bir protein bağlayıcı site veya kök-döngü yapısı içeriyorsa, bu bölgelerden kaçınmak oligonükleotidler tasarlar.
- Deneylerin hedeflerine bağlı olarak, intronic dizive bunun için bir anti-sense oligonükleotid tasarlamak için olup olmadığını düşünün.
- İlgi alanı benzersiz bir yineleme bölgesi içeriyorsa, bu özellikten yararlanın ve yineleme dizisini hedeflemek için bir anti-sense oligonükleotid tasarlayın.
- Bağlayıcı özgüllüğü sağlamak ve anti-duyu oligonükleotidin agregasyonunu azaltmak için seçilen anti-duyu dizileri üzerinde basit hesaplamalı analizler yapın.
- Diziler yaklaşık %50 GC açısından zengin (yüksek guanin ve sitozin içeriği) olmalı ve 60 ila 70 °C aralığında erime sıcaklığına sahip olmalıdır.
- Melezleme sıcaklığı olan 37 °C'nin üzerinde erime sıcaklığına sahip saç tokalarını kendi kendine dağıtmayan veya şekillendirmeyen dizileri seçmek için bir dizi analiz aracı kullanın.
- 'Biraz benzer' ayarı kullanarak hem ana bilgisayara hem de viral transkripsiymelere karşı seçilen dizilerin bir NCBI BLASTn (Ulusal Biyoteknoloji Bilgi Merkezi Temel Yerel Hizalama Arama Aracı) gerçekleştirin. Bu arama, diğer ev sahibi veya viral transkriptler muhtemelen bağlamak olmaz benzersiz anti-sense oligonükleotidler tanımlayacak.
NOT: Transkripsiyon lar mevcut değilse, BLAST aramasını genomik dizilerle gerçekleştirin. Vahşi suşları çeşitlendirmek ve farklı laboratuvar suşları dizileri bir arada içeren eğilimindedir, çünkü aramalar deneyde kullanılan enfekte hücrelerden izole virüs dizileri üzerinde yapılırsa idealdir.
- Sipariş, hesaplamalı olarak doğrulanan anti-duyu dizisine karşılık gelen DNA oligonükleotidlerinin tek ilisine alinmis ve hedef RNA' ya basılanmis tir. Oligonükleotidlerde özel bir değişiklik yapılmasına gerek yoktur.
- FISH ve Northern blot tarafından bağlayıcı özgüllük için tasarlanmış anti-sense oligonükleotidleri test edin.
- Aynı konak türünden (örneğin, 293T) ve ideal olarak aynı hücre tipinden enfekte olmayan bir hücre hattı kullanarak, sağlam bir organizatörden (CMV, sitomegalovirüs) ve boş vektöre (örn. pcDNA3) ilgi rna'sını ifade eden bir plazmidile bir transfeksiyon gerçekleştirin. GFP (yeşil floresan protein) plazmidi (örn. pmaxGFP) veya GFP içeren vektör ile birlikte transfeksiyon gibi transfeksiyon için pozitif bir kontrol kullanın.
NOT: Herpesvirüs Epstein-Barr virüsü (EBV) kullanılarak ölümsüzleştirilen hücre hatlarından kaçınmak önemlidir, çünkü herpesvirüsler arasında dizi benzerlikleri vardır. - Bu protokolde açıklandığı gibi anti-sense oligonükleotidler ile hücrelerin her iki kümeüzerinde bölüm 3 açıklandığı gibi FISH gerçekleştirin. Fish deneylerini tek tek, çiftler veya anti-duyu saleloligonükleotid kümeleriyle karşılaştırarak başarılı adayları edin. İnsan hücre çekirdeği 6 başına 500.000 kopya mevcut olan U2 snRNA (küçük nükleer RNA) FISH protokolü için pozitif kontrol kullanın( Tablo 1).
- Floresan sinyal, rna içeren hücrede özel ve güçlü olmalıdır. Sinyali güçlendirmek ve özellikle dikkate olmayan bağlamak anti-sense oligonükleotidkaldırmak için ek anti-sense oligonükleotidler tasarla. Sinyal gücü arka plan ve otofloresan üzerinde olmalıdır.
- Kuzey lekesi ile bağlayıcı özgüllüğü test edin.
- Aynı konak türünden (örneğin, 293T) ve ideal olarak aynı hücre tipinden enfekte olmayan bir hücre hattı kullanarak, sağlam bir organizatörden (CMV, sitomegalovirüs) ve boş vektöre (örn. pcDNA3) ilgi rna'sını ifade eden bir plazmidile bir transfeksiyon gerçekleştirin. GFP (yeşil floresan protein) plazmidi (örn. pmaxGFP) veya GFP içeren vektör ile birlikte transfeksiyon gibi transfeksiyon için pozitif bir kontrol kullanın.
2. Oligonükleotid ve hücre hazırlığı
- Üreticinin talimatlarına uyarak, anti-sense oligonükleotidleri dioksigenin(DIG)-dUTP ile veya güçlü bir şekilde bağlıysa, doğrudan Alexa Fluor 594-5-dUTP gibi floresan nükleotitle etiketlemek için terminal transferazını kullanın. Etiketlemeden sonra, ek arıtma gerekli değildir. Fotokopi yi önlemek için doğrudan etiketlenmişse - 20 °C'de ve birkaç yıla kadar folyo da etiketlenmiş oligonükleotidleri saklayın.
DİkKAT: Etiketleme çözeltisi toksik bir madde, potasyum kacodylate içerir. Etiketleme reaksiyonlarını eldivenlerle kullanın.
NOT: Kaynakları korumak için, birkaç farklı anti-sense oligonükleotidler tek bir reaksiyonda etiketlenebilir. Bu protokol 3'-end etiketleme kullanır. Kimyasal grup (örneğin, DIG veya florofor) yakalandığı veya DNA polimerazın aktif bölgesine giremediği için dahili etiketleme zordur. İki farklı RNA ile yapılan bir deney, doğrudan etiketlenmiş anti-sense oligonükleotidler ve farklı bir florofor (örneğin, FITC (floresan) veya Alexa Fluor 488 ile Alexa Fluor 594 ile anti-DIG immünorfloresans kullanılarak yapılabilir. - Hücreleri sekiz bölmeli slaytlara yapıştırın.
NOT: Sekiz odalı slaytlar antikorlar gibi değerli kaynakları en aza indirirken birkaç eşzamanlı deneye izin verir. Sekiz odalı slaytlara alternatif olarak, her ikisi de steril olan standart kapaklı (22 mm x 22 mm) altı kuyulu doku kültür plakasi bulunur. Benzer bir düzenleme dairesel kapaklar ve 24 kuyulu doku kültür plakası ile mümkündür. Her ikisi için de, bu protokolde belirtilen hacimleri sırasıyla 10-15x (örn. 1,75 mL hibridizasyon çözümü) ve 4x (örn. 600 μL hibridizasyon çözümü) artırın.- Yapışık litik hücreler için, hücreleri askıya almak ve %60 birleşmeyi seyreltmek için 37 °C'de 1x tripsin/PBS ve 10 dakika için %5 CO2 kullanın.
NOT: FISH deneylerinde kullanılan yapışık hücre hatları 293T, iSLK.2197ve iSLK-BAC36 hücreleri 8'dir. - Steril sekiz odalı slaytların her bir odasına 200 μL hücre süspansiyonu uygulayın ve 37 °C'de 12-24 saat ve %5 CO 2'de tohum büyümesine izin verin. Yavaş veya hızlı büyüyen hücreler ve tripsin tarafından kolayca zarar gören hücreler için gerektiği gibi ayarlayın.
NOT: Amaç, eşit aralıklı hücrelerin slayta sıkıca bağlanmasıdır. Litik hücreler kırılgansa yapışma sonrası litik faz ısklamayı düşünün. iSLK hücreleri ile yapılan deneylerden elde edilen sonuçlar9ile sınırlıdır. - Litik süspansiyon hücreleri için, doku kültürü başlık altında 5 dakika için 1:10 poli L-lizin ile sekiz odalı slaytlar ön tedavi. Daha sonra kaydırakça oda sıcaklığında veya 65 °C'de 1 saat kurumaya bırakın. 1 x 106 hücre/mL konsantrasyonda 800 μL likit hücreleri kuluçkaya yatırın ve 30 dk ile 1 saat arasında 37 °C'de ve %5 CO 2'de odacıklı slaytlar .
NOT: Süspansiyon hücreleri tek katmanlı bir yerleşir, poli L-lizin yapışmasını ve böylece aşırı hücreler yapışık hücrelere göre bir endişe değildir. Mümkünse üzüm kümelerini oluşturan likit hücreler nazik girdap veya kimyasal yollarla ayrılmalıdır. Kuşkusuz, yazarlar litik BJAB-RRV-GFP hücreleri durumunda bu tür öneriler ile çok başarılı olmadı. Süspansiyon hücreleri iyi yapışmaz ise, poli L-lizin kuluçka ya zaman veya konsantrasyonu artırmayı düşünün.
- Yapışık litik hücreler için, hücreleri askıya almak ve %60 birleşmeyi seyreltmek için 37 °C'de 1x tripsin/PBS ve 10 dakika için %5 CO2 kullanın.
3. Fiksasyon, İmmünofluoresans (İsteğe Bağlı), Hibridizasyon ve Viral RNA'ların Görselleştirilmesi
- Ortam ve fazla hücreleri kaldırın. Bu protokol boyunca, çözümleri kaldırmak için vakum emme ve çözümler eklemek için nazik mikropipetleme kullanın.
NOT: Bir vakumun mukavemeti, cam Pasteur pipetinin üzerine 200 μL mikropipet ucu yerleştirilerek azaltılabilir. Kontaminasyonu önlemek için mikropipet ucunu yıkama adımları arasında değiştirin. Hücrelerin asla kurumaması zorunlu olduğundan her yıkama adımı hızlı bir şekilde yapılmalıdır. - Hemen, 30 dakika boyunca buz üzerinde önceden soğutulmuş% 4 formaldehit / PBS (fosfat-tamponlu salin) ile hücreleri düzeltmek.
- 200 μL önceden soğutulmuş %0,5 Triton-X/PBS (fosfat tamponlu salin) ile 10 dakika buz üzerinde veya 750 μL önceden soğutulmuş %70 etanol ile 4 °C'de 1 saat (dk) ila 7 d (max) arasında permeabilize edin.
NOT: Görüntüler ve biyokimyasal tahliller arasında tutarlılık sağlamak için fiksasyon noktasında protein, total RNA ve genomik DNA örneklerini toplayın. Aksi belirtilmedikçe, bu protokol deki tüm yıkıntılar aynı şekilde gerçekleştirilir. %70 etanol, daha sonra ayırmayı kolaylaştıran ve protokolde önemli bir duraklama sağlayan tutkal ile slayt arasındaki tutkalgevşetini gevşetir. Bununla birlikte, buharlaşmayı azaltmak için oda slayt etrafında parafin film kullanın ve her odada etanol seviyesini kontrol yaklaşık her 8 h. 70% etanol de hücreleri düzleştirir, daha canlı bir görüntü yapma, Triton-X hücreleri dehidratasyon ve değiştirmek hücrenin boyutları.
- 200 μL önceden soğutulmuş %0,5 Triton-X/PBS (fosfat tamponlu salin) ile 10 dakika buz üzerinde veya 750 μL önceden soğutulmuş %70 etanol ile 4 °C'de 1 saat (dk) ila 7 d (max) arasında permeabilize edin.
- Slaytın çatlamasını önlemek için odaları dikkatlice çıkarın. Deney, poliklonal primer antikoriçeren bir viral veya konak proteinin immünofloresansını (IF) içeriyorsa, RNA FISH'e geçmeden önce aşağıda açıklandığı gibi IF'yi gerçekleştirin. İmmün floresan monoklonal primer antikor kullanıyorsa, 3.11'den sonra adım 3.3.1'de açıklandığı gibi immünororesans gerçekleştirin.
NOT: Yeni bir kaldırma cihazı veya üretici tarafından sağlanan çok az artık yapıştırıcı içeren bir cihaz kullanın ve kaydırağın çatlamasını önlemek için odaları hafifçe gevşetin. 4 saat için permeabilizing reaktif olarak% 70 etanol kullanarak büyük ölçüde çatlama olasılığını azaltır. Bir çatlak durumunda, çatlaktan etkilenmeyen bölmelerde protokolü devam edin ve kusurlu mühürlü slaytların daha yüksek oksidasyon oranına (yani depolama ömrünün azaldığına) dikkat edin.- Önceden soğutulmuş 1x PBS ile hücreleri durulayın ve önceden soğutulmuş 4% BSA (sığır serum albumin)/1x PBS ile 30 dakika 4 °C'de.
NOT: Bu protokol boyunca BSA'nın kullanımı spesifik olmayan etiketlemeyi sınırlar. - Bloklama çözeltisini çıkarın ve hücreleri 1:200 veya başka bir poliklonal primer antikor ile %0,1 BSA/1x PBS'de 1 saat 4 °C'de inküler. Sonra 1x PBS ile üç kez yıkayın.
NOT: 1:200 seyreltmede SSB/ORF6 (viral tek iplikli DNA bağlayıcı protein) tespiti için 10 antikor kullanıldı. - 4 °C'de 1 saat boyunca FISH-detecting antikor ile uyumlu florosofor ile ikincil bir antikor ile hücreleri kuluçkaya yatırın. 1x PBS ile üç kez yıkayın. Daha sonra 10-15 dakika için% 4 formaldehit/1x PBS ile düzeltmek ve daha önce FISH geçmeden önce açıklandığı gibi ya Triton-X veya% 70 etanol ile permeabilize. Floresan sinyali korumak ve fotobeyazlmayı önlemek için kaydırağı folyo ile kaplayın.
- Önceden soğutulmuş 1x PBS ile hücreleri durulayın ve önceden soğutulmuş 4% BSA (sığır serum albumin)/1x PBS ile 30 dakika 4 °C'de.
- Hücreleri 2x SSC (tuzlu sodyum sitrat) ile bir kez yıkayın ve daha sonra %50 formamid, %10 dekstran sülfat, 2x SSC, %0.1 BSA, 500 μg/mL somon sperm DNA,125 μg/mL E. coli tRNA ve 1 mM vanadyl ribonucleoside'dan oluşan 45 μL hibridizasyon solüsyonu uygulayın Külliyesi. Nemlendirilmiş steril mendillerle 150 mm Petri kabı olabilecek bir nem haznesinde 37 °C'de 1 saat kuluçka.
NOT: Kullanmadan en az bir saat önce taze hibridizasyon çözümü hazırlayın. Dekstran sülfatı önce suda çözün, sık sık girdap ve 37 °C'lik bir su banyosunda kuluçkaya yatırın. - Oda başına 35-uL hibridizasyon çözeltisinde önerilen 25 μM oligonükleotid konsantrasyonuna sahip olduğunu hesaplayın. Anti-sense oligonükleotid konsantrasyonu gerektiği gibi ayarlayın. Denatürasyon hacmini 10 μL'ye çıkarmak için oligonükleotidlere distile su ekleyin.
NOT: Etiketleme reaksiyonu sonrasında, oligonükleotidler 0.18 M potasyum kacodylate, 23 mM Tris-HCl, 0.23 mg/mL BSA, 4.5 mM CoCl2,18 mM EDTA, 2.7 mM K-fosfat ve 6.8 mM KCl, 45 μM 2-Mercaptoethanol, %0.02 Triton X-100 ve %2 gliserol. Konsantrasyonlar, su ile seyreltmenin denatürasyon çözeltisini standart bir oligonükleotid denatürasyon tamponu olan 1x TE 'ye (10 mM Tris-HCl ve 1 mM EDTA) yakın konsantrasyonlara getirecek kadar yüksektir. - DIG ve/veya Alexa Fluor 594 etiketli oligonükleotidleri 95 °C'de 5 dk.'da denatüre edin. Daha sonra denatüre oligonükleotidlere amaçlanan oda başına 35 μL taze hibridizasyon çözeltisi ekleyin. Çift FISH yapıyorsa, her iki anti-duyu lu oligonükleotid kümesi dedenave melezlenebilir.
- Ön hibridizasyon çözümlerini çıkarın ve sonra hücrelere etiketli oligonükleotidleri içeren hibridizasyon çözeltisi ekleyin. Florofor etiketli oligonükleotidleri korumak için 37 °C'de nem odasında bir gecede tüp folyo ile inkübed.
NOT: Kuluçka en az 10 saat ve en fazla 24 saat olmalıdır. - Ertesi gün hücreleri 37 °C'de 10 dk 2x SSC ile iki kez, 25 °C'de 10 dakika 1x SSC ile iki kez yıkayın.
- Önceden soğutulmuş% 4 formaldehit/1x PBS ile hücreleri düzeltin 10-15 dakika buz üzerinde. Daha sonra hücreleri PBS ile üç kez yıkayın ve 1 saat önceden soğutulmuş %70 etanol ile veya 10 dakika önceden soğutulmuş %0,5 Triton-X/1x PBS ile 4 °C'de permeabilize edin.
- Hücreleri 1:200 anti-DIG FITC ile önceden soğutulmuş %0,1 BSA/1x PBS'de 1 saat 4 °C'de 1 saat kuluçkaya yatırın. Antikor solüsyonu çıkarın ve 1x PBS ile üç kez yıkayın.
- 4 °C'de 10-15 dakika önceden soğutulmuş %4 formaldehit/1x PBS ile düzeltin ve 1x PBS ile üç kez yıkayın. Monoklonal primer antikoriçeren bir konak veya viral protein için immünofloresan gerçekleştiriyorsanız, hücreleri permeabilize edin ve daha sonra adım 3.3.1'de belirtilen IF protokolünü uygulayın. Aksi takdirde DAPI boyama devam edin.
- Hücreleri önceden soğutulmuş %0.5 Triton-X/1x PBS'de %0.4 g/mL DAPI ile 15 dakika buzüzerinde kuluçkaya yatırın ve 1x PBS ile üç kez yıkayın.
- Floresan boncuk (isteğe bağlı) ve montaj ortamı ile montaj slaytlar. Sonra açık oje ile slayt için coverslip mühür.
- Bir konfokal mikroskop kullanarak, 630x büyütme protokolü gerçekleştirmek ten sonra bir saat ila bir hafta içinde örneklerin görüntülerini toplamak. Kapak fişini kapatmak ve oksidasyon oranını azaltarak florofor ömrünü uzatmak için birden fazla kat oje uygulayın.
NOT: DAPI içeren montaj ortamı kullanmayın. Görüntüleri toplarken, daha sonra niceleme için her görüntünün ölçek çubuğunu ekleyin. Floresan boncuklar slaytlar ve örnek preparatlar arasında floresan yoğunluğu kontrolleri olarak hizmetvermektedir 11. Adım 4'te iki boyutlu (2D) nicelik için hücrenin orta kısmından görüntüler edinin.
4. Subsellüler lokalizasyonu vurgulamak ve floresannün nükleositoplazmik oranını belirlemek için FISH ve IF görüntülerinin sayısallaştırılması
- Tutarlılık sağlamak için çeşitli floresan lekeli ve birleştirilmiş görüntülerin birleştirilmiş yığınında görüntü analizi gerçekleştirin. Görüntüler toplandığında dahil edilen ölçek çubuğunu kullanarak görüntü çözümleme yazılımının ölçeğini ayarlayın.
- Çeşitli kanallarda ve nükleer DAPI lekesine atıfta bulunarak floresan yoğunluğunu ölçmek için bir çizgi aracı ve çizim profili işlevi kullanın. Ardından, görüntüleyenin yargısını engellemeyen veya etkilemeyen işaretçiler kullanarak görüntünün bir kopyasındaki satırı kalıcı olarak belirtin.
- Merkezi bir eksen veya aşırı doymamış alanlardan geçmeyen bir çizgi boyunca topografik özellikler, zirveler ve vadiler çeşitliliğini yakalayan bir iz gibi çizginin çizildiği yeri yönlendirmek için ölçütler belirleyin.
NOT: Bu çizgi izleri bir hücrede ham floresan tasvir ve bu nedenle bir leke değil, yoğunluk konumları karşılaştırmaları ile sınırlıdır. Slaytlar, tedaviler veya preparatlar arasında aynı lekenin yoğunluklarını karşılaştırmak için, adım 3.13 sırasında bir iç kontrol olarak slayt bir floresan boncuk ekleyin. Floresan boncuk montaj işlemi sırasında eklenmeli ve uyarma lazer ve fotoçarpan tüpü (konfokal) üzerinde aynı ayarları ile tespit edilmelidir.
- Merkezi bir eksen veya aşırı doymamış alanlardan geçmeyen bir çizgi boyunca topografik özellikler, zirveler ve vadiler çeşitliliğini yakalayan bir iz gibi çizginin çizildiği yeri yönlendirmek için ölçütler belirleyin.
- Hücre altı lokalizasyonundaki değişimi ölçmek için, farklı tedaviler uygulanan hücrelerin nükleositoplazmik oranlarını hesaplayın.
- İç sınırı ayarlamak için nükleer DAPI lekesini kullanarak hem çekirdeğin hem de sitoplazmanın alanını ve ham floresan yoğunluğunu ölçün. Nükleer RNA (örneğin, KSHV PAN RNA) ve sitoplazmik RNA (örneğin, ev sahibi GAPDH mRNA) gibi nükleer ve sitoplazmik kontrolleri içerir. Ayrıca, üç hücre benzeri alan için arka plan yoğunluğunu hesaplayın ve piksel veya μm2başına değerleri ortalama.
NOT: Yoğunluk değerleri birim eksikliği eğilimindedir ve bu nedenle 'birimler' terimi kullanılır. - Önce aynı alan için ortalama arka planı belirleyerek ve daha sonra alanın ham yoğunluğundan bu bireyselleştirilmiş değeri çıkararak hem nükleer hem de hücresel ham yoğunluk değerlerini normalleştirin.
- Örneğin, bir litik B hücresinin çekirdeği 133,4 μm2 alana ve ham yoğunluğu 75976 birim iken, aynı floresan sinyalin arka plan yoğunluğu nun μm başına 0,67 birim2olarak belirlenmiştir. Normalleşen nükleer yoğunluk,
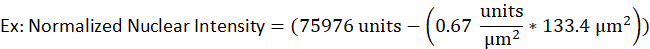
- Örneğin, bir litik B hücresinin çekirdeği 133,4 μm2 alana ve ham yoğunluğu 75976 birim iken, aynı floresan sinyalin arka plan yoğunluğu nun μm başına 0,67 birim2olarak belirlenmiştir. Normalleşen nükleer yoğunluk,
- Aşağıdaki denklemdeki değerleri girin.

NOT: Bu hesaplama hücre altı alanındaki değişiklikleri denetler. Litik indüksiyon ve ilaç tedavileri çekirdeği büyütebilir veya hücrenin boyutunu değiştirebilir, sırasıyla. - Sonuçları yorumlamak için bir kutu bıyık çizimi oluşturun. Floresan sinyalin eşit dağılımı sıfıra yakın olurken, nükleer dağılım pozitif bir oran değerini ve sitoplazmik dağılımı negatif oran değerine doğru eğilim gösterir.
- İç sınırı ayarlamak için nükleer DAPI lekesini kullanarak hem çekirdeğin hem de sitoplazmanın alanını ve ham floresan yoğunluğunu ölçün. Nükleer RNA (örneğin, KSHV PAN RNA) ve sitoplazmik RNA (örneğin, ev sahibi GAPDH mRNA) gibi nükleer ve sitoplazmik kontrolleri içerir. Ayrıca, üç hücre benzeri alan için arka plan yoğunluğunu hesaplayın ve piksel veya μm2başına değerleri ortalama.
Sonuçlar
Bu el yazmasında ayrıntılı olarak açıklanan FISH ve IF yöntemleri, floresan yoğunluğunun çizgi izleriyle sonuçların ölçülmesi ile birlikte Şekil 1'de gösterilmiştir. Burada sunulan sonuçlar yarı-nicel ve deneyler slayt hazırlanmasında floresan boncuk içermemelidir çünkü farklı floresan lekelerin yoğunlukları arasındaki karşılaştırmalar yerine, lokalizasyon içine fikir sunuyoruz. Şekil 1 ayrıca...
Tartışmalar
Bu raporda açıklanan protokol farklı hücre tiplerine uyarlanabilir ve çift RNA FISH ve RNA FISH için hem monoklonal hem de poliklonal primer antikorlar kullanılarak adımlarını içerir. Hazırlanan slaytlar genellikle konfokal mikroskop ile görüntülense de, artmış antikor konsantrasyonu ve farklı montaj ortamı modifikasyonları yapıldıktan sonra sted (uyarılmış emisyon tükenmesi) mikroskopile görüntüleme yapılabilir. Bireysel hücrelerin gelişmiş analizi için, bu protokol ile hazırlanan örn...
Açıklamalar
Yazarların ifşa etmesi gereken çıkar çatışmaları yok.
Teşekkürler
Biz veri analizi konusunda tavsiye için Jonathan Rodenfels, Kazimierz Tycowski ve Johanna B. Withers teşekkür ederiz. Ayrıca G. Hayward'a anti-SSB antikorları için teşekkür ederiz. Bu çalışma, Ulusal Sağlık Enstitüleri (TKV'ye) ve NIH hibe (CA16038) (JAS'e) T32GM007223 ve T32AI055403 hibeleri ile desteklenmiştir. JAS, Howard Hughes Tıp Enstitüsü'nün bir araştırmacısı. Şekil 1-3 ve Tablo 1 Aşağıdaki yayından Creative Commons Atıf lisansı altında Amerikan Mikrobiyoloji Derneği izni ile çoğaltıldı: Vallery, T. K., Withers, J. B., Andoh, J. A., Steitz, J. A. Kaposi's Sarcoma-Associated Nükleer Foci Herpesvirus mRNA Birikimi Viral DNA Replikasyonu ve Viral Noncoding Polyadenylated Nükleer RNA etkilenir. Viroloji Dergisi. 92 (13), doi:10.1128/JVI.00220-18, (2018).
Malzemeler
| Name | Company | Catalog Number | Comments |
| AlexaFluor594-5-dUTP | Life Technologies | C1100 | |
| anti-DIG FITC | Jackson Lab Immunologicals | 200-092-156 | |
| Anti-Rabbit Secondary AlexaFluor594 Monoclonal Antibody | Invitrogen | A-11037 | Goat |
| Anti-SSB Antibody | N/A | N/A | Ref. Chiou et al. 2002 |
| BLASTn | NIH NCBI | N/A | Free Sequence Alignment Software |
| Dextran Sulfate | Sigma Aldrich | D8906 | Molecular Biology Grade |
| DIG-Oligonucleotide Tailing Kit | Sigma Roche | #03353583910 | 2nd Gen |
| Eight-Chamber Slides | Nunc Lab Tek II | #154453 | Blue seal promotes surface tension but separation by clear gel is also available. |
| Formamide | Sigma Aldrich | F9037 | Molecular Biology Grade |
| GAPDH Probes | Stellaris | SMF-2019-1 | Compatible with protocol, Quasar 670 |
| ImageJ | NIH, Bethesda, MD | N/A | Free Image Analysis Software, [http:rsb.info.nih.gov/ij/] |
| OligoAnalyzer | IDT | N/A | Free Oligonucleotide Analyzer |
| pcDNA3 | Invitrogen | A-150228 | |
| pmaxGFP | Amaxa | VDF-1012 | |
| Poly L-Lysine | Sigma Aldrich | P8920 | |
| Terminal Transferase | Sigma Roche | #003333574001 | |
| Vanadyl Ribonucleoside Complexes | NEB | S1402S | |
| Vectashield | Vector Laboratories, Inc. | H-1000 | DAPI within the mounting media scatters the light and reduces contrast. |
Referanslar
- Amen, M. A., Griffiths, A. Packaging of Non-Coding RNAs into Herpesvirus Virions: Comparisons to Coding RNAs. Frontiers in Genetics. 2, 81 (2011).
- Schmid, M., Speiseder, T., Dobner, T., Gonzalez, R. A. DNA virus replication compartments. Journal of Virology. 88 (3), 1404-1420 (2014).
- Pawlicki, J. M., Steitz, J. A. Primary microRNA transcript retention at sites of transcription leads to enhanced microRNA production. Journal of Cell Biology. 182 (1), 61-76 (2008).
- Borah, S., Darricarrere, N., Darnell, A., Myoung, J., Steitz, J. A. A viral nuclear noncoding RNA binds re-localized poly(A) binding protein and is required for late KSHV gene expression. Public Library of Science Pathogens. 7 (10), e1002300 (2011).
- Tycowski, K. T., Shu, M. D., Borah, S., Shi, M., Steitz, J. A. Conservation of a triple-helix-forming RNA stability element in noncoding and genomic RNAs of diverse viruses. Cell Reports. 2 (1), 26-32 (2012).
- Weinberg, R. A., Penman, S. Small molecular weight monodisperse nuclear RNA. Journal of Molecular Biology. 38 (3), 289-304 (1968).
- Myoung, J., Ganem, D. Generation of a doxycycline-inducible KSHV producer cell line of endothelial origin: maintenance of tight latency with efficient reactivation upon induction. Journal of Virology Methods. 174 (1-2), 12-21 (2011).
- Brulois, K. F., et al. Construction and manipulation of a new Kaposi's sarcoma-associated herpesvirus bacterial artificial chromosome clone. Journal of Virology. 86 (18), 9708-9720 (2012).
- Sturzl, M., Gaus, D., Dirks, W. G., Ganem, D., Jochmann, R. Kaposi's sarcoma-derived cell line SLK is not of endothelial origin, but is a contaminant from a known renal carcinoma cell line. International Journal of Cancer. 132 (8), 1954-1958 (2013).
- Chiou, C. J., et al. Patterns of gene expression and a transactivation function exhibited by the vGCR (ORF74) chemokine receptor protein of Kaposi's sarcoma-associated herpesvirus. Journal of Virology. 76 (7), 3421-3439 (2002).
- Cole, R. W., Jinadasa, T., Brown, C. M. Measuring and interpreting point spread functions to determine confocal microscope resolution and ensure quality control. Nature Protocols. 6 (12), 1929-1941 (2011).
- Nakamura, H., et al. Global changes in Kaposi's sarcoma-associated virus gene expression patterns following expression of a tetracycline-inducible Rta transactivator. Journal of Virology. 77 (7), 4205-4220 (2003).
- Majerciak, V., Yamanegi, K., Zheng, Z. M. Gene structure and expression of Kaposi's sarcoma-associated herpesvirus ORF56, ORF57, ORF58, and ORF59. Journal of Virology. 80 (24), 11968-11981 (2006).
- Sun, R., Lin, S. F., Gradoville, L., Miller, G. Polyadenylylated nuclear RNA encoded by Kaposi sarcoma-associated herpesvirus. Proceedings of the National Academy Sciences U S A. 93 (21), 11883-11888 (1996).
- Vallery, T. K., Withers, J. B., Andoh, J. A., Steitz, J. A. Kaposi's Sarcoma-Associated Herpesvirus mRNA Accumulation in Nuclear Foci Is Influenced by Viral DNA Replication and Viral Noncoding Polyadenylated Nuclear RNA. Journal of Virology. 92 (13), (2018).
- Borah, S., Nichols, L. A., Hassman, L. M., Kedes, D. H., Steitz, J. A. Tracking expression and subcellular localization of RNA and protein species using high-throughput single cell imaging flow cytometry. RNA. 18 (8), 1573-1579 (2012).
- Bruce, A. G., et al. Quantitative Analysis of the KSHV Transcriptome Following Primary Infection of Blood and Lymphatic Endothelial Cells. Pathogens. 6 (1), (2017).
- Chen, C. P., et al. Kaposi's Sarcoma-Associated Herpesvirus Hijacks RNA Polymerase II To Create a Viral Transcriptional Factory. Journal of Virology. 91 (11), (2017).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır