Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Fluorescence quantitative In Situ Hybridization (FISH) et Immunofluorescence (IF) de produits génétiques spécifiques dans les cellules infectées par le KSHV
Dans cet article
Résumé
Nous décrivons un protocole utilisant l'hybridation in situ de fluorescence (FISH) pour visualiser les ARN herpèsvirs multiples dans les cellules humaines lytically infectées, en suspension ou adhérentes. Ce protocole comprend la quantification de la fluorescence produisant un rapport nucléocytoplasmique et peut être étendu pour la visualisation simultanée des protéines hôtes et virales avec immunofluorescence (IF).
Résumé
La perspicacité mécanique provient d'une étude minutieuse et d'une quantification d'ARN et de protéines spécifiques. Les emplacements relatifs de ces biomolécules dans toute la cellule à des moments précis peuvent être capturés avec la fluorescence in situ hybridation (FISH) et l'immunofluorescence (IF). Pendant l'infection par l'herpèsvirus lytique, le virus détourne la cellule hôte pour exprimer de préférence les gènes viraux, provoquant des changements dans la morphologie cellulaire et le comportement des biomolécules. Les activités lytiques sont centrées dans les usines nucléaires, appelées compartiments de réplication virale, qui ne sont discernables qu'avec FISH et IF. Ici nous décrivons un protocole adaptable des techniques de FISH d'ARN et de IF pour kaposi' s sarcoma-associé à l'herpèsvirus (KSHV)-infecté cellules, adhérentes et en suspension. La méthode comprend des étapes pour le développement d'oligonucléotides anti-sens spécifiques, DOUBLE ARN FISH, ARN FISH avec IF, et des calculs quantitatifs des intensités de fluorescence. Ce protocole a été appliqué avec succès à plusieurs types de cellules, cellules non infectées, cellules latentes, cellules lytiques, cours temporels, et cellules traitées avec des inhibiteurs pour analyser les activités spatiotemporal esdedeuses de RNA et de protéines spécifiques de l'hôte humain et Le KSHV.
Introduction
Dans leur phase lytique (active), les herpèsvirus détournent la cellule hôte, provoquant des changements dans la morphologie cellulaire et la localisation des molécules biologiques, pour produire des virions. La base des opérations est le noyau, où le génome viral de l'ADN à double brin est reproduit et emballé dans une coquille de protéine, appelée capside1. Pour commencer, le virus exprime ses propres protéines, détournant les machines hôtes et empêchant l'expression de gènes hôtes non essentiels, un processus appelé l'effet d'arrêt de l'hôte. La majorité de cette activité est localisée dans des régions nucléaires spécifiques, exemptes de réplication virale, composées de protéines hôtes et virales, d'ARNet d'ADN viral 2. La cellule est révisée pour fournir de l'espace et des ressources pour les compartiments de réplication et donc l'assemblage de capsides virales. Une fois que la capsite sort du noyau, la façon dont la capside est enveloppée dans le cytoplasme pour produire une particule virale liée à la membrane, également connue sous le nom de virion, n'est pas claire. La compréhension de la localisation et des changements spatiaux des biomolécules hôtes et virales pendant la phase lytique fournit un aperçu mécaniste plus profond de l'arrangement du compartiment de réplication, de l'effet d'arrêt de l'hôte, de la voie d'évacuation des virions et d'autres processus liés à l'infection et à la réplication de l'herpèsviral.
Actuellement, la meilleure méthode pour détecter et étudier ces changements est la visualisation des protéines et des ARN dans les cellules infectées avec immunofluorescence (IF) et l'hybridation in situ fluorescente (FISH), respectivement. L'utilisation d'un cours de temps avec ces techniques révèle la localisation des biomolécules à des points clés de la phase lytique ou tout simplement, des données spatiotemporales. FISH et IF complètent d'autres techniques biochimiques, telles que l'inhibition d'un processus cellulaire (p. ex., inhibition de la réplication virale de l'ADN), RT-qPCR (réaction en chaîne de polymérase en temps réel), séquençage de l'ARN, taches nordiques, spectrométrie de masse, ballonnements occidentaux et l'analyse de la production d'ADN viral, qui peut fournir une image plus globale des activités cellulaires.
Nous avons développé des stratégies RNA FISH pour examiner les produits à base d'ARN à partir de gènes spécifiques et une analyse computationnelle qui calcule quantitativement le rapport nucléocytoplasmique d'un produit génétique spécifique. La préparation de l'échantillon, modifiée à partir de publications antérieures par Steitz et ses collègues3,4, est relativement facile et peut être utilisé pour les cellules adhérentes et suspendues. Le protocole est également adaptable pour l'utilisation simultanée de plusieurs stratégies ARN FISH (double ARN FISH) ou RNA FISH avec des stratégies IF. L'élaboration d'une stratégie FISH spécifique est difficile, mais des suggestions pour améliorer le succès sont esquissées. L'analyse des données décrite ici est quantitative si des perles fluorescentes et des marqueurs solides des limites des compartiments sont utilisés et offre un aperçu supplémentaire des micrographes, un aperçu qui élimine les biais d'observation. Le protocole détaillé est conçu pour les cellules latentes et lytiques infectées par l'herpèsvirus associé au sarcome de Kaposi (KSHV) et peut être utilisé avec des cellules non infectées ou des cellules infectées par d'autres herpèsvirus5. Les méthodes de quantitation s'appliquent aux études sur les décalages nucléocytoplasmiques ou la relocalisation entre les compartiments subcellulaires dans la plupart des cellules.
Protocole
1. Conception de la fluorescence in situ (FISH) oligonucléotides anti-sens pour détecter une transcription herpèsviral spécifique
- Sélectionnez 25 à 40 segments nt de la séquence de l'ARN d'intérêt et convertir pour être anti-sens. Une stratégie FISH réussie peut contenir d'un jusqu'à dix ou plus différents oligonucléotides anti-sens. Lors de la sélection des séquences, considérez ce qui suit :
- Si l'ARN d'intérêt contient une région de répétition unique, puis capitaliser sur cette fonctionnalité et concevoir un oligonucléotide anti-sens pour cibler la séquence de répétition.
REMARQUE : Tycowski et ses collègues5 donnent un exemple de cette stratégie avec l'ARN nucléaire polyadenylated (PAN) polyadenylated de rhésus de rhésus (RRV). - Si l'ARN d'intérêt contient un site de liaison protéique connu ou une structure en boucle de tige, concevez des oligonucléotides qui évitent ces régions.
- Selon les objectifs des expériences, considérez la séquence intronique et si oui ou non de concevoir un oligonucléotide anti-sens pour elle.
- Si l'ARN d'intérêt contient une région de répétition unique, puis capitaliser sur cette fonctionnalité et concevoir un oligonucléotide anti-sens pour cibler la séquence de répétition.
- Effectuer des analyses computationnelles simples sur les séquences anti-sens sélectionnées afin d'assurer une spécificité contraignante et de réduire l'agrégation de l'oligonucléotide antisens.
- Les séquences doivent être riches d'environ 50 % de GC (teneur élevée en guanine et en cytosine) et avoir une température de fonte de l'ordre de 60 à 70 oC.
- Utilisez un outil d'analyseur de séquences pour sélectionner des séquences qui ne s'auto-dimèrent pas ou ne forment pas d'épingles à cheveux avec des températures de fonte supérieures à 37 oC, la température d'hybridation.
- Effectuer une recherche NCBI BLASTn (National Center for Biotechnology Information Basic Local Alignment Search Tool for nucleotide alignments) recherche des séquences sélectionnées contre l'hôte et les transcriptomes viraux en utilisant le réglage « assez similaire ». Cette recherche permettra d'identifier des oligonucléotides antisens uniques qui ne se lieront probablement pas à d'autres transcriptions d'hôte ou virales.
REMARQUE : Si les transcriptomes ne sont pas disponibles, effectuez la recherche BLAST avec les séquences génomiques. Il est idéal si les recherches sont effectuées sur les séquences du virus isolé s'agit des cellules infectées utilisées dans l'expérience parce que les souches sauvages ont tendance à se diversifier et contiennent une combinaison de séquences provenant de différentes souches de laboratoire.
- Commandez des oligonucléotides d'ADN purifiés correspondant à la séquence anti-sens qui a été vérifiée par calcul pour être unique et susceptible de se lier à l'ARN cible. Aucune modification spéciale ne doit être introduite dans les oligonucléotides.
- Testez les oligonucléotides anti-sens conçus pour une spécificité de liaison par FISH et Northern blot.
- À l'aide d'une lignée cellulaire non infectée de la même espèce hôte (p. ex. 293T) et idéalement du même type de cellule, effectuez une transfection avec un plasmide exprimant l'ARN d'intérêt d'un promoteur robuste (CMV, cytomégalovirus) et un autre avec le vecteur vide (p. ex., pcDNA3). Utilisez un contrôle positif pour la transfection comme la cotransfection avec un plasmide GFP (protéine fluorescente verte) (p.p. ex., pmaxGFP) ou le vecteur contenant du GFP.
REMARQUE : Il est important d'éviter les lignées cellulaires qui ont été immortalisées à l'aide du virus de l'herpèsvirus Epstein-Barr (EBV) puisqu'il existe des similitudes de séquences entre les herpèsvirus. - Effectuer FISH tel que décrit dans la section 3 sur les deux ensembles de cellules avec les oligonucléotides anti-sens comme décrit dans ce protocole. Déduisez les candidats retenus en comparant les expériences FISH avec des oligonucléotides individuels, paires ou ensembles d'oligonucléotides antisensés. Utilisez un contrôle positif pour le protocole FISH tels que U2 snRNA (petit ARN nucléaire) FISH, présent à 500 000 exemplaires par noyau cellulaire humain6 (tableau 1).
- Le signal fluorescent doit être spécifique et fort dans la cellule contenant l'ARN d'intérêt. Concevoir des oligonucléotides anti-sens supplémentaires pour renforcer le signal et supprimer les oligonucléotides anti-sens qui lient non spécifiquement de la considération. La force du signal doit être au-dessus du fond et de l'autofluorescence.
- Spécificité de liaison d'essai par tache nordique.
- À l'aide d'une lignée cellulaire non infectée de la même espèce hôte (p. ex. 293T) et idéalement du même type de cellule, effectuez une transfection avec un plasmide exprimant l'ARN d'intérêt d'un promoteur robuste (CMV, cytomégalovirus) et un autre avec le vecteur vide (p. ex., pcDNA3). Utilisez un contrôle positif pour la transfection comme la cotransfection avec un plasmide GFP (protéine fluorescente verte) (p.p. ex., pmaxGFP) ou le vecteur contenant du GFP.
2. Oligonucléotide et préparation cellulaire
- En suivant les instructions du fabricant, utilisez le terminal transferase pour étiqueter les oligonucléotides antisens avec de la dioxigénie (DIG)-dUTP ou, si fortement liant, directement avec un nucléotide fluorescent comme Alexa Fluor 594-5-dUTP. Après l'étiquetage, une purification supplémentaire n'est pas nécessaire. Conserver les oligonucléotides étiquetés à -20 oC jusqu'à plusieurs années et dans du papier d'aluminium s'ils sont directement étiquetés pour éviter le photoblanchiment.
CAUTION: La solution d'étiquetage contient un matériau toxique, le cacodylate de potassium. Gérer les réactions d'étiquetage avec des gants.
REMARQUE : Pour conserver les ressources, plusieurs oligonucléotides anti-sens différents peuvent être étiquetés en une seule réaction. Ce protocole utilise l'étiquetage de 3'-extrémité. L'étiquetage interne est difficile parce que le groupe chimique (p. ex., DIG ou le fluorophore) se fait attraper ou est incapable d'entrer dans le site actif d'une polymérase d'ADN. Une expérience avec deux ARN différents peut être effectuée à l'aide d'oligonucléotides antisens directement étiquetés et d'immunofluorescence anti-DIG avec un fluorophore différent (p. ex. FITC (fluorescein) ou Alexa Fluor 488 avec Alexa Fluor 594). - Adhérer les cellules aux diapositives à huit chambres.
REMARQUE : Les diapositives à huit chambres permettent plusieurs expériences simultanées tout en minimisant les ressources précieuses comme les anticorps. Une alternative aux toboggans à huit chambres est une plaque de culture tissulaire à six puits avec des plaques de couverture standard (22 mm x 22 mm) qui sont toutes deux stériles. Un arrangement semblable est possible avec des couvertures circulaires et une plaque de culture de tissu de 24 puits. Pour les deux, augmenter les volumes mentionnés dans ce protocole de 10-15x (p. ex., 1,75 mL de solution d'hybridation) et de 4x (p. ex., une solution d'hybridation de 600 l) respectivement.- Pour les cellules lytiques adhérentes, utilisez 1x trypsine/PBS à 37 oC et 5 % de CO2 pendant 10 min pour suspendre les cellules et diluer à 60 % de confluence.
REMARQUE : Les lignées cellulaires adhérentes utilisées dans les expériences FISH comprenaient 293T, iSLK.2197, et iSLK-BAC36 cellules8. - Appliquer 200 ll de suspension cellulaire dans chaque chambre des lames stériles à huit chambres et permettre la croissance des graines de 12 à 24 h à 37 oC et 5 % de CO2. Ajuster au besoin pour les cellules à croissance lente ou rapide et pour les cellules qui sont facilement endommagées par la trypsine.
REMARQUE : L'objectif est d'avoir des cellules également espacées fermement attachées à la diapositive. Envisager d'induire la phase lytique après l'adhérence si les cellules lytiques sont fragiles. Les conclusions tirées d'expériences avec des cellules iSLK sont limitées9. - Pour les cellules de suspension lytiques, prétraitez des glissières à huit chambres avec 1:10 poly L-lysine pendant 5 min sous le capot de culture tissulaire. Laissez ensuite sécher les lames pendant la nuit à température ambiante ou 1 h à 65 oC. Incuber 800 l de cellules lytiques à une concentration de 1 x 106 cellules/mL avec les lames chambrées de 30 min à 1 h à 37 oC et 5 % de CO2.
REMARQUE : Les cellules de suspension s'installeront dans une monocouche, s'en tenant à la poly L-lysine et donc les cellules excédentaires ne sont pas une préoccupation par rapport aux cellules adhérentes. Les cellules lytiques qui forment des grappes de raisin, si possible, doivent être séparées par un tourbillon doux ou des moyens chimiques. Certes, les auteurs n'ont pas eu beaucoup de succès avec de telles recommandations dans le cas des cellules lytiques de BJAB-RRV-GFP. Si les cellules de suspension n'adhèrent pas bien, envisagez d'augmenter le temps ou la concentration de l'incubation poly llysine.
- Pour les cellules lytiques adhérentes, utilisez 1x trypsine/PBS à 37 oC et 5 % de CO2 pendant 10 min pour suspendre les cellules et diluer à 60 % de confluence.
3. Fixation, immunofluorescence (facultatif), hybridation et visualisation des ARN virales
- Enlever les médias et les cellules excédentaires. Tout au long de ce protocole, utilisez l'aspiration sous vide pour enlever les solutions et le micropipetting doux pour ajouter des solutions.
REMARQUE : La résistance d'un aspirateur peut être réduite en plaçant une pointe de micropipette de 200 l sur la pipette Pasteur en verre. Remplacer la pointe de micropipette entre les étapes de lavage pour prévenir la contamination. Chaque étape de lavage doit être effectuée rapidement parce qu'il est impératif que les cellules ne se dessèchent jamais. - Immédiatement, fixer les cellules avec du formaldéhyde/PBS préréfrigéré 4 % (saline tamponné par le phosphate) sur la glace pendant 30 min. Laver les cellules trois fois avec 200 L 1x PBS refroidis à 4 oC et couver pendant 5 min à température ambiante ou sur glace.
- Perméabiliser les cellules fixes avec 200 l de Triton-X/PBS pré-réfrigéré 0,5 % (saline tamponné par phosphate) pendant 10 min sur la glace ou 750 l d'éthanol préréfrigéré de 70 % à 4 oC pour 1 h (min) à 7 d (max).
REMARQUE : Recueillir des échantillons de protéines, d'ARN total et d'ADN génomique au point de fixation pour assurer la cohérence entre les images et les analyses biochimiques. Tous les lavages tout au long de ce protocole sont exécutés de la même manière, sauf indication contraire. 70% d'éthanol desserre la colle entre les chambres et la glissière, ce qui facilite la séparation ultérieure, et fournit également une pause significative dans le protocole. Néanmoins, utilisez du film de paraffine autour de la glissière de chambre pour réduire l'évaporation et vérifier le niveau de l'éthanol dans chaque chambre environ tous les 8 h. 70% d'éthanol aplatit également les cellules, faisant une image plus nette, tandis que Triton-X ne déshydrate pas les cellules et ne change pas le dimensions de la cellule.
- Perméabiliser les cellules fixes avec 200 l de Triton-X/PBS pré-réfrigéré 0,5 % (saline tamponné par phosphate) pendant 10 min sur la glace ou 750 l d'éthanol préréfrigéré de 70 % à 4 oC pour 1 h (min) à 7 d (max).
- Retirez soigneusement les chambres pour éviter de casser la glissière. Si l'expérience comprend l'immunofluorescence (IF) d'une protéine virale ou hôte avec un anticorps primaire polyclonal, effectuez la FI telle que décrite ci-dessous avant de passer à l'ARN FISH. Si l'immunofluorescence utilise un anticorps primaire monoclonal, alors exécutez l'immunofluorescence comme décrit dans l'étape 3.3.1 après étape 3.11.
REMARQUE : Utilisez un dispositif d'enlèvement frais ou un dispositif avec très peu d'adhésif de restefourni par le fabricant et soulagez doucement les chambres pour empêcher la glissière de se fissurer. L'utilisation de 70 % d'éthanol comme réactif perméabilisant pendant 4 h réduit considérablement la probabilité de fissuration. Dans le cas d'une fissure, continuez le protocole sur les chambres non touchées par la fissure et soyez conscient du taux d'oxydation plus élevé des glissières scellées imparfaitement (c.-à-d. diminution de la durée de conservation).- Rincer les cellules avec du PBS 1x préréfrigéré et bloquer avec du BSA (albumine de sérum bovin) préréfrigéré à 4 % pendant 30 min à 4 oC.
REMARQUE : L'utilisation de BSA tout au long de ce protocole limite l'étiquetage non spécifique. - Enlever la solution de blocage et incuber les cellules avec 1:200 ou un autre anticorps primaire polyclonal dans 0.1% BSA/1x PBS pendant 1 h à 4 oC. Ensuite, laver trois fois avec 1x PBS.
REMARQUE : Un anticorps10 pour la détection du SSB/ORF6 (protéine virale de liaison d'ADN unique-échoué) a été employé à la dilution 1:200. - Incuber les cellules avec un anticorps secondaire avec fluorophore compatible avec l'anticorps de détection FISH pendant 1 h à 4 oC. Laver trois fois avec 1x PBS. Ensuite, fixer avec 4% de formaldéhyde/ 1x PBS pour 10-15 min et perméabilize avec soit Triton-X ou 70% d'éthanol comme décrit précédemment avant de procéder à FISH. Couvrir la glissière de papier d'aluminium pour préserver le signal fluorescent et prévenir le photoblanchiment.
- Rincer les cellules avec du PBS 1x préréfrigéré et bloquer avec du BSA (albumine de sérum bovin) préréfrigéré à 4 % pendant 30 min à 4 oC.
- Laver les cellules avec 2x SSC (citrate de sodium salin) une fois, puis appliquer 45 L de solution d'hybridation composée de 50% de formamide, 10% de sulfate de dextran, 2x SSC, 0,1% BSA, 500 g/mL d'ADN de sperme de saumon, 125 g/mL E. coli tRNA, et 1 mM vanadyl ribonucleoside complexes. Incuber pendant 1 h à 37 oC dans une chambre d'humidité qui peut être un plat Petri de 150 mm avec des lingettes stériles humidifiées.
REMARQUE : Préparer une solution d'hybridation fraîche au moins une heure avant l'utilisation. Dissoudre d'abord le sulfate de dextran dans l'eau, en tourbillonnant fréquemment et en couvant dans un bain d'eau de 37 oC. - Calculez pour avoir une concentration suggérée de 25 oligonucléotides m dans une solution d'hybridation de 35 uL par chambre. Ajuster la concentration d'oligonucléotide antisens au besoin. Ajouter de l'eau distillée aux oligonucléotides pour porter le volume de dénaturation à 10 l.
REMARQUE : À la suite de la réaction d'étiquetage, les oligonucléotides sont stockés dans la solution étanche contenant 0,18 M de cacodylate de potassium, 23 mM Tris-HCl, 0,23 mg/mL BSA, 4,5 mM CoCl2, 18 mM EDTA, 2,7 mM K-phosphate, et 6,8 mM KCl, 45 M 2-Mer 0,02% Triton X-100, et 2% de glycérol. Les concentrations sont suffisamment élevées pour que la dilution avec de l'eau amène la solution de dénaturation à des concentrations proches de 1x TE (10 mM Tris-HCl et 1 mM EDTA), un tampon standard de dénaturation de l'oligonucléotide. - Dénaturer les oligonucléotides étiquetés DIG- et/ou Alexa Fluor 594 à 95 oC pendant 5 min. Ajoutez ensuite une solution d'hybridation fraîche de 35 L par chambre prévue aux oligonucléotides dénaturés. Si vous effectuez un double FISH, les deux ensembles d'oligonucléotides anti-sens peuvent être dénaturés et hybridés ensemble.
- Retirez la solution de pré-hybridation, puis ajoutez la solution d'hybridation contenant les oligonucléotides étiquetés aux cellules. Incuber toute la nuit dans la chambre d'humidité à 37 oC avec du papier d'aluminium pour protéger les oligonucléotides étiquetés fluorophore.
REMARQUE : L'incubation doit être d'au moins 10 h et pas plus de 24 h. - Le lendemain, lavez les cellules deux fois avec 2x SSC pendant 10 min à 37 oC, puis deux fois avec 1x SSC pendant 10 min à 25 oC.
- Fixer les cellules avec du formaldéhyde préréfrigéré 4%/1x PBS pendant 10-15 min sur la glace. Ensuite, lavez les cellules avec du PBS trois fois et perméabilize pendant 1 h avec de l'éthanol pré-réfrigéré 70% ou pendant 10 min avec pré-réfrigéré 0,5% Triton-X/1x PBS à 4 oC.
- Incuber les cellules avec 1:200 anti-DIG FITC en BSA/1x PBS pré-réfrigéré s'est réfrigérer pendant 1 h à 4 oC. Retirez la solution d'anticorps et lavez-la trois fois avec 1x PBS.
- Fixer avec du formaldéhyde préréfrigéré 4%/1x PBS pendant 10-15 min à 4 oC, puis laver trois fois avec 1x PBS. Si vous effectuez l'immunofluorescence d'un hôte ou d'une protéine virale avec un anticorps primaire monoclonal, perméabilisez les cellules et exécutez ensuite le protocole DE FI décrit à l'étape 3.3.1. Sinon, procéder à la coloration DAPI.
- Incuber les cellules avec 0,4 G/mL DAPI en Pré-réfrigéré 0,5% Triton-X/1x PBS pendant 15 min sur la glace, puis laver trois fois avec 1x PBS.
- Montez des glissières avec des perles fluorescentes (facultatif) et un milieu de montage. Ensuite, scellez la glissière à la glissière avec du vernis à ongles transparent.
- À l'aide d'un microscope confocal, recueillir des images des échantillons dans l'heure à une semaine d'effectuer le protocole à 630x grossissement. Appliquer plusieurs couches de vernis à ongles pour sceller la feuille de couverture et de prolonger la vie fluorophore en réduisant le taux d'oxydation.
REMARQUE : N'utilisez pas de support de montage contenant du DAPI. Lors de la collecte des images, inclure la barre d'échelle sur chaque image pour une quantification ultérieure. Les perles fluorescentes servent de contrôle de l'intensité de la fluorescence entre les glissements et les préparations d'échantillons11. Acquérir des images à la section médiane de la cellule pour une quantification bidimensionnelle (2D) à l'étape 4.
4. Quantification des images FISH et IF pour mettre en évidence la localisation subcellulaire et pour déterminer le rapport nucléocytoplasmique de fluorescence
- Effectuez l'analyse d'image sur une pile assemblée des différentes images fluorescentes et fusionnées pour assurer la cohérence. Définir l'échelle du logiciel d'analyse d'images à l'aide de la barre d'échelle incluse lors de la collecte des images.
- Pour quantifier l'intensité de la fluorescence sur plusieurs canaux et en référence à la tache DAPI nucléaire, utilisez un outil de ligne et une fonction de profil de parcelle. Indiquez ensuite la ligne en permanence sur une copie de l'image à l'aide de marqueurs qui n'entravent pas ou n'influencent pas le jugement du spectateur.
- Établir des critères pour guider l'endroit où la ligne est tracée, comme une trace qui capture une diversité de caractéristiques topographiques, de pics et de vallées, le long d'un axe central ou d'une ligne qui ne traverse pas les zones sursaturées.
REMARQUE : Ces traces linéaires représentent la fluorescence brute dans une cellule et se limitent donc à des comparaisons des emplacements d'une tache, et non de l'intensité. Pour comparer les intensités d'une même tache entre les diapositives, les traitements ou les préparations, ajoutez une perle fluorescente à la glissière comme contrôle interne pendant l'étape 3.13. La perle fluorescente doit être ajoutée pendant le processus de montage et détectée avec les mêmes réglages sur le laser d'excitation et le tube photomultiplicateur (confocal).
- Établir des critères pour guider l'endroit où la ligne est tracée, comme une trace qui capture une diversité de caractéristiques topographiques, de pics et de vallées, le long d'un axe central ou d'une ligne qui ne traverse pas les zones sursaturées.
- Pour quantifier un changement dans la localisation subcellulaire, calculer les ratios nucléocytoplasmiques des cellules subissant différents traitements.
- Mesurez la zone et l'intensité de fluorescence brute du noyau et du cytoplasme à l'aide de la tache DAPI nucléaire pour définir la limite intérieure. Inclure les contrôles nucléaires et cytoplasmiques tels qu'un ARN nucléaire (p. ex., ARN KSHV PAN) et l'ARN cytoplasmique (p. ex., l'ARNm GAPDH hôte). De plus, calculez l'intensité de fond pour trois zones semblables à des cellules et calculez les valeurs moyennes par pixel ou2m .
REMARQUE : Les valeurs d'intensité ont tendance à manquer d'unités et le terme « unités » est donc utilisé. - Normaliser les valeurs d'intensité brute nucléaire et cellulaire en déterminant d'abord l'arrière-plan moyen pour la même zone, puis en soustrayant cette valeur individualisée de l'intensité brute de la zone.
- Par exemple, un noyau d'une cellule B lytique a une superficie de 133,4 m2 et une intensité brute de 75976 unités, tandis que l'intensité de fond pour le même signal fluorescent a été déterminée à 0,67 unité par m2. L'intensité nucléaire normalisée serait
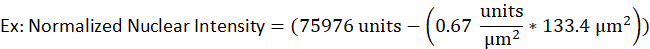
- Par exemple, un noyau d'une cellule B lytique a une superficie de 133,4 m2 et une intensité brute de 75976 unités, tandis que l'intensité de fond pour le même signal fluorescent a été déterminée à 0,67 unité par m2. L'intensité nucléaire normalisée serait
- Entrez les valeurs dans l'équation suivante.

REMARQUE : Ce calcul contrôle les changements dans la zone subcellulaire. L'induction lytique et les traitements médicamenteux peuvent agrandir le noyau ou modifier la taille de la cellule, respectivement. - Pour interpréter les résultats, créez une parcelle de moustaches boîte. Une distribution égale du signal fluorescent serait proche de zéro, tandis qu'une distribution nucléaire favoriserait une valeur de ratio positive et une distribution cytoplasmique tendrait vers une valeur de ratio négative.
- Mesurez la zone et l'intensité de fluorescence brute du noyau et du cytoplasme à l'aide de la tache DAPI nucléaire pour définir la limite intérieure. Inclure les contrôles nucléaires et cytoplasmiques tels qu'un ARN nucléaire (p. ex., ARN KSHV PAN) et l'ARN cytoplasmique (p. ex., l'ARNm GAPDH hôte). De plus, calculez l'intensité de fond pour trois zones semblables à des cellules et calculez les valeurs moyennes par pixel ou2m .
Résultats
Les méthodes FISH et IF détaillées dans ce manuscrit sont présentées à la figure 1 ainsi que la quantification des résultats par ligne des traces d'intensité fluorescente. Les résultats présentés ici sont semi-quantitatifs et offrent un aperçu de la localisation, plutôt que dans les comparaisons entre les intensités de différentes taches fluorescentes parce que les expériences n'ont pas inclus une perle fluorescente dans la préparation de dia...
Discussion
Le protocole décrit dans ce rapport peut être adapté à différents types de cellules et inclut des étapes pour le FISH et le FISH d'ARN doubles avec IF utilisant les anticorps primaires monoclonaux et polyclonal. Bien que les diapositives préparées soient généralement représentées à l'effile avec un microscope confocal, l'imagerie peut être réalisée avec un microscope STED (épuisement des émissions stimulées) après des modifications de la concentration accrue des anticorps et d'un milieu de montage dif...
Déclarations de divulgation
Les auteurs n'ont aucun conflit d'intérêts à divulguer.
Remerciements
Nous remercions Jonathan Rodenfels, Kazimierz Tycowski et Johanna B. Withers pour leurs conseils sur l'analyse des données. Nous remercions également G. Hayward pour l'anticorps anti-SSB. Ce travail a été soutenu par des subventions T32GM007223 et T32AI055403 des National Institutes of Health (to TKV) et des NIH (CA16038) (à JAS). JAS est un chercheur du Howard Hughes Medical Institute. Les figures 1-3 et le tableau 1 ont été reproduits avec la permission de l'American Society for Microbiology sous une licence Creative Commons Attribution de la publication suivante : Vallery, T. K., Withers, J. B., Andoh, J. A., Steitz, J. A. Kaposi's Sarcoma-Associated L'accumulation d'ARNm d'herpèsvirus dans le foci nucléaire est influencée par la réplication virale d'ADN et l'ARN nucléaire polyadenylated de non-codage viral. Journal of Virology. 92 (13), doi:10.1128/JVI.00220-18, (2018).
matériels
| Name | Company | Catalog Number | Comments |
| AlexaFluor594-5-dUTP | Life Technologies | C1100 | |
| anti-DIG FITC | Jackson Lab Immunologicals | 200-092-156 | |
| Anti-Rabbit Secondary AlexaFluor594 Monoclonal Antibody | Invitrogen | A-11037 | Goat |
| Anti-SSB Antibody | N/A | N/A | Ref. Chiou et al. 2002 |
| BLASTn | NIH NCBI | N/A | Free Sequence Alignment Software |
| Dextran Sulfate | Sigma Aldrich | D8906 | Molecular Biology Grade |
| DIG-Oligonucleotide Tailing Kit | Sigma Roche | #03353583910 | 2nd Gen |
| Eight-Chamber Slides | Nunc Lab Tek II | #154453 | Blue seal promotes surface tension but separation by clear gel is also available. |
| Formamide | Sigma Aldrich | F9037 | Molecular Biology Grade |
| GAPDH Probes | Stellaris | SMF-2019-1 | Compatible with protocol, Quasar 670 |
| ImageJ | NIH, Bethesda, MD | N/A | Free Image Analysis Software, [http:rsb.info.nih.gov/ij/] |
| OligoAnalyzer | IDT | N/A | Free Oligonucleotide Analyzer |
| pcDNA3 | Invitrogen | A-150228 | |
| pmaxGFP | Amaxa | VDF-1012 | |
| Poly L-Lysine | Sigma Aldrich | P8920 | |
| Terminal Transferase | Sigma Roche | #003333574001 | |
| Vanadyl Ribonucleoside Complexes | NEB | S1402S | |
| Vectashield | Vector Laboratories, Inc. | H-1000 | DAPI within the mounting media scatters the light and reduces contrast. |
Références
- Amen, M. A., Griffiths, A. Packaging of Non-Coding RNAs into Herpesvirus Virions: Comparisons to Coding RNAs. Frontiers in Genetics. 2, 81 (2011).
- Schmid, M., Speiseder, T., Dobner, T., Gonzalez, R. A. DNA virus replication compartments. Journal of Virology. 88 (3), 1404-1420 (2014).
- Pawlicki, J. M., Steitz, J. A. Primary microRNA transcript retention at sites of transcription leads to enhanced microRNA production. Journal of Cell Biology. 182 (1), 61-76 (2008).
- Borah, S., Darricarrere, N., Darnell, A., Myoung, J., Steitz, J. A. A viral nuclear noncoding RNA binds re-localized poly(A) binding protein and is required for late KSHV gene expression. Public Library of Science Pathogens. 7 (10), e1002300 (2011).
- Tycowski, K. T., Shu, M. D., Borah, S., Shi, M., Steitz, J. A. Conservation of a triple-helix-forming RNA stability element in noncoding and genomic RNAs of diverse viruses. Cell Reports. 2 (1), 26-32 (2012).
- Weinberg, R. A., Penman, S. Small molecular weight monodisperse nuclear RNA. Journal of Molecular Biology. 38 (3), 289-304 (1968).
- Myoung, J., Ganem, D. Generation of a doxycycline-inducible KSHV producer cell line of endothelial origin: maintenance of tight latency with efficient reactivation upon induction. Journal of Virology Methods. 174 (1-2), 12-21 (2011).
- Brulois, K. F., et al. Construction and manipulation of a new Kaposi's sarcoma-associated herpesvirus bacterial artificial chromosome clone. Journal of Virology. 86 (18), 9708-9720 (2012).
- Sturzl, M., Gaus, D., Dirks, W. G., Ganem, D., Jochmann, R. Kaposi's sarcoma-derived cell line SLK is not of endothelial origin, but is a contaminant from a known renal carcinoma cell line. International Journal of Cancer. 132 (8), 1954-1958 (2013).
- Chiou, C. J., et al. Patterns of gene expression and a transactivation function exhibited by the vGCR (ORF74) chemokine receptor protein of Kaposi's sarcoma-associated herpesvirus. Journal of Virology. 76 (7), 3421-3439 (2002).
- Cole, R. W., Jinadasa, T., Brown, C. M. Measuring and interpreting point spread functions to determine confocal microscope resolution and ensure quality control. Nature Protocols. 6 (12), 1929-1941 (2011).
- Nakamura, H., et al. Global changes in Kaposi's sarcoma-associated virus gene expression patterns following expression of a tetracycline-inducible Rta transactivator. Journal of Virology. 77 (7), 4205-4220 (2003).
- Majerciak, V., Yamanegi, K., Zheng, Z. M. Gene structure and expression of Kaposi's sarcoma-associated herpesvirus ORF56, ORF57, ORF58, and ORF59. Journal of Virology. 80 (24), 11968-11981 (2006).
- Sun, R., Lin, S. F., Gradoville, L., Miller, G. Polyadenylylated nuclear RNA encoded by Kaposi sarcoma-associated herpesvirus. Proceedings of the National Academy Sciences U S A. 93 (21), 11883-11888 (1996).
- Vallery, T. K., Withers, J. B., Andoh, J. A., Steitz, J. A. Kaposi's Sarcoma-Associated Herpesvirus mRNA Accumulation in Nuclear Foci Is Influenced by Viral DNA Replication and Viral Noncoding Polyadenylated Nuclear RNA. Journal of Virology. 92 (13), (2018).
- Borah, S., Nichols, L. A., Hassman, L. M., Kedes, D. H., Steitz, J. A. Tracking expression and subcellular localization of RNA and protein species using high-throughput single cell imaging flow cytometry. RNA. 18 (8), 1573-1579 (2012).
- Bruce, A. G., et al. Quantitative Analysis of the KSHV Transcriptome Following Primary Infection of Blood and Lymphatic Endothelial Cells. Pathogens. 6 (1), (2017).
- Chen, C. P., et al. Kaposi's Sarcoma-Associated Herpesvirus Hijacks RNA Polymerase II To Create a Viral Transcriptional Factory. Journal of Virology. 91 (11), (2017).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon