È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Quantitative Fluorescence In Situ Hybridization (FISH) e Immunofluorescence (IF) di prodotti genici specifici nelle cellule infette da KSHV
In questo articolo
Riepilogo
Descriviamo un protocollo che utilizza la fluorescenza nell'ibridazione situ (FISH) per visualizzare più RNA herpesvirali all'interno di cellule umane lytically infette, sia in sospensione che aderenti. Questo protocollo include la quantificazione della fluorescenza producendo un rapporto nucleocitoplasmatica e può essere esteso per la visualizzazione simultanea di proteine ospite e virali con immunofluorescenza (IF).
Abstract
Le informazioni meccanicistiche arrivano da un attento studio e quantificazione di RNA e proteine specifici. Le posizioni relative di queste biomolecole in tutta la cellula in momenti specifici possono essere catturate con la fluorescenza nell'ibridazione situ (FISH) e l'immunofluorescenza (IF). Durante l'infezione da herpesvirus littico, il virus dirotta la cellula ospite per esprimere preferibilmente geni virali, causando cambiamenti nella morfologia cellulare e nel comportamento delle biomolecole. Le attività littiche sono incentrate in fabbriche nucleari, definite compartimenti di replicazione virale, che sono distinguibili solo con FISH e IF. Qui descriviamo un protocollo adattabile delle tecniche RNA FISH e IF per le cellule infettate associate al sarcoma (KSHV) di Kaposi, sia aderenti che in sospensione. Il metodo include passaggi per lo sviluppo di specifici oligonucleotidi anti-senso, doppio RNA FISH, RNA FISH con IF, e calcoli quantitativi di intensità di fluorescenza. Questo protocollo è stato applicato con successo a più tipi di cellule, cellule non infette, cellule latenti, cellule littiche, corsi temporali e cellule trattate con inibitori per analizzare le attività spatiotemporali di RNA specifici e proteine sia dall'ospite umano che il kSHV.
Introduzione
Nella loro fase litetica (attiva), gli herpesvirus dirottano la cellula ospite, causando cambiamenti nella morfologia cellulare e nella localizzazione delle molecole biologiche, per produrre virioni. La base delle operazioni è il nucleo, dove il genoma virale del DNA a doppio filamento viene replicato e confezionato in un guscio proteico, chiamato capside1. Per cominciare, il virus esprime le proprie proteine, dirottando i macchinari ospiti e impedendo l'espressione di geni ospiti non essenziali, un processo chiamato effetto di chiusura dell'ospite. La maggior parte di questa attività è localizzata in specifiche regioni nucleari prive di 4,6-diamidino_2 -fenylindole (DAPI) chiamate compartimenti di replicazione virale, composti da proteine host e virali, RNA e DNA virale2. La cella viene revisionata per fornire spazio e risorse per i compartimenti di replicazione e quindi l'assemblaggio di capsidi virali. Una volta che il capside esce dal nucleo, non è chiaro come il capside sia avvolto nel citoplasma per produrre una particella virale legata alla membrana, nota anche come virione. La comprensione della localizzazione e degli spostamenti spaziali delle biomolecole sia dell'ospite che di quella virale durante la fase littica fornisce una visione più profonda meccanistica della disposizione del compartimento di replicazione, dell'effetto di spegnimento dell'ospite, della via virione-uscita e di altri processi correlati all'infezione da herpesviral e alla replicazione.
Attualmente il metodo migliore per rilevare e studiare questi cambiamenti è la visualizzazione di proteine e RNA nelle cellule infette con immunofluorescenza (IF) e l'ibridazione fluorescente in situ (FISH), rispettivamente. L'uso di un corso temporale con queste tecniche rivela la localizzazione delle biomolecole nei punti chiave della fase littica o semplicemente, dati spatiotemporali. FISH e IF completano altre tecniche biochimiche, come l'inibizione di un processo cellulare (ad esempio, l'inibizione della replicazione del DNA virale), RT-qPCR (reazione a catena polimerasi in tempo reale), il sequenziamento dell'RNA, le macchie settentrionali, la spettrometria di massa, l'oscillazione occidentale e l'analisi della produzione di DNA virale, che può fornire un quadro più globale delle attività cellulari.
Abbiamo sviluppato strategie di RNA FISH per esaminare i prodotti dell'RNA da geni specifici e un'analisi computazionale che calcola quantitativamente il rapporto nucleocitoplasmatico di un prodotto genetico specifico. La preparazione di esempio, modificata da pubblicazioni precedenti da Steitz e colleghi3,4, è relativamente semplice e può essere utilizzata sia per le cellule aderenti che per le cellule sospese. Il protocollo è anche adattabile per l'uso simultaneo di più strategie DI RNA FISH (double RNA FISH) o RNA FISH con strategie IF. Lo sviluppo di una strategia FISH specifica è impegnativo, ma vengono delineati suggerimenti per migliorare il successo. L'analisi dei dati qui descritta è quantitativa se vengono utilizzate perline fluorescenti e forti indicatori di confini di compartimenti e offre ulteriori informazioni sulle micrografie, informazioni che eliminano la distorsione di osservazione. Il protocollo dettagliato è progettato per cellule latenti e litine infettate dall'herpesvirus associato al sarcoma di Kaposi (KSHV) e può essere utilizzato con cellule non infette o cellule infettate da altri herpesvirus5. I metodi di quantitazione sono applicabili agli studi sui spostamenti nucleocitoplasmatici o sulla rilocalizzazione tra compartimenti subcellulari nella maggior parte delle cellule.
Protocollo
1. Progettazione di oligonucleotidi antisenso a fluorescenza in situ (FISH) per rilevare una specifica trascrizione dell'herpesvirale
- Selezionare da 25 a 40 nt segmenti dalla sequenza di RNA di interesse e convertire in modo anti-senso. Una strategia FISH di successo può contenere da uno fino a dieci o più oligonucleotidi anti-senso diversi. Quando si selezionano le sequenze, tenere presente quanto segue:
- Se l'RNA di interesse contiene una regione di ripetizione unica, sfrutta questa funzione e progetta un oligonucleotide antisenso per indirizzare la sequenza di ripetizione.
NOTA: Tycowski e colleghi5 forniscono un esempio di questa strategia con rhesus rhadinovirus (RRV) RNA nucleare polianegato (PAN). - Se l'RNA di interesse contiene un sito di legame proteico noto o una struttura del ciclo stelo, progettare oligonucleotidi che evitano queste regioni.
- A seconda degli obiettivi degli esperimenti, considerare la sequenza intronica e se progettare o meno un oligonucleotide anti-senso per esso.
- Se l'RNA di interesse contiene una regione di ripetizione unica, sfrutta questa funzione e progetta un oligonucleotide antisenso per indirizzare la sequenza di ripetizione.
- Eseguire semplici analisi computazionali sulle sequenze anti-senso selezionate per garantire specificità di legame e ridurre l'aggregazione dell'oligonucleotide anti-senso.
- Le sequenze devono essere ricche di circa il 50% di GC (alto contenuto di guanina e citosina) e avere una temperatura di fusione compresa tra 60 e 70 gradi centigradi.
- Utilizzare uno strumento analizzatore di sequenze per selezionare le sequenze che non si autodismerano o formano forcine con temperature di fusione superiori a 37 gradi centigradi, la temperatura di ibridazione.
- Eseguire una ricerca NCBI BLASTn (National Center for Biotechnology Information Basic Alignment Tool for nucleotides) delle sequenze selezionate sia contro i trascriptomi host che virali utilizzando l'impostazione "un po' simile". Questa ricerca identificherà oligonucleotidi anti-sense unici che probabilmente non si legheranno ad altre trascrizioni dell'ospite o dei virali.
NOTA: se i trascrittomi non sono disponibili, eseguire la ricerca BLAST con le sequenze genomiche. È ideale se le ricerche vengono eseguite sulle sequenze del virus isolato dalle cellule infette utilizzate nell'esperimento perché i ceppi selvatici tendono a diversificarsi e contengono una combinazione di sequenze da diversi ceppi di laboratorio.
- Ordinare oligonucleotidi di DNA purificato corrispondenti alla sequenza anti-senso che è stata verificata computazionalmente per essere unica e suscettibile di legarsi all'RNA bersaglio. Non è necessario introdurre modifiche speciali negli oligonucleotidi.
- Testare gli oligonucleotidi anti-senso progettati per la specificità di legame da FISH e Northern blot.
- Utilizzando una linea cellulare non infetta della stessa specie ospite (ad esempio, 293T) e idealmente dello stesso tipo di cellula, condurre una trasfezione con un plasmide che esprime l'RNA di interesse da un promotore robusto (CMV, citomegalovirus) e uno con il vettore vuoto (ad esempio, pcDNA3). Utilizzare un controllo positivo per la trasfezione, ad esempio la co-trasfezione con un plasmid GFP (proteina fluorescente verde) plasmid (ad esempio, pmaxGFP) o il vettore contenente GFP.
NOTA: È importante evitare le linee cellulari che sono state immortalate utilizzando il virus Epstein-Barr (EBV) dell'herpesvirus (EBV) poiché ci sono somiglianze di sequenza tra le herpesvirus. - Eseguire FISH come descritto nella sezione 3 su entrambi i set di cellule con gli oligonucleotidi anti-senso come descritto in questo protocollo. Deduci i candidati di successo confrontando gli esperimenti FISH con singoli, coppie o serie di oligonucleotidi anti-senso. Utilizzare un controllo positivo per il protocollo FISH, come U2 snRNA (small nuclear RNA) FISH, presente a 500.000 copie per nucleo cellulare umano6 (Tabella 1).
- Il segnale fluorescente deve essere specifico e forte nella cellula contenente l'RNA di interesse. Progettare oligonucleotidi anti-senso aggiuntivi per rafforzare il segnale e rimuovere gli oligonucleotidi anti-senso che si legano in modo non specifico dalla considerazione. La potenza del segnale deve essere al di sopra dello sfondo e dell'autofluorescenza.
- Testare la specificità di legame per macchia settentrionale.
- Utilizzando una linea cellulare non infetta della stessa specie ospite (ad esempio, 293T) e idealmente dello stesso tipo di cellula, condurre una trasfezione con un plasmide che esprime l'RNA di interesse da un promotore robusto (CMV, citomegalovirus) e uno con il vettore vuoto (ad esempio, pcDNA3). Utilizzare un controllo positivo per la trasfezione, ad esempio la co-trasfezione con un plasmid GFP (proteina fluorescente verde) plasmid (ad esempio, pmaxGFP) o il vettore contenente GFP.
2. Oligonucleotide e preparazione delle cellule
- Seguendo le istruzioni del produttore, utilizzare il transferasi terminale per etichettare gli oligonucleotidi anti-senso con diossigenina (DIG)-dUTP o, se fortemente legante, direttamente con un nucleotide fluorescente come Alexa Fluor 594-5-dUTP. Dopo l'etichettatura, non è necessaria ulteriore purificazione. Conservare oligonucleotidi etichettati a -20 gradi centigradi fino a diversi anni e in lamina di latta se etichettati direttamente per evitare il fotosbiancamento.
AVVISO: La soluzione di etichettatura contiene un materiale tossico, cacodialato di potassio. Gestire le reazioni di etichettatura con i guanti.
NOTA: Per risparmiare risorse, diversi oligonucleotidi anti-senso possono essere etichettati in una sola reazione. Questo protocollo utilizza l'etichettatura a 3'-end. L'etichettatura interna è difficile perché il gruppo chimico (ad esempio, DIG o fluoroforo) viene catturato o non è in grado di entrare nel sito attivo di una polimerasi del DNA. Un esperimento con due RNA diversi può essere eseguito utilizzando oligonucleotidi anti-senso direttamente etichettati e immunofluorescenza anti-DIG con un fluoroforo diverso (ad esempio, FITC (fluorescein) o Alexa Fluor 488 con Alexa Fluor 594). - Aderire le cellule alle diapositive di otto camere.
NOTA: i vetrini a otto camere consentono diversi esperimenti simultanei riducendo al minimo le risorse preziose come gli anticorpi. Un'alternativa ai vetrini a otto camere è una piastra di coltura tissutale a sei pozzelmi con copricapi standard (22 mm x 22 mm) che sono entrambi sterili. Una disposizione simile è possibile con coperture circolari e una piastra di coltura tissutale 24-pozzo. Per entrambi, aumentare i volumi menzionati in questo protocollo rispettivamente di 10-15x (ad esempio, soluzione di ibridazione da 1,75 mL) e 4x (ad es., 600 soluzione di ibridazione l).- Per le cellule litiliche aderenti, utilizzare 1x trypsin/PBS a 37 e 5% di CO2 per 10 min per sospendere le cellule e diluire al 60% la confluenza.
NOTA: le linee cellulari aderenti utilizzate negli esperimenti FISH includevano 293T, iSLK.2197e iSLK-BAC36 celle8. - Applicare 200 litri di sospensione cellulare in ogni camera dei vetrini sterili a otto camere e consentire la crescita del seme per 12-24 h a 37 e 5% di CO2. Regolare in base alle esigenze per le cellule a crescita lenta o veloce e per le cellule che sono facilmente danneggiate dalla trypsin.
NOTA: L'obiettivo è quello di avere celle distanziate uniformemente saldamente al vetrino. Considerare l'induzione fase litica dopo l'adesione se le cellule littiche sono fragili. Le conclusioni tratte da esperimenti con cellule iSLK sono limitate9. - Per le cellule sospese littiche, pre-trattare i vetrini a otto camere con 1:10 poli L-lisina per 5 min sotto il cofano di coltura dei tessuti. Lasciare quindi asciugare i vetrini durante la notte a temperatura ambiente o 1 h a 65 gradi centigradi. Incubare 800 celle littiche a una concentrazione di 1 x 106 cellule/mL con i vetrini camerati per 30 min a 1 h a 37 e 5% di CO2.
NOTA: Le cellule di sospensione si depositano in un monostrato, attaccandosi alla polil-lisina e quindi le cellule in eccesso non sono una preoccupazione rispetto alle cellule aderenti. Le cellule litiliche che formano grappoli d'uva, se possibile, dovrebbero essere separate da un leggero vortice o da mezzi chimici. Certo, gli autori non hanno avuto molto successo con tali raccomandazioni nel caso di cellule littiche BJAB-RRV-GFP. Se le cellule sospese non aderiscono bene, prendere in considerazione l'aumento del tempo o la concentrazione dell'incubazione poli-lisina.
- Per le cellule litiliche aderenti, utilizzare 1x trypsin/PBS a 37 e 5% di CO2 per 10 min per sospendere le cellule e diluire al 60% la confluenza.
3. Fissazione, immunofluorescenza (opzionale), ibridazione e visualizzazione di RNA virali
- Rimuovere i supporti e le cellule in eccesso. In questo protocollo, utilizzare l'aspirazione del vuoto per rimuovere le soluzioni e micropipetta delicata per aggiungere soluzioni.
NOTA: La resistenza di un vuoto può essere ridotta posizionando una punta di micropipette da 200 l sopra la pipetta Pasteur in vetro. Sostituire la punta della micropipetta tra le fasi di lavaggio per evitare la contaminazione. Ogni passo di lavaggio deve essere eseguito rapidamente perché è imperativo che le cellule non si asciughino mai. - Immediatamente, fissare le cellule con formaldeide/PBS (salina tamponata da fosfato) sul ghiaccio per 30 min.
- Percentoabilizzare le celle fisse con 200 l di 0,5% Triton-X/PBS (fosfato buffersalto salina) per 10 min sul ghiaccio o 750 L di etanolo pre-raffreddato a 4 gradi centigradi per 1 h (min) a 7 d (max).
NOTA: Raccogli campioni di proteine, RNA totale e DNA genomico nel punto di fissazione per garantire la coerenza tra immagini e analisi biochimiche. Tutti gli osisti in tutto questo protocollo vengono eseguiti nello stesso modo, se non diversamente specificato. 70% etanolo allenta la colla tra le camere e lo scivolo, che facilita la separazione successiva, e fornisce anche una pausa significativa nel protocollo. Tuttavia, utilizzare pellicola di paraffina intorno al vetrino della camera per ridurre l'evaporazione e controllare il livello dell'etanolo in ogni camera circa ogni 8 h. 70% etanolo appiattisce anche le cellule, rendendo un'immagine più nitida, mentre Triton-X non disidrata le cellule e cambia il dimensioni della cella.
- Percentoabilizzare le celle fisse con 200 l di 0,5% Triton-X/PBS (fosfato buffersalto salina) per 10 min sul ghiaccio o 750 L di etanolo pre-raffreddato a 4 gradi centigradi per 1 h (min) a 7 d (max).
- Rimuovere le camere con attenzione per evitare di screpolature del vetrino. Se l'esperimento include l'immunofluorescenza (IF) di una proteina virale o ospite con un anticorpo primario policlonale, eseguire l'IF come descritto di seguito prima di procedere all'RNA FISH. Se l'immunofluorescenza utilizza un anticorpo primario monoclonale, eseguire l'immunofluorescenza come descritto al punto 3.3.1 dopo il passaggio 3.11.
NOTA: Utilizzare un dispositivo di rimozione fresco o uno con pochissimo adesivo resimo fornito dal produttore e facilitare delicatamente le camere fuori per evitare che il vetrino da crepitio. L'utilizzo del 70% di etanolo come reagente permeabilizzante per 4 h riduce notevolmente la probabilità di fessurazione. Nel caso di una crepa, continuare il protocollo sulle camere non interessate dalla fessura ed essere consapevoli del più alto tasso di ossidazione di vetrini imperfettamente sigillati (cioè diminuzione della durata di stoccaggio).- Sciacquare le cellule con 1x PBS pre-raffreddato e bloccare con bsA (albumina del siero bovino) pre-raffreddata 4% /1x PBS per 30 min a 4 gradi centigradi.
NOTA: l'uso di BSA in questo protocollo limita l'etichettatura non specifica. - Rimuovere la soluzione di blocco e incubare le cellule con 1:200 o un altro anticorpo primario policlonale in 0.1% BSA/1x PBS per 1 h a 4 gradi centigradi. Quindi lavare tre volte con 1x PBS.
NOTA: un anticorpo10 per la rilevazione di SSB/ORF6 (proteina virale per legame del DNA a singolo filamento) è stato utilizzato a 1:200 diluizione. - Incubare le cellule con un anticorpo secondario con fluoroforo compatibile con l'anticorpo che rileva FISH per 1 h a 4 gradi centigradi. Lavare tre volte con 1x PBS. Quindi fissare con 4% formaldeide/1x PBS per 10-15 min e permeabilizzare con triton-X o 70% etanolo come descritto in precedenza prima di procedere a FISH. Coprire il vetrino con lamina di stagnoper preservare il segnale fluorescente ed evitare il fotosbiancamento.
- Sciacquare le cellule con 1x PBS pre-raffreddato e bloccare con bsA (albumina del siero bovino) pre-raffreddata 4% /1x PBS per 30 min a 4 gradi centigradi.
- Lavare le cellule con 2x SSC (citrato di sodio salina) una volta e quindi applicare 45 -L di soluzione di ibridazione costituita da 50% formamide, 10% dextran solfato, 2x SSC, 0,1% BSA, 500 g/mL di spermatozoi di salmone, 125 g/mL E. colitRNA e 1 m. vanadyl ribonucleo Complessi. Incubare per 1 h a 37 gradi centigradi in una camera di umidità che può essere un piatto Petri da 150 mm con salviette sterili inumidite.
NOTA: preparare una nuova soluzione di ibridazione almeno un'ora prima dell'uso. Sciogliere prima il solfato dextran in acqua, vortice frequentemente e incubare in un bagno d'acqua a 37 gradi centigradi. - Calcolare per avere una concentrazione suggerita di 25 oligonucleotidi M in soluzione di ibridazione 35-uL per camera. Regolare la concentrazione di oligonucleotide anti-senso in base alle esigenze. Aggiungere acqua distillata alle oligonucleotidi per portare il volume di denaturazione a 10 gradi.
NOTA: dopo la reazione di etichettatura, gli oligonucleotidi sono conservati nella soluzione di quenched contenente 0,18 M cacodilate di potassio, 23 mM Tris-HCl, 0,23 mg/mL BSA, 4,5 mM CoCl2, 18 mM EDTA, 2,7 mM K-fosfato e 6,8 mM KCl, 45 m 2-Mertothaneo 0,02% Triton X-100 e 2% glicerolo. Le concentrazioni sono abbastanza alte che la diluizione con acqua porterà la soluzione di denaturazione a concentrazioni vicine a 1x TE (10 mM Tris-HCl e 1 mM EDTA), un buffer di denaturazione oligonucleotide standard. - Denaturare gli oligonucleotidi etichettati DIG e/o Alexa Fluor 594 a 95 gradi centigradi per 5 min. Aggiungere quindi 35 gradi di ibridazione freschi per ogni camera prevista agli oligonucleotidi denaturati. Se si esegue il doppio FISH, entrambi i set di oligonucleotidi anti-senso possono essere denaturati e ibridati insieme.
- Rimuovere la soluzione di pre-ibridazione e quindi aggiungere la soluzione di ibridazione contenente gli oligonucleotidi etichettati alle cellule. Incubare durante la notte nella camera di umidità a 37 gradi centigradi con lamina di stagno per proteggere gli oligonucleotidi con etichettatura fluoroforo.
NOTA: L'incubazione deve essere di almeno 10 h e non superiore a 24 h. - Il giorno successivo, lavare le cellule due volte con 2x SSC per 10 min a 37 gradi centigradi e poi due volte con 1x SSC per 10 min a 25 .
- Fissare le cellule con formaldeide 4% pre-raffreddato/1x PBS per 10-15 min sul ghiaccio. Quindi lavare le celle con PBS tre volte e permeabilizzare per 1 h con etanolo pre-raffreddato 70% o per 10 min con pre-raffreddato 0.5% Triton-X/1x PBS a 4 gradi centigradi.
- Incubare le cellule con 1:200 anti-DIG FITC in pre-raffreddato 0,1% BSA/1x PBS per 1 h a 4 gradi centigradi. Rimuovere la soluzione anticorpale e lavare tre volte con 1x PBS.
- Fissare con pre-freddo 4% formaldeide/1x PBS per 10-15 min a 4 gradi centigradi e poi lavare tre volte con 1x PBS. Se si esegue l'immunofluorescenza per un ospite o una proteina virale con un anticorpo primario monoclonale, permetto le cellule e quindi eseguire il protocollo IF delineato nel passaggio 3.3.1. In caso contrario, procedere alla colorazione DAPI.
- Incubare le cellule con DAPI a 0,4 g/mL in TRIton-X/1x PBS pre-raffreddati per 15 min sul ghiaccio e poi lavare tre volte con 1x PBS.
- Montare scivoli con perline fluorescenti (opzionale) e un supporto di montaggio. Quindi sigillare il coperchio alla diapositiva con smalto trasparente.
- Utilizzando un microscopio confocale, raccogliere le immagini dei campioni entro un'ora e una settimana dall'esecuzione del protocollo con ingrandimento 630x. Applicare più strati di smalto per sigillare lo slittamento di copertura e prolungare la durata del fluoroforo riducendo il tasso di ossidazione.
NOTA: non utilizzare un supporto di montaggio contenente DAPI. Quando raccogli le immagini, includi la barra della scala su ogni immagine per una successiva quantificazione. Perline fluorescenti servono come controlli di intensità di fluorescenza tra i vetrini e preparati campione11. Acquisire le immagini nella sezione centrale della cella per la quantificazione bidimensionale (2D) nel passaggio 4.
4. Quantificazione delle immagini FISH e IF per evidenziare la localizzazione subcellulare e per determinare il rapporto nucleocitoplasmatico della fluorescenza
- Eseguire l'analisi delle immagini su una pila assemblata delle varie immagini fluorescenti e unite per garantire la coerenza. Impostare la scala del software di analisi delle immagini utilizzando la barra di scala inclusa quando le immagini sono state raccolte.
- Per quantificare l'intensità della fluorescenza su più canali e in riferimento alla macchia NUCLEARe DAPI, utilizzare uno strumento di linea e una funzione di profilo di trama. Quindi indicare la linea in modo permanente su una copia dell'immagine utilizzando marcatori che non ostacolano o influenzano il giudizio dello spettatore.
- Stabilire criteri per guidare la posizione in cui viene tracciata la linea, ad esempio una traccia che cattura una varietà di caratteristiche topografiche, picchi e valli, lungo un asse centrale o una linea che non attraversa aree supersaturate.
NOTA: Queste tracce di linea raffigurano la fluorescenza grezza in una cella e quindi sono limitate al confronto delle posizioni di una macchia, non dell'intensità. Per confrontare le intensità della stessa macchia tra vetrini, trattamenti o preparazioni, aggiungere una perlina fluorescente alla diapositiva come controllo interno durante la fase 3.13. Il tallone fluorescente deve essere aggiunto durante il processo di montaggio e rilevato con le stesse impostazioni sul laser di eccitazione e sul tubo fotomoltiplicatore (confocale).
- Stabilire criteri per guidare la posizione in cui viene tracciata la linea, ad esempio una traccia che cattura una varietà di caratteristiche topografiche, picchi e valli, lungo un asse centrale o una linea che non attraversa aree supersaturate.
- Per quantificare uno spostamento nella localizzazione subcellulare, calcolare i rapporti nucleocitoplasmatici delle cellule sottoposte a diversi trattamenti.
- Misurare l'area e l'intensità di fluorescenza grezza del nucleo e del citoplasma utilizzando la macchia DAPI nucleare per impostare il confine interno. Includere controlli nucleari e citoplasmamici come un RNA nucleare (ad esempio, KSHV PAN RNA) e RNA citoplasmico (ad esempio, l'mRNA GAPDH ospite). Inoltre, calcolare l'intensità dello sfondo per tre aree simili a celle e calcolare la media dei valori per pixel o .m2.
NOTA: i valori di intensità tendono a mancare di unità e quindi viene utilizzato il termine "unità". - Normalizzare i valori di intensità grezza sia nucleari che cellulari determinando innanzitutto lo sfondo medio per la stessa area e quindi sottraendo tale valore individualizzato dall'intensità grezza dell'area.
- Ad esempio, un nucleo di una cellula B litetica ha un'area di 133,4 m2 e un'intensità grezza di 75976 unità, mentre l'intensità dello sfondo per lo stesso segnale fluorescente è stata determinata per essere 0,67 unità per2. L'intensità nucleare normalizzata sarebbe
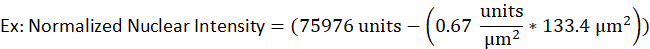
- Ad esempio, un nucleo di una cellula B litetica ha un'area di 133,4 m2 e un'intensità grezza di 75976 unità, mentre l'intensità dello sfondo per lo stesso segnale fluorescente è stata determinata per essere 0,67 unità per2. L'intensità nucleare normalizzata sarebbe
- Immettere i valori nell'equazione seguente.

NOTA: questo calcolo controlla le modifiche nell'area subcellulare. L'induzione litetica e i trattamenti farmacologici possono ingrandire il nucleo o modificare le dimensioni della cellula, rispettivamente. - Per interpretare i risultati, creare un grafico baffi a scatola. Una distribuzione equa del segnale fluorescente sarebbe prossima allo zero, mentre una distribuzione nucleare favorirebbe un rapporto positivo e una distribuzione citoplasmica andrebbero verso un valore di rapporto negativo.
- Misurare l'area e l'intensità di fluorescenza grezza del nucleo e del citoplasma utilizzando la macchia DAPI nucleare per impostare il confine interno. Includere controlli nucleari e citoplasmamici come un RNA nucleare (ad esempio, KSHV PAN RNA) e RNA citoplasmico (ad esempio, l'mRNA GAPDH ospite). Inoltre, calcolare l'intensità dello sfondo per tre aree simili a celle e calcolare la media dei valori per pixel o .m2.
Risultati
I metodi FISH e IF descritti in questo manoscritto sono illustrati nella Figura 1 insieme alla quantificazione dei risultati per traccia di linea di intensità fluorescente. I risultati qui presentati sono semi-quantitativi e offrono una visione della localizzazione, piuttosto che nel confronto tra intensità di diverse macchie fluorescenti perché gli esperimenti non includevano una perla fluorescente nella preparazione del vetrino. La f...
Discussione
Il protocollo descritto in questo rapporto può essere adattato a diversi tipi di cellule e include passi per raddoppiaRE RNA FISH e RNA FISH con IF utilizzando anticorpi primari monoclonali e policlonali. Anche se i vetrini preparati sono tipicamente immagini con un microscopio confocale, l'imaging può essere eseguito con un microscopio STED (esaurimento delle emissioni stimolato) dopo le modifiche di una maggiore concentrazione di anticorpi e di un diverso mezzo di montaggio. Per un'analisi avanzata delle singole cell...
Divulgazioni
Gli autori non hanno conflitti di interesse da divulgare.
Riconoscimenti
Ringraziamo Jonathan Rodenfels, Kazimierz Tycowski e Johanna B. Withers per consigli sull'analisi dei dati. Ringraziamo anche G. Hayward per l'anticorpo anti-SSB. Questo lavoro è stato sostenuto da sovvenzioni T32GM007223 e T32AI055403 dai National Institutes of Health (a TKV) e niH grant (CA16038) (a JAS). JAS è un investigatore dell'Howard Hughes Medical Institute. Le figure 1-3 e 1 tabella sono state riprodotte con il permesso dell'American Society for Microbiology sotto una licenza di attribuzione Creative Commons della seguente pubblicazione: Vallery, T. K., Withers, J. B., Andoh, J. A., Steitz, Sarcoma-Associated di J. A. Kaposi L'accumulo di mRNA da herpesvirus nei foci nucleari è influenzato dalla replicazione del DNA virale e dall'RNA nucleare policodificato virale non codificante. Giornale di Virologia. 92 (13), doi:10.1128/JVI.00220-18, (2018).
Materiali
| Name | Company | Catalog Number | Comments |
| AlexaFluor594-5-dUTP | Life Technologies | C1100 | |
| anti-DIG FITC | Jackson Lab Immunologicals | 200-092-156 | |
| Anti-Rabbit Secondary AlexaFluor594 Monoclonal Antibody | Invitrogen | A-11037 | Goat |
| Anti-SSB Antibody | N/A | N/A | Ref. Chiou et al. 2002 |
| BLASTn | NIH NCBI | N/A | Free Sequence Alignment Software |
| Dextran Sulfate | Sigma Aldrich | D8906 | Molecular Biology Grade |
| DIG-Oligonucleotide Tailing Kit | Sigma Roche | #03353583910 | 2nd Gen |
| Eight-Chamber Slides | Nunc Lab Tek II | #154453 | Blue seal promotes surface tension but separation by clear gel is also available. |
| Formamide | Sigma Aldrich | F9037 | Molecular Biology Grade |
| GAPDH Probes | Stellaris | SMF-2019-1 | Compatible with protocol, Quasar 670 |
| ImageJ | NIH, Bethesda, MD | N/A | Free Image Analysis Software, [http:rsb.info.nih.gov/ij/] |
| OligoAnalyzer | IDT | N/A | Free Oligonucleotide Analyzer |
| pcDNA3 | Invitrogen | A-150228 | |
| pmaxGFP | Amaxa | VDF-1012 | |
| Poly L-Lysine | Sigma Aldrich | P8920 | |
| Terminal Transferase | Sigma Roche | #003333574001 | |
| Vanadyl Ribonucleoside Complexes | NEB | S1402S | |
| Vectashield | Vector Laboratories, Inc. | H-1000 | DAPI within the mounting media scatters the light and reduces contrast. |
Riferimenti
- Amen, M. A., Griffiths, A. Packaging of Non-Coding RNAs into Herpesvirus Virions: Comparisons to Coding RNAs. Frontiers in Genetics. 2, 81 (2011).
- Schmid, M., Speiseder, T., Dobner, T., Gonzalez, R. A. DNA virus replication compartments. Journal of Virology. 88 (3), 1404-1420 (2014).
- Pawlicki, J. M., Steitz, J. A. Primary microRNA transcript retention at sites of transcription leads to enhanced microRNA production. Journal of Cell Biology. 182 (1), 61-76 (2008).
- Borah, S., Darricarrere, N., Darnell, A., Myoung, J., Steitz, J. A. A viral nuclear noncoding RNA binds re-localized poly(A) binding protein and is required for late KSHV gene expression. Public Library of Science Pathogens. 7 (10), e1002300 (2011).
- Tycowski, K. T., Shu, M. D., Borah, S., Shi, M., Steitz, J. A. Conservation of a triple-helix-forming RNA stability element in noncoding and genomic RNAs of diverse viruses. Cell Reports. 2 (1), 26-32 (2012).
- Weinberg, R. A., Penman, S. Small molecular weight monodisperse nuclear RNA. Journal of Molecular Biology. 38 (3), 289-304 (1968).
- Myoung, J., Ganem, D. Generation of a doxycycline-inducible KSHV producer cell line of endothelial origin: maintenance of tight latency with efficient reactivation upon induction. Journal of Virology Methods. 174 (1-2), 12-21 (2011).
- Brulois, K. F., et al. Construction and manipulation of a new Kaposi's sarcoma-associated herpesvirus bacterial artificial chromosome clone. Journal of Virology. 86 (18), 9708-9720 (2012).
- Sturzl, M., Gaus, D., Dirks, W. G., Ganem, D., Jochmann, R. Kaposi's sarcoma-derived cell line SLK is not of endothelial origin, but is a contaminant from a known renal carcinoma cell line. International Journal of Cancer. 132 (8), 1954-1958 (2013).
- Chiou, C. J., et al. Patterns of gene expression and a transactivation function exhibited by the vGCR (ORF74) chemokine receptor protein of Kaposi's sarcoma-associated herpesvirus. Journal of Virology. 76 (7), 3421-3439 (2002).
- Cole, R. W., Jinadasa, T., Brown, C. M. Measuring and interpreting point spread functions to determine confocal microscope resolution and ensure quality control. Nature Protocols. 6 (12), 1929-1941 (2011).
- Nakamura, H., et al. Global changes in Kaposi's sarcoma-associated virus gene expression patterns following expression of a tetracycline-inducible Rta transactivator. Journal of Virology. 77 (7), 4205-4220 (2003).
- Majerciak, V., Yamanegi, K., Zheng, Z. M. Gene structure and expression of Kaposi's sarcoma-associated herpesvirus ORF56, ORF57, ORF58, and ORF59. Journal of Virology. 80 (24), 11968-11981 (2006).
- Sun, R., Lin, S. F., Gradoville, L., Miller, G. Polyadenylylated nuclear RNA encoded by Kaposi sarcoma-associated herpesvirus. Proceedings of the National Academy Sciences U S A. 93 (21), 11883-11888 (1996).
- Vallery, T. K., Withers, J. B., Andoh, J. A., Steitz, J. A. Kaposi's Sarcoma-Associated Herpesvirus mRNA Accumulation in Nuclear Foci Is Influenced by Viral DNA Replication and Viral Noncoding Polyadenylated Nuclear RNA. Journal of Virology. 92 (13), (2018).
- Borah, S., Nichols, L. A., Hassman, L. M., Kedes, D. H., Steitz, J. A. Tracking expression and subcellular localization of RNA and protein species using high-throughput single cell imaging flow cytometry. RNA. 18 (8), 1573-1579 (2012).
- Bruce, A. G., et al. Quantitative Analysis of the KSHV Transcriptome Following Primary Infection of Blood and Lymphatic Endothelial Cells. Pathogens. 6 (1), (2017).
- Chen, C. P., et al. Kaposi's Sarcoma-Associated Herpesvirus Hijacks RNA Polymerase II To Create a Viral Transcriptional Factory. Journal of Virology. 91 (11), (2017).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon